Method Article
종양 미세환경의 공간 면역 세포 풍경에 대한 다중 면역조직화학적 분석
* 이 저자들은 동등하게 기여했습니다
요약
이 프로토콜은 다중 면역조직화학을 사용하여 종양 미세환경의 면역 세포 특성 분석을 수행하는 방법을 자세히 설명합니다.
초록
종양 미세환경의 면역 세포 환경에는 예후 및 예측 바이오마커의 발견을 위한 정보가 포함될 수 있습니다. Multiplex immunohistochemistry는 공간 정보를 유지하면서 종양 조직에서 다양한 유형의 면역 세포를 시각화하고 식별할 수 있는 유용한 도구입니다. 여기에서는 조직 절편에서 림프구, 골수성 및 수지상 세포 집단을 분석하기 위한 자세한 프로토콜을 제공합니다. 포르말린 고정 파라핀이 포매된 절편 절단, 자동화된 플랫폼에서의 자동 멀티플렉스 염색 절차, 다중 스펙트럼 이미징 현미경으로 슬라이드 스캔, 자체 개발한 머신 러닝 알고리즘 ImmuNet을 사용한 이미지 분석에 이르기까지. 이러한 프로토콜은 단순히 종양 마커를 전환하여 샘플의 다른 구획(종양 대 침습적 가장자리)에 있는 면역 세포를 분석하고 가장 가까운 이웃 분석을 적용함으로써 다양한 종양 검체에 적용할 수 있습니다. 이 분석은 종양 샘플에 국한되지 않고 다른 (비)병원성 조직에도 적용할 수 있습니다. 지난 몇 년 동안 장비 및 작업 흐름이 개선되어 처리 시간이 크게 단축되었으며, 이는 향후 진단 환경에서 이 절차를 쉽게 적용할 수 있도록 합니다.
서문
면역 세포는 바이러스 및 박테리아와 같은 병원체뿐만 아니라 암세포에 대한 보호에도 중요한 역할을 합니다1. 따라서 종양미세환경(tumor microenvironment, TME) 내의 면역체계는 예후 및 예측 바이오마커를 발견하는 데 많은 가능성을 가지고 있다2. 면역 세포 침윤은 다양한 유형의 암에서 예후와 상관 관계가 있지만 아직 임상 치료에서 구현되지 않았습니다 3,4. 대부분의 종양 유형에서 세포독성 T 세포와 T helper 1 세포의 수가 많거나 조절 T 세포의 수가 적으면 예후가 양호합니다. 소위 "면역 점수"를 대장암의 TNM 병기에 통합하여 TNM-I 병기 5,6으로 전환하기 위한 노력이 진행 중입니다. 면역 점수는 서로 다른 두 종양 영역, 즉 종양 코어와 종양의 침습적 가장자리(IM)에 있는 T 세포(CD3로 검출)와 세포독성 T 세포(CD8로 검출)의 총 수에서 파생됩니다. 면역 점수는 또한 흑색종, 폐암 및 유방암과 같은 다른 유형의 암에서 예후 가치가 있는 것으로 제안되었습니다 6,7,8,9. 또한, 면역세포 침투는 관문봉쇄면역요법에 대한 반응과도 상관관계가 있을 수있다 10. 그러나 이러한 예측 바이오마커는 임상 실습에서 일상적으로 구현되기 전에 전향적 연구에서 검증되어야 합니다. 또한, 단일 바이오마커가 의미 있는 예측에 불충분하다는 것도 제안되었다11. 따라서 서로 다른 바이오마커를 결합하여 환자 샘플의 완전한 맵을 생성하는 것이 소위 "암 면역도"12에서 보다 포괄적인 예측 바이오마커로 제안되었습니다.
TME 내에서 면역 세포를 연구하는 방법 중 가장 오래되고 가장 잘 알려진 기술은 면역조직화학(IHC)으로, 여러 질병, 특히 암의 진단 검사에 일상적으로 사용된다13. 이 기법은 오랫동안 하나 또는 소수의 마커(14 )의 사용으로 제한되었기 때문에, 유세포분석 및 유전자 발현 프로파일링(GEP)과 같은 다른 기법에 의해 연구 환경에서 경쟁에서 뒤처졌다. 그러나 일상적인 진단 및 연구에 일반적으로 사용되는 포르말린 고정 및 파라핀 포매(FFPE) 종양 조직은 유세포 분석 및 GEP에 (최적으로) 적합하지 않습니다. 또한 GEP 및 유세포 분석은 세포 표현형 및 기능에 대한 많은 통찰력을 제공하지만 공간 정보가 부족하다는 것이 주요 단점입니다. 따라서 종양의 면역 세포 침투 영역과 면역 세포 배제 영역의 차이와 같은 샘플 내 이질성은 감지되지 않을 수 있습니다15. 조직 절편16 내에서 여러 마커를 동시에 검출하는 데 사용할 수 있는 멀티플렉스 IHC, 이미징 질량 세포 분석 및 CO-Detection by indEXing(CO-Detection by indEXing)과 같은 FFPE 조직의 다중 분석을 위해 새로운 플랫폼이 개발되었습니다. TME의 면역 세포는 면역 요법을 위한 최상의 바이오마커를 찾기 위해 널리 연구되고 있습니다. 그러나 멀티플렉스 기술과 자동화된 이미지 분석은 그 자체로 장애물이 있습니다.
우리 실험실은 Opal/Tyramide 신호 증폭(TSA) 방법을 사용한 멀티플렉스 IHC 염색에 대한 광범위한 경험을 보유하고 있으며 IHC 플랫폼(재료 표 참조)에서 이를 자동화했습니다.17,18,19,20,21,22,23,24,25,26,27,28, 29,30,31. 당사는 림프구, 골수성 세포 및 수지상 세포(DC)의 다양한 하위 집합을 검출하기 위해 면역 세포 패널을 최적화했습니다. 림프구 또는 복잡한 세포 형태(즉, 골수성 세포 및 DC)와 같은 조밀한 면역 세포 영역을 포함하는 조직은 존재하는 면역 세포의 수를 과대평가하거나 과소평가할 위험이 있어 분석이 특히 까다롭습니다. 이 문제를 극복하기 위해 그룹32는 ImmuNet 분석 소프트웨어를 개발했으며, 이 머신 러닝 파이프라인은 이러한 다양한 유형의 면역 세포의 검출 품질을 크게 향상시켰습니다. FFPE 물질을 얻는 것부터 다양한 조직 구획의 면역 세포 밀도 및 면역 세포 유형 간의 거리 분석에 이르는 자세한 프로토콜이 여기에 설명되어 있습니다.
이 프로토콜은 2022년 디지털 병리학 이미저가 구현된 이후 Radboud University Medical Center에서 멀티플렉스 IHC 패널이 어떻게 수행되는지 간략하게 설명합니다. 설명된 멀티플렉스 IHC 패널은 종양 마커로서 팬-사이토케라틴 항체를 사용하는 다양한 암종(예를 들어, 폐, 전립선, 결장직장, 방광, 유방)에 사용할 수 있으며, 멜라닌 세포 관련 항체를 종양 마커로 사용하는 흑색종에 사용할 수 있다. 이러한 멀티플렉스 IHC 프로토콜은 1차 항체 농도, 형광단 조합 및 염색 절차의 순서 측면에서 신중하게 최적화되었습니다. 우리와 다른 사람들은 이전에 17,33,34,35 년에 멀티플렉스 IHC 패널 최적화에 대해 설명했습니다. 멀티플렉스 IHC 패널은 조정할 수 있지만, 설명된 분석 파이프라인을 평가하고 그에 따라 잠재적으로 조정하거나 재교육해야 합니다. 설명된 7색 멀티플렉스 IHC 프로토콜은 Opal 형광단 Opal480, Opal520, Opal570, Opal620, Opal690, Opal780 및 4',6-diamidino-2-phenylindole(DAPI)을 사용하므로 "Multispectral One Touch ImmunoFluorescence"(MOTiF)를 통해 이미저에서 쉬운 혼합 해제 및 빠른 스캔이 가능합니다. 9색 염색 및 스캐닝은 실험 설정을 더욱 미세 조정하고 액정 조정 가능 필터를 사용하는 이미저에서 다른 스캐닝 모드를 더 세밀하게 조정해야 하기 때문에 이 프로토콜에서는 설명되지 않습니다.
프로토콜
이 프로토콜에 대해 표시된 환자 자료는 이전에 수행된 연구의 일부였으며 네덜란드 법률(파일 번호 2017-3164)과 동시에 현지 Radboudumc 의료 윤리 위원회에서 공식적으로 의료 윤리 승인이 면제된 것으로 간주되었습니다(파일 번호 2017-3164)30.
1. FFPE 물질 수집, 블록 선택 및 샘플 준비
- 치료 의사 또는 병리학자를 통해 환자 파일에서 FFPE 블록 식별자를 검색합니다. 윤리적 허가가 필요한지 현지 규정을 확인하십시오.
- 지역 병리학 아카이브 또는 외부 병원에서 FFPE 블록을 요청합니다.
참고: 특정 연구를 위해 종양 물질 또는 생검을 획득할 수도 있습니다. 이는 소규모 임상시험이나 동물 연구의 경우일 수 있습니다. 이러한 경우 조직 샘플의 처리는 연구자의 책임일 수 있습니다. - 여러 FFPE 블록을 사용할 수 있는 경우, 헤마톡실린 및 에오신(HE) 염색 슬라이드를 평가하여 존재하는 주변 기질 조직과 함께 생존 가능한 종양 조직을 포함하는 가장 대표적인 FFPE 블록을 선택합니다(그림 1).
참고: 이 선택에 대한 전문가 의견(예: 병리학자)을 얻는 것이 좋습니다. FFPE 블록의 함량을 평가하기 위해 HE를 사용할 수 없으며 선택을 위해 새로운 HE를 만들어야 할 수 있습니다. 설명을 보려면 섹션 2로 이동합니다. - 마이크로톰에서 4μm 두께의 FFPE 리본을 자릅니다.
참고: 두께는 눈에 띄는 염색 영향 없이 1μm에서 6μm 사이일 수 있습니다. 그러나 4μm가 가장 표준입니다. - 아래 설명된 방법 중 하나를 사용하여 자동 염색기(그림 2A-C)의 유체에 유리한 위치에 샘플을 유리 슬라이드에 장착합니다.
- 증류된 40°C 물의 표면에 단면을 수조에 넣고 펴서 유리 슬라이드로 들어 올립니다.
또는
40°C 가열판에 유리 슬라이드를 놓고 슬라이드에 단면이 장착될 위치를 증류수 한 방울로 덮어야 합니다. 이 방울 위에 집게로 섹션을 놓고 펴십시오. 종이 타월을 사용하여 증류수를 흡수하고 슬라이드를 두드려 여분의 물을 제거합니다.
참고: 조직 절편을 슬라이드 라벨에 너무 가깝게 배치하면 최적이 아닌 염색이 발생합니다(그림 2D,E). 우리는 샘플당 6-10개의 유리 슬라이드를 장착하여 다양한 멀티플렉스 IHC 패널을 수행하고 백업을 유지하는 경향이 있습니다.
- 증류된 40°C 물의 표면에 단면을 수조에 넣고 펴서 유리 슬라이드로 들어 올립니다.
- 장착된 유리 슬라이드를 56°C에서 1시간 동안 건조시키거나 37°C에서 밤새 건조시킵니다.
- 실험을 위해 장착된 유리 슬라이드를 사용하거나 4°C의 상자에 보관하십시오.
참고: 지금까지의 경험에 따르면 이러한 장착된 슬라이드는 멀티플렉스 IHC 염색이 수행되기 전에 수년 동안 보관할 수 있습니다.
2. 헤마톡실린 및 에오신 염색 슬라이드 생성
알림: 섹션 2의 다음 단계는 모두 흄 후드에서 수행해야 합니다.
- 슬라이드를 자일렌으로 탈파라핀화합니다(2 x 5분).
- 에탄올로 재수화합니다(99.6% 1 x 5분, 95% 1 x 5분, 70% 1 x 2분). 또는 슬라이드를 99.6% 에탄올에 3번 담그십시오.
- 슬라이드를 증류수(2분)로 세척합니다.
- 헤마톡실린으로 핵을 염색합니다(10분).
- 증류된 H2O(5분)로 슬라이드를 세척합니다.
- 슬라이드를 에오신으로 염색합니다(5분).
- 3% 에탄올에 99.6배를 담궈 슬라이드를 탈수합니다.
- 슬라이드를 크실렌에 2번 담궈냅니다.
- 장착 매체 몇 방울을 추가하고 커버슬립으로 밀봉합니다.
- 슬라이드를 굳히고 모든 화학 물질이 증발했을 때 흄 후드에서 슬라이드를 꺼냅니다.
3. 자동 염색기에서 모노플렉스 및 멀티플렉스 IHC 수행
- 염색할 샘플의 수에 따라 필요한 시약의 양을 계산합니다.
참고: 실행당 자동 염색기는 30개의 슬라이드 용량을 가지며 6개의 항체로 다중 IHC 프로토콜을 완료하는 데 ~18시간이 걸립니다. 더 많은 슬라이드를 염색해야 하는 경우 (작업) 주의 매일 밤에 여러 배치를 넣을 수 있습니다. 4박 30개 슬라이드 = 주당 120개 슬라이드.- 한 주가 시작될 때 필요한 모든 시약을 준비하십시오. 자동 염색 시스템은 슬라이드당 150 μL의 시약을 분주합니다. 항체 및 Opal 시약에는 6mL 적정 용기를 사용하고 차단 시약 및 보조 항체인 양 고추냉이 과산화효소에는 30mL 용기를 사용하십시오.
참고: 6mL 용기에는 필요할 때 쉽게 꺼내고 교체할 수 있는 편리한 인서트가 있습니다. 시약 계산에서는 30mL 용기 또는 6mL 적정 용기에 대해 각각 1.6mL 또는 300μL의 데드 부피를 고려해야 합니다. - 제공된 희석제에 모든 Opal fluorophores 및 digoxigenin (DIG) 1:100을 희석하십시오. 항체 희석액에 Opal780 1:25를 희석하십시오. 모든 1차 항체를 항체 희석액에 희석하고, 보충 파일 1에 명시된 희석액을 사용합니다.
- 한 주가 시작될 때 필요한 모든 시약을 준비하십시오. 자동 염색 시스템은 슬라이드당 150 μL의 시약을 분주합니다. 항체 및 Opal 시약에는 6mL 적정 용기를 사용하고 차단 시약 및 보조 항체인 양 고추냉이 과산화효소에는 30mL 용기를 사용하십시오.
- 이 프로토콜을 따르려면 실제 멀티플렉스 IHC 실험을 시작하기 전에 편도 대조 조직과 기타 (종양) 관심 조직 유형이 모두 포함된 슬라이드에서 모노플렉스 IHC(보충 파일 2)를 실행하여 모든 시약이 잘 준비되었는지 확인하십시오.
알림: Monoplex IHC는 ~3.5시간이 소요되며 신호 패턴과 강도에 대해 하루가 끝나기 전에 확인할 수 있습니다. 특정 신호가 너무 약하면(그림 3) 시약을 조정할 수 있습니다. - 자가형광 교정을 위해 혈액 및 콜라겐과 같은 자가형광 구조를 포함하는 (종양) 조직이 있는 슬라이드를 준비합니다. 이 슬라이드를 모노플렉스 IHC 슬라이드와 동시에 준비하되, 항체 및 오팔 시약을 대체하는 차단 시약을 사용합니다(보충 파일 3).
참고: 원칙적으로 이러한 슬라이드는 자가형광 보정이 더 이상 최적이 아닐 때까지 다중 스펙트럼 이미징에 재사용할 수 있습니다. 그러나 뇌 및 간과 같은 자가형광성이 높은 조직의 경우 자가형광 보정을 위해 해당 조직을 사용하는 것이 좋습니다. - 각 멀티플렉스 IHC 실행에서 하나의 대조군 슬라이드를 사용하여 29개의 샘플을 자동 염색기 시스템에 로드하여 각 멀티플렉스 IHC 실행의 성능을 확인합니다.
- 다운로드 탭의 자동 염색기 웹사이트에서 멀티플렉스 IHC 프로토콜을 다운로드하고 각각의 맞춤형 멀티플렉스 IHC 패널(36)에 맞게 조정합니다. 멀티플렉스 IHC의 경우 프로토콜에 대한 보충 파일 4를 참조하고 맞춤형 멀티플렉스 IHC 패널에 대해서는 추가 파일 1을 참조하십시오.
- 염색 프로토콜이 완료되면 자동 염색기에서 슬라이드를 꺼내 세척 버퍼가 있는 용기에 넣습니다.
- 샘플이 이미 매우 낮은 농도로 염색되어 있으므로 자동 염색기 시스템이 DIPI로 오염되는 것을 방지하려면 슬라이드를 커버슬립으로 덮기 전에 수동으로 DIPI를 적용하십시오. 세척 완충액 mL당 DAPI 2방울을 넣고 어두운 어두운 실온에서 5분 동안 배양합니다.
NOTE: 스펙트럼 라이브러리를 구축하기 위해서는 샘플에 DAPI가 염색되지 않는 것이 중요합니다. 세척 완충액 mL당 DAPI 1방울과 RT에서 10분 배양도 가능합니다. - 세척 버퍼로 슬라이드를 3번 세척합니다.
- 슬라이드를 종이 타월 위에 놓고 슬라이드에서 여분의 세척 버퍼를 두드립니다.
- 조직에 장착 매체 몇 방울을 피펫으로 떨어뜨립니다.
- 기포를 피하기 위해 슬라이드를 비스듬히 덮기 위해 장착 매체 위에 유리 커버슬립을 부드럽게 놓습니다.
- 집게나 깨끗한 피펫 팁으로 유리 커버슬립을 부드럽게 밀어 과도한 장착 매체와 기포를 제거합니다.
- 장착 매체가 응고되기 전에 현미경 슬라이드 보드에서 수평으로 또는 이미징을 위해 현미경에 직접 로드하여 슬라이드를 ~24시간 동안 방해하지 않도록 두십시오.
- 장착 매체가 응고된 후 또는 슬라이드를 이미징한 후 슬라이드를 4°C의 현미경 상자에 보관합니다.
4. 디지털 병리학 이미저를 사용한 이미징 및 스캔 파일 주석
- 기기 오른쪽에 있는 전원 버튼을 눌러 이미저를 켭니다. 최소 20초 후에 소프트웨어를 시작합니다.
주: 하드웨어가 올바르게 시작될 때까지 20초 동안 기다립니다. - 슬라이드를 4개의 슬라이드당 카세트에 로드합니다.
- 선택 사항: 템플릿을 다운로드할 수 있는 .csv 파일에 슬라이드를 입력합니다(보충 파일 5). .csv 파일을 프로그램에로드하려면 C:\Users\Public\Akoya\VectraPolaris\States에 저장하십시오.
참고: 최대 20개의 카세트 또는 80개의 슬라이드를 동시에 로드할 수 있습니다.
- 선택 사항: 템플릿을 다운로드할 수 있는 .csv 파일에 슬라이드를 입력합니다(보충 파일 5). .csv 파일을 프로그램에로드하려면 C:\Users\Public\Akoya\VectraPolaris\States에 저장하십시오.
- 참조 설정
- 메인 메뉴에서 Check Dashboard 를 엽니다.
알림: 참조 슬라이드가 있는 카세트는 제조업체에서 제공하며 선택적으로 슬롯 20에 영구적으로 보관할 수 있습니다. - 제조업체의 지침에 따라 일주일에 한 번 제공된 슬라이드의 명시야 참조를 설정합니다(몇 분 소요).
- 제조업체의 지침에 따라 한 달에 한 번 제공된 슬라이드에 형광 참조를 설정합니다(1시간 이상 소요).
- 메인 메뉴에서 Check Dashboard 를 엽니다.
- 프로토콜 수립 또는 조정
- 메인 메뉴로 돌아가서 Edit Protocol(프로토콜 편집 )을 클릭하여 프로토콜을 만듭니다.
- New...를 클릭하고 Staining(염색) 옵션에서 Fluorescence(형광)를 Imaging Mode(이미징 모드), Multispectral Slide Scan(다중 스펙트럼 슬라이드 스캔) 및 Opal Polaris 5, 6 및 7 color(오팔 폴라리스 5, 6 및 7 색상)로 선택합니다.
- 프로토콜 이름(Protocol Name)에서 프로토콜 이름을 지정하고 사용 가능한 스터디(Available Studies)에서 스터디를 선택하여 스터디 아래에 저장하거나 새 스터디 생성(Create New Study) | 스터디 이름.
- Create Protocol(프로토콜 생성)을 선택하여 완료합니다.
- 이러한 유형의 스캔에는 왼쪽 창만 사용하십시오. 다중 스펙트럼 슬라이드 스캔 설정; 오른쪽 Multispectral Field Settings 창을 무시합니다.
- 슬라이드를 다양한 배율로 스캔합니다. 이 프로토콜을 따르려면 픽셀 해상도 를 0.50μm(20x)로 유지하여 20배 배율로 스캔합니다.
- 스캔 노출(Scan Exposures)을 선택하여 노출 시간을 설정합니다.
- Load Carrier(캐리어 로드) 옵션에서 올바른 슬롯을 선택하여 슬라이드가 보관된 카세트를 로드합니다.
- 슬라이드를 탐색하는 데 도움이 되도록 Take Overview(개요 수행)를 선택하여 캐리어가 로드된 후 슬라이드가 포함된 캐리어의 개요 이미지를 가져옵니다. 이 기능을 자동으로 켜거나 끄려면 오른쪽 상단의 톱니바퀴 아이콘을 클릭하고 기본 설정...으로 이동한 다음 Navigation Overview Image(탐색 개요 이미지)에서 옵션을 켜거나 끄는 옵션을 선택하여 대화형 작업을 위해 로드할 때 자동으로 이미지 캐리어를 활성화합니다.
- 스캔 노출 설정을 선택하고 포지티브 신호가 있는 다른 지점을 찾아 해당 모노플렉스 IHC 염색 슬라이드의 필터당 노출 시간을 설정합니다. 수동으로 초점을 맞추거나 자동 초점을 사용하고 해당 신호에 대해 호환되는 필터로 전환한 후 자동 노출을 선택합니다. 노출 과다를 방지하기 위해 가장 낮은 노출 시간을 선택하고 모든 노출 시간이 설정된 후 참조용으로 각 슬라이드의 스냅샷을 찍습니다(그림 3).
참고: 이 유형의 스캔에 대해서는 Set Field Exposures(필드 노출 설정 ) 옵션을 무시하십시오. - 포지티브 신호가 있는 몇 개 위치의 모든 필터를 확인하여 멀티플렉스 염색 슬라이드의 노출 시간을 설정합니다. 노출 과다를 방지하기 위해 가장 낮은 자동 노출 시간을 10% 줄이고 모든 노출 시간이 설정된 후 몇 장의 스냅샷을 찍습니다.
- Sample AF 필터를 사용하여 탐색하여 자가형광 보정을 위해 염색되지 않은 슬라이드의 스냅샷을 촬영합니다(그림 3H).
참고: 적혈구와 콜라겐 구조가 있는 위치가 중요합니다. 강한 자가형광 영역의 경우 Opal480 필터의 노출 시간을 줄여야 할 수 있습니다. Opal480 신호가 충분히 강한 경우에도 독점적인 Sample AF 필터의 구현으로 인해 자동 형광 구조에서 잘 분리되어야 합니다(섹션 6 참조). - 소프트웨어를 사용하여 염색 및 이미징의 품질을 평가합니다(섹션 5 및 6 참조; 그림 4, 보충 파일 6: 보충 그림 S1 및 추가 그림 S2).
- 저장... 버튼을 선택하여 프로토콜과 조정된 노출 시간이 프로토콜에 저장되도록 합니다.
알림: 프로토콜이 이미 저장된 경우 지금까지 소프트웨어에서 저장되지 않은 조정에 대한 추가 알림을 제공하지 않습니다.
- 슬라이드 자동 스캔
- 메인 메뉴로 돌아가서 슬라이드 스캔 을 클릭하여 슬라이드를 스캔합니다.
- Configure Tasks에서 슬라이드 이름/ID와 해당 작업 및 프로토콜을 수동으로 입력하거나 Load Setup을 사용하여 이전에 만든 .csv 파일에서 자동으로 입력합니다.
- 스캔을 클릭하여 스캔을 시작합니다.
- 스캔 설정을 저장하기 위해 창이 나타날 때까지 기다립니다. Save(저장 )를 클릭하여 기본 설정을 사용하고 스캔을 시작합니다.
참고: 이 방법을 사용하여 스캔하는 데는 슬라이드당 ~10-20분이 소요됩니다. 슬라이드 수에 따라 스캔하는 데 최대 하루가 걸릴 수 있습니다. - 오류 메시지를 찾아 모든 슬라이드에 대해 슬라이드 스캔이 성공했는지 확인합니다. 스캔이 성공했는지 확인하려면 스캔의 저장된 Akoya 전체 슬라이드 스캔 파일 (.qptiff)과 스캔의 전체 조직을 찾으십시오.
5. 슬라이드 뷰어를 사용하여 데이터 주석 달기
- 메인 메뉴로 돌아가서 Phenochart 실행 을 클릭하여 슬라이드 뷰어를 엽니다.
- 스캔 파일이 직접 표시되지 않는 경우 먼저 오른쪽 상단 모서리에 있는 톱니바퀴 아이콘을 클릭하여 해당 위치를 지정하고 브라우저 위치 변경... 관심 있는 데이터 세트의 .qptiff 파일 중 하나를 임의로 선택합니다.
참고: 데이터는 기본적으로 D:\Data\VectraPolaris에 저장됩니다. - 슬라이드를 선택하고 오른쪽 상단 모서리에 있는 로드 를 클릭하거나 슬라이드를 두 번 클릭하여 로드합니다.
- 오른쪽 상단 모서리에 있는 로그인 버튼을 클릭하여 로그인합니다.
참고: 사용자 이름은 이니셜 또는 이름일 수 있으며 누가 어떤 주석을 만들었는지 추적하는 데 사용됩니다. - 언믹싱을 수행하려면 상단의 언믹싱 버튼을 클릭하고 Opal + AF 옵션을 선택합니다.
참고: 이것은 Opal 480 채널 근처의 자동 형광 신호 중 일부를 제거하는 데 유용하지만 전부는 아닙니다. - 데이터를 일괄 처리하기 위한 알고리즘을 생성하려면 for inForm Projects 1 x 1 images (image size: 928 μm x 696 μm) 옵션을 사용하여 스탬프를 사용하여 대표 이미지를 선택합니다.
참고: 종양, 기질, 배경 및 다양한 유형의 면역 세포를 포함하는 몇 가지 대표적인 스탬프가 데이터 세트 전체에서 선택되어 ~20-30개의 이미지로 끝납니다. - 조직에서 분석해야 하는 항목에 따라 ROI 옵션을 사용하여 관심 영역을 선택하고 inForm Batch를 선택합니다. 분석할 필요가 없는 이미지(예: 종양에서 너무 멀리 떨어져 있거나 배경에 있는 이미지)를 수동으로 삭제합니다.
참고: 우리는 전체 종양 주위에 ROI를 그리고 종양 영역에서 하나 떨어진 추가 이미지를 선택하여 ~0.5mm의 IM을 분석할 수 있도록 하는 경향이 있습니다.
그려진 ROI가 상대적으로 작은 경우 ROI는 2-9개의 병합된 20x 이미지로 구성됩니다. 이것은 우리가 선호하지 않으므로 이를 피하기 위해 관심 조직(inForm Batch에 대해 선택됨)을 수동으로 스탬프하십시오. - 주석 달기가 완료되면 주석이 자동으로 저장되고 다음 슬라이드를 로드합니다.
- 주석 프로세스 중에 슬라이드가 올바르게 스캔되었는지 확인하십시오.
- .qptiff 파일이 없거나 슬라이드가 성공적으로 스캔되지 않은 경우 슬라이드에 조직이 있는지 확인하고 70% 에탄올로 슬라이드를 청소한 다음 다시 스캔합니다.
- 조직을 완전히 스캔하지 않아 잠재적으로 중요한(종양) 부위를 놓치거나 중요한 부위를 스캔할 때 초점이 맞지 않는 경우 슬라이드를 70% 에탄올로 세척하고 다시 스캔합니다.
참고: 두 경우 모두 커버슬립 상단에 마커로 조직을 둘러싸면 시스템이 조직을 찾고 다시 스캔을 시도하는 데 도움이 될 수 있습니다(보충 파일 6: 보충 그림 S3). 우리 손에서는 얇은 빨간색 마커가 두꺼운 검은색 마커보다 더 잘 작동했습니다.
- 모든 샘플의 스캔 및 주석 달기가 완료되면 다른 컴퓨터나 외부 디스크에 저장하여 데이터를 백업합니다.
6. 스펙트럼 불혼합
- inForm 자동 이미지 분석 소프트웨어를 엽니다.
- File(파일) | 이미지 열기; .qptiff 파일을 선택합니다. 5.6단계에서 inForm Projects로 표시된 스탬프를 프로젝트에 로드합니다.
- 자가형광 보정을 위해 이미징된 .qptiff 파일을 불러옵니다.
- 자가형광을 보정하려면 이미지에서 자가형광 선택 도구를 사용하여 적혈구 및 콜라겐과 같은 자가형광성 다양한 유형의 구조를 통해 염색되지 않은 슬라이드의 이미지에 선을 그립니다.
- Edit Markers and Colors...(마커 및 색상 편집) 섹션에서 Opal 형광단에 해당하는 마커 이름을 할당하고 원하는 색상으로 조정합니다.
- 형광단을 혼합 해제하려면 왼쪽 하단 모서리에서 Prepare All 을 선택합니다.
- 이미지를 살펴보고 이미지에서 모든 신호를 볼 수 있는지, 혼합 해제가 잘 이루어졌는지 확인합니다. 눈알 아이콘을 선택하여 모든 마커를 하나씩 끄고 켜서 품질을 확인합니다.
- 선택적으로, 조직 분할, 세포 분할 및 표현형을 위한 알고리즘을 훈련시킵니다.
- Export 탭으로 이동하여 Export Directory 아래의 Browse... 버튼을 클릭하여 비어 있는 새 내보내기 디렉터리를 만듭니다.
- 내보낼 이미지:에서 Composite Image and Component Images (multi-image TIFF)를 선택합니다.
- 파일 | 저장 | 특정 위치에 알고리즘을 저장하는 프로젝트입니다.
- 슬라이드의 일괄 처리를 위해 왼쪽의 Batch Analysis 탭으로 세로로 이동합니다.
- Export Options(내보내기 옵션)에서 Create separate directories for each item(각 항목에 대해 별도의 디렉터리 만들기)을 선택합니다.
- 해석할 슬라이드를 추가하려면 슬라이드 추가... 버튼에서 .qptiff 파일을 선택하고 배치 해석에 로드합니다.
- [실행]을 선택하여 슬라이드의 일괄 처리를 시작합니다.
7. ROI 그림
- 섹션 6의 구성 요소 파일만 있는 폴더를 만들지만 계층적 폴더 구조는 그대로 유지합니다(구성 요소 파일은 sample/slide로 명명된 폴더에 있음).
- QuPath 전체 슬라이드 뷰어 소프트웨어를 엽니다.
- 왼쪽에서 프로젝트 만들기 를 클릭하고 적절한 이름의 빈 폴더를 새로 선택/만듭니다.
- 자동화를 클릭하고 스크립트 편집기 표시를 선택합니다.
- 보충 파일 7에서 사용할 수 있는 스크립트를 복사하여 붙여넣습니다. 34행에서 모든 구성 요소 파일이 포함된 슬라이드 폴더(7.1단계에서 만든 폴더)가 있는 위치로 변경합니다.
- 슬라이드의 일괄 스티칭이 완료되면(다음 날 또는 그 이후) 실행 을 선택하고 돌아와서 계속합니다.
- 생성된 .ome.tif 파일을 QuPath 프로젝트로 끌어다 놓고 프로젝트로 저장합니다.
- 새 창이 자동으로 나타나면 이미지 형식 설정 | Fluorescence(형광) 를 선택하고 Import(가져오기)를 클릭합니다.
- 왼쪽 메뉴에서 샘플 목록을 관찰하십시오. 하나를 두 번 클릭하여 샘플을 엽니다(그림 5A).
- 채널이 더 잘 보이도록 채널의 강도를 조정하려면 대비 아이콘을 클릭합니다.
- 모든 채널을 선택하고 재설정을 클릭합니다.
- 자가형광을 끕니다.
- 종양에 대한 ROI를 그리기 시작하려면 대비 아이콘을 클릭하고 그레이스케일 표시를 선택합니다. 종양 마커 채널을 선택하고 최적으로 보이도록 강도를 조정합니다(그림 5B).
- 브러시 도구를 클릭하여 종양 ROI를 대략적으로 그립니다.
- 지팡이 도구를 선택하는 동안 ALT 키를 누른 상태에서 ROI 외부를 클릭하여 외부에서 ROI를 매끄럽게 합니다(그림 5C).
- 동일한 ROI를 가진 분리된 종양 조각을 병합합니다.
- 왼쪽 목록의 주석을 마우스 오른쪽 버튼으로 클릭하여 ROI에 종양 과 같은 적절한 이름을 지정합니다. 속성 설정을 선택하고 이름을 입력합니다.
- IM에 대한 ROI를 만들려면 다음을 선택하여 종양 영역에서 기존 ROI를 확장합니다. 객체 | 주석... | 주석을 확장합니다.
- 확장 반경의 크기를 선택하고 내부 제거 및 상위로 제한을 선택합니다(그림 5D).
- 대비 아이콘을 클릭하고, 자가형광 채널을 선택하고, 최적으로 보이도록 강도를 조정합니다.
- 지팡이를 클릭하고 alt 키를 누른 상태에서 ROI를 조정하여 외부에서 ROI를 매끄럽게 하고 이 ROI의 일부가 아니어야 하는 배경을 제거합니다.
- 왼쪽 목록에서 주석을 마우스 오른쪽 버튼으로 클릭하여 ROI에 invasive margin 또는 IM 과 같은 적절한 이름을 지정하고, 속성 설정을 선택하고, 이름을 입력하고, 필요에 따라 색상을 녹색으로 변경합니다.
- 주석 저장: 파일 | 개체 내보내기 | 모든 개체를 내보내고 FeatureCollection으로 내보내기에서 기본 선택으로 확인을 클릭하고 원하는 위치에 저장합니다.
8. 면역 세포 검출
- ImmuNet은 훈련 및 추론 모두에 구성 요소 데이터(다중 채널 TIFF 파일)를 사용하므로 주석을 훈련 및 검증 세트로 분할합니다. 모델을 학습시키려면 리포지토리의 Readme 파일에 설명된 단계를 수행하여 예제 데이터 세트와 주석을 원하는 데이터로 대체합니다. 다른 면역 세포 외에도 관심 세포로 인식되어서는 안 되는 부위(종양 세포, 다른 세포 또는 "세포 없음"(관심 세포와 혼동될 수 있는 구조))에 배경 주석을 달아 모델에 부정적인 예를 제공합니다. 자세한 내용은 ImmuNet 간행물을 참조하십시오32.
- 유효성 검사 주석을 사용하여 성능이 만족스러운지 확인합니다. 가장 간단한 평가 지표인 어노테이션 유형별 오류율(모델이 감지하지 못한 검증 어노테이션의 점유율)을 확인합니다. 몇 가지 완전히 주석이 달린 ROI를 만들고 F-점수를 계산하여 거짓 긍정에 대한 성능을 평가합니다.
- 정량적 평가 외에도 예측을 육안으로 검사하여 모델이 범하는 경향이 있는 오류에 대한 정성적 감각을 얻습니다(그림 6, 보충 파일 6: 보충 그림 S4 및 보충 그림 S5). 모델 성능이 충분하지 않다고 판단되는 경우 리포지토리에 설명된 대로 일부 타일에 대한 예측을 시각화하고 오류가 가장 발생하기 쉬운 사이트를 확인합니다. 이러한 사이트에서 더 많은 주석을 만들고 모델 학습 및 평가를 다시 실행합니다.
- 목표 성능이 달성되면 리포지토리 Readme의 전체 데이터 세트에 대한 추론 섹션에 설명된 대로 전체 데이터 세트에 대한 추론을 실행합니다. 모델 예측과 함께 얻은 .csv 파일을 데이터 분석을 위한 입력으로 사용합니다(이를 위해 Python 또는 R 스크립트 작성).
9. 예측 표현형 분석 및 데이터 분석
참고: 이 섹션에서는 림프구 패널로 염색된 단일 흑색종 샘플에 대한 간단한 데이터 분석의 예를 제공하며, 이는 ImmuNet(섹션 8)으로 식별된 면역 세포의 위치와 QuPath(섹션 7)로 설명된 ROI를 결합합니다. 분석은 R 4.1.1에서 수행되었습니다 (스크립트는 보충 파일 8로 제공됨). 스크립트에는 plyr 1.8.8, dplyr 1.0.8, tidyr 1.2.0, sf 1.0-7, ggplot2 3.4.0, RANN 2.6.1 및 RColorBrewer 1.1-2 패키지가 필요하며 install.packages() 명령으로 설치할 수 있습니다. 입력으로는 ImmuNet의 샘플 예측이 포함된 .csv 파일과 QuPath에서 내보낸 ROI가 있는 파일을 사용합니다. 9.1-9.6단계는 제공된 스크립트에서 수행된 단일 샘플의 분석을 설명하고, 9.7-9.9단계는 여러 샘플의 분석 옵션을 설명합니다.
- ImmuNet의 예측을 R에 로드한 후, 서로에 대해 표현형을 정의하는 마커를 플로팅하고 모집단을 가장 잘 구분하는 임계값을 선택하여 예측된 마커 발현에 대한 임계값을 결정합니다.
참고: 주어진 샘플에 사용된 게이팅 전략은 그림 7B에 나와 있습니다. 골수성 및 수지상 세포 패널에 대한 게이팅 전략은 보충 파일 6: 보충 그림 S6 및 추가 그림 S7에 나와 있습니다. - 임계값을 결정한 후 이를 사용하여 각 ImmuNet 예측에 패널에 정의된 표현형을 할당합니다. 일부 예측에서는 예측된 마커 중 어느 것도 임계값을 초과하지 않거나 임계값 설정 후에 발현된 것으로 간주되는 마커의 조합이 일관되지 않을 수 있음을 관찰합니다(예: 림프구 패널의 CD3+ CD20+ 예측). 8.3단계에서 양호한 모델 성능이 달성되면 이러한 예측의 비율이 작아집니다. 분석하기 전에 필터링하십시오.
- QuPath에서 추출한 종양 및 최대 100μm의 침습성 마진에 대한 ROI를 별도로 분석하려면 R에서 해당 GeoJSON 파일을 로드하고 각 예측에 대해 예측이 적용되는 ROI를 결정합니다.
- 온전성 검사를 위해 탐색적 데이터 분석의 일환으로 샘플에서 발견되는 면역 세포를 ROI의 경계와 함께 해당 ROI에서 별도로 시각화합니다(그림 7A).
- 이제 각 ROI에 대해 서로 다른 면역 세포의 밀도를 개별적으로 계산합니다. 주어진 샘플에서 발견되는 밀도는 표 1에 나와 있습니다.
- 여러 샘플을 사용할 수 있는 경우 세포 밀도의 분포를 시각화합니다. 밀도 값을 로그 변환하여 정규 분포 값을 얻습니다.
참고: 특정 표현형의 개수가 0이면 Log-transformed할 수 없으므로 값이 누락됩니다. 이 문제를 극복하기 위해 표면적으로 나누기 전에 먼저 모든 셀 수에 0.5를 추가하여 LaPlacian 평활화를 적용할 수 있습니다. - 밀도 값을 분석하고 선택한 소프트웨어를 사용하여 플롯합니다(그림 8).
- 셀의 보존된 위치를 통해 공간 분석을 수행할 수 있습니다. 예를 들어, 검출된 각 면역 세포에 대해 가장 가까운 이웃을 찾은 다음 각 표현형에 대해 서로 다른 표현형이 가장 가까운 이웃으로 발생하는 사례의 백분율을 계산합니다.
참고: 이 샘플에서 발견된 자연살해(NK) 세포의 수가 매우 적었기 때문에 이 분석에서 제외했습니다. 종양 및 IM ROI에 대해 얻은 결과는 그림 9에 나와 있습니다.
결과
종양 조직을 포함하는 FFPE 블록은 병리학 보고서 및 HE 염색 슬라이드를 기반으로 선택되었습니다. 환자로부터 여러 종양 병변을 절제하거나 종양 샘플이 큰 경우, 이들은 여러 FFPE 블록으로 나뉩니다. 우리는 종양 구획과 종양의 침습적 가장자리(IM)로 알려진 것 모두에서 면역 세포를 분석하는 것을 선호합니다. IM은 종양에 인접한 비암성 기질 조직입니다. 따라서 하나의 종양 샘플에 사용할 수 있는 여러 FFPE 블록이 있는 경우 두 조직 유형을 모두 포함하는 FFPE 블록이 선택됩니다. HE 염색 슬라이드에서 볼 수 있듯이 하나의 FFPE 블록에는 종양에 인접한 종양 조직과 기질 조직이 포함되어 있습니다(그림 1A). 동일한 종양의 또 다른 FFPE 블록은 주변 기질 조직을 훨씬 적게 함유하고 있었습니다(그림 1B). 그러나, 일부 조직 샘플의 경우, FFPE 블록에 선택의 여지가 없거나 IM이 FFPE 블록에 존재하지 않습니다. 이것은 종종 (바늘) 생검의 경우이며, 데이터 해석 중에 염두에 두어야 합니다.
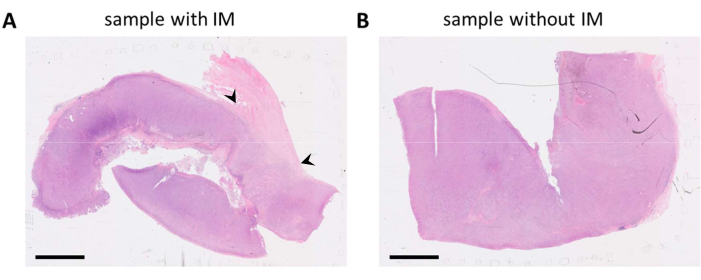
그림 1: 흑색종 종양 표본의 HE 염색 슬라이드. (A) 샘플의 오른쪽 상단 모서리에 종양(IM)에 인접한 기질 조직이 있는 종양 샘플의 예(검은색 화살촉으로 표시). (B) 샘플에 기질 조직이 거의 또는 전혀 존재하지 않는 동일한 종양 병변의 다른 샘플. 축척 막대 = 5mm. 약어: HE = 헤마톡실린 및 에오신; IM = 침습적 마진. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
제안된 7색 패널(보충 파일 4)을 사용한 멀티플렉스 IHC 염색은 3일 염색 과정(정상 근무 시간 고려)에서 수동으로 수행하거나 자동 염색기에서 야간에 수행할 수 있습니다. 자동 염색기를 사용할 때는 시스템의 최적 유체 역학을 가능하게 하는 유리 슬라이드의 특정 위치에 섹션을 장착해야 합니다(그림 2A). 단면이 슬라이드에 올바르게 장착되면(그림 2B) 균일하게 염색됩니다(그림 2C). 단면이 유리 슬라이드에 최적으로 장착되지 않으면(그림 2D), 자동 염색기의 유체가 (완전한) 조직에 도달하지 못하기 때문에 종종 차선의 염색 패턴(그림 2E)이 발생합니다. 이것은 샘플이 매우 크거나 이 문제를 인식하지 못하는 사람이 장착된 슬라이드를 제공할 때 발생할 수 있습니다. 이러한 경우 슬라이드에서 잘 얼룩진 부분만 분석을 위해 선택해야 합니다. 이러한 유형의 샘플에 대한 또 다른 선택은 액체를 최적으로 퍼뜨리기 위해 수동으로 염색하는 것입니다.
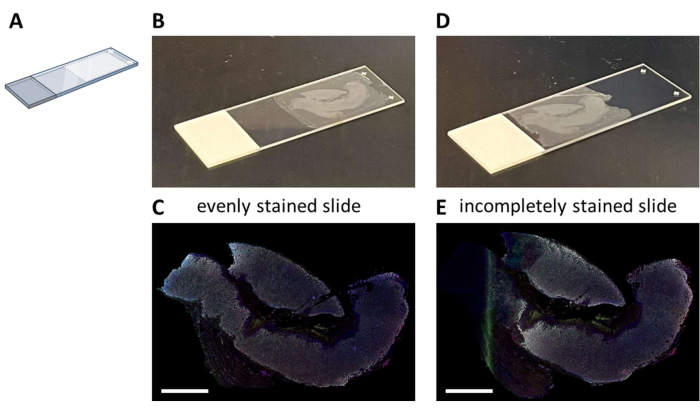
그림 2: 유리 슬라이드에 FFPE 섹션을 장착하고 충격을 가하는 모습. (A) 자동 염색기에서 최적의 염색을 위해 유리 슬라이드에 장착할 위치에 대한 개략도. (B) 예amp올바르게 장착된 슬라이드의 파일. (C) 슬라이드를 올바르게 장착하면 조직 부분이 고르게 염색됩니다. (D) 전amp최적이 아닌 장착 슬라이드의 예. (E) 최적화되지 않은 장착 슬라이드는 이 그림의 왼쪽에서 볼 수 있듯이 불완전한 염색 조직 절편을 초래할 수 있습니다. 축척 막대 = 5mm. 약어: FFPE = 포르말린 고정 및 파라핀 내장. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
대규모 멀티플렉스 IHC 실험이 여러 염색 라운드로 수행되고 많은 양의 용액을 준비해야 하는 경우, 멀티플렉스 IHC로 진행하기 전에 먼저 이러한 시약을 모노플렉스 IHC 실행에서 테스트하는 것이 가장 좋습니다. Monoplex IHC는 디지털 병리학 이미저로 예상 염색 패턴을 확인하고 노출 시간은 대조군 슬라이드의 해당 필터로 설정합니다(그림 3A-H). 편도선 조직은 대부분의 면역 세포 마커에 대한 양성 대조군으로 사용됩니다. 편도선 조절 조직에서의 DAPI 노출 시간은 항상 다른 조직보다 높기 때문에(그림 3G), 연구할 조직 유형에 대해 DAPI 노출 시간을 설정해야 합니다. 이러한 유형의 스캐닝에서 정기적인 노출 시간은 형광단과 필터에 따라 1ms에서 30ms 사이입니다(그림 3I). 모노플렉스 IHC가 이 수치를 초과하거나 염색 패턴이 예상만큼 명확하지 않은 경우 항체 용액을 조정하거나 교체해야 합니다. 여기에 표시된 예에서는 FOXP3(그림 3C 및 그림 3I)의 농도를 증가시켜 다른 마커와 더 많은 범위를 갖도록 했습니다. 자가형광은 편도선 대조 조직보다 다른 조직에서도 더 강할 수 있습니다. 이 설정에서 샘플 AF 필터의 노출 시간은 25ms에서 50ms 사이입니다(그림 3H,I).

그림 3: monoplex IHC 및 염색되지 않은 대조군 샘플에 대한 노출 시간 설정. (A) CD20 - 편도선 조절 조직의 오팔 480 신호. (B) CD3 - 편도선 조절 조직의 오팔 520 신호. (C) FOXP3 - 편도선 조절 조직의 오팔 570 신호. (D) CD56 - 편도선 조절 조직의 오팔 620 신호 (E) CD8 - 편도 조절 조직의 오팔 690 신호. (F) 종양 마커 - 편도선 조절 조직의 Opal 780 신호. (G) 편도 조절 조직의 DAPI 신호는 종종 관심 조직 유형보다 약합니다. (H) 자가형광 - 종양 조절 조직의 샘플 AF 신호. (I) 10%로 조정하고 멀티플렉스 IHC 염색 슬라이드를 확인하기 전의 노출 시간 스크린샷. 스케일 바 = 100 μm. 약어: AF = autofluorescence; IHC = 면역조직화학; DAPI = 4'6-디아미디노-2-페닐린돌. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
멀티플렉스 IHC를 수행한 후 몇 개의 멀티플렉스 IHC 슬라이드를 확인하고 자동 노출을 선택하여 모노플렉스 IHC 설정에서 노출 시간을 조정합니다. 이러한 유형의 스캔에는 채도 보호 옵션이 없으므로 노출을 너무 높게 설정하지 않도록 하여 과다 노출을 피하는 것이 매우 중요합니다. 과다 노출은 fluorophores의 스펙트럼 불혼합을 방해합니다. 우리는 종종 모노플렉스 IHC를 기반으로 한 노출 시간을 초과하는 노출 시간을 설정하지 않으며 다중 IHC에서 더 강한 마커에 대해서만 노출 시간을 줄입니다(그림 3G 및 그림 4A). 몇 개의 슬라이드에서 다른 위치에 자동 노출함으로써 일부 필터의 노출 시간이 여전히 너무 높다는 것을 관찰할 수 있습니다. 이 값은 자동 노출 설정을 사용할 때 관찰되는 가장 낮은 숫자로 조정해야 하며 보이지 않는 다른 위치에서 과다 노출을 방지하기 위해 값의 10%를 더 빼야 합니다(그림 4A). 이 방법을 사용하면 모노플렉스 IHC에 설정된 것보다 특정 필터에 대한 노출 시간이 더 낮을 수 있습니다. 그러나 성공적인 멀티플렉스 IHC 실험을 통해 모든 마커는 적어도 대조군 슬라이드에서 관찰할 수 있어야 합니다(그림 4B-H, 보충 파일 6: 추가 그림 S1 및 추가 그림 S2). 특정 마커가 모든 샘플에 존재하지 않을 수 있다는 점을 고려하십시오. 최소한 편도 단면을 포함하는 대조군 슬라이드를 포함함으로써 표준 패널의 모든 마커와 신호 강도의 성공적인 염색을 확인할 수 있습니다.
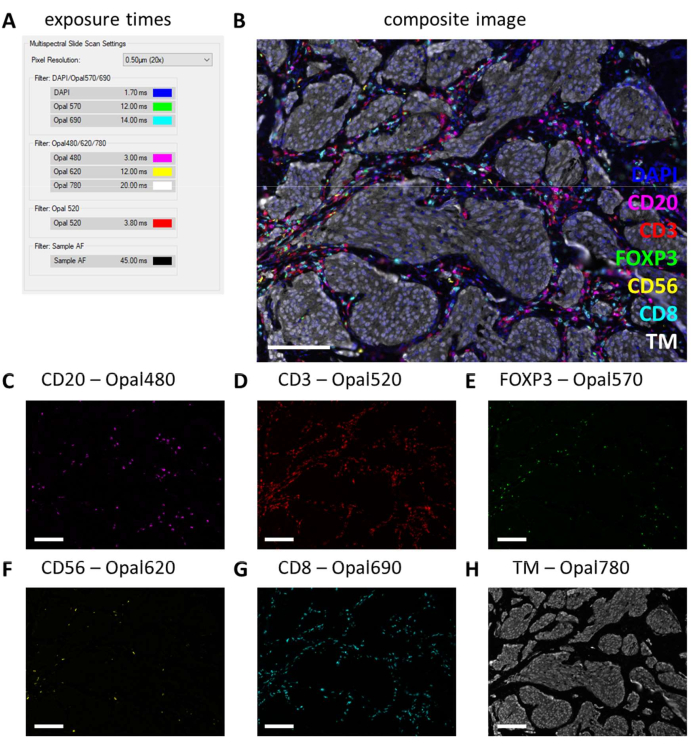
그림 4: 흑색종 종양 표본에서 림프구 패널로 성공적으로 염색된 절편의 예. (A) 이 멀티플렉스 IHC 샘플을 기록하는 데 사용된 노출 시간. (B) 종양 조직 내 다중성 IHC 림프구 패널의 합성 이미지. (C) CD20 - 마젠타색의 오팔 480 신호. (D) CD3 - 빨간색의 오팔 520 신호. (E) FOXP3 - 녹색의 오팔 570 신호. (F) CD56 - 노란색의 오팔 620 신호. (G) CD8 - 청록색의 오팔 690 신호. (H) TM - 화이트 오팔 780. 스케일 바 = 100 μm. 약어: TM = 종양 표지자; IHC = 면역조직화학. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
멀티플렉스 IHC 슬라이드는 디지털 이미저에 의해 완전히 스캔됩니다. 후속 분석을 위한 타일이 슬라이드 뷰어에서 선택됩니다. 그러나 종양 대 IM과 같이 보다 구체적인 영역을 분석해야 하는 경우 QuPath를 사용하여 이러한 관심 영역(ROI)을 그릴 수 있습니다. 슬라이드 뷰어에서 선택한 타일의 일괄 처리가 완료되면 구성 요소 파일이 다시 병합됩니다(그림 5A 및 추가 파일 7). 종양 마커 채널(그림 5B)과 QuPath의 마술 지팡이 도구를 사용하여 종양 윤곽을 추적하여 "종양 ROI"(그림 5C)를 형성할 수 있습니다. 다음으로, 종양 ROI를 특정 거리(이 경우 500μm)로 확장하여 "침습적 마진 ROI"를 생성할 수 있습니다(그림 5D). 원치 않는 배경(비조직)은 자가형광 신호를 확인하여 마술 지팡이 도구를 사용하여 이 ROI에서 제거합니다(그림 5E). Tumor ROI와 IM ROI는 모두 추가 처리를 위해 GeoJSON 파일로 저장됩니다(그림 5F).
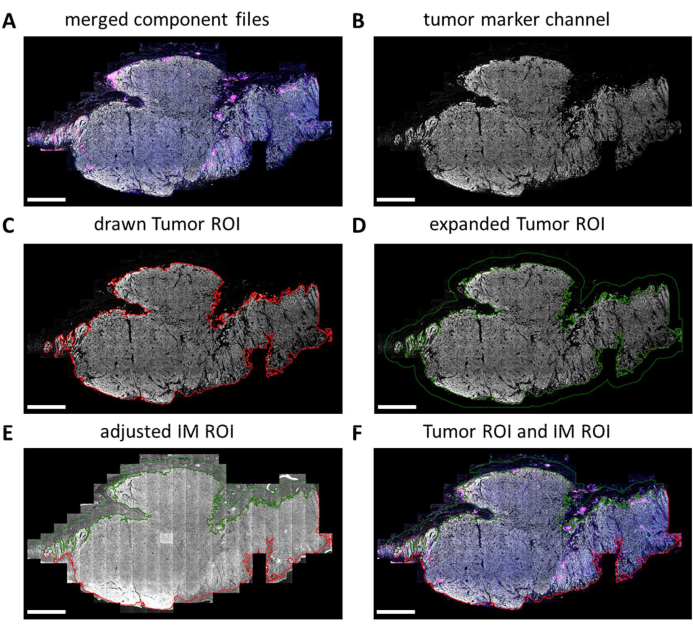
그림 5: QuPath의 종양 ROI 및 침습적 마진 ROI 드로잉 프로세스. (A) 병합된 구성 요소 파일. (B) 종양 마커 채널만 보여주는 회색조 이미지. (C) 종양 ROI는 종양 마커 신호 주위에 그려집니다. (D) 종양 ROI를 100-500 μm 확장하여 IM ROI를 형성함으로써 새로운 ROI를 만듭니다. (E) IM ROI는 배경(음성 신호) 및 지방, 혈관 및 모낭과 같은 기타 큰 조직 구조를 제외하고 기질 조직만 포함하도록 조정됩니다. (F) 결과 종양 ROI 및 IM ROI는 영역의 추가 처리를 위해 저장되고 GeoJSON 파일로 내보내집니다. 종양 ROI는 빨간색 윤곽선으로 표시되고 IM ROI는 녹색 윤곽선으로 표시됩니다. 축척 막대 = 2mm. 약어: ROI = 관심 영역; IM = 침습적 마진; GeoJSON = 지리적 JavaScript 객체 표기법. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
ImmuNet 네트워크는 면역 세포를 검출하는 데 사용할 수 있습니다. 림프구 패널의 경우 실험용 복합 이미지(그림 6A)를 소프트웨어에서 감지한 면역 세포와 육안으로 비교할 수 있습니다(그림 6B). 골수성 패널(보충 파일 6: 보충 그림 S4)과 수지상 세포 패널(보충 파일 6: 보충 그림 S5)에 대해서도 유사한 시각적 비교가 이루어질 수 있습니다.
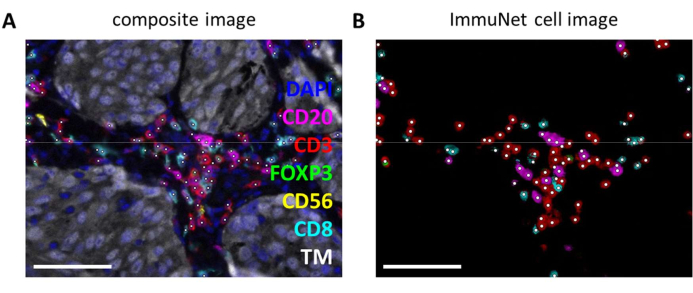
그림 6: ImmuNet에 의해 인식된 림프구 . (A) ImmuNet에 의해 인식된 세포를 흰색 점으로 보여주는 그림 4B 의 합성 이미지. (B) ImmuNet에 의해 인식된 세포 및 이후에 검출된 마커 발현. 스케일 바 = 50 μm. 약어: TM = 종양 마커. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
ImmuNet에 의해 검출되고 .csv 형식으로 저장된 면역 세포는 추가 분석을 위해 모든 프로그래밍 언어로 가져올 수 있습니다. 공간 시각화와 게이팅을 R에서 수행했습니다(보충 파일 8). 그런 다음 검출된 세포를 공간적으로 시각화할 수 있습니다(그림 7A, 추가 파일 6: 추가 그림 S6 및 추가 그림 S7). pseudomarker 발현에 대한 게이팅을 수행하여 개별 면역 세포를 표현형화할 수 있습니다(그림 7B).
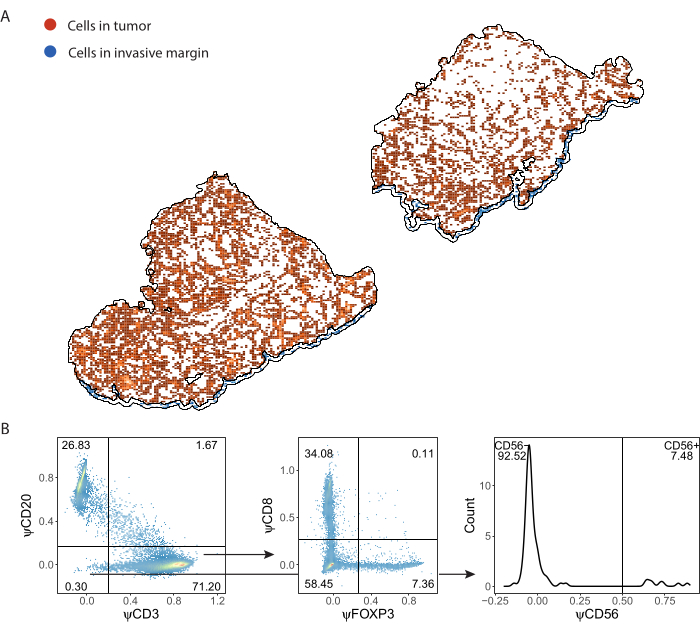
그림 7: 림프구 패널의 게이팅 전략. (A) QuPath로 묘사된 종양 및 침습적 마진 관심 영역에서 검출된 면역 세포. (B) 파트 A에서 ImmuNet에 의해 검출된 모든 세포의 게이팅. 림프구는 먼저 CD20+ B 세포와 CD3+ T 세포에 의해 조절됩니다. CD3+ T 세포는 CD8 및 FOXP3 발현을 위해 추가로 gated됩니다. CD20-CD3- 집단은 CD56+ 자연살해세포(natural killer cell)에 대해 게이트(gateated)되어 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
예측된 세포의 표현형이 게이팅(gating)으로 결정될 때, 서로 다른 표현형의 세포 밀도는 서로 다른 ROI 내에서 계산될 수 있습니다. 이는 표현형당 총 세포 수를 ROI의 표면적으로 나누어 계산합니다(표 1, 그림 8 및 추가 파일 8). 여기서 B세포는 CD3-CD20+, 도우미T세포는 CD3+CD20-CD8-FoxP3-, 조절T세포는 CD3+CD20-CD8-FoxP3+, 세포독성T세포는 CD3+CD20-CD8+FoxP3-, NK세포는 CD3-CD20-CD56+로 정의된다.
| 표현형 | 종양의 밀도(cells/mm2) | IM의 밀도(cells/mm2) |
| B 세포 | 185.74 | 145.62 |
| 도우미 T 세포 | 301.46 | 157.51 |
| 조절 T 세포 | 38.53 | 19.53 |
| 세포독성 T 세포 | 185.35 | 83.21 |
| NK 세포 | 0.18 | 0 |
표 1: ROI의 표현형 밀도. 림프구 패널로 염색된 단일 흑색종 샘플에서 발견되는 다양한 표현형의 세포 밀도. 밀도는 종양 및 IM ROI에서 별도로 계산됩니다. 약어: IM = invasive margin; ROI = 관심 영역.
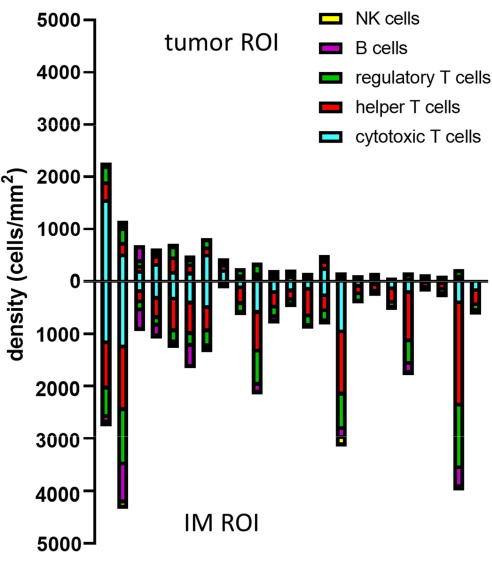
그림 8: 여러 샘플에 대한 데이터 분석의 예. 23개의 원발성 흑색종 종양의 종양 및 IM에서 다양한 림프구 표현형의 밀도 분석. 약어: IM = invasive margin; ROI = 관심 영역. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이러한 면역 세포의 공간 정보를 더 자세히 알아보기 위해 샘플에서 식별된 표현형 사이의 거리 또는 가장 가까운 이웃의 표현형 비율을 결정할 수도 있습니다(그림 9).
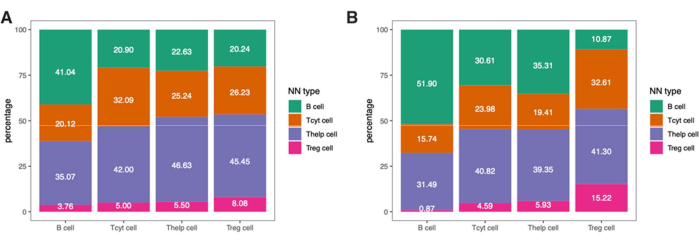
그림 9: 단일 샘플에 대한 최근접 이웃 분석의 예. 림프구 패널로 염색된 단일 흑색종 샘플에서 발견된 (A) 종양 및 (B) IM ROI의 다양한 세포 유형에 대한 가장 가까운 이웃 표현형의 백분율. 약어: IM = invasive margin; ROI = 관심 영역. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 파일 1: 프로토콜 사양을 요약한 멀티플렉스 IHC. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 2: 모노플렉스에 대한 자동 염색 프로토콜. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 3: 자가형광 보정을 위한 자동염색기 프로토콜. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 4: 다중 면역조직화학을 위한 자동염색기 프로토콜. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 5: 템플릿 .csv 파일. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 6: 흑색종 조직 샘플의 골수성 및 수지상 세포 패널; 스캐닝 실패 시 슬라이드 표시; ImmuNet에 의해 인식된 골수성 및 수지상 세포; 골수성 및 수지상 세포 패널의 게이팅 전략. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 7: QuPath 스티치 스크립트. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 8: 데이터 분석 스크립트. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
TME의 공간 분석은 특히 면역 종양학 분야에서 면역 세포 구획에 대해 자세히 알아보고 새로운 예후 및 예측 바이오마커를 발견하기 위해 인기 있는 기법입니다16. 이러한 목적을 위해 단백질, mRNA 전사체 또는 이 둘의 조합을 검출하는 등 최대 100-1,000개의 표적을 추정하는 다양한 기술이 개발되고 있습니다. 그러나 멀티플렉싱이 높을수록 처리량이 적은 실험, 더 높은 실험 비용 및 기술적 문제가 발생하며 TME의 작은 부분만 분석할 수 있는 경우가 많습니다. 여기에서 설명하는 TSA 기반 방법을 사용하는 멀티플렉스 IHC는 6개의 서로 다른 마커 + DAPI를 동시에 검출하고, 수행 비용이 상대적으로 저렴하며, 전체 조직 절편이 20분 이내에 이미징되어 완전히 분석할 준비가 됩니다. 이 기술은 염색 절차의 자동화로 덜 복잡해졌습니다. 두 개의 추가 필터 추가를 포함한 다중 스펙트럼 현미경의 개선으로 스펙트럼 불혼합 및 스캔 시간이 크게 개선되었습니다. 최대 8개의 서로 다른 마커 + DAPI를 동시에 감지할 수 있습니다. 그러나 더 많은 마커로 멀티플렉싱을 확장하면 스펙트럼 불혼합이 더 어려워지고 전체 슬라이드의 스캔 시간이 크게 증가함에 따라 앞서 언급한 이점이 사라집니다. 진단 환경에서 보다 쉽게 구현할 수 있도록 서로 다른 기관 간의 멀티플렉스 IHC를 표준화하기 위한 노력이 진행되고 있습니다. 이러한 멀티플렉스 IHC의 표준화를 위해 사용자는 6개의 서로 다른 마커 + DAPI를 사용하여 보다 접근하기 쉬운 프로토콜을 준수하는 것이 좋습니다. 그럼에도 불구하고 여전히 상당한 기술적 노하우가 필요하고 다운스트림 분석이 어려울 수 있으므로 이 프로토콜에 설명된 방법론을 개발했습니다.
표준화는 멀티플렉스 IHC 패널 개발에서 시작됩니다. 특정 단백질 표적을 검출하는 1차 항체 선택의 중요성은17년 이전부터 강조되어 왔습니다. 당사의 멀티플렉스 IHC 패널은 대부분 당사의 진단 부서에서 IHC에 사용되고 검증된 1차 항체 클론으로 개발됩니다. 그러나, 수지상세포 다중화 IHC 패널의 경우, 대부분의 항체는 진단 환경에서 사용되지 않았다(van der Hoorn et al., manuscript in submission). 특이성을 보장하고 배치 차이를 최소화하기 위해 다클론 항체보다 단클론 항체를 사용하기로 결정했으며 transfection된 세포주와 일차 세포를 사용하여 대부분의 항체를 검증했습니다. 수년에 걸쳐 Vectra 3 시스템 18,21,23,24,25,26,27,28,29,30,31,32를 사용하는 수많은 연구에서 다양한 버전의 멀티플렉스 IHC 패널이 사용되었습니다.. PhenoImager HT 시스템에서 이러한 멀티플렉스 IHC 패널을 최적으로 구현하기 위해 1차 항체 및 형광단 조합에서 일부 조정이 이루어져야 했습니다. 더 나은 스펙트럼 불혼합과 전체 조직 절편의 더 빠른 스캐닝 시간을 활용하려면 최신 Opal480 및 Opal780 형광단을 구현하고 7색 다중 IHC 패널에서 Opal540 및 Opal650 형광단을 사용하지 않아야 합니다. 스캐닝 시간은 조직 절편의 크기에 따라 3~10배 더 빠릅니다. 멀티플렉스 IHC 패널 조정은 매우 쉽게 달성할 수 있었지만 몇 가지 고려 사항을 염두에 두어야 합니다. Opal480의 형광 스펙트럼은 자가형광 스펙트럼과 많이 겹치므로 적혈구 및 기타 자가형광 구조의 스펙트럼 불혼합을 방해합니다. Opal480과 함께 증가된 농도의 1차 항체를 사용하면 대부분의 경우 이 문제가 해결되었습니다. PhenoImager HT에 독점적인 샘플 AF 필터를 구현하면 Opal480과 자가형광의 언믹스가 용이해집니다. 그러나 Opal480과 함께 사용할 때 명확한 신호를 생성하는 1차 항체를 사용하여 신호가 자가형광보다 높도록 하는 것이 가장 좋습니다.
이러한 멀티플렉스 IHC 패널이 구축되더라도 배치 간 변동을 고려해야 합니다. 전체 멀티플렉스 IHC 실험을 시작하기 전에 모노플렉스 IHC 대조군을 수행함으로써, 우리는 때때로 1차 항체가 실험마다 더 강하거나 더 약하게 수행되는 것을 관찰했습니다. 그 이유는 피펫팅 오류, 최적화되지 않은 시약 보관 조건 및 보관 수명일 수 있습니다. 우리는 우리의 경험을 바탕으로 1차 항체 용액을 조정하여 이 문제를 해결했습니다. 앞서 언급한 조정 중 어느 것도 할 필요가 없는 경우에도 모든 멀티플렉스 IHC 배치 실험에서 모노플렉스 IHC 염색 대조군 슬라이드를 기반으로 노출 시간을 설정하는 것이 중요합니다.
우리의 연구는 초기에 다양한 유형의 암종과 흑색종에 초점을 맞추었기 때문에 최소한의 조정으로 종양 유형 간에 상호 교환할 수 있는 멀티플렉스 IHC 패널이 필요했습니다. 따라서 우리는 항상 최적화 과정에 여러 (종양) 조직 유형을 포함시켰고 면역 세포 마커에 대한 1차 항체에 대한 희석이 서로 다른 종양 유형 간에 유사하게 유지될 수 있음을 관찰했습니다. 그러나 암종과 흑색종 사이의 종양 조직 검출에는 다른 종양 마커가 필요합니다. 따라서, 종양 마커는 항상 각 멀티플렉스 IHC 패널의 끝에서 작동하도록 최적화되었으며, 현재 항상 Opal780과 함께 사용되며, 이는 공교롭게도 멀티플렉스 IHC 염색 절차에서 마지막 형광단에 있어야 합니다. 결과적으로 멀티플렉스 IHC의 말단에 종양 마커를 사용함으로써, 이러한 멀티플렉스 IHC 패널은 교모세포종(즉, GFAP) 및 호지킨 림프종(즉, CD30)과 같은 다른 종양 유형으로 쉽게 교환될 수 있습니다. 혈관육종의 경우, 적혈구 형질전환 특이적 유전자(ERG)가 있는 이 림프구 다중성 IHC 패널을 종양 마커로 사용했으며, 단 두 번의 최적화 실험으로25. 최적화에는 ERG 1차 항체의 적정과 끝에 ERG가 있는 멀티플렉스 IHC 패널 테스트가 포함되었습니다.
이러한 멀티플렉스 IHC 패널에 대한 다른 조정은 특정 면역 세포 마커를 다른 면역 또는 기능 마커로 교환함으로써 이루어질 수도 있습니다. 모든 변화에는 최적화가 필요합니다. 최적화를 위한 프로토콜은 앞서 설명한 바와 같이 따를 수 있다17. 제안된 멀티플렉스 IHC 패널에 대한 특정 변경 사항은 우리가 만든 ImmuNet 알고리즘을 방해할 것입니다. 이러한 변경 사항을 알고리즘에 구현하기 위해 충분한 데이터가 생성되어야 하며 시간이 소요되어야 합니다(모든 새로운 마커 및/또는 세포 표현형에 대해 최소 750개의 주석, 이전에 훈련된 마커의 검증을 위한 150개의 주석). 여기에 제시된 패널에는 기능성 마커가 포함되어 있지 않지만, PD-1 및 PD-L1과 같은 면역 관문 마커를 다중 IHC 패널에 구현하는 작업이 우리 실험실에서 수행됩니다. 그러나 음수 신호와 양수 신호에서 덜 이분법적인 마커를 분석하는 것은 더 어려운 것으로 입증되었으며 우리 그룹에서 활발한 연구 영역입니다.
멀티플렉스 IHC로 동시에 평가할 수 있는 마커의 수는 다른 새로운 기술에 비해 제한적입니다. FFPE 블록의 연속된 슬라이스에서 서로 다른 패널을 분석하여 이를 피할 수 있지만 이러한 슬라이스를 공간적으로 비교하는 것은 어렵습니다. 방향과 접힌 아티팩트는 슬라이드 준비 후에 동일하지 않을 수 있습니다. 그럼에도 불구하고 멀티플렉스 IHC는 접근성이 뛰어나 더 많은 기관과 연구자에게 매력적인 도구가 될 수 있으므로 향후 진단 환경에서 구현하기에 더 적합합니다. 여러 종양 유형 및 다운스트림 분석 파이프라인에 대한 멀티플렉스 IHC 면역 세포 패널의 표준화를 통해 환자와 종양 유형 간의 TME의 차이에 대해 더 많은 지식을 얻을 수 있습니다. 예를 들어, 이는 특정 치료에 대한 항종양 반응에서 TME의 역할에 대한 더 많은 통찰력으로 이어질 수 있습니다. 이는 치료에 대한 반응 및 예상 생존과 같은 요인을 예측하기 위한 새로운 바이오마커를 발생시킬 수도 있습니다. 전반적으로, 이를 통해 멀티플렉스 IHC는 맞춤형 의료 접근 방식에서 임상 의사 결정을 지원하는 임상 도구가 될 수 있습니다. 물론, 분석 절차의 더 많은 단계를 자동화하고 표준화하여 일상적인 진단 환경에서 사용할 수 있도록 해야 하므로 아직까지는 대부분 미래 지향적인 관점입니다.
단일 샘플 슬라이드에서 여러 마커를 분석하는 것은 기술적 어려움에도 불구하고 매우 강력한 도구가 될 수 있습니다. 여기에서 ImmuNet을 사용하여 설명한 바와 같이 표준화된 실험 프로토콜과 강력한 분석 방법을 사용하면 여러 마커의 정량화가 기존 IHC보다 더 많은 정보를 제공하는 반면, 멀티플렉스 IHC는 새로운 고플렉스 실험 방법에 비해 상대적으로 높은 처리량을 유지합니다.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
PhenoImager HT는 Radboud University Medical Center와 Radboud Technology Center for Microscopy에서 제공하는 자금을 통해 구입했습니다. CF는 네덜란드 암 협회 보조금(10673)과 ERC Adv 보조금 ARTimmune (834618)의 재정 지원을 받습니다. JT는 NWO Vidi 보조금(VI.Vidi.192.084)으로 재정적으로 지원됩니다. 저자들은 멀티플렉스 IHC 데이터를 저장하기 위한 워크플로우를 만드는 데 도움을 준 Eric van Dinther와 Ankur Ankan에게 감사를 표하고, Bengt Phung은 ROI 도면을 위해 QuPath에서 멀티플렉스 IHC 데이터를 구현하는 방법에 대한 지침을 제공해 주었습니다.
자료
| Name | Company | Catalog Number | Comments |
| anti-CD14 | Cell Marque | 114R-16 | section 3, clone EPR3653 |
| anti-CD163 | Cell Marque | 163M-15 | section 3, clone MRQ-26 |
| anti-CD19 | Abcam | ab134114 | section 3, clone EPR5906 |
| anti-CD1c (BDCA1) | Thermo Scientific | TA505411 | section 3, clone OTI2F4 |
| anti-CD20 | Thermo Scientific | MS-340-S | section 3, clone L26 |
| anti-CD3 | Thermo Scientific | RM-9107 | section 3, clone sp7 |
| anti-CD303/BDCA2 | Dendritics via Enzo Lifesciences/Axxora | DDX0043 | section 3, clone 124B3.13 |
| anti-CD56 | Cell Marque | 156R-94 | section 3, clone MRQ-42 |
| anti-CD66b | BD Biosciences | 555723 | section 3, clone G10F5 |
| anti-CD68 | Dako Agilent | M087601 | section 3, clone PG-M1 |
| anti-CD8 | Dako Agilent | M7103 | section 3, clone C8/144B |
| anti-Foxp3 | Thermo Scientific | 14-4777 | section 3, clone 236A/E7 |
| anti-Gp100 | Dako Agilent | M063401 | section 3, clone HMB45 |
| anti-HLA-DR, DP, DQ | Santa Cruz | sc-53302 | section 3, clone CR3/43 |
| anti-MART-1 | Thermo Scientific | MS-799 | section 3, clone A103 |
| anti-pan cytokeratin | Abcam | ab86734 | section 3, clone AE1/AE3 + 5D3 |
| anti-SOX10 | Sigma Aldrich | 383R | section 3, clone EP268 |
| anti-Tyrosinase | Sanbio | MONX10591 | section 3, clone T311 |
| anti-XCR1 | Cell Signaling Technologies via Bioké | 44665S | section 3, clone D2F8T |
| antibody diluent | Akoya BioSciences | SKU ARD1001EA | section 3, from Opal 7-Color Automation IHC Kit 50 slide (can optionally also be replaced by TBST with 10% BSA) |
| Bond Aspirating Probe | Leica Biosciences | S21.0605 | section 3 |
| Bond Aspirating Probe Cleaning | Leica Biosciences | CS9100 | section 3 |
| Bond Dewax Solution | Leica Biosciences | AR9222 | section 3 |
| Bond Objectglas label + print lint | Leica Biosciences | S21.4564.A | section 3 |
| Bond Research Detection System 2 | Leica Biosciences | DS9777 | section 3 |
| Bond RX autostainer | Leica Biosciences | - | section 3, automated platform |
| Bond TM Epitope Retrieval 1 - 1 L | Leica Biosciences | AR9961 | section 3 |
| Bond TM Epitope Retrieval 2 - 1 L | Leica Biosciences | AR9640 | section 3 |
| Bond TM Wash Solution 10x - 1 L | Leica Biosciences | AR9590 | section 3 |
| BOND Universal Covertile | Leica Biosciences | S21.4611 | section 3 |
| Bond(TM) Titration Kit | Leica Biosciences | OPT9049 | section 3 |
| Coverslip 24 x 32 mm #1 (0.13-0.16 mm) | Fisher Scientific | 15717592 | section 2 |
| coverslip 24 x 50 mm | VWR | 631-0146 | section 2 |
| DAPI Fluoromount-G | VWR | 0100-20 | section 3, whenever monoplex slides need to be quickly checked, not for official analysis, then DAPI is stained seperately for better results |
| Eosine | section 2, home made | ||
| Ethanol 99.5% | VWR | 4099.9005 | section 2 |
| Fluoromount-G | VWR | 0100-01 | section 3 |
| haematoxyline | - | section 2, home made | |
| ImmuNet | - | immune cell detection and phenotyping pipeline | |
| inForm software 2.4.10 | Akoya BioSciences | - | section 4 & 6 |
| OPAL 480 reagent pack | Akoya BioSciences | FP1500001KT | section 3 |
| OPAL 520 reagent pack | Akoya BioSciences | FP1487001KT | section 3 |
| OPAL 570 reagent pack | Akoya BioSciences | FP1488001KT | section 3 |
| OPAL 620 reagent pack | Akoya BioSciences | FP1495001KT | section 3 |
| OPAL 690 reagent pack | Akoya BioSciences | FP1497001KT | section 3 |
| OPAL 780 reagent pack | Akoya BioSciences | FP1501001KT | section 3 |
| Opal 7-Color Automation IHC Kit 50 slide | Akoya BioSciences | NEL821001KT | section 3 |
| PhenoChart 1.1.0 | Akoya BioSciences | - | section 5 |
| PhenoImagerHT | Akoya BioSciences | CLS143455 | section 4, digital pathology imager with slide viewer and imaging software (formerly known as Vectra Polaris) |
| Quick-D mounting medium | Klinipath | 7280 | section 2 |
| QuPath 0.3.2 | whole slide image analysis software platform | ||
| R 4.1.1 | |||
| Slide boxes | VWR | 631-0737 | section 1 |
| SuperFrost Plus | Thermo Scientific through VWR | 631-9483 | section 1 |
| Vectra Polaris software 1.0.13 | Akoya BioSciences | - | section 4 |
| Xylene | VWR | 4055-9005 | section 2 |
참고문헌
- Dunn, G. P., Bruce, A. T., Ikeda, H., Old, L. J., Schreiber, R. D. Cancer immunoediting: from immunosurveillance to tumor escape. Nature Immunology. 3 (11), 991-998 (2002).
- van der Woude, L. L., Gorris, M. A. J., Halilovic, A., Figdor, C. G., de Vries, I. J. M. Migrating into the tumor: a roadmap for T cells. Trends in Cancer. 3 (11), 797-808 (2017).
- Fridman, W. H., Pages, F., Sautes-Fridman, C., Galon, J. The immune contexture in human tumours: impact on clinical outcome. Nature Reviews. Cancer. 12 (4), 298-306 (2012).
- Fridman, W. H., et al. The immune microenvironment of human tumors: general significance and clinical impact. Cancer Microenvironment. 6 (2), 117-122 (2013).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Angell, H. K., Bruni, D., Barrett, J. C., Herbst, R., Galon, J. The Immunoscore: colon cancer and beyond. Clinical Cancer Research. 26 (2), 332-339 (2020).
- Angell, H., Galon, J. From the immune contexture to the Immunoscore: the role of prognostic and predictive immune markers in cancer. Current Opinion in Immunology. 25 (2), 261-267 (2013).
- Galon, J., et al. Cancer classification using the Immunoscore: a worldwide task force. Journal of Translational Medicine. 10, 205(2012).
- Galon, J., et al. World-wide Immunoscore Task Force: meeting report from the 34;Melanoma Bridge", Napoli, November 30th-December 3rd, 2016. Journal of Translational Medicine. 15 (1), 212(2017).
- Tumeh, P. C., et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance. Nature. 515 (7528), 568-571 (2014).
- Creemers, J. H. A., et al. A tipping point in cancer-immune dynamics leads to divergent immunotherapy responses and hampers biomarker discovery. Journal for Immunotherapy of Cancer. 9 (5), e002032(2021).
- Blank, C. U., Haanen, J. B., Ribas, A., Schumacher, T. N. CANCER IMMUNOLOGY. The "cancer immunogram". Science. 352 (6286), 658-660 (2016).
- Teruya-Feldstein, J. The immunohistochemistry laboratory looking at molecules and preparing for tomorrow. Archives of Pathology & Laboratory Medicine. 134 (11), 1659-1665 (2010).
- Ramos-Vara, J. A., Miller, M. A., et al. When tissue antigens and antibodies get along: revisiting the technical aspects of immunohistochemistry--the red, brown, and blue technique. Veterinary Pathology. 51 (1), 42-87 (2014).
- Hegde, P. S., Karanikas, V., Evers, S. The where, the when, and the how of immune monitoring for cancer immunotherapies in the era of checkpoint inhibition. Clinical Cancer Research. 22 (8), 1865-1874 (2016).
- Parra, E. Novel platforms of multiplexed immunofluorescence for study of paraffin tumor tissues. Journal of Cancer Treatment and Diagnosis. 2 (1), 43-53 (2018).
- Gorris, M. A. J., et al. Eight-color multiplex immunohistochemistry for simultaneous detection of multiple immune checkpoint molecules within the tumor microenvironment. Journal of Immunology. 200 (1), 347-354 (2018).
- Roelofsen, T., et al. Spontaneous regression of ovarian carcinoma after septic peritonitis; a unique case report. Frontiers in Oncology. 8, 562(2018).
- van den Brand, D., et al. Peptide-mediated delivery of therapeutic mRNA in ovarian cancer. European Journal of Pharmaceutics and Biopharmaceutics. 141, 180-190 (2019).
- van den Brand, D., et al. EpCAM-binding DARPins for targeted photodynamic therapy of ovarian cancer. Cancers. 12 (7), 1762(2020).
- Di Blasio, S., et al. The tumour microenvironment shapes dendritic cell plasticity in a human organotypic melanoma culture. Nature Communications. 11 (1), 2749(2020).
- van Beek, J. J. P., et al. Human pDCs are superior to cDC2s in attracting cytolytic lymphocytes in melanoma patients receiving DC vaccination. Cell Reports. 30 (4), 1027-1038 (2020).
- Rodriguez-Rosales, Y. A., et al. Immunomodulatory aged neutrophils are augmented in blood and skin of psoriasis patients. Journal of Allergy and Clinical Immunology. 148 (4), 1030-1040 (2021).
- Hoeijmakers, Y. M., et al. Immune cell composition in the endometrium of patients with a complete molar pregnancy: Effects on outcome. Gynecologic Oncology. 160 (2), 450-456 (2021).
- van Ravensteijn, S. G., et al. Immunological and genomic analysis reveals clinically relevant distinctions between angiosarcoma subgroups. Cancers. 14 (23), 5938(2022).
- van der Woude, L. L., et al. Tumor microenvironment shows an immunological abscopal effect in patients with NSCLC treated with pembrolizumab-radiotherapy combination. Journal for Immunotherapy of Cancer. 10 (10), e005248(2022).
- Graham Martinez, C., et al. The immune microenvironment landscape shows treatment-specific differences in rectal cancer patients. Frontiers in Immunology. 13, 1011498(2022).
- Cortenbach, K. R. G., et al. Topography of immune cell infiltration in different stages of coronary atherosclerosis revealed by multiplex immunohistochemistry. International Journal of Cardiology. Heart & Vasculature. 44, 101111(2023).
- van Wilpe, S., et al. Homologous recombination repair deficient prostate cancer represents an immunologically distinct subtype. Oncoimmunology. 11 (1), 2094133(2022).
- Gorris, M. A. J., et al. Paired primary and metastatic lesions of patients with ipilimumab-treated melanoma: high variation in lymphocyte infiltration and HLA-ABC expression whereas tumor mutational load is similar and correlates with clinical outcome. Journal for Immunotherapy of Cancer. 10 (5), e004329(2022).
- van Wilpe, S., et al. Intratumoral T cell depletion following neoadjuvant chemotherapy in patients with muscle-invasive bladder cancer is associated with poor clinical outcome. Cancer Immunology, Immunotherapy. 72 (1), 137-149 (2023).
- Sultan, S., Gorris, M. A. J., Buytenhuijs, F., Lvan de Woude, L. A Segmentation-free machine learning architecture for immune land-scape phenotyping in solid tumors by multichannel imaging. bioRxiv. , (2021).
- Parra, E. R., et al. Immuno-profiling and cellular spatial analysis using five immune oncology multiplex immunofluorescence panels for paraffin tumor tissue. Scientific Reports. 11 (1), 8511(2021).
- Parra, E. R., et al. Validation of multiplex immunofluorescence panels using multispectral microscopy for immune-profiling of formalin-fixed and paraffin-embedded human tumor tissues. Scientific Reports. 7 (1), 13380(2017).
- Sun, Z., Nyberg, R., Wu, Y., Bernard, B., Redmond, W. L. Developing an enhanced 7-color multiplex IHC protocol to dissect immune infiltration in human cancers. PLoS One. 16 (2), e0247238(2021).
- LeicaBiosystems. BOND RX Fully Automated Research Stainer Protocols. , https://www.leicabiosystems.com/en-nl/ihc-ish/ihc-ish-instruments/bond-rx/ (2022).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유