Method Article
Analyse immunohistochimique multiplex du paysage cellulaire immunitaire spatial du microenvironnement tumoral
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit en détail comment la caractérisation des cellules immunitaires du microenvironnement tumoral à l’aide de l’immunohistochimie multiplex est effectuée.
Résumé
Le paysage cellulaire immunitaire du microenvironnement tumoral contient potentiellement des informations pour la découverte de biomarqueurs pronostiques et prédictifs. L’immunohistochimie multiplex est un outil précieux pour visualiser et identifier différents types de cellules immunitaires dans les tissus tumoraux tout en conservant ses informations spatiales. Nous fournissons ici des protocoles détaillés pour analyser les populations de cellules lymphocytaires, myéloïdes et dendritiques dans des coupes de tissus. De la découpe de sections fixées au formol et noyées en paraffine, des procédures de coloration multiplex automatique sur une plate-forme automatisée, du balayage des lames sur un microscope d’imagerie multispectrale, à l’analyse d’images à l’aide d’un algorithme d’apprentissage automatique développé en interne, ImmuNet. Ces protocoles peuvent être appliqués à une variété d’échantillons tumoraux en changeant simplement de marqueurs tumoraux pour analyser les cellules immunitaires dans différents compartiments de l’échantillon (tumeur ou marge invasive) et appliquer une analyse du voisin le plus proche. Cette analyse ne se limite pas aux échantillons tumoraux mais peut également être appliquée à d’autres tissus (non-)pathogènes. Les améliorations apportées à l’équipement et au flux de travail au cours des dernières années ont considérablement réduit les délais de traitement, ce qui facilite l’application future de cette procédure dans le cadre du diagnostic.
Introduction
Les cellules immunitaires jouent un rôle crucial dans la protection contre les agents pathogènes tels que les virus et les bactéries, mais aussi contre les cellules cancéreuses1. Par conséquent, le système immunitaire au sein du microenvironnement tumoral (TME) est très prometteur pour la découverte de biomarqueurs pronostiques et prédictifs2. Les infiltrats des cellules immunitaires ont été corrélés au pronostic dans divers types de cancer, bien que cela n’ait pas encore été mis en œuvre dans les soins cliniques 3,4. Dans la plupart des types de tumeurs, un nombre élevé de lymphocytes T cytotoxiques et de lymphocytes T auxiliaires 1 et/ou un faible nombre de lymphocytes T régulateurs sont liés à de bons pronostics. Des efforts sont en cours pour intégrer ce qu’on appelle un « immunoscore » dans la stadification TNM du cancer colorectal, en la transformant en stadification TNM-I 5,6. L’immunoscore est dérivé du nombre total de lymphocytes T (détectés avec CD3) et de lymphocytes T cytotoxiques (détectés avec CD8) dans deux régions tumorales différentes : le noyau tumoral par rapport à la marge invasive (IM) des tumeurs. Il a également été proposé que l’immunoscore ait une valeur pronostique dans d’autres types de cancer, tels que le mélanome, le cancer du poumon et le cancer du sein 6,7,8,9. De plus, les infiltrats des cellules immunitaires peuvent également être corrélés à la réponse à l’immunothérapie par blocage de point de contrôle10. Cependant, ces biomarqueurs prédictifs doivent être validés dans des études prospectives avant de pouvoir être systématiquement mis en œuvre dans la pratique clinique. De plus, il a également été proposé qu’un seul biomarqueur soit insuffisant pour une prédiction significative11. Par conséquent, la création d’une carte complète d’un échantillon de patient en combinant différents biomarqueurs a été proposée comme un biomarqueur prédictif plus complet dans ce que l’on appelle « l’immunogramme du cancer »12.
Parmi les méthodes d’étude des cellules immunitaires au sein de l’EUT, la technique la plus ancienne et la plus connue est l’immunohistochimie (IHC), couramment utilisée pour les tests de diagnostic dans plusieurs maladies, en particulier le cancer13. Cette technique a longtemps été limitée à l’utilisation d’un ou de quelques marqueurs14 et a donc été supplantée dans le cadre de la recherche par d’autres techniques telles que la cytométrie en flux et le profilage d’expression génique (GEP). Cependant, les tissus tumoraux fixés au formol et inclus en paraffine (FFPE) généralement utilisés dans les diagnostics et la recherche de routine ne sont pas (de manière optimale) adaptés à la cytométrie en flux et à la GEP. De plus, bien que la GEP et la cytométrie en flux fournissent beaucoup d’informations sur le phénotype et la fonction cellulaires, le manque d’informations spatiales est un inconvénient majeur. Par conséquent, l’hétérogénéité au sein d’un échantillon, telle que les différences entre les zones infiltrées par les cellules immunitaires et les zones exclues des cellules immunitaires d’une tumeur, pourrait passer inaperçue15. De nouvelles plateformes ont été développées pour l’analyse multiplex des tissus FFPE, telles que l’IHC multiplex, la cytométrie de masse par imagerie et la détection de CO par indEXing (CODEX) qui peuvent être utilisées pour détecter plusieurs marqueurs simultanément dans une section de tissu16. Les cellules immunitaires de l’EUT font l’objet d’études approfondies afin de trouver les meilleurs biomarqueurs pour l’immunothérapie. Cependant, les techniques de multiplexage et l’analyse automatisée des images posent leurs propres obstacles.
Notre laboratoire possède une vaste expérience de la coloration IHC multiplex à l’aide de la méthode d’amplification du signal Opal/Tyramide (TSA) et l’a automatisée sur une plate-forme IHC (voir le tableau des matériaux)17,18,19,20,21,22,23,24,25,26,27,28, 29,30,31. Nous avons optimisé les panels de cellules immunitaires pour la détection de différents sous-ensembles de lymphocytes, de cellules myéloïdes et de cellules dendritiques (DC). Les tissus qui contiennent des zones de cellules immunitaires denses - pour les lymphocytes ou les morphologies cellulaires complexes (c’est-à-dire les cellules myéloïdes et les DC) - sont particulièrement difficiles à analyser, avec un risque de sur- ou de sous-estimer le nombre de cellules immunitaires présentes. Pour pallier à ce problème, le logiciel d’analyse ImmuNet a été développé par notre groupe32, et ce pipeline d’apprentissage automatique a considérablement amélioré la qualité de la détection de ces différents types de cellules immunitaires. Un protocole détaillé allant de l’obtention du matériel FFPE à l’analyse des densités de cellules immunitaires dans différents compartiments tissulaires et des distances entre les types de cellules immunitaires est décrit ici.
Ce protocole décrit la manière dont les panels IHC multiplexes sont réalisés au Centre médical de l’Université Radboud depuis la mise en œuvre de l’imageur numérique de pathologie en 2022. Les panels IHC multiplexes décrits peuvent être utilisés pour différents carcinomes (par exemple, poumon, prostate, colorectal, vessie, sein) avec l’utilisation d’un anticorps pan-cytokératine comme marqueur tumoral ou pour le mélanome avec l’utilisation d’anticorps associés aux mélanocytes comme marqueurs tumoraux. Ces protocoles IHC multiplex ont été soigneusement optimisés en termes de concentration d’anticorps primaires, de combinaisons de fluorophores et de séquence de la procédure de coloration. Nous et d’autres avons décrit l’optimisation du panel IHC multiplex plus tôt 17,33,34,35. Les panneaux IHC multiplex peuvent être adaptés, mais les pipelines d’analyse décrits doivent être évalués et éventuellement ajustés ou réentraînés en conséquence. Les protocoles IHC multiplexes à sept couleurs décrits utilisent les fluorophores Opal Opal480, Opal520, Opal570, Opal620, Opal690, Opal780 et 4',6-diamidino-2-phénylindole (DAPI), de sorte qu’un démélange facile et un balayage rapide sur l’imageur sont activés avec l’immunofluorescence multispectrale à une touche (MOTiF). La coloration et le balayage en neuf couleurs ne sont pas décrits dans ce protocole car cela nécessite encore plus de réglage précis de la configuration expérimentale et un autre mode de balayage sur l’imageur qui utilise le filtre accordable à cristaux liquides.
Protocole
Le matériel destiné aux patients qui est présenté pour ce protocole faisait partie d’une étude précédemment menée et a été officiellement considéré comme exempté de l’approbation éthique médicale par le comité local d’éthique médicale Radboudumc conformément à la législation néerlandaise (numéro de dossier 2017-3164)30.
1. Collecte du matériel FFPE, sélection des blocs et préparation des échantillons
- Récupérez les identifiants de bloc FFPE à partir des dossiers patients auprès des médecins traitants ou des pathologistes. Vérifiez auprès des réglementations locales si une autorisation éthique est requise.
- Demandez des blocs FFPE auprès des archives de pathologie locales ou d’un ou plusieurs hôpitaux externes.
REMARQUE : Il est également possible qu’un matériel tumoral ou une biopsie soit acquis pour une étude particulière. Cela peut être le cas pour de petits essais cliniques ou des études sur des animaux. Dans ces cas, le traitement de l’échantillon de tissu peut être la responsabilité du chercheur. - Lorsque plusieurs blocs FFPE sont disponibles, sélectionnez le bloc FFPE le plus représentatif contenant du tissu tumoral viable, de préférence avec le tissu stromal environnant présent en évaluant les lames colorées à l’hématoxyline et à l’éosine (HE) (Figure 1).
REMARQUE : Il est conseillé d’obtenir l’avis d’un expert pour cette sélection (par exemple, un pathologiste). Il est possible que les HE ne soient pas disponibles pour l’évaluation du contenu d’un bloc FFPE et que de nouveaux doivent être créés pour la sélection. Allez à la section 2 pour une description. - Découper des rubans FFPE de 4 μm d’épaisseur sur un microtome.
REMARQUE : L’épaisseur peut être comprise entre 1 μm et 6 μm sans impact notable sur les taches ; Cependant, 4 μm est le plus standard. - Montez les échantillons sur des lames de verre à une position favorable à la fluidique de l’autocolorant (Figure 2A-C) en utilisant l’une des méthodes décrites ci-dessous :
- Placez les sections à la surface de l’eau distillée à 40 °C dans un bain-marie pour les étirer et ramassez-les avec une lame de verre.
OU
Placez les lames de verre sur une plaque chauffante à 40 °C, en veillant à couvrir l’endroit où la section doit être montée sur la lame avec une goutte d’eau distillée. Placez la section au-dessus de cette goutte à l’aide d’une pince et laissez-la s’étirer. Absorbez l’eau distillée à l’aide d’une serviette en papier et retirez l’excès d’eau en tapotant la lame.
REMARQUE : Placer des sections de tissu trop près de l’étiquette de la lame entraînera une coloration sous-optimale (Figure 2D,E). Nous avons tendance à monter 6 à 10 lames de verre par échantillon pour réaliser les différents panels IHC multiplex et pour avoir une sauvegarde.
- Placez les sections à la surface de l’eau distillée à 40 °C dans un bain-marie pour les étirer et ramassez-les avec une lame de verre.
- Laissez sécher les lames de verre montées à 56 °C pendant 1 h ou toute la nuit à 37 °C.
- Utilisez les lames de verre montées pour l’expérience ou stockez-les dans des boîtes à 4 °C.
REMARQUE : D’après notre expérience jusqu’à présent, ces lames montées peuvent être stockées pendant des années avant que la coloration IHC multiplex ne soit effectuée.
2. Génération de lames colorées à l’hématoxyline et à l’éosine
REMARQUE : Toutes les étapes suivantes de la section 2 doivent être effectuées dans une hotte.
- Déparaffiniser les lames dans du xylène (2 x 5 min).
- Réhydrater dans de l’éthanol (99,6 % 1 x 5 min ; 95 % 1 x 5 min ; 70 % 1 x 2 min). Vous pouvez également tremper les lames 3 fois dans de l’éthanol à 99,6 %.
- Lavez les lames à l’eau distillée (2 min).
- Teintez les noyaux avec de l’hématoxyline (10 min).
- Lavez les lames avec du H2O distillé (5 min).
- Colorer les lames avec de l’éosine (5 min).
- Déshydratez les lames en les trempant 3x dans de l’éthanol à 99,6%.
- Trempez les lames 2x dans du xylène.
- Ajoutez quelques gouttes de support de montage et scellez avec une lamelle.
- Laissez les lames durcir et retirez-les de la hotte lorsque tous les produits chimiques se sont évaporés.
3. Réalisation d’IHC monoplex et multiplex dans l’autocolorateur
- Calculez la quantité de réactif nécessaire en fonction du nombre d’échantillons à colorer.
REMARQUE : Par cycle, l’autocolorant a une capacité de 30 lames et prend ~18 h pour compléter le protocole IHC multiplex avec six anticorps. Lorsque plus de lames doivent être colorées, plusieurs lots peuvent être mis en place chaque soir de la semaine (de travail) ; 4 nuits de 30 toboggans = 120 toboggans par semaine.- Préparez tous les réactifs nécessaires au début de la semaine. Le système d’autocoloration distribue 150 μL de réactif par lame. Utilisez les récipients de titrage de 6 mL pour les réactifs d’anticorps et d’opale et les récipients de 30 mL pour le réactif de blocage et l’anticorps secondaire-peroxydase de raifort.
REMARQUE : Les contenants de 6 ml ont des inserts pratiques qui peuvent facilement être retirés et remplacés si nécessaire. Avec les calculs de réactifs, il faut tenir compte du volume mort de 1,6 mL ou 300 μL pour le récipient de titrage de 30 mL ou 6 mL, respectivement. - Diluer tous les fluorophores d’opale et la digoxigénine (DIG) 1:100 dans le diluant fourni ; diluer Opal780 1:25 dans le diluant de l’anticorps. Diluer tous les anticorps primaires dans le diluant d’anticorps, avec les dilutions spécifiées dans le fichier supplémentaire 1.
- Préparez tous les réactifs nécessaires au début de la semaine. Le système d’autocoloration distribue 150 μL de réactif par lame. Utilisez les récipients de titrage de 6 mL pour les réactifs d’anticorps et d’opale et les récipients de 30 mL pour le réactif de blocage et l’anticorps secondaire-peroxydase de raifort.
- Pour suivre ce protocole, exécutez l’IHC monoplex (Supplemental File 2) sur des lames contenant à la fois du tissu de contrôle des amygdales et d’autres types de tissus (tumoraux) d’intérêt avant de commencer l’expérience IHC multiplex réelle pour vous assurer que tous les réactifs sont bien préparés.
REMARQUE : Le monoplex IHC prend ~3,5 h et peut être vérifié avant la fin de cette journée pour les modèles de signal et l’intensité. Si certains signaux sont trop faibles (Figure 3), des ajustements peuvent être apportés aux réactifs. - Pour la correction de l’autofluorescence, préparez une lame avec du tissu (tumoral) contenant des structures autofluorescentes, telles que le sang et le collagène. Préparez cette lame en même temps avec des lames IHC monoplexes, mais avec un réactif bloquant remplaçant l’anticorps et les réactifs Opal (Dossier supplémentaire 3).
REMARQUE : En principe, une telle lame peut être réutilisée pour l’imagerie multispectrale jusqu’à ce que la correction de l’autofluorescence ne soit plus optimale. Cependant, avec des tissus hautement autofluorescents, tels que le cerveau et le foie, il est conseillé d’utiliser ces tissus pour la correction de l’autofluorescence. - À chaque cycle IHC multiplex, chargez 29 échantillons dans le système d’autocoloration avec une lame de tissu de contrôle pour vérifier les performances de chaque cycle IHC multiplex.
- Téléchargez les protocoles IHC multiplex depuis le site Web de l’autostainer sous l’onglet Téléchargements et ajustez-les pour s’adapter à chaque panneau IHC multiplex personnalisé36. Pour l’IHC multiplex, voir le fichier supplémentaire 4 pour le protocole et pour les panneaux IHC multiplex personnalisés, voir le fichier supplémentaire 1.
- Une fois le protocole de coloration terminé, sortez les lames de l’autocolorateur et mettez-les dans un récipient avec un tampon de lavage.
- Pour éviter la contamination du système d’autocoloration par le DAPI, car les échantillons sont déjà colorés à de très faibles concentrations, appliquez le DAPI manuellement avant de recouvrir les lames avec des lamelles. Ajouter deux gouttes de DAPI par mL de tampon de lavage et incuber pendant 5 min à température ambiante dans l’obscurité.
REMARQUE : Pour la construction de bibliothèques spectrales, il est important de ne pas avoir de DAPI coloré dans les échantillons. Une goutte de DAPI par mL de tampon de lavage et 10 min d’incubation à RT sont également possibles. - Lavez les lames 3x avec un tampon de lavage.
- Placez les lames sur du papier absorbant et tapotez l’excédent de tampon de lavage sur les diapositives.
- Piquez quelques gouttes de milieu de montage sur le tissu.
- Placez doucement une lamelle en verre sur le support de montage pour couvrir la diapositive à un angle afin d’éviter les bulles d’air.
- Éliminez l’excès de fluide de montage et les bulles d’air en appuyant doucement sur la lamelle en verre à l’aide d’une pince ou d’une pointe de pipette propre.
- Laissez les lames intactes pendant ~24 h avant que le support de montage ne se solidifie, soit horizontalement sur une planche à lames de microscopie, soit en les chargeant directement dans le microscope pour l’imagerie.
- Une fois le support de montage solidifié ou après l’imagerie des lames, stockez les lames dans des boîtes de microscopie à 4 °C.
4. Imagerie à l’aide de l’imageur de pathologie numérique et annotation des fichiers de numérisation
- Allumez l’imageur en appuyant sur le bouton d’alimentation situé à droite de l’appareil. Après au moins 20 s, démarrez le logiciel.
REMARQUE : Attendez 20 s pour permettre au matériel de démarrer correctement. - Chargez les diapositives dans les cassettes par quatre diapositives.
- Facultatif : Entrez les diapositives dans un fichier .csv pour lequel un modèle peut être téléchargé (Fichier supplémentaire 5). Pour charger le fichier .csv dans le programme, enregistrez-le dans C :\Users\Public\Akoya\VectraPolaris\States.
REMARQUE : Un maximum de 20 cassettes ou 80 diapositives peuvent être chargées simultanément.
- Facultatif : Entrez les diapositives dans un fichier .csv pour lequel un modèle peut être téléchargé (Fichier supplémentaire 5). Pour charger le fichier .csv dans le programme, enregistrez-le dans C :\Users\Public\Akoya\VectraPolaris\States.
- Paramètres de référence
- Ouvrez Check Dashboard dans le menu principal.
REMARQUE : Une cassette avec des glissières de référence est fournie par le fabricant et peut être maintenue en option en permanence dans l’emplacement 20. - Réglez les références en fond clair sur la diapositive fournie une fois par semaine selon les instructions du fabricant (cela prend quelques minutes).
- Réglez les références de fluorescence sur la lame fournie une fois par mois selon les instructions du fabricant (cela prend plus de 1 h).
- Ouvrez Check Dashboard dans le menu principal.
- Élaboration ou ajustement du protocole
- Revenez au menu principal et cliquez sur Modifier le protocole pour créer un protocole.
- Cliquez sur Nouveau... et sélectionnez Fluorescence comme mode d’imagerie, Balayage multispectral de lames et Opal Polaris 5, 6 et 7 couleurs sous l’option Coloration .
- Donnez un nom au protocole sous Nom du protocole et enregistrez-le dans une étude en sélectionnant une étude dans Études disponibles ou créez une étude sous Créer une nouvelle étude | Nom de l’étude.
- Terminez en sélectionnant Créer un protocole.
- Pour ce type de numérisation, utilisez uniquement la fenêtre de gauche Paramètres de numérisation de diapositives multispectrales ; ignorez la fenêtre de droite Paramètres de champ multispectral.
- Numérisez les lames à différents grossissements. Pour suivre ce protocole, balayez à un grossissement de 20x en laissant la résolution des pixels à 0,50 μm (20x).
- Réglez les temps d’exposition en sélectionnant Expositions de balayage.
- Chargez la cassette dans laquelle les glissières sont conservées en sélectionnant la bonne fente sous l’option Porte-charge .
- Pour vous aider à naviguer dans les diapositives, sélectionnez Prendre une vue d’ensemble pour acquérir une image d’ensemble du support contenant les diapositives après le chargement du support. Pour l’activer ou la désactiver automatiquement, cliquez sur l’icône d’engrenage en haut à droite, allez dans Préférences..., et cochez l’option activée ou désactivée sous Vue d’ensemble de la navigation Image pour activer le support d’image automatique lors du chargement des tâches interactives.
- Réglez les temps d’exposition par filtre sur les lames colorées IHC monoplex correspondantes en sélectionnant Définir les expositions de balayage et en trouvant différents points avec un signal positif. Faites la mise au point manuellement ou utilisez la mise au point automatique et sélectionnez Exposition automatique après avoir basculé vers le filtre compatible pour ce signal. Sélectionnez le temps d’exposition le plus bas pour éviter la surexposition et prenez des instantanés de chaque diapositive à titre de référence une fois que tous les temps d’exposition sont réglés (Figure 3).
REMARQUE : Ignorez l’option Définir l’exposition des champs pour ce type de numérisation. - Réglez les temps d’exposition sur une lame colorée multiplex en vérifiant tous les filtres à quelques endroits avec un signal positif. Réduisez le temps d’exposition automatique le plus bas de 10 % pour éviter la surexposition et prenez quelques clichés une fois que tous les temps d’exposition sont réglés.
- Prenez des instantanés de la lame non colorée pour la compensation de l’autofluorescence à l’aide du filtre AF échantillon pour naviguer (Figure 3H).
REMARQUE : Les emplacements avec des érythrocytes et des structures de collagène sont intéressants. Il peut être nécessaire de réduire le temps d’exposition du filtre Opal480 pour les régions autofluorescentes fortes. Si le signal Opal480 est suffisamment fort, il doit toujours être bien séparé (voir section 6) des structures autofluorescentes en raison de la mise en œuvre du filtre propriétaire Sample AF. - Évaluer la qualité de la coloration et de l’imagerie à l’aide du logiciel (voir rubriques 5 et 6 ; Figure 4, Fichier supplémentaire 6 : Figure supplémentaire S1 et Figure supplémentaire S2).
- Sélectionnez le bouton Enregistrer... pour vous assurer que le protocole et ses temps d’exposition ajustés sont enregistrés dans le protocole.
REMARQUE : Lorsque le protocole est déjà enregistré, aucune notification supplémentaire des ajustements non enregistrés n’est donnée par le logiciel jusqu’à présent.
- Numérisation automatique des diapositives
- Revenez au menu principal et cliquez sur Numériser les diapositives pour numériser les diapositives.
- Entrez manuellement les noms/ID des diapositives ainsi que les tâches et le protocole correspondants sous Configurer les tâches ou automatiquement à partir du fichier .csv précédemment créé avec Charger la configuration.
- Cliquez sur Analyser pour lancer l’analyse.
- Attendez qu’une fenêtre s’ouvre pour enregistrer la configuration de l’analyse. Cliquez sur Enregistrer pour utiliser les paramètres par défaut et lancer l’analyse.
REMARQUE : La numérisation à l’aide de cette méthode prend ~10-20 min par lame. Selon le nombre de diapositives, la numérisation peut prendre jusqu’à une journée entière. - Vérifiez si l’analyse des diapositives a réussi pour toutes les diapositives en recherchant des messages d’erreur. Pour savoir si l’analyse a réussi, recherchez un fichier de numérisation de diapositives entières Akoya enregistré (.qptiff) de la numérisation et le tissu complet de la numérisation.
5. Annotation des données à l’aide de la visionneuse de diapositives
- Revenez au menu principal et cliquez sur Lancer le Phenochart pour ouvrir le visualiseur de diapositives.
- Si les fichiers numérisés ne sont pas directement visibles, indiquez leur emplacement en cliquant d’abord sur l’icône d’engrenage dans le coin supérieur droit, allez dans Modifier l’emplacement du navigateur... et sélectionnez au hasard l’un des fichiers .qptiff de l’ensemble de données qui vous intéresse.
REMARQUE : Les données sont stockées par défaut dans D :\Data\VectraPolaris. - Chargez une diapositive en la sélectionnant et en cliquant sur Charger dans le coin supérieur droit ou en double-cliquant dessus.
- Connectez-vous en cliquant sur le bouton Connexion dans le coin supérieur droit.
REMARQUE : Le nom d’utilisateur peut être simplement les initiales ou le nom et est utilisé pour garder une trace de qui a fait quelles annotations. - Pour effectuer le démixage , cliquez sur le bouton Décalage en haut et sélectionnez l’option Opale + AF .
REMARQUE : Ceci est utile pour se débarrasser d’une partie du signal autofluorescent près du canal Opal 480, mais pas tous. - Pour générer un algorithme de traitement par lots des données, sélectionnez des images représentatives à l’aide du tampon à l’aide de l’option pour les images 1 x 1 des projets inForm (taille de l’image : 928 μm x 696 μm).
REMARQUE : Quelques tampons représentatifs contenant la tumeur, le stroma, l’arrière-plan et différents types de cellules immunitaires sont sélectionnés tout au long de l’ensemble de données pour aboutir à ~20-30 images. - En fonction de ce qui doit être analysé dans le tissu, sélectionnez une région d’intérêt à l’aide de l’option ROI et sélectionnez inForm Batch. Supprimez manuellement les images qui n’ont pas besoin d’être analysées, telles que les images qui sont trop éloignées de la tumeur ou en arrière-plan.
REMARQUE : Nous avons tendance à dessiner un retour d’intérêt autour de l’ensemble de la tumeur et à sélectionner une image supplémentaire loin de la région tumorale pour pouvoir analyser un IM de ~0,5 mm.
Si le retour sur investissement dessiné est relativement faible, il se composera de 2 à 9 images fusionnées 20x. Comme ce n’est pas notre choix, tamponnez manuellement le tissu d’intérêt (sélectionné pour inForm Batch) pour contourner ce problème. - Une fois l’annotation terminée, les annotations sont automatiquement enregistrées et chargent la diapositive suivante.
- Pendant le processus d’annotation, vérifiez que les diapositives sont correctement numérisées.
- S’il manque un fichier .qptiff ou si une lame n’est pas numérisée avec succès, vérifiez s’il y a du tissu sur la lame, nettoyez la lame avec de l’éthanol à 70 % et numérisez à nouveau.
- Si le tissu n’est pas entièrement scanné, manquant ainsi une région potentiellement importante (tumeur), ou si le balayage de la région importante était flou, nettoyez la lame avec de l’éthanol à 70% et scannez à nouveau.
REMARQUE : Dans les deux cas, il peut également être utile d’encercler le tissu avec un marqueur sur le dessus de la lamelle pour aider le système à localiser le tissu et à essayer de le scanner à nouveau (Fichier supplémentaire 6 : Figure supplémentaire S3). Dans nos mains, un marqueur rouge fin fonctionnait mieux qu’un marqueur noir épais.
- Une fois la numérisation et l’annotation de tous les échantillons terminées, sauvegardez les données en les stockant sur un autre ordinateur ou un disque externe.
6. Démixage spectral
- Ouvrez le logiciel d’analyse automatisée d’images inForm.
- Chargez les images dans le logiciel à l’aide de Fichier | Ouvrir l’image ; Sélectionnez les fichiers .qptiff. Laissez les tampons, marqués comme projets inForm à l’étape 5.6, être chargés dans le projet.
- Chargez les fichiers .qptiff qui sont imagés pour la compensation d’autofluorescence.
- Pour compenser l’autofluorescence, utilisez l’outil Sélectionner l’autofluorescence sur l’image pour tracer une ligne sur l’image à partir de la lame non colorée à travers différents types de structures autofluorescentes, telles que les érythrocytes et le collagène.
- Dans la section Modifier les marqueurs et les couleurs... , attribuez des noms de marqueurs qui correspondent au fluorophore Opale et ajustez la couleur à celle que vous préférez.
- Pour démélanger les fluorophores, sélectionnez Tout préparer dans le coin inférieur gauche.
- Parcourez les images et vérifiez si tous les signaux sont visibles dans les images et si le démixage s’est bien passé. Sélectionnez l’icône du globe oculaire pour éteindre et rallumez tous les marqueurs un par un pour vérifier la qualité.
- Si vous le souhaitez, entraînez les algorithmes pour la segmentation des tissus, la segmentation cellulaire et le phénotypage.
- Allez dans l’onglet Exporter et créez un nouveau répertoire d’exportation vide en cliquant sur le bouton Parcourir... sous le répertoire d’exportation.
- Sous Images à exporter :, sélectionnez Image composite et Images de composant (TIFF multi-images).
- Sélectionnez Fichier | Enregistrer | Projet d’enregistrement de l’algorithme à un certain emplacement.
- Allez dans l’onglet Analyse par lots verticalement à gauche pour le traitement par lots des diapositives.
- Sélectionnez Créer des répertoires distincts pour chaque élément sous les options d’exportation.
- Pour ajouter des diapositives à analyser, sélectionnez des fichiers .qptiff sous le bouton Ajouter des diapositives... et chargez-les dans l’analyse par lots.
- Sélectionnez Exécuter pour démarrer le traitement par lots des diapositives.
7. Dessin du retour sur investissement
- Créez un dossier avec uniquement les fichiers de composant de la section 6, mais conservez la structure hiérarchique des dossiers intacte (les fichiers de composant se trouvent dans des dossiers nommés par sample/slide).
- Ouvrez le logiciel de visualisation de diapositives QuPath entier.
- Cliquez sur Créer un projet sur la gauche et sélectionnez/créez un nouveau dossier vide avec un nom approprié.
- Cliquez sur Automatiser et sélectionnez Afficher l’éditeur de script.
- Copiez-collez le script disponible dans le fichier supplémentaire 7. À la ligne 34, modifiez l’emplacement des dossiers de diapositives contenant tous les fichiers de composant (le dossier créé à l’étape 7.1.
- Sélectionnez Exécuter et revenir lorsque l’assemblage par lots des diapositives est terminé (le lendemain ou plus tard) pour continuer.
- Faites glisser les fichiers .ome.tif générés dans le projet QuPath et enregistrez-les en tant que projet.
- Lorsqu’une nouvelle fenêtre s’affiche automatiquement, sélectionnez Définir le type d’image | Fluorescence et cliquez sur Importer.
- Dans le menu de gauche, observez la liste des échantillons ; double-cliquez sur l’un d’entre eux pour ouvrir l’échantillon (Figure 5A).
- Pour ajuster l’intensité des canaux afin de les rendre plus visibles, cliquez sur l’icône de contraste.
- Sélectionnez toutes les chaînes et cliquez sur Réinitialiser.
- Désactivez l’autofluorescence.
- Pour commencer à dessiner une ROI pour la tumeur, cliquez sur l’icône de contraste et sélectionnez Afficher les niveaux de gris. Sélectionnez le canal marqueur de la tumeur et ajustez l’intensité pour le rendre visible de manière optimale (Figure 5B).
- Cliquez sur l’outil pinceau pour dessiner approximativement le retour sur investissement d’une tumeur.
- Tout en sélectionnant l’outil baguette, cliquez à l’extérieur de la zone d’intérêt tout en appuyant sur la touche Alt pour lisser la zone d’intérêt de l’extérieur (Figure 5C).
- Fusionnez des morceaux de tumeur séparés avec le même retour sur investissement.
- Donnez au ROI un nom approprié, tel que tumeur, en cliquant avec le bouton droit de la souris sur l’annotation dans la liste de gauche ; sélectionnez Définir les propriétés et entrez le nom.
- Pour créer un retour sur investissement pour l’IM, développez le retour sur investissement existant à partir de la région tumorale en sélectionnant : Objets | Annotations... | Développez les annotations.
- Sélectionnez la taille du rayon d’expansion, puis sélectionnez Supprimer l’intérieur et Contraindre au parent (Figure 5D).
- Cliquez sur l’icône de contraste, sélectionnez le canal d’autofluorescence et ajustez l’intensité pour le rendre visible de manière optimale.
- Cliquez sur la baguette et ajustez le retour sur investissement tout en appuyant sur la touche alt pour lisser le retour sur investissement de l’extérieur et supprimer tout arrière-plan qui ne devrait pas faire partie de ce retour sur investissement.
- Donnez au retour d’investissement un nom approprié, tel que marge invasive ou IM , en cliquant avec le bouton droit de la souris sur l’annotation dans la liste de gauche, en sélectionnant Définir les propriétés, en saisissant le nom et en remplaçant sa couleur par du vert.
- Enregistrez les annotations : Fichier | Exportation d’objets | Exporter tous les objets et cliquez sur OK avec la sélection par défaut dans Exporter en tant que FeatureCollection et enregistrez-le à l’emplacement de votre choix.
8. Détection des cellules immunitaires
- Comme ImmuNet utilise des données de composant (fichiers TIFF multicanaux) pour l’entraînement et l’inférence, divisez les annotations en ensembles d’entraînement et de validation. Pour entraîner le modèle, suivez les étapes décrites dans le fichier Readme du référentiel, en remplaçant l’exemple de jeu de données et les annotations par les données souhaitées. En dehors des différentes cellules immunitaires, fournissez au modèle des exemples négatifs en faisant des annotations d’arrière-plan sur des sites qui ne doivent pas être reconnus comme une cellule d’intérêt : cellules tumorales, autres cellules ou « aucune cellule » (structures qui pourraient être confondues avec des cellules d’intérêt) ; voir la publication ImmuNet pour plus de détails32.
- À l’aide des annotations de validation, assurez-vous que les performances sont satisfaisantes. Examinez le taux d’erreur par type d’annotation (une part d’annotations de validation que le modèle n’a pas détectées), la mesure d’évaluation la plus simple. Évaluez les performances par rapport aux faux positifs en faisant quelques retours sur investissement entièrement annotés et en calculant les scores F.
- En plus de l’évaluation quantitative, inspectez visuellement la prédiction pour avoir une idée qualitative des erreurs que le modèle a tendance à faire (Figure 6, Fichier supplémentaire 6 : Figure supplémentaire S4 et Figure supplémentaire S5). Si les performances du modèle sont jugées insuffisantes, visualisez la prédiction de certaines vignettes comme décrit dans le référentiel et vérifiez quels sites sont les plus sujets aux erreurs. Faites plus d’annotations sur ces sites et réexécutez l’entraînement et l’évaluation du modèle.
- Lorsque les performances cibles sont atteintes, exécutez l’inférence pour l’ensemble du jeu de données comme décrit dans la section Inférence pour l’ensemble du jeu de données du fichier Readme du référentiel. Utilisez les fichiers .csv obtenus avec la prédiction du modèle comme entrée pour l’analyse des données (écrivez un script Python ou R pour cela).
9. Prédiction, phénotypage et analyse des données
REMARQUE : Dans cette section, nous donnons un exemple d’analyse de données simple pour un seul échantillon de mélanome coloré avec le panel de lymphocytes, qui combine les emplacements des cellules immunitaires identifiées par ImmuNet (section 8) et les zones d’intérêt délimitées avec QuPath (section 7). L’analyse a été effectuée dans R 4.1.1 (un script est fourni en tant que fichier supplémentaire 8). Le script nécessite les paquets : plyr 1.8.8, dplyr 1.0.8, tidyr 1.2.0, sf 1.0-7, ggplot2 3.4.0, RANN 2.6.1 et RColorBrewer 1.1-2, qui peuvent être installés avec la commande install.packages(). En entrée, il prend un fichier .csv avec la prédiction d’un échantillon d’ImmuNet et un fichier avec des ROI exportés depuis QuPath. Les étapes 9.1 à 9.6 décrivent l’analyse d’un seul échantillon effectuée dans le script fourni, et les sections 9.7 à 9.9 décrivent les options d’analyse de plusieurs échantillons.
- Après avoir chargé la prédiction d’ImmuNet dans R, déterminez les seuils d’expression des marqueurs prédits en traçant les marqueurs qui définissent les phénotypes les uns par rapport aux autres et en sélectionnant les seuils qui séparent le mieux les populations.
REMARQUE : La stratégie de contrôle utilisée pour l’échantillon donné est illustrée à la figure 7B. Les stratégies de contrôle pour les panels de cellules myéloïdes et dendritiques sont présentées dans le fichier supplémentaire 6 : figure supplémentaire S6 et figure supplémentaire S7. - Après avoir déterminé les seuils, utilisez-les pour attribuer à chaque prédiction ImmuNet un phénotype défini dans un panneau. Dans certaines prédictions, observez qu’aucun des marqueurs prédits n’est au-dessus du seuil ou que la combinaison de marqueurs considérés comme exprimés après le seuillage peut être incohérente (par exemple, les prédictions CD3+ CD20+ dans les panels de lymphocytes). Si le rendement du modèle est bon à l’étape 8.3, la fraction de ces prédictions sera faible ; Filtrez-les avant l’analyse.
- Pour analyser séparément les retours sur investissement de la tumeur et sa marge invasive jusqu’à 100 μm dessinée dans QuPath, chargez les fichiers GeoJSON correspondants dans R et, pour chaque prédiction, déterminez le retour sur investissement dans lequel la prédiction se situe.
- Pour une vérification de l’intégrité et dans le cadre de l’analyse exploratoire des données, visualisez les cellules immunitaires trouvées dans un échantillon séparément dans les zones d’intérêt correspondantes ainsi que les limites des zones d’intérêt (figure 7A).
- Maintenant, calculez les densités des différentes cellules immunitaires séparément pour chaque retour sur investissement. Les densités trouvées dans l’échantillon donné sont indiquées dans le tableau 1.
- Si plusieurs échantillons sont disponibles, visualisez la distribution des densités cellulaires. Transformez log-transformer les valeurs de densité pour obtenir des valeurs normalement distribuées.
REMARQUE : Lorsque le nombre de certains phénotypes est égal à 0, ceux-ci ne peuvent pas être transformés en log, ce qui entraîne des valeurs manquantes. Pour surmonter ce problème, le lissage LaPlacian peut être appliqué en ajoutant 0,5 à tous les comptages cellulaires avant de diviser par la surface. - Analysez les valeurs de densité et tracez-les à l’aide du logiciel de votre choix (Figure 8).
- Les emplacements préservés des cellules permettent l’analyse spatiale. Par exemple, pour chaque cellule immunitaire détectée, trouvez un voisin le plus proche, puis pour chaque phénotype, calculez le pourcentage de cas où les différents phénotypes se produisent comme le voisin le plus proche.
REMARQUE : Étant donné que le nombre de cellules tueuses naturelles (NK) trouvées dans cet échantillon était très faible, nous les avons exclues de cette analyse. Les résultats obtenus pour les ROI de la tumeur et de l’IM sont donnés à la figure 9.
Résultats
Des blocs FFPE contenant du tissu tumoral ont été sélectionnés sur la base de rapports de pathologie et de lames colorées à l’HE. Lorsque plusieurs lésions tumorales sont réséquées chez le patient et/ou que les échantillons tumoraux sont volumineux, ceux-ci sont divisés sur plusieurs blocs FFPE. Nous préférons analyser les cellules immunitaires à la fois dans le compartiment tumoral et dans ce que l’on appelle la marge invasive (IM) de la tumeur. L’IM est un tissu stromal non cancéreux qui est adjacent à la tumeur. Par conséquent, lorsqu’il y a plusieurs blocs FFPE disponibles pour un échantillon de tumeur, les blocs FFPE qui contiennent les deux types de tissus sont sélectionnés. Comme on le voit sur les lames colorées à l’HE, un bloc FFPE contenait du tissu tumoral et du tissu stromal adjacents à la tumeur (Figure 1A). Un autre bloc FFPE de la même tumeur contenait beaucoup moins de tissu stromal environnant (Figure 1B). Cependant, pour certains échantillons de tissus, il n’y a pas de choix dans les blocs FFPE ou l’IM n’est présent dans aucun des blocs FFPE. C’est souvent le cas pour les biopsies (à l’aiguille), qui doivent être prises en compte lors de l’interprétation des données.
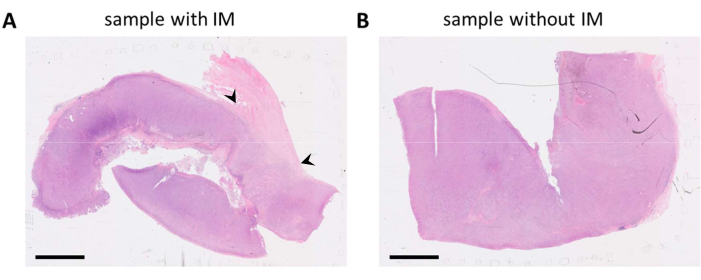
Figure 1 : Lames colorées à l’HE d’un échantillon de tumeur de mélanome. (A) Un exemple d’échantillon de tumeur avec du tissu stromal adjacent à la tumeur (IM) dans le coin supérieur droit de l’échantillon (indiqué par des pointes de flèches noires). (B) Un autre échantillon de la même lésion tumorale avec peu ou pas de tissu stromal présent dans l’échantillon. Barres d’échelle = 5 mm. Abréviations : HE = hématoxyline et éosine ; IM = marge invasive. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La coloration IHC multiplex avec un panneau à sept couleurs proposé (Fichier supplémentaire 4) peut être effectuée soit manuellement dans un processus de coloration de 3 jours (compte tenu des heures de travail normales), soit pendant la nuit dans un autocolorant. Lors de l’utilisation de l’autocolorateur, les sections doivent être montées à un endroit particulier sur la lame de verre qui permet une fluidique optimale du système (Figure 2A). Lorsque les sections sont correctement montées sur des glissières (Figure 2B), elles seront uniformément colorées (Figure 2C). Si les sections ne sont pas montées de manière optimale sur la lame de verre (Figure 2D), il en résulte souvent un motif de coloration sous-optimal (Figure 2E) car la fluidique de l’autocolorateur n’atteint pas le tissu (complet). Cela peut se produire lorsque les échantillons sont très volumineux ou lorsque les lames montées sont fournies par quelqu’un qui n’est pas au courant de ce problème. Dans ces cas, seule la partie bien colorée de la lame doit être sélectionnée pour l’analyse. Un autre choix pour ces types d’échantillons pourrait être de les colorer manuellement pour répartir les liquides de manière optimale.
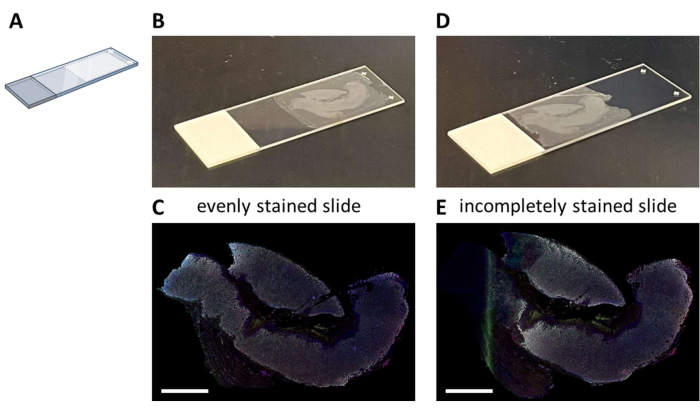
Figure 2 : Montage de la section FFPE sur la lame de verre et impact. (A) Schéma de l’endroit où monter sur la lame de verre pour une coloration optimale sur l’autostainer. (B) Exemple d’une glissière correctement montée. (C) Des lames correctement montées permettent d’obtenir une section de tissu uniformément colorée. (D) Exemple d’une glissière montée non optimale. (E) Des lames montées de manière non optimale peuvent entraîner une coupe de tissu colorée incomplète, comme on le voit sur le côté gauche de cette image. Barres d’échelle = 5 mm. Abréviation : FFPE = fixé au formol et enrobé de paraffine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Lorsque de grandes expériences IHC multiplex sont réalisées en plusieurs cycles de coloration et que de grandes quantités de solutions doivent être préparées, il est préférable de tester d’abord ces réactifs dans un cycle IHC monoplexe avant de passer à l’IHC multiplex. L’IHC Monoplex est vérifié à l’aide de l’imageur de pathologie numérique pour les modèles de coloration attendus et les temps d’exposition sont réglés avec les filtres correspondants sur les lames de contrôle (Figure 3A-H). Le tissu amygdalien est utilisé comme contrôle positif pour la plupart des marqueurs de cellules immunitaires. Comme le temps d’exposition au DAPI dans les tissus de contrôle des amygdales est toujours plus élevé que dans d’autres tissus (Figure 3G), le temps d’exposition au DAPI doit être fixé au type de tissu à étudier. Les temps d’exposition réguliers avec ce type de balayage sont compris entre 1 ms et 30 ms, selon le fluorophore et le filtre (Figure 3I). Lorsqu’un monoplexe IHC dépasse ces chiffres ou que le motif de coloration n’est pas aussi clair que prévu, la solution d’anticorps doit être ajustée ou remplacée. Dans l’exemple présenté ici, nous avons décidé d’augmenter la concentration de FOXP3 (Figure 3C et Figure 3I) pour avoir une intensité plus proche de celle des autres marqueurs. L’autofluorescence peut également être plus forte dans d’autres tissus que dans les tissus de contrôle des amygdales. Dans notre réglage, le temps d’exposition du filtre AF de l’échantillon est compris entre 25 ms et 50 ms (Figure 3H,I).

Figure 3 : Réglage des temps d’exposition sur des échantillons de contrôle monoplex IHC et non colorés. (A) CD20 - Signal Opal 480 dans le tissu témoin des amygdales. (B) Signal CD3 - Opal 520 dans le tissu de contrôle des amygdales. (C) FOXP3 - Signal Opal 570 dans le tissu de contrôle des amygdales. (D) CD56 - Signal de l’opale 620 dans le tissu de contrôle des amygdales (E) CD8 - Signal de l’opale 690 dans le tissu de contrôle des amygdales. (F) Marqueur tumoral - Signal Opal 780 dans le tissu de contrôle des amygdales. (G) Le signal DAPI dans le tissu de contrôle des amygdales est souvent plus faible que le type de tissu d’intérêt. (H) Autofluorescence - échantillon de signal AF dans le tissu de contrôle de la tumeur. (I) Capture d’écran des temps d’exposition avant de l’ajuster à 10% et de vérifier sur des lames multiplex colorées IHC. Barres d’échelle = 100 μm. Abréviations : AF = autofluorescence ; IHC = immunohistochimie ; DAPI = 4'6-diamidino-2-phénylindol. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Après l’exécution de l’IHC multiplex, les temps d’exposition sont ajustés à partir des paramètres IHC monoplex en vérifiant quelques lames IHC multiplex et en sélectionnant l’exposition automatique. Avec ce type de numérisation, il n’y a pas d’option de protection contre la saturation et, par conséquent, il est extrêmement important d’éviter de régler l’exposition trop élevée, évitant ainsi la surexposition. La surexposition entrave le démélange spectral des fluorophores. Souvent, nous ne fixons pas les temps d’exposition dépassant les temps d’exposition qui étaient basés sur l’IHC monoplexe et nous ne diminuons les temps d’exposition que pour les marqueurs qui sont plus forts dans l’IHC multiplex (Figure 3G et Figure 4A). En s’exposant automatiquement à différents endroits sur quelques diapositives, on peut observer que les temps de pose de quelques filtres sont encore trop élevés. Ceux-ci doivent être ajustés au nombre le plus bas observé lors de l’utilisation du réglage d’exposition automatique et soustraire 10 % supplémentaires de la valeur pour éviter une surexposition dans d’autres endroits invisibles (Figure 4A). Avec cette méthode, les temps d’exposition peuvent être plus courts pour certains filtres que ceux qui ont été réglés sur l’IHC monoplexe. Cependant, avec une expérience IHC multiplex réussie, tous les marqueurs devraient être observables, au moins sur la lame de contrôle (Figure 4B-H, Fichier supplémentaire 6 : Figure supplémentaire S1 et Figure supplémentaire S2). Considérez que certains marqueurs peuvent ne pas être présents dans tous les échantillons. En incluant une glissière de contrôle contenant au moins une section d’amygdale, il est possible de vérifier la coloration réussie de tous les marqueurs des panneaux standard et l’intensité du signal.
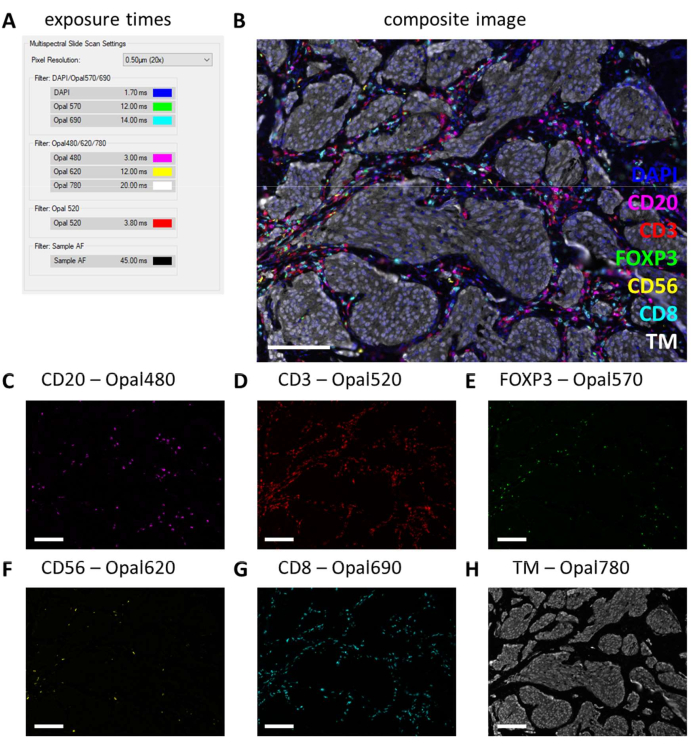
Figure 4 : Exemple d’une coupe colorée avec succès avec le panel de lymphocytes dans un échantillon de tumeur de mélanome. (A) Temps d’exposition utilisés pour enregistrer cet échantillon IHC multiplex. (B) Image composite d’un panel de lymphocytes IHC multiplex dans le tissu tumoral. (C) CD20 - Signal Opal 480 en magenta. (D) CD3 - Signal Opal 520 en rouge. (E) FOXP3 - Signal Opal 570 en vert. (F) CD56 - Signal Opal 620 en jaune. (G) CD8 - Signal Opal 690 en cyan. (H) TM - Opal 780 en blanc. Barres d’échelle = 100 μm. Abréviation : TM = marqueur tumoral ; IHC = immunohistochimie. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les lames IHC multiplex sont entièrement numérisées par l’imageur numérique. Les tuiles pour l’analyse ultérieure sont sélectionnées dans le visualiseur de diapositives. Cependant, lorsque des régions plus spécifiques doivent être analysées, telles que la tumeur par rapport à l’IM, ces régions d’intérêt (ROI) peuvent être dessinées à l’aide de QuPath. Une fois le traitement par lots des tuiles sélectionnées dans le visualiseur de diapositives terminé, les fichiers de composant sont fusionnés à nouveau (Figure 5A et Fichier supplémentaire 7). À l’aide du canal marqueur de tumeur (Figure 5B) et de l’outil de baguette magique dans QuPath, le contour de la tumeur peut être tracé pour former le « ROI de la tumeur » (Figure 5C). Ensuite, le ROI de la tumeur peut être étendu à une certaine distance, dans ce cas, 500 μm, pour créer un « ROI de marge invasive » (Figure 5D). Tout arrière-plan indésirable (non tissulaire) est supprimé de ce retour sur investissement à l’aide de l’outil de la baguette magique en regardant le signal d’autofluorescence (Figure 5E). Le ROI de la tumeur et le ROI IM sont enregistrés sous forme de fichier GeoJSON pour un traitement ultérieur (Figure 5F).
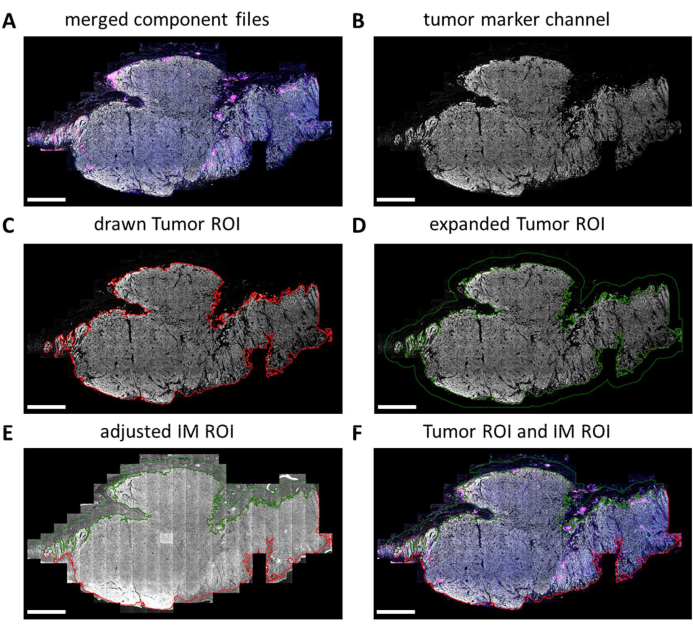
Figure 5 : Processus de dessin du retour sur investissement de la tumeur et de la marge invasive dans QuPath. (A) Fichiers de composants fusionnés. (B) Image en niveaux de gris montrant uniquement le canal marqueur de la tumeur. (C) Le retour sur investissement de la tumeur est dessiné autour du signal du marqueur tumoral. (D) Un nouveau retour sur investissement est obtenu en augmentant le retour sur investissement de la tumeur de 100 à 500 μm pour former le retour sur investissement IM. (E) Le retour d’intérêt IM est ajusté pour n’inclure que le tissu stromal en excluant le bruit de fond (signal négatif) et d’autres grandes structures tissulaires telles que la graisse, les vaisseaux sanguins et les follicules pileux. (F) Le retour sur investissement de la tumeur et le retour sur investissement de la tumeur qui en résultent sont enregistrés et exportés dans des fichiers GeoJSON pour un traitement ultérieur des régions. Le retour sur investissement de la tumeur est affiché avec un contour rouge et le retour sur investissement de la tumeur avec un contour vert. Barres d’échelle = 2 mm. Abréviations : ROI = région d’intérêt ; IM = marge envahissante ; GeoJSON = Notation d’objet JavaScript géographique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les réseaux ImmuNet peuvent être utilisés pour détecter les cellules immunitaires. Pour le panel de lymphocytes, l’image composite expérimentale (Figure 6A) peut être comparée visuellement avec les cellules immunitaires détectées par le logiciel (Figure 6B). Des comparaisons visuelles similaires peuvent être faites pour le panel myéloïde (Fichier supplémentaire 6 : Figure supplémentaire S4) et le panel de cellules dendritiques (Fichier supplémentaire 6 : Figure supplémentaire S5).
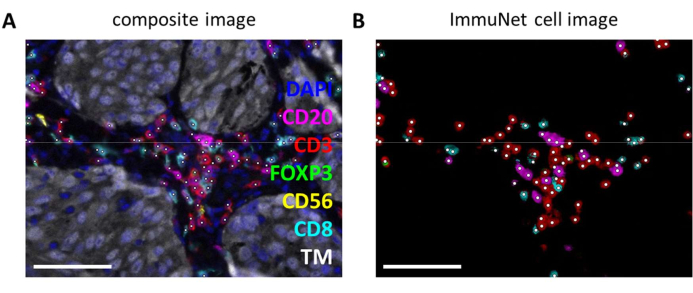
Figure 6 : Lymphocytes reconnus par ImmuNet. (A) Image composite de la figure 4B montrant les cellules reconnues par ImmuNet avec des points blancs. (B) Cellules reconnues par ImmuNet et expression ultérieure du marqueur détecté. Barres d’échelle = 50 μm. Abréviation : TM = marqueur tumoral. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les cellules immunitaires détectées par ImmuNet et enregistrées dans .csv format peuvent être importées dans n’importe quel langage de programmation pour une analyse plus approfondie. Nous avons effectué une visualisation spatiale et un gating dans R (Fichier supplémentaire 8). Les cellules détectées peuvent ensuite être visualisées spatialement (Figure 7A, Fichier supplémentaire 6 : Figure supplémentaire S6 et Figure supplémentaire S7). Un contrôle de l’expression des pseudomarqueurs peut être effectué pour phénotyper les cellules immunitaires individuelles (Figure 7B).
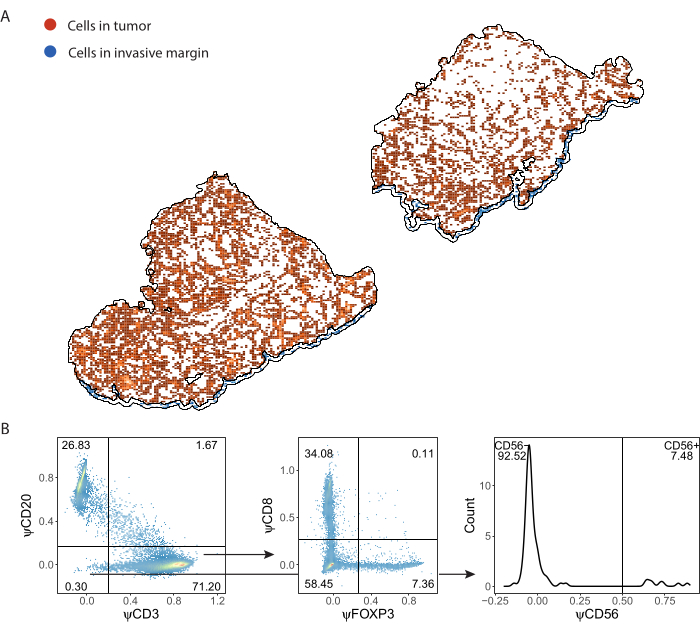
Figure 7 : Stratégie de déclenchement d’un panel de lymphocytes. (A) Cellules immunitaires détectées dans les régions d’intérêt de la tumeur et de la marge invasive délimitées avec QuPath. (B) Gating de toutes les cellules détectées par ImmuNet à partir de la partie A. Les lymphocytes sont d’abord bloqués sur les lymphocytes B CD20+ et les lymphocytes T CD3+. Les lymphocytes T CD3+ sont en outre bloqués pour l’expression de CD8 et FOXP3. La population CD20-CD3- est contrôlée pour les cellules tueuses naturelles CD56+. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Lorsque les phénotypes des cellules prédites sont déterminés à l’aide d’un gating, les densités cellulaires de différents phénotypes peuvent être calculées dans différents ROI. Ceci est calculé en divisant le nombre total de cellules par phénotype par la surface du ROI (tableau 1, figure 8 et fichier supplémentaire 8). Ici, les lymphocytes B sont définis comme CD3-CD20+, les lymphocytes T auxiliaires comme CD3+CD20-CD8-FoxP3-, les lymphocytes T régulateurs comme CD3+CD20-CD8-FoxP3+, les lymphocytes T cytotoxiques comme CD3+CD20-CD8+FoxP3-, et les lymphocytes NK comme CD3-CD20-CD56+.
| Phénotype | Densité dans la tumeur (cellules/mm2) | Masse volumique en IM (cellules/mm2) |
| Lymphocytes B | 185.74 | 145.62 |
| Lymphocyte T auxiliaire | 301.46 | 157.51 |
| Lymphocytes T régulateurs | 38.53 | 19.53 |
| Lymphocytes T cytotoxiques | 185.35 | 83.21 |
| Cellule NK | 0.18 | 0 |
Tableau 1 : Densités des phénotypes dans les ROI. Densités de cellules de différents phénotypes trouvées dans un seul échantillon de mélanome coloré avec le panel de lymphocytes. Les densités sont calculées séparément dans les ROI tumoral et IM. Abréviations : IM = marge envahissante ; ROI = région d’intérêt.
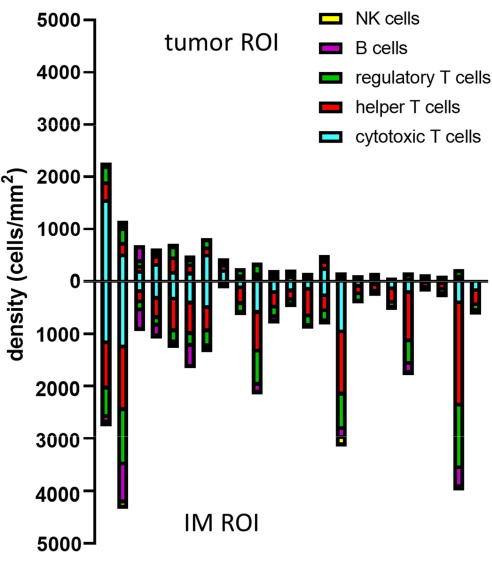
Figure 8 : Exemple d’analyse de données pour plusieurs échantillons. Analyse de la densité de différents phénotypes lymphocytaires dans la tumeur et IM de 23 tumeurs primaires de mélanome. Abréviations : IM = marge envahissante ; ROI = région d’intérêt. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Pour approfondir l’information spatiale de ces cellules immunitaires, il est également possible de déterminer les distances entre les phénotypes identifiés ou les pourcentages de phénotypes des plus proches voisins dans un échantillon (Figure 9).
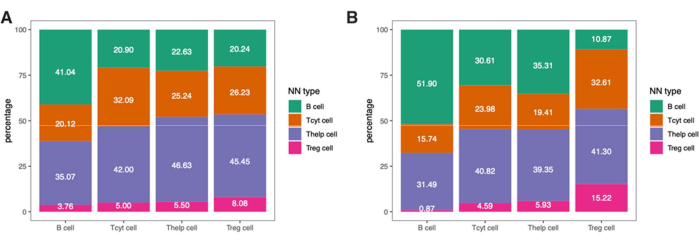
Figure 9 : Exemple d’analyse du voisin le plus proche pour un seul échantillon. Pourcentage de phénotypes du voisin le plus proche pour différents types de cellules dans (A) la tumeur et (B) les ROI IM trouvés dans un seul échantillon de mélanome coloré avec le panel de lymphocytes. Abréviations : IM = marge envahissante ; ROI = région d’intérêt. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Fichier supplémentaire 1 : IHC multiplex résumant les spécifications du protocole. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 2 : Protocole Autostainer pour monoplex. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 3 : Protocole d’autocoloration pour la compensation d’autofluorescence. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 4 : Protocole d’autocoloration pour l’immunohistochimie multiplex. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 5 : Modèle .csv fichier. Veuillez cliquer ici pour télécharger ce fichier.
Dossier supplémentaire 6 : Panels de cellules myéloïdes et dendritiques dans un échantillon de tissu de mélanome ; marquage des lames en cas d’échec du balayage ; cellules myéloïdes et dendritiques reconnues par ImmuNet ; stratégies de contrôle des panels de cellules myéloïdes et dendritiques. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 7 : script de point QuPath. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 8 : Script d’analyse de données. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
L’analyse spatiale de l’EUT est une technique recherchée pour en savoir plus sur le compartiment cellulaire immunitaire et découvrir de nouveaux biomarqueurs pronostiques et prédictifs, notamment dans le domaine de l’immuno-oncologie16. De nombreuses techniques différentes sont en cours de développement à cet effet, impliquant la détection de protéines, de transcrits d’ARNm ou d’une combinaison des deux, avec des estimations allant jusqu’à 100 à 1 000 cibles. Cependant, un multiplexage plus élevé se fait au prix d’une réduction du nombre d’expériences à haut débit, de coûts expérimentaux plus élevés et de défis techniques, et souvent, seule une petite partie du TME peut être analysée. L’IHC multiplexe utilisant la méthode basée sur la TSA que nous décrivons ici, détecte simultanément six marqueurs différents + DAPI, est relativement moins coûteux à réaliser et des coupes de tissus entiers sont imagées en moins de 20 minutes, prêtes à être entièrement analysées. Cette technique est devenue moins complexe avec l’automatisation de la procédure de coloration. Les améliorations apportées au microscope multispectral, qui comprennent l’ajout de deux filtres supplémentaires, ont considérablement amélioré les temps de démélange et de balayage spectrals. Il est possible de détecter jusqu’à huit marqueurs différents + DAPI simultanément. Cependant, en étendant le multiplexage avec plus de marqueurs, les avantages susmentionnés disparaissent à mesure que le démixage spectral devient plus difficile et que les temps de balayage de lames entières augmentent considérablement. Des efforts sont entrepris pour normaliser les CSI multiplexes entre les différents établissements afin de faciliter la mise en œuvre dans le cadre du diagnostic. Pour cette standardisation de l’IHC multiplex, nous conseillons aux utilisateurs d’adhérer au protocole plus accessible avec six marqueurs différents + DAPI. Néanmoins, un certain savoir-faire technique est encore nécessaire et l’analyse en aval peut être difficile, pour laquelle nous avons développé des méthodologies qui sont décrites dans ce protocole.
La normalisation commence par le développement de panels IHC multiplex. L’importance du choix des anticorps primaires détectant des cibles protéiques particulières a été soulignée avant17. Nos panels IHC multiplex sont principalement développés avec des clones d’anticorps primaires qui sont également utilisés et validés pour l’IHC dans notre service de diagnostic. Cependant, dans le cas du panel IHC multiplexe de cellules dendritiques, la plupart des anticorps n’ont pas été utilisés dans le cadre du diagnostic (van der Hoorn et al., manuscrit en cours de soumission). Pour garantir la spécificité et minimiser les différences de lot, nous avons choisi d’utiliser des anticorps monoclonaux plutôt que des anticorps polyclonaux et avons également validé la plupart des anticorps en utilisant des lignées cellulaires transfectées et des cellules primaires. Au fil des ans, différentes versions de panels IHC multiplexes ont été utilisées dans de nombreuses études utilisant le système Vectra 3 18,21,23,24,25,26,27,28,29,30,31,32. Pour mettre en œuvre ces panels IHC multiplex de manière optimale sur le système PhenoImager HT, certains ajustements ont dû être effectués dans les combinaisons d’anticorps primaires et de fluorophores. Pour bénéficier d’un meilleur démélange spectral et de temps de balayage plus rapides de coupes de tissus entiers, il est nécessaire de mettre en œuvre les derniers fluorophores Opal480 et Opal780 et d’éviter l’utilisation de fluorophores Opal540 et Opal650 dans des panels IHC multiplexes à sept couleurs. Les temps de balayage sont ~3 à 10 fois plus rapides selon la taille de la section de tissu. Les ajustements de panel IHC multiplex ont été assez faciles à réaliser, mais certaines considérations doivent être gardées à l’esprit. Le spectre fluorescent de l’Opal480 chevauche beaucoup le spectre de l’autofluorescence et, par conséquent, interfère avec le démélange spectral des érythrocytes et d’autres structures autofluorescentes. L’utilisation d’une concentration accrue de l’anticorps primaire associé à Opal480 a résolu ce problème dans la plupart des cas. La mise en œuvre du filtre propriétaire Sample AF sur le PhenoImager HT facilite le démélange de l’Opal480 et de l’autofluorescence. Cependant, il est préférable d’utiliser un anticorps primaire qui donne un signal clair lorsqu’il est utilisé avec Opal480 afin que son signal soit supérieur à l’autofluorescence.
Même si ces panels IHC multiplex sont établis, la variation d’un lot à l’autre est quelque chose qui doit être pris en compte. En effectuant des contrôles IHC monoplexes avant de commencer l’expérience IHC multiplex complète, nous avons parfois observé que les anticorps primaires se comportent plus ou moins bien d’une expérience à l’autre. Les raisons peuvent être des erreurs de pipetage, des conditions de stockage des réactifs non optimales et une durée de conservation. Nous avons résolu ce problème en ajustant la solution d’anticorps primaires sur la base de notre expérience. Même lorsqu’aucun des ajustements susmentionnés n’a dû être effectué, pour chaque expérience de lot multiplex IHC, il est important de définir les temps d’exposition en fonction des lames de contrôle monoplex colorées à l’IHC.
Étant donné que notre recherche s’est initialement concentrée sur différents types de carcinomes et de mélanomes, les panels IHC multiplex devaient être interchangeables entre les types de tumeurs avec un minimum d’ajustements. Par conséquent, nous avons toujours inclus plusieurs types de tissus (tumoraux) dans le processus d’optimisation et observé que les dilutions des anticorps primaires pour les marqueurs de cellules immunitaires peuvent être maintenues similaires entre différents types de tumeurs. Cependant, la détection du tissu tumoral entre les carcinomes et le mélanome nécessite des marqueurs tumoraux différents. En conséquence, le marqueur tumoral a toujours été optimisé pour fonctionner à la fin de chaque panel IHC multiplex et est actuellement toujours utilisé en conjonction avec Opal780, qui, par coïncidence, doit également être au dernier fluorophore dans une procédure de coloration IHC multiplex. En utilisant le marqueur tumoral par conséquent à la fin de l’IHC multiplex, ces panels IHC multiplex peuvent être facilement échangés contre d’autres types de tumeurs, tels que le glioblastome (c’est-à-dire GFAP) et le lymphome hodgkinien (c’est-à-dire CD30). Pour l’angiosarcome, nous avons utilisé ce panel IHC multiplexe de lymphocytes avec le gène lié à la transformation de l’érythroblaste (ERG) comme marqueur tumoral avec seulement deux expériences d’optimisation25. L’optimisation comprenait le titrage de l’anticorps primaire ERG et le test du panel IHC multiplex avec ERG à la fin.
D’autres ajustements à ces panels IHC multiplex peuvent également être effectués en échangeant un certain marqueur de cellules immunitaires contre un autre marqueur immunitaire ou fonctionnel. Chaque changement nécessite une optimisation. Le protocole d’optimisation pourrait être suivi comme décrit précédemment17. Certaines modifications apportées aux panneaux multiplexes IHC proposés interféreront avec les algorithmes ImmuNet que nous avons créés. Il faut générer suffisamment de données et consacrer du temps à la mise en œuvre de ces modifications dans l’algorithme (au moins 750 annotations pour chaque nouveau marqueur et/ou phénotype cellulaire, et 150 annotations pour la validation des marqueurs précédemment formés). Les panels présentés ici ne contiennent pas de marqueurs fonctionnels, bien que l’implémentation de marqueurs de point de contrôle immunitaire tels que-1 et-L1 dans des panels IHC multiplex soit réalisée dans notre laboratoire. Cependant, l’analyse de marqueurs moins binaires dans les signaux négatifs et positifs s’est avérée plus difficile et constitue un domaine de recherche actif dans notre groupe.
Le nombre de marqueurs pouvant être évalués simultanément avec l’IHC multiplex est limité par rapport à d’autres nouvelles techniques. Bien que cela puisse être contourné en analysant différents panneaux sur des tranches consécutives d’un bloc FFPE, il sera difficile de comparer ces tranches spatialement. L’orientation et les artefacts pliés ne sont probablement pas les mêmes après la préparation des diapositives. Néanmoins, l’IHC multiplex est assez accessible, ce qui en fait un outil attrayant pour davantage d’institutions et de chercheurs et donc, plus adapté à une mise en œuvre future dans un cadre de diagnostic. Avec la normalisation des panels de cellules immunitaires IHC multiplex pour plusieurs types de tumeurs et les pipelines d’analyse en aval, davantage de connaissances pourraient être acquises sur les différences de TME entre les patients et les types de tumeurs. Cela peut, par exemple, conduire à plus de connaissances sur le rôle de l’EUT dans la réponse antitumorale à des traitements spécifiques. Cela pourrait même donner naissance à de nouveaux biomarqueurs pour prédire des facteurs tels que la réponse au traitement et la survie attendue. Dans l’ensemble, cela peut permettre à l’IHC multiplex de devenir un outil clinique d’aide à la prise de décision clinique, dans une approche de médecine personnalisée. Certes, davantage d’étapes de la procédure d’analyse devraient probablement être automatisées et standardisées pour qu’elle puisse être utilisée dans un cadre de diagnostic quotidien, de sorte qu’il s’agit pour l’instant d’une perspective principalement futuriste.
L’analyse de plusieurs marqueurs sur une seule lame d’échantillon peut être un outil très puissant malgré ses défis techniques. Avec des protocoles expérimentaux standardisés et une méthode d’analyse robuste, comme nous l’avons décrit ici à l’aide d’ImmuNet, la quantification de plusieurs marqueurs la rend plus informative que l’IHC classique, tandis que l’IHC multiplex reste relativement à haut débit par rapport aux nouvelles méthodes expérimentales à haut plex.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Le PhenoImager HT a été acheté grâce à un financement fourni par le Centre médical de l’Université Radboud et le Centre technologique Radboud pour la microscopie. La mucoviscidose est soutenue financièrement par une bourse de la Société néerlandaise du cancer (10673) et une bourse ERC Adv ARTimmune (834618). JT est soutenu financièrement par une subvention de NWO Vidi (VI.Vidi.192.084). Les auteurs tiennent à remercier Eric van Dinther et Ankur Ankan pour leur aide dans la création de flux de travail pour stocker des données IHC multiplexes et Bengt Phung est remercié pour ses instructions sur la façon d’implémenter des données IHC multiplex dans QuPath pour le dessin du retour sur investissement.
matériels
| Name | Company | Catalog Number | Comments |
| anti-CD14 | Cell Marque | 114R-16 | section 3, clone EPR3653 |
| anti-CD163 | Cell Marque | 163M-15 | section 3, clone MRQ-26 |
| anti-CD19 | Abcam | ab134114 | section 3, clone EPR5906 |
| anti-CD1c (BDCA1) | Thermo Scientific | TA505411 | section 3, clone OTI2F4 |
| anti-CD20 | Thermo Scientific | MS-340-S | section 3, clone L26 |
| anti-CD3 | Thermo Scientific | RM-9107 | section 3, clone sp7 |
| anti-CD303/BDCA2 | Dendritics via Enzo Lifesciences/Axxora | DDX0043 | section 3, clone 124B3.13 |
| anti-CD56 | Cell Marque | 156R-94 | section 3, clone MRQ-42 |
| anti-CD66b | BD Biosciences | 555723 | section 3, clone G10F5 |
| anti-CD68 | Dako Agilent | M087601 | section 3, clone PG-M1 |
| anti-CD8 | Dako Agilent | M7103 | section 3, clone C8/144B |
| anti-Foxp3 | Thermo Scientific | 14-4777 | section 3, clone 236A/E7 |
| anti-Gp100 | Dako Agilent | M063401 | section 3, clone HMB45 |
| anti-HLA-DR, DP, DQ | Santa Cruz | sc-53302 | section 3, clone CR3/43 |
| anti-MART-1 | Thermo Scientific | MS-799 | section 3, clone A103 |
| anti-pan cytokeratin | Abcam | ab86734 | section 3, clone AE1/AE3 + 5D3 |
| anti-SOX10 | Sigma Aldrich | 383R | section 3, clone EP268 |
| anti-Tyrosinase | Sanbio | MONX10591 | section 3, clone T311 |
| anti-XCR1 | Cell Signaling Technologies via Bioké | 44665S | section 3, clone D2F8T |
| antibody diluent | Akoya BioSciences | SKU ARD1001EA | section 3, from Opal 7-Color Automation IHC Kit 50 slide (can optionally also be replaced by TBST with 10% BSA) |
| Bond Aspirating Probe | Leica Biosciences | S21.0605 | section 3 |
| Bond Aspirating Probe Cleaning | Leica Biosciences | CS9100 | section 3 |
| Bond Dewax Solution | Leica Biosciences | AR9222 | section 3 |
| Bond Objectglas label + print lint | Leica Biosciences | S21.4564.A | section 3 |
| Bond Research Detection System 2 | Leica Biosciences | DS9777 | section 3 |
| Bond RX autostainer | Leica Biosciences | - | section 3, automated platform |
| Bond TM Epitope Retrieval 1 - 1 L | Leica Biosciences | AR9961 | section 3 |
| Bond TM Epitope Retrieval 2 - 1 L | Leica Biosciences | AR9640 | section 3 |
| Bond TM Wash Solution 10x - 1 L | Leica Biosciences | AR9590 | section 3 |
| BOND Universal Covertile | Leica Biosciences | S21.4611 | section 3 |
| Bond(TM) Titration Kit | Leica Biosciences | OPT9049 | section 3 |
| Coverslip 24 x 32 mm #1 (0.13-0.16 mm) | Fisher Scientific | 15717592 | section 2 |
| coverslip 24 x 50 mm | VWR | 631-0146 | section 2 |
| DAPI Fluoromount-G | VWR | 0100-20 | section 3, whenever monoplex slides need to be quickly checked, not for official analysis, then DAPI is stained seperately for better results |
| Eosine | section 2, home made | ||
| Ethanol 99.5% | VWR | 4099.9005 | section 2 |
| Fluoromount-G | VWR | 0100-01 | section 3 |
| haematoxyline | - | section 2, home made | |
| ImmuNet | - | immune cell detection and phenotyping pipeline | |
| inForm software 2.4.10 | Akoya BioSciences | - | section 4 & 6 |
| OPAL 480 reagent pack | Akoya BioSciences | FP1500001KT | section 3 |
| OPAL 520 reagent pack | Akoya BioSciences | FP1487001KT | section 3 |
| OPAL 570 reagent pack | Akoya BioSciences | FP1488001KT | section 3 |
| OPAL 620 reagent pack | Akoya BioSciences | FP1495001KT | section 3 |
| OPAL 690 reagent pack | Akoya BioSciences | FP1497001KT | section 3 |
| OPAL 780 reagent pack | Akoya BioSciences | FP1501001KT | section 3 |
| Opal 7-Color Automation IHC Kit 50 slide | Akoya BioSciences | NEL821001KT | section 3 |
| PhenoChart 1.1.0 | Akoya BioSciences | - | section 5 |
| PhenoImagerHT | Akoya BioSciences | CLS143455 | section 4, digital pathology imager with slide viewer and imaging software (formerly known as Vectra Polaris) |
| Quick-D mounting medium | Klinipath | 7280 | section 2 |
| QuPath 0.3.2 | whole slide image analysis software platform | ||
| R 4.1.1 | |||
| Slide boxes | VWR | 631-0737 | section 1 |
| SuperFrost Plus | Thermo Scientific through VWR | 631-9483 | section 1 |
| Vectra Polaris software 1.0.13 | Akoya BioSciences | - | section 4 |
| Xylene | VWR | 4055-9005 | section 2 |
Références
- Dunn, G. P., Bruce, A. T., Ikeda, H., Old, L. J., Schreiber, R. D. Cancer immunoediting: from immunosurveillance to tumor escape. Nature Immunology. 3 (11), 991-998 (2002).
- van der Woude, L. L., Gorris, M. A. J., Halilovic, A., Figdor, C. G., de Vries, I. J. M. Migrating into the tumor: a roadmap for T cells. Trends in Cancer. 3 (11), 797-808 (2017).
- Fridman, W. H., Pages, F., Sautes-Fridman, C., Galon, J. The immune contexture in human tumours: impact on clinical outcome. Nature Reviews. Cancer. 12 (4), 298-306 (2012).
- Fridman, W. H., et al. The immune microenvironment of human tumors: general significance and clinical impact. Cancer Microenvironment. 6 (2), 117-122 (2013).
- Pages, F., et al. International validation of the consensus Immunoscore for the classification of colon cancer: a prognostic and accuracy study. Lancet. 391 (10135), 2128-2139 (2018).
- Angell, H. K., Bruni, D., Barrett, J. C., Herbst, R., Galon, J. The Immunoscore: colon cancer and beyond. Clinical Cancer Research. 26 (2), 332-339 (2020).
- Angell, H., Galon, J. From the immune contexture to the Immunoscore: the role of prognostic and predictive immune markers in cancer. Current Opinion in Immunology. 25 (2), 261-267 (2013).
- Galon, J., et al. Cancer classification using the Immunoscore: a worldwide task force. Journal of Translational Medicine. 10, 205 (2012).
- Galon, J., et al. World-wide Immunoscore Task Force: meeting report from the 34;Melanoma Bridge", Napoli, November 30th-December 3rd, 2016. Journal of Translational Medicine. 15 (1), 212 (2017).
- Tumeh, P. C., et al. PD-1 blockade induces responses by inhibiting adaptive immune resistance. Nature. 515 (7528), 568-571 (2014).
- Creemers, J. H. A., et al. A tipping point in cancer-immune dynamics leads to divergent immunotherapy responses and hampers biomarker discovery. Journal for Immunotherapy of Cancer. 9 (5), e002032 (2021).
- Blank, C. U., Haanen, J. B., Ribas, A., Schumacher, T. N. CANCER IMMUNOLOGY. The "cancer immunogram". Science. 352 (6286), 658-660 (2016).
- Teruya-Feldstein, J. The immunohistochemistry laboratory looking at molecules and preparing for tomorrow. Archives of Pathology & Laboratory Medicine. 134 (11), 1659-1665 (2010).
- Ramos-Vara, J. A., Miller, M. A., et al. When tissue antigens and antibodies get along: revisiting the technical aspects of immunohistochemistry--the red, brown, and blue technique. Veterinary Pathology. 51 (1), 42-87 (2014).
- Hegde, P. S., Karanikas, V., Evers, S. The where, the when, and the how of immune monitoring for cancer immunotherapies in the era of checkpoint inhibition. Clinical Cancer Research. 22 (8), 1865-1874 (2016).
- Parra, E. Novel platforms of multiplexed immunofluorescence for study of paraffin tumor tissues. Journal of Cancer Treatment and Diagnosis. 2 (1), 43-53 (2018).
- Gorris, M. A. J., et al. Eight-color multiplex immunohistochemistry for simultaneous detection of multiple immune checkpoint molecules within the tumor microenvironment. Journal of Immunology. 200 (1), 347-354 (2018).
- Roelofsen, T., et al. Spontaneous regression of ovarian carcinoma after septic peritonitis; a unique case report. Frontiers in Oncology. 8, 562 (2018).
- van den Brand, D., et al. Peptide-mediated delivery of therapeutic mRNA in ovarian cancer. European Journal of Pharmaceutics and Biopharmaceutics. 141, 180-190 (2019).
- van den Brand, D., et al. EpCAM-binding DARPins for targeted photodynamic therapy of ovarian cancer. Cancers. 12 (7), 1762 (2020).
- Di Blasio, S., et al. The tumour microenvironment shapes dendritic cell plasticity in a human organotypic melanoma culture. Nature Communications. 11 (1), 2749 (2020).
- van Beek, J. J. P., et al. Human pDCs are superior to cDC2s in attracting cytolytic lymphocytes in melanoma patients receiving DC vaccination. Cell Reports. 30 (4), 1027-1038 (2020).
- Rodriguez-Rosales, Y. A., et al. Immunomodulatory aged neutrophils are augmented in blood and skin of psoriasis patients. Journal of Allergy and Clinical Immunology. 148 (4), 1030-1040 (2021).
- Hoeijmakers, Y. M., et al. Immune cell composition in the endometrium of patients with a complete molar pregnancy: Effects on outcome. Gynecologic Oncology. 160 (2), 450-456 (2021).
- van Ravensteijn, S. G., et al. Immunological and genomic analysis reveals clinically relevant distinctions between angiosarcoma subgroups. Cancers. 14 (23), 5938 (2022).
- van der Woude, L. L., et al. Tumor microenvironment shows an immunological abscopal effect in patients with NSCLC treated with pembrolizumab-radiotherapy combination. Journal for Immunotherapy of Cancer. 10 (10), e005248 (2022).
- Graham Martinez, C., et al. The immune microenvironment landscape shows treatment-specific differences in rectal cancer patients. Frontiers in Immunology. 13, 1011498 (2022).
- Cortenbach, K. R. G., et al. Topography of immune cell infiltration in different stages of coronary atherosclerosis revealed by multiplex immunohistochemistry. International Journal of Cardiology. Heart & Vasculature. 44, 101111 (2023).
- van Wilpe, S., et al. Homologous recombination repair deficient prostate cancer represents an immunologically distinct subtype. Oncoimmunology. 11 (1), 2094133 (2022).
- Gorris, M. A. J., et al. Paired primary and metastatic lesions of patients with ipilimumab-treated melanoma: high variation in lymphocyte infiltration and HLA-ABC expression whereas tumor mutational load is similar and correlates with clinical outcome. Journal for Immunotherapy of Cancer. 10 (5), e004329 (2022).
- van Wilpe, S., et al. Intratumoral T cell depletion following neoadjuvant chemotherapy in patients with muscle-invasive bladder cancer is associated with poor clinical outcome. Cancer Immunology, Immunotherapy. 72 (1), 137-149 (2023).
- Sultan, S., Gorris, M. A. J., Buytenhuijs, F., Lvan de Woude, L. A Segmentation-free machine learning architecture for immune land-scape phenotyping in solid tumors by multichannel imaging. bioRxiv. , (2021).
- Parra, E. R., et al. Immuno-profiling and cellular spatial analysis using five immune oncology multiplex immunofluorescence panels for paraffin tumor tissue. Scientific Reports. 11 (1), 8511 (2021).
- Parra, E. R., et al. Validation of multiplex immunofluorescence panels using multispectral microscopy for immune-profiling of formalin-fixed and paraffin-embedded human tumor tissues. Scientific Reports. 7 (1), 13380 (2017).
- Sun, Z., Nyberg, R., Wu, Y., Bernard, B., Redmond, W. L. Developing an enhanced 7-color multiplex IHC protocol to dissect immune infiltration in human cancers. PLoS One. 16 (2), e0247238 (2021).
- LeicaBiosystems. BOND RX Fully Automated Research Stainer Protocols. , (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon