Method Article
オープンソースの液滴マイクロ流体工学によるプラスミド安定性解析
要約
細菌におけるプラスミド保持の並列分析のために、アクセス可能なオープンソースのマイクロ流体ワークフローが紹介されています。蛍光顕微鏡法を用いてゲル微小液滴内の単一細胞微小コロニーにおけるプラスミドの存在を定量することにより、この方法は、従来のプレートカウントに代わる、正確でアクセスしやすく、スケーラブルな方法を提供します。
要約
プラスミドは、さまざまな生物への外来遺伝子の導入と発現を可能にし、細胞集団内および細胞集団間の生物学的回路と経路の構築を促進することにより、合成生物学において重要な役割を果たします。多くのアプリケーションでは、抗生物質を選択せずに機能性プラスミドを維持することが非常に重要です。この研究では、ゲルマイクロ液滴中の単一細胞を培養し、蛍光顕微鏡を使用してマイクロコロニーを定量することにより、プラスミド保持を解析するためのオープンハードウェアベースのマイクロ流体ワークフローを紹介します。このアプローチにより、多数の液滴とマイクロコロニーの並行分析が可能になり、従来のプレートカウントと比較してより大きな統計的検出力が得られ、アッセイを他の液滴マイクロ流体ワークフローに統合することができます。蛍光タンパク質を発現するプラスミドを非特異的な蛍光DNA染色剤と併用することで、プラスミドの損失や蛍光マーカーの発現に基づいて単一コロニーを同定し、鑑別することができます。特に、オープンソースのハードウェアで実装されたこの高度なワークフローは、サンプルとマイクロ流体チップの両方の正確なフロー制御と温度管理を提供します。これらの機能により、ワークフローの使いやすさ、堅牢性、アクセシビリティが向上します。この研究では、実験モデルとして 大腸菌 に焦点を当てていますが、この方法の真の可能性は、その汎用性にあります。プラスミドや染色剤からの蛍光シグナル定量を必要とするさまざまな研究や、その他のアプリケーションにも適応できます。オープンソースのハードウェアの採用により、さまざまな研究環境でアクセス可能な技術を使用してハイスループットバイオアナリシスを行う可能性が広がります。
概要
プラスミドは原核細胞の必須遺伝要素であり、DNAの横方向導入と環境変化への迅速な適応を通じて微生物の進化に大きく貢献しています1,2。これらの染色体外DNA分子は、抗生物質耐性、代謝機能、病原性因子などの有利な形質を提供する遺伝子を持っているため、微生物学研究、合成生物学、進化研究に有用です2。しかし、細胞集団におけるプラスミドの維持は、複製と分離の代謝負荷のために困難であり、選択圧なしではプラスミドが失われることがよくあります3。さらに、安定した遺伝には、毒素-抗毒素や分配システムなどのメカニズムが必要であり、プラスミドの維持が複雑になります。さまざまな条件下でのプラスミドの安定性を評価することは、プラスミドを主要な研究要素として利用する基礎研究と実用化の両方にとって重要です4,5。プラスミドの安定性を評価するための現在の方法のほとんどは、大きな限界があります:フローサイトメトリーベースの方法は、間接的な集団レベルのデータを提供し、高価な機器を必要とし、コロニーの直接的な可視化を欠いています6。バルクトランスクリプトミクスおよびプロテオミクス法は高価で、平均的な細胞応答しか得られず、個々のコロニーにおけるプラスミド保持を直接定量化することはできません6。段階希釈や継代などの従来の方法は単純ですが時間がかかり、精度と表現性に欠けています7。全体として、特定の機能的プラスミドを経時的または選択的圧力にわたって保持するコロニーの数を定量的に推定または予測することは依然として困難です。
これらの課題に対処するために、 大腸菌 をモデルとして使用して、細菌の複数の分離されたコロニーの蛍光シグナルを定量化するための、オープンハードウェア研究機器を利用した新しいマイクロ流体ワークフローが提示されます。この方法により、さまざまな条件や選択圧力にわたるプラスミド保持のハイスループットで正確な分析が可能になります。シングルセル分解能解析は、単離されたコロニーを正確に操作する方法を提供し、プラスミド定量に関する高感度データが得られ、保持率と損失率の評価に役立ちます4。
マイクロ流体工学、特に液滴マイクロ流体工学は、制御された環境で個々の細胞をカプセル化および操作するための強力なツールとして浮上しています8。具体的には、マイクロゲル液滴は、油中に懸濁した液滴を維持することなく、ハイスループットで正確な分析のために単一細胞をカプセル化することができ9、定義された微小環境におけるプラスミドダイナミクスの制御された研究を可能にします。ピペットチップ10から直接細胞懸濁液をカプセル化した後、蛍光技術を使用して液滴内のマイクロコロニーの成長をモニターすることができ、異なる選択圧力下でのプラスミドの保持および分離の詳細な分析を可能にする3。
従来のバルク培養技術と比較した場合のこの方法の利点には、精度の向上、ばらつきの低減、ハイスループット分析の実施能力などがあります。オープンソースのマイクロ流体工学技術は、アクセシビリティ、適応性、メンテナンスの問題など、研究の進行を妨げることが多い高価な独自システムの限界を克服します11,12,13。オープンソースの装置を使用して、マイクロゲルにおけるプラスミド保持分析の高度な実験ワークフローを適用する方法を実証することにより、プラスミド生物学、合成生物学アプリケーション、およびマイクロ流体液滴分析技術の研究に利用しやすく信頼性の高い方法が提供されます。
要約すると、この記事では、 大腸菌 のプラスミド保持を高い統計的検出力で定量的に評価するためのアクセス可能な方法を紹介します。この分析法の能力は、プラスミドバイオロジーの理解を深め、合成バイオロジーのアプリケーションを改善するための貴重なツールとなっています。
プロトコル
図1は、 大腸菌 のプラスミドの安定性を評価するための概略図を示しています。試薬および使用した機器の詳細は 、材料表に記載されています。生データと視覚化スクリプトは、https://doi.org/10.17605/OSF.IO/6YWJK で入手できます。
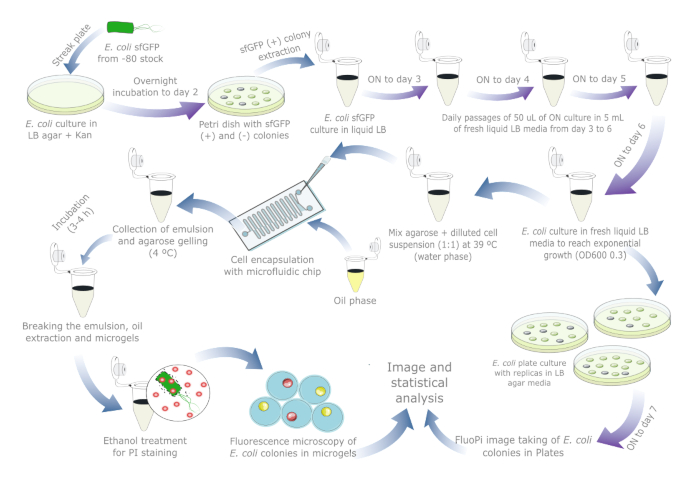
図1:大腸菌のプラスミド安定性を評価するための日々のプロトコル。青い矢印は日中の歩数を示し、紫色の矢印は一晩のインキュベーションを示します。すべての液体および寒天インキュベーションは、別々のネガティブコントロールチューブ/プレートとともに37°Cで行いました。プラスミド損失がすでに発生している可能性のある実際のサンプルには、細胞培養の調製と継代は必要ないため、プレート参照培養が含まれている場合は、プロトコルを1日または2日に短縮する必要があります。この図の拡大版を表示するには、ここをクリックしてください。
1. マイクロ流体チップの調製
注:このプロトコルでは、滴下領域で直径100μm未満の油中水滴を生成できるセルカプセル化に、さまざまな商用またはカスタムチップ設計を使用することができます。この研究では、チップは、以前に公開されたレポート14で報告されたのと同じ設計および製造方法に従って設計および製造されました(記事データ https://doi.org/10.17605/OSF.IO/6YWJK を参照)。
- ゲルマイクロ液滴生成用に設計されたマスターモールドを使用して、PDMS-on-glassマイクロ流体チップを取得または調製します。
- 撥水溶液(フルオロアルキルシラン)をチップに注入し、内側のマイクロチャネルを疎水性にします。溶液をインレットに注入し、すべてのチャネルが液体で満たされていることを確認します。満たされたチャネルを約30〜60秒間休ませます。
- 内部のマイクロチャネルに空気を排出することにより、デバイスから溶液を取り出します。空のエアシリンジを使用して空気を洗い流し、他のポートに吸収性ワイプを使用して飛散を防ぎます。
- 処理したデバイスをホットプレートで65°Cで15分間焼き、余分な溶液を蒸発させます。または、デバイスを冷蔵庫(4°C)に一晩保管します。
注:マイクロ流体チップはすぐに使用できます。ここでプロトコルを一時停止できます。
2. サンプル調製
- 細胞回収
注:液滴中の細胞培養は、プラスミドダイナミクスに関する関連データを提供できます。実験細菌モデルとして、 大腸菌 TOP10株を、スーパーフォルダー緑色蛍光タンパク質(sfGFP)およびカナマイシン耐性15をコードするプラスミドpCA_Odd1(寄託データ https://doi.org/10.17605/OSF.IO/6YWJK を参照)とともに使用する。バクテリア、プラスミド、および増殖培地は、実験システムによって異なります。- 25 gの予混合LBと12 gの寒天を800 mLの蒸留水(dH2O)に溶解して、Luria Bertoni(LB)-寒天を調製します。また、25gの予め混合したLBを800mLのdH2Oに溶解して液体LB培地を調製し、溶液をオートクレーブして約60°Cまで冷却します。 次のステップのために、フローフードなどの無菌環境に移行してください。
注:予め混合されたLB粉末には、10 gのトリプトン、5 gの酵母抽出物、および10 gのNaClが含まれています。自己混合LB寒天培地も使用できます。 - 50 mLの液体LB-Agar(最終濃度:100 μg/mL)に50 μLのカナマイシン(100 mg / mLで調製)を加えます。.チューブを数回反転させて溶液を混合します。
- ペトリ皿(90 mm x 15 mm)あたり約15 mLの液体LB寒天を注ぎます。ネガティブコントロール(コンタミネーションモニタリング)用と実験培養用の2枚のプレートを用意します。LB寒天溶液を冷まし、両方のプレートで色が暗い色から透明に変わるまで固めます。
- 滅菌ループを使用して、ストリークプレート技術を使用して大 腸菌 株を培養プレート上に広げます。使用後すぐにグリセロールストック の大腸菌 を-80°Cに戻します。両方のプレートを閉じ、37°Cで一晩インキュベートします(1日目)。
- 2日目に、汚染のシグナルとしてコロニーのネガティブコントロールプレートを確認します(その場合は、2.1.4の手順を繰り返します)。青色光トランスイルミネーターを使用して大 腸菌 プレート上の単一の蛍光コロニーを同定し、液体培養ストックを調製します。
- 滅菌条件下で、プレートを開き、滅菌ループまたは200 μLピペットチップを使用して選択したコロニーを選択し、5 mLの新鮮液体LB培地と5 μLのカナマイシン(100 mg/mLで調製)を入れた培養チューブに移します。コンタミネーションモニタリング用に、接種していないネガティブコントロールチューブを調製します。培養チューブを37°Cで一晩インキュベートし、220rpmで振とうします。
- 抗生物質を含まない培地でサンプルをさらに3日間通過させ、プラスミドの損失が発生する可能性のある条件をシミュレートします。3日目、4日目、5日目に、50 μLの一晩培養液を、抗生物質を含まない5 mLの液体LBを含む新しい培養チューブに移します。220 rpmで振とうしながら、37°Cで一晩インキュベートします。このステップを6日目まで繰り返し、合計4回の継代後に最終的な5mLの培養物を得る。
- 最終一晩培養液から50 μLを2 mLの液体LBを保持した新しい培養チューブに移し、分光光度計を使用して培養物を光学密度(OD600)0.3(約3〜4時間)に達するまで待ちます。
注:OD600が増加する前に新鮮な培養物を使用して、細菌が対数成長段階にあることを確認する必要があります。目的の濃度に達しない場合は、手順2.1.8から繰り返します。
- 25 gの予混合LBと12 gの寒天を800 mLの蒸留水(dH2O)に溶解して、Luria Bertoni(LB)-寒天を調製します。また、25gの予め混合したLBを800mLのdH2Oに溶解して液体LB培地を調製し、溶液をオートクレーブして約60°Cまで冷却します。 次のステップのために、フローフードなどの無菌環境に移行してください。
- カプセル化用の細胞-アガロースミックス
注:濃度制御は、単一細胞の液滴カプセル化を確保するために不可欠です。必要な細胞濃度は、次の例に示すように、目標カプセル化速度と特定の液滴量に対して計算できます。
液滴量(V液滴):液滴が50μm発生した場合
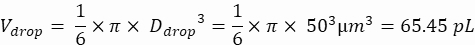
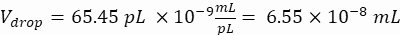
液滴あたりの所望細胞数(Cpd):平均して5液滴につき1細胞(0.2細胞/液滴)
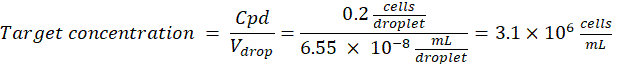
希釈係数:初期細胞濃度はステップ2.1.8で取得します。
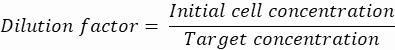
ここでは、50 μmまたは100 μm(液滴分割なし)の液滴(65〜520 pL)が生成され、液滴5個あたり約1個の細胞がカプセル化され、分裂せずに液滴あたり1.6個の細胞が生成されます。 大腸菌については、1 OD600 ユニット≈ 7.8 x 108 cells/mL16 の変換係数を使用します。ステップ 2.1.8 の OD600 値に換算係数を掛けて、培養物の初期濃度 (細胞数/mL) を求めます。- 調製した培養液から 大腸菌 を、カナマイシンを含まない液体LBに6.2e+6細胞/mL(直径50μmの液滴の目標濃度)で再懸濁します。バクテリア懸濁液を室温に保ち、アガロースと混合します。
注:抗生物質は負のマイクロコロニー形成を抑制するため、プラスミド損失実験では培地から抗生物質を除外することが不可欠です。 - 超低ゲル化温度のアガロースを液体LB中で2%(w/v)の濃度で90°Cに加熱することにより、アガロースを調製します。温度制御されたシェーカーで混合物を10分間振とうします。
- サーモシェーカーの温度を39°Cに下げて、アガロース溶液を冷却します。並行して、バクテリア懸濁液チューブをサーモシェーカーに4分間入れて、39°Cに温めます。
- 細菌とアガロース懸濁液を1:1の比率で混合し、アガロース濃度1%(w/v)と細胞懸濁液を3.1e+6細胞/mLで取得します。ネガティブコントロール溶液(コンタミネーションモニタリング)は、細菌懸濁液の代わりに液体LBを使用して、同じアガロース濃度で調製します。
注:アガロース細胞懸濁液は、細菌の増殖による濃度変化を避けるために迅速に使用する必要があります。サーモシェーカーを39°Cに維持して、液滴発生のためにチップヒーターにロードされるまでアガロース液を保持します。
- 調製した培養液から 大腸菌 を、カナマイシンを含まない液体LBに6.2e+6細胞/mL(直径50μmの液滴の目標濃度)で再懸濁します。バクテリア懸濁液を室温に保ち、アガロースと混合します。
3. ハイスループットなシングルコロニー培養
- 実験セットアップ
- ガス圧力ドライバーやフローセンサーを含む、完全なオープンソースのフロープラットフォーム(https://doi.org/10.17605/OSF.IO/6YWJK を参照)を構築または入手します。または、よりシンプルなオープンソースのハードウェアストロボ強化顕微鏡ステージをビルドします(ビルド手順 https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ およびプロジェクトリポジトリ https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage を参照)。
注:市販の顕微鏡、高速度カメラ、およびサンプル加熱機能を備えた従来のマイクロ流体セットアップを使用することもできます。 - 指定されたリポジトリ(https://github.com/wenzel-lab/modular-microfluidics-workstation-controller)に記載されているように、オープンソースのRaspberry Piベースの圧力および流量コントローラーシステムを統合します。
メモ: モジュール制御は図示されており、データリポジトリ(https://doi.org/10.17605/OSF.IO/6YWJK)にバックアップされています。別の方法として、従来の圧力コントローラーまたは高トルクシリンジポンプを採用することができます。 - スライドガラスとピペットチップヒーター(https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring)をセットアップに含めて、アガロースセルサンプルがチップに入るときの温度を制御します。
注:データリポジトリ(https://github.com/wenzel-lab/flow-microscopy-platform および https://doi.org/10.17605/OSF.IO/6YWJK)に示されているこれらのヒーターは、アガロースでの作業を可能にするオープンソースコントローラーシステムの重要な機能であり、他の商用システムでは利用できない場合があります。
- ガス圧力ドライバーやフローセンサーを含む、完全なオープンソースのフロープラットフォーム(https://doi.org/10.17605/OSF.IO/6YWJK を参照)を構築または入手します。または、よりシンプルなオープンソースのハードウェアストロボ強化顕微鏡ステージをビルドします(ビルド手順 https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ およびプロジェクトリポジトリ https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage を参照)。
- シングルセルカプセル化
- マイクロ流体チップをストロボ強化顕微鏡ステージに配置し、液滴生成接合部(水相と油相の交点)が見えるようにします。
- 制御ソフトウェアインターフェースを使用して、ピペットチップヒーターとスライドガラスヒーターを40°Cに設定します。
- チューブとPDMSプラグ付きのシリンジを使用して、細胞懸濁液を含む1%アガロースミックスを200 μLピペットチップにロードします。チップをチップヒーターに挿入し、マイクロ流体チップの水相の入口に置きます。チップのPDMSシールをフローコントロールシステムチューブに接続されたものと交換し、細胞懸濁液の注入を開始します。
- 出口チューブの端を廃液チューブに挿入し、ユーザーインターフェースで2段階の流量または圧力を設定して、流体をマイクロ流体チャネルにゆっくりと供給します。水相には200μL/h(180mbar)、油相には1700μL/h(320mbar)を使用します。液滴発生が安定するまで1分間お待ちください。
注:圧力値はチップ設計のチャネルサイズによって異なり、液滴生成ジャンクション設計ごとに流量値を調整する必要がある場合があります。 - 液滴の発生が安定したら、廃棄物と収集チューブを収集チューブに移します。サンプルリザーバーが空になるまで液滴の収集を続けます。手順3.2.3から3.2.5を繰り返して、手順2.2.4のネガティブコントロールソリューションをカプセル化します。
注:サンプル収集は15分以内に完了する必要があります。 - 液滴生成中は採取チューブを氷上に保管するか、実験後に4°Cで1時間放置して、アガロースが液滴内でゲル化します。
注:マイクロ流体チップは、マイクロチャネルが詰まっていないままで、同じサスペンションがロードされている場合に再利用できます。エマルジョンの生成が完了したら、ピペットチップを廃棄します(ステップ3.2.3〜3.2.5)。
- コロニーの成長とエマルジョンからの放出
- 細菌を含むゲル微小液滴と液滴中のネガティブコントロールを37°Cに設定されたインキュベーションチャンバーに移します。
- マイクロ液滴を少なくとも4時間、または一晩インキュベートして、十分なコロニー成長を確保します。ネガティブコントロールが明視野顕微鏡 で 汚染の兆候を示さないことを確認します。
- エマルジョンからコロニーを放出するために、ピペットまたは針付きシリンジ(ここではサイズ21Gの針を使用)を使用して、ゲルマイクロ液滴エマルジョンの下からできるだけ多くの油を取り除きます。
- 50 μLのゲルマイクロ液滴を新しいマイクロチューブに移し、さらに液滴分析のために4°Cで保存します。残りのエマルジョンに、エマルジョンに等しい容量のフッ素化油と1H、1H、2H、2H-パーフルオロ-1-オクタノール(PFO)の1:1混合物を加えます。
- エマルジョンの上に約200 μLのリン酸緩衝生理食塩水(PBS)緩衝液、または0.9% w/v NaCl緩衝液を加えます。混合物をボルテックスし、固定速度の遠心分離機で短時間スピンダウンします。
- 液界面の底部から油相を慎重に取り除き、上部から100μLのPBSを捨てます。ステップ3.3.4〜3.3.5を繰り返して、油の残留物が最小限またはまったくないPBSバッファーで洗浄されたマイクロゲルを取得します。
注:マイクロゲルは液体界面に沈殿します。油相と一緒にそれらを取り除くことは避けてください。
4. シングルコロニー解析
- 細胞染色
注:このプロトコルでは、多くの異なる染色の組み合わせが機能します。基本的には、プラスミドによってコードされる蛍光タンパク質とは異なる色を持つDNAまたは細胞壁染色剤を選択し、蛍光顕微鏡で利用可能なフィルターの組み合わせを使用して分析する必要があります。ここでは、細胞のDNAをヨウ化プロピジウム(PI)で染色して、プラスミドにコードされた緑色蛍光タンパク質と蛍光を区別していますが、他の多くのDNA染色も使用できます。
注意: PIは発がん性物質の可能性があるため、適切な個人用保護具で取り扱う必要があります。染料は安全に、地域の規制に準拠して廃棄してください。- 洗浄したマイクロゲルを約80 x g で室温で5分間遠心分離します。ピペットを使用して上清を捨てます。
- 50 uL のマイクロゲルを新しいマイクロチューブに移し、エタノール処理のネガティブコントロールとして使用します。
注:PI染色は、エタノール処理の影響を受けた細胞など、膜が損傷した細胞にのみ入ります。 - 残りのマイクロゲルに等量の70%エタノールを加え、ボルテックスで短時間混合します。室温で15分間インキュベートし、PI染色のために細菌膜を透過化します。手順4.1.1を繰り返します。
- 等量の0.9% w/v NaClをマイクロゲルに加え、短時間ボルテックスします。手順4.1.1を繰り返します。
- 2 μL の PI (1 mg/mL) を両方のマイクロチューブサンプルに加えます。十分に混合し、暗所で室温で15分間インキュベートします。
注:ネガティブコントロールが蛍光顕微鏡 法で 赤色蛍光シグナルを示す場合、コロニーの完全性は、前のステップで他の溶液への曝露中に損なわれている可能性があります。
- 顕微鏡
注:液滴とマイクロゲルを倒立型落射蛍光顕微鏡でイメージングし、マイクロゲル内の細菌コロニーの液滴サイズ分布と蛍光を取得します(図2)。ここでは、オープンソースの倒立顕微鏡プラットフォーム(https://github.com/wenzel-lab/SQUID-bioimaging-platform)17 を、10x 0.3NA対物レンズ、明視野照明用の白色LEDアレイ、励起用の470nm LEDとともに使用しています。市販の落射蛍光顕微鏡は、液滴やマイクロゲルのイメージングに使用できます。照明とフィルターは、使用するモデル、ブランド、蛍光タンパク質によって異なるため、キャリブレーションが必要です。- 2 μL のゲル微小液滴をイメージングチャンバースライドに移し、5 μL のフッ素化オイルを添加して液滴の単層を形成し、最適なイメージングを実現します。
注:細胞計数チャンバーまたは単純なマイクロ流体チャンバーは、エマルジョンを薄く広げ、乾燥プロセスを遅らせるのに役立ちます。 - 顕微鏡で、明視野イメージングのために上から白色LEDマトリックス照明をアクティブにします。調製したスライドをセットし、サンプルに焦点を合わせ、液滴の単層を見つけます。明視野画像をキャプチャします。
- サンプルを動かさずに、励起用の470 nm LEDに切り替えてコロニーの蛍光画像をキャプチャします。フィルターホイールを調整して、sfGFPイメージング用の緑色の波長フィルターに合わせます。液滴の単層を含むすべての領域をスキャンし、手順4.2.2〜4.2.3を繰り返して、液滴分析の統計的堅牢性を確認します。
- 染色したマイクロゲル2μLをイメージングチャンバーチップに移し、0.9%w/v NaClの5μLを移し替えて、マイクロゲルの単層を形成します。イメージング中の蒸発を防ぐために、チップの入口と出口を密閉します。
注:蒸発は蛍光標識の共局在に影響を与える可能性があります。マイクロ流体チャンバーは、長時間のイメージングに推奨されます。 - 手順 4.2.2 から 4.2.3 を繰り返します。PIイメージング用の赤色の波長間隔フィルターを調整し、それぞれの画像をキャプチャします。
- 1つの場所でイメージングが完了したら、マイクロゲルの単層を使用してスライド上の次の適切な領域を見つけ、イメージングプロセスを繰り返して、サンプルの統計的堅牢性と包括的な分析を確保します。
- 2 μL のゲル微小液滴をイメージングチャンバースライドに移し、5 μL のフッ素化オイルを添加して液滴の単層を形成し、最適なイメージングを実現します。
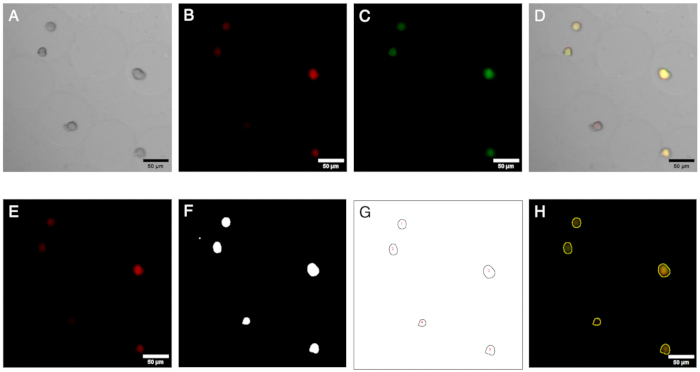
図2:顕微鏡画像とその分析。マイクロゲル中のコロニーの蛍光イメージングと分析。(A-C)倒立顕微鏡による明視野顕微鏡および蛍光顕微鏡 で 取得した画像チャンネル。合成画像(D)は、マイクロゲル内にネガティブコロニー(赤色蛍光のみ)が存在することを示しています。(E-H)画像解析ワークフローの結果。ROIを生成することにより、赤と緑のチャネルでコロニーを同定し、シグナルを定量化してネガティブコロニーの存在を定義することができます。スケールバー:50μmこの 図の拡大版を表示するには、ここをクリックしてください。
- 画像解析
注:カプセル化されたコロニーからの蛍光シグナルを分析し、まれなイベントを特定するために、明視野および蛍光イメージングからの画像をFiji/ImageJで処理できます(図3)。これらの手順はマクロ スクリプトに実装でき、パラメーター値は光学構成によって異なる場合があります。- 緑と赤のチャンネルから画像を開きます。座標 (500, 500) から始まる四角形の関心領域 (ROI) を、幅と高さがそれぞれ 2000 ピクセルで定義します。
注:これらのパラメータは3000 x 3000ピクセルの画像に適用され、ROIはより良い照明で領域を定義します。 - 定義された長方形のROIに画像をトリミングします。各ピクセルの強度から定数値 10 を減算し、画像にスムージング フィルターを適用して、バックグラウンド ノイズを減らし、関心のあるオブジェクトをより明確にします。
注:この領域のみが詳細な分析に使用されます。 - 赤チャネルから画像を複製し、バイナリマスクに変換して、プラスミドに関連するまれなイベントを特定します。しきい値を 10 から 255 の間で設定します。この範囲内のピクセルは前景 (関心のあるオブジェクト) と見なされ、他のピクセルは背景として扱われます。
- 形態学的演算を実行して、小さなギャップを埋め、関心のあるオブジェクト内の穴を埋めます。集水域アルゴリズムを適用して、バイナリ マスク内の重なり合うオブジェクトを分離します。
- バイナリ マスク内のパーティクルを解析します。サイズが 30 ピクセルを超え、真円度が 0.50 から 1.00 の粒子のみを考慮してください。結果が集計され、結果テーブルに追加されます。粒子解析によって検出されたROIのセットを保存して、さらに視覚化します。
- 保存されたROIを緑と赤のチャンネルからの画像に表示します。これらの画像内のROIの強度またはその他のプロパティを測定します。測定値を結果テーブルに記録し、結果を個別のCSVファイルに保存して、さらに統計分析を行います。
- 緑と赤のチャンネルから画像を開きます。座標 (500, 500) から始まる四角形の関心領域 (ROI) を、幅と高さがそれぞれ 2000 ピクセルで定義します。
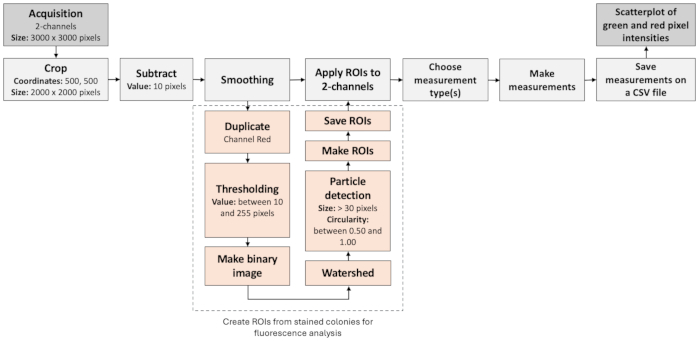
図3:ネガティブコロニーを同定するための画像解析ワークフロー。この図は、蛍光画像を自動的に処理および評価するための段階的なワークフローを示しています。このワークフローは、蛍光標識の共局在と粒子分析に基づいています。 この図の拡大版を表示するには、ここをクリックしてください。
5. 寒天プレート比較アッセイ
注:液滴法を従来のプレートアッセイと比較するために、ステップ2.1.8で同じ 大腸菌 株の蛍光コロニーの定量を得ました。ペトリ皿を使用して行われました。これは、sfGFPプラスミドの安定性を測定するためのアナログ制御法として機能しました。 図 1 のメソッドの図も参照してください。
- LB寒天培地(抗生物質なし)を、Cell Harvestingセクションのステップ2.1.1で説明したように調製します。寒天を60°C以下に冷却し、均質化します。
- 無菌環境で、各シャーレに15mLの寒天培地を注ぎます。プレートが使用できるようになるまで、蓋を部分的に開いた状態でプレートを固めます。
- LB培地中の 大腸菌 培養物の3つのプレートにそれぞれ10μLを、OD6000±0.002で接種します。L字型スプレッダーを使用して均等に広げます。プレートを37°Cでインキュベートし、閉じて反転させます。同じ条件下でバクテリアのないコントロールプレートを準備します。
- 24時間後、オープンソースの蛍光イメージングシステムであるFluoPiを使用してプレートの蛍光画像をキャプチャします(https://github.com/RudgeLab/FluoPi)15。
注:FluoPiは、470nmを中心とする波長の青色励起光とアクリル励起および発光フィルターを備えたRaspberry Piカメラで構成されています。 - キャプチャした画像を使用して、各プレートの蛍光コロニーと非蛍光コロニーを手動でカウントします。
結果
細胞カプセル化とマイクロコロニー形成の検証
細胞のカプセル化は、エマルジョンを破砕してマイクロゲルを洗浄する前に、ゲルの微小液滴に対して明視野顕微鏡を行うことで視覚的に確認することができます。このステップでのエマルジョンの代表的な結果を 図4に示します。
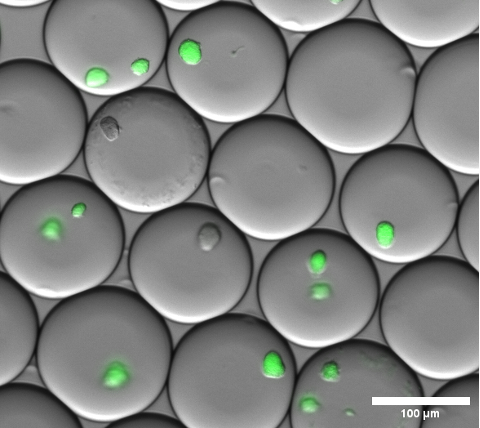
図4:蛍光顕微鏡の重ね合わせ画像の断面。一晩のインキュベーション後、ゲル微小液滴の内部に 大腸菌 コロニーを発現するsfGFPの代表的なマイクロコロニー。倍率10倍、NA0.30の顕微鏡対物レンズを使用しました。スケールバー:100μmこの 図の拡大版を表示するには、ここをクリックしてください。
画像解析結果
マイクロゲルを染色し、明視野、およびいくつかの位置で取得した蛍光チャネルを染色すると、元の画像で陰性と同定されたコロニーを視覚化できます( 図5Aを参照)。1つの実験のすべての画像から抽出したデータをプロットして、さまざまなコロニーの蛍光比を示すことができ、プラスミドでコードされた蛍光を失ったコロニーを強調表示できます( 図5Bを参照)。結果は、分析された合計2785のマイクロコロニーのうち、100のコロニーがプラスミドまたはプラスミドの機能を失ったことを示しており、これは3.6%に相当します。
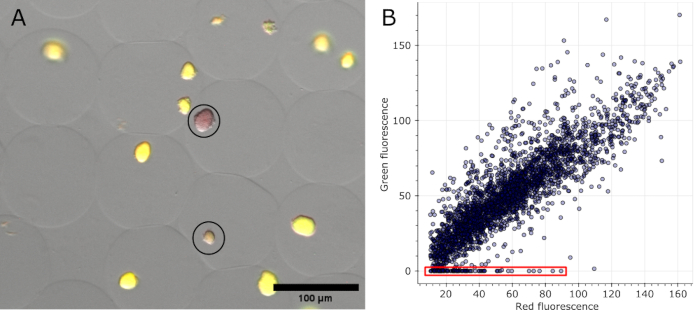
図5:ネガティブマイクロコロニーの定量化。(A)蛍光顕微鏡オーバーレイ画像のセクション。油の除去および染色後、sfGFPを発現するマイクロゲル中の代表コロニーおよび2つのネガティブコロニーは、DNA染色の赤色蛍光を示しました(黒丸で囲んだ部分)。スケールバー:100μm.(B)16のマルチチャンネル顕微鏡画像から抽出した個々のマイクロコロニーの蛍光値の散布図。緑色の蛍光がないコロニーは、グラフの赤で示されているように、陰性としてカウントされました。 この図の拡大版を表示するには、ここをクリックしてください。
寒天プレートの定量
三重プレートの画像を 図6に示し、非蛍光コロニーを白い矢印で示しています。最初のプレート(図6A)は合計213のコロニーを示し、そのうち1つは蛍光性ではありませんでした。2番目のプレート(図6B)には合計49のコロニーがあり、非蛍光コロニーはありませんでした。3番目のプレート(図6C)は合計252のコロニーを示し、そのうち6つは蛍光性ではありませんでした。これらの結果は、平均コロニープラスミド損失率が2.3%で、標準偏差が3.2と大きいことに相当します。
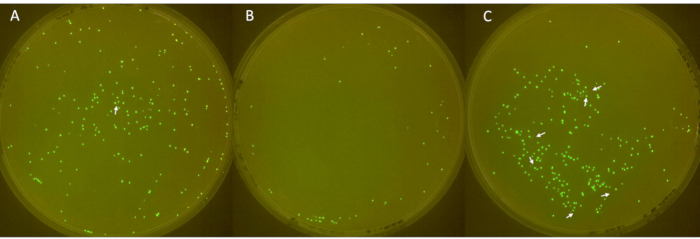
図6:プレート上のネガティブコロニーの同定。(A-C)LB寒天プレート上の蛍光性および非蛍光性大腸菌コロニー(直径:90 mm、高さ:15 mm)。大腸菌に由来し、-80°CストックのsfGFPを含む接種物を、1日目に線条状にし、2日目に抗生物質で培養し、3日目から6日目まで毎日1:100に希釈してプラスミドの損失を可能にしました。コロニーを37°Cで24時間インキュベートし、FluoPiチャンバーでイメージングしました。非蛍光性コロニーをGIMPで増強し、白い矢印で示しました。この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
ゲルマイクロドロップレットベースの分析法により、sfGFPなどの蛍光タンパク質のプラスミドコード遺伝的発現の有無にかかわらず、コロニーを効果的に同定および定量できることが実証されています。プラスミド産物を十分に発現しないコロニーは、すべてのコロニーを染色し、異なる発光波長を特徴とする蛍光DNA染色剤(ここではヨウ化プロピジウム)を用いて同定します。この液滴マイクロ流体工学、ゲル化、および蛍光顕微鏡法の統合は、オープンソース技術を利用して、多くの研究環境で高度なワークフローを実行することを可能にします11,13。ゲルマイクロ液滴の生成が成功したことで、細胞溶解、単一ゲノム増幅、代謝細胞相互作用スクリーニング、培地交換など、高度な分子生物学の単一細胞ワークフローが可能になります8,9。これらの利点は、このプロトコルで、従来のプレートベースのアッセイよりもスケーラブルな方法でマイクロコロニーを増殖、染色、および分析するために使用されます。
重要なステップ
カプセル化プロセスは、プロトコルの重要でデリケートな部分です。特定のサイズ範囲内で均一なマイクロゲルを作製し、液滴あたりの平均細胞数を制御するためには、成分の濃度、流量、圧力を正確に制御する必要があります。さらに、細胞-アガロースミックスの濃度と温度を維持することで、凝集や早期のゲル化を防ぐことができます。ピペットチップ内の液体アガロース細胞懸濁液の温度制御は、シリンジポンプやチューブの温度を制御する努力と比較して、はるかに簡単で堅牢なマイクロゲル生成を提供するオープンソースのハードウェアマイクロ流体ワークステーションの特に有利な実装です。細胞はカプセル化および培養前にアガロース増殖培地と混合されるため、細胞濃度の大きな変化を避けるために、アガロースマイクロゲルを迅速に生成する必要があります。この目的のために、Abateらに触発された液滴分裂マイクロ流体チップの設計が最適化されました18。
変更とトラブルシューティング
元のプロトコルを改良するために、いくつかのキャリブレーションと修正が必要でした。アガロースのカプセル化は、通常の油中水滴よりもはるかに困難であり、アガロースを液体状態に維持しながら、水相流が均一な粒子サイズ範囲を達成することを保証するシステムの設計が必要です。ゲル化によるアガロース粘度の変化は流量に影響を与え、粒子サイズが大きくなります。顕微鏡検査では、励起信号と発光信号が重ならないように、フィルターと光源を慎重に選択して、明確な区別を行う必要があります。当初、細菌の染色にはDAPIが選択されましたが、その発光シグナルがsfGFPと重なったため、青色の検出チャネルでsfGFPが検出されました。PIに切り替えたのは、その発光が長波長(赤色光)でsfGFPから十分に分離されているためです。
提案法を用いてプラスミド損失を定量化した一方で、使用したsfGFPプラスミドは予想外に安定しており、抗生物質を使わずに培養した第1世代の細胞では、pH9培地や40°Cでのインキュベーションなどのストレス条件下でもプラスミド損失の事例はほとんど見られませんでした。 この観察結果は、他の研究グループ1,19の調査結果と一致しています。プラスミドの安定性により、初期細胞培養世代におけるこの分析法の完全な定量能力の実証には限界がありましたが、この分析法がプラスミド保持のわずかな違いでも検出できるほどの感度があることを実証しました。初期世代における高プラスミド安定性の観察は、標的細菌の阻害などのネガティブセレクションアッセイを用いた液滴マイクロ流体スクリーニングに重要な意味を持ちます。これは、選択ターゲットのプラスミド損失が偽陽性の選択結果の原因が少ないことを意味します。液滴マイクロ流体スクリーンは、通常、ピペッティングロボットワークフローなどの他のハイスループットスクリーニングよりもスループットが桁違いに高いため、これらのまれなイベントを評価し、考慮する必要があります。
制限
その利点にもかかわらず、提示された方法には制限があります。マイクロ流体デバイスの製造には、専門知識と細部への細心の注意、および決定論的なカプセル化効率を確保するための流量の厳密な実験的制御が必要です。これらの側面は、さまざまな実験設定の最適化が必要になる場合があります。この方法は、シグナル検出に蛍光顕微鏡に依存しているため、適切なイメージング機器にアクセスする必要がありますが、この機器はオープンソースのハードウェアを使用して製造できるため、よりアクセスしやすくなっています。さらに、マイクロゲルは大型ノズルを使用した市販のフローサイトメトリーで処理できるため、アクセシビリティと実験スループットがさらに向上します。液滴ソーターは、このサイトメトリー分析にも使用できます。
さらに、この方法はプラスミド、染色剤、またはその他のマーカーからの蛍光シグナルを検出するように設計されていますが、蛍光標識が可能な細胞に限定されており、すべての細菌株や実験条件に当てはまるとは限りません。ただし、この方法は、位相差顕微鏡や明視野顕微鏡などの他のタイプの顕微鏡を組み込むように適合させることができ、蛍光以外の表現型アプリケーションが可能になります。さらに、FTIRやラマン分光法などの分光技術と組み合わせることができるため、カプセル化された細胞の化学組成や構造情報を分析するための能力が広がります。これらの適応により、その適用範囲が広がり、多様な研究環境に対応する汎用性の高いツールとなっています。
意義と応用
プラスミド損失19 の従来のアッセイでは、発現を失った細胞の比率を適切に定量化することはできませんが、この情報は実験方法の設計やさまざまな生物学的アプリケーションにおいて非常に重要になる可能性があります。通常、コロニータイプは寒天プレートアッセイで列挙され、 図4に示すように、明確に分離されたコロニーを取得できます。ただし、重複するコロニーは、自信を持って特定するのが困難です。私たちの手では、常に最適なコロニー密度が得られるわけではなく、低周波プラスミド損失イベントの良好な統計を得るためには多くのプレートが必要です。提案された方法は、微小液滴ではコロニーが別々に発生し、小さく、イメージングチャンバーにロードしやすいため、コロニー数の多い単離コロニーからの蛍光シグナルを正確に定量するためのより堅牢なアプローチを提供します。これにより、分析法の統計的表現が大幅に向上し、他のゲルマイクロドロップレットワークフローへの統合が可能になります。
オープンソースのハードウェア11,20を使用すると、研究者はマイクロ流体ワークステーションの設計をカスタマイズし、フローラットを正確に調整できます。そのため、粒子径は様々な細胞種や実験条件に対応しています。この柔軟性は、位相差法や分光法などの他の顕微鏡法を組み込む可能性にも及び、この分析法の適用範囲を広げます。さまざまな条件下でプラスミドの安定性を評価するこの分析法の能力は、特定のストレス条件下で、またはさまざまな培養世代の下で、抗生物質を選択しないプラスミド保持を必要とするアプリケーションにとって非常に重要です。提示された方法の汎用性と適応性は、合成生物学、環境モニタリング、臨床診断2などの分野での多様な研究アプリケーションにとって価値があります。
開示事項
著者らは、競合する金銭的利益や個人的な関係が、この論文で報告された研究に影響を与えた可能性はないと宣言します。
謝辞
この研究は、ANID FONDECYT Regular 1241621 と Chang Zuckerberg Initiative プロジェクト「Latin American Hub for Bioimaging Through Open Hardware」から T.W. に付与された資金提供プロジェクトの一部です。T.W.は、CIFARマクミランマルチスケールヒューマンプログラムのアズリーリグローバル奨学生として、CIFARからの資金提供にも感謝しています。
資料
| Name | Company | Catalog Number | Comments |
| 1H,1H,2H,2H-Perfluoro-1-octanol | Sigma-Aldrich | 370533-25G | For breaking emulsion |
| 70% ethanol | For cell permeabilization | ||
| Agar-Agar | Winkler | 9002-18-0 | |
| Biopsy Punch | 0.75 mm and 1.8 mm | ||
| Blue LED transilluminator | IO Rodeo | ||
| Culture tube | 15 mL | ||
| Desiccator | With vacuum pump | ||
| Disposable cup | For mixing PDMS | ||
| Disposable fork | For mixing PDMS | ||
| E. coli TOP10 strain | |||
| FluoPi microscope | https://github.com/wenzel-lab/FluoPi | Green fluorescence imaging system for analyzing plates | |
| Fluorinated Oil | 3M | Novec 7500 | |
| Glass slide heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of microfluidic chip | |
| Glass Slides | |||
| Hotplate | Mechanic | For evaporating Aquapel | |
| Image analysis software | Fiji/ImageJ | 2.14.0/1.54f | |
| Incubator | Mundo Lab | MLAB Scientific / For incubation of plates and microgels | |
| Isopropanol | For cleaning glass slides | ||
| Kanamycin | 100 ug/mL concentration | ||
| L-shaped spreader | For spreading bacteria on agar plates | ||
| Master mold | Chip design on silicone or glass wafer | ||
| Microtubes | 2 mL | ||
| NaCl solution | Sodium chloride 0.9% w/v | ||
| Open-source hardware strobe-enhanced microscopy stage | https://github.com/wenzel-lab/flow-microscopy-platform | For bright-field microscopy | |
| Petri dish | Citotest | 2303-1090 | 90 x 15 mm |
| Pipette tip heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of pipette tip | |
| Plasma Cleaner | Diener Electronic | 117056 | For bonding PDMS with a glass slide |
| Plasmid pCA_Odd1 | Encodes sfGFP and kanamycin resistance | ||
| Polytetrafluoroethylene (PTFE) tubing | Adtech Polymer Engineering Ltd | ||
| Pre-mixed Luria Bertoni medium | US Biological Life Science | L1520 | |
| Propidium iodide (PI) | For staining | ||
| Raspberry Pi-based pressure and flow controller system | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller | For controlling pressure and flow rates | |
| Silicone elastomer base | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Silicone elastomer curing agent | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Spectrophotometer | For measuring absorbance | ||
| SQUID microscope | https://github.com/wenzel-lab/SQUID-bioimaging-platform | Multi-fluorescence imaging system for analyzing stained cells | |
| Sterile loop | For picking a colony and streaking plating | ||
| Surfactant | Sphere Fluidics | Pico-Surf | |
| Syringes | NIPRO | With filters and tubing | |
| Temperature-controlled shaker | Mundo Lab | DLAB HCM100-Pro | |
| Tweezer | |||
| Ultra-low gelling temperature agarose | Sigma-Aldrich | A2576-5G | For generating hydrogel beads |
| Water repelent solution (fluoroalkyl silane) | Aquapel | For treating microchannels of PDMS device |
参考文献
- Wein, T., Hülter, N. F., Mizrahi, I., Dagan, T. Emergence of plasmid stability under non-selective conditions maintains antibiotic resistance. Nat Commun. 10 (1), 2595(2019).
- Rodríguez-Beltrán, J., DelaFuente, J., León-Sampedro, R., MacLean, R. C., Millán, ÁS. Beyond horizontal gene transfer: The role of plasmids in bacterial evolution. Nat Rev Microbiol. 19 (6), 347(2021).
- Wein, T., Dagan, T. Plasmid evolution. Curr Biol. 30 (19), R1158-R1163 (2020).
- Chen, S., Larsson, M., Robinson, R. C., Chen, S. L. Direct and convenient measurement of plasmid stability in lab and clinical isolates of E. coli. Sci Rep. 7 (1), 4788(2017).
- Rouches, M. V., Xu, Y., Cortes, L. B. G., Lambert, G. A plasmid system with tunable copy number. Nat Commun. 13 (1), 3908(2022).
- Silva, F., Queiroz, J. A., Domingues, F. C. Evaluating metabolic stress and plasmid stability in plasmid DNA production by Escherichia coli. Biotechnol Adv. 30 (3), 691-708 (2012).
- Wang, R., et al. Construction of novel pJRD215-derived plasmids using chloramphenicol acetyltransferase (cat) gene as a selection marker for Acidithiobacillus caldus. PLoS ONE. 12 (8), e0183307(2017).
- Moragues, T., et al. Droplet-based microfluidics. Nat Rev Methods Primers. 3 (1), 32(2023).
- Vitalis, C., Wenzel, T. Leveraging interactions in microfluidic droplets for enhanced biotechnology screens. Current Opinion in Biotechnology. 82, 102966(2023).
- Sinha, N., Subedi, N., Wimmers, F., Soennichsen, M., Tel, J. A pipette-tip based method for seeding cells to droplet microfluidic platforms. J Vis Exp. (144), e57848(2019).
- Wenzel, T. Open hardware: From DIY trend to global transformation in access to laboratory equipment. PLOS Biol. 21 (1), e3001931(2023).
- Murillo, L. F. R., Wenzel, T. Welcome to the journal of open hardware. J Open Hardware. 1 (1), (2017).
- Shin, J. H., Choi, S. Open-source and do-it-yourself microfluidics. Sens Actuators B Chem. 347, 130624(2021).
- Pryszlak, A., et al. Enrichment of gut microbiome strains for cultivation-free genome sequencing using droplet microfluidics. Cell Rep Methods. 2 (1), 100137(2021).
- Pollak, B., et al. Universal loop assembly: open, efficient and cross-kingdom DNA fabrication. Biology. 5 (1), (2020).
- Volkmer, B., Heinemann, M. Condition-dependent cell volume and concentration of Escherichia coli to facilitate data conversion for systems biology modeling. PLoS ONE. 6 (7), e23126(2011).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , (2020).
- Abate, A. R., Weitz, D. A. Faster multiple emulsification with drop splitting. Lab Chip. 11 (11), 1911-1915 (2011).
- Lau, B. T. C., Malkus, P., Paulsson, J. New quantitative methods for measuring plasmid loss rates reveal unexpected stability. Plasmid. 70 (3), 353-361 (2013).
- Oellermann, M., et al. Open hardware in science: The benefits of open electronics. Integr Comp Biol. 62 (4), 1061-1075 (2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved