Method Article
Analyse de la stabilité des plasmides avec la microfluidique de gouttelettes open source
Dans cet article
Résumé
Un flux de travail microfluidique accessible et open-source est présenté pour l’analyse parallélisée de la rétention plasmidique chez les bactéries. En utilisant la microscopie à fluorescence pour quantifier la présence de plasmides dans des microcolonies unicellulaires au sein de microgouttelettes de gel, cette méthode offre une alternative précise, accessible et évolutive au comptage traditionnel sur plaque.
Résumé
Les plasmides jouent un rôle essentiel dans la biologie synthétique en permettant l’introduction et l’expression de gènes étrangers dans divers organismes, facilitant ainsi la construction de circuits et de voies biologiques au sein et entre les populations cellulaires. Pour de nombreuses applications, il est essentiel de maintenir des plasmides fonctionnels sans sélection d’antibiotiques. Cette étude introduit un flux de travail microfluidique basé sur du matériel ouvert pour analyser la rétention des plasmides en cultivant des cellules uniques dans des microgouttelettes de gel et en quantifiant les microcolonies à l’aide de la microscopie à fluorescence. Cette approche permet l’analyse parallèle de nombreuses gouttelettes et microcolonies, offrant une plus grande puissance statistique par rapport au comptage traditionnel sur plaque et permettant l’intégration du test dans d’autres flux de travail microfluidiques de gouttelettes. En utilisant des plasmides exprimant des protéines fluorescentes avec une coloration d’ADN fluorescente non spécifique, des colonies uniques peuvent être identifiées et différenciées en fonction de la perte de plasmide ou de l’expression de marqueurs fluorescents. Notamment, ce flux de travail avancé, mis en œuvre avec du matériel open source, offre un contrôle précis du flux et une gestion de la température de l’échantillon et de la puce microfluidique. Ces fonctionnalités améliorent la facilité d’utilisation, la robustesse et l’accessibilité du flux de travail. Bien que l’étude se concentre sur Escherichia coli comme modèle expérimental, le véritable potentiel de la méthode réside dans sa polyvalence. Il peut être adapté pour diverses études nécessitant la quantification du signal de fluorescence à partir de plasmides ou de colorants, ainsi que pour d’autres applications. L’adoption de matériel open source élargit le potentiel de réalisation de bioanalyses à haut débit à l’aide d’une technologie accessible dans divers contextes de recherche.
Introduction
Les plasmides sont des éléments génétiques essentiels dans les cellules procaryotes, contribuant de manière significative à l’évolution microbienne par transfert latéral d’ADN et adaptation rapide aux changements environnementaux 1,2. Ces molécules d’ADN extrachromosomiques portent des gènes qui offrent des caractéristiques avantageuses, telles que la résistance aux antibiotiques, les fonctions métaboliques et les facteurs de virulence, ce qui les rend précieuses pour la recherche en microbiologie, la biologie synthétique et les études de l’évolution2. Cependant, le maintien des plasmides dans les populations cellulaires est difficile en raison de la charge métabolique de la réplication et de la ségrégation, entraînant souvent une perte de plasmide sans pression de sélection3. De plus, l’hérédité stable nécessite des mécanismes tels que les systèmes toxine-antitoxine et de partition, ce qui ajoute de la complexité à la maintenance des plasmides. L’évaluation de la stabilité des plasmides dans des conditions variables est cruciale à la fois pour la recherche fondamentale et les applications pratiques qui utilisent les plasmides comme élément de recherche principal 4,5. La plupart des méthodes actuelles d’évaluation de la stabilité des plasmides ont des limites importantes : les méthodes basées sur la cytométrie en flux fournissent des données indirectes au niveau de la population, nécessitent un équipement coûteux et manquent de visualisation directe des colonies6. Les méthodes de transcriptomique et de protéomique en vrac sont coûteuses, ne fournissent que des réponses cellulaires moyennes et ne peuvent pas quantifier directement la rétention des plasmides dans les colonies individuelles6. Les méthodes traditionnelles telles que la dilution en série et le passage sont simples mais chronophages et manquent de précision et de représentabilité7. Dans l’ensemble, il reste difficile d’inférer ou de prévoir quantitativement le nombre de colonies qui conservent un plasmide fonctionnel spécifique au fil du temps ou des pressions sélectives.
Pour relever ces défis, un nouveau flux de travail microfluidique utilisant des instruments de recherche à matériel ouvert est présenté pour quantifier les signaux fluorescents dans plusieurs colonies isolées de bactéries, en utilisant Escherichia coli comme modèle. Cette méthode permet une analyse précise et à haut débit de la rétention plasmidique dans diverses conditions ou pressions sélectives. L’analyse de la résolution d’une cellule unique fournit une méthode précise pour manipuler des colonies isolées, produisant des données sensibles sur la quantification des plasmides qui peuvent aider à évaluer les taux de rétention et de perte4.
La microfluidique, en particulier la microfluidique des gouttelettes, est apparue comme un outil puissant pour encapsuler et manipuler des cellules individuelles dans des environnements contrôlés8. Plus précisément, les gouttelettes de microgel peuvent encapsuler des cellules uniques pour une analyse précise et à haut débit sans qu’il soit nécessaire de maintenir les gouttelettes en suspension dans l’huile9, ce qui permet une étude contrôlée de la dynamique des plasmides dans un microenvironnement défini. Après l’encapsulation de suspensions cellulaires directement à partir d’une pointe de pipette10, des techniques de fluorescence peuvent être utilisées pour surveiller la croissance des microcolonies dans les gouttelettes, ce qui permet une analyse détaillée de la rétention et de la ségrégation des plasmides sous différentes pressions de sélection3.
Les avantages de cette méthode par rapport aux techniques traditionnelles de culture en vrac comprennent une précision accrue, une variabilité réduite et la possibilité d’effectuer des analyses à haut débit. La technologie microfluidique open source surmonte les limites des systèmes propriétaires coûteux, telles que les problèmes d’accessibilité, d’adaptabilité et de maintenance, qui entravent souvent les progrès de la recherche 11,12,13. En démontrant comment appliquer le flux de travail expérimental avancé de l’analyse de la rétention plasmidique dans des microgels avec des instruments open source, une méthode accessible et fiable est fournie pour la recherche en biologie des plasmides, les applications de biologie synthétique et les techniques d’analyse microfluidique des gouttelettes.
En résumé, cet article présente une méthode accessible pour évaluer quantitativement la rétention plasmidique chez E. coli avec une puissance statistique élevée. Les capacités de cette méthode en font un outil précieux pour faire progresser la compréhension de la biologie des plasmides et améliorer les applications de la biologie synthétique.
Protocole
La figure 1donne un aperçu schématique de l’évaluation de la stabilité plasmidique chez E. coli. Les détails des réactifs et de l’équipement utilisé sont répertoriés dans la table des matériaux. Les données brutes et les scripts de visualisation sont disponibles à l’https://doi.org/10.17605/OSF.IO/6YWJK.
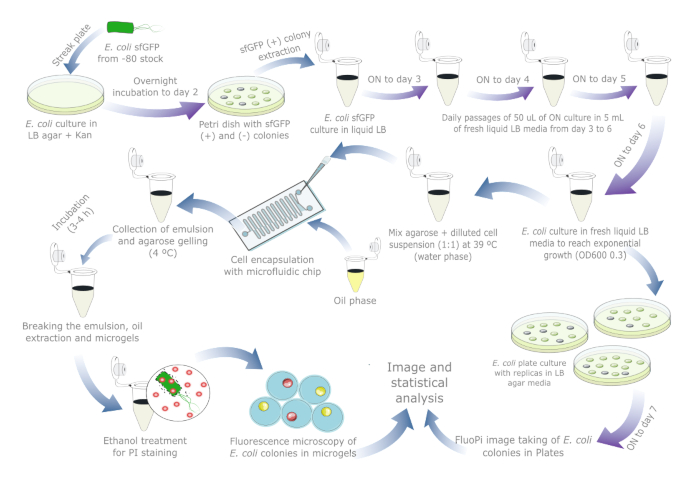
Figure 1 : Protocole quotidien d’évaluation de la stabilité plasmidique chez E. coli. Les flèches bleues indiquent les étapes de la journée et les flèches violettes indiquent l’incubation pendant la nuit. Chaque incubation liquide et gélose a été effectuée à 37 °C avec un tube/une plaque de contrôle négatif séparé. Notez que la préparation et les passages de la culture cellulaire ne sont pas nécessaires pour les échantillons du monde réel dans lesquels la perte de plasmide peut déjà s’être produite, de sorte que le protocole doit être réduit à un jour ou deux si une culture de référence sur plaque est incluse. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Préparation de la puce microfluidique
REMARQUE : Dans ce protocole, différentes conceptions de puces commerciales ou personnalisées peuvent être utilisées pour l’encapsulation de cellules capables de générer des gouttelettes d’eau dans l’huile de moins de 100 μm de diamètre dans un régime d’égouttement. Pour cette étude, la puce a été conçue et fabriquée (voir les données de l’article https://doi.org/10.17605/OSF.IO/6YWJK) selon la même méthode de conception et de fabrication que celle rapportée dans un rapport14 précédemment publié.
- Obtenez ou préparez une puce microfluidique PDMS sur verre à l’aide d’un moule maître conçu pour la génération de microgouttelettes de gel.
- Injectez une solution hydrofuge (fluoroalkyl silane) dans la puce pour rendre les microcanaux internes hydrophobes. Infusez la solution dans les entrées et assurez-vous que tous les canaux sont remplis de liquide. Laissez reposer les canaux remplis pendant environ 30 à 60 s.
- Retirez la solution de l’appareil en expulsant de l’air dans les microcanaux internes. Utilisez une seringue à air vide pour rincer l’air et une lingette absorbante sur les autres orifices pour éviter les éclaboussures.
- Cuire l’appareil traité à 65 °C pendant 15 min sur une plaque chauffante pour évaporer l’excès de solution. Vous pouvez également conserver l’appareil au réfrigérateur (4 °C) pendant la nuit.
REMARQUE : La puce microfluidique est prête à l’emploi. Le protocole peut être mis en pause ici.
2. Préparation de l’échantillon
- Prélèvement cellulaire
REMARQUE : La culture cellulaire dans des gouttelettes peut fournir des données pertinentes sur la dynamique des plasmides. En tant que modèle bactérien expérimental, une souche TOP10 d’E. coli est utilisée avec le plasmide pCA_Odd1 (voir données déposées https://doi.org/10.17605/OSF.IO/6YWJK) qui code pour une protéine fluorescente verte superfolder (sfGFP) et une résistance à la kanamycine15. Les bactéries, les plasmides et les milieux de croissance peuvent varier en fonction du système expérimental.- Préparez la gélose Luria Bertoni (LB) en dissolvant 25 g de LB prémélangés et 12 g de gélose dans 800 mL d’eau distillée (dH2O). Préparez également un milieu liquide en dissolvant 25 g de LB pré-mélangé dans 800 mL de dH2O. Autoclavez les solutions et laissez-les refroidir à environ 60 °C. Déplacez-vous vers un environnement stérile tel qu’une hotte pour les prochaines étapes.
REMARQUE : La poudre LB pré-mélangée contient 10 g de tryptone, 5 g d’extrait de levure et 10 g de NaCl. Un milieu de gélose LB auto-mélangé peut également être utilisé. - Ajouter 50 μL de kanamycine (préparée à 100 mg/mL) à 50 mL de gélose LB-Agar liquide (concentration finale : 100 μg/mL). Mélangez la solution en inversant le tube plusieurs fois.
- Verser environ 15 mL de gélose LB liquide par boîte de Pétri (90 mm x 15 mm). Préparez deux plaques : une pour le contrôle négatif (surveillance de la contamination) et une pour la culture expérimentale. Laissez la solution de gélose LB refroidir et se solidifier jusqu’à ce que la couleur passe de sombre à claire sur les deux plaques.
- À l’aide d’une boucle stérile, répartir la souche d’E. coli sur la plaque de culture à l’aide de la technique de la plaque striée. Remettez le stock de glycérol E. coli à -80 °C immédiatement après utilisation. Fermez les deux plaques et incuberez-les toute la nuit à 37 °C (jour 1).
- Le jour 2, vérifiez la plaque de contrôle négative pour toute colonie comme un signal de contamination (si c’est le cas, répétez les étapes de 2.1.4.). Identifiez les colonies fluorescentes uniques sur la plaque d’E. coli à l’aide d’un transilluminateur à lumière bleue pour préparer un stock de culture liquide.
- Dans des conditions stériles, ouvrir la plaque et utiliser une boucle stérile ou une pointe de pipette de 200 μL pour prélever la colonie sélectionnée et la transférer dans un tube de culture contenant 5 ml de milieu liquide frais LB et 5 μL de kanamycine (préparé à 100 mg/mL). Préparez un tube témoin négatif sans inoculation pour la surveillance de la contamination. Incuber les tubes de culture à 37 °C pendant la nuit en les secouant à 220 tr/min.
- Passez l’échantillon pendant 3 jours supplémentaires dans un milieu sans antibiotiques pour simuler les conditions dans lesquelles la perte de plasmide peut se produire. Les jours 3, 4 et 5, transférez 50 μL de la culture de nuit dans un nouveau tube de culture contenant 5 ml de LB liquide sans antibiotiques. Incuber à 37 °C pendant la nuit en secouant à 220 tr/min. Répétez cette étape jusqu’au jour 6, en obtenant une culture finale de 5 mL après quatre passages au total.
- Transférez 50 μL de la dernière culture de nuit dans un nouveau tube de culture contenant 2 mL de LB liquide. Laissez la culture atteindre une densité optique (OD600) de 0,3 à l’aide d’un spectrophotomètre (3 à 4 h environ).
REMARQUE : La culture fraîche doit être utilisée avant que la DO 600 n’augmente pour s’assurer que les bactéries sont en phase de croissance logarithmique. Si la concentration souhaitée n’est pas atteinte, répéter l’opération à partir de l’étape 2.1.8.
- Préparez la gélose Luria Bertoni (LB) en dissolvant 25 g de LB prémélangés et 12 g de gélose dans 800 mL d’eau distillée (dH2O). Préparez également un milieu liquide en dissolvant 25 g de LB pré-mélangé dans 800 mL de dH2O. Autoclavez les solutions et laissez-les refroidir à environ 60 °C. Déplacez-vous vers un environnement stérile tel qu’une hotte pour les prochaines étapes.
- Mélange cellule-agarose pour l’encapsulation
REMARQUE : Le contrôle de la concentration est essentiel pour assurer l’encapsulation des gouttelettes d’une seule cellule. La concentration cellulaire requise peut être calculée pour un taux d’encapsulation cible et un volume de gouttelettes spécifique, comme illustré dans l’exemple suivant :
Volume des gouttelettes (goutte en V) : si des gouttelettes de 50 μm sont générées
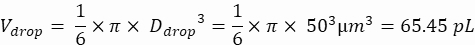
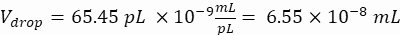
Cellules souhaitées par gouttelette (C) : Une cellule pour cinq gouttelettes en moyenne (0,2 cellule/gouttelette)
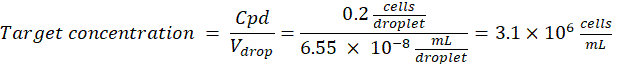
Facteur de dilution : La concentration cellulaire initiale est obtenue à l’étape 2.1.8
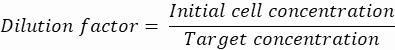
Ici, des gouttelettes de 50 μm ou 100 μm (sans séparation des gouttelettes) (65 à 520 pL) sont générées où environ une cellule sur cinq gouttelettes est encapsulée, soit 1,6 cellule par gouttelette sans séparation. Pour E. coli, utilisez le facteur de conversion de 1 unité OD600 ≈ 7,8 x 108 cellules/mL16. Multipliez la valeur OD600 de l’étape 2.1.8 par le facteur de conversion pour obtenir la concentration initiale (cellules/mL) de la culture.- Remettre E . coli en suspension du matériel de culture préparé dans des LB liquides sans kanamycine à une concentration de 6,2e + 6 cellules/mL (concentration cible pour des gouttelettes de 50 μm de diamètre). Conservez la suspension bactérienne à température ambiante jusqu’à ce qu’elle soit mélangée à l’agarose.
REMARQUE : Les antibiotiques supprimeront la formation de microcolonies négatives, il est donc essentiel de l’exclure du milieu pour les expériences de perte de plasmide. - Préparez de l’agarose à très basse température de gélification en la chauffant à 90 °C dans de la LB liquide à une concentration de 2 % (p/v). Agitez le mélange pendant 10 min dans un agitateur à température contrôlée.
- Réduire la température de l’agitateur thermique à 39 °C pour refroidir la solution d’agarose. En parallèle, placez le tube de suspension des bactéries dans le thermo-shaker pendant 4 min pour le réchauffer à 39 °C.
- Mélangez les bactéries et les suspensions d’agarose dans un rapport de 1:1 pour obtenir une concentration d’agarose de 1 % (p/v) avec une suspension cellulaire à 3,1e + 6 cellules/mL. Préparez la solution de contrôle négatif (surveillance de la contamination) avec la même concentration d’agarose en utilisant du LB liquide au lieu de la suspension bactérienne.
REMARQUE : La suspension de cellules d’agarose doit être utilisée rapidement pour éviter les changements de concentration dus à la croissance de bactéries. Maintenez le thermo-agitateur à 39 °C pour maintenir l’agarose liquide jusqu’à ce qu’il soit chargé dans le réchauffeur de pointe pour la génération de gouttelettes.
- Remettre E . coli en suspension du matériel de culture préparé dans des LB liquides sans kanamycine à une concentration de 6,2e + 6 cellules/mL (concentration cible pour des gouttelettes de 50 μm de diamètre). Conservez la suspension bactérienne à température ambiante jusqu’à ce qu’elle soit mélangée à l’agarose.
3. Culture monocolonie à haut débit
- Dispositif expérimental
- Construisez ou obtenez la plate-forme de débit open source complète (voir https://doi.org/10.17605/OSF.IO/6YWJK), y compris les pilotes de pression de gaz et les capteurs de débit. Vous pouvez également créer l’étape de microscopie stroboscopique matérielle open source plus simple (voir les instructions de construction https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ et le référentiel du projet https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage).
REMARQUE : On peut également utiliser une configuration microfluidique traditionnelle avec un microscope commercial, une caméra à grande vitesse et des capacités de chauffage d’échantillon. - Intégrez le système de contrôleur de pression et de débit open source basé sur Raspberry Pi, comme indiqué dans le référentiel désigné (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller).
REMARQUE : Le contrôle du module est illustré et sauvegardé dans le référentiel de données (https://doi.org/10.17605/OSF.IO/6YWJK. En alternative, des régulateurs de pression traditionnels ou des pousse-seringues à couple élevé peuvent être utilisés. - Inclure une lame en verre et des réchauffeurs de pointe de pipette (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring) dans la configuration pour contrôler la température de l’échantillon de cellule d’agarose lorsqu’il entre dans la puce.
REMARQUE : Ces réchauffeurs, illustrés dans le référentiel de données (https://github.com/wenzel-lab/flow-microscopy-platform et https://doi.org/10.17605/OSF.IO/6YWJK), sont une caractéristique importante du système de contrôleur open source qui permet de travailler avec l’agarose et peuvent ne pas être disponibles dans d’autres systèmes commerciaux.
- Construisez ou obtenez la plate-forme de débit open source complète (voir https://doi.org/10.17605/OSF.IO/6YWJK), y compris les pilotes de pression de gaz et les capteurs de débit. Vous pouvez également créer l’étape de microscopie stroboscopique matérielle open source plus simple (voir les instructions de construction https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ et le référentiel du projet https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage).
- Encapsulation de cellules uniques
- Positionnez la puce microfluidique sur la platine de microscopie stroboscopique, en veillant à ce que la jonction de génération de gouttelettes (intersection des phases aqueuse et huileuse) soit visible.
- Réglez le réchauffeur de pointe de pipette et le réchauffeur de diapositive en verre à 40 °C à l’aide de l’interface du logiciel de commande.
- À l’aide d’une seringue munie d’un tube et d’un bouchon PDMS, chargez le mélange d’agarose à 1 % avec suspension cellulaire dans une pointe de pipette de 200 μL. Insérez l’embout dans le réchauffeur d’embout et placez-le sur l’entrée de la phase aqueuse dans la puce microfluidique. Remplacez le joint PDMS de l’embout par un joint connecté à la tubulure du système de contrôle de débit et démarrez l’infusion de la suspension cellulaire.
- Insérez l’extrémité du tube de sortie dans un tube d’évacuation et réglez les débits ou les pressions de deux phases sur l’interface utilisateur pour fournir lentement le fluide au canal microfluidique. Utilisez 200 μL/h (180 mbar) pour la phase aqueuse et 1700 μL/h (320 mbar) pour la phase huileuse. Comptez 1 min pour la stabilisation de la génération de gouttelettes.
REMARQUE : Les valeurs de pression dépendent de la taille des canaux de la conception de la puce, et les valeurs de débit peuvent devoir être ajustées pour différentes conceptions de jonction de génération de gouttelettes. - Une fois que la génération de gouttelettes est stable, transférez les déchets et le tube de collecte dans le tube de collecte. Continuer à recueillir les gouttelettes jusqu’à ce que le réservoir d’échantillon soit vide. Répétez les étapes 3.2.3 à 3.2.5 pour encapsuler la solution témoin négative de l’étape 2.2.4.
REMARQUE : Le prélèvement de l’échantillon doit être terminé dans les 15 minutes. - Conservez les tubes de collecte sur de la glace pendant la génération des gouttelettes ou placez-les à 4 °C après l’expérience pendant 1 h pour permettre à l’agarose de se gélifier à l’intérieur des gouttelettes.
REMARQUE : La puce microfluidique peut être réutilisée si les microcanaux restent débouchés et que la même suspension est chargée. Jetez la pointe de la pipette une fois la génération de l’émulsion terminée (étapes 3.2.3 à 3.2.5).
- Croissance de la colonie et libération de l’émulsion
- Transférez les microgouttelettes de gel contenant des bactéries et le contrôle négatif dans les gouttelettes dans une chambre d’incubation réglée à 37 °C.
- Incuber les microgouttelettes pendant au moins 4 h, ou toute la nuit, pour permettre une croissance suffisante de la colonie. Confirmez que le témoin négatif ne montre aucun signe de contamination par microscopie à fond clair.
- Pour libérer les colonies de l’émulsion, prélevez autant d’huile que possible sous l’émulsion de microgouttelettes de gel à l’aide d’une pipette ou d’une seringue avec une aiguille (ici, une aiguille de taille 21 G a été utilisée).
- Transférez 50 μL de microgouttelettes de gel dans un nouveau microtube et stockez-le à 4 °C pour une analyse ultérieure des gouttelettes. À l’émulsion restante, ajoutez un mélange 1:1 d’huile fluorée avec du 1H, 1H, 2H, 2H-perfluoro-1-octanol (PFO) dans un volume égal à l’émulsion.
- Ajoutez environ 200 μL de tampon salin tamponné au phosphate (PBS), ou de tampon NaCl à 0,9 % p/v, sur l’émulsion. Vortex le mélange et faites-le tourner brièvement dans une centrifugeuse à vitesse fixe.
- Retirez soigneusement la phase huileuse du bas de l’interface liquide et jetez 100 μL de PBS par le haut. Répétez les étapes 3.3.4 à 3.3.5 pour obtenir des microgels lavés dans un tampon PBS avec un minimum ou pas de résidus d’huile.
REMARQUE : Les microgels se déposent à l’interface liquide ; Évitez de les enlever en même temps que la phase huileuse.
4. Analyse d’une seule colonie
- Coloration cellulaire
REMARQUE : De nombreuses combinaisons de colorants différentes fonctionnent pour ce protocole. Essentiellement, il faut choisir une coloration d’ADN ou de paroi cellulaire dont la couleur est distincte de la protéine fluorescente codée par le plasmide et qui peut être analysée à l’aide des combinaisons de filtres disponibles sur le microscope à fluorescence. Ici, l’ADN des cellules a été coloré avec de l’iodure de propidium (PI) pour distinguer sa fluorescence de la protéine fluorescente verte codée sur les plasmides, mais de nombreuses autres colorations d’ADN peuvent être utilisées.
ATTENTION : L’IP est un cancérogène potentiel et doit être manipulé avec un équipement de protection individuelle approprié. Éliminez le colorant en toute sécurité et conformément aux réglementations locales.- Centrifugez les microgels lavés à environ 80 x g pendant 5 min à température ambiante. Jetez le surnageant à l’aide d’une pipette.
- Transférez 50 μL des microgels dans un nouveau microtube pour servir de contrôle négatif pour le traitement à l’éthanol.
REMARQUE : La coloration PI ne pénètre que dans les cellules dont les membranes sont compromises, telles que celles affectées par le traitement à l’éthanol. - Ajoutez un volume égal d’éthanol à 70 % aux microgels restants et mélangez brièvement avec un vortex. Incuber à température ambiante pendant 15 min pour perméabiliser les membranes bactériennes pour la coloration PI. Répétez l’étape 4.1.1.
- Ajouter un volume égal de 0,9 % p/v de NaCl dans les microgels et agiter brièvement. Répétez l’étape 4.1.1.
- Ajouter 2 μL d’IP (1 mg/mL) aux deux échantillons de microtubes. Bien mélanger et incuber à l’obscurité à température ambiante pendant 15 min.
REMARQUE : Si le témoin négatif présente un signal fluorescent rouge via la microscopie à fluorescence, l’intégrité des colonies peut avoir été compromise lors de leur exposition à d’autres solutions lors des étapes précédentes.
- Microscopie
REMARQUE : Les gouttelettes et les microgels sont imagés à l’aide d’un microscope à épifluorescence inversée afin d’obtenir la distribution de la taille des gouttelettes et la fluorescence des colonies bactériennes dans les microgels (figure 2). Ici, une plate-forme de microscopie inversée (https://github.com/wenzel-lab/SQUID-bioimaging-platform)17 open source est utilisée avec un objectif 10x 0,3NA, un réseau de LED blanches pour l’éclairage en fond clair et une LED 470nm pour l’excitation. Les microscopes à épifluorescence commerciaux peuvent être utilisés pour imager des gouttelettes et des microgels. L’étalonnage est nécessaire car l’éclairage et les filtres varient en fonction du modèle, de la marque et des protéines fluorescentes utilisées.- Transférez 2 μL de microgouttelettes de gel dans une lame de chambre d’imagerie et ajoutez 5 μL d’huile fluorée pour aider à former une monocouche de gouttelettes pour une imagerie optimale.
REMARQUE : Les chambres de comptage de cellules ou de simples chambres microfluidiques peuvent aider à étaler l’émulsion finement et à ralentir le processus de séchage. - Sur le microscope, activez l’éclairage matriciel LED blanc par le haut pour l’imagerie en fond clair. Montez la lame préparée, concentrez-vous sur l’échantillon et localisez une monocouche de gouttelettes. Capturez une image en fond clair.
- Sans déplacer l’échantillon, capturez une image de fluorescence des colonies en passant à la LED de 470 nm pour l’excitation. Ajustez la roue à filtres pour l’aligner avec le filtre de longueur d’onde vert pour l’imagerie sfGFP. Scannez toutes les zones contenant des monocouches de gouttelettes et répétez les étapes 4.2.2 à 4.2.3 pour assurer la robustesse statistique de l’analyse des gouttelettes.
- Transférez 2 μL des microgels colorés dans une puce de chambre d’imagerie et 5 μL de NaCl à 0,9 % p/v pour aider à former une monocouche de microgels. Scellez l’entrée et la sortie de la puce pour éviter l’évaporation pendant l’imagerie.
REMARQUE : L’évaporation peut affecter la colocalisation des marqueurs fluorescents. Les chambres microfluidiques sont recommandées pour l’imagerie prolongée. - Répétez les étapes 4.2.2 à 4.2.3. Ajustez le filtre d’intervalle de longueur d’onde rouge pour l’imagerie PI et capturez les images correspondantes.
- Après avoir terminé l’imagerie à un endroit, trouvez la zone appropriée suivante sur la lame avec une monocouche de microgels et répétez le processus d’imagerie pour garantir la robustesse statistique et l’analyse complète de l’échantillon.
- Transférez 2 μL de microgouttelettes de gel dans une lame de chambre d’imagerie et ajoutez 5 μL d’huile fluorée pour aider à former une monocouche de gouttelettes pour une imagerie optimale.
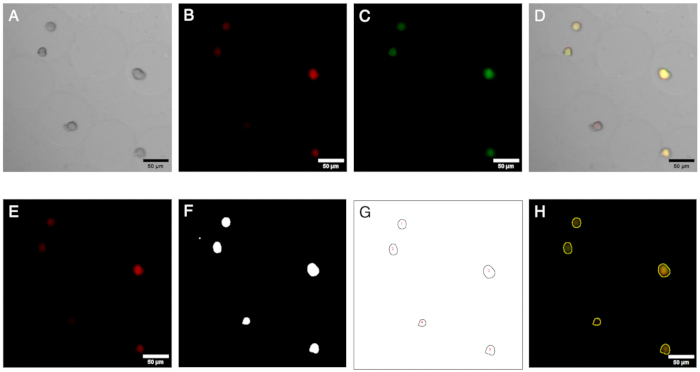
Figure 2 : Images de microscopie et leur analyse. Imagerie par fluorescence et analyse de colonies dans des microgels. (A-C) Canaux d’image acquis par microscopie à fond clair et à fluorescence avec le microscope inversé. L’image composite (D) montre la présence d’une colonie négative (fluorescence rouge uniquement) au sein des microgels. (E–H) Résultats du flux de travail d’analyse d’images. En générant des ROI, les colonies peuvent être identifiées sur les canaux rouge et vert, et les signaux peuvent être quantifiés pour définir la présence de colonies négatives. Barres d’échelle : 50 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Analyse d’images
REMARQUE : Pour analyser les signaux de fluorescence des colonies encapsulées et identifier les événements rares, les images de l’imagerie en fond clair et de fluorescence peuvent être traitées avec Fiji/ImageJ (Figure 3). Ces étapes peuvent être implémentées dans un script de macro, et les valeurs des paramètres peuvent varier en fonction de la configuration optique.- Ouvrez les images à partir des canaux vert et rouge. Définissez une zone d’intérêt (ROI) rectangulaire commençant aux coordonnées (500, 500) d’une largeur et d’une hauteur de 2000 pixels chacune.
REMARQUE : Ces paramètres s’appliquent aux images de 3000 x 3000 pixels, et le retour d’intérêt définit la zone avec le meilleur éclairage. - Recadrez l’image à la valeur d’intérêt rectangulaire définie. Soustrayez une valeur constante de 10 de l’intensité de chaque pixel et appliquez un filtre de lissage à l’image pour réduire le bruit de fond et rendre les objets d’intérêt plus distincts.
REMARQUE : Seule cette région sera utilisée pour une analyse plus approfondie. - Dupliquez l’image du canal rouge et convertissez-la en un masque binaire pour identifier les événements rares liés au plasmide. Réglez les valeurs de seuil entre 10 et 255. Les pixels situés dans cette plage seront considérés comme des objets d’intérêt, tandis que les autres seront considérés comme des objets d’arrière-plan.
- Effectuer des opérations morphologiques pour fermer les petits espaces et remplir les trous à l’intérieur des objets d’intérêt. Appliquez l’algorithme de plage horaire pour séparer les objets qui se chevauchent dans le masque binaire.
- Analysez les particules dans le masque binaire. Ne considérez que les particules d’une taille supérieure à 30 pixels et d’une circularité comprise entre 0,50 et 1,00. Les résultats sont résumés et ajoutés au tableau des résultats. Enregistrez l’ensemble des retours sur investissement détectés par l’analyse des particules pour une visualisation plus approfondie.
- Affichez les retours sur investissement enregistrés sur les images des canaux vert et rouge. Mesurez l’intensité ou d’autres propriétés des retours sur investissement dans ces images. Enregistrez les mesures dans le tableau des résultats et enregistrez les résultats dans des fichiers CSV séparés pour une analyse statistique plus approfondie.
- Ouvrez les images à partir des canaux vert et rouge. Définissez une zone d’intérêt (ROI) rectangulaire commençant aux coordonnées (500, 500) d’une largeur et d’une hauteur de 2000 pixels chacune.
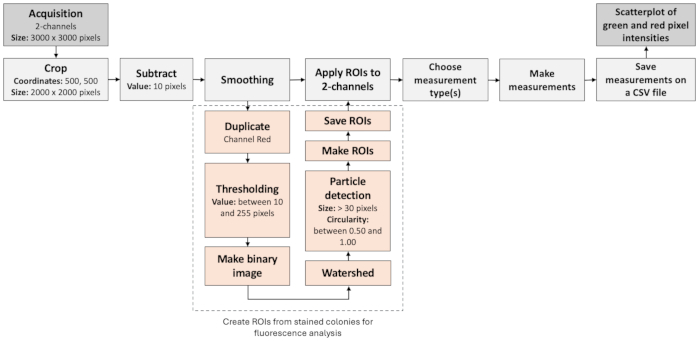
Figure 3 : Flux de travail d’analyse d’images pour identifier les colonies négatives. La figure illustre un flux de travail étape par étape pour traiter et évaluer automatiquement les images de fluorescence. Le flux de travail est basé sur la colocalisation des marqueurs de fluorescence ainsi que sur l’analyse des particules. Veuillez cliquer ici pour voir une version agrandie de cette figure.
5. Essai de comparaison de plaque d’agarle
REMARQUE : Pour comparer la méthode des gouttelettes à un dosage sur plaque traditionnel, la quantification des colonies fluorescentes de la même souche d’E. coli a été obtenue à l’étape 2.1.8. a été réalisée à l’aide de boîtes de Pétri. Il s’agissait d’une méthode de contrôle analogique pour mesurer la stabilité du plasmide sfGFP. Voir aussi l’illustration de la méthode à la figure 1.
- Préparez la gélose LB (sans antibiotique) comme décrit à l’étape 2.1.1 de la section Récolte de cellules. Refroidir l’agar à une température inférieure à 60 °C et homogénéiser.
- Dans un environnement stérile, verser 15 ml de milieu gélosé dans chaque boîte de Pétri. Laissez les plaques se solidifier avec les couvercles partiellement ouverts jusqu’à ce qu’elles soient prêtes à l’emploi.
- Inoculer trois plaques de 10 μL chacune d’une culture d’E. coli dans un milieu LB avec une DO 600 de 0,007 ± 0,002. Répartissez uniformément à l’aide d’un écarteur en forme de L. Incuber les plaques à 37 °C, fermées et inversées. Préparez une plaque de contrôle sans bactéries dans les mêmes conditions.
- Après 24 h, capturez des images de fluorescence des plaques à l’aide du FluoPi, un système d’imagerie par fluorescence (https://github.com/RudgeLab/FluoPi)15.
REMARQUE : Le FluoPi se compose d’une caméra Raspberry Pi avec une lumière d’excitation bleue à une longueur d’onde centrée à 470 nm et des filtres d’excitation et d’émission acryliques. - Comptez manuellement les colonies fluorescentes et non fluorescentes dans chaque plaque à l’aide des images capturées.
Résultats
Validation de l’encapsulation cellulaire et de la formation de microcolonies
L’encapsulation cellulaire peut être confirmée visuellement en effectuant une microscopie à fond clair sur les microgouttelettes de gel avant de briser l’émulsion et de laver les microgels. Un résultat représentatif de l’émulsion à cette étape est illustré à la figure 4.
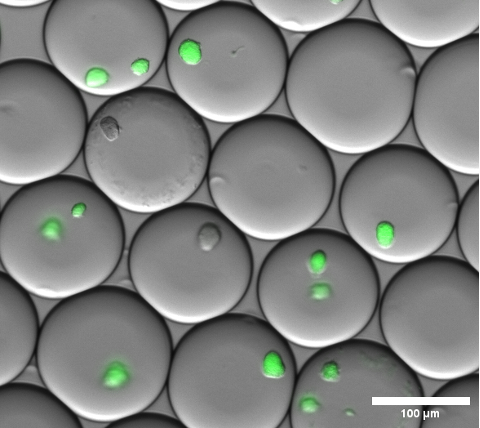
Figure 4 : Coupe d’une image de superposition de microscopie à fluorescence. Après une nuit d’incubation, des microcolonies représentatives de sfGFP exprimant E . coli se colonisent à l’intérieur de microgouttelettes de gel. Un objectif de microscope avec un grossissement de 10x et une NA de 0,30 a été utilisé. Barre d’échelle : 100 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats de l’analyse d’images
Une fois les microgels colorés et en fond clair, ainsi que les canaux de fluorescence acquis à plusieurs positions, les colonies identifiées comme négatives dans les images originales peuvent être visualisées (voir Figure 5A). Les données extraites de toutes les images d’une expérience peuvent être tracées pour montrer le rapport de fluorescence de diverses colonies, en mettant en évidence celles qui ont perdu la fluorescence codée par plasmide (voir Figure 5B). Les résultats indiquent que 100 colonies ont perdu leur plasmide ou leur fonctionnalité plasmidique sur un total de 2785 microcolonies analysées, ce qui correspond à 3,6 %.
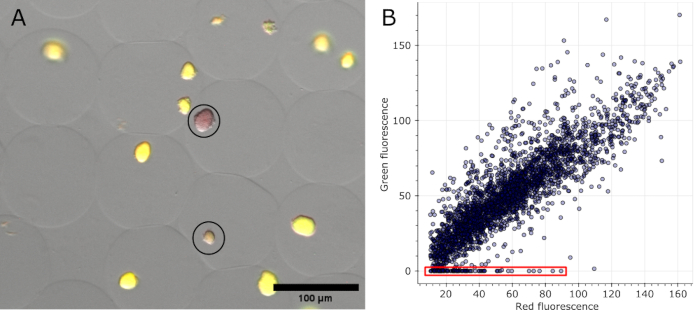
Figure 5 : Quantification des microcolonies négatives. (A) Coupe d’une image de superposition de microscopie à fluorescence. Après l’élimination de l’huile et la coloration, des colonies représentatives dans des microgels exprimant sfGFP et deux colonies négatives ont montré la fluorescence rouge de la coloration de l’ADN (entourée en noir). Barre d’échelle : 100 μm. (B) Nuage de points des valeurs de fluorescence de microcolonies individuelles extraites de 16 images de microscopie multicanaux. Les colonies sans fluorescence verte ont été comptées comme négatives, comme l’indique le rouge dans le graphique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Quantification de la plaque de gélose
Des images des plaques triples sont montrées à la figure 6, avec des colonies non fluorescentes indiquées par des flèches blanches. La première plaque (figure 6A) montrait un total de 213 colonies, dont 1 n’était pas fluorescente. La deuxième plaque (figure 6B) comportait un total de 49 colonies, sans colonies non fluorescentes. La troisième plaque (figure 6C) montrait un total de 252 colonies, dont 6 n’étaient pas fluorescentes. Ces résultats correspondent à un taux moyen de perte de plasmide de colonie de 2,3 %, avec un écart-type important de 3,2.
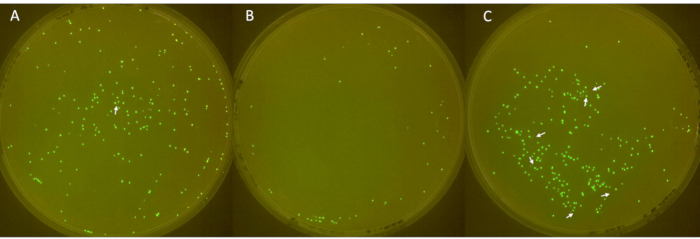
Figure 6 : Identification des colonies négatives sur les plaques. (A-C) Colonies d’E. coli fluorescentes et non fluorescentes sur plaques de gélose LB (diamètre : 90 mm, hauteur : 15 mm). L’inoculum, dérivé d’E. coli avec sfGFP à partir d’un stock de -80 °C, a été strié le jour 1, cultivé avec un antibiotique le jour 2 et dilué à 1:100 par jour du jour 3 au 6 pour permettre la perte de plasmide. Les colonies ont été incubées à 37 °C pendant 24 h et imagées dans une chambre FluoPi. Les colonies non fluorescentes ont été améliorées avec GIMP et indiquées par des flèches blanches. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Il a été démontré qu’une méthode basée sur des microgouttelettes de gel permet d’identifier et de quantifier efficacement les colonies avec et sans expression génétique codée par plasmide de protéines fluorescentes, telles que la sfGFP. Les colonies qui n’expriment pas suffisamment le produit plasmidique sont identifiées à l’aide d’une coloration d’ADN fluorescente (ici, l’iodure de propidium) qui colore toutes les colonies et présente une longueur d’onde d’émission différente. Cette intégration de la microfluidique des gouttelettes, de la gélification et de la microscopie à fluorescence, à l’aide d’une technologie open source, permet d’exécuter un flux de travail avancé dans de nombreux contextes de recherche11,13. La génération réussie de gel-microgouttelettes permet des flux de travail unicellulaires avancés en biologie moléculaire, y compris la lyse cellulaire, l’amplification du génome unique, les criblages d’interaction cellulaire métabolique, l’échange de milieux, etc. 8,9. Ces avantages sont utilisés dans ce protocole pour cultiver, colorer et analyser les microcolonies de manière plus évolutive que dans les tests traditionnels sur plaque.
Étapes critiques
Le processus d’encapsulation est une partie critique et délicate du protocole. Un contrôle précis des concentrations d’ingrédients, des débits et des pressions est nécessaire pour générer des microgels uniformes dans une plage de taille spécifique et contrôler le nombre moyen de cellules par gouttelette. De plus, le maintien de la concentration et de la température du mélange cellule-agarose empêche l’agglutination ou la gélification prématurée. Le contrôle de la température de la suspension de cellules d’agarose liquide dans une pointe de pipette est une mise en œuvre particulièrement avantageuse de notre station de travail microfluidique matérielle open source qui permet une génération de microgel beaucoup plus facile et plus robuste par rapport aux efforts de contrôle de la température des pousse-seringues et des tubes. Étant donné que les cellules sont mélangées au milieu de croissance de l’agarose avant l’encapsulation et la culture, les microgels d’agarose doivent être générés rapidement afin d’éviter des changements majeurs de concentration cellulaire. À cette fin, la conception d’une puce microfluidique à séparation de gouttelettes inspirée d’Abate et al. a été optimisée18.
Modifications et dépannage
Plusieurs étalonnages et modifications ont été nécessaires pour affiner le protocole original. L’encapsulation de l’agarose est beaucoup plus difficile que celle des gouttelettes d’eau dans l’huile ordinaires, nécessitant la conception d’un système pour maintenir l’agarose à l’état liquide tout en veillant à ce que le flux de phase aqueuse atteigne une plage de taille de particule homogène. Les changements de viscosité de l’agarose dus à la gélification affectent le débit, entraînant des particules de plus grande taille. La microscopie nécessite une sélection minutieuse de filtres et de sources lumineuses pour garantir des signaux d’excitation et d’émission non chevauchants pour une différenciation claire. Initialement, le DAPI a été choisi pour colorer les bactéries, mais son signal d’émission chevauchait le sfGFP, ce qui entraînait la détection du sfGFP dans le canal de détection bleu. Nous sommes passés à PI car son émission est bien séparée de sfGFP aux grandes longueurs d’onde (lumière rouge).
Alors que la perte de plasmide a été quantifiée à l’aide de la méthode proposée, le plasmide sfGFP utilisé était étonnamment stable, ne montrant pratiquement aucun cas de perte de plasmide dans la première génération de cellules cultivées sans antibiotiques, même dans des conditions de stress telles que les milieux de pH 9 et l’incubation à 40 °C. Cette observation est cohérente avec les résultats d’autres groupes de recherche 1,19. La stabilité du plasmide a limité la démonstration des capacités complètes de quantification de la méthode pour les premières générations de cultures cellulaires, mais elle a démontré que la méthode est suffisamment sensible pour détecter même de petites différences dans la rétention du plasmide. L’observation d’une stabilité plasmidique élevée chez les jeunes générations a une implication importante pour les criblages microfluidiques de gouttelettes utilisant un test de sélection négative, comme l’inhibition des bactéries cibles. Cela signifie que la perte plasmidique des cibles de sélection est une source faible de résultats de sélection faussement positifs. Étant donné que les cribles microfluidiques à gouttelettes dépassent généralement d’autres cribles à haut débit, tels que les flux de travail des robots de pipetage, de plusieurs ordres de grandeur en termes de débit, ces événements rares doivent être évalués et pris en compte.
Limitations
Malgré ses avantages, la méthode présentée présente des limites. La fabrication de dispositifs microfluidiques nécessite une expertise et une attention méticuleuse aux détails, ainsi qu’un contrôle expérimental strict des débits pour garantir une efficacité d’encapsulation déterministe. Ces aspects peuvent nécessiter une optimisation pour différentes configurations expérimentales. Bien que cette méthode repose sur la microscopie à fluorescence pour la détection des signaux, nécessitant l’accès à un équipement d’imagerie approprié, cet équipement peut être fabriqué à l’aide de matériel open source, ce qui le rend plus accessible. De plus, les microgels peuvent être traités en cytométrie en flux commerciale avec de grandes buses, ce qui améliore encore l’accessibilité et le débit expérimental. Des trieurs de gouttelettes peuvent également être utilisés pour cette analyse cytométrique.
De plus, bien que la méthode soit conçue pour détecter les signaux fluorescents des plasmides, des colorants ou d’autres marqueurs, elle est limitée aux cellules qui peuvent être marquées par fluorescence, ce qui peut ne pas s’appliquer à toutes les souches bactériennes ou à toutes les conditions expérimentales. Cependant, la méthode peut être adaptée pour incorporer d’autres types de microscopies, telles que la microscopie à contraste de phase ou la microscopie à fond clair, permettant des applications de phénotypage au-delà de la fluorescence. De plus, il peut être combiné avec des techniques spectroscopiques telles que la spectroscopie FTIR ou Raman, élargissant ainsi ses capacités d’analyse des compositions chimiques et des informations structurelles des cellules encapsulées. Ces adaptations élargissent le champ d’application de la recherche, ce qui en fait un outil polyvalent pour divers contextes de recherche.
Importance et applications
Les tests traditionnels de perte de plasmide19 ne permettent pas une bonne quantification du rapport des cellules qui ont perdu leur expression, une information qui peut être très importante dans la conception de méthodes expérimentales et diverses applications biologiques. Habituellement, les types de colonies sont énumérés dans les essais sur plaque de gélose, où des colonies isolées bien définies peuvent être obtenues, comme le montre la figure 4. Cependant, il est difficile d’identifier avec certitude les colonies qui se chevauchent ; Entre nos mains, nous n’obtenons pas toujours une densité de colonie optimale, et de nombreuses plaques sont nécessaires pour obtenir de bonnes statistiques des événements de perte de plasmide à basse fréquence. La méthode proposée offre une approche plus robuste pour quantifier avec précision les signaux fluorescents provenant de colonies isolées avec un nombre plus élevé de colonies que les méthodes analogiques sur plaque gélosée, car, dans les microgouttelettes, les colonies se développent séparément, sont plus petites et sont faciles à charger dans des chambres d’imagerie, ce qui permet la quantification d’un grand nombre de colonies basée sur la microscopie ou la cytométrie en flux. Cela peut améliorer considérablement la représentation statistique de la méthode et permettre l’intégration dans d’autres flux de travail gel-microgouttelettes.
L’utilisation de matériel open source 11,20 permet aux chercheurs de personnaliser la conception de la station de travail microfluidique et d’ajuster précisément le débit ; Par conséquent, la taille des particules prend en charge différents types de cellules et conditions expérimentales. Cette flexibilité s’étend à l’incorporation potentielle d’autres types de microscopie, tels que le contraste de phase ou la spectroscopie, élargissant ainsi l’applicabilité de la méthode. La capacité de la méthode à évaluer la stabilité des plasmides dans diverses conditions est cruciale pour les applications nécessitant une rétention des plasmides sans sélection d’antibiotiques, dans des conditions de stress particulières ou dans diverses générations de cultures. La polyvalence et l’adaptabilité de la méthode présentée la rendent précieuse pour diverses applications de recherche dans des domaines tels que la biologie synthétique, la surveillance de l’environnement et le diagnostic clinique2.
Déclarations de divulgation
Les auteurs déclarent qu’aucun intérêt financier ou relation personnelle concurrent n’aurait pu influencer les travaux rapportés dans cet article.
Remerciements
Ce travail s’inscrit dans le cadre des projets financés par l’ANID FONDECYT Regular 1241621 et le projet de l’Initiative Chang Zuckerberg « Latin American Hub for Bioimaging Through Open Hardware ». T.W. est également reconnaissant du financement de l’ICRA, en tant que chercheur mondial Azrieli dans le programme MacMillan Multiscale Human de CIFAR.
matériels
| Name | Company | Catalog Number | Comments |
| 1H,1H,2H,2H-Perfluoro-1-octanol | Sigma-Aldrich | 370533-25G | For breaking emulsion |
| 70% ethanol | For cell permeabilization | ||
| Agar-Agar | Winkler | 9002-18-0 | |
| Biopsy Punch | 0.75 mm and 1.8 mm | ||
| Blue LED transilluminator | IO Rodeo | ||
| Culture tube | 15 mL | ||
| Desiccator | With vacuum pump | ||
| Disposable cup | For mixing PDMS | ||
| Disposable fork | For mixing PDMS | ||
| E. coli TOP10 strain | |||
| FluoPi microscope | https://github.com/wenzel-lab/FluoPi | Green fluorescence imaging system for analyzing plates | |
| Fluorinated Oil | 3M | Novec 7500 | |
| Glass slide heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of microfluidic chip | |
| Glass Slides | |||
| Hotplate | Mechanic | For evaporating Aquapel | |
| Image analysis software | Fiji/ImageJ | 2.14.0/1.54f | |
| Incubator | Mundo Lab | MLAB Scientific / For incubation of plates and microgels | |
| Isopropanol | For cleaning glass slides | ||
| Kanamycin | 100 ug/mL concentration | ||
| L-shaped spreader | For spreading bacteria on agar plates | ||
| Master mold | Chip design on silicone or glass wafer | ||
| Microtubes | 2 mL | ||
| NaCl solution | Sodium chloride 0.9% w/v | ||
| Open-source hardware strobe-enhanced microscopy stage | https://github.com/wenzel-lab/flow-microscopy-platform | For bright-field microscopy | |
| Petri dish | Citotest | 2303-1090 | 90 x 15 mm |
| Pipette tip heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of pipette tip | |
| Plasma Cleaner | Diener Electronic | 117056 | For bonding PDMS with a glass slide |
| Plasmid pCA_Odd1 | Encodes sfGFP and kanamycin resistance | ||
| Polytetrafluoroethylene (PTFE) tubing | Adtech Polymer Engineering Ltd | ||
| Pre-mixed Luria Bertoni medium | US Biological Life Science | L1520 | |
| Propidium iodide (PI) | For staining | ||
| Raspberry Pi-based pressure and flow controller system | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller | For controlling pressure and flow rates | |
| Silicone elastomer base | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Silicone elastomer curing agent | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Spectrophotometer | For measuring absorbance | ||
| SQUID microscope | https://github.com/wenzel-lab/SQUID-bioimaging-platform | Multi-fluorescence imaging system for analyzing stained cells | |
| Sterile loop | For picking a colony and streaking plating | ||
| Surfactant | Sphere Fluidics | Pico-Surf | |
| Syringes | NIPRO | With filters and tubing | |
| Temperature-controlled shaker | Mundo Lab | DLAB HCM100-Pro | |
| Tweezer | |||
| Ultra-low gelling temperature agarose | Sigma-Aldrich | A2576-5G | For generating hydrogel beads |
| Water repelent solution (fluoroalkyl silane) | Aquapel | For treating microchannels of PDMS device |
Références
- Wein, T., Hülter, N. F., Mizrahi, I., Dagan, T. Emergence of plasmid stability under non-selective conditions maintains antibiotic resistance. Nat Commun. 10 (1), 2595(2019).
- Rodríguez-Beltrán, J., DelaFuente, J., León-Sampedro, R., MacLean, R. C., Millán, ÁS. Beyond horizontal gene transfer: The role of plasmids in bacterial evolution. Nat Rev Microbiol. 19 (6), 347(2021).
- Wein, T., Dagan, T. Plasmid evolution. Curr Biol. 30 (19), R1158-R1163 (2020).
- Chen, S., Larsson, M., Robinson, R. C., Chen, S. L. Direct and convenient measurement of plasmid stability in lab and clinical isolates of E. coli. Sci Rep. 7 (1), 4788(2017).
- Rouches, M. V., Xu, Y., Cortes, L. B. G., Lambert, G. A plasmid system with tunable copy number. Nat Commun. 13 (1), 3908(2022).
- Silva, F., Queiroz, J. A., Domingues, F. C. Evaluating metabolic stress and plasmid stability in plasmid DNA production by Escherichia coli. Biotechnol Adv. 30 (3), 691-708 (2012).
- Wang, R., et al. Construction of novel pJRD215-derived plasmids using chloramphenicol acetyltransferase (cat) gene as a selection marker for Acidithiobacillus caldus. PLoS ONE. 12 (8), e0183307(2017).
- Moragues, T., et al. Droplet-based microfluidics. Nat Rev Methods Primers. 3 (1), 32(2023).
- Vitalis, C., Wenzel, T. Leveraging interactions in microfluidic droplets for enhanced biotechnology screens. Current Opinion in Biotechnology. 82, 102966(2023).
- Sinha, N., Subedi, N., Wimmers, F., Soennichsen, M., Tel, J. A pipette-tip based method for seeding cells to droplet microfluidic platforms. J Vis Exp. (144), e57848(2019).
- Wenzel, T. Open hardware: From DIY trend to global transformation in access to laboratory equipment. PLOS Biol. 21 (1), e3001931(2023).
- Murillo, L. F. R., Wenzel, T. Welcome to the journal of open hardware. J Open Hardware. 1 (1), (2017).
- Shin, J. H., Choi, S. Open-source and do-it-yourself microfluidics. Sens Actuators B Chem. 347, 130624(2021).
- Pryszlak, A., et al. Enrichment of gut microbiome strains for cultivation-free genome sequencing using droplet microfluidics. Cell Rep Methods. 2 (1), 100137(2021).
- Pollak, B., et al. Universal loop assembly: open, efficient and cross-kingdom DNA fabrication. Biology. 5 (1), (2020).
- Volkmer, B., Heinemann, M. Condition-dependent cell volume and concentration of Escherichia coli to facilitate data conversion for systems biology modeling. PLoS ONE. 6 (7), e23126(2011).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , (2020).
- Abate, A. R., Weitz, D. A. Faster multiple emulsification with drop splitting. Lab Chip. 11 (11), 1911-1915 (2011).
- Lau, B. T. C., Malkus, P., Paulsson, J. New quantitative methods for measuring plasmid loss rates reveal unexpected stability. Plasmid. 70 (3), 353-361 (2013).
- Oellermann, M., et al. Open hardware in science: The benefits of open electronics. Integr Comp Biol. 62 (4), 1061-1075 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon