Method Article
Análise de estabilidade de plasmídeo com microfluídica de gotículas de código aberto
Neste Artigo
Resumo
Um fluxo de trabalho microfluídico acessível e de código aberto é apresentado para a análise paralelizada da retenção de plasmídeo em bactérias. Ao empregar microscopia de fluorescência para quantificar a presença de plasmídeo em microcolônias de célula única dentro de microgotículas de gel, este método fornece uma alternativa precisa, acessível e escalável à contagem tradicional de placas.
Resumo
Os plasmídeos desempenham um papel vital na biologia sintética, permitindo a introdução e expressão de genes estranhos em vários organismos, facilitando assim a construção de circuitos e vias biológicas dentro e entre as populações de células. Para muitas aplicações, a manutenção de plasmídeos funcionais sem seleção de antibióticos é fundamental. Este estudo apresenta um fluxo de trabalho microfluídico baseado em hardware aberto para analisar a retenção de plasmídeo, cultivando células únicas em microgotículas de gel e quantificando microcolônias usando microscopia de fluorescência. Essa abordagem permite a análise paralela de várias gotículas e microcolônias, fornecendo maior poder estatístico em comparação com a contagem tradicional de placas e permitindo a integração do ensaio em outros fluxos de trabalho microfluídicos de gotículas. Usando plasmídeos que expressam proteínas fluorescentes ao lado de uma coloração de DNA fluorescente não específica, colônias únicas podem ser identificadas e diferenciadas com base na perda de plasmídeo ou na expressão de marcadores fluorescentes. Notavelmente, esse fluxo de trabalho avançado, implementado com hardware de código aberto, oferece controle de fluxo preciso e gerenciamento de temperatura da amostra e do chip microfluídico. Esses recursos aumentam a facilidade de uso, robustez e acessibilidade do fluxo de trabalho. Embora o estudo se concentre na Escherichia coli como modelo experimental, o verdadeiro potencial do método está em sua versatilidade. Ele pode ser adaptado para vários estudos que requerem quantificação de sinal de fluorescência de plasmídeos ou manchas, bem como para outras aplicações. A adoção de hardware de código aberto amplia o potencial para a realização de bioanálises de alto rendimento usando tecnologia acessível em diversos ambientes de pesquisa.
Introdução
Os plasmídeos são elementos genéticos essenciais nas células procarióticas, contribuindo significativamente para a evolução microbiana por meio da transferência lateral de DNA e rápida adaptação às mudanças ambientais 1,2. Essas moléculas de DNA extracromossômicas carregam genes que fornecem características vantajosas, como resistência a antibióticos, funções metabólicas e fatores de virulência, tornando-as valiosas para pesquisas em microbiologia, biologia sintética e estudos de evolução2. No entanto, a manutenção do plasmídeo em populações de células é desafiadora devido à carga metabólica de replicação e segregação, muitas vezes resultando em perda de plasmídeo sem pressão de seleção3. Além disso, a herança estável requer mecanismos como toxina-antitoxina e sistemas de partição, adicionando complexidade à manutenção do plasmídeo. Avaliar a estabilidade do plasmídeo sob condições variadas é crucial tanto para a pesquisa fundamental quanto para aplicações práticas que utilizam plasmídeos como elemento primáriode pesquisa 4,5. A maioria dos métodos atuais para avaliar a estabilidade do plasmídeo tem limitações significativas: os métodos baseados em citometria de fluxo fornecem dados indiretos em nível populacional, exigem equipamentos caros e carecem de visualização direta das colônias6. Os métodos transcriptômicos e proteômicos em massa são caros, fornecem apenas respostas celulares médias e não podem quantificar diretamente a retenção de plasmídeo em colônias individuais6. Métodos tradicionais como diluição serial e passagem são simples, mas demorados e carecem de precisão e representabilidade7. No geral, inferir ou projetar quantitativamente o número de colônias que retêm um plasmídeo funcional específico ao longo do tempo ou pressões seletivas continua sendo um desafio.
Para enfrentar esses desafios, um novo fluxo de trabalho microfluídico utilizando instrumentos de pesquisa de hardware aberto é apresentado para quantificar sinais fluorescentes em várias colônias isoladas de bactérias, usando Escherichia coli como modelo. Este método permite alto rendimento e análise precisa da retenção de plasmídeo em várias condições ou pressões seletivas. A análise de resolução de célula única fornece um método preciso para manipular colônias isoladas, produzindo dados sensíveis sobre a quantificação de plasmídeos que podem ajudar a avaliar as taxas de retenção e perda4.
A microfluídica, particularmente a microfluídica de gotículas, surgiu como uma ferramenta poderosa para encapsular e manipular células individuais em ambientes controlados8. Especificamente, as gotículas de microgel podem encapsular células únicas para alto rendimento e análise precisa sem a necessidade de manter gotículas suspensas no óleo9, permitindo um estudo controlado da dinâmica do plasmídeo em um microambiente definido. Após o encapsulamento de suspensões celulares diretamente de uma ponta de pipeta10, técnicas de fluorescência podem ser usadas para monitorar o crescimento de microcolônias dentro de gotículas, permitindo uma análise detalhada da retenção e segregação de plasmídeos sob diferentes pressões de seleção3.
As vantagens desse método em relação às técnicas tradicionais de cultura a granel incluem maior precisão, variabilidade reduzida e capacidade de realizar análises de alto rendimento. A tecnologia microfluídica de código aberto supera as limitações de sistemas proprietários caros, como questões de acessibilidade, adaptabilidade e manutenção, que muitas vezes dificultam o progresso da pesquisa 11,12,13. Ao demonstrar como aplicar o fluxo de trabalho experimental avançado de análise de retenção de plasmídeo em microgéis com instrumentação de código aberto, um método acessível e confiável é fornecido para pesquisa em biologia de plasmídeos, aplicações de biologia sintética e técnicas de análise de gotículas microfluídicas.
Em resumo, este artigo apresenta um método acessível para avaliar quantitativamente a retenção de plasmídeos em E. coli com alto poder estatístico. Os recursos desse método o tornam uma ferramenta valiosa para avançar na compreensão da biologia do plasmídeo e melhorar as aplicações da biologia sintética.
Protocolo
A Figura 1fornece uma visão geral esquemática para avaliar a estabilidade do plasmídeo em E. coli. Os detalhes dos reagentes e do equipamento utilizado estão listados na Tabela de Materiais. Os dados brutos e os scripts de visualização estão disponíveis em https://doi.org/10.17605/OSF.IO/6YWJK.
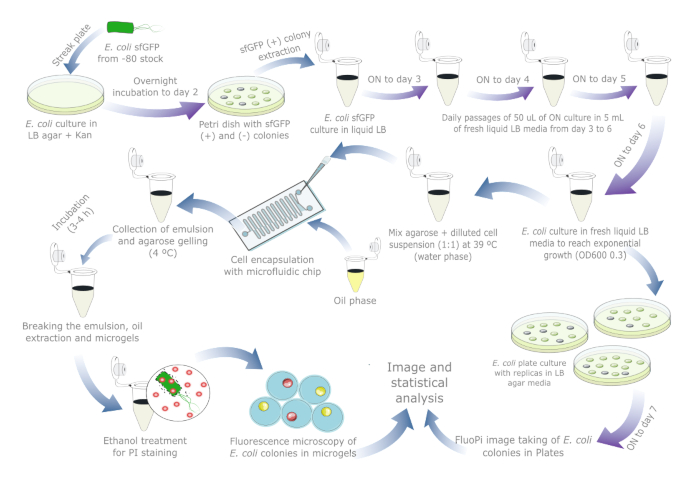
Figura 1: Protocolo diário para avaliar a estabilidade do plasmídeo em E. coli. As setas azuis indicam etapas durante o dia e as setas roxas indicam incubação durante a noite. Cada incubação de líquido e ágar foi realizada a 37 °C ao lado de um tubo/placa de controle negativo separado. Observe que a preparação e as passagens da cultura de células não são necessárias para amostras do mundo real nas quais a perda de plasmídeo já pode ter ocorrido, portanto, o protocolo deve ser reduzido para um ou dois dias se uma cultura de referência em placa for incluída. Clique aqui para ver uma versão maior desta figura.
1. Preparação microfluídica de chips
NOTA: Neste protocolo, diferentes designs de chips comerciais ou personalizados podem ser usados para encapsulamento de células que é capaz de gerar gotículas de água em óleo com menos de 100 μm de diâmetro em um regime de gotejamento. Para este estudo, o chip foi projetado e fabricado (ver dados do artigo https://doi.org/10.17605/OSF.IO/6YWJK) seguindo o mesmo método de design e fabricação relatado em um relatório publicado anteriormente14.
- Obtenha ou prepare um chip microfluídico PDMS-on-glass usando um molde mestre projetado para geração de microgotículas de gel.
- Injete uma solução repelente de água (fluoroalquil silano) no chip para tornar os microcanais internos hidrofóbicos. Infundir a solução nas entradas e garantir que todos os canais estão cheios de fluido. Deixe os canais cheios descansarem por cerca de 30 a 60 s.
- Remova a solução do dispositivo expelindo o ar para os microcanais internos. Use uma seringa de ar vazia para lavar o ar e um pano absorvente sobre as outras portas para evitar respingos.
- Asse o dispositivo tratado a 65 °C por 15 min em uma placa de aquecimento para evaporar o excesso de solução. Como alternativa, guarde o dispositivo na geladeira (4 °C) durante a noite.
NOTA: O chip microfluídico está pronto para uso. O protocolo pode ser pausado aqui.
2. Preparação da amostra
- Colheita de células
NOTA: A cultura de células em gotículas pode fornecer dados relevantes sobre a dinâmica do plasmídeo. Como um modelo experimental de bactérias, uma cepa de E. coli TOP10 é usada com o plasmídeo pCA_Odd1 (ver dados depositados https://doi.org/10.17605/OSF.IO/6YWJK) que codifica uma proteína fluorescente verde superpasta (sfGFP) e resistência à canamicina15. Bactérias, plasmídeos e meios de crescimento podem variar de acordo com o sistema experimental.- Preparar o ágar Luria Bertoni (LB) dissolvendo 25 g de LB pré-misturado e 12 g de ágar em 800 ml de água destilada (dH2O). Além disso, prepare o meio LB líquido dissolvendo 25 g de LB pré-misturado em 800 mL de dH2O. Autoclave as soluções e deixe-as esfriar até cerca de 60 ° C. Mude para um ambiente estéril, como uma coifa de fluxo, para as próximas etapas.
NOTA: O pó LB pré-misturado contém 10 g de triptona, 5 g de extrato de levedura e 10 g de NaCl. Meios de ágar LB auto-misturados também podem ser usados. - Adicionar 50 μL de canamicina (preparada a 100 mg/ml) a 50 ml de ágar LB líquido (concentração final: 100 μg/ml). Misturar a solução invertendo o tubo algumas vezes.
- Despeje cerca de 15 mL de ágar LB líquido por placa de Petri (90 mm x 15 mm). Preparar duas placas: uma para o controlo negativo (monitorização da contaminação) e outra para a cultura experimental. Deixe a solução de ágar LB esfriar e solidificar até que a cor mude de escura para clara em ambas as placas.
- Use uma alça estéril para espalhar a cepa de E. coli na placa de cultura usando a técnica da placa de listra. Coloque o estoque de glicerol E. coli de volta a –80 °C imediatamente após o uso. Feche ambas as placas e incube-as durante a noite a 37 °C (dia 1).
- No dia 2, verifique a placa de controle negativo para qualquer colônia como um sinal de contaminação (em caso afirmativo, repita as etapas de 2.1.4.). Identifique colônias fluorescentes únicas na placa de E. coli usando um transiluminador de luz azul para preparar um estoque de cultura líquida.
- Em condições estéreis, abra a placa e use uma alça estéril ou ponta de pipeta de 200 μL para pegar a colônia selecionada e transferi-la para um tubo de cultura com 5 mL de meio LB líquido fresco e 5 μL de canamicina (preparado a 100 mg / mL). Preparar um tubo de controlo negativo sem inoculação para a monitorização da contaminação. Incubar os tubos de cultura a 37 °C durante a noite, agitando a 220 rpm.
- Passe a amostra por mais 3 dias em meio sem antibióticos para simular as condições sob as quais a perda de plasmídeo pode ocorrer. Nos dias 3, 4 e 5, transfira 50 μL da cultura noturna para um novo tubo de cultura contendo 5 mL de LB líquido sem antibióticos. Incubar a 37 °C durante a noite, agitando a 220 rpm. Repita esta etapa até o dia 6, obtendo uma cultura final de 5 mL após quatro passagens totais.
- Transfira 50 μL da cultura noturna final para um novo tubo de cultura contendo 2 mL de LB líquido. Deixe a cultura atingir uma densidade óptica (OD600) de 0,3 usando um espectrofotômetro (3–4 h aproximadamente).
NOTA: A cultura fresca deve ser usada antes que o OD600 aumente para garantir que as bactérias estejam na fase de crescimento logarítmico. Se a concentração desejada não for alcançada, repita a partir da etapa 2.1.8.
- Preparar o ágar Luria Bertoni (LB) dissolvendo 25 g de LB pré-misturado e 12 g de ágar em 800 ml de água destilada (dH2O). Além disso, prepare o meio LB líquido dissolvendo 25 g de LB pré-misturado em 800 mL de dH2O. Autoclave as soluções e deixe-as esfriar até cerca de 60 ° C. Mude para um ambiente estéril, como uma coifa de fluxo, para as próximas etapas.
- Mistura de célula-agarose para encapsulamento
NOTA: O controle de concentração é essencial para garantir o encapsulamento de gotículas de célula única. A concentração de células necessária pode ser calculada para uma taxa de encapsulamento alvo e um volume específico de gotículas, conforme mostrado no exemplo a seguir:
Volume de gotículas (gota V): Se gotículas de 50 μm forem geradas
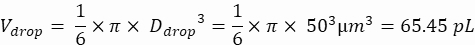
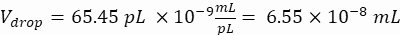
Células desejadas por gotícula (Cpd): Uma célula por cinco gotículas em média (0,2 células/gota)
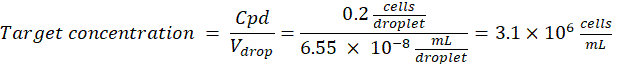
Factor de diluição: A concentração inicial da célula é obtida na etapa 2.1.8
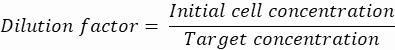
Aqui, gotículas de 50 μm ou 100 μm (sem divisão de gotículas) (65–520 pL) são geradas onde cerca de uma célula por cinco gotículas é encapsulada, ou 1,6 células por gotícula sem divisão. Para E. coli, use o fator de conversão de 1 unidade OD600 ≈ 7,8 x 108 células/mL16. Multiplique o valor OD600 da etapa 2.1.8 pelo fator de conversão para obter a concentração inicial (células/mL) da cultura.- Ressuspender E. coli da massa de cultura preparada em LB líquido sem canamicina a uma concentração de 6,2e+6 células/ml (concentração alvo para gotículas de 50 μm de diâmetro). Mantenha a suspensão bacteriana em temperatura ambiente até misturar com agarose.
NOTA: Os antibióticos suprimirão a formação de microcolônias negativas, por isso é essencial excluí-las do meio para experimentos de perda de plasmídeo. - Prepare agarose de temperatura gelificante ultrabaixa aquecendo-a a 90 °C em LB líquido a uma concentração de 2% (p/v). Agite a mistura por 10 min em um agitador com temperatura controlada.
- Reduza a temperatura do termo-agitador para 39 °C para arrefecer a solução de agarose. Paralelamente, coloque o tubo de suspensão de bactérias no termo-agitador por 4 min para aquecê-lo a 39 °C.
- Misture as bactérias e as suspensões de agarose na proporção de 1:1 para obter uma concentração de agarose de 1% (p/v) com suspensão celular a 3,1e+6 células/mL. Preparar a solução de controlo negativo (monitorização da contaminação) com a mesma concentração de agarose, utilizando LB líquido em vez da suspensão bacteriana.
NOTA: A suspensão de células de agarose deve ser usada rapidamente para evitar mudanças de concentração devido ao crescimento de bactérias. Mantenha o termoagitador a 39 °C para manter o líquido de agarose até ser carregado no aquecedor de ponta para geração de gotículas.
- Ressuspender E. coli da massa de cultura preparada em LB líquido sem canamicina a uma concentração de 6,2e+6 células/ml (concentração alvo para gotículas de 50 μm de diâmetro). Mantenha a suspensão bacteriana em temperatura ambiente até misturar com agarose.
3. Cultivo de colônia única de alto rendimento
- Configuração experimental
- Construa ou obtenha a plataforma de fluxo de código aberto completa (consulte https://doi.org/10.17605/OSF.IO/6YWJK), incluindo drivers de pressão de gás e sensores de fluxo. Como alternativa, crie o estágio de microscopia aprimorada por estroboscópio de hardware de software livre mais simples (consulte as instruções de compilação https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ e repositório do projeto https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage).
NOTA: Também é possível usar uma configuração microfluídica tradicional com um microscópio comercial, câmera de alta velocidade e recursos de aquecimento de amostras. - Integre o sistema de controlador de pressão e fluxo baseado em Raspberry Pi de código aberto, conforme documentado no repositório designado (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller).
NOTA: O controle do módulo é ilustrado e copiado para o repositório de dados (https://doi.org/10.17605/OSF.IO/6YWJK. Como alternativa, controladores de pressão tradicionais ou bombas de seringa de alto torque podem ser empregados. - Inclua uma lâmina de vidro e aquecedores de ponta de pipeta (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring) na configuração para controlar a temperatura da amostra de célula de agarose à medida que ela entra no chip.
NOTA: Esses aquecedores, ilustrados no repositório de dados (https://github.com/wenzel-lab/flow-microscopy-platform e https://doi.org/10.17605/OSF.IO/6YWJK), são um recurso importante do sistema controlador de código aberto que permite o trabalho com agarose e podem não estar disponíveis em outros sistemas comerciais.
- Construa ou obtenha a plataforma de fluxo de código aberto completa (consulte https://doi.org/10.17605/OSF.IO/6YWJK), incluindo drivers de pressão de gás e sensores de fluxo. Como alternativa, crie o estágio de microscopia aprimorada por estroboscópio de hardware de software livre mais simples (consulte as instruções de compilação https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ e repositório do projeto https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage).
- Encapsulamento de célula única
- Posicione o chip microfluídico no estágio de microscopia aprimorada por estroboscópio, garantindo que a junção de geração de gotículas (interseção das fases aquosa e oleosa) seja visível.
- Ajuste o aquecedor da ponteira da pipeta e o aquecedor da lâmina de vidro para 40 °C usando a interface do software de controle.
- Use uma seringa com tubo e plugue PDMS para carregar a mistura de agarose a 1% com suspensão celular em uma ponta de pipeta de 200 μL. Insira a ponta no aquecedor de ponta e coloque-a na entrada da fase aquosa no chip microfluídico. Troque o selo PDMS da ponta por um conectado à tubulação do sistema de controle de fluxo e inicie a infusão da suspensão celular.
- Insira a extremidade do tubo de saída em um tubo de resíduos e defina taxas de fluxo ou pressões de duas fases na interface do usuário para fornecer fluido lentamente ao canal microfluídico. Use 200 μL / h (180 mbar) para a fase aquosa e 1700 μL / h (320 mbar) para a fase oleosa. Aguarde 1 min para a estabilização da geração de gotículas.
NOTA: Os valores de pressão dependem dos tamanhos dos canais do projeto do cavaco, e os valores de fluxo podem ter que ser ajustados para diferentes projetos de junção de geração de gotículas. - Quando a geração de gotículas estiver estável, transfira os resíduos e a tubulação de coleta para o tubo de coleta. Continuar coletando gotículas até que o reservatório de amostra esteja vazio. Repetir os passos 3.2.3 a 3.2.5 para encapsular a solução de controlo negativo do passo 2.2.4.
NOTA: A coleta de amostras deve ser concluída em 15 minutos. - Armazenar os tubos de recolha no gelo durante a produção das gotículas ou colocá-los a 4 °C após a experiência durante 1 h para permitir que a agarose gelifique no interior das gotículas.
NOTA: O chip microfluídico pode ser reutilizado se os microcanais permanecerem desobstruídos e a mesma suspensão estiver carregada. Descarte a ponta da pipeta após concluir a geração da emulsão (etapas 3.2.3 a 3.2.5).
- Crescimento da colônia e liberação da emulsão
- Transferir as microgotículas de gel contendo bactérias e o controlo negativo em gotículas para uma câmara de incubação regulada para 37 °C.
- Incube as microgotículas por pelo menos 4 h, ou durante a noite, para permitir o crescimento suficiente da colônia. Confirmar que o controlo negativo não apresenta sinais de contaminação por microscopia de campo claro.
- Para liberar as colônias da emulsão, remova o máximo de óleo possível de baixo da emulsão de microgotículas de gel usando uma pipeta ou uma seringa com uma agulha (aqui, foi usada agulha tamanho 21 G).
- Transfira 50 μL das microgotículas de gel para um novo microtubo e armazene-o a 4 °C para posterior análise de gotículas. À emulsão restante, adicione uma mistura 1:1 de óleo fluorado com 1H,1H,2H,2H-perfluoro-1-octanol (PFO) em um volume igual à emulsão.
- Adicione aproximadamente 200 μL de tampão salino tampão fosfato (PBS) ou 0,9% p / v de tampão NaCl, em cima da emulsão. Vortex a mistura e gire brevemente em uma centrífuga de velocidade fixa.
- Remova cuidadosamente a fase oleosa da parte inferior da interface líquida e descarte 100 μL de PBS da parte superior. Repita as etapas 3.3.4–3.3.5 para obter microgéis lavados em tampão PBS com o mínimo ou nenhum resíduo de óleo.
NOTA: Os microgéis se depositam na interface do líquido; Evite removê-los junto com a fase oleosa.
4. Análise de colônia única
- Coloração celular
NOTA: Muitas combinações diferentes de manchas funcionam para este protocolo. Em essência, uma coloração de DNA ou parede celular que tenha uma cor distinta da proteína fluorescente codificada pelo plasmídeo deve ser escolhida e que possa ser analisada usando as combinações de filtros disponíveis no microscópio de fluorescência. Aqui, o DNA das células foi corado com iodeto de propídio (PI) para distinguir sua fluorescência da proteína fluorescente verde codificada nos plasmídeos, mas muitas outras colorações de DNA podem ser usadas.
CUIDADO: O IP é um potencial cancerígeno e deve ser manuseado com equipamento de proteção individual adequado. Descarte o corante com segurança e em conformidade com os regulamentos locais.- Centrifugue os microgéis lavados a aproximadamente 80 x g por 5 min em temperatura ambiente. Descarte o sobrenadante usando uma pipeta.
- Transferir 50 uL dos microgéis para um novo microtubo para servir como controle negativo para o tratamento com etanol.
NOTA: A coloração PI só entra em células com membranas comprometidas, como as afetadas pelo tratamento com etanol. - Adicione um volume igual de etanol a 70% aos microgéis restantes e misture brevemente com um vórtice. Incube em temperatura ambiente por 15 min para permeabilizar as membranas bacterianas para coloração PI. Repita a etapa 4.1.1.
- Adicione um volume igual de 0,9% p/v de NaCl aos microgéis e rapidamente vórtice. Repita a etapa 4.1.1.
- Adicione 2 μL de PI (1 mg / mL) a ambas as amostras de microtubos. Misture bem e incube no escuro em temperatura ambiente por 15 min.
NOTA: Se o controle negativo apresentar algum sinal fluorescente vermelho via microscopia de fluorescência, a integridade das colônias pode ter sido comprometida durante sua exposição a outras soluções em etapas anteriores.
- Microscopia
NOTA: Gotículas e microgéis são fotografados com um microscópio de epifluorescência invertida para obter a distribuição do tamanho das gotículas e a fluorescência das colônias bacterianas em microgéis (Figura 2). Aqui, uma plataforma de microscopia invertida de código aberto (https://github.com/wenzel-lab/SQUID-bioimaging-platform)17 é usada com uma objetiva de 10x 0,3NA, uma matriz de LED branco para iluminação de campo claro e um LED de 470 nm para excitação. Microscópios de epifluorescência comerciais podem ser usados para obter imagens de gotículas e microgéis. A calibração é necessária porque a iluminação e os filtros variam dependendo do modelo, marca e proteínas fluorescentes usadas.- Transfira 2 μL de microgotículas de gel para uma lâmina de câmara de imagem e adicione 5 μL de óleo fluorado para ajudar a formar uma monocamada de gotículas para uma imagem ideal.
NOTA: Câmaras de contagem de células ou câmaras microfluídicas simples podem ajudar a espalhar a emulsão e retardar o processo de secagem. - No microscópio, ative a iluminação de matriz de LED branca de cima para imagens de campo claro. Monte a lâmina preparada, concentre-se na amostra e localize uma monocamada de gotículas. Capture uma imagem de campo claro.
- Sem mover a amostra, capture uma imagem de fluorescência das colônias mudando para o LED de 470 nm para excitação. Ajuste a roda do filtro para alinhar com o filtro de comprimento de onda verde para imagens sfGFP. Examine todas as áreas que contêm monocamadas de gotículas e repita as etapas 4.2.2 a 4.2.3 para garantir a robustez estatística na análise de gotículas.
- Transfira 2 μL dos microgéis corados para um chip de câmara de imagem e 5 μL de NaCl a 0,9% p/v para ajudar a formar uma monocamada de microgéis. Vede a entrada e a saída do chip para evitar a evaporação durante a imagem.
NOTA: A evaporação pode afetar a colocalização das etiquetas fluorescentes. As câmaras microfluídicas são recomendadas para imagens prolongadas. - Repita as etapas 4.2.2 a 4.2.3. Ajuste o filtro de intervalo de comprimento de onda vermelho para imagens PI e capture as respectivas imagens.
- Depois de concluir a imagem em um local, encontre a próxima área adequada na lâmina com uma monocamada de microgéis e repita o processo de imagem para garantir robustez estatística e análise abrangente da amostra.
- Transfira 2 μL de microgotículas de gel para uma lâmina de câmara de imagem e adicione 5 μL de óleo fluorado para ajudar a formar uma monocamada de gotículas para uma imagem ideal.
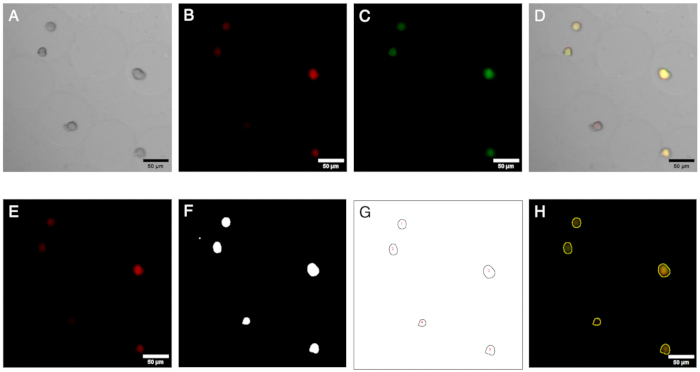
Figura 2: Imagens de microscopia e sua análise. Imagem de fluorescência e análise de colônias em microgéis. (A-C) Canais de imagem adquiridos por microscopia de campo claro e fluorescência com o microscópio invertido. A imagem composta (D) mostra a presença de uma colônia negativa (apenas fluorescência vermelha) dentro dos microgéis. (E-H) Resultados do fluxo de trabalho de análise de imagem. Ao gerar ROIs, as colônias podem ser identificadas em canais vermelhos e verdes, e os sinais podem ser quantificados para definir a presença de colônias negativas. Barras de escala: 50 μm. Clique aqui para ver uma versão maior desta figura.
- Análise de imagem
NOTA: Para analisar os sinais de fluorescência das colônias encapsuladas e identificar eventos raros, imagens de campo claro e fluorescência podem ser processadas com Fiji/ImageJ (Figura 3). Essas etapas podem ser implementadas em um script de macro e os valores dos parâmetros podem variar dependendo da configuração óptica.- Abra as imagens dos canais verde e vermelho. Defina uma região retangular de interesse (ROI) começando nas coordenadas (500, 500) com largura e altura de 2000 pixels cada.
NOTA: Esses parâmetros se aplicam a imagens de 3000 x 3000 pixels, e o ROI define a área com melhor iluminação. - Corte a imagem para o ROI retangular definido. Subtraia um valor constante de 10 da intensidade de cada pixel e aplique um filtro de suavização à imagem para reduzir o ruído de fundo e tornar os objetos de interesse mais distintos.
NOTA: Somente esta região será usada para análise posterior. - Duplique a imagem do canal vermelho e converta-a em uma máscara binária para identificar os eventos raros relacionados ao plasmídeo. Defina os valores de limite entre 10 e 255. Os pixels dentro desse intervalo serão considerados em primeiro plano (objetos de interesse), enquanto outros serão tratados como plano de fundo.
- Execute operações morfológicas para fechar pequenas lacunas e preencher quaisquer lacunas dentro dos objetos de interesse. Aplique o algoritmo de bacia hidrográfica para separar objetos sobrepostos dentro da máscara binária.
- Analise as partículas na máscara binária. Considere apenas partículas com tamanho superior a 30 pixels e circularidade entre 0,50 e 1,00. Os resultados são resumidos e adicionados à tabela de resultados. Salve o conjunto de ROIs detectados pela análise de partículas para visualização adicional.
- Exiba os ROIs salvos nas imagens dos canais verde e vermelho. Meça a intensidade ou outras propriedades dos ROIs nessas imagens. Registre as medições na tabela de resultados e salve os resultados em arquivos CSV separados para análise estatística adicional.
- Abra as imagens dos canais verde e vermelho. Defina uma região retangular de interesse (ROI) começando nas coordenadas (500, 500) com largura e altura de 2000 pixels cada.
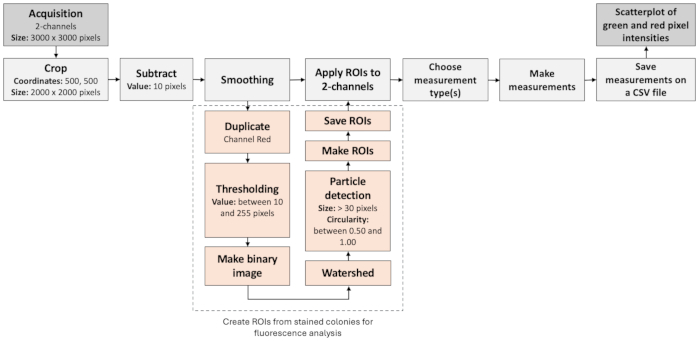
Figura 3: Fluxo de trabalho de análise de imagem para identificar colônias negativas. A figura ilustra um fluxo de trabalho passo a passo para processar e avaliar automaticamente imagens de fluorescência. O fluxo de trabalho é baseado na colocalização de rótulos de fluorescência, bem como na análise de partículas. Clique aqui para ver uma versão maior desta figura.
5. Ensaio de comparação de placa de ágar
NOTA: Para comparar o método de gotículas com um ensaio tradicional em placa, a quantificação de colônias fluorescentes da mesma cepa de E. coli foi obtida na etapa 2.1.8. foi realizado usando placas de Petri. Isso serviu como um método de controle analógico para medir a estabilidade do plasmídeo sfGFP. Veja também a ilustração do método na Figura 1.
- Prepare o ágar LB (sem antibiótico) conforme descrito na etapa 2.1.1 da seção Coleta de células. Arrefecer o ágar-ágar a uma temperatura inferior a 60 °C e homogeneizar.
- Em um ambiente estéril, despeje 15 mL do meio de ágar em cada placa de Petri. Deixe as placas solidificarem com as tampas parcialmente abertas até que estejam prontas para uso.
- Inocular três placas com 10 μL cada de uma cultura de E. coli em meio LB com um OD600 de 0,007 ± 0,002. Espalhe uniformemente usando um espalhador em forma de L. Incubar as placas a 37 °C, fechadas e invertidas. Prepare uma placa de controle sem bactérias nas mesmas condições.
- Após 24 h, capture imagens de fluorescência das placas usando o FluoPi, um sistema de imagem de fluorescência de código aberto (https://github.com/RudgeLab/FluoPi)15.
NOTA: O FluoPi consiste em uma câmera Raspberry Pi com uma luz de excitação azul em um comprimento de onda centrado em 470 nm e filtros de excitação e emissão de acrílico. - Conte manualmente as colônias fluorescentes e não fluorescentes em cada placa usando as imagens capturadas.
Resultados
Validação do encapsulamento celular e formação de microcolônias
O encapsulamento celular pode ser confirmado visualmente realizando microscopia de campo claro nas microgotículas de gel antes de quebrar a emulsão e lavar os microgéis. Um resultado representativo da emulsão nesta etapa é mostrado na Figura 4.
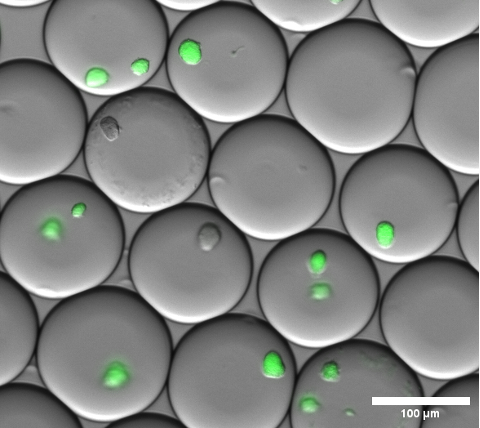
Figura 4: Seção de uma imagem de sobreposição de microscopia de fluorescência. Após incubação durante a noite, microcolônias representativas de sfGFP expressando colônias de E. coli dentro de microgotículas de gel. Foi utilizada uma objetiva microscópica com aumento de 10x e 0,30 NA. Barra de escala: 100 μm. Clique aqui para ver uma versão maior desta figura.
Resultados da análise de imagem
Uma vez que os microgéis são corados e de campo claro, bem como os canais de fluorescência adquiridos em várias posições, as colônias identificadas como negativas nas imagens originais podem ser visualizadas (ver Figura 5A). Os dados extraídos de todas as imagens de um experimento podem ser plotados para mostrar a taxa de fluorescência de várias colônias, destacando aquelas que perderam a fluorescência codificada por plasmídeo (ver Figura 5B). Os resultados indicam que 100 colônias perderam seu plasmídeo ou funcionalidade de plasmídeo de um total de 2785 microcolônias analisadas, correspondendo a 3,6%.
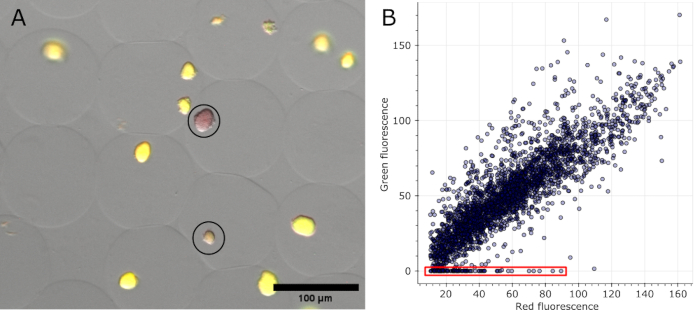
Figura 5: Quantificação de microcolônias negativas. (A) Seção de uma imagem de sobreposição de microscopia de fluorescência. Após a remoção e coloração do óleo, colônias representativas em microgéis expressando sfGFP e duas colônias negativas mostraram a fluorescência vermelha da coloração de DNA (circulada em preto). Barra de escala: 100 μm. (B) Gráfico de dispersão dos valores de fluorescência de microcolônias individuais extraídas de 16 imagens de microscopia multicanal. As colônias sem fluorescência verde foram contadas como negativas, conforme indicado em vermelho no gráfico. Clique aqui para ver uma versão maior desta figura.
Quantificação de placas de ágar
As imagens das placas triplicadas são mostradas na Figura 6, com colônias não fluorescentes indicadas por setas brancas. A primeira placa (Figura 6A) exibiu um total de 213 colônias, das quais 1 não era fluorescente. A segunda placa (Figura 6B) tinha um total de 49 colônias, sem colônias não fluorescentes. A terceira placa (Figura 6C) mostrou um total de 252 colônias, 6 das quais não eram fluorescentes. Esses resultados correspondem a uma taxa média de perda de plasmídeo de colônia de 2,3%, com um grande desvio padrão de 3,2.
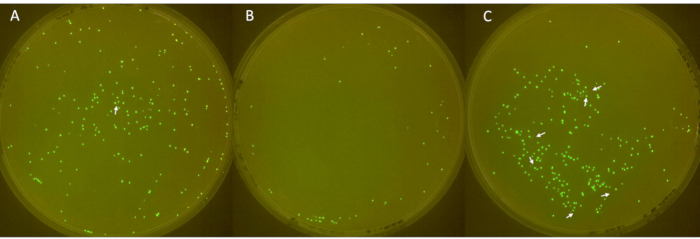
Figura 6: Identificação de colônias negativas em placas. (A-C) Colônias fluorescentes e não fluorescentes de E. coli em placas de ágar LB (diâmetro: 90 mm, altura: 15 mm). O inóculo, derivado de E. coli com sfGFP a partir do estoque de -80 °C, foi listrado no dia 1, cultivado com antibiótico no dia 2 e diluído 1:100 diariamente do dia 3 ao 6 para permitir a perda de plasmídeo. As colônias foram incubadas a 37 ° C por 24 h e fotografadas em uma câmara FluoPi. As colônias não fluorescentes foram aprimoradas com GIMP e indicadas com setas brancas. Clique aqui para ver uma versão maior desta figura.
Discussão
Um método baseado em microgotículas de gel é demonstrado para identificar e quantificar efetivamente colônias com e sem expressão genética codificada por plasmídeo de proteínas fluorescentes, como sfGFP. As colônias que não expressam suficientemente o produto do plasmídeo são identificadas usando uma coloração de DNA fluorescente (aqui, iodeto de propídio) que cora todas as colônias e apresenta um comprimento de onda de emissão diferente. Essa integração de microfluídica de gotículas, gelificação e microscopia de fluorescência, utilizando tecnologia de código aberto, permite a execução de um fluxo de trabalho avançado em muitos ambientes de pesquisa11,13. A geração bem-sucedida de microgotículas de gel permite fluxos de trabalho avançados de biologia molecular de célula única, incluindo lise celular, amplificação de genoma único, telas de interação celular metabólica, troca de mídia e muito mais 8,9. Essas vantagens são usadas neste protocolo para cultivar, corar e analisar microcolônias de maneira mais escalável do que nos ensaios tradicionais baseados em placas.
Etapas críticas
O processo de encapsulamento é uma parte crítica e delicada do protocolo. O controle preciso das concentrações, taxas de fluxo e pressões dos ingredientes é necessário para gerar microgéis uniformes dentro de uma faixa de tamanho específica e controlar o número médio de células por gota. Além disso, manter a concentração e a temperatura da mistura célula-agarose evita a aglomeração ou gelificação prematura. O controle de temperatura da suspensão de células de agarose líquida em uma ponta de pipeta é uma implementação particularmente vantajosa de nossa estação de trabalho de microfluídica de hardware de código aberto que fornece uma geração de microgel muito mais fácil e robusta em comparação com os esforços para controlar a temperatura das bombas de seringa e tubulações. Como as células são misturadas com o meio de crescimento de agarose antes do encapsulamento e cultivo, os microgéis de agarose devem ser gerados rapidamente para evitar grandes mudanças na concentração celular. Para este propósito, um design de chip microfluídico de divisão de gotículas inspirado em Abate et al. foi otimizado18.
Modificações e solução de problemas
Várias calibrações e modificações foram necessárias para refinar o protocolo original. O encapsulamento da agarose é muito mais desafiador do que as gotículas regulares de água em óleo, exigindo o projeto de um sistema para manter a agarose no estado líquido, garantindo que o fluxo da fase aquosa atinja uma faixa homogênea de tamanho de partícula. Mudanças na viscosidade da agarose devido à gelificação afetam a taxa de fluxo, levando a tamanhos de partícula maiores. A microscopia requer uma seleção cuidadosa de filtros e fontes de luz para garantir sinais de excitação e emissão não sobrepostos para uma diferenciação clara. Inicialmente, o DAPI foi escolhido para corar bactérias, mas seu sinal de emissão se sobrepôs ao sfGFP, fazendo com que o sfGFP fosse detectado no canal de detecção azul. Mudamos para PI porque sua emissão é bem separada do sfGFP em comprimentos de onda longos (luz vermelha).
Enquanto a perda de plasmídeo foi quantificada usando o método proposto, o plasmídeo sfGFP usado foi inesperadamente estável, exibindo quase nenhum caso de perda de plasmídeo na primeira geração de células cultivadas sem antibióticos, mesmo sob condições de estresse, como meio pH9 e incubação a 40 ° C. Essa observação é consistente com os achados de outros grupos de pesquisa 1,19. A estabilidade do plasmídeo limitou a demonstração das capacidades completas de quantificação do método para as gerações iniciais de cultura de células, mas demonstrou que o método é sensível o suficiente para detectar até mesmo pequenas diferenças na retenção do plasmídeo. A observação de alta estabilidade de plasmídeo nas primeiras gerações tem uma implicação importante para telas microfluídicas de gotículas usando um ensaio de seleção negativa, como a inibição de bactérias-alvo. Isso significa que a perda de plasmídeo de alvos de seleção é uma fonte baixa de resultados de seleção falso-positivos. Como as telas microfluídicas de gotículas normalmente excedem outras telas de alto rendimento, como fluxos de trabalho de robôs de pipetagem, em ordens de grandeza no rendimento, esses eventos raros precisam ser avaliados e levados em consideração.
Limitações
Apesar de suas vantagens, existem limitações no método apresentado. A fabricação de dispositivos microfluídicos requer experiência e atenção meticulosa aos detalhes, bem como controle experimental rígido das taxas de fluxo para garantir a eficiência do encapsulamento determinístico. Esses aspectos podem exigir otimização para diferentes configurações experimentais. Embora esse método dependa da microscopia de fluorescência para detecção de sinal, necessitando de acesso a equipamentos de imagem adequados, esse equipamento pode ser fabricado usando hardware de código aberto, tornando-o mais acessível. Além disso, os microgéis podem ser processados em citometria de fluxo comercial com bicos grandes, melhorando ainda mais a acessibilidade e o rendimento experimental. Classificadores de gotículas também podem ser usados para esta análise citométrica.
Além disso, embora o método seja projetado para detectar sinais fluorescentes de plasmídeos, manchas ou outros marcadores, ele é limitado a células que podem ser marcadas com fluorescência, o que pode não se aplicar a todas as cepas bacterianas ou condições experimentais. No entanto, o método pode ser adaptado para incorporar outros tipos de microscopias, como contraste de fase ou microscopia de campo claro, permitindo aplicações de fenotipagem além da fluorescência. Além disso, pode ser combinado com técnicas espectroscópicas como FTIR ou espectroscopia Raman, expandindo suas capacidades para analisar composições químicas e informações estruturais das células encapsuladas. Essas adaptações ampliam o alcance de sua aplicabilidade, tornando-o uma ferramenta versátil para diversos ambientes de pesquisa.
Significado e aplicações
Os ensaios tradicionais de perda de plasmídeo19 não permitem uma boa quantificação da proporção de células que perderam sua expressão, informação que pode ser muito importante no desenho de métodos experimentais e em diversas aplicações biológicas. Normalmente, os tipos de colônias são enumerados em ensaios de placa de ágar, onde colônias isoladas bem definidas podem ser obtidas, conforme demonstrado na Figura 4. No entanto, as colônias sobrepostas são difíceis de identificar com confiança; Em nossas mãos, nem sempre obtemos uma densidade de colônia ideal, e muitas placas são necessárias para obter boas estatísticas de eventos de perda de plasmídeo de baixa frequência. O método proposto oferece uma abordagem mais robusta para quantificar com precisão os sinais fluorescentes provenientes de colônias isoladas com um número maior de colônias do que os métodos análogos da placa de ágar porque, em microgotículas, as colônias se desenvolvem separadamente, são menores e são fáceis de carregar em câmaras de imagem, permitindo a quantificação baseada em microscopia ou citometria de fluxo de grandes números de colônias. Isso pode melhorar significativamente a representação estatística do método e permitir a integração em outros fluxos de trabalho de microgotículas de gel.
O uso de hardware de código aberto 11,20 permite que os pesquisadores personalizem o design da estação de trabalho microfluídica e ajustem com precisão o fluxo rat; portanto, o tamanho da partícula suporta vários tipos de células e condições experimentais. Essa flexibilidade se estende à incorporação potencial de outros tipos de microscopia, como contraste de fase ou espectroscopia, ampliando a aplicabilidade do método. A capacidade do método de avaliar a estabilidade do plasmídeo sob várias condições é crucial para aplicações que requerem retenção de plasmídeo sem seleção de antibióticos, sob condições particulares de estresse ou várias gerações de cultura. A versatilidade e adaptabilidade do método apresentado o tornam valioso para diversas aplicações de pesquisa em áreas como biologia sintética, monitoramento ambiental e diagnóstico clínico2.
Divulgações
Os autores declaram que nenhum interesse financeiro ou relacionamento pessoal conflitante poderia ter influenciado o trabalho relatado neste artigo.
Agradecimentos
Este trabalho faz parte dos projetos financiados a T.W. pela ANID FONDECYT Regular 1241621 e pelo projeto da Iniciativa Chang Zuckerberg 'Latin American Hub for Bioimaging Through Open Hardware'. T.W. também agradece o financiamento do CIFAR, como Azrieli Global Scholar no programa CIFAR MacMillan Multiscale Human.
Materiais
| Name | Company | Catalog Number | Comments |
| 1H,1H,2H,2H-Perfluoro-1-octanol | Sigma-Aldrich | 370533-25G | For breaking emulsion |
| 70% ethanol | For cell permeabilization | ||
| Agar-Agar | Winkler | 9002-18-0 | |
| Biopsy Punch | 0.75 mm and 1.8 mm | ||
| Blue LED transilluminator | IO Rodeo | ||
| Culture tube | 15 mL | ||
| Desiccator | With vacuum pump | ||
| Disposable cup | For mixing PDMS | ||
| Disposable fork | For mixing PDMS | ||
| E. coli TOP10 strain | |||
| FluoPi microscope | https://github.com/wenzel-lab/FluoPi | Green fluorescence imaging system for analyzing plates | |
| Fluorinated Oil | 3M | Novec 7500 | |
| Glass slide heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of microfluidic chip | |
| Glass Slides | |||
| Hotplate | Mechanic | For evaporating Aquapel | |
| Image analysis software | Fiji/ImageJ | 2.14.0/1.54f | |
| Incubator | Mundo Lab | MLAB Scientific / For incubation of plates and microgels | |
| Isopropanol | For cleaning glass slides | ||
| Kanamycin | 100 ug/mL concentration | ||
| L-shaped spreader | For spreading bacteria on agar plates | ||
| Master mold | Chip design on silicone or glass wafer | ||
| Microtubes | 2 mL | ||
| NaCl solution | Sodium chloride 0.9% w/v | ||
| Open-source hardware strobe-enhanced microscopy stage | https://github.com/wenzel-lab/flow-microscopy-platform | For bright-field microscopy | |
| Petri dish | Citotest | 2303-1090 | 90 x 15 mm |
| Pipette tip heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of pipette tip | |
| Plasma Cleaner | Diener Electronic | 117056 | For bonding PDMS with a glass slide |
| Plasmid pCA_Odd1 | Encodes sfGFP and kanamycin resistance | ||
| Polytetrafluoroethylene (PTFE) tubing | Adtech Polymer Engineering Ltd | ||
| Pre-mixed Luria Bertoni medium | US Biological Life Science | L1520 | |
| Propidium iodide (PI) | For staining | ||
| Raspberry Pi-based pressure and flow controller system | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller | For controlling pressure and flow rates | |
| Silicone elastomer base | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Silicone elastomer curing agent | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Spectrophotometer | For measuring absorbance | ||
| SQUID microscope | https://github.com/wenzel-lab/SQUID-bioimaging-platform | Multi-fluorescence imaging system for analyzing stained cells | |
| Sterile loop | For picking a colony and streaking plating | ||
| Surfactant | Sphere Fluidics | Pico-Surf | |
| Syringes | NIPRO | With filters and tubing | |
| Temperature-controlled shaker | Mundo Lab | DLAB HCM100-Pro | |
| Tweezer | |||
| Ultra-low gelling temperature agarose | Sigma-Aldrich | A2576-5G | For generating hydrogel beads |
| Water repelent solution (fluoroalkyl silane) | Aquapel | For treating microchannels of PDMS device |
Referências
- Wein, T., Hülter, N. F., Mizrahi, I., Dagan, T. Emergence of plasmid stability under non-selective conditions maintains antibiotic resistance. Nat Commun. 10 (1), 2595 (2019).
- Rodríguez-Beltrán, J., DelaFuente, J., León-Sampedro, R., MacLean, R. C., Millán, &. #. 1. 9. 3. ;. S. Beyond horizontal gene transfer: The role of plasmids in bacterial evolution. Nat Rev Microbiol. 19 (6), 347 (2021).
- Wein, T., Dagan, T. Plasmid evolution. Curr Biol. 30 (19), R1158-R1163 (2020).
- Chen, S., Larsson, M., Robinson, R. C., Chen, S. L. Direct and convenient measurement of plasmid stability in lab and clinical isolates of E. coli. Sci Rep. 7 (1), 4788 (2017).
- Rouches, M. V., Xu, Y., Cortes, L. B. G., Lambert, G. A plasmid system with tunable copy number. Nat Commun. 13 (1), 3908 (2022).
- Silva, F., Queiroz, J. A., Domingues, F. C. Evaluating metabolic stress and plasmid stability in plasmid DNA production by Escherichia coli. Biotechnol Adv. 30 (3), 691-708 (2012).
- Wang, R., et al. Construction of novel pJRD215-derived plasmids using chloramphenicol acetyltransferase (cat) gene as a selection marker for Acidithiobacillus caldus. PLoS ONE. 12 (8), e0183307 (2017).
- Moragues, T., et al. Droplet-based microfluidics. Nat Rev Methods Primers. 3 (1), 32 (2023).
- Vitalis, C., Wenzel, T. Leveraging interactions in microfluidic droplets for enhanced biotechnology screens. Current Opinion in Biotechnology. 82, 102966 (2023).
- Sinha, N., Subedi, N., Wimmers, F., Soennichsen, M., Tel, J. A pipette-tip based method for seeding cells to droplet microfluidic platforms. J Vis Exp. (144), e57848 (2019).
- Wenzel, T. Open hardware: From DIY trend to global transformation in access to laboratory equipment. PLOS Biol. 21 (1), e3001931 (2023).
- Murillo, L. F. R., Wenzel, T. Welcome to the journal of open hardware. J Open Hardware. 1 (1), (2017).
- Shin, J. H., Choi, S. Open-source and do-it-yourself microfluidics. Sens Actuators B Chem. 347, 130624 (2021).
- Pryszlak, A., et al. Enrichment of gut microbiome strains for cultivation-free genome sequencing using droplet microfluidics. Cell Rep Methods. 2 (1), 100137 (2021).
- Pollak, B., et al. Universal loop assembly: open, efficient and cross-kingdom DNA fabrication. Biology. 5 (1), (2020).
- Volkmer, B., Heinemann, M. Condition-dependent cell volume and concentration of Escherichia coli to facilitate data conversion for systems biology modeling. PLoS ONE. 6 (7), e23126 (2011).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , (2020).
- Abate, A. R., Weitz, D. A. Faster multiple emulsification with drop splitting. Lab Chip. 11 (11), 1911-1915 (2011).
- Lau, B. T. C., Malkus, P., Paulsson, J. New quantitative methods for measuring plasmid loss rates reveal unexpected stability. Plasmid. 70 (3), 353-361 (2013).
- Oellermann, M., et al. Open hardware in science: The benefits of open electronics. Integr Comp Biol. 62 (4), 1061-1075 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados