Method Article
Analisi della stabilità dei plasmidi con microfluidica a goccioline open-source
In questo articolo
Riepilogo
Viene presentato un flusso di lavoro microfluidico accessibile e open source per l'analisi parallelizzata della ritenzione plasmidica nei batteri. Utilizzando la microscopia a fluorescenza per quantificare la presenza di plasmidi in microcolonie di singole cellule all'interno di microgoccioline di gel, questo metodo fornisce un'alternativa precisa, accessibile e scalabile al tradizionale conteggio su piastra.
Abstract
I plasmidi svolgono un ruolo vitale nella biologia sintetica consentendo l'introduzione e l'espressione di geni estranei in vari organismi, facilitando così la costruzione di circuiti biologici e percorsi all'interno e tra le popolazioni cellulari. Per molte applicazioni, il mantenimento di plasmidi funzionali senza la selezione di antibiotici è fondamentale. Questo studio introduce un flusso di lavoro microfluidico basato su hardware aperto per l'analisi della ritenzione plasmidica mediante coltura di singole cellule in microgoccioline di gel e la quantificazione delle microcolonie mediante microscopia a fluorescenza. Questo approccio consente l'analisi parallela di numerose goccioline e microcolonie, fornendo una maggiore potenza statistica rispetto al tradizionale conteggio su piastra e consentendo l'integrazione del saggio in altri flussi di lavoro microfluidici delle goccioline. Utilizzando plasmidi che esprimono proteine fluorescenti insieme a una colorazione di DNA fluorescente non specifica, le singole colonie possono essere identificate e differenziate in base alla perdita di plasmidi o all'espressione di marcatori fluorescenti. In particolare, questo flusso di lavoro avanzato, implementato con hardware open source, offre un controllo preciso del flusso e una gestione della temperatura sia del campione che del chip microfluidico. Queste funzionalità migliorano la facilità d'uso, la robustezza e l'accessibilità del flusso di lavoro. Sebbene lo studio si concentri sull'Escherichia coli come modello sperimentale, il vero potenziale del metodo risiede nella sua versatilità. Può essere adattato per vari studi che richiedono la quantificazione del segnale di fluorescenza da plasmidi o coloranti, nonché per altre applicazioni. L'adozione di hardware open source amplia il potenziale per condurre bioanalisi ad alto rendimento utilizzando una tecnologia accessibile in diversi contesti di ricerca.
Introduzione
I plasmidi sono elementi genetici essenziali nelle cellule procariotiche e contribuiscono in modo significativo all'evoluzione microbica attraverso il trasferimento laterale del DNA e il rapido adattamento ai cambiamenti ambientali 1,2. Queste molecole di DNA extracromosomico trasportano geni che forniscono tratti vantaggiosi, come la resistenza agli antibiotici, le funzioni metaboliche e i fattori di virulenza, rendendole preziose per la ricerca microbiologica, la biologia sinteticae gli studi sull'evoluzione. Tuttavia, il mantenimento dei plasmidi nelle popolazioni cellulari è impegnativo a causa del carico metabolico della replicazione e della segregazione, che spesso si traduce in perdita di plasmidi senza pressione selettiva3. Inoltre, l'ereditarietà stabile richiede meccanismi come la tossina-antitossina e i sistemi di partizionamento, aggiungendo complessità al mantenimento dei plasmidi. La valutazione della stabilità dei plasmidi in condizioni variabili è fondamentale sia per la ricerca di base che per le applicazioni pratiche che utilizzano i plasmidi come elemento di ricerca primario 4,5. La maggior parte dei metodi attuali per valutare la stabilità dei plasmidi presenta limitazioni significative: i metodi basati sulla citometria a flusso forniscono dati indiretti a livello di popolazione, richiedono attrezzature costose e mancano di visualizzazione diretta delle colonie6. I metodi di trascrittomica e proteomica di massa sono costosi, forniscono solo risposte cellulari medie e non possono quantificare direttamente la ritenzione plasmidica nelle singole colonie6. I metodi tradizionali come la diluizione seriale e il passamento sono semplici ma richiedono molto tempo e mancano di precisione e rappresentabilità7. Nel complesso, dedurre quantitativamente o proiettare il numero di colonie che mantengono uno specifico plasmide funzionale nel tempo o nelle pressioni selettive rimane una sfida.
Per affrontare queste sfide, viene presentato un nuovo flusso di lavoro microfluidico che utilizza strumenti di ricerca hardware aperti per quantificare i segnali fluorescenti in più colonie isolate di batteri, utilizzando Escherichia coli come modello. Questo metodo consente un'analisi precisa e ad alta produttività della ritenzione plasmidica in varie condizioni o pressioni selettive. L'analisi della risoluzione di singole cellule fornisce un metodo preciso per manipolare colonie isolate, fornendo dati sensibili sulla quantificazione dei plasmidi che possono aiutare a valutare i tassi di ritenzione e perdita4.
La microfluidica, in particolare la microfluidica a goccioline, è emersa come un potente strumento per incapsulare e manipolare singole cellule in ambienti controllati8. In particolare, le goccioline di microgel possono incapsulare singole cellule per un'analisi precisa e ad alto rendimento senza la necessità di mantenere goccioline sospese nell'olio9, consentendo uno studio controllato della dinamica dei plasmidi in un microambiente definito. Dopo l'incapsulamento delle sospensioni cellulari direttamente da un puntale di pipetta10, le tecniche di fluorescenza possono essere utilizzate per monitorare la crescita delle microcolonie all'interno delle goccioline, consentendo un'analisi dettagliata della ritenzione e della segregazione dei plasmidi a diverse pressioni di selezione3.
I vantaggi di questo metodo rispetto alle tradizionali tecniche di coltura di massa includono una maggiore precisione, una minore variabilità e la capacità di condurre analisi ad alto rendimento. La tecnologia microfluidica open source supera i limiti dei costosi sistemi proprietari, come i problemi di accessibilità, adattabilità e manutenzione, che spesso ostacolano il progresso della ricerca 11,12,13. Dimostrando come applicare il flusso di lavoro sperimentale avanzato dell'analisi della ritenzione plasmidica in microgel con strumentazione open-source, viene fornito un metodo accessibile e affidabile per la ricerca nella biologia dei plasmidi, nelle applicazioni della biologia sintetica e nelle tecniche di analisi delle goccioline microfluidiche.
In sintesi, questo articolo presenta un metodo accessibile per valutare quantitativamente la ritenzione plasmidica in E. coli con un'elevata potenza statistica. Le capacità di questo metodo lo rendono uno strumento prezioso per far progredire la comprensione della biologia dei plasmidi e migliorare le applicazioni della biologia sintetica.
Protocollo
La Figura 1fornisce una panoramica schematica per valutare la stabilità dei plasmidi in E. coli. I dettagli dei reagenti e delle attrezzature utilizzate sono elencati nella Tabella dei Materiali. I dati grezzi e gli script di visualizzazione sono disponibili all'indirizzo https://doi.org/10.17605/OSF.IO/6YWJK.
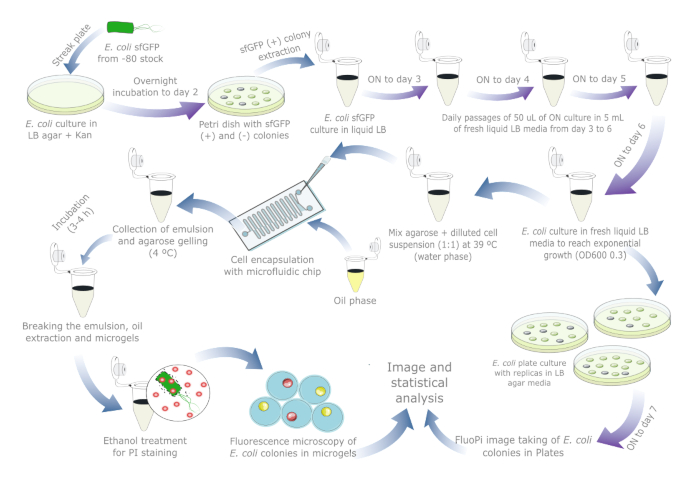
Figura 1: Protocollo giorno per giorno per la valutazione della stabilità dei plasmidi in E. coli. Le frecce blu indicano i passaggi durante il giorno e le frecce viola indicano l'incubazione notturna. Ogni incubazione di liquidi e agar è stata eseguita a 37 °C insieme a una provetta/piastra di controllo negativa separata. Si noti che la preparazione e i passaggi della coltura cellulare non sono necessari per i campioni del mondo reale in cui potrebbe essersi già verificata la perdita di plasmidi, quindi il protocollo dovrebbe essere ridotto a uno o due giorni se è inclusa una coltura di riferimento su piastra. Clicca qui per visualizzare una versione più grande di questa figura.
1. Preparazione del chip microfluidico
NOTA: In questo protocollo, è possibile utilizzare diversi design di chip commerciali o personalizzati per l'incapsulamento delle celle in grado di generare goccioline di acqua in olio di diametro inferiore a 100 μm in un regime di gocciolamento. Per questo studio, il chip è stato progettato e fabbricato (si veda l'articolo dati https://doi.org/10.17605/OSF.IO/6YWJK) seguendo lo stesso metodo di progettazione e fabbricazione riportato in un rapporto precedentemente pubblicato14.
- Ottenere o preparare un chip microfluidico PDMS su vetro utilizzando uno stampo master progettato per la generazione di microgocce di gel.
- Iniettare una soluzione idrorepellente (fluoroalchil silano) nel chip per rendere idrofobici i microcanali interni. Infondere la soluzione negli ingressi e assicurarsi che tutti i canali siano pieni di liquido. Lasciare riposare i canali riempiti per circa 30-60 s.
- Rimuovere la soluzione dal dispositivo espellendo l'aria nei microcanali interni. Utilizzare una siringa d'aria vuota per sciacquare l'aria e una salvietta assorbente sulle altre porte per evitare schizzi.
- Cuocere il dispositivo trattato a 65 °C per 15 minuti su una piastra riscaldante per far evaporare la soluzione in eccesso. In alternativa, conservare il dispositivo in frigorifero (4 °C) per una notte.
NOTA: Il chip microfluidico è pronto per l'uso. Il protocollo può essere messo in pausa qui.
2. Preparazione del campione
- Prelievo di cellule
NOTA: La coltura cellulare in goccioline può fornire dati rilevanti sulla dinamica dei plasmidi. Come modello sperimentale di batteri, un ceppo TOP10 di E. coli viene utilizzato con il plasmide pCA_Odd1 (vedi dati depositati https://doi.org/10.17605/OSF.IO/6YWJK) che codifica per una proteina fluorescente verde supercartella (sfGFP) e la resistenza alla kanamicina15. Batteri, plasmidi e mezzi di crescita possono variare in base al sistema sperimentale.- Preparare Luria Bertoni (LB)-Agar sciogliendo 25 g di LB premiscelato e 12 g di agar in 800 mL di acqua distillata (dH2O). Inoltre, preparare il terreno LB liquido sciogliendo 25 g di LB premiscelato in 800 mL di dH2O. Autoclavare le soluzioni in autoclave e lasciarle raffreddare a circa 60 °C. Spostarsi in un ambiente sterile come una cappa a flusso per i passaggi successivi.
NOTA: La polvere LB premiscelata contiene 10 g di triptone, 5 g di estratto di lievito e 10 g di NaCl. È possibile utilizzare anche terreni LB-agar automiscelati. - Aggiungere 50 μL di kanamicina (preparata a 100 mg/mL) a 50 mL di LB-Agar liquido (concentrazione finale: 100 μg/mL). Mescolare la soluzione capovolgendo il tubo alcune volte.
- Versare circa 15 ml di LB-agar liquido per piastra di Petri (90 mm x 15 mm). Preparare due piastre: una per il controllo negativo (monitoraggio della contaminazione) e una per la coltura dell'esperimento. Lasciare raffreddare la soluzione di LB-agar e solidificare fino a quando il colore cambia da scuro a chiaro su entrambe le piastre.
- Utilizzare un anello sterile per diffondere il ceppo di E. coli sulla piastra di coltura utilizzando la tecnica della piastra a strisce. Riposizionare il brodo di glicerolo E. coli a –80 °C subito dopo l'uso. Chiudere entrambe le piastre e incubarle per una notte a 37 °C (giorno 1).
- Il giorno 2, controllare la piastra di controllo negativa per eventuali colonie come segnale di contaminazione (in tal caso, ripetere i passaggi dal punto 2.1.4.). Identificare le singole colonie fluorescenti sulla piastra di E. coli utilizzando un transilluminatore a luce blu per preparare un stock di coltura liquida.
- In condizioni sterili, aprire la piastra e utilizzare un ansa sterile o un puntale per pipetta da 200 μL per prelevare la colonia selezionata e trasferirla in una provetta di coltura con 5 mL di terreno LB liquido fresco e 5 μL di kanamicina (preparata a 100 mg/mL). Preparare una provetta di controllo negativo senza inoculazione per il monitoraggio della contaminazione. Incubare le provette di coltura a 37 °C per una notte agitando a 220 giri/min.
- Far passare il campione per altri 3 giorni in terreni senza antibiotici per simulare le condizioni in cui può verificarsi la perdita di plasmidi. Nei giorni 3, 4 e 5, trasferire 50 μl di coltura notturna in una nuova provetta contenente 5 mL di LB liquido senza antibiotici. Incubare a 37 °C per una notte con agitazione a 220 giri/min. Ripetere questo passaggio fino al giorno 6, ottenendo una coltura finale di 5 ml dopo quattro passaggi totali.
- Trasferire 50 μl dalla coltura finale durante la notte a una nuova provetta contenente 2 mL di LB liquido. Lasciare che la coltura raggiunga una densità ottica (OD600) di 0,3 utilizzando uno spettrofotometro (3-4 ore circa).
NOTA: La coltura fresca deve essere utilizzata prima che l'OD600 aumenti per garantire che i batteri siano nella fase di crescita logaritmica. Se non si raggiunge la concentrazione desiderata, ripetere dal passaggio 2.1.8.
- Preparare Luria Bertoni (LB)-Agar sciogliendo 25 g di LB premiscelato e 12 g di agar in 800 mL di acqua distillata (dH2O). Inoltre, preparare il terreno LB liquido sciogliendo 25 g di LB premiscelato in 800 mL di dH2O. Autoclavare le soluzioni in autoclave e lasciarle raffreddare a circa 60 °C. Spostarsi in un ambiente sterile come una cappa a flusso per i passaggi successivi.
- Miscela di agarosio cellulare per incapsulamento
NOTA: Il controllo della concentrazione è essenziale per garantire l'incapsulamento di goccioline a singola cellula. La concentrazione di celle richiesta può essere calcolata per una velocità di incapsulamento target e un volume specifico di goccioline, come mostrato nell'esempio seguente:
Volume delle goccioline (goccia V): se vengono generate goccioline da 50 μm
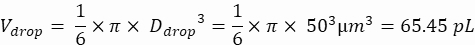
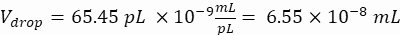
Cellule desiderate per gocciolina (Cpd): una cellula per cinque goccioline in media (0,2 cellule/gocciolina)
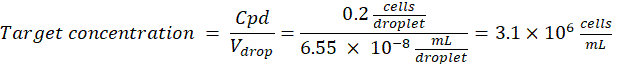
Fattore di diluizione: la concentrazione iniziale della cella si ottiene al punto 2.1.8
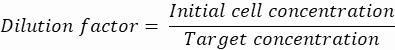
Qui, vengono generate goccioline da 50 μm o 100 μm (senza scissione delle goccioline) (65-520 pL) in cui è incapsulata circa una cellula ogni cinque goccioline, o 1,6 cellule per gocciolina senza scissione. Per E. coli, utilizzare il fattore di conversione di 1 unità OD600 ≈ 7,8 x 108 cellule/mL16. Moltiplicare il valore OD600 del passaggio 2.1.8 per il fattore di conversione per ottenere la concentrazione iniziale (cellule/mL) della coltura.- Risospendere E. coli dal materiale di coltura preparato in LB liquido senza kanamicina a una concentrazione di 6,2e+6 cellule/mL (concentrazione target per goccioline di 50 μm di diametro). Conservare la sospensione batterica a temperatura ambiente fino a quando non si mescola con l'agarosio.
NOTA: Gli antibiotici sopprimono la formazione di microcolonie negative, quindi è essenziale escluderle dai terreni per gli esperimenti di perdita di plasmidi. - Preparare l'agarosio a bassissima temperatura di gelificazione riscaldandolo a 90 °C in LB liquido a una concentrazione del 2% (p/v). Agitare il composto per 10 minuti in uno shaker a temperatura controllata.
- Ridurre la temperatura del termo-agitatore a 39 °C per raffreddare la soluzione di agarosio. Parallelamente, posizionare il tubo di sospensione batterica nel termo-shaker per 4 minuti per riscaldarlo a 39 °C.
- Mescolare i batteri e le sospensioni di agarosio in rapporto 1:1 per ottenere una concentrazione di agarosio dell'1% (p/v) con la sospensione cellulare a 3,1e+6 cellule/mL. Preparare la soluzione di controllo negativo (monitoraggio della contaminazione) con la stessa concentrazione di agarosio utilizzando LB liquido al posto della sospensione batterica.
NOTA: La sospensione di cellule di agarosio deve essere utilizzata rapidamente per evitare variazioni di concentrazione dovute alla crescita dei batteri. Mantenere il termo-shaker a 39 °C per mantenere l'agarosio liquido fino a quando non viene caricato nel riscaldatore del puntale per la generazione di goccioline.
- Risospendere E. coli dal materiale di coltura preparato in LB liquido senza kanamicina a una concentrazione di 6,2e+6 cellule/mL (concentrazione target per goccioline di 50 μm di diametro). Conservare la sospensione batterica a temperatura ambiente fino a quando non si mescola con l'agarosio.
3. Coltivazione di una singola colonia ad alto rendimento
- Configurazione sperimentale
- Costruisci o ottieni la piattaforma di flusso open source completa (vedi https://doi.org/10.17605/OSF.IO/6YWJK), inclusi i driver di pressione del gas e i sensori di flusso. In alternativa, crea la più semplice fase di microscopia stroboscopica hardware open source (vedi le istruzioni di compilazione https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ e l'https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage del repository del progetto).
NOTA: È inoltre possibile utilizzare una configurazione microfluidica tradizionale con un microscopio commerciale, una fotocamera ad alta velocità e funzionalità di riscaldamento del campione. - Integra il sistema di controllo della pressione e del flusso open source basato su Raspberry Pi come documentato nel repository designato (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller).
NOTA: Il controllo del modulo viene illustrato e sottoposto a backup nel repository dei dati (https://doi.org/10.17605/OSF.IO/6YWJK. In alternativa, è possibile utilizzare regolatori di pressione tradizionali o pompe a siringa a coppia elevata. - Includere un vetrino in vetro e riscaldatori per puntali per pipette (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring) nella configurazione per controllare la temperatura del campione di cellule di agarosio quando entra nel chip.
NOTA: Questi riscaldatori, illustrati nell'archivio dati (https://github.com/wenzel-lab/flow-microscopy-platform e https://doi.org/10.17605/OSF.IO/6YWJK), sono una caratteristica importante del sistema di controllo open source che consente il lavoro con l'agarosio e potrebbero non essere disponibili in altri sistemi commerciali.
- Costruisci o ottieni la piattaforma di flusso open source completa (vedi https://doi.org/10.17605/OSF.IO/6YWJK), inclusi i driver di pressione del gas e i sensori di flusso. In alternativa, crea la più semplice fase di microscopia stroboscopica hardware open source (vedi le istruzioni di compilazione https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ e l'https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage del repository del progetto).
- Incapsulamento a cella singola
- Posizionare il chip microfluidico sullo stadio di microscopia stroboscopica, assicurandosi che la giunzione di generazione delle goccioline (intersezione delle fasi acquosa e oleosa) sia visibile.
- Impostare il riscaldatore del puntale della pipetta e il riscaldatore del vetrino in vetro a 40 °C utilizzando l'interfaccia del software di controllo.
- Utilizzare una siringa con tubo e tappo PDMS per caricare la miscela di agarosio all'1% con sospensione cellulare in un puntale per pipetta da 200 μl. Inserire la punta nel riscaldatore della punta e posizionarla sull'ingresso della fase acquosa nel chip microfluidico. Sostituire la guarnizione PDMS del puntale con una collegata al tubo del sistema di controllo del flusso e iniziare l'infusione della sospensione cellulare.
- Inserire l'estremità del tubo di scarico in un tubo di scarico e impostare le portate o le pressioni di due fasi sull'interfaccia utente per erogare lentamente il fluido al canale microfluidico. Utilizzare 200 μL/h (180 mbar) per la fase acquosa e 1700 μL/h (320 mbar) per la fase oleosa. Attendere 1 minuto per la stabilizzazione della generazione di goccioline.
NOTA: I valori di pressione dipendono dalle dimensioni dei canali del design del chip e potrebbe essere necessario regolare i valori di flusso per diversi design di giunzione per la generazione di goccioline. - Una volta che la generazione di goccioline è stabile, trasferire i rifiuti e il tubo di raccolta nel tubo di raccolta. Continuare a raccogliere le goccioline fino a quando il serbatoio del campione non è vuoto. Ripetere i passaggi da 3.2.3 a 3.2.5 per incapsulare la soluzione di controllo negativo del passaggio 2.2.4.
NOTA: La raccolta del campione deve essere completata entro 15 minuti. - Conservare le provette di raccolta in ghiaccio durante la generazione di goccioline o posizionarle a 4 °C dopo l'esperimento per 1 ora per consentire all'agarosio di gelificare all'interno delle goccioline.
NOTA: Il chip microfluidico può essere riutilizzato se i microcanali rimangono non ostruiti e viene caricata la stessa sospensione. Eliminare il puntale della pipetta dopo aver completato la generazione dell'emulsione (passaggi 3.2.3-3.2.5).
- Crescita e rilascio delle colonie dall'emulsione
- Trasferire le microgoccioline di gel contenenti batteri e il controllo negativo nelle goccioline in una camera di incubazione impostata a 37 °C.
- Incubare le microgoccioline per almeno 4 ore, o durante la notte, per consentire una crescita sufficiente delle colonie. Confermare che il controllo negativo non mostri segni di contaminazione tramite microscopia a campo chiaro.
- Per rilasciare le colonie dall'emulsione, rimuovere quanto più olio possibile da sotto l'emulsione di microgocce di gel utilizzando una pipetta o una siringa con ago (qui è stato utilizzato un ago da 21 G).
- Trasferire 50 μl di microgoccioline di gel in una nuova microprovetta e conservarla a 4 °C per un'ulteriore analisi delle goccioline. All'emulsione rimanente, aggiungere una miscela 1:1 di olio fluorurato con 1H,1H,2H,2H-perfluoro-1-ottanolo (PFO) in un volume uguale all'emulsione.
- Aggiungere circa 200 μl di tampone salino tamponato con fosfato (PBS) o 0,9% p/v di tampone NaCl, sopra l'emulsione. Agitare il composto e farlo girare brevemente in una centrifuga a velocità fissa.
- Rimuovere con cautela la fase oleosa dalla parte inferiore dell'interfaccia liquida e scartare 100 μl di PBS dalla parte superiore. Ripetere i passaggi 3.3.4–3.3.5 per ottenere microgel lavati in tampone PBS con residui di olio minimi o nulli.
NOTA: I microgel si depositano all'interfaccia liquida; Evitare di rimuoverli insieme alla fase oleosa.
4. Analisi di singole colonie
- Colorazione cellulare
NOTA: Molte diverse combinazioni di coloranti funzionano per questo protocollo. In sostanza, dovrebbe essere scelto un colorante di DNA o di parete cellulare che abbia un colore distinto dalla proteina fluorescente codificata dal plasmide e che possa essere analizzato utilizzando le combinazioni di filtri disponibili sul microscopio a fluorescenza. Qui, il DNA delle cellule è stato colorato con ioduro di propidio (PI) per distinguere la sua fluorescenza dalla proteina fluorescente verde codificata sui plasmidi, ma possono essere utilizzate molte altre colorazioni di DNA.
ATTENZIONE: Il PI è un potenziale cancerogeno e deve essere maneggiato con adeguati dispositivi di protezione individuale. Smaltire il colorante in modo sicuro e nel rispetto delle normative locali.- Centrifugare i microgel lavati a circa 80 x g per 5 minuti a temperatura ambiente. Eliminare il surnatante utilizzando una pipetta.
- Trasferire 50 uL dei microgel in una nuova microprovetta che funge da controllo negativo per il trattamento con etanolo.
NOTA: La colorazione PI entra solo nelle cellule con membrane compromesse, come quelle colpite dal trattamento con etanolo. - Aggiungere un volume uguale di etanolo al 70% ai microgel rimanenti e mescolare brevemente con un vortice. Incubare a temperatura ambiente per 15 minuti per permeabilizzare le membrane batteriche per la colorazione PI. Ripetere il passaggio 4.1.1.
- Aggiungere un volume uguale dello 0,9% p/v di NaCl ai microgel e agitare brevemente. Ripetere il passaggio 4.1.1.
- Aggiungere 2 μL di PI (1 mg/mL) a entrambi i campioni di microprovette. Mescolare accuratamente e incubare al buio a temperatura ambiente per 15 minuti.
NOTA: Se il controllo negativo presenta un segnale fluorescente rosso tramite microscopia a fluorescenza, l'integrità delle colonie potrebbe essere stata compromessa durante la loro esposizione ad altre soluzioni nelle fasi precedenti.
- Microscopia
NOTA: Le goccioline e i microgel vengono visualizzati con un microscopio a epifluorescenza invertita per ottenere la distribuzione delle dimensioni delle goccioline e la fluorescenza delle colonie batteriche nei microgel (Figura 2). In questo caso, viene utilizzata una piattaforma di microscopia invertita (https://github.com/wenzel-lab/SQUID-bioimaging-platform)17 open source con un obiettivo 10x 0,3NA, un array di LED bianchi per l'illuminazione in campo chiaro e un LED da 470 nm per l'eccitazione. I microscopi a epifluorescenza commerciali possono essere utilizzati per l'imaging di goccioline e microgel. La calibrazione è necessaria perché l'illuminazione e i filtri variano a seconda del modello, della marca e delle proteine fluorescenti utilizzate.- Trasferire 2 μL di microgoccioline di gel in un vetrino della camera di imaging e aggiungere 5 μL di olio fluorurato per aiutare a formare un monostrato di goccioline per un imaging ottimale.
NOTA: Le camere di conteggio delle cellule o le semplici camere microfluidiche possono aiutare a stendere l'emulsione in modo sottile e rallentare il processo di essiccazione. - Sul microscopio, attivare l'illuminazione a matrice di LED bianca dall'alto per l'imaging in campo chiaro. Montare il vetrino preparato, mettere a fuoco il campione e individuare un monostrato di goccioline. Cattura un'immagine in campo chiaro.
- Senza spostare il campione, è possibile acquisire un'immagine a fluorescenza delle colonie passando al LED da 470 nm per l'eccitazione. Regolare la ruota portafiltri in modo che si allinei con il filtro verde della lunghezza d'onda per l'imaging sfGFP. Scansiona tutte le aree contenenti monostrati di goccioline e ripeti i passaggi 4.2.2–4.2.3 per garantire la robustezza statistica dell'analisi delle goccioline.
- Trasferire 2 μl di microgel colorati in un chip della camera di imaging e 5 μl di NaCl allo 0,9% p/v per formare un monostrato di microgel. Sigillare l'ingresso e l'uscita del chip per evitare l'evaporazione durante l'imaging.
NOTA: L'evaporazione può influenzare la colocalizzazione delle etichette fluorescenti. Le camere microfluidiche sono consigliate per l'imaging prolungato. - Ripetere i passaggi 4.2.2–4.2.3. Regolare il filtro dell'intervallo della lunghezza d'onda rossa per l'imaging PI e acquisire le rispettive immagini.
- Dopo aver completato l'imaging in una posizione, trovare l'area adatta successiva sul vetrino con un monostrato di microgel e ripetere il processo di imaging per garantire la robustezza statistica e l'analisi completa del campione.
- Trasferire 2 μL di microgoccioline di gel in un vetrino della camera di imaging e aggiungere 5 μL di olio fluorurato per aiutare a formare un monostrato di goccioline per un imaging ottimale.
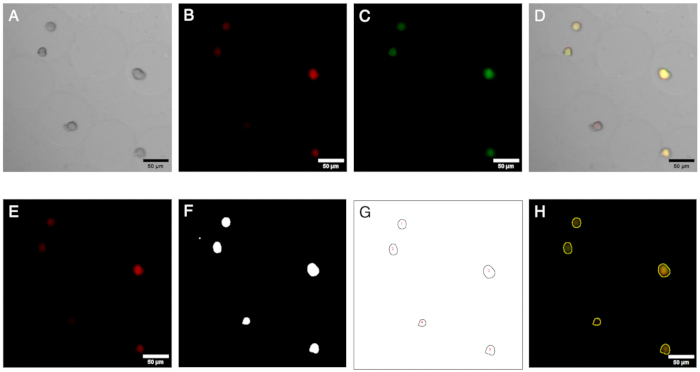
Figura 2: Immagini al microscopio e loro analisi. Imaging a fluorescenza e analisi di colonie in microgel. (A-C) Canali di immagine acquisiti tramite microscopia in campo chiaro e a fluorescenza con il microscopio invertito. L'immagine composita (D) mostra la presenza di una colonia negativa (solo fluorescenza rossa) all'interno dei microgel. (E-H) Risultati del flusso di lavoro di analisi delle immagini. Generando ROI, le colonie possono essere identificate sui canali rosso e verde e i segnali possono essere quantificati per definire la presenza di colonie negative. Barre della scala: 50 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
- Analisi delle immagini
NOTA: Per analizzare i segnali di fluorescenza provenienti dalle colonie incapsulate e identificare eventi rari, le immagini provenienti dall'imaging in campo chiaro e a fluorescenza possono essere elaborate con Fiji/ImageJ (Figura 3). Questi passaggi possono essere implementati in uno script macro e i valori dei parametri possono variare a seconda della configurazione ottica.- Apri le immagini dai canali verde e rosso. Definisci un'area rettangolare di interesse (ROI) a partire dalle coordinate (500, 500) con una larghezza e un'altezza di 2000 pixel ciascuna.
NOTA: Questi parametri si applicano alle immagini da 3000 x 3000 pixel e il ROI definisce l'area con una migliore illuminazione. - Ritaglia l'immagine in base alla ROI rettangolare definita. Sottrai un valore costante di 10 dall'intensità di ciascun pixel e applica un filtro di levigatura all'immagine per ridurre il rumore di fondo e rendere più distinti gli oggetti di interesse.
NOTA: solo questa regione verrà utilizzata per ulteriori analisi. - Duplica l'immagine dal canale rosso e convertila in una maschera binaria per identificare i rari eventi legati al plasmide. Impostare i valori di soglia tra 10 e 255. I pixel all'interno di questo intervallo saranno considerati in primo piano (oggetti di interesse), mentre gli altri saranno trattati come sfondo.
- Esegui operazioni morfologiche per chiudere piccoli spazi vuoti e riempire eventuali buchi all'interno degli oggetti di interesse. Applicare l'algoritmo spartiacque per separare gli oggetti sovrapposti all'interno della maschera binaria.
- Analizza le particelle nella maschera binaria. Considera solo le particelle con una dimensione superiore a 30 pixel e una circolarità compresa tra 0,50 e 1,00. I risultati vengono riepilogati e aggiunti alla tabella dei risultati. Salva l'insieme di ROI rilevate dall'analisi delle particelle per un'ulteriore visualizzazione.
- Visualizza le ROI salvate sulle immagini dei canali verde e rosso. Misura l'intensità o altre proprietà delle ROI all'interno di queste immagini. Registra le misurazioni nella tabella dei risultati e salva i risultati in file CSV separati per ulteriori analisi statistiche.
- Apri le immagini dai canali verde e rosso. Definisci un'area rettangolare di interesse (ROI) a partire dalle coordinate (500, 500) con una larghezza e un'altezza di 2000 pixel ciascuna.
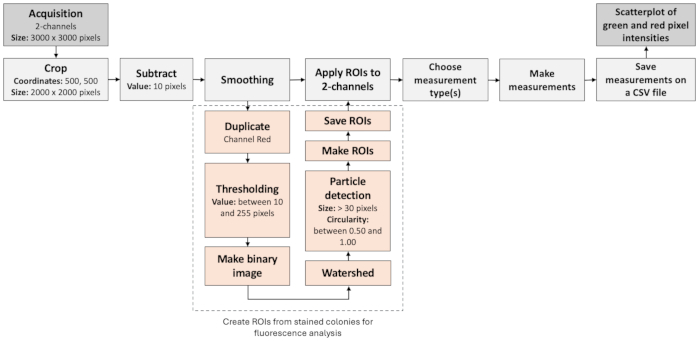
Figura 3: Flusso di lavoro dell'analisi delle immagini per identificare le colonie negative. La figura illustra un flusso di lavoro passo-passo per elaborare e valutare automaticamente le immagini a fluorescenza. Il flusso di lavoro si basa sulla colocalizzazione delle etichette di fluorescenza e sull'analisi delle particelle. Clicca qui per visualizzare una versione più grande di questa figura.
5. Saggio comparativo su piastra di agar
NOTA: Per confrontare il metodo delle gocce con un saggio tradizionale su piastra, la quantificazione delle colonie fluorescenti dello stesso ceppo di E. coli è stata ottenuta nella fase 2.1.8. è stato eseguito utilizzando piastre di Petri. Questo è servito come metodo di controllo analogico per misurare la stabilità del plasmide sfGFP. Vedere anche l'illustrazione del metodo nella Figura 1.
- Preparare LB-agar (senza antibiotico) come descritto al passaggio 2.1.1 della sezione Raccolta cellulare. Raffreddare l'agar agar a una temperatura inferiore a 60 °C e omogeneizzare.
- In un ambiente sterile, versare 15 mL di terreno di agar in ciascuna piastra di Petri. Lasciare solidificare le piastre con i coperchi parzialmente aperti fino a quando non sono pronte per l'uso.
- Inoculare tre piastre con 10 μl ciascuna di una coltura di E. coli in terreno LB con un OD600 di 0,007 ± 0,002. Stendere uniformemente con uno spandiconcime a forma di L. Incubare le piastre a 37 °C, chiuse e capovolte. Preparare una piastra di controllo senza batteri nelle stesse condizioni.
- Dopo 24 ore, acquisisci immagini a fluorescenza delle piastre utilizzando FluoPi, un sistema di imaging a fluorescenza (https://github.com/RudgeLab/FluoPi)15 open source.
NOTA: Il FluoPi è costituito da una fotocamera Raspberry Pi con una luce di eccitazione blu a una lunghezza d'onda centrata a 470 nm e filtri di eccitazione ed emissione acrilici. - Contare manualmente le colonie fluorescenti e non fluorescenti in ciascuna piastra utilizzando le immagini acquisite.
Risultati
Validazione dell'incapsulamento cellulare e della formazione di microcolonie
L'incapsulamento cellulare può essere confermato visivamente eseguendo la microscopia in campo chiaro sulle microgoccioline di gel prima di rompere l'emulsione e lavare i microgel. Un risultato rappresentativo dell'emulsione in questa fase è mostrato nella Figura 4.
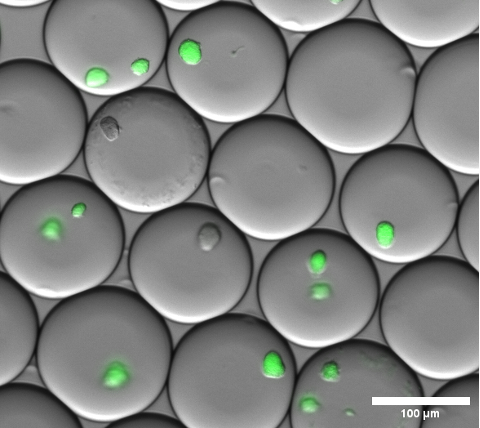
Figura 4: Sezione di un'immagine di sovrapposizione al microscopio a fluorescenza. Dopo l'incubazione notturna, microcolonie rappresentative di sfGFP esprimono colonie di E. coli all'interno di microgoccioline di gel. È stato utilizzato un obiettivo da microscopio con ingrandimento 10x e 0,30 NA. Barra della scala: 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Risultati dell'analisi delle immagini
Una volta che i microgel sono colorati e in campo chiaro, così come i canali di fluorescenza acquisiti in diverse posizioni, è possibile visualizzare le colonie identificate come negative nelle immagini originali (vedi Figura 5A). I dati estratti da tutte le immagini di un esperimento possono essere tracciati per mostrare il rapporto di fluorescenza di varie colonie, evidenziando quelle che hanno perso la fluorescenza codificata dai plasmidi (vedi Figura 5B). I risultati indicano che 100 colonie hanno perso la loro funzionalità plasmidica o plasmidica su un totale di 2785 microcolonie analizzate, corrispondenti al 3,6%.
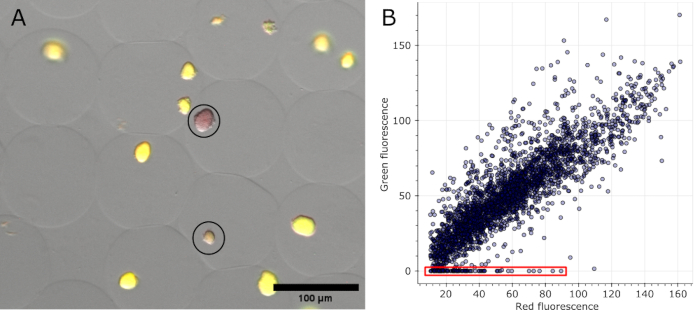
Figura 5: Quantificazione delle microcolonie negative. (A) Sezione di un'immagine di sovrapposizione al microscopio a fluorescenza. Dopo la rimozione dell'olio e la colorazione, colonie rappresentative in microgel che esprimono sfGFP e due colonie negative hanno mostrato la fluorescenza rossa della colorazione del DNA (cerchiata in nero). Barra della scala: 100 μm. (B) Grafico a dispersione dei valori di fluorescenza di singole microcolonie estratte da 16 immagini di microscopia multicanale. Le colonie senza alcuna fluorescenza verde sono state contate come negative, come indicato in rosso nel grafico. Clicca qui per visualizzare una versione più grande di questa figura.
Quantificazione della piastra di agar
Le immagini delle piastre triplicate sono mostrate nella Figura 6, con le colonie non fluorescenti indicate da frecce bianche. La prima lastra (Figura 6A) mostrava un totale di 213 colonie, di cui 1 non fluorescente. La seconda piastra (Figura 6B) aveva un totale di 49 colonie, senza colonie non fluorescenti. La terza lastra (Figura 6C) mostrava un totale di 252 colonie, 6 delle quali non erano fluorescenti. Questi risultati corrispondono a un tasso medio di perdita di plasmidi di colonie del 2,3%, con un'ampia deviazione standard di 3,2.
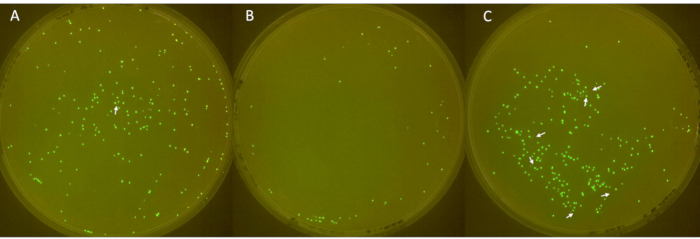
Figura 6: Identificazione di colonie negative su piastre. (A-C) Colonie di E. coli fluorescenti e non fluorescenti su piastre LB-agar (diametro: 90 mm, altezza: 15 mm). L'inoculo, derivato da E. coli con sfGFP da stock di -80 °C, è stato striato il giorno 1, coltivato con antibiotico il giorno 2 e diluito 1:100 al giorno dal giorno 3 al 6 per consentire la perdita di plasmidi. Le colonie sono state incubate a 37 °C per 24 ore e sottoposte a riprese in una camera FluoPi. Le colonie non fluorescenti sono state potenziate con GIMP e indicate con frecce bianche. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
È stato dimostrato che un metodo basato su microgoccioline di gel identifica e quantifica efficacemente le colonie con e senza l'espressione genetica codificata da plasmidi di proteine fluorescenti, come sfGFP. Le colonie che non esprimono sufficientemente il prodotto plasmidico vengono identificate utilizzando un colorante fluorescente di DNA (qui, ioduro di propidio) che colora tutte le colonie e presenta una diversa lunghezza d'onda di emissione. Questa integrazione di microfluidica a goccioline, gelificazione e microscopia a fluorescenza, utilizzando la tecnologia open source, consente l'esecuzione di un flusso di lavoro avanzato in molti contesti di ricerca11,13. La generazione di successo di microgoccioline di gel consente flussi di lavoro avanzati di biologia molecolare su singole cellule, tra cui la lisi cellulare, l'amplificazione di un singolo genoma, gli screening di interazione cellulare metabolica, lo scambio di terreni e altro ancora 8,9. Questi vantaggi vengono utilizzati in questo protocollo per coltivare, colorare e analizzare le microcolonie in modo più scalabile rispetto ai tradizionali saggi basati su piastra.
Passaggi critici
Il processo di incapsulamento è una parte critica e delicata del protocollo. È necessario un controllo preciso delle concentrazioni, delle portate e delle pressioni degli ingredienti per generare microgel uniformi all'interno di un intervallo di dimensioni specifico e controllare il numero medio di cellule per gocciolina. Inoltre, il mantenimento della concentrazione e della temperatura della miscela di cellule e agarosio previene la formazione di grumi o la gelificazione prematura. Il controllo della temperatura della sospensione liquida di cellule di agarosio in un puntale per pipette è un'implementazione particolarmente vantaggiosa della nostra workstation di microfluidica hardware open source che fornisce una generazione di microgel molto più semplice e robusta rispetto agli sforzi per controllare la temperatura delle pompe a siringa e dei tubi. Poiché le cellule vengono mescolate con il terreno di crescita dell'agarosio prima dell'incapsulamento e della coltivazione, i microgel di agarosio devono essere generati rapidamente per evitare importanti cambiamenti nella concentrazione cellulare. A tale scopo, è stato ottimizzato un design di chip microfluidico con divisione delle gocce ispirato ad Abate et al.18.
Modifiche e risoluzione dei problemi
Sono state necessarie diverse calibrazioni e modifiche per perfezionare il protocollo originale. L'incapsulamento dell'agarosio è molto più impegnativo rispetto alle normali goccioline di acqua in olio, poiché richiede la progettazione di un sistema per mantenere l'agarosio allo stato liquido, garantendo al contempo che il flusso in fase acquosa raggiunga un intervallo omogeneo di dimensioni delle particelle. Le variazioni della viscosità dell'agarosio dovute alla gelificazione influiscono sulla portata, portando a particelle di dimensioni maggiori. La microscopia richiede un'attenta selezione di filtri e sorgenti luminose per garantire segnali di eccitazione ed emissione non sovrapposti per una chiara differenziazione. Inizialmente, DAPI è stato scelto per la colorazione dei batteri, ma il suo segnale di emissione si sovrapponeva a sfGFP, causando il rilevamento di sfGFP nel canale di rilevamento blu. Siamo passati al PI perché la sua emissione è ben separata dalla sfGFP a lunghezze d'onda lunghe (luce rossa).
Mentre la perdita di plasmidi è stata quantificata utilizzando il metodo proposto, il plasmide sfGFP utilizzato è risultato inaspettatamente stabile, mostrando quasi nessun caso di perdita di plasmidi nella prima generazione di cellule coltivate senza antibiotici, anche in condizioni di stress come il terreno pH9 e l'incubazione a 40 °C. Questa osservazione è coerente con i risultati di altri gruppi di ricerca 1,19. La stabilità del plasmide ha limitato la dimostrazione delle capacità di quantificazione complete del metodo per le generazioni iniziali di colture cellulari, ma ha dimostrato che il metodo è abbastanza sensibile da rilevare anche piccole differenze nella ritenzione plasmidica. L'osservazione di un'elevata stabilità plasmidica nelle prime generazioni ha un'importante implicazione per gli schermi microfluidici delle goccioline utilizzando un saggio di selezione negativa, come l'inibizione dei batteri bersaglio. Significa che la perdita plasmidica dei bersagli di selezione è una bassa fonte di risultati di selezione falsi positivi. Poiché gli schermi microfluidici a goccioline in genere superano altri schermi ad alta produttività, come i flussi di lavoro dei robot di pipettaggio, di ordini di grandezza in termini di produttività, questi eventi rari devono essere valutati e presi in considerazione.
Limitazioni
Nonostante i suoi vantaggi, ci sono limitazioni al metodo presentato. La fabbricazione di dispositivi microfluidici richiede esperienza e meticolosa attenzione ai dettagli, nonché uno stretto controllo sperimentale delle portate per garantire l'efficienza deterministica dell'incapsulamento. Questi aspetti possono richiedere l'ottimizzazione per diverse configurazioni sperimentali. Sebbene questo metodo si basi sulla microscopia a fluorescenza per il rilevamento del segnale, che richiede l'accesso ad apparecchiature di imaging adeguate, queste apparecchiature possono essere fabbricate utilizzando hardware open source, rendendole più accessibili. Inoltre, i microgel possono essere processati in citometria a flusso commerciale con ugelli di grandi dimensioni, migliorando ulteriormente l'accessibilità e la produttività sperimentale. Per questa analisi citometrica possono essere utilizzati anche i selezionatori di gocce.
Inoltre, mentre il metodo è progettato per rilevare segnali fluorescenti da plasmidi, colorazioni o altri marcatori, è limitato alle cellule che possono essere marcate in fluorescenza, il che potrebbe non essere applicabile a tutti i ceppi batterici o alle condizioni sperimentali. Tuttavia, il metodo può essere adattato per incorporare altri tipi di microscopie, come la microscopia a contrasto di fase o a campo chiaro, consentendo applicazioni di fenotipizzazione oltre la fluorescenza. Inoltre, può essere combinato con tecniche spettroscopiche come la spettroscopia FTIR o Raman, ampliando le sue capacità di analizzare le composizioni chimiche e le informazioni strutturali delle cellule incapsulate. Questi adattamenti ampliano la gamma della sua applicabilità, rendendolo uno strumento versatile per diversi contesti di ricerca.
Significato e applicazioni
I saggi tradizionali per la perdita di plasmidi19 non consentono una buona quantificazione del rapporto tra cellule che hanno perso la loro espressione, informazioni che possono essere molto importanti nella progettazione di metodi sperimentali e in varie applicazioni biologiche. Di solito, i tipi di colonie sono enumerati in saggi su piastra di agar, dove è possibile ottenere colonie isolate ben definite, come dimostrato nella Figura 4. Tuttavia, le colonie sovrapposte sono difficili da identificare con sicurezza; Nelle nostre mani, non sempre otteniamo una densità di colonia ottimale e sono necessarie molte piastre per ottenere buone statistiche sugli eventi di perdita di plasmidi a bassa frequenza. Il metodo proposto offre un approccio più robusto per quantificare con precisione i segnali fluorescenti provenienti da colonie isolate con un numero maggiore di colonie rispetto ai metodi analoghi della piastra di agar perché, nelle microgoccioline, le colonie si sviluppano separatamente, sono più piccole e sono facili da caricare nelle camere di imaging, consentendo la quantificazione basata sulla microscopia o sulla citometria a flusso di grandi numeri di colonie. Ciò può migliorare significativamente la rappresentazione statistica del metodo e consentire l'integrazione in altri flussi di lavoro gel-microdroplet.
L'utilizzo di hardware open-source11,20 consente ai ricercatori di personalizzare il design della workstation microfluidica e regolare con precisione il flusso; Pertanto, la dimensione delle particelle supporta vari tipi di cellule e condizioni sperimentali. Questa flessibilità si estende alla potenziale incorporazione di altri tipi di microscopia, come il contrasto di fase o la spettroscopia, ampliando l'applicabilità del metodo. La capacità del metodo di valutare la stabilità dei plasmidi in varie condizioni è fondamentale per le applicazioni che richiedono la ritenzione dei plasmidi senza selezione di antibiotici, in particolari condizioni di stress o in varie generazioni di colture. La versatilità e l'adattabilità del metodo presentato lo rendono prezioso per diverse applicazioni di ricerca in campi quali la biologia sintetica, il monitoraggio ambientale e la diagnostica clinica2.
Divulgazioni
Gli autori dichiarano che nessun interesse finanziario o relazione personale in competizione può aver influenzato il lavoro riportato in questo articolo.
Riconoscimenti
Questo lavoro fa parte dei progetti finanziati a T.W. da ANID FONDECYT Regular 1241621 e dal progetto Chang Zuckerberg Initiative "Latin American Hub for Bioimaging Through Open Hardware". T.W. è anche grato per il finanziamento del CIFAR, in qualità di Azrieli Global Scholar nel programma CIFAR MacMillan Multiscale Human.
Materiali
| Name | Company | Catalog Number | Comments |
| 1H,1H,2H,2H-Perfluoro-1-octanol | Sigma-Aldrich | 370533-25G | For breaking emulsion |
| 70% ethanol | For cell permeabilization | ||
| Agar-Agar | Winkler | 9002-18-0 | |
| Biopsy Punch | 0.75 mm and 1.8 mm | ||
| Blue LED transilluminator | IO Rodeo | ||
| Culture tube | 15 mL | ||
| Desiccator | With vacuum pump | ||
| Disposable cup | For mixing PDMS | ||
| Disposable fork | For mixing PDMS | ||
| E. coli TOP10 strain | |||
| FluoPi microscope | https://github.com/wenzel-lab/FluoPi | Green fluorescence imaging system for analyzing plates | |
| Fluorinated Oil | 3M | Novec 7500 | |
| Glass slide heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of microfluidic chip | |
| Glass Slides | |||
| Hotplate | Mechanic | For evaporating Aquapel | |
| Image analysis software | Fiji/ImageJ | 2.14.0/1.54f | |
| Incubator | Mundo Lab | MLAB Scientific / For incubation of plates and microgels | |
| Isopropanol | For cleaning glass slides | ||
| Kanamycin | 100 ug/mL concentration | ||
| L-shaped spreader | For spreading bacteria on agar plates | ||
| Master mold | Chip design on silicone or glass wafer | ||
| Microtubes | 2 mL | ||
| NaCl solution | Sodium chloride 0.9% w/v | ||
| Open-source hardware strobe-enhanced microscopy stage | https://github.com/wenzel-lab/flow-microscopy-platform | For bright-field microscopy | |
| Petri dish | Citotest | 2303-1090 | 90 x 15 mm |
| Pipette tip heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of pipette tip | |
| Plasma Cleaner | Diener Electronic | 117056 | For bonding PDMS with a glass slide |
| Plasmid pCA_Odd1 | Encodes sfGFP and kanamycin resistance | ||
| Polytetrafluoroethylene (PTFE) tubing | Adtech Polymer Engineering Ltd | ||
| Pre-mixed Luria Bertoni medium | US Biological Life Science | L1520 | |
| Propidium iodide (PI) | For staining | ||
| Raspberry Pi-based pressure and flow controller system | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller | For controlling pressure and flow rates | |
| Silicone elastomer base | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Silicone elastomer curing agent | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Spectrophotometer | For measuring absorbance | ||
| SQUID microscope | https://github.com/wenzel-lab/SQUID-bioimaging-platform | Multi-fluorescence imaging system for analyzing stained cells | |
| Sterile loop | For picking a colony and streaking plating | ||
| Surfactant | Sphere Fluidics | Pico-Surf | |
| Syringes | NIPRO | With filters and tubing | |
| Temperature-controlled shaker | Mundo Lab | DLAB HCM100-Pro | |
| Tweezer | |||
| Ultra-low gelling temperature agarose | Sigma-Aldrich | A2576-5G | For generating hydrogel beads |
| Water repelent solution (fluoroalkyl silane) | Aquapel | For treating microchannels of PDMS device |
Riferimenti
- Wein, T., Hülter, N. F., Mizrahi, I., Dagan, T. Emergence of plasmid stability under non-selective conditions maintains antibiotic resistance. Nat Commun. 10 (1), 2595(2019).
- Rodríguez-Beltrán, J., DelaFuente, J., León-Sampedro, R., MacLean, R. C., Millán, ÁS. Beyond horizontal gene transfer: The role of plasmids in bacterial evolution. Nat Rev Microbiol. 19 (6), 347(2021).
- Wein, T., Dagan, T. Plasmid evolution. Curr Biol. 30 (19), R1158-R1163 (2020).
- Chen, S., Larsson, M., Robinson, R. C., Chen, S. L. Direct and convenient measurement of plasmid stability in lab and clinical isolates of E. coli. Sci Rep. 7 (1), 4788(2017).
- Rouches, M. V., Xu, Y., Cortes, L. B. G., Lambert, G. A plasmid system with tunable copy number. Nat Commun. 13 (1), 3908(2022).
- Silva, F., Queiroz, J. A., Domingues, F. C. Evaluating metabolic stress and plasmid stability in plasmid DNA production by Escherichia coli. Biotechnol Adv. 30 (3), 691-708 (2012).
- Wang, R., et al. Construction of novel pJRD215-derived plasmids using chloramphenicol acetyltransferase (cat) gene as a selection marker for Acidithiobacillus caldus. PLoS ONE. 12 (8), e0183307(2017).
- Moragues, T., et al. Droplet-based microfluidics. Nat Rev Methods Primers. 3 (1), 32(2023).
- Vitalis, C., Wenzel, T. Leveraging interactions in microfluidic droplets for enhanced biotechnology screens. Current Opinion in Biotechnology. 82, 102966(2023).
- Sinha, N., Subedi, N., Wimmers, F., Soennichsen, M., Tel, J. A pipette-tip based method for seeding cells to droplet microfluidic platforms. J Vis Exp. (144), e57848(2019).
- Wenzel, T. Open hardware: From DIY trend to global transformation in access to laboratory equipment. PLOS Biol. 21 (1), e3001931(2023).
- Murillo, L. F. R., Wenzel, T. Welcome to the journal of open hardware. J Open Hardware. 1 (1), (2017).
- Shin, J. H., Choi, S. Open-source and do-it-yourself microfluidics. Sens Actuators B Chem. 347, 130624(2021).
- Pryszlak, A., et al. Enrichment of gut microbiome strains for cultivation-free genome sequencing using droplet microfluidics. Cell Rep Methods. 2 (1), 100137(2021).
- Pollak, B., et al. Universal loop assembly: open, efficient and cross-kingdom DNA fabrication. Biology. 5 (1), (2020).
- Volkmer, B., Heinemann, M. Condition-dependent cell volume and concentration of Escherichia coli to facilitate data conversion for systems biology modeling. PLoS ONE. 6 (7), e23126(2011).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , (2020).
- Abate, A. R., Weitz, D. A. Faster multiple emulsification with drop splitting. Lab Chip. 11 (11), 1911-1915 (2011).
- Lau, B. T. C., Malkus, P., Paulsson, J. New quantitative methods for measuring plasmid loss rates reveal unexpected stability. Plasmid. 70 (3), 353-361 (2013).
- Oellermann, M., et al. Open hardware in science: The benefits of open electronics. Integr Comp Biol. 62 (4), 1061-1075 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon