Method Article
Plasmidstabilitätsanalyse mit Open-Source-Tröpfchen-Mikrofluidik
In diesem Artikel
Zusammenfassung
Es wird ein zugänglicher, quelloffener mikrofluidischer Workflow für die parallelisierte Analyse der Plasmidretention in Bakterien vorgestellt. Durch den Einsatz von Fluoreszenzmikroskopie zur Quantifizierung des Plasmidvorhandenseins in einzelligen Mikrokolonien in Gel-Mikrotröpfchen bietet diese Methode eine präzise, zugängliche und skalierbare Alternative zur herkömmlichen Plattenzählung.
Zusammenfassung
Plasmide spielen eine wichtige Rolle in der synthetischen Biologie, indem sie die Einführung und Expression fremder Gene in verschiedenen Organismen ermöglichen und so den Aufbau biologischer Schaltkreise und Signalwege innerhalb und zwischen Zellpopulationen erleichtern. Für viele Anwendungen ist die Aufrechterhaltung funktionsfähiger Plasmide ohne Antibiotikaauswahl von entscheidender Bedeutung. In dieser Studie wird ein Open-Hardware-basierter mikrofluidischer Arbeitsablauf zur Analyse der Plasmidretention durch die Kultivierung einzelner Zellen in Gel-Mikrotröpfchen und die Quantifizierung von Mikrokolonien mittels Fluoreszenzmikroskopie vorgestellt. Dieser Ansatz ermöglicht die parallele Analyse zahlreicher Tröpfchen und Mikrokolonien, was im Vergleich zur herkömmlichen Plattenzählung eine größere statistische Aussagekraft bietet und die Integration des Assays in andere mikrofluidische Tröpfchen-Arbeitsabläufe ermöglicht. Durch die Verwendung von Plasmiden, die fluoreszierende Proteine exprimieren, zusammen mit einer unspezifischen fluoreszierenden DNA-Färbung können einzelne Kolonien identifiziert und anhand des Plasmidverlusts oder der Fluoreszenzmarkerexpression unterschieden werden. Insbesondere bietet dieser fortschrittliche Workflow, der mit Open-Source-Hardware implementiert wurde, eine präzise Durchflussregelung und ein präzises Temperaturmanagement sowohl der Probe als auch des mikrofluidischen Chips. Diese Funktionen verbessern die Benutzerfreundlichkeit, Robustheit und Zugänglichkeit des Workflows. Während sich die Studie auf Escherichia coli als experimentelles Modell konzentriert, liegt das wahre Potenzial der Methode in ihrer Vielseitigkeit. Es kann für verschiedene Studien angepasst werden, die eine Quantifizierung von Fluoreszenzsignalen aus Plasmiden oder Färbungen erfordern, sowie für andere Anwendungen. Die Einführung von Open-Source-Hardware erweitert das Potenzial für die Durchführung von Hochdurchsatz-Bioanalysen mit zugänglicher Technologie in verschiedenen Forschungsumgebungen.
Einleitung
Plasmide sind essentielle genetische Elemente in prokaryotischen Zellen und tragen durch lateralen DNA-Transfer und schnelle Anpassung an Umweltveränderungen wesentlich zur mikrobiellen Evolution bei 1,2. Diese extrachromosomalen DNA-Moleküle tragen Gene, die vorteilhafte Eigenschaften wie Antibiotikaresistenz, Stoffwechselfunktionen und Virulenzfaktoren aufweisen, was sie für die mikrobiologische Forschung, die synthetische Biologie und Evolutionsstudien wertvoll macht2. Die Aufrechterhaltung von Plasmiden in Zellpopulationen ist jedoch aufgrund der metabolischen Belastung durch Replikation und Segregation eine Herausforderung, was häufig zu einem Plasmidverlust ohne Selektionsdruck führt3. Darüber hinaus erfordert eine stabile Vererbung Mechanismen wie Toxin-Antitoxin- und Partitionierungssysteme, was die Komplexität der Plasmidpflege erhöht. Die Beurteilung der Plasmidstabilität unter verschiedenen Bedingungen ist sowohl für die Grundlagenforschung als auch für praktische Anwendungen, bei denen Plasmide als primäres Forschungselement verwendet werden, von entscheidender Bedeutung 4,5. Die meisten aktuellen Methoden zur Beurteilung der Plasmidstabilität weisen erhebliche Einschränkungen auf: Auf Durchflusszytometrie basierende Methoden liefern indirekte Daten auf Populationsebene, erfordern teure Geräte und es fehlt an direkter Visualisierung von Kolonien6. Bulk-Transkriptomik- und Proteomik-Methoden sind kostspielig, liefern nur durchschnittliche zelluläre Reaktionen und können die Plasmidretention in einzelnen Kolonien nicht direkt quantifizieren6. Traditionelle Methoden wie serielle Verdünnung und Passage sind einfach, aber zeitaufwändig und es mangelt an Präzision und Darstellbarkeit7. Insgesamt bleibt die quantitative Ableitung oder Projektion der Anzahl der Kolonien, die ein spezifisches funktionelles Plasmid über die Zeit oder den Selektionsdruck beibehalten, eine Herausforderung.
Um diese Herausforderungen zu bewältigen, wird ein neuartiger mikrofluidischer Arbeitsablauf unter Verwendung von Open-Hardware-Forschungsinstrumenten vorgestellt, um Fluoreszenzsignale in mehreren isolierten Bakterienkolonien zu quantifizieren, wobei Escherichia coli als Modell dient. Diese Methode ermöglicht einen hohen Durchsatz und eine präzise Analyse der Plasmidretention über verschiedene Bedingungen oder Selektivdrücke. Die Analyse mit Einzelzellauflösung bietet eine präzise Methode zur Manipulation isolierter Kolonien und liefert empfindliche Daten zur Plasmidquantifizierung, die bei der Beurteilung der Retentions- und Verlustraten helfen können4.
Die Mikrofluidik, insbesondere die Tröpfchenmikrofluidik, hat sich als leistungsfähiges Werkzeug für die Verkapselung und Manipulation einzelner Zellen in kontrollierten Umgebungen erwiesen8. Insbesondere können Mikrogel-Tröpfchen einzelne Zellen für einen hohen Durchsatz und eine präzise Analyse einkapseln, ohne dass Tröpfchen in Öl9 suspendiert werden müssen, was eine kontrollierte Untersuchung der Plasmiddynamik in einer definierten Mikroumgebung ermöglicht. Nach der Verkapselung von Zellsuspensionen direkt von einer Pipettenspitze10 können Fluoreszenztechniken verwendet werden, um das Wachstum von Mikrokolonien innerhalb von Tröpfchen zu überwachen, was eine detaillierte Analyse der Plasmidretention und -seigerung unter verschiedenen Selektionsdrücken ermöglicht3.
Zu den Vorteilen dieser Methode gegenüber herkömmlichen Bulk-Kulturtechniken gehören eine höhere Präzision, eine geringere Variabilität und die Möglichkeit, Hochdurchsatzanalysen durchzuführen. Die Open-Source-Mikrofluidik-Technologie überwindet die Einschränkungen teurer proprietärer Systeme, wie z. B. Probleme der Zugänglichkeit, Anpassungsfähigkeit und Wartung, die oft den Forschungsfortschritt behindern 11,12,13. Durch die Demonstration der Anwendung des fortschrittlichen experimentellen Arbeitsablaufs der Plasmid-Retentionsanalyse in Mikrogelen mit Open-Source-Instrumenten wird eine zugängliche und zuverlässige Methode für die Forschung in der Plasmidbiologie, in Anwendungen der synthetischen Biologie und in mikrofluidischen Tröpfchenanalysetechniken bereitgestellt.
Zusammenfassend stellt dieser Artikel eine zugängliche Methode zur quantitativen Bewertung der Plasmidretention in E. coli mit hoher statistischer Aussagekraft vor. Die Fähigkeiten dieser Methode machen sie zu einem wertvollen Werkzeug, um das Verständnis der Plasmidbiologie zu verbessern und die Anwendungen der synthetischen Biologie zu verbessern.
Protokoll
Abbildung 1bietet einen schematischen Überblick zur Beurteilung der Plasmidstabilität in E. coli. Einzelheiten zu den verwendeten Reagenzien und den verwendeten Geräten sind in der Materialtabelle aufgeführt. Die Rohdaten und die Visualisierungsskripte sind unter https://doi.org/10.17605/OSF.IO/6YWJK verfügbar.
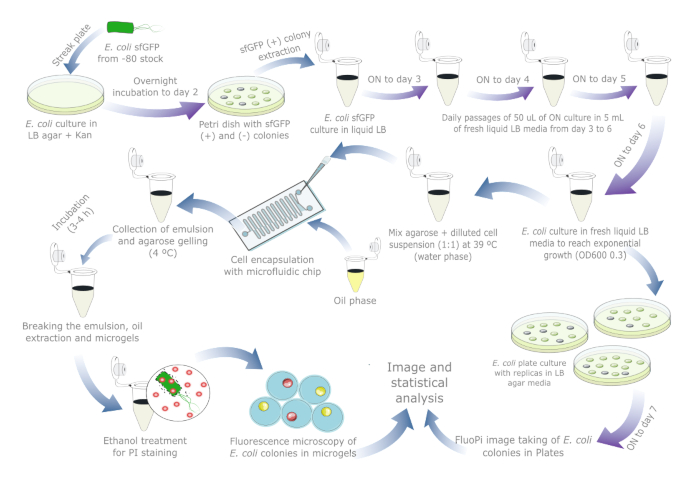
Abbildung 1: Tagweises Protokoll zur Beurteilung der Plasmidstabilität in E. coli. Blaue Pfeile zeigen die Schritte während des Tages an, und violette Pfeile zeigen die Inkubation über Nacht an. Jede Inkubation von Flüssigkeit und Agar wurde bei 37 °C zusammen mit einem separaten Negativkontrollröhrchen/einer separaten Negativkontrollplatte durchgeführt. Beachten Sie, dass die Vorbereitung und die Passagen der Zellkultur für reale Proben, bei denen möglicherweise bereits ein Plasmidverlust aufgetreten ist, nicht erforderlich sind, sodass das Protokoll auf ein oder zwei Tage reduziert werden sollte, wenn eine Plattenreferenzkultur enthalten ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. Mikrofluidische Chipvorbereitung
HINWEIS: In diesem Protokoll können verschiedene kommerzielle oder kundenspezifische Chipdesigns für die Zellverkapselung verwendet werden, die in der Lage ist, Wasser-in-Öl-Tröpfchen mit einem Durchmesser von weniger als 100 μm in einem Tropfregime zu erzeugen. Für diese Studie wurde der Chip nach der gleichen Design- und Herstellungsmethode entworfen und hergestellt (siehe Artikeldaten https://doi.org/10.17605/OSF.IO/6YWJK), über die in einem zuvor veröffentlichten Berichtberichtet wurde 14.
- Gewinnen oder bereiten Sie einen PDMS-auf-Glas-Mikrofluidik-Chip mit einer Masterform vor, die für die Erzeugung von Gel-Mikrotröpfchen entwickelt wurde.
- Injizieren Sie eine wasserabweisende Lösung (Fluoralkylsilan) in den Chip, um die inneren Mikrokanäle hydrophob zu machen. Gießen Sie die Lösung in die Einlässe und stellen Sie sicher, dass alle Kanäle mit Flüssigkeit gefüllt sind. Lassen Sie die gefüllten Kanäle etwa 30–60 s ruhen.
- Entfernen Sie die Lösung aus dem Gerät, indem Sie Luft in die inneren Mikrokanäle ausstoßen. Verwenden Sie eine leere Luftspritze, um die Luft zu spülen, und ein saugfähiges Tuch über die anderen Anschlüsse, um Spritzer zu vermeiden.
- Das behandelte Gerät bei 65 °C 15 min auf einer Herdplatte backen, um die überschüssige Lösung zu verdampfen. Alternativ können Sie das Gerät über Nacht im Kühlschrank (4 °C) lagern.
HINWEIS: Der mikrofluidische Chip ist einsatzbereit. Das Protokoll kann hier pausiert werden.
2. Vorbereitung der Probe
- Zellernte
HINWEIS: Zellkulturen in Tröpfchen können relevante Daten zur Plasmiddynamik liefern. Als experimentelles Bakterienmodell wird ein E. coli TOP10-Stamm mit dem Plasmid pCA_Odd1 (siehe hinterlegte Daten https://doi.org/10.17605/OSF.IO/6YWJK) verwendet, der für ein grün fluoreszierendes Superfolder-Protein (sfGFP) und Kanamycin-Resistenz15 kodiert. Bakterien, Plasmide und Wachstumsmedien können je nach Versuchssystem variieren.- Bereiten Sie Luria Bertoni (LB)-Agar zu, indem Sie 25 g vorgemischtes LB und 12 g Agar in 800 mL destilliertem Wasser (dH2O) auflösen. Bereiten Sie außerdem flüssige LB-Medien vor, indem Sie 25 g vorgemischtes LB in 800 mL dH2O auflösen. Autoklavieren Sie die Lösungen und lassen Sie sie auf etwa 60 °C abkühlen. Wechseln Sie für die nächsten Schritte in eine sterile Umgebung, z. B. in eine Flow-Hood.
HINWEIS: Das vorgemischte LB-Pulver enthält 10 g Trypton, 5 g Hefeextrakt und 10 g NaCl. Es können auch selbstgemischte LB-Agar-Medien verwendet werden. - 50 μl Kanamycin (hergestellt mit 100 mg/ml) zu 50 mL flüssigem LB-Agar (Endkonzentration: 100 μg/ml) hinzufügen. Mischen Sie die Lösung, indem Sie das Röhrchen einige Male umdrehen.
- Gießen Sie etwa 15 mL flüssiges LB-Agar pro Petrischale (90 mm x 15 mm). Bereiten Sie zwei Platten vor: eine für die Negativkontrolle (Kontaminationsüberwachung) und eine für die Versuchskultur. Lassen Sie die LB-Agar-Lösung abkühlen und erstarren, bis sich die Farbe auf beiden Platten von dunkel zu klar ändert.
- Verwenden Sie eine sterile Schlaufe, um den E. coli-Stamm mit der Streifenplattentechnik auf der Kulturplatte zu verteilen. Stellen Sie den Glycerinfond E. coli sofort nach Gebrauch wieder in die -80 °C. Verschließen Sie beide Platten und inkubieren Sie sie über Nacht bei 37 °C (Tag 1).
- An Tag 2 ist die Negativkontrollplatte auf eine Kolonie zu überprüfen, um eine Kontamination zu signalisieren (wenn ja, wiederholen Sie die Schritte aus 2.1.4.). Identifizieren Sie einzelne fluoreszierende Kolonien auf der E. coli-Platte mit einem Blaulicht-Transilluminator, um einen Flüssigkulturbestand vorzubereiten.
- Öffnen Sie unter sterilen Bedingungen die Platte und verwenden Sie eine sterile Schlaufe oder eine 200-μl-Pipettenspitze, um die ausgewählte Kolonie zu entnehmen und sie in ein Kulturröhrchen mit 5 mL frischem flüssigem LB-Medium und 5 μL Kanamycin (hergestellt mit 100 mg/ml) zu übertragen. Bereiten Sie ein Negativkontrollröhrchen ohne Inokulation für die Kontaminationsüberwachung vor. Inkubieren Sie die Kulturröhrchen über Nacht bei 37 °C unter Schütteln bei 220 U/min.
- Die Probe wird für weitere 3 Tage in Medien ohne Antibiotika behandelt, um die Bedingungen zu simulieren, unter denen ein Plasmidverlust auftreten kann. An den Tagen 3, 4 und 5 werden 50 μl der Übernachtkultur ohne Antibiotika in ein neues Kulturröhrchen mit 5 ml flüssigem LB umgefüllt. Über Nacht bei 37 °C und Schütteln bei 220 U/min inkubieren. Wiederholen Sie diesen Schritt bis zum 6. Tag und erhalten Sie nach insgesamt vier Passagen eine letzte 5-ml-Kultur.
- 50 μl aus der letzten Übernachtkultur in ein neues Kulturröhrchen mit 2 ml flüssigem LB überführen. Lassen Sie die Kultur mit einem Spektralphotometer eine optische Dichte (OD600) von 0,3 erreichen (ca. 3–4 Stunden).
HINWEIS: Die Frischkultur sollte verwendet werden, bevor der OD600-Gehalt ansteigt, um sicherzustellen, dass sich die Bakterien in der logarithmischen Wachstumsphase befinden. Wenn die gewünschte Konzentration nicht erreicht wird, wiederholen Sie den Vorgang ab Schritt 2.1.8.
- Bereiten Sie Luria Bertoni (LB)-Agar zu, indem Sie 25 g vorgemischtes LB und 12 g Agar in 800 mL destilliertem Wasser (dH2O) auflösen. Bereiten Sie außerdem flüssige LB-Medien vor, indem Sie 25 g vorgemischtes LB in 800 mL dH2O auflösen. Autoklavieren Sie die Lösungen und lassen Sie sie auf etwa 60 °C abkühlen. Wechseln Sie für die nächsten Schritte in eine sterile Umgebung, z. B. in eine Flow-Hood.
- Zell-Agarose-Mischung zur Verkapselung
HINWEIS: Eine Konzentrationskontrolle ist unerlässlich, um eine Tröpfchenverkapselung mit einzelnen Zellen zu gewährleisten. Die erforderliche Zellkonzentration kann für eine Zielverkapselungsrate und ein spezifisches Tröpfchenvolumen berechnet werden, wie im folgenden Beispiel gezeigt:
Tröpfchenvolumen (V-Tropfen): Wenn 50 μm Tröpfchen erzeugt werden
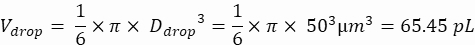
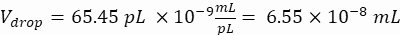
Gewünschte Zellen pro Tröpfchen (Cpd): Durchschnittlich eine Zelle pro fünf Tröpfchen (0,2 Zellen/Tröpfchen)
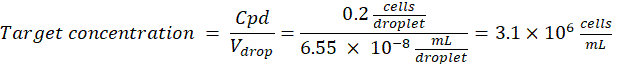
Verdünnungsfaktor: Die anfängliche Zellkonzentration wird in Schritt 2.1.8 ermittelt
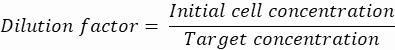
Hier werden 50 μm oder 100 μm (ohne Tröpfchenteilung) Tröpfchen (65–520 pL) erzeugt, wobei etwa eine Zelle pro fünf Tröpfchen verkapselt wird, oder 1,6 Zellen pro Tröpfchen ohne Teilung. Für E. coli ist der Umrechnungsfaktor von 1 OD600-Einheit ≈ 7,8 x 108 Zellen/ml16 zu verwenden. Multiplizieren Sie den OD600-Wert aus Schritt 2.1.8 mit dem Umrechnungsfaktor, um die Anfangskonzentration (Zellen/ml) der Kultur zu erhalten.- E. coli aus dem vorbereiteten Kulturbestand in flüssigem LB ohne Kanamycin in einer Konzentration von 6,2e+6 Zellen/ml (Zielkonzentration für Tröpfchen mit einem Durchmesser von 50 μm) resuspendieren. Bewahren Sie die Bakteriensuspension bei Raumtemperatur auf, bis sie mit Agarose vermischt wird.
HINWEIS: Antibiotika unterdrücken die Bildung negativer Mikrokolonien, daher ist es wichtig, sie aus den Medien für Plasmidverlustexperimente auszuschließen. - Agarose mit extrem niedriger Geliertemperatur herstellen, indem es in flüssiger LB bei einer Konzentration von 2 % (w/v) auf 90 °C erhitzt wird. Schütteln Sie die Mischung 10 Minuten lang in einem temperaturkontrollierten Shaker.
- Reduzieren Sie die Temperatur des Thermoschüttlers auf 39 °C, um die Agaroselösung abzukühlen. Legen Sie parallel dazu das Bakteriensuspensionsrohr für 4 Minuten in den Thermoschüttler, um es auf 39 °C zu erwärmen.
- Mischen Sie die Bakterien und die Agarosesuspensionen im Verhältnis 1:1, um eine Agarosekonzentration von 1% (w/v) mit der Zellsuspension bei 3,1e+6 Zelle/ml zu erhalten. Die Negativkontrolllösung (Kontaminationsüberwachung) mit der gleichen Agarosekonzentration wird mit flüssigem LB anstelle der Bakteriensuspension hergestellt.
HINWEIS: Die Agarose-Zellsuspension sollte schnell verwendet werden, um Konzentrationsänderungen aufgrund von Bakterienwachstum zu vermeiden. Halten Sie den Thermoschüttler bei 39 °C, um die Agaroseflüssigkeit zu halten, bis sie zur Tröpfchenerzeugung in die Spitzenheizung geladen wird.
- E. coli aus dem vorbereiteten Kulturbestand in flüssigem LB ohne Kanamycin in einer Konzentration von 6,2e+6 Zellen/ml (Zielkonzentration für Tröpfchen mit einem Durchmesser von 50 μm) resuspendieren. Bewahren Sie die Bakteriensuspension bei Raumtemperatur auf, bis sie mit Agarose vermischt wird.
3. Anbau von Einzelkolonien mit hohem Durchsatz
- Versuchsaufbau
- Erstellen oder beziehen Sie die vollständige Open-Source-Durchflussplattform (siehe https://doi.org/10.17605/OSF.IO/6YWJK), einschließlich Gasdrucktreibern und Durchflusssensoren. Alternativ können Sie die einfachere Open-Source-Hardware-Stroboskop-Mikroskopiestufe erstellen (siehe Build-Anweisungen https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ und Projekt-Repository https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage).
HINWEIS: Man kann auch einen herkömmlichen Mikrofluidik-Aufbau mit einem kommerziellen Mikroskop, einer Hochgeschwindigkeitskamera und Probenerhitzungsfunktionen verwenden. - Integrieren Sie das Open-Source-System auf Raspberry Pi-basiertem Druck- und Durchflussreglersystem, wie im dafür vorgesehenen Repository (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller) dokumentiert.
HINWEIS: Das Modulsteuerelement wird veranschaulicht und im Datenrepository (https://doi.org/10.17605/OSF.IO/6YWJK. Alternativ können traditionelle Druckregler oder Spritzenpumpen mit hohem Drehmoment eingesetzt werden. - Fügen Sie einen Glasobjektträger und Pipettenspitzenheizungen (https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring) in das Setup ein, um die Temperatur der Agarosezellenprobe beim Eintritt in den Chip zu steuern.
HINWEIS: Diese Heizungen, die im Datenrepository (https://github.com/wenzel-lab/flow-microscopy-platform und https://doi.org/10.17605/OSF.IO/6YWJK) abgebildet sind, sind ein wichtiges Merkmal des Open-Source-Steuerungssystems, das die Arbeit mit Agarose ermöglicht und in anderen kommerziellen Systemen möglicherweise nicht verfügbar ist.
- Erstellen oder beziehen Sie die vollständige Open-Source-Durchflussplattform (siehe https://doi.org/10.17605/OSF.IO/6YWJK), einschließlich Gasdrucktreibern und Durchflusssensoren. Alternativ können Sie die einfachere Open-Source-Hardware-Stroboskop-Mikroskopiestufe erstellen (siehe Build-Anweisungen https://wenzel-lab.github.io/strobe-enhanced-microscopy-stage/ und Projekt-Repository https://github.com/wenzel-lab/strobe-enhanced-microscopy-stage).
- Einzelzell-Verkapselung
- Positionieren Sie den Mikrofluidik-Chip auf dem stroboskopverstärkten Mikroskopietisch und stellen Sie sicher, dass der Übergang zur Tröpfchenerzeugung (Schnittpunkt der wässrigen und der Ölphase) sichtbar ist.
- Stellen Sie die Pipettenspitzenheizung und die Glasschiebeheizung über die Steuerungssoftwareschnittstelle auf 40 °C ein.
- Verwenden Sie eine Spritze mit Schlauch und PDMS-Stopfen, um die 1%ige Agarosemischung mit Zellsuspension in eine 200-μl-Pipettenspitze zu füllen. Setzen Sie die Spitze in die Spitzenheizung ein und platzieren Sie sie auf dem Einlass der wässrigen Phase im Mikrofluidik-Chip. Tauschen Sie die PDMS-Dichtung der Spitze gegen eine Dichtung aus, die an den Schlauch des Durchflusskontrollsystems angeschlossen ist, und starten Sie die Infusion der Zellsuspension.
- Führen Sie das Ende des Auslassschlauchs in einen Abflussschlauch ein und stellen Sie die Durchflussraten oder Drücke von zwei Phasen auf der Benutzeroberfläche ein, um die Flüssigkeit langsam in den mikrofluidischen Kanal zu leiten. Verwenden Sie 200 μL/h (180 mbar) für die wässrige Phase und 1700 μL/h (320 mbar) für die Ölphase. Warten Sie 1 Minute für die Stabilisierung der Tröpfchenbildung.
HINWEIS: Die Druckwerte hängen von den Kanalgrößen des Chipdesigns ab, und die Durchflusswerte müssen möglicherweise für verschiedene Designs der Tropfenerzeugungsstelle angepasst werden. - Sobald die Tröpfchenerzeugung stabil ist, übertragen Sie den Abfall und den Auffangschlauch in das Auffangrohr. Fahren Sie mit dem Sammeln von Tröpfchen fort, bis das Probenreservoir leer ist. Wiederholen Sie die Schritte 3.2.3 bis 3.2.5, um die Negativkontrolllösung aus Schritt 2.2.4 einzukapseln.
HINWEIS: Die Probenentnahme sollte innerhalb von 15 Minuten abgeschlossen sein. - Lagern Sie die Sammelröhrchen während der Tröpfchenerzeugung auf Eis oder stellen Sie sie nach dem Experiment für 1 h bei 4 °C auf, damit Agarose in den Tröpfchen gelieren kann.
HINWEIS: Der Mikrofluidik-Chip kann wiederverwendet werden, wenn die Mikrokanäle nicht verstopft bleiben und dieselbe Suspension geladen wird. Entsorgen Sie die Pipettenspitze nach Abschluss der Emulsionserzeugung (Schritte 3.2.3–3.2.5).
- Koloniewachstum und Freisetzung aus der Emulsion
- Die bakterienhaltigen Gel-Mikrotröpfchen und die Negativkontrolle in Tröpfchen werden in eine auf 37 °C eingestellte Inkubationskammer überführt.
- Inkubieren Sie die Mikrotröpfchen mindestens 4 Stunden lang oder über Nacht, um ein ausreichendes Koloniewachstum zu ermöglichen. Bestätigen Sie, dass die Negativkontrolle keine Anzeichen einer Kontamination durch Hellfeldmikroskopie zeigt.
- Um die Kolonien aus der Emulsion zu lösen, entfernen Sie mit einer Pipette oder einer Spritze mit einer Nadel (hier wurde die Nadel der Größe 21 G verwendet) so viel Öl wie möglich von unterhalb der Gel-Mikrotröpfchenemulsion.
- Übertragen Sie 50 μl der Gel-Mikrotröpfchen in ein neues Mikroröhrchen und lagern Sie es bei 4 °C für die weitere Tröpfchenanalyse. Zu der restlichen Emulsion wird eine 1:1-Mischung aus fluoriertem Öl mit 1H,1H,2H,2H-Perfluor-1-octanol (PFO) in einem Volumen hinzugefügt, das der Emulsion entspricht.
- Geben Sie ca. 200 μl phosphatgepufferten Kochsalzpuffer (PBS) oder 0,9 % w/v NaCl-Puffer auf die Emulsion. Wirbeln Sie die Mischung und schleudern Sie sie kurz in einer Zentrifuge mit fester Drehzahl herunter.
- Entfernen Sie vorsichtig die Ölphase von der Unterseite der Flüssigkeitsschnittstelle und entsorgen Sie 100 μl PBS von der Oberseite. Wiederholen Sie die Schritte 3.3.4 bis 3.3.5, um gewaschene Mikrogele in PBS-Puffer mit minimalen oder keinen Ölrückständen zu erhalten.
HINWEIS: Die Mikrogele setzen sich an der Flüssigkeitsgrenzfläche ab; Vermeiden Sie es, sie zusammen mit der Ölphase zu entfernen.
4. Analyse einzelner Völker
- Zellfärbung
HINWEIS: Für dieses Protokoll funktionieren viele verschiedene Fleckenkombinationen. Im Wesentlichen sollte eine DNA- oder Zellwandfärbung gewählt werden, die eine andere Farbe als das vom Plasmid kodierte fluoreszierende Protein aufweist und die unter Verwendung der verfügbaren Filterkombinationen am Fluoreszenzmikroskop analysiert werden kann. Hier wurde die DNA von Zellen mit Propidiumiodid (PI) gefärbt, um ihre Fluoreszenz von dem grün fluoreszierenden Protein zu unterscheiden, das auf den Plasmiden kodiert ist, aber es können viele andere DNA-Färbungen verwendet werden.
ACHTUNG: PI ist ein potentielles Karzinogen und muss mit geeigneter persönlicher Schutzausrüstung gehandhabt werden. Entsorgen Sie den Farbstoff sicher und in Übereinstimmung mit den örtlichen Vorschriften.- Zentrifugieren Sie die gewaschenen Mikrogele bei ca. 80 x g für 5 min bei Raumtemperatur. Entsorgen Sie den Überstand mit einer Pipette.
- Übertragen Sie 50 μl der Mikrogele in ein neues Mikroröhrchen, das als Negativkontrolle für die Ethanolbehandlung dient.
HINWEIS: PI-Färbung dringt nur in Zellen mit kompromittierten Membranen ein, wie z. B. solche, die von der Ethanolbehandlung betroffen sind. - Geben Sie ein gleiches Volumen von 70% Ethanol zu den restlichen Mikrogelen und mischen Sie es kurz mit einem Wirbel. 15 Minuten bei Raumtemperatur inkubieren, um die Bakterienmembranen für die PI-Färbung zu permeabilisieren. Wiederholen Sie Schritt 4.1.1.
- Geben Sie ein gleiches Volumen von 0,9 % w/v NaCl zu den Mikrogelen und wirbeln Sie sie kurz vortexen. Wiederholen Sie Schritt 4.1.1.
- Geben Sie 2 μl PI (1 mg/ml) in beide Mikroröhrchenproben. Gründlich mischen und im Dunkeln bei Raumtemperatur 15 min inkubieren.
HINWEIS: Wenn die Negativkontrolle über die Fluoreszenzmikroskopie ein rotes Fluoreszenzsignal zeigt, könnte die Integrität der Kolonien während der Exposition gegenüber anderen Lösungen in den vorangegangenen Schritten beeinträchtigt worden sein.
- Mikroskopie
HINWEIS: Tröpfchen und Mikrogele werden mit einem inversen Epifluoreszenzmikroskop abgebildet, um die Tröpfchengrößenverteilung und die Fluoreszenz von Bakterienkolonien in Mikrogelen zu ermitteln (Abbildung 2). Hier kommt eine Open-Source-Plattform für inverse Mikroskopie (https://github.com/wenzel-lab/SQUID-bioimaging-platform)17 mit einem 10x 0,3NA-Objektiv, einem weißen LED-Array für die Hellfeldbeleuchtung und einer 470nm LED für die Anregung zum Einsatz. Kommerzielle Epi-Fluoreszenzmikroskope können zur Abbildung von Tröpfchen und Mikrogelen verwendet werden. Eine Kalibrierung ist erforderlich, da die Beleuchtung und die Filter je nach Modell, Marke und verwendetem fluoreszierendem Protein variieren.- Übertragen Sie 2 μl Gel-Mikrotröpfchen in einen Objektträger der Bildgebungskammer und fügen Sie 5 μl fluoriertes Öl hinzu, um eine Monoschicht aus Tröpfchen für eine optimale Bildgebung zu bilden.
HINWEIS: Zellzählkammern oder einfache mikrofluidische Kammern können helfen, die Emulsion dünn zu verteilen und den Trocknungsprozess zu verlangsamen. - Aktivieren Sie am Mikroskop die weiße LED-Matrixbeleuchtung von oben für Hellfeld-Bildgebung. Montieren Sie den vorbereiteten Objektträger, fokussieren Sie sich auf die Probe und lokalisieren Sie eine Monoschicht aus Tröpfchen. Nehmen Sie ein Hellfeldbild auf.
- Ohne die Probe zu bewegen, können Sie ein Fluoreszenzbild der Kolonien aufnehmen, indem Sie zur Anregung auf die 470-nm-LED umschalten. Stellen Sie das Filterrad so ein, dass es mit dem grünen Wellenlängenfilter für die sfGFP-Bildgebung ausgerichtet ist. Scannen Sie alle Bereiche, die Monoschichten von Tröpfchen enthalten, und wiederholen Sie die Schritte 4.2.2 bis 4.2.3, um die statistische Robustheit der Tröpfchenanalyse sicherzustellen.
- Übertragen Sie 2 μl der gefärbten Mikrogele in einen Chip der Bildgebungskammer und 5 μl 0,9 % w/v NaCl, um eine Monoschicht aus Mikrogelen zu bilden. Versiegeln Sie den Ein- und Auslass des Chips, um eine Verdunstung während der Bildgebung zu verhindern.
HINWEIS: Verdunstung kann die Kolokalisation der fluoreszierenden Markierungen beeinträchtigen. Mikrofluidische Kammern werden für eine längere Bildgebung empfohlen. - Wiederholen Sie die Schritte 4.2.2 bis 4.2.3. Passen Sie den Filter für das rote Wellenlängenintervall für die PI-Bildgebung an und nehmen Sie die entsprechenden Bilder auf.
- Nachdem Sie die Bildgebung an einer Stelle abgeschlossen haben, suchen Sie den nächsten geeigneten Bereich auf dem Objektträger mit einer Monoschicht aus Mikrogelen und wiederholen Sie den Bildgebungsvorgang, um die statistische Robustheit und eine umfassende Analyse der Probe zu gewährleisten.
- Übertragen Sie 2 μl Gel-Mikrotröpfchen in einen Objektträger der Bildgebungskammer und fügen Sie 5 μl fluoriertes Öl hinzu, um eine Monoschicht aus Tröpfchen für eine optimale Bildgebung zu bilden.
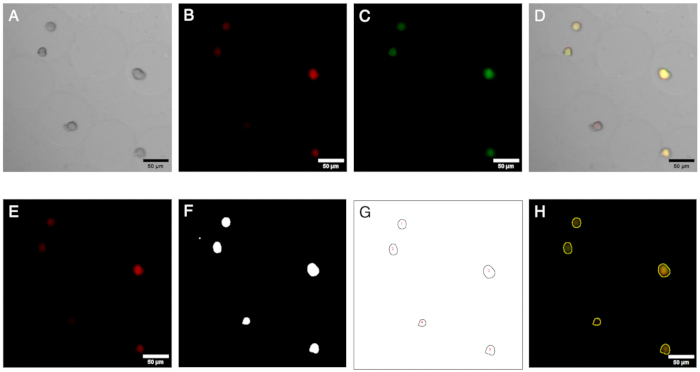
Abbildung 2: Mikroskopiebilder und deren Analyse. Fluoreszenzbildgebung und Analyse von Kolonien in Mikrogelen. (A–C) Bildkanäle, die mittels Hellfeld- und Fluoreszenzmikroskopie mit dem inversen Mikroskop aufgenommen wurden. Das Kompositbild (D) zeigt das Vorhandensein einer negativen Kolonie (nur rote Fluoreszenz) innerhalb der Mikrogele. (E–H) Ergebnisse des Bildanalyse-Workflows. Durch die Generierung von ROIs können Kolonien auf roten und grünen Kanälen identifiziert und die Signale quantifiziert werden, um das Vorhandensein negativer Kolonien zu definieren. Maßstabsstäbe: 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Bildanalyse
HINWEIS: Um die Fluoreszenzsignale der verkapselten Kolonien zu analysieren und seltene Ereignisse zu identifizieren, können Bilder aus der Hellfeld- und Fluoreszenzbildgebung mit Fiji/ImageJ verarbeitet werden (Abbildung 3). Diese Schritte können in ein Makroskript implementiert werden, und die Parameterwerte können je nach optischer Konfiguration variieren.- Öffnen Sie die Bilder aus dem grünen und roten Kanal. Definieren Sie einen rechteckigen Interessenbereich (ROI) beginnend bei den Koordinaten (500, 500) mit einer Breite und Höhe von jeweils 2000 Pixeln.
HINWEIS: Diese Parameter gelten für Bilder mit einer Größe von 3000 x 3000 Pixeln, und der ROI definiert den Bereich mit besserer Beleuchtung. - Schneiden Sie das Bild auf den definierten rechteckigen ROI zu. Subtrahieren Sie einen konstanten Wert von 10 von der Intensität jedes Pixels und wenden Sie einen Glättungsfilter auf das Bild an, um Hintergrundrauschen zu reduzieren und die interessierenden Objekte deutlicher hervorzuheben.
HINWEIS: Nur dieser Bereich wird für die weitere Analyse verwendet. - Duplizieren Sie das Bild aus dem roten Kanal und konvertieren Sie es in eine binäre Maske, um die seltenen Ereignisse im Zusammenhang mit dem Plasmid zu identifizieren. Stellen Sie die Schwellenwerte zwischen 10 und 255 ein. Pixel innerhalb dieses Bereichs werden als Vordergrund (Objekte von Interesse) betrachtet, während andere als Hintergrund behandelt werden.
- Führen Sie morphologische Operationen durch, um kleine Lücken zu schließen und Löcher in den interessierenden Objekten zu füllen. Wenden Sie den Wendepunktalgorithmus an, um überlappende Objekte innerhalb der Binärmaske zu trennen.
- Analysieren Sie die Partikel in der Binärmaske. Berücksichtigen Sie nur Partikel mit einer Größe von mehr als 30 Pixeln und einer Zirkularität zwischen 0,50 und 1,00. Die Ergebnisse werden zusammengefasst und der Ergebnistabelle hinzugefügt. Speichern Sie die von der Partikelanalyse erkannten ROIs zur weiteren Visualisierung.
- Zeigen Sie die gespeicherten ROIs auf den Bildern aus dem grünen und roten Kanal an. Messen Sie die Intensität oder andere Eigenschaften der ROIs in diesen Bildern. Erfassen Sie die Messungen in der Ergebnistabelle und speichern Sie die Ergebnisse in separaten CSV-Dateien für die weitere statistische Analyse.
- Öffnen Sie die Bilder aus dem grünen und roten Kanal. Definieren Sie einen rechteckigen Interessenbereich (ROI) beginnend bei den Koordinaten (500, 500) mit einer Breite und Höhe von jeweils 2000 Pixeln.
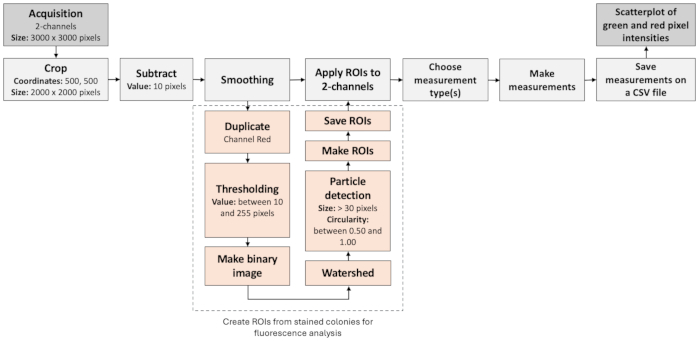
Abbildung 3: Arbeitsablauf bei der Bildanalyse zur Identifizierung negativer Kolonien. Die Abbildung zeigt einen Schritt-für-Schritt-Workflow zur automatischen Verarbeitung und Bewertung von Fluoreszenzbildern. Der Workflow basiert auf der Kolokalisierung von Fluoreszenzmarkierungen sowie der Partikelanalyse. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Agarplatten-Vergleichsassay
HINWEIS: Um die Tröpfchenmethode mit einem herkömmlichen Plattenassay zu vergleichen, wurde die Quantifizierung der fluoreszierenden Kolonien desselben E. coli-Stammes in Schritt 2.1.8 durchgeführt. wurde mit Petrischalen durchgeführt. Dies diente als analoge Kontrollmethode zur Messung der Stabilität von sfGFP-Plasmiden. Siehe auch die Methodenabbildung in Abbildung 1.
- Bereiten Sie LB-Agar (ohne Antibiotikum) vor, wie in Schritt 2.1.1 aus dem Abschnitt Zellernte beschrieben. Den Agar unter 60 °C abkühlen lassen und homogenisieren.
- Gießen Sie in einer sterilen Umgebung 15 ml des Agarmediums in jede Petrischale. Lassen Sie die Platten bei teilweise geöffneten Deckeln festigen, bis sie gebrauchsfertig sind.
- Beimpfen Sie drei Platten mit je 10 μl einer E. coli-Kultur in LB-Medien mit einem OD600 von 0,007 ± 0,002. Mit einem L-förmigen Streuer gleichmäßig verteilen. Die Platten bei 37 °C inkubieren, geschlossen und umgedreht. Bereiten Sie unter den gleichen Bedingungen eine Kontrollplatte ohne Bakterien vor.
- Nach 24 Stunden können Sie Fluoreszenzbilder der Platten mit dem FluoPi aufnehmen, einem Open-Source-Fluoreszenzbildgebungssystem (https://github.com/RudgeLab/FluoPi)15.
HINWEIS: Der FluoPi besteht aus einer Raspberry Pi Kamera mit blauem Anregungslicht bei einer Wellenlänge zentriert bei 470 nm und Anregungs- und Emissionsfiltern aus Acryl. - Zählen Sie manuell die fluoreszierenden und nicht fluoreszierenden Kolonien in jeder Platte anhand der aufgenommenen Bilder.
Ergebnisse
Validierung der Zellverkapselung und Mikrokoloniebildung
Die Zellverkapselung kann visuell bestätigt werden, indem eine Hellfeldmikroskopie an den Gel-Mikrotröpfchen durchgeführt wird, bevor die Emulsion gebrochen und die Mikrogele gewaschen werden. Ein repräsentatives Ergebnis der Emulsion in diesem Schritt ist in Abbildung 4 dargestellt.
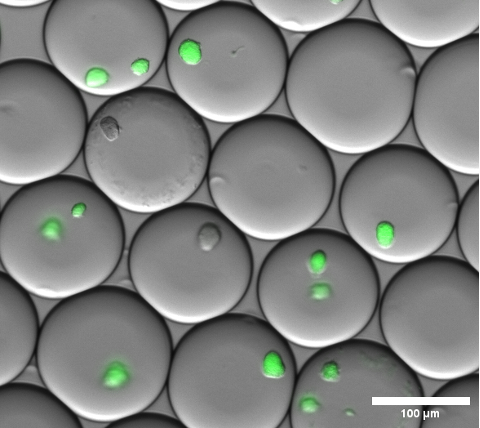
Abbildung 4: Ausschnitt aus einem Fluoreszenzmikroskopie-Overlay-Bild. Nach der Inkubation über Nacht wurden repräsentative Mikrokolonien von sfGFP-exprimierenden E. coli-Kolonien in Gel-Mikrotröpfchen gebildet. Zum Einsatz kam ein Mikroskopobjektiv mit 10-facher Vergrößerung und einem 0,30 NA. Maßstab: 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse der Bildanalyse
Sobald Mikrogele gefärbt und das Hellfeld sowie die Fluoreszenzkanäle an mehreren Positionen aufgenommen wurden, können die Kolonien, die in den Originalbildern als negativ identifiziert wurden, sichtbar gemacht werden (siehe Abbildung 5A). Daten, die aus allen Bildern eines Experiments extrahiert wurden, können grafisch dargestellt werden, um das Fluoreszenzverhältnis verschiedener Kolonien zu zeigen, wobei diejenigen hervorgehoben werden, die die Plasmid-kodierte Fluoreszenz verloren haben (siehe Abbildung 5B). Die Ergebnisse zeigen, dass 100 Kolonien von insgesamt 2785 analysierten Mikrokolonien ihr Plasmid oder ihre Plasmidfunktionalität verloren haben, was 3,6% entspricht.
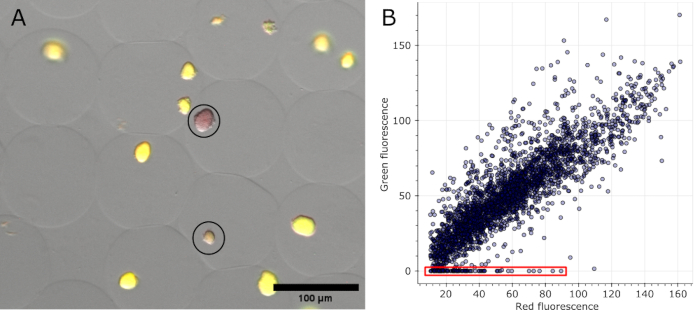
Abbildung 5: Quantifizierung negativer Mikrokolonien. (A) Ausschnitt aus einem Fluoreszenzmikroskopie-Overlay-Bild. Nach der Ölentfernung und -färbung zeigten repräsentative Kolonien in Mikrogelen, die sfGFP exprimieren, und zwei negative Kolonien die rote Fluoreszenz der DNA-Färbung (schwarz eingekreist). Maßstabsbalken: 100 μm. (B) Streudiagramm der Fluoreszenzwerte einzelner Mikrokolonien, extrahiert aus 16 Mehrkanal-Mikroskopiebildern. Kolonien ohne grüne Fluoreszenz wurden als negativ gezählt, wie in der Grafik rot dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Quantifizierung von Agarplatten
Bilder der dreifachen Platten sind in Abbildung 6 dargestellt, wobei die nicht fluoreszierenden Kolonien durch weiße Pfeile gekennzeichnet sind. Die erste Platte (Abbildung 6A) zeigte insgesamt 213 Kolonien, von denen 1 nicht fluoreszierend war. Die zweite Platte (Abbildung 6B) hatte insgesamt 49 Kolonien, ohne nicht-fluoreszierende Kolonien. Die dritte Platte (Abbildung 6C) zeigte insgesamt 252 Kolonien, von denen 6 nicht fluoreszierend waren. Diese Ergebnisse entsprechen einer mittleren Kolonieplasmidverlustrate von 2,3 % mit einer großen Standardabweichung von 3,2.
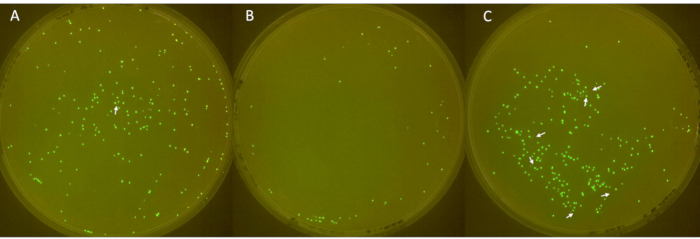
Abbildung 6: Identifizierung negativer Kolonien auf Platten. (A–C) Fluoreszierende und nicht-fluoreszierende E. coli-Kolonien auf LB-Agarplatten (Durchmesser: 90 mm, Höhe: 15 mm). Das Inokulum, das aus E. coli mit sfGFP aus einem Stamm von -80 °C gewonnen wurde, wurde an Tag 1 gestreift, an Tag 2 mit einem Antibiotikum kultiviert und von Tag 3 bis 6 täglich 1:100 verdünnt, um einen Plasmidverlust zu ermöglichen. Die Kolonien wurden 24 Stunden lang bei 37 °C inkubiert und in einer FluoPi-Kammer abgebildet. Nicht fluoreszierende Kolonien wurden mit GIMP verbessert und mit weißen Pfeilen gekennzeichnet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Es wird demonstriert, dass eine auf Gel-Mikrotröpfchen basierende Methode zur effektiven Identifizierung und Quantifizierung von Kolonien mit und ohne Plasmid-kodierte genetische Expression von fluoreszierenden Proteinen, wie z. B. sfGFP, durchgeführt werden kann. Kolonien, die das Plasmidprodukt nicht ausreichend exprimieren, werden mit Hilfe einer fluoreszierenden DNA-Färbung (hier Propidiumiodid) identifiziert, die alle Kolonien anfärbt und eine andere Emissionswellenlänge aufweist. Diese Integration von Tröpfchenmikrofluidik, Gelierung und Fluoreszenzmikroskopie unter Verwendung von Open-Source-Technologie ermöglicht die Ausführung eines fortschrittlichen Arbeitsablaufs in vielen Forschungsumgebungen11,13. Die erfolgreiche Erzeugung von Gel-Mikrotröpfchen ermöglicht fortschrittliche molekularbiologische Einzelzell-Arbeitsabläufe, einschließlich Zelllyse, Einzelgenomamplifikation, metabolische Zellinteraktionsscreens, Medienaustausch und mehr 8,9. Diese Vorteile werden in diesem Protokoll genutzt, um Mikrokolonien skalierbarer zu züchten, zu färben und zu analysieren als bei herkömmlichen plattenbasierten Assays.
Kritische Schritte
Der Verkapselungsprozess ist ein kritischer und heikler Teil des Protokolls. Eine präzise Steuerung der Inhaltsstoffkonzentrationen, Durchflussraten und Drücke ist erforderlich, um gleichmäßige Mikrogele innerhalb eines bestimmten Größenbereichs zu erzeugen und die durchschnittliche Anzahl der Zellen pro Tröpfchen zu steuern. Darüber hinaus verhindert die Aufrechterhaltung der Konzentration und Temperatur des Zell-Agarose-Gemisches ein Verklumpen oder eine vorzeitige Gelierung. Die Temperaturregelung der flüssigen Agarose-Zellsuspension in einer Pipettenspitze ist eine besonders vorteilhafte Implementierung unserer Open-Source-Hardware-Mikrofluidik-Workstation, die eine viel einfachere und robustere Mikrogelerzeugung im Vergleich zu den Bemühungen zur Temperaturkontrolle von Spritzenpumpen und Schläuchen ermöglicht. Da die Zellen vor der Verkapselung und Kultivierung mit dem Agarose-Wachstumsmedium vermischt werden, müssen die Agarose-Mikrogele schnell erzeugt werden, um größere Veränderungen der Zellkonzentration zu vermeiden. Zu diesem Zweck wurde ein von Abate et al. inspiriertes tröpfchenspaltendes mikrofluidisches Chipdesign optimiert18.
Modifikationen und Fehlerbehebung
Mehrere Kalibrierungen und Modifikationen waren notwendig, um das ursprüngliche Protokoll zu verfeinern. Die Verkapselung von Agarose ist viel schwieriger als bei herkömmlichen Wasser-in-Öl-Tröpfchen und erfordert die Konstruktion eines Systems, um die Agarose in einem flüssigen Zustand zu halten und gleichzeitig sicherzustellen, dass die wässrige Phasenströmung einen homogenen Partikelgrößenbereich erreicht. Änderungen der Agaroseviskosität aufgrund der Gelierung beeinflussen die Durchflussrate, was zu größeren Partikelgrößen führt. Die Mikroskopie erfordert eine sorgfältige Auswahl von Filtern und Lichtquellen, um sicherzustellen, dass sich die Anregungs- und Emissionssignale nicht überlappen und eine klare Unterscheidung ermöglichen. Ursprünglich wurde DAPI für die Färbung von Bakterien gewählt, aber sein Emissionssignal überlappte sich mit sfGFP, was dazu führte, dass sfGFP im blauen Detektionskanal nachgewiesen wurde. Wir sind auf PI umgestiegen, weil seine Emission bei langen Wellenlängen (rotes Licht) gut von sfGFP getrennt ist.
Während der Plasmidverlust mit der vorgeschlagenen Methode quantifiziert werden konnte, war das verwendete sfGFP-Plasmid unerwartet stabil und zeigte in der ersten Generation von Zellen, die ohne Antibiotika kultiviert wurden, selbst unter Stressbedingungen wie pH9-Medien und Inkubation bei 40 °C kaum Fälle von Plasmidverlust. Diese Beobachtung stimmt mit den Ergebnissen anderer Forschungsgruppenüberein 1,19. Die Plasmidstabilität schränkte die Demonstration der vollen Quantifizierungsmöglichkeiten der Methode für erste Zellkulturgenerationen ein, zeigte jedoch, dass die Methode empfindlich genug ist, um selbst kleine Unterschiede in der Plasmidretention zu erkennen. Die Beobachtung einer hohen Plasmidstabilität in frühen Generationen hat eine wichtige Bedeutung für Tröpfchen-Mikrofluidik-Screenings mit einem negativen Selektionsassay, wie z. B. die Hemmung von Zielbakterien. Dies bedeutet, dass der Plasmidverlust von Selektionszielen eine geringe Quelle für falsch-positive Selektionsergebnisse darstellt. Da Tröpfchen-Mikrofluidik-Bildschirme in der Regel andere Hochdurchsatz-Siebe, wie z. B. die Arbeitsabläufe von Pipettierrobotern, um Größenordnungen im Durchsatz übertreffen, müssen diese seltenen Ereignisse bewertet und berücksichtigt werden.
Begrenzungen
Trotz ihrer Vorteile gibt es Einschränkungen bei der vorgestellten Methode. Die Herstellung mikrofluidischer Geräte erfordert Fachwissen und akribische Liebe zum Detail sowie eine strenge experimentelle Kontrolle der Durchflussraten, um die Effizienz der deterministischen Verkapselung zu gewährleisten. Diese Aspekte können eine Optimierung für verschiedene Versuchsaufbauten erfordern. Während diese Methode auf der Fluoreszenzmikroskopie zur Signaldetektion beruht und den Zugang zu geeigneten Bildgebungsgeräten erfordert, kann dieses Gerät mit Open-Source-Hardware hergestellt werden, was es zugänglicher macht. Darüber hinaus können Mikrogele in der kommerziellen Durchflusszytometrie mit großen Düsen verarbeitet werden, was die Zugänglichkeit und den experimentellen Durchsatz weiter verbessert. Auch Tröpfchensortierer können für diese zytometrische Analyse eingesetzt werden.
Darüber hinaus ist die Methode zwar darauf ausgelegt, Fluoreszenzsignale von Plasmiden, Färbungen oder anderen Markern zu detektieren, aber auf Zellen beschränkt, die fluoreszenzmarkiert werden können, was möglicherweise nicht auf alle Bakterienstämme oder experimentellen Bedingungen zutrifft. Die Methode kann jedoch angepasst werden, um andere Arten von Mikroskopie, wie z. B. Phasenkontrast- oder Hellfeldmikroskopie, einzubeziehen, was Phänotypisierungsanwendungen über die Fluoreszenz hinaus ermöglicht. Darüber hinaus kann es mit spektroskopischen Techniken wie FTIR- oder Raman-Spektroskopie kombiniert werden, wodurch seine Möglichkeiten zur Analyse der chemischen Zusammensetzung und strukturellen Informationen der verkapselten Zellen erweitert werden. Diese Anpassungen erweitern das Anwendungsspektrum und machen es zu einem vielseitigen Werkzeug für verschiedene Forschungsumgebungen.
Bedeutung und Anwendungen
Herkömmliche Assays für den Plasmidverlust19 erlauben keine gute Quantifizierung des Verhältnisses von Zellen, die ihre Expression verloren haben, eine Information, die bei der Gestaltung experimenteller Methoden und verschiedener biologischer Anwendungen sehr wichtig sein kann. In der Regel werden Kolonietypen in Agarplatten-Assays gezählt, bei denen gut definierte isolierte Kolonien erhalten werden können, wie in Abbildung 4 gezeigt. Es ist jedoch schwierig, überlappende Kolonien mit Sicherheit zu identifizieren. In unseren Händen erhalten wir nicht immer eine optimale Koloniedichte, und viele Platten sind notwendig, um eine gute Statistik von niederfrequenten Plasmidverlustereignissen zu erhalten. Die vorgeschlagene Methode bietet einen robusteren Ansatz zur genauen Quantifizierung von Fluoreszenzsignalen, die von isolierten Kolonien mit einer höheren Anzahl von Kolonien stammen, als die analogen Agarplattenmethoden, da sich die Kolonien in Mikrotröpfchen getrennt entwickeln, kleiner sind und leicht in Bildgebungskammern geladen werden können, was eine mikroskopische oder durchflusszytometrische Quantifizierung großer Kolonienzahlen ermöglicht. Dies kann die statistische Darstellung der Methode deutlich verbessern und die Integration in andere Gel-Mikrotröpfchen-Arbeitsabläufe ermöglichen.
Die Verwendung von Open-Source-Hardware11,20 ermöglicht es den Forschern, das mikrofluidische Workstation-Design anzupassen und die Durchflussrate präzise einzustellen; Daher unterstützt die Partikelgröße verschiedene Zelltypen und experimentelle Bedingungen. Diese Flexibilität erstreckt sich auch auf die mögliche Einbeziehung anderer Mikroskopietypen, wie z. B. Phasenkontrast oder Spektroskopie, wodurch die Anwendbarkeit der Methode erweitert wird. Die Fähigkeit der Methode, die Plasmidstabilität unter verschiedenen Bedingungen zu bewerten, ist entscheidend für Anwendungen, die eine Plasmidretention ohne Antibiotikaauswahl, unter bestimmten Stressbedingungen oder in verschiedenen Kulturgenerationen erfordern. Die Vielseitigkeit und Anpassungsfähigkeit der vorgestellten Methode machen sie wertvoll für vielfältige Forschungsanwendungen in Bereichen wie synthetische Biologie, Umweltüberwachung und klinische Diagnostik2.
Offenlegungen
Die Autoren erklären, dass keine konkurrierenden finanziellen Interessen oder persönlichen Beziehungen die in dieser Arbeit berichtete Arbeit beeinflusst haben könnten.
Danksagungen
Diese Arbeit ist Teil von Projekten, die T.W. von ANID FONDECYT Regular 1241621 und dem Projekt der Chang Zuckerberg Initiative "Latin American Hub for Bioimaging Through Open Hardware" erhalten haben. T.W. ist auch dankbar für die Finanzierung durch CIFAR, als Azrieli Global Scholar im CIFAR MacMillan Multiscale Human Programm.
Materialien
| Name | Company | Catalog Number | Comments |
| 1H,1H,2H,2H-Perfluoro-1-octanol | Sigma-Aldrich | 370533-25G | For breaking emulsion |
| 70% ethanol | For cell permeabilization | ||
| Agar-Agar | Winkler | 9002-18-0 | |
| Biopsy Punch | 0.75 mm and 1.8 mm | ||
| Blue LED transilluminator | IO Rodeo | ||
| Culture tube | 15 mL | ||
| Desiccator | With vacuum pump | ||
| Disposable cup | For mixing PDMS | ||
| Disposable fork | For mixing PDMS | ||
| E. coli TOP10 strain | |||
| FluoPi microscope | https://github.com/wenzel-lab/FluoPi | Green fluorescence imaging system for analyzing plates | |
| Fluorinated Oil | 3M | Novec 7500 | |
| Glass slide heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of microfluidic chip | |
| Glass Slides | |||
| Hotplate | Mechanic | For evaporating Aquapel | |
| Image analysis software | Fiji/ImageJ | 2.14.0/1.54f | |
| Incubator | Mundo Lab | MLAB Scientific / For incubation of plates and microgels | |
| Isopropanol | For cleaning glass slides | ||
| Kanamycin | 100 ug/mL concentration | ||
| L-shaped spreader | For spreading bacteria on agar plates | ||
| Master mold | Chip design on silicone or glass wafer | ||
| Microtubes | 2 mL | ||
| NaCl solution | Sodium chloride 0.9% w/v | ||
| Open-source hardware strobe-enhanced microscopy stage | https://github.com/wenzel-lab/flow-microscopy-platform | For bright-field microscopy | |
| Petri dish | Citotest | 2303-1090 | 90 x 15 mm |
| Pipette tip heater | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller/tree/master/module-heating-and-stirring | For controlling the temperature at 40 °C of pipette tip | |
| Plasma Cleaner | Diener Electronic | 117056 | For bonding PDMS with a glass slide |
| Plasmid pCA_Odd1 | Encodes sfGFP and kanamycin resistance | ||
| Polytetrafluoroethylene (PTFE) tubing | Adtech Polymer Engineering Ltd | ||
| Pre-mixed Luria Bertoni medium | US Biological Life Science | L1520 | |
| Propidium iodide (PI) | For staining | ||
| Raspberry Pi-based pressure and flow controller system | https://github.com/wenzel-lab/modular-microfluidics-workstation-controller | For controlling pressure and flow rates | |
| Silicone elastomer base | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Silicone elastomer curing agent | Sylgard | PDMS kit - 184 Silicone Elastomer Kit | |
| Spectrophotometer | For measuring absorbance | ||
| SQUID microscope | https://github.com/wenzel-lab/SQUID-bioimaging-platform | Multi-fluorescence imaging system for analyzing stained cells | |
| Sterile loop | For picking a colony and streaking plating | ||
| Surfactant | Sphere Fluidics | Pico-Surf | |
| Syringes | NIPRO | With filters and tubing | |
| Temperature-controlled shaker | Mundo Lab | DLAB HCM100-Pro | |
| Tweezer | |||
| Ultra-low gelling temperature agarose | Sigma-Aldrich | A2576-5G | For generating hydrogel beads |
| Water repelent solution (fluoroalkyl silane) | Aquapel | For treating microchannels of PDMS device |
Referenzen
- Wein, T., Hülter, N. F., Mizrahi, I., Dagan, T. Emergence of plasmid stability under non-selective conditions maintains antibiotic resistance. Nat Commun. 10 (1), 2595 (2019).
- Rodríguez-Beltrán, J., DelaFuente, J., León-Sampedro, R., MacLean, R. C., Millán, &. #. 1. 9. 3. ;. S. Beyond horizontal gene transfer: The role of plasmids in bacterial evolution. Nat Rev Microbiol. 19 (6), 347 (2021).
- Wein, T., Dagan, T. Plasmid evolution. Curr Biol. 30 (19), R1158-R1163 (2020).
- Chen, S., Larsson, M., Robinson, R. C., Chen, S. L. Direct and convenient measurement of plasmid stability in lab and clinical isolates of E. coli. Sci Rep. 7 (1), 4788 (2017).
- Rouches, M. V., Xu, Y., Cortes, L. B. G., Lambert, G. A plasmid system with tunable copy number. Nat Commun. 13 (1), 3908 (2022).
- Silva, F., Queiroz, J. A., Domingues, F. C. Evaluating metabolic stress and plasmid stability in plasmid DNA production by Escherichia coli. Biotechnol Adv. 30 (3), 691-708 (2012).
- Wang, R., et al. Construction of novel pJRD215-derived plasmids using chloramphenicol acetyltransferase (cat) gene as a selection marker for Acidithiobacillus caldus. PLoS ONE. 12 (8), e0183307 (2017).
- Moragues, T., et al. Droplet-based microfluidics. Nat Rev Methods Primers. 3 (1), 32 (2023).
- Vitalis, C., Wenzel, T. Leveraging interactions in microfluidic droplets for enhanced biotechnology screens. Current Opinion in Biotechnology. 82, 102966 (2023).
- Sinha, N., Subedi, N., Wimmers, F., Soennichsen, M., Tel, J. A pipette-tip based method for seeding cells to droplet microfluidic platforms. J Vis Exp. (144), e57848 (2019).
- Wenzel, T. Open hardware: From DIY trend to global transformation in access to laboratory equipment. PLOS Biol. 21 (1), e3001931 (2023).
- Murillo, L. F. R., Wenzel, T. Welcome to the journal of open hardware. J Open Hardware. 1 (1), (2017).
- Shin, J. H., Choi, S. Open-source and do-it-yourself microfluidics. Sens Actuators B Chem. 347, 130624 (2021).
- Pryszlak, A., et al. Enrichment of gut microbiome strains for cultivation-free genome sequencing using droplet microfluidics. Cell Rep Methods. 2 (1), 100137 (2021).
- Pollak, B., et al. Universal loop assembly: open, efficient and cross-kingdom DNA fabrication. Biology. 5 (1), (2020).
- Volkmer, B., Heinemann, M. Condition-dependent cell volume and concentration of Escherichia coli to facilitate data conversion for systems biology modeling. PLoS ONE. 6 (7), e23126 (2011).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , (2020).
- Abate, A. R., Weitz, D. A. Faster multiple emulsification with drop splitting. Lab Chip. 11 (11), 1911-1915 (2011).
- Lau, B. T. C., Malkus, P., Paulsson, J. New quantitative methods for measuring plasmid loss rates reveal unexpected stability. Plasmid. 70 (3), 353-361 (2013).
- Oellermann, M., et al. Open hardware in science: The benefits of open electronics. Integr Comp Biol. 62 (4), 1061-1075 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten