このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
免疫蛍光法による哺乳類細胞の核ブレブとDNA漏出の検出
要約
ラミノパチー障害は、しばしば核膜の変化を引き起こし、核のブレブや漏出を引き起こす可能性があります。この研究は、二本鎖DNA(dsDNA)とともに核ラミナを視覚化する免疫蛍光法を提示し、哺乳類細胞の核構造と完全性を評価する手段を提供します。
要約
核ラミナは、核膜の下にあるフィラメントのネットワークであり、ラミンとラミン関連タンパク質で構成されています。核構造、核孔の位置決め、遺伝子発現調節、クロマチン組織化、DNA複製、DNA修復において重要な役割を果たします。ラミンタンパク質の発現または翻訳後プロセシングに関与する遺伝子の突然変異は、ラミンパチーとして知られる遺伝性疾患を引き起こします。具体的には、 LMNA 遺伝子または ZMPSTE24 遺伝子の突然変異は、完全に処理されたラミンAには存在しないファルネシル基とメチル基を保持する不完全に処理されたラミンAの蓄積につながる可能性があります。これらの不完全に処理されたラミンAタンパク質は、成熟ラミンAが存在する核ラミナではなく、内核膜に局在します。誤って局在化したラミンタンパク質は、核の機能と構造を深く破壊し、しばしば核のブレブを引き起こします。重症の場合、核の破裂が起こり、区画化が失われ、ゲノムDNAが細胞質に漏れる可能性があります。異常な核構造と区画化の喪失は、固定細胞上の間接免疫蛍光法(IF)によって特定できます。本研究では、ラミンタンパク質と二本鎖DNA(dsDNA)に対する特異的抗体を用いて、核膜とDNAを同時に可視化する手法について概説します。このアプローチにより、核の構造的完全性と、細胞質への核DNAの潜在的な漏出を迅速に評価することができます。
概要
核ラミナは、核膜の下にあるフィラメントのネットワークであり、ラミンと呼ばれるタンパク質で構成されています。核ラミナは、核の構造、核孔の位置決め、遺伝子発現制御、クロマチン組織化、DNA複製、およびDNA修復において重要な役割を果たします1,2,3。ラミンタンパク質の発現に関与する遺伝子の突然変異は、ラミノパチー3と呼ばれる遺伝性疾患を引き起こします。
拘束性皮膚症(RD)は、主に ZMPSTE24 遺伝子4に早期終結コドンを生じさせる複合ヘテロ接合体変異によって引き起こされ、メタロプロテアーゼZMPSTE24の欠如につながる重度のラミノパチー障害です。このラミノパチーは、子宮内発育遅延、皮膚の引き締まり、硬直、口が小さい、髪の毛が薄い、骨の石灰化障害1を特徴としています。RD患者は通常、肺機能不全のため、生後1週間を過ぎて生きられません1。非定型ハッチンソン・ギルフォード早老症症候群(AT-HGPS)および下顎骨異形成B型(MAD-B)として知られる他の2つのラミノパシー障害は、ZMPSTE24発現の低下を伴い、寿命の短縮と関連しており、早期老化障害との類似性を示しています5。これらのラミノパチーで影響を受けるZMPSTE24プロテアーゼは、核ラミナの重要な成分であるラミンAの翻訳後修飾に不可欠です。ZMPSTE24欠乏症は、プレラミンA2として知られる不完全に処理されたファルネシル化形態のラミンAの蓄積をもたらします。
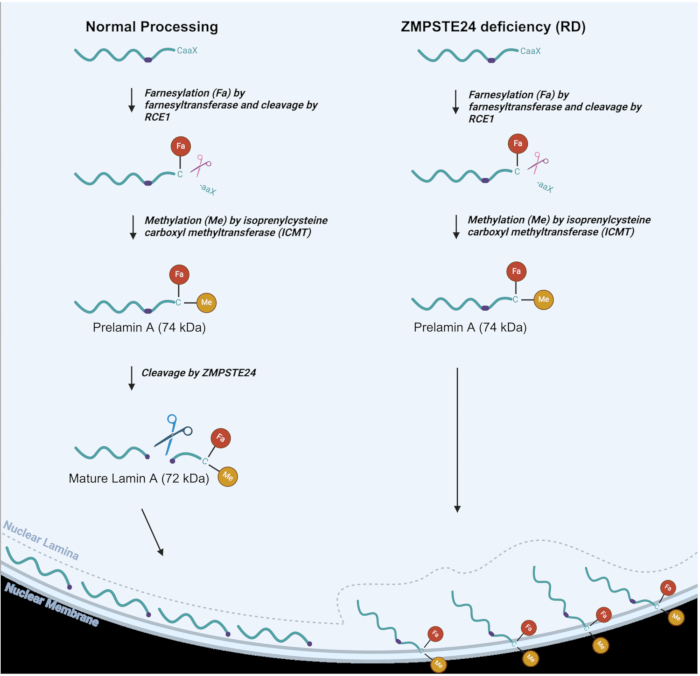
図1:通常のラミンA処理とラミンA処理 RD細胞。正常細胞におけるラミンAプロセッシング経路(左)とRDまたはZMPSTE欠損細胞におけるラミンAプロセッシングの変化(右)。ファルネシル基(Fa)基とメチル基(Me基)が示されています。この図の拡大版を表示するには、ここをクリックしてください。
図1に見られるように、通常のラミンAプロセシングでは、C末端付近のシステイン残基にファルネシル(脂質)基が結合し、続いて3つのC末端アミノ酸3,6がタンパク質分解的に切断されます。その後、システイン残基はメチル化されます。これら2つの修飾により、プレラミンAを内核膜2に標的とすることができる。次に、ZMPSTE24、最後の15個のC末端アミノ酸がファルネシル基およびメチル基とともに除去されて成熟ラミンAタンパク質が生成され、これが核ラミナ6,7,8に送達される切断反応を行います。RD、AT-HGPS、およびMAD-Bでは、ZMPSTE24メタロプロテイナーゼが不足しているか、完全に機能していないため、処理の最終ステップが効果的に行われません8,9。これにより、プレラミンAが蓄積し、プレラミンAは永久にファルネシル化およびメチル化されたままになります9。これらの後者のグループにより、プレラミンAは、図1に示すように、成熟したラミンAが最終的に存在するはずの核ラミナに局在するのではなく、内側の核膜に付着します。したがって、ZMPSTE欠損症は、核構造10,11,12,13,14,15とともに、さまざまな核機能に深刻な影響を及ぼします。これらの核構造の変化には、核のブレブや核の破裂さえ含まれ、その結果、DNAが細胞質に漏れたり、細胞質が核に侵入したりする可能性があります。異常形状の核は、プレラミンAプロセッシング16,17の欠陥によって引き起こされる他の多くの表現型とともに、確かにラミノパシー障害の特徴です。核ラミナの欠損は、核タンパク質の品質管理や核質中のDNA修復タンパク質の誤局在化など、多くの有害な影響をもたらし、その結果、核機能に多数の欠陥が生じる18。ラミノパチーのこれらの特徴を特定および監視するのに役立つ簡単な技術を持つことが重要です ラミノパチーの表現型を改善することを目的とした治療アプローチの開発に関する研究を促進します。ラミノパチーのマウスでは、核ブレブの消失がラミノパチーの表現型の一般化された消失と相関することが示されています16。ヒト細胞の核完全性のモニタリングを可能にする技術は、疾患の表現型を排除したり、大幅に改善したりするための潜在的な治療法の研究を強化する可能性があります。
間接免疫蛍光法(IF)は、一次非標識抗体と、一次抗体を認識する蛍光色素標識二次抗体の両方を使用して目的の標的を検出する高感度で広く使用されている技術です19。IF法は、特定の細胞内成分や構造を可視化するための強力な手段を提供します。また、複数の二次抗体分子が一次抗体と相互作用し、シグナル19の増幅をもたらすことも可能である。間接IFは、比較的小さな二次抗体のセットで複数の一次抗体を検出できる汎用性の高い手法であり、二次抗体は一次抗体のFcドメインに対して産生され、これは分子種19内で保存されています。
この記事では、二本鎖DNA(dsDNA)に対する抗体(dsDNA)とラミンB1を使用してDNAと核ラミナをそれぞれ検出し、ZMPSTE24欠損細胞の核ブレブとDNA漏出を評価する間接的(IF)方法について説明します。このアプローチの有用性を実証するために、この手順をノックアウトされたZMPSTE24発現を持つHeLa細胞株と、ZMPSTE24を発現するHeLa細胞株に適用し、2つの細胞株の結果を比較しました。
プロトコル
試薬および使用した機器の詳細は、材料表に記載されています。
1. 資料の準備
- 少なくとも10個の22 mm x 22 mm角のガラスカバースリップと鉗子をオートクレーブして、セルをカバーガラスにメッキします。
2.溶液の調製
- 補充培地:500 mLのダルベッコ改変イーグル培地(DMEM)を10%ウシ胎児血清(FBS)、100ユニット/ mLのペニシリン、および100 μg / mLストレプトマイシンと共に調製します。.
- 抗体希釈バッファー(ADB):2 gのウシ血清アルブミン(BSA)と1 gのフィッシュゼラチンを80 mLの1xリン酸緩衝生理食塩水(PBS)に溶解します。BSAと魚ゼラチンが溶解したら、PBSで100mLにしてください。
- 1x PBS-Tween(PBS-T):500mLのPBSと0.5mLのTweenを準備します。Tweenが溶液に簡単に混ざるまで、37°Cのウォーターバスに入れます。
3. 目的の細胞を成長させる
注:HeLa細胞は、このプロトコルでT75フラスコ内でコンフルエントになるように増殖しました。
- 6枚のカバースリップに70%エタノールをスプレーし、糸くずの出ないワイプで風乾させます。鉗子を使用して、カバースリップを滅菌済みの6ウェルプレートに、ウェルごとに1枚のカバースリップを入れます。
- 2 mLの添加DMEMを6ウェルプレートの各ウェルに加えます。
- 細胞から培地を吸引し、15 mLのPBSをフラスコに加えて細胞を洗浄し、穏やかに渦巻かせます。PBSを吸引し、2 mLのトリプシン-EDTA(トリプシン)を追加します。.
- 細胞をトリプシンでコーティングし、フラスコを37°Cおよび5%CO2 加湿インキュベーターに2分間戻して、細胞がフラスコから分離できるようにします。
- 補充したDMEMを8mLフラスコに加え、トリプシン処理した細胞を採取します。ピペットで数回上下に動かして、凝集細胞を分離します。細胞懸濁液をフラスコに集め、15mLのポリスチレンチューブに入れます。
- 血球計算盤を使用して細胞をカウントします。
- 500,000個の細胞を1滴ずつ、6ウェルプレートの各カバースリップに直接加えます。加湿インキュベーターで細胞を37°C、5%CO2 で一晩増殖させます。
4. 細胞の固定と透過化
- 使用前に4%ホルムアルデヒド溶液を新鮮に調製する:2mLの37.5%ホルムアルデヒド溶液を18mLのPBSに加えます。よく混ぜます。
- 使用前に透過処理溶液を新鮮に調製する:100 μLのTriton-Xを20 mLのPBSに加えます。Triton-Xが溶液に溶解するまで、37°Cのウォーターバスで数分間インキュベートします。よく混ぜます。
- プレートのウェルでメディアを吸引します。細胞を2mLのPBSで1回洗浄します。井戸からすべてのPBSを吸引します。
- 各ウェルに2mLの4%ホルムアルデヒド溶液を加えます。室温(RT)で10分間放置します。4%ホルムアルデヒドをウェルから完全に吸引します。
- 細胞を2mLのPBSで3回洗浄します。各洗浄後にPBSをウェルから完全に取り除きます。
- 各ウェルに2 mLの透過化溶液を加えます。RTで10分間座らせます。
- 細胞を2mLのPBSで3回洗浄します。最初の2回の洗浄後にPBSを完全に吸引し、3回目の洗浄後にPBSをウェルに残します。
注:細胞は、このステップの後、4°Cでの最後のPBS洗浄で数週間保存することができ、プレートがパラフィンフィルムで密封されて液体の蒸発を防ぎます。手順を続行できる場合は、この手順をスキップできます。
5. 免疫蛍光染色
- ウェルからすべてのPBSを取り外します。各ウェルに2 mLのADBを添加して細胞をブロックし、室温で30分間振盪しながらインキュベートします。
- 30分間のブロッキング時間の終わりに向かって、ADBで一次抗体希釈液を混合します(Lamin B1抗体とdsDNA抗体の両方で1:1,000希釈、 表1)。各カバーガラスには75 μLが必要であるため、600 μLのADB、0.6 μLのLamin B一次抗体、および0.6 μLのdsDNA抗体を6枚のカバーガラスに混合する必要があります。徹底的に渦を巻きます。
- 井戸からADBを取り除きます。平らな面に、処理中のすべてのカバーガラスを敷くのに十分な大きさのパラフィンフィルムをテープで留めます。
- 各カバースリップについて、希釈した一次抗体75 μLをパラフィンフィルムに加え、ADB内で気泡がピペットで移動しないようにします。次に、鉗子を使用して、カバースリップセルを下にしてADBの液滴に置きます。
- インキュベーションカバースリップをウェルプレートの蓋で覆い、室温で1時間インキュベートします。
- 鉗子を使用してパラフィンフィルムからカバースリップをはがし、セル側を上に向けてカバースリップを6ウェルプレートに戻します。
- カバーガラスを2mLのPBS-Tで3回洗い、毎回3〜5分間揺さぶります。各洗浄後に、すべてのPBS-Tをウェルから取り出します。
- 最後の洗浄では、二次抗体の希釈液を調製します(それぞれの二次抗体ごとに1:1,000、 表1)。各カバーガラスには75 μLが必要であるため、600 μLのADBと0.6 μLのAlexa Fluorタグ付き二次抗体を6枚のカバーガラスに混合する必要があります。徹底的に渦を巻きます。二次抗体の希釈液は、できるだけ光で覆ってください。
| 抗体 | 源 | 会社 | 参考 | ||
| dsDNA Marker 抗体 (HYB331-01) | マウスモノクローナル | サンタクルス | SC-58749 | ||
| ヤギ抗マウスIgG(H+L)高交差吸着二次抗体、Alexa Fluor Plus 488 | 山羊 | サーモフィッシャー | A32723 (英語) | ||
| ヤギ抗マウスIgG(H+L)高交差吸着二次抗体、Alexa Fluor Plus 405 | 山羊 | インビトゲン | A31553 (英語) | ||
| Lamin B1 ポリクローナル抗体 | 兎 | プロテインテック | 12987-1-AP | ||
| ヤギ抗ウサギIgG(H+L)高交差吸着二次抗体、Alexa Fluor 594 | 山羊 | インビトゲン | A11037 (英語) | ||
表1:抗体。 このプロトコルで使用されるすべての抗体のリスト。
- 最後の洗浄からすべてのPBS-Tを取り外してください。ステップ5と同様に、パラフィンフィルム上の二次抗体希釈液でカバースリップをRTで30分間インキュベートします。小さな箱で光を遮蔽できます。必要に応じて、ウェルに溶液を追加したり、ウェルから溶液を削除したりするために、迅速に作業するようにしてください。
- 鉗子を使用してパラフィンフィルムからカバースリップをはがし、セル側を上にしてカバースリップを6ウェルプレートに戻します。
- カバースリップを2mLのPBS-Tで3回洗います。各洗浄後に、すべてのPBS-Tをウェルから取り出します。
- スライドをエタノールシリーズ(70%、90%、100%)で脱水します。まず、2mLの70%エタノールを各ウェルに加えます。1〜2分間放置し、ウェルからすべてのエタノールを取り除きます。残りのエタノールの割合で繰り返します。
- 鉗子を使用してウェルからカバースリップを取り外し、糸くずの出ない拭き取りで、光から保護して風乾させます。
- カバースリップのセル面を下にして、カバースリップあたり20 μLの封入剤を使用して、ガラス顕微鏡スライドにマウントします。光から保護して、一晩乾燥させます。
6. 画像取得
- イメージングする前に、カバースリップの端を透明なマニキュアの薄い層でコーティングします(どのブランドやタイプでも十分です)。光から保護して、風乾させます。
注意: 顕微鏡を使用して画像化する方法は、使用する顕微鏡によって異なります。ここで使用する顕微鏡のカメラの解像度は1280 x 960ピクセルです。同等またはそれ以上の解像度で十分です。 - 顕微鏡ステージにスライドを載せる前に、ファイル保存の設定を調整する必要があります。顕微鏡の電源を入れ、USBドライブを挿入します。顕微鏡の設定内で保存設定を調整し、すべてのチャンネル(GFPとRFP)が別々に保存され、画像がUSBドライブの正しい場所に送られることを確認します。
注意: USBドライブにフォルダを作成し、後で参照できるように各カバースリップに独自の名前を付けます。 - イメージングの前に、糸くずの出ないワイプに70%エタノールをスプレーし、スライドの裏側をやさしく拭き取ります。スライドの前面を優しく清掃し、圧力がかかるのを最小限に抑えます。
- きれいなスライドを顕微鏡に置き、カバースリップを下に向けて置きます。遮光ボックスで覆います。
- 40倍対物レンズを使用して、適切なチャンネル(ここで使用するラミンB1抗体のRFP)を使用して、顕微鏡画面上の核を特定します。光の強度を約50%に調整して開始します。
- 飽和状態の領域をチェックするには、[カラーオフ]オプションを選択し、白黒の設定でカバーガラス全体の領域に赤信号がないことを確認します。赤信号が多い場合は、最小限の赤信号が表示されるまで光の強度を下げます。
注意: 赤い信号は飽和度が高いことを示します。核がほとんど見えない場合は、過飽和にならずに適切な強度が得られるまで、強度を10%ずつ増やします。 - 他のすべてのカバースリップを同じ強度でチェックして、飽和していないことを確認します。上記と同じプロセスを使用して、他のチャネルですべてのカバースリップを確認します(ここで使用するdsDNA抗体のGFP)。
- 各チャンネルの光の強度を記録します。イメージングプロセス全体を通じて、各チャンネルのすべての光強度が一定に保たれていることを確認してください。
- 各カバースリップを個別にイメージ化します。最初のチャネルから始めて、原子核に焦点を合わせ、画像をキャプチャします。
- チャンネルを慎重に切り替えて、細胞に再び焦点を合わせます。ピントが合ったら、画像をキャプチャします。
- 必要なチャネル(GFPおよびRFP)で画像をキャプチャした後、[ オーバーレイ ]を選択して結合された画像を観察し、画像の撮影時にスライドが動かなかったことを確認します。チャンネルが重なっていない場合は、画像が揃うまで必要なチャンネルをやり直してください。
- フラッシュドライブのアイコンをクリックして画像を保存します。カバーガラスの別の場所に移動して、イメージングを続行します。
注意: 同じ領域を繰り返しキャプチャしないように、カバースリップの異なる領域が毎回画像化されていることを確認してください。カバースリップの左上隅からイメージングを開始し、「S」字型に移動して、重複なく完全にカバーすることをお勧めします。
7. データ分析
- この方法で取得されたデータは視覚的です。DNAの漏出や核のブレブの兆候がないか画像を調べます。ブレブと漏れの例は、代表的な結果セクションに示されています。
結果
本研究では、二本鎖DNA(dsDNA)と併せて核ラミナを可視化するIF法を紹介します。IFが実行されると、キャプチャされた画像に核ブレブの兆候やDNA漏洩の兆候がないか調べることができます。
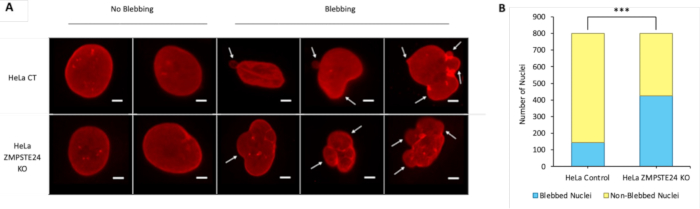
図2:HeLa CT細胞お?...
ディスカッション
提示されたプロトコルにはいくつかの重要なステップが含まれており、最も重要なのは、固定細胞を一次抗体と二次抗体で処理することです。目的のターゲットに対して良質の一次抗体を、使用する顕微鏡の範囲内で蛍光色素を用いた正しい対応する二次抗体とともに使用することで、最適な結果が得られる19。抗体の品質は、欠陥のある抗体が非?...
開示事項
著者は何も開示していません。
謝辞
この研究は、ASWへの国立老化研究所の助成金R03AG064525によって支援されました。技術指導にご協力いただいたJason A. Stewart博士と、使用した顕微鏡を提供してくださったHui Chen研究室に感謝いたします。また、これらの実験で使用したHeLa細胞株について、Fabio Martinon氏にも感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 22 mm Square Glass Coverslips | Propper Manufacturing Company | M8710 | Any size or shape can be used as long as they can be fixed to a standard microscope slide. |
| 36.5% Formaldehyde | Sigma Aldrich | F8775 | Fixation Reagent. |
| 75 cm 2 Flasks | Corning | 430725U | For cell culture. |
| Bovine Serum Albinum (BSA) | Fisher Scientific | 9048-46-8 | Antibody dilution buffer. |
| DMEM with 1 g/L glucose, L-glutamine & sodium pyruvate | Corning | 10-014-CV | For cell culture. |
| Ethanol (200 Proof) | Decon Laboratories | 2701 | For dehydration of samples before mounting, diluted to make multiple concentrations. |
| EVOS FL Digital Inverted Fluorescence Microscope | Fisher Scientific | 12-563-460 | Imaging. |
| Fish Gelatin | Sigma Aldrich | G7041 | Antibody dilution buffer. |
| Fluoromount-G | Southern Biotech | 0100-01 | Mounting medium to prevent photobleaching. |
| Parafilm (4 in) | Fisher Scientific | 13-374-12 | Any size can be substituted, as long as the coverslips being used can fit. |
| Penicillin-Streptomycin | Gibco | 15-140-122 | For cell culture. |
| Phosphate Buffered Saline (PBS) | N/A | N/A | Made and sterilized in the lab for tissue culture and solutions. |
| Premium Fetal Bovine Serum (FBS) | Atlanta Biologicals | S11150 | For cell culture. |
| Superfrost Premium Microscope Slides | Fisher Scientific | 12-544-7 | Any standard microscope slides can be used. |
| Tissue Culture Treated 6-well Flat Bottom Plates | Falcon | 353046 | For cell culture. |
| TritonX-100 | Thermofisher Scientific | A16046.AP | For washing. |
| Trypsin-EDTA (0.5%) | Gibco | 15-400-054 | For cell culture. |
| Tween 20 | Fisher Scientific | 9005-64-5 | Cell Permeation reagent. |
参考文献
- Navarro, C. L., et al. New ZMPSTE24 (FACE1) mutations in patients affected with restrictive dermopathy or related progeroid syndromes and mutation update. Eur J Hum Genet. 22 (8), 1002-1011 (2014).
- Dittmer, T. A., Misteli, T. The lamin protein family. Genome Biol. 12 (222), 1-16 (2011).
- Gruenbaum, Y., Margalit, A., Goldman, R. D., Shumaker, D. K., Wilson, K. L. The nuclear lamina comes of age. Nat Rev Mol Cell Biol. 6, 21-31 (2005).
- Navarro, C. L., et al. Lamin A and ZMPSTE24 (FACE-1) defects cause nuclear disorganization and identify restrictive dermopathy as a lethal neonatal laminopathy. Hum Mol Genet. 13 (20), 2493-2503 (2004).
- Barrowman, J., Wiley, P. A., Hudon-Miller, S. E., Hrycyna, C. A., Michaelis, S. Human ZMPSTE24 disease mutations: Residual proteolytic activity correlates with disease severity. Hum Mol Genet. 21 (18), 4084-4093 (2012).
- Richards, S., Muter, J., Ritchie, P., Lattanzi, G., Hutchison, C. The accumulation of un-repairable DNA damage in laminopathy progeria fibroblasts is caused by ROS generation and is prevented by treatment with N-acetyl cysteine. Hum Mol Genet. 20 (20), 3997-4004 (2011).
- Davies, B. S., Fong, L. G., Yang, S. H., Coffinier, C., Young, S. G. The post-translational processing of prelamin A and disease. Annu Rev Genomics Hum Genet. 10, 153-174 (2009).
- Barrowman, J., Hamblet, C., George, C. M., Michaelis, S. Analysis of prelamin A biogenesis reveals the nucleus to be a CaaX processing compartment. Mol Biol Cell. 19, 5398-5408 (2008).
- Young, S., Meta, M., Yang, S. H., Fong, L. G. Prelamin A farnesylation and progeroid syndromes. J Biol Chem. 281 (52), 39741-39745 (2006).
- Houthaeve, G., Robijns, J., Braeckmans, K., De Vos, W. H. Bypassing border control: Nuclear envelope rupture in disease. Physiol. 33 (1), 39-49 (2017).
- Schreiber, K. H., Kennedy, B. K. When lamins go bad: Nuclear structure and disease. Cell. 152 (6), 1365-1375 (2013).
- Fong, L. G., et al. Heterozygosity for Lmna deficiency eliminates the progeria-like phenotypes in Zmpste24-deficient mice. Proc Natl Acad Sci USA. 101 (52), 18111-18116 (2004).
- Pendas, A. M., et al. Defective prelamin A processing and muscular and adipocyte alterations in Zmpste24 metalloproteinase-deficient mice. Nat Genet. 31, 94-99 (2002).
- Goldman, R. D., et al. Accumulation of mutant lamin A causes progressive changes in nuclear architecture in Hutchinson-Gilford progeria syndrome. Proc Natl Acad Sci USA. 101, 8963-8968 (2004).
- Eriksson, M., et al. Recurrent de novo point mutations in lamin A cause Hutchinson–Gilford progeria syndrome. Nature. 423, 293-298 (2003).
- Young, S. G., Fong, L. G., Michaelis, S. Thematic review series: Lipid post-translational modifications. Prelamin A, Zmpste24, misshapen cell nuclei, and progeria—new evidence suggesting that protein farnesylation could be important for disease pathogenesis. J Lipid Res. 46 (12), 2531-2558 (2005).
- Worman, H. J., Michaelis, S. Prelamin A and ZMPSTE24 in premature and physiological aging. Nucleus. 14 (1), 1-17 (2023).
- Pande, S., Ghosh, D. K. Nuclear proteostasis imbalance in laminopathy-associated premature aging diseases. FASEB J. 37 (8), 1025-1035 (2023).
- Odell, I. D., Cook, D. Immunofluorescence techniques. J Invest Dermatol. 133 (4), 1-12 (2013).
- Kumar, V. Immunofluorescence and enzyme immunomicroscopy methods. J Immunoassay Immunochem. 21 (2-3), 235-253 (2000).
- Tjaden, A., Giessmann, R. T., Knapp, S., Schroder, M., Muller, S. High-content live-cell multiplex screen for chemogenomic compound annotation based on nuclear morphology. STAR Protoc. 3 (101791), 1-15 (2022).
- Gunn, A. L., Yashchenko, A. I., Dubrulle, J., Johnson, J., Hatch, E. M. A high-content screen reveals new regulators of nuclear membrane stability. Sci Rep. 14, 6013 (2024).
- Yu, L., Liu, P. Cytosolic DNA sensing by cGAS: Regulation, function, and human diseases. Nat Rev Mol Cell Biol. 6 (170), 1-14 (2021).
- Odinammadu, K. O., et al. The farnesyl transferase inhibitor (FTI) lonafarnib improves nuclear morphology in ZMPSTE24-deficient fibroblasts from patients with the progeroid disorder MAD-B. Nucleus. 14 (1), 1-14 (2023).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved