É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Detecção de Bolhas Nucleares e Vazamento de DNA em Células de Mamíferos por Imunofluorescência
Neste Artigo
Resumo
Os distúrbios da laminopatia geralmente levam a alterações no envelope nuclear, o que pode causar bolhas e vazamentos nucleares. Este estudo apresenta um método de imunofluorescência para visualizar a lâmina nuclear juntamente com o DNA de fita dupla (dsDNA), fornecendo um meio de avaliar a estrutura e integridade nuclear em células de mamíferos.
Resumo
A lâmina nuclear é uma rede de filamentos subjacentes à membrana nuclear, composta por laminas e proteínas associadas à lamina. Ele desempenha papéis críticos na arquitetura nuclear, posicionamento de poros nucleares, regulação da expressão gênica, organização da cromatina, replicação do DNA e reparo do DNA. Mutações em genes envolvidos na expressão ou processamento pós-traducional de proteínas laminadas resultam em distúrbios genéticos conhecidos como laminopatias. Especificamente, mutações nos genes LMNA ou ZMPSTE24 podem levar ao acúmulo de formas incompletamente processadas de lamina A que retêm grupos farnesil e metil, que estão ausentes na lamina A totalmente processada. Essas proteínas lamina A processadas de forma incompleta localizam-se na membrana nuclear interna em vez da lâmina nuclear, onde reside a lâmina A madura. As proteínas laminadas mal localizadas interrompem profundamente a função e a estrutura nuclear, muitas vezes resultando em bolhas nucleares. Em casos graves, pode ocorrer ruptura nuclear, causando perda de compartimentalização e vazamento de DNA genômico para o citosol. A estrutura nuclear anormal e a perda de compartimentalização podem ser identificadas por meio de imunofluorescência indireta (FI) em células fixas. Este estudo descreve esse método, empregando anticorpos específicos contra uma proteína lamina e DNA de fita dupla (dsDNA) para visualizar simultaneamente o envelope nuclear e o DNA. Essa abordagem permite uma avaliação rápida da integridade estrutural nuclear e do potencial vazamento de DNA nuclear para o citosol.
Introdução
A lâmina nuclear é uma rede de filamentos que sustenta a membrana nuclear e é composta de proteínas chamadas laminas. A lâmina nuclear desempenha papéis essenciais na arquitetura nuclear, posicionamento dos poros nucleares, regulação da expressão gênica, organização da cromatina, replicação do DNA e reparo do DNA 1,2,3. Mutações em genes que desempenham um papel na expressão das proteínas laminadas levam a distúrbios genéticos chamados laminopatias3.
A dermopatia restritiva (DR) é um distúrbio grave da laminopatia predominantemente causado por mutações heterozigóticas compostas que criam códons de terminação prematura no geneZMPSTE24 4, levando à ausência da metaloprotease ZMPSTE24. Essa laminopatia é caracterizada por retardo de crescimento intrauterino, pele rígida e firme, boca pequena, cabelos finos e defeitos de mineralização óssea1. Os pacientes com DR geralmente não vivem além da primeira semana de vida devido à insuficiência pulmonar1. Dois outros distúrbios de laminopatia conhecidos como síndrome de progeria de Hutchinson-Gilford atípica (AT-HGPS) e displasia mandibuloacral tipo B (MAD-B) envolvem expressão ZMPSTE24 reduzida e estão associados à redução da expectativa de vida e mostram semelhanças com distúrbios de envelhecimento prematuro5. A protease ZMPSTE24 que é impactada nessas laminopatias é vital na modificação pós-tradução da lamina A, que é um componente crítico da lâmina nuclear. ZMPSTE24 deficiência resulta no acúmulo de uma forma farnesilada incompletamente processada de lamina A, conhecida como prelamina A2.
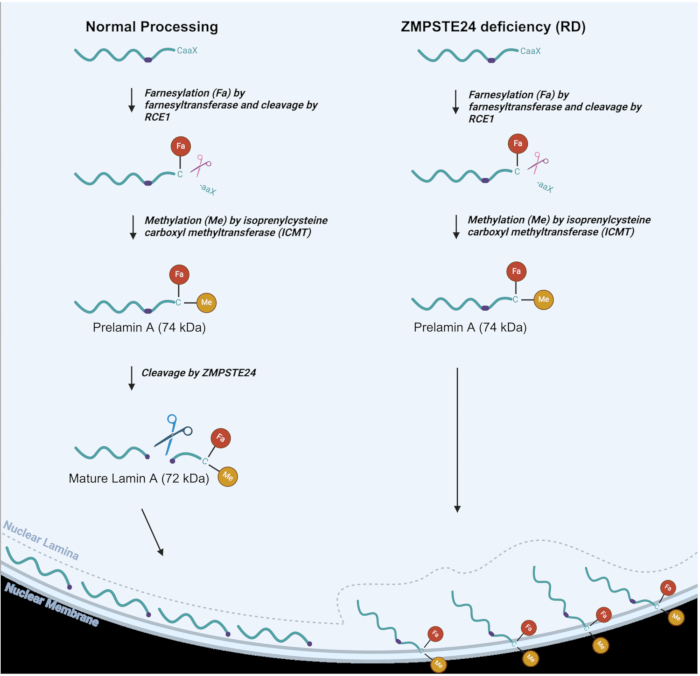
Figura 1: Processamento de lamina A em normal vs. Células RD. Via de processamento da lamina A em células normais (esquerda) e processamento alterado da lâmina A em RD ou células deficientes em ZMPSTE (direita). Os grupos farnesil (Fa) e metil (Me) são indicados. Clique aqui para ver uma versão maior desta figura.
Como visto na Figura 1, no processamento normal da lamina A, um grupo farnesil (lipídio) é ligado a um resíduo de cisteína próximo ao terminal C, seguido por clivagem proteolítica de três aminoácidos C-terminais 3,6. O resíduo de cisteína é então metilado. Essas duas modificações permitem que a prelamina A seja direcionada para a membrana nuclear interna2. ZMPSTE24 então realiza uma reação de clivagem pela qual os últimos 15 aminoácidos C-terminais, juntamente com os grupos farnesil e metil, são removidos para produzir a proteína Lamina A madura, que é entregue à lâmina nuclear 6,7,8. Em RD, AT-HGPS e MAD-B, a etapa final do processamento não ocorre de forma eficaz, uma vez que a metaloproteinase ZMPSTE24 está ausente ou não está totalmente funcional 8,9. Isso resulta no acúmulo de prelamina A, que permanece permanentemente farnesilada e metilada9. Esses últimos grupos fazem com que a prelamina A grude na membrana nuclear interna em vez de se localizar na lâmina nuclear, onde a lâmina A madura deve residir, como visto na Figura 1. Assim, a deficiência de ZMPSTE tem efeitos profundos em uma variedade de funções nucleares, juntamente com a estrutura nuclear10,11,12,13,14,15. Essas alterações na estrutura nuclear podem incluir bolhas nucleares e até rupturas nucleares, que podem resultar no vazamento de DNA para o citosol, bem como na entrada de citosol no núcleo. Núcleos de formato aberrante são de fato uma marca registrada de distúrbios de lamaminopatia, juntamente com vários outros fenótipos que são causados pelo processamento defeituoso da prelamina A16,17. Defeitos na lâmina nuclear levam a uma infinidade de impactos prejudiciais, incluindo a localização incorreta do controle de qualidade da proteína nuclear e proteínas de reparo de DNA no nucleoplasma, o que, por sua vez, resulta em vários defeitos na função nuclear18. É importante ter uma técnica simples que possa ajudar a identificar e monitorar essas características das laminopatias para facilitar a pesquisa sobre o desenvolvimento de abordagens terapêuticas destinadas a melhorar os fenótipos da laminopatia. Para camundongos com laminopatias, foi demonstrado que a eliminação da bolha nuclear se correlaciona com a eliminação generalizada dos fenótipos de laminopatia16. Uma técnica que permite o monitoramento da integridade nuclear em células humanas pode aprimorar o estudo de possíveis tratamentos para eliminar ou melhorar significativamente os fenótipos da doença.
A imunofluorescência indireta (FI) é uma técnica sensível e amplamente utilizada que utiliza tanto um anticorpo primário não marcado quanto um anticorpo secundário marcado com fluoróforo que reconhece o anticorpo primário para detectar um alvo de interesse19. Os métodos de jejum intermitente podem fornecer um meio poderoso para visualizar componentes e estruturas intracelulares específicos. Também é possível que mais de uma molécula de anticorpo secundário possa interagir com o anticorpo primário, resultando na amplificação do sinal19. O FI indireto é uma técnica versátil que também permite a detecção de múltiplos anticorpos primários com um conjunto relativamente pequeno de anticorpos secundários, uma vez que os anticorpos secundários são elevados contra o domínio Fc do anticorpo primário, que é conservado dentro da espécie19.
Este artigo descreve um método indireto (IF) para avaliar o sangramento nuclear e o vazamento de DNA em células deficientes em ZMPSTE24, usando anticorpos contra DNA de fita dupla (dsDNA) e lamina B1 para detectar DNA e lâmina nuclear, respectivamente. Para demonstrar a utilidade dessa abordagem, o procedimento foi aplicado a uma linhagem celular HeLa com expressão de ZMPSTE24 nocauteada, bem como a uma linhagem celular HeLa que expressa ZMPSTE24, e os resultados para as duas linhagens celulares foram comparados.
Protocolo
Os detalhes dos reagentes e do equipamento utilizado estão listados na Tabela de Materiais.
1. Preparação de materiais
- Autoclave pelo menos dez lamínulas e pinças de vidro quadradas de 22 mm x 22 mm antes de revestir as células nas lamínulas.
2. Preparação das soluções
- Meio suplementado: Prepare 500 mL de Dulbecco's Modified Eagle Medium (DMEM) com 10% de soro fetal bovino (FBS), 100 unidades/mL de penicilina e 100 μg/mL de estreptomicina.
- Tampão de diluição de anticorpos (ADB): Dissolva 2 g de albumina de soro bovino (BSA) e 1 g de gelatina de peixe em 80 mL de 1x solução salina tamponada com fosfato (PBS). Leve a 100 mL com PBS assim que o BSA e a gelatina de peixe estiverem dissolvidos.
- 1x PBS-Tween (PBS-T): Prepare 500 mL de PBS e 0,5 mL de Tween. Colocar num banho-maria a 37 °C até que a interpolação possa ser facilmente misturada na solução.
3. Cultivando as células desejadas
NOTA: As células HeLa foram cultivadas neste protocolo para confluência dentro de um frasco T75.
- Pulverize seis lamínulas com etanol 70% e deixe-as secar ao ar em um lenço sem fiapos. Coloque as lamínulas em uma placa estéril de 6 poços usando uma pinça, uma lamínula por poço.
- Adicione 2 mL de DMEM suplementado a cada poço na placa de 6 poços.
- Aspire o meio das células e lave as células adicionando 15 mL de PBS ao frasco, girando suavemente. Aspire o PBS e adicione 2 mL de tripsina-EDTA (tripsina).
- Cubra as células com tripsina e coloque o frasco de volta em uma incubadora umidificada a 37 ° C e CO5 a 5% 2 por 2 min para permitir que as células se desprendam do frasco.
- Adicione 8 mL de DMEM suplementado ao frasco para coletar as células tripsinizadas. Pipete para cima e para baixo algumas vezes para separar as células aglomeradas. Recolher a suspensão celular no balão e colocá-la num tubo de poliestireno de 15 ml.
- Conte as células usando um hemocitômetro.
- Adicione 500.000 células, gota a gota, diretamente em cada uma das lamínulas na placa de 6 poços. Deixe as células crescerem durante a noite a 37 °C e 5% de CO2 em uma incubadora umidificada.
4. Fixação e permeabilização celular
- Prepare a solução de formaldeído a 4% antes de usar: Adicione 2 mL de solução de formaldeído a 37,5% em 18 mL de PBS. Homogeneizar.
- Prepare a solução de permeabilização fresca antes de usar: Adicione 100 μL de Triton-X em 20 mL de PBS. Deixar incubar num banho-maria a 37 °C durante alguns minutos até que o Triton-X se dissolva na solução. Homogeneizar.
- Aspire a mídia nos poços da placa. Lave as células uma vez com 2 mL de PBS. Aspire todo o PBS dos poços.
- Adicione 2 mL de solução de formaldeído a 4% em cada poço. Deixe descansar por 10 min em temperatura ambiente (RT). Aspire formaldeído a 4% completamente dos poços.
- Lave as células 3 vezes com 2 mL de PBS. Remova o PBS completamente dos poços após cada lavagem.
- Adicione 2 mL de solução de permeabilização a cada poço. Deixe descansar por 10 min em RT. Aspire completamente a solução de permeabilização.
- Lave as células 3 vezes com 2 mL de PBS. Aspire o PBS completamente após as duas primeiras lavagens, deixando o PBS nos poços após a terceira lavagem.
NOTA: As células podem ser armazenadas na última lavagem PBS a 4 °C por algumas semanas após esta etapa, garantindo que a placa seja selada com filme de parafina para evitar a evaporação do líquido. Se o procedimento puder ser continuado, esta etapa pode ser ignorada.
5. Coloração de imunofluorescência
- Remova todo o PBS dos poços. Bloqueie as células adicionando 2 mL de ADB a cada poço e incube em RT, balançando por 30 min.
- No final do tempo de bloqueio de 30 minutos, misture a diluição do anticorpo primário no ADB (diluição de 1:1.000 para anticorpos Lamin B1 e dsDNA, Tabela 1). São necessários 75 μL para cada lamínula, portanto, 600 μL de ADB, 0,6 μL de anticorpo primário Lamin B e 0,6 μL de anticorpo dsDNA devem ser misturados para seis lamínulas. Vórtice completamente.
- Remova o ADB dos poços. Em uma superfície plana, prenda com fita adesiva um pedaço de filme de parafina que seja grande o suficiente para depositar todas as lamínulas que estão sendo processadas.
- Para cada lamínula, adicione 75 μL de diluição de anticorpo primário no filme de parafina, garantindo que nenhuma bolha seja pipetada no ADB. Em seguida, coloque as lamínulas com o lado da célula voltado para baixo na gota de ADB usando uma pinça.
- Cobrir as lamínulas de incubação com a tampa da placa do poço e deixar incubar durante 1 h em RT.
- Remova as lamínulas do filme de parafina usando uma pinça e coloque as lamínulas de volta na placa de 6 poços, com o lado da célula voltado para cima.
- Lave as lamínulas 3 vezes com 2 mL de PBS-T, balançando por 3-5 min de cada vez. Remova todo o PBS-T dos poços após cada lavagem.
- Durante a última lavagem, prepare a diluição do anticorpo secundário (1:1.000 para ambos os respectivos anticorpos secundários, Tabela 1). São necessários 75 μL para cada lamínula, portanto, 600 μL de ADB e 0,6 μL de ambos os anticorpos secundários marcados com Alexa Fluor devem ser misturados para seis lamínulas. Vórtice completamente. Cubra a diluição do anticorpo secundário da luz o máximo possível.
| Anticorpo | Fonte | Companhia | Referência | ||
| Anticorpo marcador dsDNA (HYB331-01) | Monoclonal de camundongo | Santa Cruz | SC-58749 | ||
| Anticorpo secundário altamente adsorvido anti-camundongo IgG (H+L) de cabra, Alexa Fluor Plus 488 | Cabra | ThermoFisher | Rolamento A32723 | ||
| Anticorpo secundário altamente adsorvido anti-camundongo IgG (H + L) de cabra, Alexa Fluor Plus 405 | Cabra | Invitrogen | Rolamento A31553 | ||
| Anticorpo policlonal lamina B1 | Coelho | Tecnologia de proteína | 12987-1-AP | ||
| Anticorpo secundário altamente adsorvido anti-coelho IgG (H+L) de cabra, Alexa Fluor 594 | Cabra | Invitrogen | A11037 | ||
Tabela 1: Anticorpos. Lista de todos os anticorpos usados neste protocolo.
- Remova todo o PBS-T da última lavagem. Incubar as lamínulas na diluição de anticorpos secundários em película de parafina, como no passo 5, durante 30 min à RT. Proteger da luz durante este passo e todos os restantes passos. Uma pequena caixa pode ser usada para proteger a luz. Certifique-se de trabalhar rapidamente quando necessário para adicionar ou remover soluções dos poços.
- Remova as lamínulas do filme de parafina usando uma pinça e coloque as lamínulas de volta na placa de 6 poços, com a célula voltada para cima.
- Lave as lamínulas 3 vezes com 2 mL de PBS-T. Remova todo o PBS-T dos poços após cada lavagem.
- Desidrate as lâminas com uma série de etanol (70%, 90%, 100%). Comece adicionando 2 mL de etanol a 70% em cada poço. Deixe descansar por 1-2 min e remova todo o etanol dos poços. Repita com as porcentagens de etanol restantes.
- Remova as lamínulas dos poços com uma pinça e deixe-as secar ao ar, protegidas da luz, em um lenço sem fiapos.
- Monte o lado da célula da lamínula voltado para baixo nas lâminas de microscópio de vidro usando 20 μL de meio de montagem por lamínula. Deixe secar durante a noite, protegido da luz.
6. Aquisição de imagem
- Antes de fazer a imagem, cubra as bordas das lamínulas com uma fina camada de esmalte transparente (qualquer marca ou tipo será suficiente). Deixe secar ao ar, protegido da luz.
NOTA: As instruções para usar um microscópio para obter imagens variam, dependendo do microscópio usado. A resolução da câmera no microscópio usado aqui é de 1280 x 960 pixels. Resolução semelhante ou melhor será suficiente. - Antes de colocar uma lâmina na platina do microscópio, as configurações de salvamento de arquivos devem ser ajustadas. Ligue o microscópio e insira uma unidade USB. Ajuste as configurações de salvamento nas configurações do microscópio e certifique-se de que todos os canais (GFP e RFP) sejam salvos separadamente e que as imagens estejam indo para o local correto na unidade USB.
NOTA: Crie uma pasta na unidade USB e dê a cada lamínula seu próprio nome para referência futura. - Antes da imagem, borrife um pano sem fiapos com etanol 70% e limpe suavemente a parte de trás das lâminas. Limpe suavemente a frente das corrediças, garantindo que uma pressão mínima seja exercida.
- Coloque uma lâmina limpa no microscópio, com as lamínulas voltadas para baixo. Cubra com a caixa de proteção contra luz.
- Usando uma lente objetiva de 40x, use o canal apropriado (RFP para o anticorpo lamina B1 empregado aqui) para localizar os núcleos na tela do microscópio. Ajuste a intensidade da luz para cerca de 50% para começar.
- Para verificar se há áreas supersaturadas, selecione a opção Cor desativada e certifique-se de que nenhuma área na lamínula tenha um sinal vermelho na configuração preto e branco. Se houver muito sinal vermelho, diminua a intensidade da luz até que o sinal vermelho mínimo seja exibido.
NOTA: Um sinal vermelho indica supersaturação. Se os núcleos parecerem pouco visíveis, aumente a intensidade em incrementos de 10% até que uma intensidade apropriada seja alcançada sem supersaturação. - Verifique todas as outras lamínulas com a mesma intensidade para garantir que não estejam supersaturadas. Verifique todas as lamínulas com o outro canal usando o mesmo processo acima (GFP para o anticorpo dsDNA usado aqui).
- Registre as intensidades de luz para cada canal. Certifique-se de que todas as intensidades de luz para cada canal sejam mantidas constantes durante todo o processo de imagem.
- Visualize cada lamínula individualmente. Comece com o primeiro canal, foque os núcleos e capture a imagem.
- Troque cuidadosamente os canais e coloque as células em foco novamente. Quando focado, capture a imagem.
- Depois de capturar imagens nos canais necessários (GFP e RFP), selecione Sobreposição para observar a imagem mesclada e garantir que o slide não se moveu ao tirar a imagem. Se os canais não se sobrepuserem, refaça o canal necessário até que as imagens se alinhem.
- Salve a imagem clicando no ícone da unidade flash . Vá para um local diferente na lamínula para continuar a geração de imagens.
NOTA: Certifique-se de que uma área diferente da lamínula seja fotografada a cada vez para evitar capturar a mesma região repetidamente. Recomenda-se iniciar a aquisição de imagens no canto superior esquerdo da lamínula e mover-se em um padrão "S" para garantir uma cobertura completa sem duplicação.
7. Análise dos dados
- Os dados obtidos com este método são visuais. Examine as imagens em busca de sinais de vazamento de DNA ou bolhas nucleares. Exemplos de bolhas e vazamentos são mostrados na seção de resultados representativos.
Resultados
Este estudo apresenta um método IF para visualizar a lâmina nuclear em conjunto com DNA de fita dupla (dsDNA). Uma vez que o IF é realizado, as imagens capturadas podem ser examinadas em busca de sinais de bolhas nucleares e vazamento de DNA.
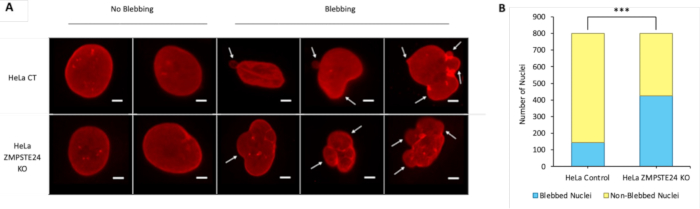
Figura 2: Bolhas nucleares em...
Discussão
O protocolo apresentado contém várias etapas críticas, sendo a mais importante o tratamento das células fixadas com anticorpos primários e secundários. Garantir o uso de um anticorpo primário de boa qualidade contra o alvo de interesse com um anticorpo secundário correspondente correto com fluoróforos dentro da faixa do microscópio usado produzirá ótimos resultados19. A qualidade do anticorpo pode impactar muito os resultados dessa técnica, pois antic...
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho foi apoiado pelo National Institute on Aging conceder R03AG064525 à ASW. Gostaríamos de agradecer ao Dr. Jason A. Stewart pela ajuda na orientação técnica e ao laboratório Hui Chen por fornecer o microscópio usado. Também gostaríamos de agradecer a Fabio Martinon pelas linhagens celulares HeLa usadas nesses experimentos.
Materiais
| Name | Company | Catalog Number | Comments |
| 22 mm Square Glass Coverslips | Propper Manufacturing Company | M8710 | Any size or shape can be used as long as they can be fixed to a standard microscope slide. |
| 36.5% Formaldehyde | Sigma Aldrich | F8775 | Fixation Reagent. |
| 75 cm 2 Flasks | Corning | 430725U | For cell culture. |
| Bovine Serum Albinum (BSA) | Fisher Scientific | 9048-46-8 | Antibody dilution buffer. |
| DMEM with 1 g/L glucose, L-glutamine & sodium pyruvate | Corning | 10-014-CV | For cell culture. |
| Ethanol (200 Proof) | Decon Laboratories | 2701 | For dehydration of samples before mounting, diluted to make multiple concentrations. |
| EVOS FL Digital Inverted Fluorescence Microscope | Fisher Scientific | 12-563-460 | Imaging. |
| Fish Gelatin | Sigma Aldrich | G7041 | Antibody dilution buffer. |
| Fluoromount-G | Southern Biotech | 0100-01 | Mounting medium to prevent photobleaching. |
| Parafilm (4 in) | Fisher Scientific | 13-374-12 | Any size can be substituted, as long as the coverslips being used can fit. |
| Penicillin-Streptomycin | Gibco | 15-140-122 | For cell culture. |
| Phosphate Buffered Saline (PBS) | N/A | N/A | Made and sterilized in the lab for tissue culture and solutions. |
| Premium Fetal Bovine Serum (FBS) | Atlanta Biologicals | S11150 | For cell culture. |
| Superfrost Premium Microscope Slides | Fisher Scientific | 12-544-7 | Any standard microscope slides can be used. |
| Tissue Culture Treated 6-well Flat Bottom Plates | Falcon | 353046 | For cell culture. |
| TritonX-100 | Thermofisher Scientific | A16046.AP | For washing. |
| Trypsin-EDTA (0.5%) | Gibco | 15-400-054 | For cell culture. |
| Tween 20 | Fisher Scientific | 9005-64-5 | Cell Permeation reagent. |
Referências
- Navarro, C. L., et al. New ZMPSTE24 (FACE1) mutations in patients affected with restrictive dermopathy or related progeroid syndromes and mutation update. Eur J Hum Genet. 22 (8), 1002-1011 (2014).
- Dittmer, T. A., Misteli, T. The lamin protein family. Genome Biol. 12 (222), 1-16 (2011).
- Gruenbaum, Y., Margalit, A., Goldman, R. D., Shumaker, D. K., Wilson, K. L. The nuclear lamina comes of age. Nat Rev Mol Cell Biol. 6, 21-31 (2005).
- Navarro, C. L., et al. Lamin A and ZMPSTE24 (FACE-1) defects cause nuclear disorganization and identify restrictive dermopathy as a lethal neonatal laminopathy. Hum Mol Genet. 13 (20), 2493-2503 (2004).
- Barrowman, J., Wiley, P. A., Hudon-Miller, S. E., Hrycyna, C. A., Michaelis, S. Human ZMPSTE24 disease mutations: Residual proteolytic activity correlates with disease severity. Hum Mol Genet. 21 (18), 4084-4093 (2012).
- Richards, S., Muter, J., Ritchie, P., Lattanzi, G., Hutchison, C. The accumulation of un-repairable DNA damage in laminopathy progeria fibroblasts is caused by ROS generation and is prevented by treatment with N-acetyl cysteine. Hum Mol Genet. 20 (20), 3997-4004 (2011).
- Davies, B. S., Fong, L. G., Yang, S. H., Coffinier, C., Young, S. G. The post-translational processing of prelamin A and disease. Annu Rev Genomics Hum Genet. 10, 153-174 (2009).
- Barrowman, J., Hamblet, C., George, C. M., Michaelis, S. Analysis of prelamin A biogenesis reveals the nucleus to be a CaaX processing compartment. Mol Biol Cell. 19, 5398-5408 (2008).
- Young, S., Meta, M., Yang, S. H., Fong, L. G. Prelamin A farnesylation and progeroid syndromes. J Biol Chem. 281 (52), 39741-39745 (2006).
- Houthaeve, G., Robijns, J., Braeckmans, K., De Vos, W. H. Bypassing border control: Nuclear envelope rupture in disease. Physiol. 33 (1), 39-49 (2017).
- Schreiber, K. H., Kennedy, B. K. When lamins go bad: Nuclear structure and disease. Cell. 152 (6), 1365-1375 (2013).
- Fong, L. G., et al. Heterozygosity for Lmna deficiency eliminates the progeria-like phenotypes in Zmpste24-deficient mice. Proc Natl Acad Sci USA. 101 (52), 18111-18116 (2004).
- Pendas, A. M., et al. Defective prelamin A processing and muscular and adipocyte alterations in Zmpste24 metalloproteinase-deficient mice. Nat Genet. 31, 94-99 (2002).
- Goldman, R. D., et al. Accumulation of mutant lamin A causes progressive changes in nuclear architecture in Hutchinson-Gilford progeria syndrome. Proc Natl Acad Sci USA. 101, 8963-8968 (2004).
- Eriksson, M., et al. Recurrent de novo point mutations in lamin A cause Hutchinson–Gilford progeria syndrome. Nature. 423, 293-298 (2003).
- Young, S. G., Fong, L. G., Michaelis, S. Thematic review series: Lipid post-translational modifications. Prelamin A, Zmpste24, misshapen cell nuclei, and progeria—new evidence suggesting that protein farnesylation could be important for disease pathogenesis. J Lipid Res. 46 (12), 2531-2558 (2005).
- Worman, H. J., Michaelis, S. Prelamin A and ZMPSTE24 in premature and physiological aging. Nucleus. 14 (1), 1-17 (2023).
- Pande, S., Ghosh, D. K. Nuclear proteostasis imbalance in laminopathy-associated premature aging diseases. FASEB J. 37 (8), 1025-1035 (2023).
- Odell, I. D., Cook, D. Immunofluorescence techniques. J Invest Dermatol. 133 (4), 1-12 (2013).
- Kumar, V. Immunofluorescence and enzyme immunomicroscopy methods. J Immunoassay Immunochem. 21 (2-3), 235-253 (2000).
- Tjaden, A., Giessmann, R. T., Knapp, S., Schroder, M., Muller, S. High-content live-cell multiplex screen for chemogenomic compound annotation based on nuclear morphology. STAR Protoc. 3 (101791), 1-15 (2022).
- Gunn, A. L., Yashchenko, A. I., Dubrulle, J., Johnson, J., Hatch, E. M. A high-content screen reveals new regulators of nuclear membrane stability. Sci Rep. 14, 6013 (2024).
- Yu, L., Liu, P. Cytosolic DNA sensing by cGAS: Regulation, function, and human diseases. Nat Rev Mol Cell Biol. 6 (170), 1-14 (2021).
- Odinammadu, K. O., et al. The farnesyl transferase inhibitor (FTI) lonafarnib improves nuclear morphology in ZMPSTE24-deficient fibroblasts from patients with the progeroid disorder MAD-B. Nucleus. 14 (1), 1-14 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados