Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détection de la bulle nucléaire et de la fuite d’ADN dans les cellules de mammifères par immunofluorescence
Dans cet article
Résumé
Les troubles de la laminopathie entraînent souvent des modifications de l’enveloppe nucléaire, ce qui peut provoquer des bulles et des fuites nucléaires. Cette étude présente une méthode d’immunofluorescence pour visualiser la lame nucléaire avec de l’ADN double brin (ADNdb), fournissant un moyen d’évaluer la structure et l’intégrité nucléaires dans les cellules de mammifères.
Résumé
La lame nucléaire est un réseau de filaments sous-jacent à la membrane nucléaire, composé de lamines et de protéines associées aux lamines. Il joue un rôle essentiel dans l’architecture nucléaire, le positionnement des pores nucléaires, la régulation de l’expression des gènes, l’organisation de la chromatine, la réplication de l’ADN et la réparation de l’ADN. Des mutations dans des gènes impliqués dans l’expression ou le traitement post-traductionnel des protéines de la lamine entraînent des troubles génétiques connus sous le nom de laminopathies. Plus précisément, des mutations dans les gènes LMNA ou ZMPSTE24 peuvent entraîner l’accumulation de formes incomplètement transformées de la lamine A qui conservent les groupes farnésyle et méthyle, qui sont absents dans la lamine A entièrement traitée. Ces protéines de la lamine A incomplètement traitées se localisent à la membrane nucléaire interne au lieu de la lame nucléaire, où réside la lamine A mature. Les protéines de lamine mal localisées perturbent profondément la fonction et la structure nucléaires, entraînant souvent des bulles nucléaires. Dans les cas graves, une rupture nucléaire peut se produire, entraînant une perte de compartimentation et une fuite d’ADN génomique dans le cytosol. Une structure nucléaire anormale et une perte de compartimentation peuvent être identifiées par immunofluorescence indirecte (FI) sur des cellules fixes. Cette étude décrit une telle méthode, utilisant des anticorps spécifiques contre une protéine de lamine et de l’ADN double brin (ADNdb) pour visualiser simultanément l’enveloppe nucléaire et l’ADN. Cette approche permet une évaluation rapide de l’intégrité structurelle nucléaire et de la fuite potentielle d’ADN nucléaire dans le cytosol.
Introduction
La lame nucléaire est un réseau de filaments qui sous-tend la membrane nucléaire et est composé de protéines appelées lamines. La lame nucléaire joue un rôle essentiel dans l’architecture nucléaire, le positionnement des pores nucléaires, la régulation de l’expression des gènes, l’organisation de la chromatine, la réplication de l’ADN et la réparation de l’ADN 1,2,3. Des mutations dans des gènes qui jouent un rôle dans l’expression des protéines du lamin entraînent des troubles génétiques appelés laminopathies3.
La dermopathie restrictive (DR) est une laminopathie sévère causée principalement par des mutations hétérozygotes composées qui créent des codons de terminaison prématurée dans le gèneZMPSTE24 4, conduisant à une absence de métalloprotéase ZMPSTE24. Cette laminopathie se caractérise par un retard de croissance intra-utérin, une peau tendue et rigide, une bouche de petite taille, des poils fins et des défauts de minéralisation osseuse1. Les patients atteints de DR ne survivent généralement pas au-delà de leur première semaine de vie en raison de l’insuffisance pulmonaire1. Deux autres troubles de la laminopathie connus sous le nom de syndrome de progeria atypique de Hutchinson-Gilford (AT-HGPS) et de dysplasie mandibulo-acrale de type B (MAD-B) impliquent une expression réduite de la ZMPSTE24 et sont associés à une durée de vie réduite et présentent des similitudes avec les troubles du vieillissement prématuré5. La protéase ZMPSTE24 qui est impactée dans ces laminopathies est vitale dans la modification post-traductionnelle de la lamine A, qui est un composant essentiel de la lame nucléaire. ZMPSTE24 carence entraîne l’accumulation d’une forme farnésylée incomplètement traitée de la lamine A, connue sous le nom de prélamine A2.
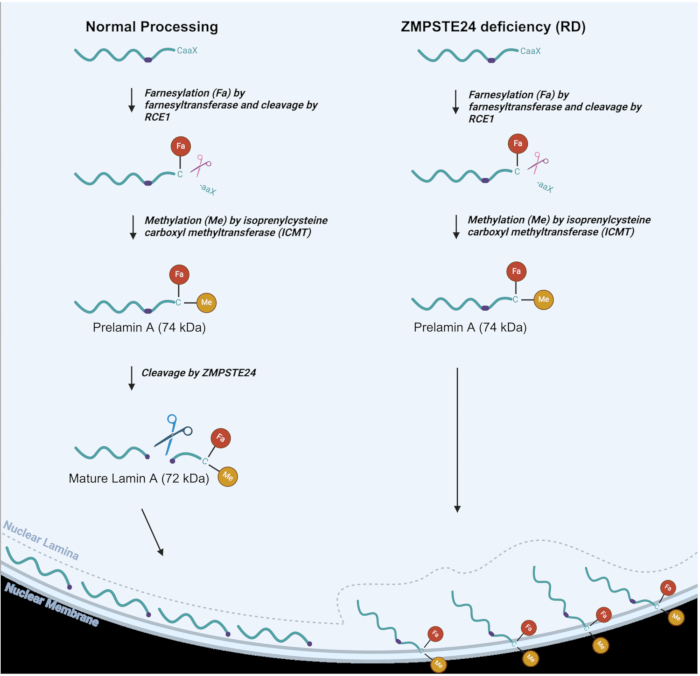
Figure 1 : Traitement de la lamine A dans les environnements normaux vs. Cellules RD. Voie de traitement de la lamine A dans les cellules normales (à gauche) et traitement de la lamine A altérée dans les cellules déficientes en RD ou ZMPSTE (à droite). Les groupes Farnésyle (Fa) et méthyle (Me) sont indiqués. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Comme le montre la figure 1, dans le traitement normal de la lamine A, un groupe farnésyle (lipide) est attaché à un résidu cystéine près de l’extrémité C-terminale, suivi d’un clivage protéolytique de trois acides aminés C-terminaux 3,6. Le résidu cystéine est ensuite méthylé. Ces deux modifications permettent au prélamin A d’être ciblé sur la membrane nucléaire interne2. ZMPSTE24 effectue ensuite une réaction de clivage par laquelle les 15 derniers acides aminés C-terminaux, ainsi que les groupes farnésyle et méthyle, sont éliminés pour produire la protéine Lamin A mature, qui est délivrée à la lame nucléaire 6,7,8. Dans RD, AT-HGPS et MAD-B, l’étape finale du traitement ne se déroule pas efficacement car la métalloprotéinase ZMPSTE24 est absente ou n’est pas entièrement fonctionnelle 8,9. Il en résulte l’accumulation de prélamine A, qui reste en permanence farnésylée et méthylée9. Ces derniers groupes font que le prélamin A adhère à la membrane nucléaire interne au lieu de se localiser dans le limbe nucléaire, où le lamin A mature devrait finalement résider, comme le montre la figure 1. Ainsi, la déficience de ZMPSTE a des effets profonds sur une variété de fonctions nucléaires, ainsi que sur la structure nucléaire10,11,12,13,14,15. Ces altérations de la structure nucléaire peuvent inclure des bulles nucléaires et même des ruptures nucléaires qui peuvent entraîner la fuite d’ADN dans le cytosol ainsi que l’entrée de cytosol dans le noyau. Les noyaux de forme aberrante sont en effet une caractéristique des troubles de la laminopathie, ainsi que de nombreux autres phénotypes causés par un traitement défectueux de la prélamine A16,17. Les défauts de la lame nucléaire entraînent une multitude d’impacts néfastes, notamment la mauvaise localisation des protéines nucléaires, le contrôle de la qualité des protéines nucléaires et les protéines de réparation de l’ADN dans le nucléoplasme, ce qui, à son tour, entraîne de nombreux défauts de la fonction nucléaire18. Il est important de disposer d’une technique simple qui puisse aider à identifier et à surveiller ces caractéristiques des laminopathies afin de faciliter la recherche dans le développement d’approches thérapeutiques visant à améliorer les phénotypes de la laminopathie. Pour les souris atteintes de laminopathies, il a été démontré que l’élimination du bulle nucléaire est corrélée à l’élimination généralisée des phénotypes de laminopathie16. Une technique qui permet de surveiller l’intégrité nucléaire dans les cellules humaines pourrait améliorer l’étude des traitements potentiels pour éliminer ou améliorer considérablement les phénotypes de la maladie.
L’immunofluorescence indirecte (FI) est une technique sensible et largement utilisée qui utilise à la fois un anticorps primaire non marqué et un anticorps secondaire marqué au fluorophore qui reconnaît l’anticorps primaire pour détecter une cible d’intérêt19. Les méthodes FI peuvent fournir un moyen puissant de visualiser des composants et des structures intracellulaires spécifiques. Il est également possible que plus d’une molécule d’anticorps secondaire interagisse avec l’anticorps primaire, entraînant une amplification du signal19. La FI indirecte est une technique polyvalente qui permet également de détecter plusieurs anticorps primaires avec un ensemble relativement petit d’anticorps secondaires, car les anticorps secondaires sont élevés contre le domaine Fc de l’anticorps primaire, qui est conservé au sein de l’espèce19.
Cet article décrit une méthode indirecte (FI) pour évaluer le gonflement nucléaire et la fuite d’ADN dans les cellules déficientes en ZMPSTE24, en utilisant des anticorps contre l’ADN double brin (ADNdb) et la lamine B1 pour détecter l’ADN et la lame nucléaire, respectivement. Pour démontrer l’utilité de cette approche, la procédure a été appliquée à une lignée cellulaire HeLa avec une expression ZMPSTE24 knock-out, ainsi qu’à une lignée cellulaire HeLa qui exprime ZMPSTE24, et les résultats des deux lignées cellulaires ont été comparés.
Protocole
Les détails des réactifs et de l’équipement utilisé sont répertoriés dans la table des matériaux.
1. Préparation des matériaux
- Autoclavez au moins dix lamelles et pinces en verre carré de 22 mm x 22 mm avant de placer les cellules sur les lamelles.
2. Préparation des solutions
- Milieu supplémenté : Préparez 500 mL de milieu Eagle modifié (DMEM) de Dulbecco avec 10 % de sérum de veau fœtal (FBS), 100 unités/mL de pénicilline et 100 μg/mL de streptomycine.
- Tampon de dilution d’anticorps (ADB) : Dissoudre 2 g d’albumine sérique bovine (BSA) et 1 g de gélatine de poisson dans 80 mL de solution saline tamponnée au phosphate (PBS). Porter à 100 mL avec du PBS une fois que le BSA et la gélatine de poisson sont dissous.
- 1x PBS-Tween (PBS-T) : Préparez 500 mL de PBS et 0,5 mL de Tween. Placer dans un bain-marie à 37 °C jusqu’à ce que Tween puisse être facilement mélangé à la solution.
3. Cultiver les cellules souhaitées
REMARQUE : Les cellules HeLa ont été cultivées dans ce protocole jusqu’à la confluence dans une fiole T75.
- Vaporisez six lamelles avec de l’éthanol à 70 % et laissez-les sécher à l’air libre sur une lingette non pelucheuse. Placez les lamelles dans une plaque stérile à 6 puits à l’aide d’une pince, une lamelle par puits.
- Ajouter 2 ml de DMEM supplémenté dans chaque puits de la plaque à 6 puits.
- Aspirez le milieu des cellules et lavez-les en ajoutant 15 ml de PBS dans le ballon, en remuant doucement. Aspirez le PBS et ajoutez 2 mL de trypsine-EDTA (trypsine).
- Enduire les cellules de trypsine et remettre le ballon dans un incubateur humidifié à 37 °C et 5 % de CO2 pendant 2 minutes pour permettre aux cellules de se détacher du ballon.
- Ajouter 8 mL de DMEM supplémenté dans la fiole pour recueillir les cellules trypsinisées. Pipetez de haut en bas plusieurs fois pour séparer les cellules agglomérantes. Recueillir la suspension cellulaire dans le ballon et la placer dans un tube de polystyrène de 15 ml.
- Comptez les cellules à l’aide d’un hémocytomètre.
- Ajoutez 500 000 cellules, goutte à goutte, directement sur chacune des lamelles de la plaque à 6 puits. Laisser les cellules se développer pendant la nuit à 37 °C et 5 % de CO2 dans un incubateur humidifié.
4. Fixation cellulaire et perméabilisation
- Préparez la solution de formaldéhyde à 4 % fraîche avant utilisation : Ajoutez 2 ml de solution de formaldéhyde à 37,5 % dans 18 ml de PBS. Mélanger.
- Préparez la solution de perméabilisation fraîche avant de l’utiliser : ajoutez 100 μL de Triton-X dans 20 mL de PBS. Laisser incuber dans un bain-marie à 37 °C pendant quelques minutes jusqu’à ce que le Triton-X se dissolve dans la solution. Mélanger.
- Aspirez le fluide dans les puits de l’assiette. Lavez les cellules une fois avec 2 ml de PBS. Aspirez tout PBS des puits.
- Ajouter 2 ml de solution de formaldéhyde à 4 % dans chaque puits. Laisser reposer 10 min à température ambiante (RT). Aspirez complètement 4 % de formaldéhyde des puits.
- Laver les cellules 3 fois avec 2 mL de PBS. Retirez complètement le PBS des puits après chaque lavage.
- Ajouter 2 mL de solution de perméabilisation dans chaque puits. Laisser reposer pendant 10 min à RT. Aspirer complètement la solution de perméabilisation.
- Laver les cellules 3 fois avec 2 mL de PBS. Aspirez complètement le PBS après les deux premiers lavages, laissant le PBS dans les puits après le troisième lavage.
REMARQUE : Les cellules peuvent être stockées dans le dernier lavage PBS à 4 °C pendant quelques semaines après cette étape, en veillant à ce que la plaque soit scellée avec un film de paraffine pour éviter l’évaporation du liquide. Si la procédure peut être poursuivie, cette étape peut être ignorée.
5. Coloration par immunofluorescence
- Retirez tout le PBS des puits. Bloquer les cellules en ajoutant 2 mL d’ADB dans chaque puits et incuber à RT, en se balançant pendant 30 min.
- Vers la fin de la période de blocage de 30 minutes, mélanger la dilution primaire de l’anticorps dans l’ADB (dilution de 1:1 000 pour les anticorps Lamin B1 et d’ADNdb, tableau 1). 75 μL sont nécessaires pour chaque lamelle, donc 600 μL d’ADB, 0,6 μL d’anticorps primaire Lamin B et 0,6 μL d’anticorps d’ADNdb doivent être mélangés pour six lamelles. Vortex à fond.
- Retirez l’ADB des puits. Sur une surface plane, collez un morceau de film de paraffine suffisamment grand pour déposer toutes les lamelles en cours de traitement.
- Pour chaque lamelle, ajoutez 75 μL de dilution d’anticorps primaires sur le film de paraffine, en veillant à ce qu’aucune bulle ne soit pipetée dans l’ADB. Ensuite, placez les lamelles côté cellule vers le bas sur la gouttelette d’ADB à l’aide d’une pince.
- Couvrez les lamelles d’incubation avec le couvercle de la plaque à puits et laissez incuber pendant 1 h à RT.
- Retirez les lamelles du film de paraffine à l’aide d’une pince et remettez-les dans la plaque à 6 puits, côté cellule vers le haut.
- Lavez les lamelles 3 fois avec 2 ml de PBS-T, en les balançant pendant 3 à 5 minutes à chaque fois. Retirez tout PBS-T des puits après chaque lavage.
- Lors du dernier lavage, préparez la dilution secondaire des anticorps (1:1 000 pour les deux anticorps secondaires respectifs, tableau 1). 75 μL sont nécessaires pour chaque lamelle, donc 600 μL d’ADB et 0,6 μL des deux anticorps secondaires marqués Alexa Fluor doivent être mélangés pour six lamelles. Vortex à fond. Couvrez autant que possible la dilution secondaire de l’anticorps à l’abri de la lumière.
| Anticorps | Source | Compagnie | Référence | ||
| Anticorps marqueur d’ADNdb (HYB331-01) | Souris monoclonale | Santa Cruz | SC-58749 | ||
| Anticorps secondaire anti-IgG de souris (H+L) hautement adsorbé croisé, Alexa Fluor Plus 488 | Chèvre | ThermoFisher | N° A32723 | ||
| Anticorps secondaire anti-Souris IgG (H+L) de chèvre à adsorption croisée, Alexa Fluor Plus 405 | Chèvre | Invitrogen | N° A31553 | ||
| Anticorps polyclonal à la lamine B1 | Lapin | Proteintech | Référence 12987-1-AP | ||
| Anticorps secondaire anti-Rabbit IgG (H+L) hautement adsorbé croisé, Alexa Fluor 594 | Chèvre | Invitrogen | N° A11037 | ||
Tableau 1 : Anticorps. Liste de tous les anticorps utilisés dans le cadre de ce protocole.
- Retirez tout PBS-T du dernier lavage. Incuber les lamelles dans la dilution secondaire de l’anticorps sur un film de paraffine, comme à l’étape 5, pendant 30 min à RT. Protéger de la lumière pour cette étape et toutes les étapes restantes. Une petite boîte peut être utilisée pour protéger la lumière. Assurez-vous de travailler rapidement au besoin pour ajouter ou retirer des solutions des puits.
- Retirez les lamelles du film de paraffine à l’aide d’une pince et remettez-les dans la plaque à 6 puits, côté cellule vers le haut.
- Lavez les lamelles 3 fois avec 2 mL de PBS-T. Retirez tout PBS-T des puits après chaque lavage.
- Déshydratez les lames avec une série d’éthanol (70 %, 90 %, 100 %). Commencez par ajouter 2 ml d’éthanol à 70 % dans chaque puits. Laisser reposer pendant 1 à 2 minutes et retirer tout l’éthanol des puits. Répétez l’opération avec les pourcentages d’éthanol restants.
- Retirez les lamelles des puits à l’aide d’une pince et laissez-les sécher à l’air, à l’abri de la lumière, sur une lingette non pelucheuse.
- Montez la lamelle côté cellule vers le bas sur des lames de microscope en verre à l’aide de 20 μL de support par lamelle. Laisser sécher toute la nuit, à l’abri de la lumière.
6. Acquisition d’images
- Avant l’imagerie, enduisez les bords des lamelles d’une fine couche de vernis à ongles transparent (n’importe quelle marque ou type suffira). Laisser sécher à l’air, à l’abri de la lumière.
REMARQUE : Les instructions d’utilisation d’un microscope pour l’imagerie varient en fonction du microscope utilisé. La résolution de la caméra du microscope utilisé ici est de 1280 x 960 pixels. Une résolution similaire ou meilleure suffira. - Avant de placer une lame sur la platine du microscope, les paramètres d’enregistrement des fichiers doivent être ajustés. Allumez le microscope et insérez une clé USB. Ajustez les paramètres d’enregistrement dans les paramètres du microscope et assurez-vous que tous les canaux (GFP et RFP) seront enregistrés séparément et que les images vont au bon emplacement sur la clé USB.
REMARQUE : Créez un dossier sur la clé USB et donnez à chaque lamelle son propre nom pour référence future. - Avant l’imagerie, vaporisez une lingette non pelucheuse avec de l’éthanol à 70 % et nettoyez doucement le dos des lames. Nettoyez délicatement l’avant des glissières, en veillant à ce qu’une pression très minimale soit exercée.
- Placez une lame propre sur le microscope, lamelles vers le bas. Couvrir avec la boîte de protection de la lumière.
- À l’aide d’une lentille d’objectif 40x, utilisez le canal approprié (RFP pour l’anticorps de lamine B1 utilisé ici) pour localiser les noyaux sur l’écran du microscope. Ajustez l’intensité lumineuse à environ 50 % pour commencer.
- Pour vérifier s’il y a des zones sursaturées, sélectionnez l’option Couleur désactivée et assurez-vous qu’aucune zone de la lamelle n’a de signal rouge en noir et blanc. S’il y a beaucoup de signal rouge, réduisez l’intensité lumineuse jusqu’à ce que le signal rouge minimal s’affiche.
REMARQUE : Un signal rouge indique une sursaturation. Si les noyaux apparaissent à peine visibles, augmentez l’intensité par incréments de 10 % jusqu’à ce qu’une intensité appropriée soit atteinte sans sursaturation. - Vérifiez toutes les autres lamelles avec la même intensité pour vous assurer qu’elles ne sont pas sursaturées. Vérifiez toutes les lamelles avec l’autre canal en utilisant le même processus que ci-dessus (GFP pour l’anticorps d’ADNdb utilisé ici).
- Enregistrez les intensités lumineuses pour chaque canal. Assurez-vous que toutes les intensités lumineuses de chaque canal sont maintenues constantes tout au long du processus d’imagerie.
- Imagez chaque lamelle individuellement. Commencez par le premier canal, concentrez les noyaux et capturez l’image.
- Changez de canal avec précaution et faites à nouveau la mise au point sur les cellules. Une fois mis au point, capturez l’image.
- Après avoir capturé les images dans les canaux nécessaires (GFP et RFP), sélectionnez Superposition pour observer l’image fusionnée et vous assurer que la diapositive n’a pas bougé lors de la prise de l’image. Si les canaux ne se chevauchent pas, refaites le canal nécessaire jusqu’à ce que les images s’alignent.
- Enregistrez l’image en cliquant sur l’icône de la clé USB . Déplacez-vous à un autre endroit sur la lamelle pour poursuivre l’imagerie.
REMARQUE : Assurez-vous qu’une zone différente de la lamelle est imagée à chaque fois pour éviter de capturer la même région à plusieurs reprises. Il est recommandé de commencer l’imagerie dans le coin supérieur gauche de la lamelle et de se déplacer en « S » pour assurer une couverture complète sans duplication.
7. Analyse des données
- Les données obtenues avec cette méthode sont visuelles. Examinez les images à la recherche de signes de fuite d’ADN ou de décoloration nucléaire. Des exemples de bulles et de fuites sont présentés dans la section des résultats représentatifs.
Résultats
Cette étude introduit une méthode IF pour visualiser la lame nucléaire en conjonction avec l’ADN double brin (ADNdb). Une fois l’IF effectuée, les images capturées peuvent être examinées à la recherche de signes de décoloration nucléaire et de fuite d’ADN.
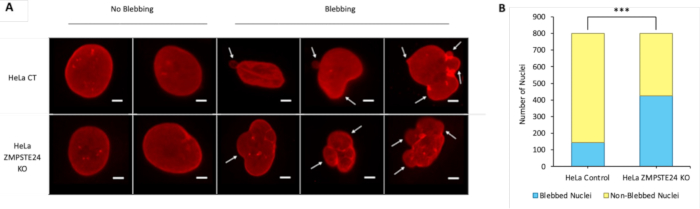
Figure 2 <...
Discussion
Le protocole présenté contient plusieurs étapes critiques, la plus importante étant le traitement des cellules fixées avec des anticorps primaires et secondaires. S’assurer de l’utilisation d’un anticorps primaire de bonne qualité contre la cible d’intérêt avec un anticorps secondaire correspondant correct avec des fluorophores dans la portée du microscope utilisé donnera des résultats optimaux19. La qualité de l’anticorps peut avoir un impac...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par la subvention du National Institute on Aging R03AG064525 à ASW. Nous tenons à remercier le Dr Jason A. Stewart pour son aide dans le mentorat technique et le laboratoire Hui Chen pour avoir fourni le microscope utilisé. Nous tenons également à remercier Fabio Martinon pour les lignées cellulaires HeLa utilisées dans ces expériences.
matériels
| Name | Company | Catalog Number | Comments |
| 22 mm Square Glass Coverslips | Propper Manufacturing Company | M8710 | Any size or shape can be used as long as they can be fixed to a standard microscope slide. |
| 36.5% Formaldehyde | Sigma Aldrich | F8775 | Fixation Reagent. |
| 75 cm 2 Flasks | Corning | 430725U | For cell culture. |
| Bovine Serum Albinum (BSA) | Fisher Scientific | 9048-46-8 | Antibody dilution buffer. |
| DMEM with 1 g/L glucose, L-glutamine & sodium pyruvate | Corning | 10-014-CV | For cell culture. |
| Ethanol (200 Proof) | Decon Laboratories | 2701 | For dehydration of samples before mounting, diluted to make multiple concentrations. |
| EVOS FL Digital Inverted Fluorescence Microscope | Fisher Scientific | 12-563-460 | Imaging. |
| Fish Gelatin | Sigma Aldrich | G7041 | Antibody dilution buffer. |
| Fluoromount-G | Southern Biotech | 0100-01 | Mounting medium to prevent photobleaching. |
| Parafilm (4 in) | Fisher Scientific | 13-374-12 | Any size can be substituted, as long as the coverslips being used can fit. |
| Penicillin-Streptomycin | Gibco | 15-140-122 | For cell culture. |
| Phosphate Buffered Saline (PBS) | N/A | N/A | Made and sterilized in the lab for tissue culture and solutions. |
| Premium Fetal Bovine Serum (FBS) | Atlanta Biologicals | S11150 | For cell culture. |
| Superfrost Premium Microscope Slides | Fisher Scientific | 12-544-7 | Any standard microscope slides can be used. |
| Tissue Culture Treated 6-well Flat Bottom Plates | Falcon | 353046 | For cell culture. |
| TritonX-100 | Thermofisher Scientific | A16046.AP | For washing. |
| Trypsin-EDTA (0.5%) | Gibco | 15-400-054 | For cell culture. |
| Tween 20 | Fisher Scientific | 9005-64-5 | Cell Permeation reagent. |
Références
- Navarro, C. L., et al. New ZMPSTE24 (FACE1) mutations in patients affected with restrictive dermopathy or related progeroid syndromes and mutation update. Eur J Hum Genet. 22 (8), 1002-1011 (2014).
- Dittmer, T. A., Misteli, T. The lamin protein family. Genome Biol. 12 (222), 1-16 (2011).
- Gruenbaum, Y., Margalit, A., Goldman, R. D., Shumaker, D. K., Wilson, K. L. The nuclear lamina comes of age. Nat Rev Mol Cell Biol. 6, 21-31 (2005).
- Navarro, C. L., et al. Lamin A and ZMPSTE24 (FACE-1) defects cause nuclear disorganization and identify restrictive dermopathy as a lethal neonatal laminopathy. Hum Mol Genet. 13 (20), 2493-2503 (2004).
- Barrowman, J., Wiley, P. A., Hudon-Miller, S. E., Hrycyna, C. A., Michaelis, S. Human ZMPSTE24 disease mutations: Residual proteolytic activity correlates with disease severity. Hum Mol Genet. 21 (18), 4084-4093 (2012).
- Richards, S., Muter, J., Ritchie, P., Lattanzi, G., Hutchison, C. The accumulation of un-repairable DNA damage in laminopathy progeria fibroblasts is caused by ROS generation and is prevented by treatment with N-acetyl cysteine. Hum Mol Genet. 20 (20), 3997-4004 (2011).
- Davies, B. S., Fong, L. G., Yang, S. H., Coffinier, C., Young, S. G. The post-translational processing of prelamin A and disease. Annu Rev Genomics Hum Genet. 10, 153-174 (2009).
- Barrowman, J., Hamblet, C., George, C. M., Michaelis, S. Analysis of prelamin A biogenesis reveals the nucleus to be a CaaX processing compartment. Mol Biol Cell. 19, 5398-5408 (2008).
- Young, S., Meta, M., Yang, S. H., Fong, L. G. Prelamin A farnesylation and progeroid syndromes. J Biol Chem. 281 (52), 39741-39745 (2006).
- Houthaeve, G., Robijns, J., Braeckmans, K., De Vos, W. H. Bypassing border control: Nuclear envelope rupture in disease. Physiol. 33 (1), 39-49 (2017).
- Schreiber, K. H., Kennedy, B. K. When lamins go bad: Nuclear structure and disease. Cell. 152 (6), 1365-1375 (2013).
- Fong, L. G., et al. Heterozygosity for Lmna deficiency eliminates the progeria-like phenotypes in Zmpste24-deficient mice. Proc Natl Acad Sci USA. 101 (52), 18111-18116 (2004).
- Pendas, A. M., et al. Defective prelamin A processing and muscular and adipocyte alterations in Zmpste24 metalloproteinase-deficient mice. Nat Genet. 31, 94-99 (2002).
- Goldman, R. D., et al. Accumulation of mutant lamin A causes progressive changes in nuclear architecture in Hutchinson-Gilford progeria syndrome. Proc Natl Acad Sci USA. 101, 8963-8968 (2004).
- Eriksson, M., et al. Recurrent de novo point mutations in lamin A cause Hutchinson–Gilford progeria syndrome. Nature. 423, 293-298 (2003).
- Young, S. G., Fong, L. G., Michaelis, S. Thematic review series: Lipid post-translational modifications. Prelamin A, Zmpste24, misshapen cell nuclei, and progeria—new evidence suggesting that protein farnesylation could be important for disease pathogenesis. J Lipid Res. 46 (12), 2531-2558 (2005).
- Worman, H. J., Michaelis, S. Prelamin A and ZMPSTE24 in premature and physiological aging. Nucleus. 14 (1), 1-17 (2023).
- Pande, S., Ghosh, D. K. Nuclear proteostasis imbalance in laminopathy-associated premature aging diseases. FASEB J. 37 (8), 1025-1035 (2023).
- Odell, I. D., Cook, D. Immunofluorescence techniques. J Invest Dermatol. 133 (4), 1-12 (2013).
- Kumar, V. Immunofluorescence and enzyme immunomicroscopy methods. J Immunoassay Immunochem. 21 (2-3), 235-253 (2000).
- Tjaden, A., Giessmann, R. T., Knapp, S., Schroder, M., Muller, S. High-content live-cell multiplex screen for chemogenomic compound annotation based on nuclear morphology. STAR Protoc. 3 (101791), 1-15 (2022).
- Gunn, A. L., Yashchenko, A. I., Dubrulle, J., Johnson, J., Hatch, E. M. A high-content screen reveals new regulators of nuclear membrane stability. Sci Rep. 14, 6013 (2024).
- Yu, L., Liu, P. Cytosolic DNA sensing by cGAS: Regulation, function, and human diseases. Nat Rev Mol Cell Biol. 6 (170), 1-14 (2021).
- Odinammadu, K. O., et al. The farnesyl transferase inhibitor (FTI) lonafarnib improves nuclear morphology in ZMPSTE24-deficient fibroblasts from patients with the progeroid disorder MAD-B. Nucleus. 14 (1), 1-14 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon