Method Article
진핵 세포에서 염색체 외 원형 DNA의 게놈 차원의 정화
요약
This paper presents a sensitive method called Circle-Seq for purifying extrachromosomal circular DNA (eccDNA). The method encompasses column purification, removal of remaining linear chromosomal DNA, rolling-circle amplification and high-throughput sequencing. Circle-Seq is applicable to genome-scale screening of eukaryotic eccDNA and studying genome instability and copy-number variation.
초록
염색체 외 원형 DNA를 (eccDNAs)는 사카로 마이 세스 세레 비지에 공통의 유전 적 요소뿐만 아니라 다른 진핵 생물에서보고됩니다. EccDNAs는 다세포 생물의 체세포들과 단세포 진핵 생물의 진화에 유전 적 변이에 기여한다. eccDNA을 검출하기위한 민감한 방법은 이러한 요소 게놈 안정성과 방법 환경 및 생물학적 요인 진핵 세포에서의 형성을 유도에 미치는 영향을 명확히 할 필요가있다. 이 동영상은 원 - 서열라는 민감한 eccDNA 정제 방법을 제시한다. 상기 방법은 원형의 DNA, 선형 염색체 DNA, eccDNA 롤링 써클 증폭 깊은 시퀀싱 매핑 잔여 제거 칼럼 정제를 포함한다. 광범위한 엑소 뉴 클레아 제 처리를 충분히 선형 염색체 DNA의 분해에 요구되었다. 하여 롤링 써클 증폭 공정오프라인 - 높이 :. 정상; "10 10 세포의 세 S. cerevisiae의 CEN.PK 인구의 원-SEQ 방법> 선형 DNA를 통해 원형 DNA에 대한 풍부한 φ (29) 중합 효소 유효성 검사는 1 킬로보다 큰 크기의 eccDNA 프로파일 수백 검출 . ASP3-1, COS111, CUP1, RSC30, HXT6, S288c 및 CEN.PK는 DNA의 회람이 유전자좌에서 균주 사이에 보존되는 것을 알 수 모두에서 원형 DNA에 HXT7 유전자. 합에서의 반복 된 결과, 서클-SEQ 방법은 광범위한있다 진핵 생물뿐만 아니라 특정 eccDNA 유형을 검출 eccDNA위한 게놈 규모 스크리닝 적용.
서문
이 세포의 큰 인구에 단일 DNA 분자의 변화를 확인이 필요하기 때문에 초기 또는 과도 염색체 증폭을 검출하는 것은 어렵다. 염색체 복사 번호 변형 (CNVs)은 일반적으로 변화 1, 2를 생성 메커니즘의 증거로 만 최종 CNV 구조를 떠나, 그들의 설립 후 잘 검출된다. 검출 및 유전자 재 배열에서 진행중인 프로세스를 규명 할 수 맥락막 신생 혈관 형성의 초기 단계에서 염색체 외 원형 DNA (eccDNA)를 복구.
이전의 eccDNA 새로이 발견 전자 현미경 3 중기 염색체 4, 2 차원 전기 영동 5 김사 염색 하였다. 이러한 방법은 원형 DNA의 서열에 대해 거의 또는 전혀 정보를 제공합니다. 이러한 남부는 현장 hybridizatio에서 PCR 8, 반대 6, 7, 또는 형광을 블로 팅으로 대상 기술N 9 특정 eccDNA 요소에 대한 증거를 제공합니다. 이러한 방법 중 어느 것도 셀 인구에있는 기존의 모든 eccDNA 유형의 순서를 제공하지 않습니다.
세포의 풀에서 발산 게놈은 게놈 서열 및 / 또는 타일 어레이 (10, 11)을 특징으로 할 수있다. 종래 DNA 정제 방법에 의해 결실 또는 증폭을 검출하는 것은 일반적으로 돌연변이 된 대립 유전자는 세포 집단 (12, 13)의 적어도 0.1 %를 나타내는 것을 필요로한다. Acentric eccDNAs 인해 자신의 중심절의 부족과 복제에서 DNA 합성의 가능성이없는 경우에 세포 배양에 더욱 과도 것으로 예상된다. 대부분은 아마도 eccDNAs 소량에 있고 그 서열이 게놈 유사 보낸 따라서, 다른 DNA 추출 방법 eccDNAs를 검출 할 필요가있다.
몇몇 원형 DNA 정제 기술은 염색체 DNA 원형의 구조적 차이를 이용한다. 예 고속 용 ultracentrifug염화 세슘 구배에 ATION 인간 헬라 암 세포주 14 350-3000 염기쌍 (BP) 큰 eccDNAs을 분리하는데 사용된다. 그러나, 고속의 침강 속도 (15) 및 eccDNA 수율 변경 닉 수퍼 코일을 원형 DNA 구조의 골격을 끊거나 할 수있다. 두타와 동료는 드 노보, 마우스 조직에서뿐만 아니라 닭의 문화와 인간의 세포 (16, 17)에서 원형 DNA의 게놈 규모 식별하는 방법을 개발했다. 그들의 방법은 플라스미드 정제 및 효소 반응 및 DNA의 추출 몇 차례 뒤에 자당 초 원심 분리에 의한 균질 조직에서 핵의 추출이다. 이들 프로토콜은 주로 200 ~ 400 염기쌍의 eccDNAs라는 microDNAs를 식별합니다. 두타와 동료는 사카로 마이 세스 세레 비지에서 microDNAs의 정화를 시도했지만이 효모 종 16 microDNA를 기록 할 수 없습니다.
우리는 새로운 방법을 개발했다원 - 서열라는 효모에서 eccDNA의 드 노보 검출. 이 방법은 전체 유전자를 수행하기에 충분히 큰 및 86 킬로 (킬로바이트) 미토콘드리아 DNA (미토콘드리아 DNA)로 큰 원형의 DNA 분자에 대한 게놈 규모의 설문 조사를 할 수 있습니다. 서클-SEQ 방법은 진핵 효모 세포에 최적화 깊은 서열과 결합 잘 설립 원핵 플라스미드 정제 방법 (18, 19)에서 개발되었다. 서클-서열 접근, 1756 다른 eccDNAs, 모든보다 큰 1킬로바이트를 사용하여 10 S.에서 검출되었다 cerevisiae의 S288c 20를 집단. 크기 컷 - 오프는 전체 유전자를 수행하기에 충분히 큰했다 eccDNA에 초점을 선택되었다. 원 - 서열은 매우 민감; 그것은 전지 (20)의 수천에서 하나의 eccDNA를 발견했습니다. 현재 연구에서, 원형 서열이 분리 다른 S. 세 생물학적 복제에서 294 eccDNAs를 식별하는 데 사용 cerevisiae의 효모 균주, CEN.PK. 데이터 eccDNA 공통 유전자 eleme이라고 밝혀S.에서 NT cerevisiae의 변종.
프로토콜
주 : 원형 DNA 정제와 시퀀싱 방법 (원-SEQ)의 개요는도 1에 도시되어있다.
1. 배양, 세포 수확 및 플라즈마 멤브레인 혼란
- 효모 펩톤 덱스 트로 오스 50 ml의 완전한 영양 배지 (YPD) 내로 O / N 문화에서 (예 : 사카로 마이 세스 세레 비지에 대한) 효모 세포를 접종한다. 1-3 X 105 세포 / ml의 낮은 초기 세포 밀도 또는 대략 0.01 OD 600의 광학 밀도로 접종한다.
- 세포가 약 24 시간 또는 OD 600> 10.0에서 광학 밀도 48 후, 약 1 × 10 10 세포의 최대 세포 밀도에 도달 할 때까지 분 (RPM) 당 150 원에서 교반하면서 30 ° C에서 세포를 품어.
주 : 낮은 세포 농도가 사용될 수있는 바와 같이, 배양 시간은 중요하지 않다.
- 세포가 약 24 시간 또는 OD 600> 10.0에서 광학 밀도 48 후, 약 1 × 10 10 세포의 최대 세포 밀도에 도달 할 때까지 분 (RPM) 당 150 원에서 교반하면서 30 ° C에서 세포를 품어.
- , 50 ML 원뿔 튜브에 능가 문화를 전송에서 원심 분리하여 세포를 펠릿800 XG에 3 분 동안 상층 액을 버린다.
- 3 분 800 XG에서 원심 분리하여 세포를 다시 펠렛 상층 액을 버리고, 10 mM 트리스 - CL, 1 mM의 EDTA, pH가 8.0 25 ml의 완충 용액으로 펠렛을 씻으십시오.
- 플라스미드 컬럼 정제 키트로부터 공급 된 1.2 ml의 재현 탁 완충액에서 세포 펠렛을 재현 탁.
- 선택 단계 : 원형 DNA 요소 (20)의 정제를 위해 컨트롤과 같은 높은 희석 플라스미드를 추가합니다.
주 : 현재의 데이터 세트에서, 7.7 μL 플라스미드 혼합물 10 10 세포를 포함하는 각 샘플에 대해 적용 하였다. 플라스미드 재고 혼합물 다른 농도의 세 가지 플라스미드 구성; 0.01 NG / 샘플의 38 NG / 샘플에서 나 pBR322, pUC19를 0.5 NG / 샘플에서, 그리고 pUG72. - 총 현탁 부피 : 3 비율이 2 ml의 마이크로 원심 분리 튜브에 세포 현탁액을 전송하고, 각각은 1에서 0.5 mm의 유리 비드로 보충.
- 플라즈마 셀을 방해 10 분간 볼텍스 최대 속도로 각 튜브막. 30 초 동안 268 XG에서 원심 분리하여 구슬을 펠렛 새로운 튜브에 두 가지의 microcentrifuge 튜브에서 1.2 ml의를 결합 뜨는을 전송합니다.
참고 : 얼터너티브 0.6 ml의 부유 완충 용액의 세포를 파괴하는 zymolyase를 사용, 1.6-1.7를 진행한다. zymolyase 십 단위는 35 ° C에서 1.5 시간에서 5 × 10 7 세포를 파괴 할 수 있습니다.
컬럼 크로마토 그래피 2. EccDNA 심화
- 플라스미드의 열 정제 키트에서 프로토콜을 따르십시오. 간단히, 1.2 ml의 알칼리 용액으로 각각의 샘플을 치료, 부드럽게 섞어 실온에서 3 분을 품어.
- 1.2 ml의 중화 버퍼를 추가 5 분 9650 XG에 부드럽게 원심 분리기 섞는다.
- 1 ML의 평형 용액과 평형을 열 상에 솔루션을로드하고 액체가 중력에 의해 열을 통과 할 수 있습니다.
- 4 ml의 세척 용액으로 열을 씻으십시오. 용액이 수지를 통과 할 때 조심스럽게 0.3 ㎖의 용출 정도 추가lution는 0.35 ml의 컬럼 공극 부피의 대부분을 대체합니다.
- 1 ㎖의 용출 용액 새로운 포집 관에 DNA를 용리 0.8 mL의 침전 혼합물을 첨가하여 DNA를 침전. 10 분 동안 9,650 XG에 원심 분리기.
- 5 ~ 15 분 동안 5 분, 공기 건조를 위해 9650 XG에 0.5 ml의 70 % 에탄올, 원심 분리기와 DNA 펠렛을 씻고 25 ㎕를 멸균 물에 정제 된 DNA를 용해.
참고 : 물에 DNA 만, 단기간의 운용을 권장합니다. 우선적으로 3 단계로 직접 진행합니다.
3. 소화 선형 염색체 DNA 남은의
- 선택 단계 : 엑소 뉴 클레아 제에 의해 선형 DNA의 특정 소화를 촉진 등을 NotI으로 드문 절단 효소로 정제 된 DNA를 치료합니다. 5 μg의 DNA를 들어, 50 μL의 총 부피로 1 단위를 NotI, 5 ㎕의 10 배 소화 버퍼와 멸균 수를 사용합니다. 16 시간 가열하여 37 ℃에서 반응물을 인큐베이션을 5 분 동안 80 ℃에서 효소를 실활.
- 20 추가단위 엑소 뉴 클레아 제 (2 μL) 4 μL의 ATP (25 mM)을 34 ㎕의 멸균 수와 50 ㎕의 효소 절단 된 DNA에 직접적으로 10 ㎕의 10 배 반응 완충액은 ATP 의존적 엑소 뉴 클레아 제를 사용하여 100 μL의 1X 반응 부피에 도달 전부.
- 오일 이상 37 ° C에서 선형 단일 가닥 및 이중 가닥 DNA의 가수 분해를 수행합니다. 추가로 4 μL의 ATP (25 mM)을 0.6 ㎕의 10 배 반응 버퍼와 1 배 반응 볼륨 효소 DNA의 소화를 계속 매 24 시간을 엑소 뉴 클레아 제 20 단위를 추가합니다.
- 선형 DNA 제거한 후, 엑소 뉴 클레아 제 처리 용액으로부터 샘플 2 μL은 액틴 유전자 ACT1 20 염색체 마커를 사용하여 정량 중합 효소 연쇄 반응 (qPCR에)에 의해 염색체 선형 DNA의 제거를 확인한다.
- 각 20 μL qPCR의 반응 부피는 2 μL의 엑소 뉴 클레아 제 처리 된 샘플을 150 nm의 ACT1 프라이머 5'-TCCGTCTGGATTGGTGGTTCTA-3 '와 5'-TGGACCACTTTCGTC를 포함GTATTC-3 ', 2 % (부피 / 부피), 디메틸 술폭 시드 및 10 ㎕의 녹색 형광 마스터 믹스.
- 반응 조건을 사용하여, 95 ° C에서 15 초 45주기와 60 ° C에서 30 초 다음에 95 ° C에서 3 분.
참고 :이 유전자의 복제 수 변이 너무 eccDNA는 ACT1을 수행하지 않아야 21-23 해로운 때문에 ACT1 선형 DNA에 특히 적합한 마커입니다. - qPCR에 의해 DNA 소화의 분석에 대한 대안은 표준 PCR (4.3) 또는 프로피 디움 요오드 염색 (4.4)입니다.
- ACT1 프라이머 5'-TGGATTCTGGTATGTTCTAGC-3 '와-GAACGACGTGAGTAACACC-3 5''와 PCR 템플릿으로 2 μL의 엑소 뉴 클레아 제 처리 된 샘플을 사용합니다. 긍정적 인 ACT1 컨트롤로, 게놈 S.을 ng를 50 ~ 100을 사용 템플릿으로 cerevisiae의 DNA. PCR 반응 조건; 95 ° C, 56 ° C에서 30 초, 72 ℃에서 1 분 30 초 35주기 다음 95 ℃에서 3 분.
- 겔 electrophore에 의해 실행 PCR은 반응0.5 μg의 1 % 아가로 오스에 동생 / ㎖ 에티 디움 브로마이드. 0.8 킬로바이트 ACT1 밴드를 찾습니다.
- 부재 또는 선형 DNA의 존재는 이전과 DNA 증폭 후 요오드화 프로피 듐 염색에 의해 검사 될 수있다.
- 20 mM의 요오드화 프로피 듐 주식의 1,000 H 2 O 희석 용액 : 1과 1 볼륨 : 1의 각 DNA 샘플을 섞는다. 실온에서 10 ~ 20 분 동안 어둠 속에서 해결책을두고 663-738 nm의 5 ~ 30 초 노출 시간에 빨간색 여기 형광 필터를 사용하여 100 배 배율에서 형광 현미경으로 DNA 염색을 분석 할 수 있습니다. DNA-염색 대조군으로 사용 효모 및 / 또는 ø29 증폭 플라스미드에서 게놈 DNA를 ø29은 증폭.
- 가열을 30 분 동안 70 ° C에서 엑소 뉴 클레아 제 용액을 비활성화한다.
4. DNA 증폭
- (가) 정제 증폭 ø29 DNA 중합 효소 24-26 협정과 단계 3.5)에서 eccDNA을 강화중합 효소 제조 업체의 프로토콜에 보내고.
- 간단히, 5 ㎕의 변성 버퍼로 eccDNA 풍부한 5 μl를 섞는다.
- RT에서 3 분 후, 10 ㎕의 중화 버퍼를 추가합니다. 부드럽게 믹스 29 μl의 반응 버퍼와 1 μl를 ø29 DNA 중합 효소를 포함하는 30 μl의 마스터 믹스를 추가합니다. (72 시간까지) 16 시간 이상 30 ° C에서 반응을 품어. 열은 3 분 동안 65 ° C에서 ø29의 DNA 폴리머 라제를 불 활성화.
5. 시퀀싱 및 데이터 분석
- 300 bp의 평균 목표 최대 크기 집속 초음파로 증폭 eccDNA 전단. 130 ㎕의 DNA 샘플에 대해 다음 설정을 사용하여 450W 피크 강도 전력, 60 초 처리, 30 %의 듀티 계수, 버스트 당 200 사이클, 온도 7 ℃로한다.
- 조각난 라이브러리 준비를위한 적절한 방법을 사용하여 시퀀싱 라이브러리의 합성 읽기에 바코드 인덱스 라벨 및 어댑터를 추가합니다.
- 141 - 염기 단일 엔드는 높은 처리량 시퀀싱 플랫폼에서 읽을 때, 예를 들어, 깊은 순서를 실행합니다.
- 지도 조사중인 효모 참조 게놈을 읽고 수 있도록 여러 영역에 매핑 읽습니다. 예를 들어, 자유롭게 사용할 워크 플로우 시스템 27, 28 및 짧은 읽기 정렬 매핑 소프트웨어 (29)를 사용합니다.
- 확인하고 간격 (20)없이, 예를 들어, 7 개 이상의 연속 읽기 읽기 연속 (> 1킬로바이트)를 사용하여 추정 eccDNAs 지역에서 읽습니다.
참고 : 소프트웨어를 사용할 수있는 매핑 탐험 27,28 관심의 게놈 영역에서 읽습니다.
결과
서클-SEQ 방법, 세 S.의 유효성을 검사하려면 세포 10 세대에 YPD에서 개별적으로 재배 된 후 1 × 10 10 세포의 cerevisiae의 CEN.PK 인구가 상영되었다. 이전 20 (데이터 미도시) 한 바와 같이 염색체 DNA 선형 제거는 ACT1 qPCR의 신호의 유무에 의해 확인 하였다. 정제 및 농축 eccDNA가 6800 만까지 서열화 된 읽기 (141 뉴클레오티드 단일 엔드 읽기)과 CEN.PK113-7D 참조 게놈 (버전 2012년 6월 19일)에 매핑. C1, C2와 C4라는 3 개의 시료에서 추정 eccDNAs의 녹음이 읽고 더 이상 1킬로바이트 이상 연속으로 매핑 게놈 영역에 할당되었다. 10,000 몬테카를로 시뮬레이션에 기초하여, 연속하여 매핑 된 각 영역의 중요성 1킬로바이트 추정 된 것보다 긴 판독한다. 이 79, 159, 56 지역은 가능성이 eccDNA 시퀀스 (p <0.1, 데이터 집합 1)로 주석되었다에서. 녹음 contiguo의 수미국> 1킬로바이트 샘플이 상기 (도 2) 서열 되었다면 더욱 eccDNA 요소가 기록되어있는 것이 시사 시퀀스 깊이의 함수로서 증가 읽는다. 예상 된 바와 같이, 원형 서열 방법은 다수의 염색체 XII의 2μ 플라스미드를 비롯한 공지 원형 DNA 요소 미토콘드리아 DNA, 리보솜 RNA 유전자의 수로부터 판독 추출한 샘플로 스파이크 된 3 개의 내부 제어 플라스미드는 pBR322, pUC19를하고 pUG72 단지 열 정화 (그림 3) 전.
동영상은 연속의 예는 염색체 IV에 HXT7 _ARS432_ HXT6 궤적에 매핑 된 것을 읽 보여줍니다. 이전에는 [HXT6 / 7 원]를 열 S288c 인구 (1 × 10 10 세포와 각)과 원형 DNA 구조에서 원 - 서열에 의해 발견 된이 역 PCR 분석 (20)에 의해 확인되었다. 에서 [HXT6 / 7 CIrcle] 또한 세 CEN.PK 인구 (그림 4A)의 각각 기록했다. 또한, CEN.PK의 복제 샘플 중 일반 eccDNA 유전자의 대부분은 S288c 데이터 세트 (그림 4B)에서 eccDNA 유전자를 중첩.
원형 DNA 정제, 30 μg의 게놈 DNA를 가진 두 개의 샘플 각각에 대한 원-SEQ 프로토콜의 특이성을 테스트하려면 시험 하였다. 하나의 샘플은 원 - 서열 프로토콜에 의해 정제 하였다 두 샘플에서 100 ng의 플라스미드 DNA와 eccDNA 보충했다. 컬럼 분리 한 후, DNA 수율은 플라스미드 (GD)과 플라스미드와 샘플 1.60 % (480 NG) (GD + P)없이 샘플 1.27 % (380 NG를)였다. 엑소 뉴 클레아 제 처리의 효율성. 29 시간 및 ACT1 대해 PCR을 사용하여 72 시간 후에 선형 DNA 함량에 대해 시험 된 어떠한 샘플 (데이타 미기재) ACT1 증폭되지 함유 하였다. 각각의 엑소 뉴 클레아 제 처리 된 샘플의 일부를 추가로했다ø29에 의해 mplified 효소 및 효소 반응의 생성물은 요오드화 프로피 듐 염색 (도 5A-F) 및 아가 로스 겔 전기 영동 (도 5G)로 분석 하였다. 엑소 뉴 클레아 제 처리 후 샘플을 최소한 프로피 듐 요오드 염색 (도 5A-B)를 나타냈다. ø29 - 만 게놈 DNA를 증폭 샘플 컨트롤 샘플 (그림 5E)와 유사한 스레드 같은 구조 (그림 5C)를 밝혔다. ø29 - 추가 플라스미드를했다 증폭 된 샘플은 플라스미드 제어 (그림 5 층) 닮은 초점 (그림 5D)를 밝혔다. 이미지는 ø29 폴리머 선형 DNA를 통해 원형 DNA에 대한 풍부한 것으로 나타났다. 대부분의 선형 염색체 DNA는 29 시간의 엑소 뉴 클레아 제 처리 (그림 5A-B, G) 후 샘플에서 제거되었습니다. 그러나 광범위한 엑소 뉴 클레아의 100 개 이상의 시간 동안 처리 및 사용하여 100 개 이상의 단위이었다 증폭 된 샘플 여전히 72 시간의 엑소 뉴 클레아 제 처리 (그림 5C-D) 후 스레드 같은 구조의 배경을 보여 주었다 - ø29 같이, 모든 염색체 선형 DNA를 제거 할 필요가 있었다.
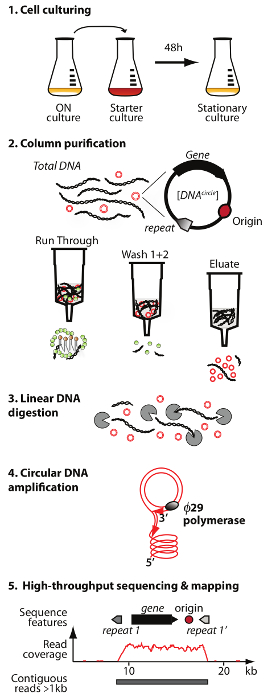
. 컬럼 크로마토 그래피의 용출액 분획 선형 염색체 DNA 잔존 3) 소화 4) 증폭 1) 세포 배양, 2) 정제 eccDNA의 농축 : 서클-SEQ 방법 그림 1. 개요 프로토콜은 5 단계를 가진다 ø29 DNA 중합 효소, 그리고 고농축 eccDNA 5) 시퀀싱 및 매핑에 의해 DNA는 S.을 읽고 cerevisiae의 참조 게놈. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
p_upload / 54239 / 54239fig2.jpg "/>
그림 2. 연속 시퀀스 깊이의 함수로> 1킬로바이트을 읽습니다. EccDNA 1 × 10 10 세포 (매핑 읽기의 수백만) 순서 깊이의 함수로 증가에서. 도시 : 반수체 CEN.PK S.에서 생물학적 삼중 10 10 세포 분열에 의해 분리 cerevisiae의 인구 (C1, C2, C4). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
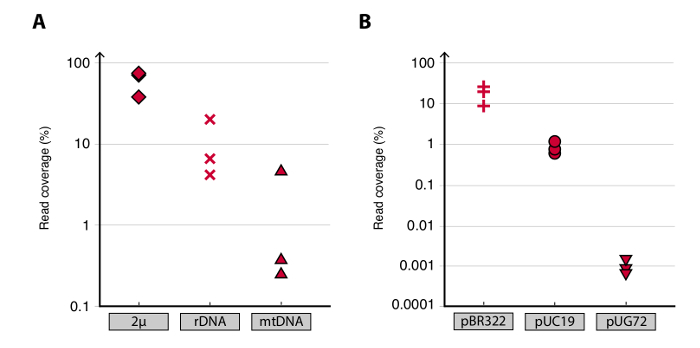
알려진 원형 DNA 요소의 그림 3. 감지. (AB)이 CEN.PK 생물의 플라스미드 백분율로 읽기 범위의 플롯을 (밀도를 읽기) 분산 형은 C1, C2와 C4를 복제합니다. 했다 매핑 (A)는 내생 효모 플라스미드를 읽 2μ을; [rDNA의 원(염색체 XII에서 리보솜 RNA 유전자); 및 미토콘드리아 DNA (미토콘드리아 DNA). (B) 플라스미드는 고유 제어 매핑 읽는다. 제어 플라스미드 칼럼 정제 전에 샘플로 스파이크 하였다. 셀 당 플라스미드 비율이었다 : 나 pBR322 (더하기 기호) 1 : 1, pUC19를 (원) 1시 50분 및 pUG72 (삼각형) 1 : 2500.
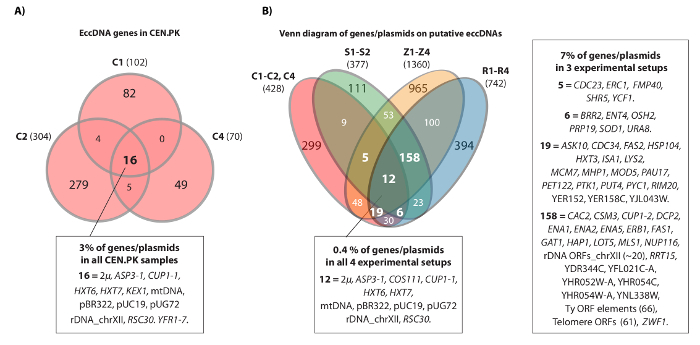
CEN.PK와 S288c 그림 4. 일반 eccDNA 요소입니다. (A) 벤 다이어그램 (476) 사이에 중첩 표시 세 CEN.PK 샘플 (C1, C2, C4)에 294 eccDNA 요소에 유전자. 16 공통 중복 eccDNA 유전자 / 플라스미드 (모든 유전자 이름이 데이터 집합 1에) 주석이 있습니다. S1-S2, R1-R4 : 10 S288c 샘플들로부터 추정 eccDNAs에 기록 된 모든 유전자에 비해 (B) 세 CEN.PK 샘플 (C1, C2, C4)에서 추정 eccDNAs에 기록 된 모든 유전자의 벤 다이어그램,, Z1-Z4는 (참고 20 참조). 유전자 / 플라스미드 및 2 변형 배경과 중 3 인 이상 실험 설정의 최소 중복 추정 eccDNA 지역 13 생물학적 복제 (S1-S2, R1-R4, Z1-Z4, C1-C3)가 표시됩니다. C 샘플, CEN.PK; R과 Z 샘플, S288c BY4741; S 샘플, S288c M3750. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
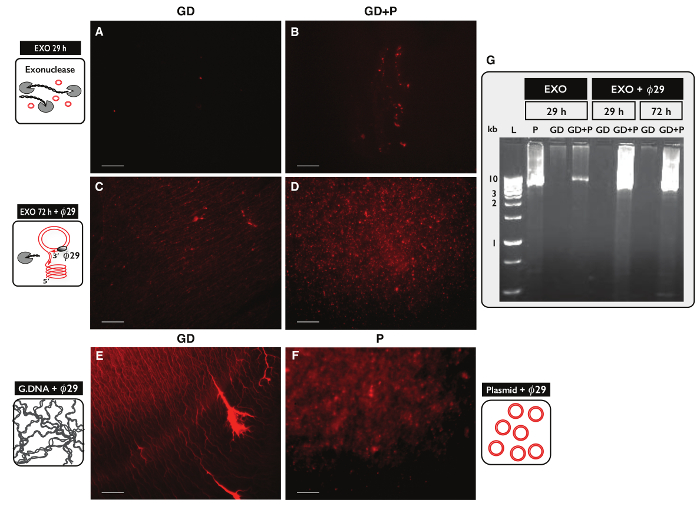
도 엑소 뉴 클레아 후 DNA 시료 5. 시각화 및 ø이 치료 구. DNA의 (AF) 요오드화 프로피 듐 염색. 스케일 바, 10 μm의. (A, C 및 E) 게놈 DNA와 시료 (GD); (B와 D) GD 플러스 플라스미드 (GD + P)와 샘플. ( 29 시간의 엑소 뉴 클레아 제 처리 후> AB) (EXO 29 시간) 옹; ø29 중합 효소 증폭 한 다음 72 시간의 엑소 뉴 클레아 제 처리 후 (CD) (EXO 72 시간 + ø29). (E) E 후 게놈 DNA 제어 : ø29 폴리머 증폭; (F) 플라스미드 제어 (5.5 KB) 후 29 중합 효소 증폭; (G의 ø) 아가로 오스 겔 eletrophoresis. L, 1킬로바이트 마커; 왼쪽부터 P, EXO 29 시간 후 플라스미드 제어 (5.5 킬로바이트) GD, EXO 29 시간 후 (A에서와 같이 샘플); GD + P, EXO 29 시간 후 (B로 샘플); GD와 GD + P, EXO 후 29 시간 + ø29; GD와 GD + P, EXO 후 72 시간 + ø29 (CD에서와 같이 샘플). 표 S1보기 추가 자세한 내용은. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
ASET 1 "SRC ="/ 파일 / ftp_upload / 54239 / 54239dataset1.jpg "/>
CEN.PK.에서 데이터 집합 1. 잠재적 인 DNA의 회람 영역 이 파일을 다운로드하려면 여기를 클릭하십시오.
시퀀스 데이터는 제시된 348 영역에 대해 분석한다. 열 AD, eccDNA 매핑입니다. 추정 eccDNA이 확인되었다되는, 샘플 A (왼쪽에서 첫 번째 열); B, 염색체; CD는 시작과 끝은 추정 eccDNAs의 좌표. EH, eccDNA 내용. E, 자율적 지역 (ARS) 시퀀스를 복제; F,이 지역의 전체 유전자; G, 지역에 포함 된 유전자의 일부; H 유전자를 BLASTN은-확인했다. IO, EccDNA 범위 및 p- 값. I, BP의 고유 주석 순서에 긴 영역; J, 모든 수 읽기 맵핑; K는, 모든 매핑의 범위는 매핑 만 읽고 (FPKM)에서 킬로바이트 당 조각으로 읽 몬테카를로 시뮬레이션에서 우연히 발생에 비해 추정 eccDNA에 대한 L, p 값이; 유엔의 M, 수iquely 읽어 맵핑; 아니; K와 L은 고유하게 매핑 사용으로하면 (UFPKM) 읽습니다. 의 매핑을위한 매개 변수 읽고 20 설명 된대로 몬테 카를로 시뮬레이션이었다.
토론
서클-SEQ 방법은 시퀀스 레벨 해상도 효모 세포 eccDNA 게놈 크기의 검출을 허용한다. 상기 방법은 집중 소용돌이 또는 피펫을 필요로하고, 다음 단계에서 엑소 뉴 클레아 제 소화에 이어질 것이다 eccDNA 파괴를 제한하는 중력 컬럼 분리를 사용하지 않는 온화한 eccDNA 정제이다. 방법의 이러한 기능은 유전자 서열을 포함하는 큰 eccDNAs을 검출하기위한 중요 할 수있다. 원 - 서열 전체 유전자 (데이터 집합 1) 등 다양한 eccDNAs를 발견했습니다. 또한 86 킬로바이트 효모 미토콘드리아 DNA를 발견했습니다. 따라서,이 프로토콜은 대형 원형 DNA 요소의 정화를 용이하게한다. 최소 DNA 추출 단계의 수를 유지하는 eccDNA 손실 위험을 감소시키고 수율을 최대화한다. 제어 결과에 기초하여, 아군 된 플라스미드, 원형 서열이 2500 세포로부터 단일 원형 DNA를 검출하는 매우 민감하다. 또한, 이러한 2μ 같은 풍부한 내인성 플라스미드를 제거; 플라스미드 또는 미토콘드리아 DNA는 크게 감도를 향상시킬 수 있습니다. 효모 문화 2μ의 경화 (30)을 설명 하였다. 또한, 2μ 및 미토콘드리아 DNA의 제거는 SwaI 같은 희귀 절단 효소, 달성 될 수 있습니다. 그러나, 제한 효소 공정은 다른 관심 eccDNAs 타겟팅 및 eccDNA 총 생산량을 제한 할 수있다.
eccDNA 검출을위한 중요한 단계는 적절한 깊이로 선형 DNA (3 단계) 및 DNA 시퀀싱 (5 단계)을 제거했다. 셀 인구에서 eccDNAs의 대부분을 기록하려면, 깊은 시퀀싱 (20)를해야 할 수도 있습니다. 원형 DNA 접합이 페어 엔드를 얻을 것으로 예상 discordantly 그지도를 읽고대로 쌍 엔드 시퀀싱, eccDNA 검출의 더 큰 신뢰를 제공해야합니다. 이러한 불일치는 원형 DNA 구조의 검색을 지원 잠재적 추가 eccDNA 검출 필터로서 사용된다.
서클 - 괜찮다Q 방법은 3 개의 독립적 인 S.를 이용하여 검증 하였다 cerevisiae의 CEN.PK 인구. 감지 서열은 이전에 eccDNAs, 내생 플라스미드를보고 아군 된 플라스미드 및 추정 eccDNAs (데이터 집합 1) 수백 포함되어 있습니다. 이러한 연구 결과는 S.에서 이전 원 - 서열 데이터 세트를 지원 cerevisiae의 S288c 20. CEN.PK 및 S288c 집단에 공통적 인 몇 가지 eccDNAs의 발견은 이러한 궤적이 같은 원형 요소 (그림 4)이 존재하는 성향이 있음을 나타냅니다. 우리는 이전에 다른 변형 배경에 [GAP1 원]의 증거가 발견되지 않았습니다 불구하고 [GAP1 원이]를 CEN.PK 배경 8 질소 제한된 조건에서 농축되는 것으로 나타났습니다. S288c 및 CEN.PK 모두에서 CUP1-1 RSC30, ASP3-1, COS111 및 HXT6 HXT7 궤적에서 eccDNA의 발견은 DNA의 회람에 대한 경향은 사기꾼입니다 제안효모 균주 사이에 봉사했다. [HXT6 / 7 원], [ASP3-1 원], [COS111 원] 및 [CUP1-1 RSC30 원]을 세포 또는 경우에 선택적 이점을 부여하는 경우 그것은 그들의 존재는 단지 높은 비율의 효과가 나타 남아 DNA의 회람.
종합하면, 결과는 원형 서열이 킬로 크기 eccDNAs 검출에 적합하고 완전한 유전자 eccDNAs를 식별하는 장점을 가지고 있음을 나타낸다. 원 - 서열은 효모에서 eccDNAs의 전체 게놈 규모의 스크린을 가능하게하는 매우 중요한 방법이다. 서클-SEQ 방법은, 유전자 증폭 및 삭제를 생성 eccDNA의 역할을 해명을 목표로 연구의 새로운 필드를 열 수있다. DNA 구조와 구조가 크게 높은 진핵 생물에 진핵 효모에서 보존되는 점을 감안, 서클-SEQ 방법은 원칙적으로 상 해당해야한다약간의 수정과 함께, 모든 진핵 세포에 전자. 현재 방법은 megabase 크기 eccDNAs를 정화하는 능력이 아직 표시하도록했지만, 어떤 제한이 나타나지 않는다. 또한, 롤링 써클 증폭 방법 (31)을 사용 ø29 DNA 중합 효소의 사용, eccDNA 정량보다 어렵게 작은 eccDNAs쪽으로의 바이어스를 생성한다. 원형 서열 인간 체세포에서 두 분 원형 DNA에 대한 연구에 적합하다, 전체 유전자를 운반하기에 충분히 큰 eccDNAs를 검출한다. 프로토 암 유전자가 이러한 요소 32-37에서 증폭 할 때 두 분은 암에 기여할 수있다. 생식선 세포 eccDNAs의 연구는 생식 계열 돌연변이 비율을 측정하고, 가축, 예를 들어, 정자의 품질을 평가하는데 사용될 수있다. 따라서, 원형 서열이 유전자 저작권법과 관련된 질환의 새로운 이해 유전 적 변이는 카피 수 변화의 형태로 발생하는 속도에 대한 통찰력을 수득하고 유도 할 가능성이있다수 변동 38-40.
공개
The authors declare that they have no competing financial interests.
감사의 말
Thanks to Kenn D. Møller and Claus Sternberg (DTU) for technical assistance and to Tue S. Jørgensen for quantitative PCR analysis.
자료
| Name | Company | Catalog Number | Comments |
| Bacto peptone | BD Difco | 211677 | Alternative product can be used. |
| Brilliant III SYBR Green PCR Master Mix | Agilent Technologies | 600882 | For qPCR analysis. Alternative product can be used. |
| Dextrose (D-glucose) | Carl Roth | HN06.4 | Alternative product can be used. |
| Disruptor Beads, 0.5 mm | Scientific Industries, Inc. | SI-BG05 | Glass beads to disrupt plasma cell membranes. Alternative product can be used. |
| Ethidium bromide | Carl Roth | 2218.2 | Agarose gel stain for detecting DNA/RNA. |
| GeneJet plasmid miniprep kit | Thermo Fisher | K0502 | Plasmid purifcation from bacteria. Alternative product can be used |
| NotI, FastDigest | Life Technologies - Thermo Fisher Scientific, USA | FD0594 | Endonuclease. Alternative product can be used. |
| Plasmid Mini AX kit | A&A Biotechnology, Poland | 010-50 | Plasmid purifcation kit used to purify eccDNA. |
| Plasmid-Safe ATP-dependent DNase kit | Epicentre, USA | E3105K | ATP-dependent exonuclease kit. Alternative product can be used. |
| Propidium iodide | Sigma-Aldrich, USA | 81845 | Alternative product can be used. |
| pUG6 plasmid | EUROSCARF, Germany | P30114 | Marker gene: loxP-PAgTEF1-kanMX-TAgTEF1-loxP. Plasmid requests: Please contact Dr. Peter Philippsen@unibas.ch |
| QIAGEN genomic-tip 100/G | Qiagen, USA | 13343 | Genomic DNA purifcation from yeast. Alternative product can be used. |
| REPLI-g Mini Kit protocol | Qiagen, USA | 150023 | Amplification of eccDNA by the phi29 polymerase |
| Yeast extract | BD Difco | 210929 | Alternative product can be used. |
| Zymolyase 100T (Lyticase, Yeast Lytic Enzyme) | Nordic BioSite, Sweden | Z1004-3 | Alternative product can be used. |
| Data access to sequence files | European Nucleotide Archive | EccDNA dataset from Saccharomyces cerevisiae CEN.PK113-7D. Study accession number PRJEB9684. 2nd accession number is ERP010820. Locus tag prefix is BN2032. | |
| Strains | |||
| Saccharomyces cerevisiae CEN.PK113-7D | Genotype MATa MAL2-8c SUC2 | ||
| Saccharomyces cerevisiae yeast deletion library pool | EUROSCARF, Germany | S288c BY4741 pool of 4400 viable single-gene deletion mutants disrubted by KanMX module. Genotypes MATa his3∆1 leu2∆0 met15∆0 ura3∆0 genexxx::KanMX. | |
| Equipments | |||
| DNA Spectrophotometer | NanoDrop 1000 Spectrophotometer, Thermo Fisher | Measuring DNA concentration. Alternative product can be used. | |
| Fluorescence microscopy | Nikon Optronics Magnafire. Red excitation fluorescence filter, 663-738 nm. | Alternative product can be used. | |
| Robotic library-build system | Apollo 324, IntegenX Inc. | DNA library preparation. Alternative product can be used. | |
| Sequencing platform | Illumina HiSeq 2000 platform, Illumina Inc. | DNA sequencing. Alternative product can be used. | |
| Ultrasonicator | Covaris LE220, microTUBE AFA Fiber tubes | Alternative product can be used. | |
| Methods | |||
| 2% YPD media | Mix 10 g Dextrose, 10 g Yeast extract, 20 g Bacto peptone and add H2O to a total volume of 1000 ml and autoclave. | ||
| Circle-Seq test on genomic DNA | Genomic DNA was purified (Qiagen) from a pool of the yeast deletion library (Euroscarf). The DNA concentration was measured by nanodrop and 30 µg genomic DNA was pipetted into two micro centrifuge tubes. One micro centrifuge tube was supplemented with 100 nanogram plasmid (pUG6). The DNA samples were purified by Circle-Seq, omitting the protocol steps 1.1-1.3 and 1.5-1.7. The eluted DNA concentrations were measured by nanodrop and the entire DNA yield from sample GD and GD+P was treated with exonuclease for a period of 29 hours. A 10% fraction was collected for phi29-amplification and PCR analysis, while the remaining DNA was subjected to 72 hour exonuclease treatment. The samples were analyzed for linear DNA content by PCR, using the ACT1 gene as chromosomal marker. A 5% fraction of each of the exonuclease treated samples was amplified by the phi29 DNA polymerase for 16 hours (Qiagen). The presence of DNA in each sample was examined by loading an equal amount (7 µl) in wells on an 0.5 µg/ml ethidium-bromide 0.9% agarose gel after running gel-electrophoresis. | ||
| Mapping software | Bowtie2 aligner, John Hopkins University | Ultrafast short read alignment. Reference: Langmead B, Salzberg S. Fast gapped-read alignment with Bowtie 2. Nature Methods. 2012, 9:357-359. | |
| Propidium iodide stain | Images of propidium iodine stained DNA were captured by fluorescence microscopy at 100x magnification (100x/1.30 oil, Nikon) in the RFP channel (red excitation fluorescence filter, 663-738 nm) using identical exposition time (5 seconds). | ||
| Workflow bioinformatic system | Galaxy, Open source. | A free web-based platform for data intensive biomedical research. References: Goecks, J, Nekrutenko, A, Taylor, J and The Galaxy Team. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biol. 2010 Aug 25;11(8):R86. Blankenberg D, Von Kuster G, Coraor N, Ananda G, Lazarus R, Mangan M, Nekrutenko A, Taylor J. "Galaxy: a web-based genome analysis tool for experimentalists". Current Protocols in Molecular Biology. 2010 Jan; Chapter 19:Unit 19.10.1-21. Giardine B, Riemer C, Hardison RC, Burhans R, Elnitski L, Shah P, Zhang Y, Blankenberg D, Albert I, Taylor J, Miller W, Kent WJ, Nekrutenko A. "Galaxy: a platform for interactive large-scale genome analysis." Genome Research. 2005 Oct; 15(10):1451-5. | |
참고문헌
- Kugelberg, E., Kofoid, E., et al. The Tandem Inversion Duplication in Salmonella enterica.: Selection Drives Unstable Precursors to Final Mutation Types. Genetics. 185 (1), 65-80 (2010).
- Reams, A. B., Kofoid, E., Savageau, M., Roth, J. R. Duplication Frequency in a Population of Salmonella enterica. Rapidly Approaches Steady State With or Without Recombination. Genetics. 184 (4), 1077-1094 (2010).
- Smith, C. A., Vinograd, J. Small polydisperse circular DNA of HeLa cells. Journal of Molecular Biology. 69 (2), 163-178 (1972).
- Carroll, S. M., DeRose, M. L., et al. Double Minute Chromosomes Can Be Produced from Precursors Derived from a Chromosomal Deletion. Molecular and cellular biology. 8 (4), 1525-1533 (1988).
- Cohen, S., Yacobi, K., Segal, D. Extrachromosomal Circular DNA of Tandemly Repeated Genomic Sequences in Drosophila. Genome research. 13 (6A), 1133-1145 (2003).
- Horowitz, H., Haber, J. E. Identification of Autonomously Replicating Circular Subtelomeric Y' Elements in Saccharomyces cerevisiae. Molecular and cellular biology. 5 (9), 2369-2380 (1985).
- Moore, I. K., Martin, M. P., Dorsey, M. J., Paquin, C. E. Formation of Circular Amplifications in Saccharomyces cerevisiae by a Breakage-Fusion-Bridge Mechanism. Environmental and molecular mutagenesis. 36 (2), 113-120 (2000).
- Gresham, D., Usaite, R., Germann, S. M., Lisby, M., Botstein, D., Regenberg, B. Adaptation to diverse nitrogen-limited environments by deletion or extrachromosomal element formation of the GAP1 locus. Proceedings of the National Academy of Sciences of the United States of America. 107 (43), 18551-18556 (2010).
- Windle, B., Draper, B. W., Yin, Y. X., O'Gorman, S., Wahl, G. M. A central role for chromosome breakage in gene amplification, deletion formation, and amplicon integration. Genes & development. 5 (2), 160-174 (1991).
- Gresham, D., Ruderfer, D. M., et al. Genome-Wide Detection of Polymorphisms at Nucleotide Resolution with a Single DNA Microarray. Science. 311 (5769), 1932-1936 (2006).
- Kidd, J. M., Cooper, G. M., et al. Mapping and sequencing of structural variation from eight human genomes. Nature. 453 (7191), 56-64 (2008).
- Gresham, D., Desai, M. M., Botstein, D., Dunham, M. J. The Repertoire and Dynamics of Evolutionary Adaptations to Controlled Nutrient-Limited Environments in Yeast. PLoS Genetics. 4 (12), 1-19 (2008).
- Lang, G. I., Botstein, D., Desai, M. M. Genetic Variation and the Fate of Beneficial Mutations in Asexual Populations. Genetics. 188 (3), 647-661 (2011).
- van Loon, N., Miller, D., Murnane, J. P. Formation of extrachromosomal circular DNA in HeLa cells by nonhomologous recombination. Nucleic Acids Research. 22 (13), 2447-2452 (1994).
- Vinograd, J., Lebowitz, J. Physical and Topological Properties of Circular Dna. Journal of General Physiology. 49 (6P2), 103 (1966).
- Shibata, Y., Kumar, P., et al. Extrachromosomal MicroDNAs and Chromosomal Microdeletions in Normal Tissues. Science. 336 (6077), 82-86 (2012).
- Dillon, L. W., Kumar, P., et al. Production of Extrachromosomal MicroDNAs Is Linked to Mismatch Repair Pathways and Transcriptional Activity. Cell Reports. 11 (11), 1749-1759 (2015).
- Li, L. L., Norman, A., Hansen, L. H., Sørensen, S. J. Metamobilomics - our knowledge on the pool of plasmid encoded traits in natural environments using high-throughput sequencing. Clinical microbiology and infection : the official publication of the European Society of Clinical Microbiology and Infectious Diseases. 18, 5-7 (2012).
- Brown Kav, A., Sasson, G., Jami, E., Doron-Faigenboim, A., Benhar, I., Mizrahi, I. Insights into the bovine rumen plasmidome. Proceedings of the National Academy of Sciences of the United States of America. 109 (14), 5452-5457 (2012).
- Møller, H. D., Parsons, L., Jørgensen, T. S., Botstein, D., Regenberg, B. Extrachromosomal circular DNA is common in yeast. Proceedings of the National Academy of Sciences of the United States of America. , 201508825 (2015).
- Drubin, D. G., Miller, K. G., Botstein, D. Yeast Actin-Binding Proteins - Evidence for a Role in Morphogenesis. The Journal of cell biology. 107 (6), 2551-2561 (1988).
- Magdolen, V., Drubin, D. G., Mages, G., Bandlow, W. High levels of profilin suppress the lethality caused by overproduction of actin in yeast cells. FEBS letters. 316 (1), 41-47 (1993).
- Sandrock, T. M., Brower, S. M., Toenjes, K. A., Adams, A. Suppressor analysis of fimbrin (Sac6p) overexpression in yeast. Genetics. 151 (4), 1287-1297 (1999).
- Blanco, L., Bernad, A., Lázaro, J. M., Martìn, G., Garmendia, C., Salas, M. Highly Efficient DNA Synthesis by the Phage ø29 DNA Polymerase. The Journal of biological chemistry. 264 (15), 8935-8940 (1989).
- Dean, F. B. Rapid Amplification of Plasmid and Phage DNA Using Phi29 DNA Polymerase and Multiply-Primed Rolling Circle Amplification. Genome research. 11 (6), 1095-1099 (2001).
- Hutchison, C. A., Smith, H. O., Pfannkoch, C., Venter, J. C. Cell-free cloning using ø29 DNA polymerase. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17332-17336 (2005).
- Goecks, J., Nekrutenko, A., Taylor, J., Galaxy Team, T. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biology. 11 (8), 86 (2010).
- Giardine, B., Riemer, C., et al. Galaxy: A platform for interactive large-scale genome analysis. Genome research. 15 (10), 1451-1455 (2005).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357 (2012).
- Tsalik, E. L., Gartenberg, M. R. Curing Saccharomyces cerevisiae of the 2 micron plasmid by targeted DNA damage. Yeast. 14 (9), 847-852 (1998).
- Norman, A., Riber, L., Luo, W., Li, L. L., Hansen, L. H., Sørensen, S. J. An Improved Method for Including Upper Size Range Plasmids in Metamobilomes. PLoS ONE. 9 (8), e104405 (2014).
- Storlazzi, C. T., Lonoce, A., et al. Gene amplification as double minutes or homogeneously staining regions in solid tumors: Origin and structure. Genome research. 20 (9), 1198-1206 (2010).
- Von Hoff, D. D., Needham-VanDevanter, D. R., Yucel, J., Windle, B. E., Wahl, G. M. Amplified human MYC localized to replicating submicroscopic circular DNA molecules. Proceedings of the National Academy of Sciences of the United States of America. 85 (13), 4804-4808 (1988).
- Raymond, E., Faivre, S., et al. Effects of hydroxyurea on extrachromosomal DNA in patients with advanced ovarian carcinomas. Clinical cancer research : an official journal of the American Association for Cancer Research. 7 (5), 1171-1180 (2001).
- Shimizu, N. Extrachromosomal Double Minutes and Chromosomal Homogeneously Staining Regions as Probes for Chromosome Research. Cytogenetic and genome research. 124 (3-4), 3-4 (2009).
- Eckhardt, S. G., Dai, A., Davidson, K. K., Forseth, B. J., Wahl, G. M., Von Hoff, D. D. Induction of differentiation in HL60 cells by the reduction of extrachromosomally amplified c-myc. Proceedings of the National Academy of Sciences of the United States of America. 91 (14), 6674-6678 (1994).
- Vogt, N., Lefèvre, S. -. H., et al. Molecular structure of double-minute chromosomes bearing amplified copies of the epidermal growth factor receptor gene in gliomas. Proceedings of the National Academy of Sciences of the United States of America. 101 (31), 11368-11373 (2004).
- Ahn, K., Gotay, N., et al. High rate of disease-related copy number variations in childhood onset schizophrenia. Molecular psychiatry. 19 (5), 568-572 (2013).
- Girirajan, S., Johnson, R. L., et al. Global increases in both common and rare copy number load associated with autism. Human molecular genetics. 22 (14), 2870-2880 (2013).
- Vogt, N., Gibaud, A., Lemoine, F., de la Grange, P., Debatisse, M., Malfoy, B. Amplicon rearrangements during the extrachromosomal and intrachromosomal amplification process in a glioma. Nucleic Acids Research. , (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유