Method Article
Ion Semiconductor Sequencing Platform을 기반으로 한 위암의 Multi-gene Single Nucleotide Polymorphism 검출
Erratum Notice
요약
이 프로토콜은 이온 반도체 염기서열 분석 플랫폼을 기반으로 위암 샘플에서 단일 뉴클레오티드 다형성을 검출하는 데 필요한 전체적인 실험실 절차를 제안합니다. target sequences, ligating adapters, library amplification and purification, 품질 관리 기준도 자세히 설명되어 있습니다.
초록
위암은 흔한 이질성 종양입니다. 대부분의 환자는 진단 시점에 위암이 진행된 상태이며 종종 화학 요법이 필요합니다. 5-플루오로우라실(5-FU)이 치료에 널리 사용되지만, 치료 민감도와 약물 내성을 결정해야 하므로 개별화된 투여의 중요성이 강조됩니다. 약물유전학은 개별화된 치료의 임상적 구현을 안내할 수 있습니다. 단일 뉴클레오티드 다형성(SNP)은 유전적 마커로서 적절한 화학요법 요법 및 투여량의 선택에 기여합니다. 일부 SNP는 5-FU의 치료 목표인 엽산 대사와 관련이 있습니다. 메틸렌테트라하이드로폴레이트 환원효소 (MTHFR) rs1801131 및 rs1801133, 디하이드로폴레이트 환원효소 (DHFR) rs1650697 및 rs442767, 메티오닌 합성효소 (MTR) rs1805087, 감마-글루타밀 가수분해 효소(GGH) rs11545078 및 용질 담체 family 19 member 1 (SLC19A1) rs1051298은 다양한 종류의 암 및 항엽산 항종양제에서 조사되었으며, 이는 잠재적인 예측 및 안내 중요성을 가지고 있습니다. 5-FU의 적용. 이온 토렌트 차세대 반도체 염기서열 분석 기술은 위암 관련 SNP를 빠르게 검출할 수 있습니다. 염기가 DNA 사슬에서 확장될 때마다 H+ 가 방출되어 국부적인 pH 변화를 일으킵니다. 이온 센서는 pH 변화를 감지하고 화학 신호를 디지털 신호로 변환하여 합성에 의한 염기서열분석을 달성합니다. 이 기술은 샘플 요구 사항이 낮고 조작이 간단하며 비용이 저렴하고 염기서열 분석 속도가 빠르기 때문에 SNP에 의한 개별화된 화학 요법을 안내하는 데 유용합니다.
서문
위암은 전 세계 공중 보건 분야에서 큰 부담입니다. 국제암연구소(IARC)가 발표한 2020년 세계 암 통계(Global Cancer Statistics 2020)에 따르면 위암은 5번째로 많이 진단되는 암이자 암 관련 사망의 4번째 주요 원인입니다. 전 세계적으로 동아시아의 연령표준화율 발생률은 남성과 여성 모두에서 가장 높다1. 위암의 발생은 잠복성이며, 이는 환자가 초기에 명확하고 구체적인 증상이 없는 경우가 많다는 것을 의미합니다. 모든 위암 환자 중 정기 검진이 없는 국가에서는 환자의 80%-90%가 종양이 수술할 수 없는 진행된 단계에서 진단되거나 수술 후 5년 이내에 재발한다2.
진행성 또는 전이성 위암의 경우 항암화학요법이 주요 치료법으로 환자의 생존율과 삶의 질을 향상시킬 수 있습니다. 전이성 위암 환자의 초기 치료에는 백금-플루오로피리미딘 요법이 1차 화학요법의 주요 선택이다3. 플루오로피리미딘은 주로 5-플루오로우라실(5-FU)과 카페시타빈, 테가푸르와 같은 경구용 플루오로피리미딘 유도체를 포함한다. 5-FU의 주요 표적은 DNA 합성을 억제하고 종양 조직의 성장을 늦추는 엽산 대사 관련 효소입니다. 약물 부작용은 설사, 점막염, 골수억제, 수족 증후군과 함께 그 효용성을 제한하며, 가장 빈번한 부작용 중 하나입니다. 치료 반응과 약물 부작용은 엽산 대사 경로의 요인과 밀접한 관련이 있는 것으로 보고되었습니다. 특히, rs1801131의 동형접합 돌연변이는 수족 증후군의 지표로 확인되었습니다(p =4.1 x 10-6, OR=9.99, 95% CI: 3.84-27.8)4. 플루오로피리미딘은 항암 화학요법에 광범위하게 사용되지만, 화학내성은 흔한 응급 상황이며, 이는 위암 치료에서 치료 실패를 초래합니다. 예를 들어, 5-FU로 치료받은 진행성 대장암 환자의 전체 반응률은10%-15%에 불과합니다. 또한 플루오로피리미딘은 무시할 수 없는 독성을 가지고 있습니다. 5-FU에 의해 유발되는 독성 반응은 주로 설사, 수족 증후군, 구내염, 호중구 감소증, 혈소판 감소증, 신경 독성 및 심지어 사망을 포함한다6. 플루오로피리미딘으로 치료받은 환자의 10%-30%에서 심각한 치료 관련 독성이 발생하고, 이 중 0.5%-1%에서 치명적인 독성이 발생한다7.
진행성 위암 환자를 대상으로 한 삶의 질 연구에 따르면 5-FU 기반 화학요법을 받은 환자의 반응률은 50% 미만이었다8. 따라서 5-FU 기반 화학요법의 민감도와 관련된 요인을 이해하는 것은 독성을 최소화하면서 반응률과 효과를 극대화하기 위한 정확한 치료를 위해 특히 중요합니다. 5-FU가 엽산 대사와 밀접한 관련이 있다는 점을 고려할 때, 엽산 대사 경로에 있는 효소의 유전적 변이가 요인 중 하나일 수 있습니다. 인간 염기서열 변이의 약 90%는 단일 뉴클레오티드 다형성(SNP)으로 알려진 DNA의 단일 염기 돌연변이에 기인합니다9. SNP가 엽산 대사의 효소 특성을 변화시키면 위암 환자에서 플루오로우라실에 대한 효능, 독성 및 화학저항성에 개인차가 발생할 수 있습니다.
메틸렌테트라하이드로폴레이트 환원효소(MTHFR)는 주로 5,10-메틸렌테트라하이드로폴레이트(5,10-MTHF)를 5-메틸테트라하이드로폴레이트로 변환하는 데 사용됩니다. 5-FU의 대사 산물인 5-FdUMP는 5,10-MTHF 및 티미딜레이트 합성효소(TS)와 함께 비활성 삼원을 형성하여 TS의 활성을 억제하고 dTMP10의 결핍을 유발합니다. 5,10-MTHF의 축적은 TS에 대한 5-FU의 억제 효과를 향상시킬 수 있으며, 이는 MTHFR의 활성과 관련이 있습니다. MTHFR rs1801131 및 rs1801133은 가장 일반적인 다형성이며, 이는 낮은 효소 활성(rs1801133의 경우 75%, rs181131의 경우 30% 감소) 및 5,10-MTHF11의 축적과 관련이 있습니다.
디하이드로폴레이트 환원효소(DHFR)는 엽산 대사와 DNA 합성의 핵심 효소입니다. DHFR은 NADPH를 사용하여 디하이드로폴레이트를 테트라하이드로폴레이트(THF)로 환원하며, 테트라하이드로폴레이트는 1탄소 단위를 운반하는 데 사용됩니다. DHFR의 SNP는 DHFR 의 발현에 영향을 미치고, THF의 활성과 풍부도를 변화시키며, 엽산 대사 및 5-FU의 민감도에 더 많은 영향을 미칠 수 있습니다. DHFR rs1650697 점 돌연변이는 DHFR 유전자의 주요 프로모터에서 발생하며, 이는 DHFR 발현을 증가시킨다12. 연구에 따르면 rs442767은 페메트렉시드 및 메토트렉세이트와 같은 항엽산 항종양제의 효능 및 독성과 관련이 있습니다. SNP rs442767과 관련하여 GT 유전자형은 각 부모의 상동 염색체에서 동일한 유전자좌에서 G 대립유전자와 T 대립유전자의 유전을 의미합니다. 유사하게, 유전자형 GG 및 TT 는 두 개의 G 대립 유전자 또는 두 개의 T 대립 유전자의 유전을 나타냅니다. 유전자형 GT+TT와 비교했을 때, GG 는 무사건 생존율 감소 및 위험 증가와 관련이 있다13. 이는 rs442767이 5-FU에 특정 잠재적 영향을 미칠 수 있음을 시사합니다.
메티오닌 합성효소(MTR)는 호모시스테인을 메티오닌으로 재메틸화하는 촉매를 하며, 메티오닌은 엽산 대사에 중요한 역할을 합니다. MTR rs1805087은 MTR 유전자의 가장 흔한 다형성입니다. MTR rs1805087은 MTR의 활성을 감소시킬 수 있는 단백질의 잠재적으로 기능적인 부위에서 아스파르트산 대신 글리신을 대체합니다. G 대립유전자를 가진 피험자는 혈장 엽산 수치가 증가하고 혈장 호모시스테인 수치가14 감소했습니다. 이에 반해 rs1805087은 5-FU의 효능과 통계적으로 유의한 연관성이 없다는 연구 결과가 나왔다. 그러나 이 연구는 대장암에 초점을 맞췄고 표본 크기가 작았습니다. rs1805087과 위암 환자에서 5-FU의 효능 사이의 관계는 아직 연구되지 않았다15.
감마-글루타밀 가수분해효소(GGH)는 세포 내 엽산 농도를 조절하는 리소좀 효소입니다. 프테로일글루탐산은 프테린, p-아미노메틸벤조산, 글루탐산으로 구성된 엽산의 동의어입니다. 유기체에는 두 가지 형태의 엽산이 있는데, 모노글루타메이트 엽산과 폴리글루타메이트 엽산입니다. THF-폴리글루타메이트는 GGH에 의해 효소로 모노글루타민산 엽산으로 전환되어 모노-글루타메이트(모노-글루) 또는 디-글루타메이트(디-글루)를 연속적으로 방출합니다16. 국소적으로 진행된 위암 환자에서 GGH 발현에 관한 연구는 높은 GGH 발현이 5,10-MTHF 및 TS를 감소시킬 수 있음을 보여주었는데, 이는 이러한 환자에서 TS 억제 효과를 달성하기 위해 5-FU의 소량만 필요하다는 것을 의미합니다17. GG는 5-FU의 전구약물인 S-1을 이용한 수술 후 보조화학요법으로 치료받은 국소 진행성 위암 환자의 예후 바이오마커이며, 엽산18의 세포내 항상성을 유지하는 데 중요한 역할을 합니다. GGH rs11545078은 오센스 변형이며 Thr-127을 Ile-127로 변경합니다. GGH의 기질 특이성에 초점을 맞춘 연구에 따르면 rs11545078은 야생형에 비해 메토트렉세이트에 대한 Km이 높고 촉매 효율이 낮으며 구조는 엽산19와 유사합니다. rs11545078과 5-FU의 임상 결과 간의 관계를 탐구하는 것은 약물 내성을 이해하는 데 유망한 전략입니다.
환원엽산 수송체(reduced folate transporter)라고도 하는 용질 담체 family 19 member 1(SLC19A1)은 포유류 세포가 de novo synthesize 능력이 부족한 환원된 엽산을 수입하는 전형적인 촉진 촉진 막관통 단백질로, 5-FU20에 대한 종양의 반응을 추정하는 것으로 알려져 있습니다. 그러나 5-FU와 SLC19A1 다형성 사이의 연관성에 관한 연구는 소수에 불과하다21. 엽산 유사체인 페메트렉시드를 투여받은 비소세포폐암 환자에서 SLC19A1 유전자의 rs1051298은 모든 약물 부작용의 위험을 증가시키고 전체 생존을 감소시키는 데 기여했습니다22,23. SLC19A1 rs1051298, 엽산 대사에 대한 3' 번역되지 않은 영역 변형은 5-FU 요법에 대한 일부 개인차를 설명하는 데 도움이 될 수 있습니다. 여기서 목표는 위암 환자에서 rs1051298과 5-FU 내성 사이의 연관성을 평가하는 것입니다.
반도체 염기서열 분석을 기반으로 하는 키트는 in vitro 유전자 검출에 사용되며(그림 1), 5개 유전자에서 선택된 7개의 SNP, MTHFR 유전자의 rs1801131 및 rs1801133 돌연변이, DHFR 유전자의 rs1650697 및 rs442767 돌연변이, MTR 유전자의 rs1805087 돌연변이, GGH 유전자의 rs11545078 돌연변이 및 SLC19A1 의 rs1051298 돌연변이를 검출할 수 있습니다. 위암 환자의 종양 조직 샘플에 있는 유전자. 먼저 샘플 핵산을 추출하고, 타겟 단편을 PCR에 의해 특이적으로 증폭하였다. 범용 염기서열분석 어댑터를 DNA 단편의 양쪽 끝에 추가하여 염기서열분석에 사용할 수 있는 라이브러리를 구축했습니다. 그런 다음 PCR에 의한 염기서열분석 라이브러리의 증폭을 수행하여 염기서열분석 템플릿을 형성하였다. 포지티브 템플릿은 염기서열분석 요구 사항을 충족하도록 강화되었습니다. 반도체 시퀀싱 시스템을 사용하여 반도체 칩의 작은 구멍에 DNA 가닥을 고정합니다. DNA 중합효소는 단일 가닥 DNA를 템플릿으로 사용하여 상보적 염기쌍의 원리에 따라 상보적 DNA 가닥을 합성합니다. 염기가 DNA 사슬에서 확장될 때마다 양성자가 방출되어 국부적인 pH 변화를 일으킵니다. 이온 센서는 pH 변화를 감지하고 화학 신호를 디지털 신호로 변환하여 염기를 실시간으로 해석할 수 있으며 최종적으로 각 DNA 세그먼트의 염기 서열을 얻을 수 있습니다. 생물정보학 분석은 이러한 염기서열을 인간 게놈의 참조 맵과 일치시키기 위해 사용되었습니다. 위암 관련 유전자가 돌연변이를 일으키면 해당 DNA 염기 서열이 변경되어 관련 유전자의 돌연변이 정보를 얻을 수 있습니다.
그 결과 유전자 돌연변이 상태를 표시할 수 있으며, 임상의가 화학요법 약물의 적절한 유형과 용량을 선택하고 위암 환자의 약물 내성을 예측할 수 있는 참고 자료를 제공할 수 있습니다. 그러나 검사 결과는 임상 참조용일 뿐이며 환자의 개별화된 치료를 위한 유일한 근거가 되는 것은 권장되지 않습니다. 임상의는 환자의 상태, 약물 적응증, 치료 반응 및 기타 실험실 검사 지표를 기반으로 종합적인 판단을 내려야 합니다.
프로토콜
본 연구에 사용된 상부 위장관 내시경 검사 또는 수술에서 얻은 위암(1단계)의 모든 프로토콜과 조직 샘플은 2023년 7월 24일 의료윤리위원회에서 검토 및 승인되었습니다(NFEC-2023-298). 게다가, 이 프로토콜은 코호트 비교에 참여하지 않고 위암에서 다중 유전자 검출을 설명하기 위해 독점적으로 설계되었기 때문에 환자를 포함하거나 제외하기 위한 특정 기준을 부과하지 않습니다. 환자/참가자는 이 연구에 참여하기 위해 서면 동의서를 제공했습니다.
1. 위암의 임베디드 블록의 준비
- 파라핀 저장소와 디스펜서로 구성된 조직 매립 시스템을 따뜻한 플레이트와 차가운 플레이트와 함께 규정된 작동 지침에 따라 구성합니다.
- 탈수기에서 매립을 위해 준비된 조직을 추출하여 매립 센터의 보관 슬롯에 넣습니다.
- 조직 크기에 따라 적합한 임베딩 금형을 선택하고 조직을 덮을 수 있도록 충분한 양의 녹은 파라핀을 부은 다음 따뜻한 판에 금형을 놓습니다.
- 카세트에서 조직을 꺼내고 위 점막을 임베딩 몰드의 바닥에 수직으로 배치하여 단면 평면이 모든 조직 층을 절단하도록 합니다. 금형에서 조직을 정렬합니다.
- 카세트를 금형 위에 놓고 파라핀을 두 번째로 부어 금형을 채웁니다.
- 내장된 금형을 냉각판에 놓고 파라핀이 응고된 후 금형에서 내장된 블록을 떼어냅니다.
2. 샘플에서 핵산 추출
- 핵산 추출 및 정제 키트를 사용하여 파라핀이 포함된 조직 샘플에서 핵산을 추출합니다.
- 핵산 정량기를 사용하여 추출된 핵산을 정량화합니다. 핵산 농도는 2ng/μL 이상인 것이 좋습니다.
참고: 여기에 사용된 재료는 재료 목차에서 확인할 수 있습니다.
3. sequencing library 준비
- 실험 전 준비 사항
- UV 램프를 켜십시오.amp 매우 깨끗한 작업대에서 30분 동안 살균합니다. 그런 다음 UV 램프를 끄고 팬을 켜서 10분 동안 환기시킵니다.
- -20 ± 5 °C 냉장고에서 핵산을 꺼내 샘플 ID, 핵산 바코드 및 샘플에 할당된 특정 라벨 태그 번호를 확인하고 기록합니다. 검증 후 상온(RT)에서 용해하기 위해 원심분리기 튜브 랙에 놓고 대기 상태용 고정 2,500 x g 으로 10초 동안 원심분리합니다.
- 타겟 단편의 PCR 증폭
- 맞춤형 위암 다중 유전자 관절 검출 키트에서 단편 포획 반응 용액, 위암 증폭 프라이머 및 융합 증폭 프라이머를 얼음 위에 올려 녹입니다. 녹인 후 흔들어 섞고 10초 동안 원심분리한 후 뉴클레아제가 없는 물을 준비합니다. 자세한 프라이머 정보는 표 1에 나와 있습니다.
알림: 이 키트는 아직 상업적으로 사용할 수 없으며 자세한 내용은 autothors에 문의하십시오. - 0.2mL PCR 반응 튜브를 준비하고, 보충 표 1에 따라 튜브에 시약을 차례로 첨가하고, 5초 동안 와류 및 혼합하고, 2,500 x g을 고정하여 10초 동안 즉시 원심분리하여 튜브 벽과 덮개에 눈에 띄는 방울이 없도록 합니다. RNA 샘플은 cDNA로 역전사된 다음 후속 라이브러리 구성에 사용됩니다. cDNA 산물은 보충 표 2에 따라 차례로 튜브에 첨가되어야 한다.
- 각 반응 튜브를 열 순환기에 놓습니다. DNA 샘플의 경우 표 2에 자세히 설명된 대로 증폭 프로그램을 실행합니다. cDNA 제품의 경우 표 3에 자세히 설명된 대로 증폭 프로그램을 실행합니다.
- 맞춤형 위암 다중 유전자 관절 검출 키트에서 단편 포획 반응 용액, 위암 증폭 프라이머 및 융합 증폭 프라이머를 얼음 위에 올려 녹입니다. 녹인 후 흔들어 섞고 10초 동안 원심분리한 후 뉴클레아제가 없는 물을 준비합니다. 자세한 프라이머 정보는 표 1에 나와 있습니다.
- primer sequence의 분해
- 프라이머 소화 효소를 꺼내 얼음에 넣어 녹입니다. 증폭 후 위의 반응 튜브를 꺼내고 각 튜브에 2μL의 프라이머 분해 효소를 추가하고 총 부피가 22μL가 되도록 합니다.
- PCR 튜브에서 반응 용액을 소용돌이 치게 하고 혼합하고 고정 2,500 x g으로 10초 동안 즉시 원심분리합니다.
- 각 반응 튜브를 열 순환기에 놓고 표 4에 설명된 대로 프로그램을 실행합니다.
- 어댑터의 결찰
- 어댑터 연결 시약을 얼음에 올려 녹입니다. 1.5mL 미세원심분리기 튜브를 준비한 후 표 5에 따라 각 성분을 혼합하고 어댑터 혼합물 X로 표시합니다.
참고: 어댑터 연결 시약에는 P1 어댑터와 1에서 48까지 번호가 매겨진 특정 어댑터 X가 포함됩니다. 이들은 다양한 샘플에 고유하게 태그를 지정하도록 설계되었습니다. 특정 어댑터를 추가할 때는 특정 어댑터의 교차 오염을 방지하기 위해 한 번에 하나의 튜브만 열어야 합니다. 희석된 특정 어댑터 혼합물은 대기 상태를 위해 -20 ± 5 °C에서 보관할 수 있습니다. 염기서열분석 워크플로우에서 샘플은 개별적으로 처리되지 않으며, 대신 각각 특정 어댑터로 태그가 지정된 여러 샘플이 염기서열분석을 위해 통합 라이브러리로 결합됩니다. 이 방법을 사용하면 특정 어댑터 시퀀스에 따라 샘플을 나중에 구별할 수 있습니다. - 열 순환기에서 소화된 프라이머 제품(22μL)을 꺼냅니다. 표 6에 따라 튜브에 시약을 차례로 넣고 와류하고 5초 동안 혼합합니다. 2,500 x g로 고정된 상태에서 10초 동안 저속으로 원심분리기를 사용하여 튜브의 벽과 덮개에 눈에 띄는 낙하가 없도록 합니다.
- 열 순환기에 반응 튜브를 놓습니다. DNA 샘플의 경우 표 7에 자세히 설명된 대로 증폭 프로그램을 실행합니다. cDNA 제품의 경우 표 8에 자세히 설명된 대로 증폭 프로그램을 실행합니다.
- 어댑터 연결 시약을 얼음에 올려 녹입니다. 1.5mL 미세원심분리기 튜브를 준비한 후 표 5에 따라 각 성분을 혼합하고 어댑터 혼합물 X로 표시합니다.
- ligation 산물의 정제 및 증폭
- DNA 정제 마그네틱 비드를 2-8 °C 냉장고에서 미리 꺼내 고르게 소용돌이치고 2,500 x g의 고정으로 10초 동안 즉시 원심분리합니다. 30분 동안 RT에서 마그네틱 비드를 평형화합니다.
- 1.5mL의 저흡착 미세원심분리기 튜브를 준비하고 결찰 반응 생성물을 해당 튜브로 옮깁니다.
- 45 μL의 DNA 정제 마그네틱 비드를 각 튜브에 넣고 와류하고 잘 섞은 다음 10초 동안 순간적으로 원심분리하고 실온에서 5분 동안 배양합니다.
- 튜브를 마그네틱 랙에 3분 동안 놓습니다. 상층액을 버리고 비드를 피펫으로 빼내지 마십시오.
- 새로 준비된 75% 에탄올 300μL를 마이크로 원심분리기 튜브에 넣고 튜브를 180°에서 4번 부드럽게 회전시킵니다. 용액이 맑아지면 상층액을 빠르게 버리십시오. 비드를 피펫팅하지 마십시오. 세탁 절차를 1배 더 반복합니다.
- 마그넷 랙에서 1.5mL 미세 원심분리기 튜브를 제거하고 짧게(10초, 고정 2,500 x g 사용). 튜브를 자석 랙에 다시 넣고 남아 있는 액체를 피펫팅합니다. 튜브 벽에 잔류 액체가 없는지 확인하십시오.
- 각 1.5mL 미세 원심분리기 튜브의 캡을 열고 비드를 실온에서 5분 동안 건조시킵니다. 구슬의 건조-습윤 상태에 주의하십시오. 마그네틱 비드를 건조시킨 후 표면에 물 얼룩이 없는지 확인하십시오. 마그네틱 비드가 너무 젖었을 경우 건조 시간을 적절하게 연장하십시오. 비드에 균열이 생기면 즉시 뚜껑을 닫고 다음 단계를 계속하여 라이브러리를 증폭하고 정제합니다.
- 라이브러리의 증폭 및 정화
- PCR 관련 시약을 미리 얼음에 넣어 용해시키고 와류시킨 후 10초간 원심분리한다. 마그넷 랙에서 1.5mL 미세원심분리기 튜브를 제거하고 표 9 에 따라 PCR 리젠트를 튜브에 피펫팅하고 캡과 볼텍스를 5초 동안 닫습니다. 튜브 벽과 덮개에 눈에 띄는 액체 방울이 떨어지지 않도록 원심분리기를 짧게(10초) 합니다.
- 위의 제품을 새 PCR 튜브에 옮깁니다. 열 순환기에서 샘플을 배양합니다. DNA 샘플의 경우, 표 10에 상세히 기술된 바와 같이 증폭 프로그램을 실행한다. cDNA 산물의 경우, 표 11에 상세히 기술된 바와 같이 증폭 프로그램을 실행한다.
- 새로운 1.5mL 저흡착 미세 원심분리기 튜브를 준비합니다. 배양 후 10초 동안 PCR 튜브를 원심분리합니다. PCR 튜브에서 EP 튜브로 제품을 이송합니다.
- 25μL의 DNA 정제 마그네틱 비드를 각 튜브에 추가합니다. 와류와 잘 혼합하고 저속으로 원심 분리하고 5 분 동안 실온에서 배양합니다.
- 튜브를 마그네틱 랙에 3분 동안 놓고 용액이 투명해질 때까지 기다립니다. 상층액을 새 미세 원심분리기 튜브로 옮깁니다. 비드를 피펫팅하지 마십시오.
- 60μL의 DNA 정제 마그네틱 비드를 각 튜브에 추가합니다. 와류와 잘 혼합하고 저속으로 원심 분리하고 5 분 동안 실온에서 배양합니다.
- 튜브를 마그네틱 랙에 3분 동안 놓고 용액이 투명해질 때까지 기다립니다. 상층액을 버립니다. 비드를 피펫팅하지 마십시오.
- 새로 준비된 75% 에탄올 300μL를 튜브에 옮기고 튜브를 180°에서 4번 부드럽게 회전시킵니다. 용액이 맑아지면 빠르게 피펫팅하여 상층액을 버립니다. 비드를 피펫팅하지 마십시오. 세탁 절차를 1번 반복합니다.
- 마그네틱 랙에서 1.5mL 미세 원심분리기 튜브를 제거하고 짧게(10초) 원심분리합니다. 튜브를 마그네틱 랙에 다시 삽입하고 남아 있는 액체를 피펫으로 빼냅니다. 튜브 벽에 잔류 액체가 없는지 확인하십시오.
- 1.5mL 튜브의 캡을 열고 비드를 실온에서 5분 동안 건조합니다. 구슬의 건조-습윤 상태에 주의하십시오. 마그네틱 비드가 건조된 후 표면에 물 얼룩이 없습니다. 마그네틱 비드가 너무 젖었을 경우 건조 시간을 적절하게 연장하십시오. 구슬에 균열이 생기면 즉시 뚜껑을 닫으십시오.
- 용리액 50 μL를 튜브에 피펫팅하고 와류시켜 잘 섞습니다. 원심분리기를 짧게(5초, 고정 2,500 x g 사용) 튜브를 5분 동안 상온에 놓습니다.
- 튜브를 마그네틱 랙에 3분 동안 놓고 용액이 투명해질 때까지 기다립니다. 액체를 새 튜브에 조심스럽게 제거하고 라이브러리 이름을 표시하십시오.
- 라이브러리를 2-8 °C의 냉장고에 임시로 보관하고 정량 측정을 기다리거나 장기 보관을 위해 -20 ± 5 °C의 냉장고에 라이브러리를 보관하십시오.
4. 구축된 라이브러리의 정량화
- 핵산 정량자를 사용하여 라이브러리를 정량화합니다. 라이브러리 농도가 0.2ng/μL≥하면 자격이 있습니다. 그렇지 않으면 라이브러리를 다시 빌드합니다.
- 동일한 부피의 DNA(또는 RNA)를 혼합하고 용액을 정량화합니다. 정량적 결과에 따라 다음 공식을 사용하여 혼합 용액을 100pmol/L로 희석합니다.
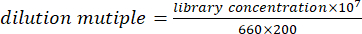
참고: 대안은 공식에 따라 라이브러리를 100 pmol/L로 희석한 다음 형광 정량 PCR로 정량화하는 것입니다. PCR 기기의 정량적 결과에 따라 DNA(또는 RNA) 라이브러리를 균등하게 혼합합니다. - 100 pmol/L DNA 라이브러리와 RNA 라이브러리를 4:1의 비율로 혼합합니다. 컴퓨터 염기서열분석을 위해 DNA와 RNA 라이브러리의 혼합 용액을 사용합니다.
5. 시퀀싱
- universal kit of sequencing reaction24,25(반도체 시퀀싱 방법)의 매뉴얼을 참조하여 시퀀싱을 수행합니다.
6. 샘플의 품질 관리
- 위암 DNA의 긍정적 인 품질 관리 : 위암의 DNA 양성 대조군을 취하고 키트의 지시에 따라 테스트를 수행합니다. 컨트롤은 키트와 함께 제공됩니다. 그 결과 MTHFR, DHFR, MTR, GGH 및 SLC19A1 유전자 돌연변이가 검출되었습니다.
- 위암 DNA의 음성 품질 관리: 위암의 DNA 음성 대조군을 취하고 키트의 지시에 따라 테스트를 수행합니다. 컨트롤은 키트와 함께 제공됩니다. 그 결과 MTHFR, DHFR, MTR, GGH 및 SLC19A1 유전자의 야생형이 검출되었음을 알 수 있습니다.
참고: 두 경우 모두에서 기준이 충족되는지 확인하고, 그렇지 않으면 다시 검색해야 합니다.
7. 데이터 분석
- Torrent Suite Software에서 해당 플러그인(Variant Caller, Coverage Analysis 및 Ion Reporter Software)을 실행하여 샘플의 분석 결과를 얻습니다. 플러그인의 분석 결과에 따라 시료 검출 결과를 판단합니다.
결과
테스트 결과의 결정은 기준 구간으로도 인식되는 긍정적 판단 값에 의존합니다. 반도체 염기서열분석 방법을 사용하여 수집된 임상 샘플을 검출합니다. MTHFR, DHFR, MTR, GGH 및 SLC19A1 유전자의 돌연변이 빈도 값이 5%≥ 경우 검출 결과는 해당 유전자의 돌연변이입니다. 돌연변이 빈도 값이 5%< 경우, 검출 결과는 해당 유전자(26)의 야생형이다.
다음 기준을 사용하여 탐지 결과를 신뢰할 수 있는지 여부를 확인할 수 있습니다. 첫째, DNA 염기서열 분석 결과의 평균 적용 범위가 500≥이고 RNA 염기서열 분석 결과의 매핑된 판독값이 20000≥ 경우 검사 결과는 신뢰할 수 있습니다27. 그렇지 않으면 다시 테스트하는 것이 좋습니다. 둘째, 위암의 DNA 양성 품질관리의 평균 적용 범위는 ≥ 500이며, 검사 결과는 MTHFR 유전자의 rs1801131 및 rs1801133 돌연변이, DHFR 유전자의 rs1650697 및 rs442767 돌연변이, MTR 유전자의 rs1805087 돌연변이, GGH 유전자의 rs11545078 돌연변이 및 rs1051298 SLC19A1의 돌연변이를 준수해야 합니다 . 유전자 양성. 그렇지 않으면 다시 테스트하는 것이 좋습니다. 셋째, 위암의 DNA 음성 품질관리의 평균 적용 범위는 ≥ 500이며, 검출 결과는 이 키트의 검출 범위 내의 모든 부위가 야생형임을 나타내야 합니다. 그렇지 않으면 다시 테스트하는 것이 좋습니다. 마지막으로, 라이브러리 농도가 0.2ng/μL 미만인 경우, 이는 샘플의 DNA 또는 RNA가 분해되었거나 실험 과정을 엄격하게 따르지 않거나 실험 중 만료된 시약을 사용했기 때문일 수 있습니다. 위의 조건으로 인해 시퀀싱 품질이 저하되거나 실패할 수 있습니다. 라이브러리를 다시 빌드하는 것이 좋습니다.
이 키트를 사용하면 임상 샘플에서 염기서열 분석 라이브러리를 구축하기 위한 일련의 단계를 따릅니다. 그런 다음 라이브러리는 이온 반도체 염기서열 분석 플랫폼에서 시퀀싱되며, ANNOVAR를 사용하여 결과에 주석을 추가합니다. 염기서열분석 후, 문서를 사용하여 각 샘플의 돌연변이 유형을 요약합니다. 돌연변이 유형이 없다는 것은 샘플이 특정 돌연변이를 겪지 않았음을 나타냅니다. 보충표 3 은 이에 대한 전형적인 예를 제공합니다. 표 12 에는 여기에서 탐지된 SNP에 대한 기본 정보가 포함되어 있습니다. 1000 Genomes Project(1000g2015aug; https://www.internationalgenome.org/; 표 13) 및 엑솜 집계 컨소시엄(ExAC; https://gnomad.broadinstitute.org/; 표 14)는 서로 다른 SNP가 서로 다른 집단에서 유의하게 존재한다는 것을 나타낼 수 있다.
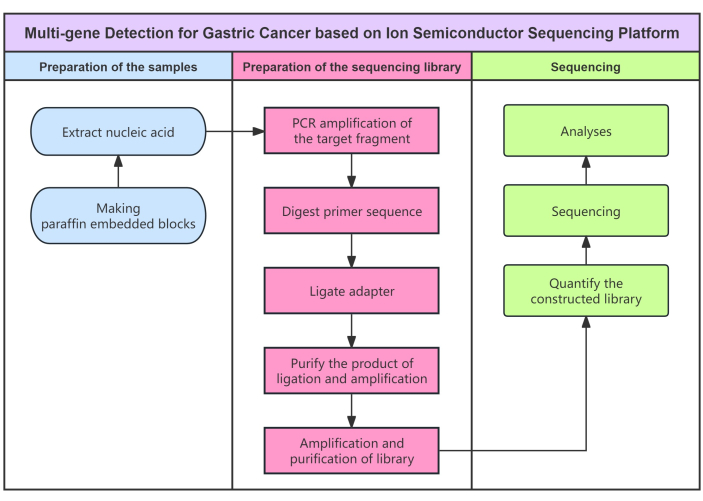
그림 1: 프로토콜의 흐름도. 반도체 염기서열분석 방법을 이용한 위암에 대한 다중 유전자 검출의 흐름도. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 염색체 | 위치 | 프라이머 디자인 간격 | 왼쪽 프라이머 | 오른쪽 프라이머 | |
| 채널1 | 11854475 | 채널1:11854175-11854775 | 아카갓그그 가그트카캅 | 아 그그카카아그 | |
| 11856377 | 채널1:11856077-11856677 | CTTCAGGTCA (주)케이터카 GCCTCAAAGC (영문) | TCCCTGT그그트 CTCTTCATCC 님 | ||
| 채널5 | 79950780 | 채널5:79950480-79951080 | CGCCGCACAT 님 AGTAGGTTCT 님 | CTTCCTCCTC 증권 시세 표시기 | |
| 79951495 | 채널5:79951195-79951795 | CTTGGGTCAC (크트그그캅) CTGCACAGTA 님 | 앳트가아그씨 (주)아큐캐치 | ||
| 채널1 | 237048499 | 채널1:237048199-237048799 | GTCAAAGGCC 아그트크크트씨 | CTCCCTTCAC (주)씨티카 CAACTGTGCT (영어) | |
| 채널8 | 63938763 | 채널8:63938463-63939063 | CAGTGAAGTT 님 CAGCGG캣 | 타트트cctgt GTGGGGCAC (영문) | |
| 채널21 | 46934825 | 채널21:46934525-46935125 | CCAACCTGAG (영어) ATGGCTTTTC | TCCTTGGTGC 님 TCTTGCTTTT | |
표 1: 5개의 유전자에서 선택된 7개의 SNP에 사용된 프라이머. 이 시트는 위암에서 5-FU 저항성과 관련된 SNP의 프라이머 위치에 대한 정보를 표시합니다.
| 온도 | 시간 | 아니요. 사이클 수 |
| 99 캜 | 2 민 | 1 사이클 |
| 99 캜 | 15들 | 22 사이클 |
| 60 캜 | 4 민 | |
| 10 캜 | 들다 | 1 사이클 |
표 2: DNA 증폭을 위한 열 사이클 프로그램. 온도, 시간 및 주기와 같은 열 반응 조건이 표시됩니다.
| 걸음 | 온도 | 시간 | 아니요. 사이클 수 |
| 효소 활성화 | 99 캜 | 2 민 | 1 사이클 |
| 변성 | 99 캜 | 15들 | 30 사이클 |
| 어닐링 및 확장 | 60 캜 | 4 민 | |
| 저장하다 | 10 캜 | 들다 | 1 사이클 |
표 3: cDNA 증폭을 위한 열 사이클 프로그램. 온도, 시간 및 주기와 같은 열 반응 조건이 표시됩니다.
| 온도 | 시간 | 아니요. 사이클 수 |
| 50 캜 | 10분 | 1 사이클 |
| 55 캜 | 10분 | 1 사이클 |
| 60 캜 | 20분 | 1 사이클 |
| 10 캜 | 들다 | 1 사이클 |
표 4: 프라이머 염기서열의 분해를 위한 열 사이클 프로그램. 온도, 시간 및 주기와 같은 열 반응 조건이 표시됩니다.
| 구성 요소 | 음량 |
| P1 어댑터 | 1.5 μL |
| 특정 어댑터 X | 1.5 μL |
| 뉴클레아제가 없는 물 | 3 μL |
| 반응 시스템의 총 부피 | 6 마이크로리터 |
| X: 특정 어댑터 번호를 나타냅니다. |
표 5: 어댑터 혼합물의 준비를 위한 시약을 첨가하는 순서. 여기에 제공된 순서대로 튜브에 시약을 추가합니다.
| 구성 요소 | 음량 |
| 버퍼 연결 | 4 μL |
| 어댑터 믹스 X | 2 μL |
| DNA 리가아제 | 2 μL |
| 반응 시스템의 총 부피 | 30 마이크로리터 |
표 6: 분해된 프라이머 생산에 시약을 추가하는 순서. 여기에 제공된 순서대로 튜브에 시약을 추가합니다.
| 온도 | 시간 | 아니요. 사이클 수 |
| 22 캜 | 30분 | 1 사이클 |
| 72 캜 | 10분 | 1 사이클 |
| 10 캜 | 들다 | 1 사이클 |
표 7: 어댑터를 DNA에 연결하기 위한 열 사이클 프로그램. 온도, 시간 및 주기와 같은 열 반응 조건이 표시됩니다.
| 온도 | 시간 | 아니요. 사이클 수 |
| 22 캜 | 30분 | 1 사이클 |
| 68 캜 | 5분 | 1 사이클 |
| 72 캜 | 5분 | 1 사이클 |
| 10 캜 | 들다 | 1 사이클 |
표 8: 어댑터를 cDNA에 연결하기 위한 열 사이클 프로그램. 온도, 시간 및 주기와 같은 열 반응 조건이 표시됩니다.
| 구성 요소 | 음량 |
| library amplification의 반응 용액 | 50 μL |
| 라이브러리 프라이머 혼합물 | 2 μL |
| 반응 시스템의 총 부피 | 52 μL |
표 9: 증폭 시약을 추가하는 순서. 여기에 제공된 순서대로 튜브에 시약을 추가합니다.
| 온도 | 시간 | 아니요. 사이클 수 |
| 98 캜 | 2 민 | 1 사이클 |
| 98 캜 | 15들 | 5 사이클 |
| 60 캜 | 1분 | |
| 10 캜 | 들다 | 1 사이클 |
표 10: DNA 라이브러리를 증폭하기 위한 열 사이클 프로그램. 온도, 시간 및 주기와 같은 열 반응 조건이 표시됩니다.
| 온도 | 시간 | 아니요. 사이클 수 |
| 98 캜 | 2 민 | 1 사이클 |
| 98 캜 | 15들 | 5 사이클 |
| 64 캜 | 1분 | |
| 10 캜 | 들다 | 1 사이클 |
표 11: cDNA 라이브러리 증폭을 위한 열 사이클 프로그램. 온도, 시간 및 주기와 같은 열 반응 조건이 표시됩니다.
| rs1801131 님 | rs1801133 | RS1650697 (영문) | rs442767 (영문) | rs1805087 | rs11545078 (영문) | RS1051298 | |
| 참고 문헌 | T | G | A | G | A | G | G |
| Alt 키 | G | A | G | T | G | A | A |
| Func.ref GeneWithVer (영문) | 엑소닉 | 엑소닉 | UTR5 | 업스트림 | 엑소닉 | 엑소닉 | UTR3 |
| 유전자.ref GeneWithVer (영문) | 증권 시세 표시기 | 증권 시세 표시기 | 디에프르 | 디에프르 | 산트리노미 역 | 그에이치 | SLC19A1 |
| 유전자세부 정보 .refGeneWithVer | . | . | NG_023304.1: g.5020T>지 | . | . | . | NM_001205206.1: c.*64C>T; NM_001205207.1: c.*746C>T; NM_194255장 2절 *746C>티 |
| 엑소닉 펑크 .refGeneWithVer | 동의어가 아닌; 증권 시세 표시기 | 동의어가 아닌; 증권 시세 표시기 | . | . | 동의어가 아닌; 증권 시세 표시기 | 동의어가 아닌; 증권 시세 표시기 | . |
| 아칸지.ref GeneWithVer (영문) | MTHFR:NM_00 1330358.1:엑소 n8:c.A1409C:p입니다. E470A; MTHFR: NM_005957.4: 엑손8:c.A1286 C:P.E429A | MTHFR:NM_00 1330358.1:엑소 n5:c.C788T:p입니다. A263V; MTHFR:N M_005957.4:e xon5:c.C665T: p.A222V | . | . | MTR:NM_0012 91939.1: 엑손2 5:c.A2603G:p. D868G; MTR:N M_001291940. 1:엑손25:c.A1 535G: p.D512G; MTR:NM_0002 54.2: 엑손26:C. A2756G:p.D91 9지 | GGH:NM_003878 .2 : 엑손 5 : c.C452T : p.T151나 | 진 |
| 사이토밴드 | 1p36.22 | 1p36.22 | 5분기14.1 | 5분기14.1 | 1Q43 | 8Q12.3 | 21Q22.3 |
표 12: SNP의 관련 정보. ANNOVAR를 사용하여 샘플에 주석 달기.
| 증권 시세 표시기 | 디에프르 | 산트리노미 역 | 그에이치 | SLC19A1 | ||||
| rs1801131 님 | rs1801133 | RS1650697 (영문) | rs442767 (영문) | rs1805087 | rs11545078 (영문) | RS1051298 | ||
| 1000g2015aug_all | 0.249401 | 0.245407 | 0.76857 | 0.290136 | 0.218251 | 0.085463 | 0.524361 | |
| 1000g2015aug_afr | 0.1513 | 0.09 | 0.9349 | 0.0318 | 0.2844 | 0.056 | 0.5772 | |
| 1000g2015aug_amr | 0.1513 | 0.4741 | 0.755 | 0.3991 | 0.1772 | 0.0403 | 0.4323 | |
| 1000g2015aug_eas | 0.2192 | 0.2956 | 0.6607 | 0.5853 | 0.1052 | 0.0873 | 0.5635 | |
| 1000g2015aug_eur | 0.3131 | 0.3648 | 0.7555 | 0.3191 | 0.173 | 0.0924 | 0.4493 | |
| 1000g2015aug_sas | 0.4172 | 0.1186 | 0.6779 | 0.228 | 0.3211 | 0.1483 | 0.5552 | |
표 13: 1000 게놈 프로젝트에서 SNP의 필터 기반 주석.
| 증권 시세 표시기 | 디에프르 | 산트리노미 역 | 그에이치 | SLC19A1 | |||
| rs1801131 님 | rs1801133 | RS1650697 (영문) | rs442767 (영문) | rs1805087 | rs11545078 (영문) | RS1051298 | |
| ExAC_ALL | 0.295 | 0.3037 | . | . | 0.2091 | 0.0936 | . |
| ExAC_AFR | 0.1588 | 0.1124 | . | . | 0.2666 | 0.0545 | . |
| ExAC_AMR | 0.1555 | 0.5141 | . | . | 0.1864 | 0.0361 | . |
| ExAC_EAS | 0.2148 | 0.3052 | . | . | 0.1132 | 0.0824 | . |
| ExAC_FIN | 0.3128 | 0.2227 | . | . | 0.1892 | 0.0581 | . |
| ExAC_NFE | 0.3191 | 0.345 | . | . | 0.1919 | 0.0969 | . |
| ExAC_OTH | 0.304 | 0.3062 | . | . | 0.2108 | 0.0973 | . |
| ExAC_SAS | 0.4153 | 0.1409 | . | . | 0.316 | 0.165 | . |
표 14: 엑솜 응집체 컨소시엄(Exome Aggregation Consortium)의 SNP에 대한 필터 기반 주석.
보충 표 1: DNA 증폭을 위한 시약을 첨가하는 순서. 여기에 제공된 순서대로 튜브에 시약을 추가합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 2: cDNA 증폭을 위한 시약을 추가하는 순서. 여기에 제공된 순서대로 튜브에 시약을 추가합니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 표 3: 임상 검체 염기서열 분석 결과의 예. 표본에 존재하는 돌연변이만 문서에 기록됩니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
임상 전문가들은 같은 유형과 병기의 위암을 앓고 있는 환자들이라도 동일한 치료 접근법에 대해 현저하게 다른 반응을 보일 수 있다는 데 만장일치로 동의합니다. 수년간의 연구를 통해 과학자들은 개인차가 주로 위암의 이질적이고 다형성이며 다양하게 분화된 세포 집단이라는 특성에 기인하며, 이로 인해 치료 반응에서 상당한 개별 불균형이 발생한다는 사실이 밝혀졌습니다28. 결과적으로 상부 위장관 내시경 검사 또는 수술을 통한 위암 검체 채취 및 혈액 검체와 유전자 분석을 위한 고처리량 염기서열 분석이 결합되어 개인 맞춤형 위암 치료가 가능합니다. 이 전략은 임상 치료 효능을 높이고 심각한 독성 부작용의 위험을 줄이기 위해 고안되었습니다. 이온 반도체 염기서열 분석 기술의 발전은 맞춤형 치료를 실제 현실로 바꾸어 놓았다29.
이 테스트 방법의 몇 가지 제한 사항은 다음과 같습니다. 여기에 사용된 키트는 주로 체외 진단을 위한 것이므로 MTHFR 유전자의 rs1801131 및 rs1801133, DHFR 유전자의 rs1650697 및 rs442767, MTR 유전자의 rs1805087, GGH 유전자의 rs11545078 및 SLC19A1의 rs1051298의 돌연변이를 검출하는 것으로 제한됩니다. 다른 섹션의 돌연변이는 감지할 수 없습니다. 종양 조직의 상당한 이질성으로 인해 다양한 샘플링 위치가 검출 결과에 영향을 미칠 수 있습니다. 파라핀 내장 조직 샘플의 경우 장기간 보관하면 DNA와 RNA가 어느 정도 분해되어 테스트 결과에 영향을 미칠 수 있습니다. 불합리한 샘플 수집, 운송 및 처리, 부적절한 실험 작업 및 실험 환경은 위음성 또는 위양성 결과를 초래할 수 있습니다. 핵산 농도가 2ng/μL 미만인 경우 검출 결과를 보장할 수 없습니다. 키트의 테스트 결과는 임상 참조용으로만 사용됩니다. 환자를 위한 맞춤형 치료법의 선택은 환자의 증상/징후, 병력, 기타 실험실 검사 및 치료 반응과 함께 고려되어야 합니다. 음성 결과는 표적 유전자 돌연변이의 존재를 완전히 배제할 수 없습니다. 음성 결과는 샘플에 종양 세포가 너무 적거나, 핵산이 과도하게 분해되거나, 증폭 반응 시스템의 표적 유전자 농도가 검출 한계 미만으로 인해 발생할 수도 있습니다.
사용된 키트의 일부 성능 지표는 설명된 것과 같습니다. 분석 감도: DNA 샘플의 경우 이 키트의 총 핵산에서 검출 가능한 최소 양은 10ng이며 5%의 돌연변이율을 검출할 수 있습니다. RNA 샘플의 경우 이 키트에서 총 핵산의 최소 검출 가능한 양은 10ng입니다. 양수 및 음수 일치율: 양수 및 음수 일치율은 100%에 이릅니다. 검출 한계(LOD): L1-L14로 번호가 매겨진 총 14개의 LOD 참조를 사용할 수 있습니다. L1-L11은 MTHFR, DHFR, MTR, GGH 및 SLC19A1 유전자의 LOD 참조이며, 이들의 검출 결과는 해당 유전자 부위의 돌연변이 유형이 양성이고 일치율이 100%여야 합니다. 반복성: 총 5개의 반복 참조 물질을 사용할 수 있으며, 번호는 R1-R5입니다. R1은 강력한 positive repetitive reference material(rs67376798 DPYD 유전자 돌연변이)입니다. 이 돌연변이를 더 적은 수로 포함하는 R2는 약한 positive repetitive reference material(rs67376798 DPYD 유전자의 돌연변이)이고, R3는 negative repetitive reference material( DPYD, MTHFR 및 ABCB1 유전자의 6개 부위가 야생형으로 검출됨)입니다. 각 참조 샘플은 10회 테스트해야 하며, 이러한 반복 평가의 결과가 사전 정의된 분류와 일치하는지 확인해야 합니다. 데이터 볼륨: DNA 및 RNA 샘플의 유효 데이터 볼륨은 0.05M 이상으로 제어해야 합니다. DNA 샘플의 염기서열 분석 깊이는 500 이상으로 제어되어야 하며 RNA 샘플의 매핑된 읽기는 20000 이상으로 제어되어야 합니다. 간섭 테스트: 이 키트는 내인성 간섭 물질(트리글리세리드 및 알부민) 및 외인성 간섭 물질(포르말린 및 탈수 알코올)의 영향을 받지 않습니다.
실험 중에는 몇 가지 예방 조치를 준수해야 합니다. 여기에 사용된 키트는 in vitro 테스트에만 사용할 수 있습니다. 실험 전에 이 설명서를 주의 깊게 읽고 유효 기간 내에 사용하십시오. 서로 다른 배치에 있는 키트의 구성 요소는 상호 교환하여 사용할 수 없습니다. 오염을 방지하기 위해 이 키트에는 일회용 소모품을 사용하는 것이 좋습니다. 이 키트를 사용하는 동안 필터 요소가 있는 흡입 헤드를 사용하는 것이 좋습니다. 샘플의 잠재적인 생물학적 위험을 피하기 위해 테스트 샘플은 피부 및 점막과의 접촉을 피하기 위해 감염 물질로 간주되어야 합니다. 에어로졸의 유출을 방지할 수 있는 생물 안전 캐비닛에서 샘플을 처리하는 것이 좋습니다. 수술에 사용된 시험관과 빨판은 폐기하기 전에 멸균해야 합니다. 샘플의 운영 및 폐기는 관련 법률 및 규정의 요구 사항을 충족해야 합니다: 미생물 생물 의학 실험실의 생물 안전성에 대한 일반 지침 및 보건부의 의료 폐기물 관리 규정30,31. 실험 인력은 전문 교육을 받고 지침에 따라 엄격하게 작동해야 하며 실험 과정에 따라 영역을 엄격하게 분리해야 합니다. 실험 작업의 각 단계에서 특수 기기 및 장비를 사용해야 하며, 각 영역의 각 단계에서 물품을 상호 교환하여 사용해서는 안 됩니다. 실험 인원은 실험 과정에 따라 영역을 엄격하게 분리해야 합니다. 실험 작업의 각 단계에서 특수 기기 및 장비를 사용해야 합니다. 필요에 따라 장갑, 작업복 등과 같은 보호 조치를 취하십시오. 폐기물 처리는 관련 국가 규정을 준수해야 합니다.
이 논문의 초점은 위암과 관련된 5개의 유전자 내에 있는 7개의 SNP에 맞춰져 있지만, 실제 응용에서의 염기서열 분석은 이 5개 유전자에만 국한되지 않습니다. 이 논문은 위암에서 7가지 SNP와 5-FU 화학요법에 대한 민감도 사이의 중요한 상관관계를 명확하게 확립합니다.
공개
저자는 밝힐 것이 없습니다.
감사의 말
이 연구는 중국 국립자연과학재단(National Nature Science Foundation of China)의 지원을 받아 위암 세포의 Apatinib-Induced Ferroptosis에 대한 내성에서 ERK2/Snai1/AGPS/PUFA-PL 경로의 메커니즘 및 역할 탐색(Item number: 82172814)의 지원을 받습니다. 고도 불포화 에테르 인지질 경로를 통해 아파티닙에 의해 유도된 페롭토시스에 대한 위암 세포 저항성을 조절하는 데 있어 징크 핑거 전사 인자 1의 역할 및 메커니즘 조사(품목 번호: 2022A1515010267), 광동성 기초 및 응용 연구 자금 지원 위원회(Guangdong Provincial Committee for the Funding of Basic and Applied Research) 지원; 위암에 대한 5-FU 화학 민감성 진단을 위한 시약의 연구 및 적용(항목 번호: 201903010072), 광저우의 과학 및 기술 프로젝트.
자료
| Name | Company | Catalog Number | Comments |
| 1.5 mL DNA LoBind Tubes | Eppendorf | 30108051 | |
| 50 mL tubes | Greiner Bio-One | 227261 | |
| Amplification primer of gastric cancer | Thermo Fisher | The primers are sythesized by Thermo Fshier according to the sequence in Table 1. | |
| Deparaffinization | Qiagen | 19093 | |
| DNA purification magnetic beads | Bechkman | A63881 | |
| Ethyl alcohol | Guangzhou Chemical Reagent Factory Thermo Fisher Scientific | http://www.chemicalreagent.com/ | |
| Ion AmpliSeq Library Kit 2.0 | Thermo Fisher | 4480441 | |
| Nuclease-Free Water | Life Technologies | AM9932 | |
| PCR tubes | Axygen | PCR-02D-C | |
| PCR tubes | Axygen | PCR-02D-C | |
| Pipette tips | Quality Scientific Products | https://www.qsptips.com/products/standard_pipette_tips.aspx | |
| PureLink RNA Mini Columns | Thermo Fisher | A29839 | |
| RecoverAll Total Nucleic Acid Isolation Kit | Thermo Fisher | AM1975 | |
| Tabletop mini centrifuge | SCILOGES | S1010E | |
| Thermal Cycler | Life Technologies | 4375786 | |
| Ultramicro nucleic acid analyzer | BEIJING ORIENTAL SCIENCE & TECHNOLOGY DEVELOPMENT LTD. | BD-1000 |
참고문헌
- Sung, H., et al. Global cancer statistics 2020: Globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 71 (3), 209-249 (2021).
- Wagner, A. D., et al. Chemotherapy for advanced gastric cancer. Cochrane Database Syst Rev. 8, (2017).
- Shah, M. A., et al. Immunotherapy and targeted therapy for advanced gastroesophageal cancer: Asco guideline. J Clin Oncol. 41 (7), 1470-1491 (2023).
- Loganayagam, A., et al. Pharmacogenetic variants in the dpyd, tyms, cda and mthfr genes are clinically significant predictors of fluoropyrimidine toxicity. Br J Cancer. 108 (12), 2505-2515 (2013).
- Giacchetti, S., et al. Phase iii multicenter randomized trial of oxaliplatin added to chronomodulated fluorouracil-leucovorin as first-line treatment of metastatic colorectal cancer. J Clin Oncol. 18 (1), 136-147 (2000).
- Hoff, P. M., et al. Comparison of oral capecitabine versus intravenous fluorouracil plus leucovorin as first-line treatment in 605 patients with metastatic colorectal cancer: Results of a randomized phase iii study. J Clin Oncol. 19 (8), 2282-2292 (2001).
- Meulendijks, D., et al. Clinical relevance of dpyd variants c.1679t>g, c.1236g>a/hapb3, and c.1601g>a as predictors of severe fluoropyrimidine-associated toxicity: A systematic review and meta-analysis of individual patient data. Lancet Oncol. 16 (16), 1639-1650 (2015).
- Sadighi, S., Mohagheghi, M. A., Montazeri, A., Sadighi, Z. Quality of life in patients with advanced gastric cancer: A randomized trial comparing docetaxel, cisplatin, 5-fu (tcf) with epirubicin, cisplatin, 5-fu (ecf). BMC Cancer. 6, 274(2006).
- Collins, F. S., Brooks, L. D., Chakravarti, A. A DNA polymorphism discovery resource for research on human genetic variation. Genome Res. 8 (12), 1229-1231 (1998).
- Ladner, R. D. The role of dutpase and uracil-DNA repair in cancer chemotherapy. Curr Protein Pept Sci. 2 (4), 361-370 (2001).
- Capitain, O., et al. The influence of fluorouracil outcome parameters on tolerance and efficacy in patients with advanced colorectal cancer. Pharmacogenomics J. 8 (4), 256-267 (2008).
- Askari, B. S., Krajinovic, M. Dihydrofolate reductase gene variations in susceptibility to disease and treatment outcomes. Curr Genomics. 11 (8), 578-583 (2010).
- Ceppi, F., et al. DNA variants in dhfr gene and response to treatment in children with childhood b all: Revisited in aieop-bfm protocol. Pharmacogenomics. 19 (2), 105-112 (2018).
- Sharp, L., Little, J. Polymorphisms in genes involved in folate metabolism and colorectal neoplasia: A huge review. Am J Epidemiol. 159 (5), 423-443 (2004).
- Yousef, A. M., et al. The association of polymorphisms in folate-metabolizing genes with response to adjuvant chemotherapy of colorectal cancer. Cancer Chemother Pharmacol. 82 (2), 237-243 (2018).
- Schneider, E., Ryan, T. J. Gamma-glutamyl hydrolase and drug resistance. Clin Chim Acta. 374 (1-2), 25-32 (2006).
- Moran, R. G. Roles of folylpoly-gamma-glutamate synthetase in therapeutics with tetrahydrofolate antimetabolites: An overview. Semin Oncol. 26 (2), 24-32 (1999).
- Maezawa, Y., et al. High gamma-glutamyl hydrolase and low folylpolyglutamate synthetase expression as prognostic biomarkers in patients with locally advanced gastric cancer who were administrated postoperative adjuvant chemotherapy with s-1. J Cancer Res Clin Oncol. 146 (1), 75-86 (2020).
- Cheng, Q., et al. A substrate specific functional polymorphism of human gamma-glutamyl hydrolase alters catalytic activity and methotrexate polyglutamate accumulation in acute lymphoblastic leukaemia cells. Pharmacogenetics. 14 (8), 557-567 (2004).
- Zhang, Q., et al. Recognition of cyclic dinucleotides and folates by human slc19a1. Nature. 612 (7938), 170-176 (2022).
- Ulrich, C. M., et al. Polymorphisms in folate-metabolizing enzymes and response to 5-fluorouracil among patients with stage ii or iii rectal cancer (int-0144; swog 9304). Cancer. 120 (21), 3329-3337 (2014).
- Zhang, X., et al. Discovery of novel biomarkers of therapeutic responses in han chinese pemetrexed-based treated advanced nsclc patients. Front Pharmacol. 10, 944(2019).
- Corrigan, A., et al. Pharmacogenetics of pemetrexed combination therapy in lung cancer: Pathway analysis reveals novel toxicity associations. Pharmacogenomics J. 14 (5), 411-417 (2014).
- Ion 520 & ion 530 ext kit - chef user guide. Thermofisherscientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https://assets.thermofisher.com/TFS-Assets%2FLSG%2Fmanuals%2FMAN0015805_Ion520_530ExTKit_UG.pdf (2023).
- Ion genestudio s5 instrument user guide. Thermofisherscientific. , Available from: https://www.thermofisher.com/document-connect/document-connect.html?url=https://assets.thermofisher.com/TFS-Assets%2FLSG%2Fmanuals%2FMAN0017528_Ion_GeneStudio_S5_Instrument_UG.pdf (2023).
- Parkin, N. T., et al. Multi-laboratory comparison of next-generation to sanger-based sequencing for hiv-1 drug resistance genotyping. Viruses. 12 (7), 694(2020).
- Ziller, M. J., Hansen, K. D., Meissner, A., Aryee, M. J. Coverage recommendations for methylation analysis by whole-genome bisulfite sequencing. Nat Methods. 12 (3), 230-232 (2015).
- Lordick, F., et al. Unmet needs and challenges in gastric cancer: The way forward. Cancer Treat Rev. 40 (6), 692-700 (2014).
- Kumar, K. R., Cowley, M. J., Davis, R. L. Next-generation sequencing and emerging technologies. Semin Thromb Hemost. 45 (7), 661-673 (2019).
- Chinese Centres for Disease Control and Prevention. Ministry of Health of the People's Republic of China. WS 233-2002. Chinese Centres for Disease Control and Prevention. , 1-64 (2002).
- China Environmental Protection Industry. Medical waste management regulations. China Environmental Protection Industry. (1), 6-10 (2004).
Erratum
Formal Correction: Erratum: Multi-Gene Single Nucleotide Polymorphism Detection in Gastric Cancer Based on Ion Semiconductor Sequencing Platform
Posted by JoVE Editors on 6/05/2024. Citeable Link.
This corrects the article 10.3791/66058
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유