É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
RNAi Mediated Gene Knockdown e Transgénese por microinjeção no Nematóides Necromenic Pristionchus pacificus
Neste Artigo
Resumo
Em organismos modelo, transgenia pode manipular as funções dos genes, enquanto RNAi pode knockdown transcrições de mRNA específicas 1-2. Este protocolo visa ilustrar as técnicas necessárias para introduzir DNA transmitidas de maneira estável e transitórios RNA de filamento duplo para o nematóide necromenic Pristionchus pacificus Para estudos em biologia evolutiva, de desenvolvimento e comportamentais.
Resumo
Embora seja cada vez mais acessíveis para os organismos modelo emergente para obter genomas completamente seqüenciados, ainda mais em profundidade a função do gene e análises de expressão por RNA de interferência e transgênese estáveis permanecem limitados em muitas espécies, devido à anatomia e biologia celular molecular do organismo. Por exemplo, fora do grupo de Caenorhabditis coroa que inclui Caenorhabditis elegans 3, transmitidas de maneira estável em linhagens transgênicas não Caenorhabditis espécies não foram relatados neste filo especiosa (Nematoda), com exceção de Strongyloides stercoralis 4 e pacificus Pristionchus 5. Para facilitar a expansão do papel dos P. pacificus no estudo do desenvolvimento, evolução e comportamento 07/06, descrevemos aqui os métodos atuais de usar microinjeção para a tomada de animais transgênicos e gene knock down por RNAi. Como as gônadas de P. pacificus é sincicial e capaz de incorporar DNA e RNA para os oócitos, quando entregue por microinjeção direta. Ao contrário de C. elegans no entanto, a herança transgene estável e expressão somática em P. pacificus requer a adição de DNA genômico digerido com endonucleases auto complementares até os confins da transgenes alvo e marcadores coinjection 5. A adição de DNA genômico transportadora é similar à exigência de expressão do transgene em Strongyloides stercoralis 4 e nas células germinativas de C. elegans. No entanto, não está claro se o requisito específico para o DNA dos animais genômica própria é porque P. pacificus soma é muito eficiente para o silenciamento não-complexo multi-cópia genes ou que arrays extracromossómicos em P. pacificus requerem seqüências genômicas para a montagem cinetocoro adequada durante a mitose. A migração ventral do (dois braçosdidelfo) em gônadas hermafroditas complica ainda mais a capacidade de injetar tanto em gônadas worms individuais 8. Nós também demonstrar o uso de microinjeção para knockdown um mutante dominante (rolo, tu92) injetando double-stranded RNA (dsRNA) para as gônadas para obter não rolando F uma progênie. Ao contrário de C. elegans, mas como a maioria dos outros nematóides, P. pacificus PS312 não é receptivo a RNAi sistêmica através de alimentação e de imersão e, portanto, dsRNA deve ser administrado por microinjeção para as gônadas sincicial. No presente estudo, esperamos para descrever o processo de microinjeção necessárias para transformar um promotor Ppa-egl-4:: GFP repórter de fusão e knockdown um rolo dominante PRL-1 (tu92) mutante em um protocolo visualmente informativo.
Protocolo
1. Transgenia: preparação DNA
- Marcador co-injeção dominante: pRL3 [Ppa-prl-1 (tu92)]
O plasmídeo pRL3 é um marcador co-injeção dominante para visualmente identificar eventos de transformação bem sucedida. Este plasmídeo codifica para um alelo dominante mutante (tu92) do gene PPA-prl-1 intimamente relacionado com o sqt-1 gene de colágeno em C. elegans e transforma a locomoção do tipo selvagem sinusoidal em movimentos de torção no sentido horário ao longo do corpo do worm eixo 1,5. O animal transformada é muito parecido com o marcador de seleção popular dominante rol-6 (su1006) usado em C. elegans, nos últimos 20 anos. - Alvo repórter transgene: Ppa-egl-4p:: gfp
Um gene de interesse pode ser ligado ou transcricionalmente traducionalmente a uma seqüência de codificação GFP 9. Neste estudo, o PPA-egl 4promoter:: gfp (egl-4, da proteína quinase dependente cGMP) was usado para determinar se um fragmento de 2 kb a montante do início da tradução pode conferir expressão GFP em P. pacificus PS312. Este vector GFP contém uma região com GFP codificação modificado
introns e do polivalente Ppa-rpl-23 3 'UTR terminator 5 gentilmente cedidas por Xiaoyue Wang e J. Ralf Sommer (Max-Planck Institute, Tuebingen, Alemanha, UE). - DNA genômico: PS312
A adição de tipo selvagem P. pacificus DNA genômico PS312 é necessário para obter a herança do transgene estável, particularmente a partir de transgênicos F 1 animais para sua progênie F 2 5. Não é ainda certo se o DNA genômico formas complexas matrizes extras cromossômicas com os dois transgenes (o marcador co-injeção PRL-3 e do PPA-egl-4p:: reporter GFP) semelhantes aos observados no estáveis F 2 linhagens transgênicas em C. elegans 3. No entanto, a exigência de ter o DNA genômico e transgenes para compartilhar identicaoverhangs l coesa sugere fortemente a formação de complexos extra-cromossômicas arrays as células hospedeiras pode reconhecer para a transmissão de DNA adequada (gDNA preparados usando Sigma G1N10 kit). - Digestão do DNA
Enzimas de restrição são usadas para linearizar o PPA-egl-4p:: gfp DNA do vetor e para produzir saliências sticky compatível com o co-injeção e marcador pRL3 PS312 gDNA. Mix tocando, não vórtex. Incubar durante uma hora a 37 ° C.
| pRL3 | 4 mg |
| 10xbuffer | 10 ml |
| PstI (10 U mL /) | 4 mL |
| dH 2 O | ~ |
| Total: | 100 L |
| Ppa-egl-4p:: gfp | 4 mg |
| Tampão 10x | 10 ml |
| SalI (10 U mL /) | 4 mL |
| dH 2 O | ~ |
| Total: | 100 l |
| gDNA PS312 | 10 mg |
| Tampão 10x | 10 ml |
| Pst I e Sal I (10 U mL /) | 8 mL |
| dH 2 O | ~ |
| Total: | 100 l |
Tabela 1. Mistura de enzima de restrição Digest.
- DNA de precipitação e de preparação
- Adicionar acetato de sódio 1:10 (3 M NaCOOCH 3 pH 5,2) e 2,5 vezes o volume de etanol a 100% para o produto digerido, adicionar 20 ng / mL de glicogênio para ver pellet mais fácil.
- Centrifugar a14.000 rpm por 15 minutos a 4 ° C.
- Decantar o sobrenadante por tombamento lentamente o tubo de centrífuga cabeça para baixo e em seguida, tocando-o em um lenço de papel, certificando-se que o pellet é segura no canto inferior do tubo e livre de etanol.
- Adicionar 1 ml de etanol 75%.
- Imediatamente giram em 14.000 rpm por 5 minutos a 4 ° C.
- Decantar o sobrenadante por lentamente derrubar o tubo de centrifugação de cabeça para baixo e tocando-o em um lenço de papel certificando-se que o pellet-la segura no canto inferior do tubo e livre de etanol.
- Seca sedimento em centrífuga a vácuo a 14.000 rpm por 5 minutos a 25-30 ° C, até secar e completamente livre de etanol.
- Adicionar 30 mL de TE, ou dH 2 O estéril e deixe durante a noite a 4 ° C antes brevemente mistura com um vórtice.
- A Tabela 2 descreve como criar a mistura de injeção de cada DNA purificado para injeção.
- Armazenar a -20 ° C. Após o descongelamento da mistura noprojecção, spin para baixo o tubo a 14.000 rpm por 5 minutos para livrar partículas detritos que possam entupir a agulha de injeção.
| Material genético | Concentração |
| Ppa-egl-4p:: gfp (SalI) | > 5 ng / mL (0.1-10ng/μl) |
| pRL3 (PstI) | > 1 ng / mL |
| gDNA PS312 (+ PstI SalI) | > 60 ng / mL |
| dH 2 O | ~ |
| Total: | 30 mL |
Mistura Tabela 2. Ppa-prl-1 injeção
2. Interferência de RNA: In vitro dupla síntese de RNA fita
- Use PCR para amplificar o gene de interesse. RHL091 5'-agtggatccGAAGGTCCATACGGGAGC-3 '(Site BamHI) e RHL092 5'-tatctgcagGTGAGGAGTACCAGGAGAG-3 '(site PstI) foram usados para amplificar um fragmento de 464 bp do genoma (posição 3-466) a partir do vetor contendo o pRL3 Ppa-prl-1 (tu92) alelo.
- Digerir o produto de PCR com BamHI e PstI.
- Ligadura o fragmento a BamHI / PstI digerida pL4440 alimentação RNAi vector 2.
- Transformar o vector inserido em células competentes e cultivá-las em placas de LB media Ampicilina (50 mg / ml).
- Selecione o vetor inserido com sucesso por PCR usando os primers T7.
- Use o T7 ladeado produto da PCR contendo o prl-1 gene para fazer double-stranded RNA dsRNA), utilizando o BLOCO-IT RNAi síntese kit (Invitrogen).
- A concentração dsRNA é melhor se> 200 ng / mL, até 1 mg / mL para injeção.
- Armazenar a -20 ° C. Após o descongelamento da mistura para injeção, girar o tubo a 14.000 rpm por 5 minutos para se livrar de partículas de detritos que possam entupir as injectioagulha n.
3. Microinjeção: Protocolo para a injeção de transgenes e / ou dsRNA
- Agulhas de injecção
- Definir o Narishige agulha extrator (modelo #: PC-10) de temperatura:
- Gire o botão de baixo para "Aquecedor No.2".
- Transformar o "Adj Aquecedor No.2". botão até painel lê 55,0-60,0 ° C para a ponta da agulha cônicos de diferentes comprimentos.
- Gire o botão inferior de volta para a "Etapa 1" (Puxa a agulha em uma única etapa).
- Força na agulha para dentro da câmara e verifique se ele é seguro.
- Pressione o botão "Iniciar" (botão vermelho) e permitir agulha para ser puxado com todos os pesos (isso deve fazer duas agulhas idênticas em sentidos opostos).
- Remova a agulha da câmara e guarde em uma placa de cultura de plástico seguros no lugar de Play-Doh para impedir que a poeira se acumula nas agulhas.
- Definir o Narishige agulha extrator (modelo #: PC-10) de temperatura:
- Pads de montagem
- Em um tubo de centrífuga de 1,5 ml, fazer uma solutio Agar 2% Noblen, adicionando 0,02 g de Agar Noble para 1 ml de H 2 O. O agar pode ser armazenado a 4 ° C por até um ano.
- Misturar bem e derreter agar em um bloco de aquecimento definida como> 88 ° C.
- Usando uma micropipeta de 1 ml, coloque uma gota de Agar 2% líquidos Noble para o meio da lâmina de vidro (Fisher, 12-544-F).
- Flatten colocando outra lâmina de vidro em cima da queda.
- Repita "Step Drop" até que haja uma camada de cinco vidro-slide.
- Retire cada lâmina de vidro, deslizando o vidro desliza para fora uns dos outros.
- Seca achatar pad ágar em lâminas de vidro em um forno a vácuo a 70 ° C por 4 horas durante a noite (ou à temperatura ambiente durante a noite).
- Fazer almofadas de injeção múltipla e armazenar para uso futuro.
- Microinjeção em gônadas hermafroditas
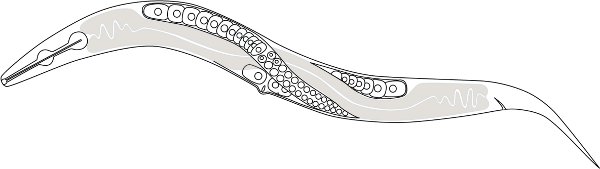
Figura 1. Um desenho esquemático do P. pacificus anatnomia. As células nucleadas claras são células germinativas femininas (ovócitos) ea parte em cinza é o intestino. Apenas uma das gônadas distal anterior é mostrado, mas observe os dois gônadas distal cruzam dorsalmente perto mid-corpo.
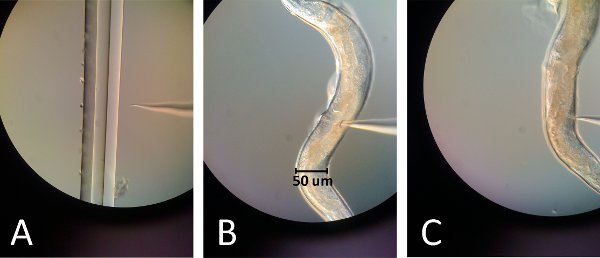
Figura 2. Imagens da microinjeção. (A) A ponta da agulha de injeção está em foco com a linha mais à direita da peça puxado capilar. Ao se tocar levemente a ponta da agulha para essa vantagem, a ponta da agulha deve quebrar, mantendo uma ponta afiada. (B) As inserções de agulhas no topo das gônadas logo acima da curvatura do intestino para a primeira injeção. (C) As inserções de agulhas para o fundo das gônadas logo abaixo da curvatura do intestino para a segunda injeção.
- Certifique-se o contraste ea definição de corte DIC é ideal para visualizar as gônadas hermafroditas.
- Carga 1,0-2,0 mL da mistura de injecção na puxadacapilar agulha.
- Coloque a agulha no manipulador de microinjeção; medidores de pressão do tanque de nitrogênio deve ser ajustado para as condições ideais (10-20 psi para 0,5-1 seg).
- Quebrar a ponta da agulha, tocando-a levemente a ponta de um tubo capilar fina puxada colocadas em uma lâmina (figura 2A).
- Agulha deslize breaker: a parte mais fina de um capilar puxado colocado sobre uma lâmina de vidro em uma gota de óleo
- Uma vez que a agulha está quebrada pode ser efectuado a uma posição de marcha lenta, enquanto você escolhe seu worm para o bloco (P. pacificus tem uma janela de 5 minutos para a injeção antes que seque-out e morrer).
- Usando um prato cheio de vermes adultos jovens-J4, despeje óleo de parafina suficiente (pesado) apenas para cobrir todos os vermes na OP50 gramado bacteriana.
- Escolha um verme e colocá-lo no bloco de injeção.
- Coloque a lâmina de vidro com o worm no microscópio; gônadas posição do worm na direção da agulha e da vulva longe da necessidadele (Figura 2B, 2C).
- Inferior a agulha para a posição da visão do microscópio.
- Posição do top das gônadas ea agulha no mesmo plano focal (Figura 2B).
- Poke o worm delicadamente e bomba na mistura da injeção; ooctyes separando para a ponta distal indicam uma injeção de sucesso (Para otimizar o transgene ou entrega dsRNA, você deve ver após a injecção de uma onda de expansão na gônada que continua até que seja próximo ao distal ponta).
- Reposicionar a agulha para injetar a menor das gônadas e injeção de repetição, mas uma vez que esta gônada é gut sob a disseminação dos oócitos não vai ser visto e é, portanto, mais difícil de alcançar (Figura 2C).
- Verme aparência normal indica uma injeção bem sucedida após a injeção, não deve haver qualquer gônadas danificado (salientes para fora da vulva ou estalou dentro da cavidade do corpo).
- Coloque a agulha na posição de marcha lenta.
- Prepare-se para resgatar o worm.
- Adicionar umqueda (0,5 L) de tampão M9 sobre o bloco onde o verme injetado é colocado.
- Usando algumas OP50 bactérias em sua escolha, toque o worm com o OP50, e ele deve ficar. Nós não usamos pipeta boca.
- Coloque o worm na placa semeada com 50 pontos mL de OP50, 2-4 worms podem ser resgatados em placas desse tipo.
- Pós-injeção de armazenamento e triagem para Transgénese
- Loja injetado worms (P 0) a 20 ° C para a 4 dias para desovar e crescer F 1.
- Worms tela no quarto dia de fenótipo selecionado (Para tela de busca por transgenia, para os animais rolando expressando o PPA-prl-1 marcador dominante).
- Escolha único independente F 1 's animais com o fenótipo observado a nova placa de sementes e armazenamento a 25 ° C; esta temperatura favorece a formação de linhas estáveis de F 1' s.
- F única de dois independentes F 1 linhas. Individuais F 2 Linhas de ter taxas de transmissão semelhantes transgene. Nós muitas vezes obter> 15 F 2 linhas por transformada F 1 animal.
- Loja gerações posteriores a 25 ° C.
- A taxa de transmissão de rolos normalmente permanece constante após a geração F 4, portanto, é necessária neste momento para determinar o nível de expressão do transgene alvo (PPA-egl-4p:: gfp) para cada linha individual. Expressão GFP pode não se correlacionar com o grau de penetrância do fenótipo rolo dominante.
- Pós-injeção de armazenamento e triagem de fenótipos RNAi
- Loja injetado worms (P 0) a 20 ° C para a 4 dias para leigos e crescer F 1.
- Tela de um worms F no quarto dia de fenótipo selecionado (Para tela para RNAi fenótipo knockdown, procure fenótipos penetrante que pode estar associada com perda de atividade dos genes-alvo, no nosso caso a perda do PPA-prl-1 fenótipo).
- Em nossa experiência, o fenótipo knockdown não-roller é perdida em> 97% da F 2.
4. Resultados representativos:
1. Resultado da Transgénese
| Sessão | Injetado P 0 | F 1 Rollers | F 2% transgênica | Transmissão% em média depois de F 2 |
| 1 | 60 | 2 | (Linha 1) 0% (Linha 2) 13% | NA 13% ± 10 |
| 2 | 40 | 1 * | (Linha 3) 0% | NA |
| 3 | 40 | 0 | 0% | NA |
| 4 | 40 | 1 | (Linha 4) 0% | NA |
| 5 | 20 | 0 | 0% | NA |
| 6 | 40 | 1 | (Linha 5) 26% | 22% ± 10 |
* Rolo de um macho que não cruz; NA: Não aplicável
. Tabela 3 Resultados do PS312 injetados com Ppa-egl-4p:: gfp (Sal eu digerida), pRL3 (rolo) (Pst I digerida), e gDNA PS312 (PstI e SalI digerida).

Figura 3 (A) do tipo selvagem (B, C) A estabilidade F 4 pRL3; Ppa-egl-4p:.: Gfp linhagem transgênica mostrando cabeça neurônio expressão GFP.
2. Resultado da interferência de RNA

Figura 4. RNA injetado PRL-1 mutantesmostrar knockdown completa de locomoção "rolling". (A) PRL-1 rolo de ganho de função-mutante tu92. (B) "derrubou" PRL-1 mutante exibem postura do tipo selvagem normal e locomoção. (C) Injected PRL-1 (baixo) também tem mais corpo do que prl-1 mutante (em cima). (D) O corpo de mais injetados PRL-1 (baixo) também é maior do que o PS312 tipo selvagem (em cima). O fenótipo mais corpo também foi observado no rol-5 (sqt-1) knockdown de RNAi C. elegans N2 (dados não mostrados).
| rolo | não-roll | |
| A PRL-1 dsRNA (200 ng / mL) | 13 | 21 |
| Prl B-1 dsRNA (1000 ng / mL) | 9 | 16 |
| controle | 20 | 2 |
Tabela 4. Resumo das Ppa-prl-1 dsRNA injeções. (A) [dsRNA] = 200 ng / mL; (B) [dsRNA] = 1000 ng / mL. P = 0,0019 e 0,041 pelo teste exato de Fisher, bicaudal, para 200 e 1000 ng injeções / mL, respectivamente.
Access restricted. Please log in or start a trial to view this content.
Discussão
Populações de P. pacificus são encontrados em estreita associação com espécies de escaravelho vários besouro todo o mundo e é um modelo intermediário entre o nematóide de vida livre e nematóides parasitas. A força do P. pacificus como um organismo modelo emergente reside na integração de seus mapas genéticos e físicos que promovem o mapeamento posicional de mutantes isolados de telas imparcial frente genética (ou seja, não apenas por genes candidatos previamente caracterizada...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Os autores são muito gratos ao RJ Sommer e Wang X para obter ajuda com microinjeção, bem como comentários perspicaz dos revisores anônimos. Este trabalho é apoiado por NIH conceder SC2GM089602.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Produto: | Catalogo: | Fornecedor: | |
|---|---|---|---|
| DMI3000 microscópio Injection | DMI3000 | Leica | |
| Microinjetor manipulador (acionamento direto) | Narashige conjunta BC-3 bolas | Tritech Research | |
| Agulha Puller | Narashige PC-10 | Tritech Research | |
| Microinjetor ™ All-Digital Sistema Multi-pressão | Narashige Minj-D | Tritech Research | |
| GeneElute ™ Genomic DNA de mamíferos Miniprep Kit | G1N70 | Sigma | |
| pJet Clonagem Jet kit | K1231 | Fermentas | |
| GeneJET plasmídeo Miniprep kit | K0502 | Fermentas | |
| DNA Clean & Concentrador ™ | D4005 | ZYMO Research | |
| BLOCK-IT ™ RNAi TOPO kit de transcrição ® | K3500-01 & K3650-01 | Invitrogen | |
| Agar Difco ™ Noble | DF0142-15-2 | Fisher cientificamente | |
| De vidro para microscópio cobrem (1,5 - 0,16 a 0,19 milímetros de espessura; Tamanho: 50 x 45mm) | 12-554-F | Fisher Scientific | |
| Capilares de vidro (filamento) | 615000 | Sistemas AM | |
| Óleo de parafina (Pesado) | O122-1 | Fisher Scientific | |
| KH 2 PO 4 | P386-500 | Fisher Scientific | |
| Na 2 HPO 4 | AC20651-5000 | Fisher Scientific | |
| NaCl | BP3581 | Fisher Scientific | |
| MgSO 4 | M80-500 | Fisher Scientific |
Referências
- Fire, A., Xu, S., Montgomery, M. K., Kostas, S. A., Driver, S. E., Mello, C. C. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Ahringer, J. Reverse Genetics. Wormbook. , (2006).
- Mello, C. C., Kramer, J. M., Stinchcomb, D., Ambros, V. Efficient gene transfer in C. elegans: extrachromosomal maintenance and integration of transforming sequences. EMBO J. 10, 3959-3970 (1991).
- Junio, A. J., Li, X., Massery, H. C., Nolan, T. J., Lamitina, S. T., Sundaram, M. V., Lok, J. B. Strongyloides stercoralis: cell- and tissue-specific transgene expression and co-transformation with vector constructs incorporating a common multifunctional 3' UTR. Exp. Parasitology. 118, 253-265 (2008).
- Schlager, B., Wang, X., Braach, G., Sommer, R. J. Molecular cloning of a dominant roller mutant and establishment of DNA-mediated transformation in the nematode Pristionchus pacificus. Genesis. 47, 300-304 (2009).
- Hong, R. L., Sommer, R. J. Pristionchus pacificus: a well-rounded nematode. Bioessays. 28, 651-659 (2006).
- Hong, R. L., Sommer, R. J. Chemoattraction in Pristionchus nematodes and implications for insect recognition. Curr. Biol. 16, 2359-2365 (2006).
- Rudel, D., Riebesell, M., Sommer, R. J. Gonadogenesis in Pristionchus pacificus and organ evolution: development, adult morphology and cell-cell interactions in the hermaphrodite gonad. Dev. Biol. 277, 200-221 (2005).
- Chalfie, M., Tu, Y., Euskirchen, G., Ward, W. W., Prasher, D. C. Green fluorescent protein as a marker for gene expression. Science. 11, 802-805 (1994).
- Chaudhuri, J., Parihar, M., Pires-daSilva, A. An Introduction to Worm Lab: from Culturing Worms to Mutagenesis. J. Vis. Exp. (47), e2293-e2293 (2011).
- Evans, T. C. Transformation and Microinjection. Wormbook. , (2006).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados