Method Article
A técnica de microscopia TIRF para imagens em tempo real, simultâneo da TCR e seus associados proteínas sinalizadoras
Neste Artigo
Resumo
A compartimentação de proteínas, quer no interior da membrana plasmática ou em localizações intracelulares é um mecanismo de regulação que pode influenciar grandemente os resultados de sinalização, daí, para compreender a sinalização é importante para estudar o comportamento espacial e temporal das proteínas envolvidas. Descrevemos aqui um sistema de microscopia TIRF base para estudar a transdução de sinal em células T, mas é amplamente aplicável.
Resumo
Sinalização é iniciada através do receptor de célula T (TCR) quando ele está envolvida por fragmentos antigénicos peptídicos ligados por Complexo Principal de Histocompatibilidade (pMHC) proteínas expressas na superfície de células apresentadoras de antigénio (APCs). O complexo TCR é composto do heterodímero TCRαβ ligação do ligando que associa não covalentemente com CD3 dímeros (os εδ e εγ heterodímeros e do homodímero ζζ) 1. Após acoplamento do receptor, as cadeias zeta CD3 são fosforilados pela quinase da família Src, Lck. Isto leva ao recrutamento da família Syk quinase, ZAP70, que é então fosforilada e activado por Lck. Depois disso, o adaptador de ZAP70 fosforila as proteínas LAT e SLP76, iniciando a formação do complexo de sinalização proximal contendo um grande número de diferentes moléculas sinalizadoras 2.
A formação deste complexo eventualmente resulta em cálcio e Ras-dependente traactivação nscription fator ea consequente início de uma série complexa de programas de expressão génica que dão origem a diferenciação de células T 2. Sinais de TCR (eo estado resultante da diferenciação) são modulados por muitos outros fatores, incluindo a potência antígeno e crosstalk com co-stimulatory/co-inhibitory, quimiocinas e receptores de citocinas 3-4. Estudar a organização espacial e temporal do complexo de sinalização proximal sob condições de estimulação diversas é, portanto, a chave para a compreensão da TCR via de sinalização, bem como sua regulamentação por outras vias de sinalização.
Um sistema de modelo muito útil para estudar a sinalização iniciada pelo TCR na membrana plasmática de células T é bicamadas lipídicas de vidro-suportados, como descrito anteriormente 5-6. Eles podem ser utilizados para apresentar complexos antigénicos pMHC, adesão e moléculas co-estimulatórias em células T, que serve como APCs artificiais. Por imagem das células T a interagir com obicamada lipídica usando microscopia de fluorescência interna total reflexão (TIRFM), podemos limitar a excitação para dentro de 100 nm do espaço entre o vidro ea superfície da célula 7-8. Isto permite-nos a imagem em primeiro lugar os eventos de sinalização que ocorre na membrana plasmática. À medida que está interessado em imagiologia o recrutamento de proteínas de sinalização para o complexo TCR, nós descrevemos um de dois câmara TIRF sistema de imagem, em que o TCR, rotulado com Fab fluorescente (antigénio fragmento de ligação) fragmentos do anticorpo H57 (purificada a partir de hibridoma H57-597 , ATCC Número, ATCC: HB-218) que é específico para TCRβ, e as proteínas de sinalização, marcado com GFP, pode ser trabalhada em simultâneo e em tempo real. Esta estratégia é necessário devido à natureza altamente dinâmico de ambas as células T e dos eventos de sinalização que estão a ocorrer no TCR. Esta modalidade de imagem tem permitido aos pesquisadores ligantes de imagens simples 9-11, bem como o recrutamento de moléculas sinalizadoras a receptores ativados eé um excelente sistema para estudar bioquímica in-situ 12-16.
Protocolo
Procedimento Experimental:
1. Transfecção utilizando Amaxa rato célula T Nucleofector Kit
Descrevemos aqui o transfecção de ingênuo ou in-vitro ativados pilhas de T com plasmídeos de expressão que codificam moléculas sinalizadoras marcados por GFP, usando o mouse Amaxa célula T Kit Nucleofector. In-vitro a activação de células T utilizando péptido-carregados APCs esplénicas é realizada como descrito antes 7. Todas as células T utilizadas nos nossos estudos expressar o TCR e que reconhece MCC péptido (88-103) ligado à molécula de MHC IE k. A transfecção é efectuada essencialmente de acordo com as recomendações do fabricante, no entanto, apresentam-se alguns dicas que promovem a viabilidade das células T após a transfecção. Tanto a viabilidade e eficiência de transfecção é muito maior em in-vitro de células T activadas do que nas células naive.
- Antes de começar, preparar 2 mL de meio de células T suplementado pela adição de 20 uL deambos os componentes do meio A e B e 100 uL de soro fetal de bovino (5%) para cada transfecção a ser executada. Equilibrar o meio em uma placa de 12 poços num humidificada 37 ° C, 5% de CO 2 incubadora durante pelo menos uma hora. Alternativamente, alíquotas de meio suplementado com soro de 5% e um componente podem ser preparados e armazenados congelados. Se este for o caso, descongelar o número necessário de alíquotas, adicionar o Componente B, e executar o equilíbrio.
- Pré-equilíbrio do meio é muito importante, como a morte celular irá resultar da re-suspensão de células após a transfecção em meio que é frio ou tem um pH diferente de 7,2.
- Em seguida, preparar a mistura de transfecção. Combinar 82 uL de rato Solução Nucleofector célula T com 18 uL de Suplemento 2 e 5μg de DNA de plasmídeo (concentração não inferior a 0,5 ug / uL). Misturar bem tocando o tubo ou com o uso de micro pipeta.
-É importante para realizar uma titulação da dose do plasmídeo por um número fixo de células para cada constructo a ser utilizado para a transfecção. Para algumas construções maiores, mais DNA pode ser necessária. DNA deve ser livre de endotoxinas.
- Transferência de 5 a 10 milhões de células a um tubo de centrífuga de 15 mL. Centrifugar a 600 rpm durante 5 minutos à temperatura ambiente. Remover o máximo do sobrenadante quanto possível, com um aspirador de vácuo.
- Os fabricantes recomendam a girar as células a menos de 90x g, no nosso caso, essa velocidade corresponde a 75x g. O escolhido força centrífuga relativa (RCF) para se obter um sedimento celular é talvez o mais importante variável, tal como centrifugação a RCF baixa impede danos para a membrana celular.
- Re-suspender o sedimento de células na mistura de transfecção por pipetagem suave e lentamente o pellet celular até que não haja grandes aglomerações de células restantes. Isto pode geralmente ser realizada por completamente aspiração das células Through a ponta da pipeta não mais do que 3-4 vezes.
- Usando uma pipeta, transferir cuidadosamente a suspensão para uma cuvete Amaxa certificada, assegurando que não existem bolhas de presentes. Tapar o cuvete.
- Seleccione o programa Nucleofector X-001 no dispositivo de Nucleofector. Insira a cubeta no suporte Cuvette Nucleofector e aplicar o programa selecionado.
- Depois electroporating das células, o melhor é imediatamente transferi-los para o meio de cultura pré-equilibrada. Os fabricantes recomendam que as células ser mantido no meio de nucleofector por não mais do que 15 minutos.
- Traga a cuvete e do pré-equilibrada, meio de cultura suplementado totalmente para a capa. Usando a pipeta de plástico fornecidos, adicionar cerca de 500 uL do meio para a cuvete e transferir a suspensão de célula para o meio na placa gota a gota.
- Incubar as células a 37 ° C durante 4 horas antes de imagem. Células naive são incubated a 30 ° C em vez de 37 ° C. Isto ajuda a manter a sua reactividade ao antigénio.
- Três a quatro horas de incubação são geralmente suficientes para as células T a detectavelmente expressar a GFP proteínas marcadas. Optou-se por células de imagem, neste ponto do tempo, porque o nível de expressão é baixa e artefactos de sobre-expressão deve ser minimizada. Se a sobre-expressão de uma proteína particular é desejado, no entanto, as células T deve ser removido a partir do meio Amaxa após 4 horas. Isto é conseguido através de novo peletização as células a RCF baixa e ressuspensão em pré-equilibrada meio de cultura de células T suplementado com 50 U / mL de interleucina 2 (IL-2) a uma densidade de 2 milhões de células / mL.
2. Microscopia TIRF
Descrição do microscópio TIRF:
- Gerais de configuração: Optical componentes foram construídos em torno de um microscópio de fluorescência Olympus IX71 que foi originalmente equipado com um módulo TIRF Olympus. Logo discovered efeitos cromáticos neste módulo, quando um estado único alinhamento não conseguir colimação coincidente para linhas de laser de diferentes comprimentos de onda. Este módulo, portanto, não permitiria a imagem TIRF simultânea em comprimentos de onda de excitação diferentes, e nos propusemos a construir um aparelho TIRF personalizado que minimiza os efeitos cromáticos no sistema, como discutiremos em detalhes nas seções seguintes. O método aqui descrito foi desenvolvido para a ampliação 150 X, 1,45 abertura numérica (NA) TIRFM objetivo (Olympus), mas funciona tão bem para a ampliação X 60, 1,45 versões de NA. Para a iluminação de campo de largura, o microscópio está ligado a uma fonte de luz haleto de metal Lambda-XL e uma roda de filtro de excitação (Sutter Instruments) equipado com filtros de excitação. Para o posicionamento automático da fase de amostra em relação ao objectivo, o microscópio é equipado com uma tradução XY motorizado, piezo-controlada Z fase (ASI). As imagens são capturadas através de dois Qu idênticoAntem elétrons Multiplicando (EM) câmeras CCD (Photometrics). O tamanho do pixel dessas câmeras combina muito bem com a ampliação oferecida pelo 150 objetivo TIRF X, dando uma resolução final de 0,1 mM por pixel. As câmeras também oferecem EMCCD a sensibilidade de visualizar transf GFP com nível de baixa expressão.
- Lasers: Para TIRF iluminação, um sistema de entrega de 5 lasers um total de 6 linhas de laser (405 nm, 440, 488, 514, 561 e 640) está acoplado a uma fibra monomodo (Technologies Solamere). O laser de Árgon e 561 nm diodo bombeado de estado sólido (DPSS) laser (linhas 488, 514 e 561) são encaminhados através de uma fibra acusto-óptica sintonizável (AOTF) para controlo de intensidade enquanto a intensidade dos lasers de diodo (405, 440 e 640 nm) é controlado por tensão do modulador de suas fontes de alimentação. A tensão na AOTF e as fontes de alimentação dos lasers de diodo é controlado através de uma placa de aquisição de dados (DAC) placa (Measurement Computing), utilizando MetaMorph software (moleculareslar Devices). A eficiência de acoplamento para os lasers de diodo é de 30% enquanto que as dos lasers de árgon e DPSS são aproximadamente 60%, devido aos seus diâmetros menores do feixe.
- TIRF módulo: A fim de simultaneamente a imagem do TCR e suas moléculas de sinalização associadas, como descrito anteriormente, foi essencial ter TIRF iluminação que foi cromaticamente corrigida e que não exigiria realinhamento ou recentragem na aquisição de emissões de fluorescência de comprimentos de onda diferentes. TIRFM foi conseguida utilizando através-da-objectivo de iluminação, como descrito anteriormente 17. O cabo de fibra óptica que fornece luz de laser para o microscópio foi fixada em um lançamento de fibra equipado com um suporte de fibra XY, montado sobre um carril micrómetro óptico orientado para Z ajustamento (Thorlabs). Para alcançar TIRF iluminação, o feixe de laser é focada no plano focal posterior da objectiva, usando um sistema de duas lentes (L1 (colimação da lente) e L2 (lente de focagem) na Figura 1), de modo que o beam surge colimado do objetivo. Isto assegura que todos os raios de luz que sai do objectivo terá o mesmo ângulo em relação ao plano da amostra. A ponta da fibra óptica é posicionada no foco da lente de colimação. Ao mover o micrómetro óptico ao longo do carril em relação à lente colimadora (na direcção Z) é possível ajustar a focagem do feixe e mover o feixe em X e Y posição usando os botões de ajuste XY. Devido à volta da abertura muito pequena do objectivo X 150, descobrimos que, para alcançar TIRF, o tamanho do ponto de laser no plano focal posterior necessário para ser pequena. Para alcançar o foco necessária para produzir a pequena mancha, as lentes de comprimento focal curta eram necessárias (Keir Neumann, comunicação pessoal), daí, utilizou-se uma lente de focagem com uma distância focal de aproximadamente 108 mm que foi colocado no cubo dicróico imediatamente anterior o objetivo. A lente de colimação foi colocado mais perto do divisor de feixe que trouxe na TIIluminação RF. Para minimizar os indesejáveis efeitos cromáticos, usamos doublets acromáticas (lente cromaticamente corrigido feita por elementos fundindo dois lentes feitas de materiais de diferentes índices de refração) com revestimentos anti-reflexo, tanto para a colimação e focagem com lentes (JML óptica). O foco pode ser verificado através da verificação de que o feixe colimado emerge fora do objectivo quando posicionado no centro da abertura para trás e devem formar um ponto apertado no tecto do quarto. A posição do ponto focado é então movido para a borda da abertura (na direcção Y), que faz com que o feixe convergir para o plano de imagem em grandes ângulos, e eventualmente, o ângulo crítico para a reflexão interna total é alcançada. Usando estes componentes ópticos, obtivemos colimação simultânea para todos os comprimentos de onda de 442 a 640 nm no mesmo alinhamento. Um alinhamento óptico único, por conseguinte, permitiu-nos para executar simultaneamente ao longo de TIRFM múltiplos comprimentos de onda.
- Appar câmera duplaATUS: aquisição simultânea da emissão de fluorescência distinta resultante de dois fluoróforos diferentes foi conseguida através da construção de um sistema de câmara dupla que divide a emissão em duas gamas de comprimento de onda através do uso de espelhos dicróicos (Figura 2). Luz emitida é reflectida a partir do objectivo para fora da porta do lado direito do microscópio. Como a maioria dos objectivos são infinito corrigida (com imagens formadas ao infinito), uma lente de tubo é necessário para focar a imagem. Para manipular dois comprimentos de onda de emissão simultânea, a lente tubo na porta do lado direito foi removido. Um adaptador personalizado contendo c-mount tópicos (Sutter Instruments) foi anexada e ligada a um detentor espelho dicróico (Edmund Optics). Espelhos dicróicos que separam a emissão de GFP do de corantes orgânicos utilizados para etiquetar o TCR (Alexa546, Alexa647, etc) foram instaladas no presente titular. Este módulo foi seguido, em ambos os caminhos de luz por um tubo de suporte de lente, um suporte de filtro de emissão e os tubos de extensão. OAs lentes de tubo (TL1 e TL2 (Figura 2)) têm um comprimento focal de 180 mm. As lacunas nos tubos de extensão que levam para as câmeras CCD moeda electrónica foram cobertas com papel preto para proteger as câmeras de luz difusa. Para permitir o alinhamento dos dois canais, as câmaras foram montadas em dois suportes de tradução personalizados XYZ (Produtos Holmarc).
Abaixo descrevemos como alinhar o microscópio para dois canais de imagem simultânea. Os passos envolvidos são alinhando a iluminação TIRF, ajustando a potência do laser, e alinhar as duas imagens de saída utilizando a resolução sub-micropérolas.
- Antes de começar, todos os componentes do microscópio estão ligados. O computador e software são então iniciado e é assegurado que todos os componentes de hardware são reconhecidos pelo software.
- Para utilização como um padrão de co-localização, diluir 1 uL de sub-resolução (0,2 um) Fluoresbrite Multi-fluorescente Microesferas em 2 mL de PBS e adicionar 0,25 mL da microesferasuspensão a cada poço de uma II Lab-Tek 8-bem câmaras sistema lamela (Nunc). Coloque o sistema de slide câmara para a plataforma acima do objetivo. Grânulos que são adsorvidos ao vidro são então postos em destaque.
- Usando o alinhamento Y do lançamento de fibra XYZ, a posição do feixe de laser é movido para o centro da abertura de trás do objectivo. É importante fazer isso, quando o objectivo é na posição onde o plano de vidro é em foco. Se o feixe não é colimada, o micrómetro Z é ajustada para atingir colimação completa. Colimação é testado individualmente para todas as linhas de laser a ser utilizadas na experiência. Nesta fase, a potência do laser, para cada linha é medido usando um medidor de energia laser e é ajustado a 20-30 mW em cada canal.
-T células são extremamente sensíveis à radiação de laser, ea iluminação está limitada a um total de 50 mW.
- Imagens das esferas são adquiridos por meio TIRF ILLUMINATíon no modo ao vivo. Ligue o micrómetro Y-alinhamento sobre o lançamento de fibra de modo a que o feixe começa afastando-se do limite máximo até que o seu ângulo com respeito às abordagens o objectivo do eixo óptico 90 graus. Eventualmente, quando o ângulo crítico para a TIR, é atingido, a luz do laser já não sair do objectivo. Alinhamento TIRF é então julgado por focagem cima e para baixo através do plano da lamela. Se grânulos que estão flutuando em solução pode ser visualizado, em seguida, o ângulo do feixe no que diz respeito às necessidades objectivas normais de ser aumentada ainda mais. No alinhamento TIRF correcta, apenas um plano óptico estarão em foco (isto é, apenas as pérolas que são adsorvidas à lamela). Um teste mais fina do alinhamento TIRF pode ser implementada quando as células T de imagem marcadas com um corante de superfície que se espalhar sobre uma superfície. (Ver ponto 3.4).
- Depois de iluminação TIRF tenha sido alcançado, os dois canais podem ser alinhadas com relação um ao outro. Focar os grânulos e comparar a posição do icaracterísticas dentifiable nas imagens produzidas em ambos os canais. Usando o X e Y parafusos micrómetros em um suporte para a câmara, trazer a posição relativa dos grânulos em como a proximidade estreita quanto possível. Salve as imagens de ambos os canais. Estas imagens será utilizado como um padrão de co-localização para alinhar os dois canais.
- As câmaras também precisam de ser alinhadas de modo que eles são parfocal com respeito a um outro. Isto é conseguido através da aquisição de um conjunto de imagens das pérolas de resolução de sub que são separados um do outro na direcção Z de 100 nm. Se o pixel intensidade máxima para as pérolas se situa no plano mesma para ambas as câmaras, em seguida, eles são parfocal.
3. Imagem
Uma vez que o microscópio está configurado, a próxima tarefa é preparar câmaras de fluxo contendo bicamadas lipídicas de vidro apoiadas apresentam moléculas de antigénio e de adesão, como descrito anteriormente 6 e em seguida, executar TIRFM de células T transfectadas interagindo com osubstrato. A bicamada publicado protocolo 5-6 foi seguido tal como publicado, excepto que os lipossomas não foram incubadas durante 20 minutos sobre a lamela após a câmara de fluxo foi montado. Em vez disso, HBS-BSA tampão foi fluiu na célula de fluxo imediatamente após a montagem.
- Montar uma câmara de fluxo com bicamadas lipídicas planas formadas na lamínula de vidro contendo Ni-NTA lipídios. As moléculas de adesão, ICAM-1 (100 moléculas / iM 2), e péptido-MHC complexos, MCC carregado IE k (moléculas 5-10 / iM 2), são incorporados na bicamada via suas etiquetas de histidina. A co-estimulatório molécula CD80 é adicionado usando uma âncora de GPI (100 moléculas / iM 2).
- Coloque a célula de fluxo sobre a fase do microscópio e estabilizá-la. Anexar o fabricante fornecido elemento de aquecimento sobre a célula de fluxo para controlar a sua temperatura. Posicione o objetivo em uma bicamada e trazer o avião bicamada em se concentrar. Habilitar aquecimento do objectivo e do fluxocélula para estabilizar a temperatura da câmara a 37 ° C.
- Transfectadas As células T são peletizadas após 4 horas de incubação a uma velocidade de 80x g, e re-suspensas em HBS-BSA e tampão suplementado com 10 ug / mL de H57 Fab ou 20 ug / mL de H57 único fragmento variável de cadeia (scFv) para etiquetar o TCR. As células são então injectados nas células de fluxo. Imagens é feito na presença contínua do Fab ou scFv, devido à sua alta constante de dissociação. A sua presença no meio não aumenta significativamente o fundo como o campo TIRF é muito fina.
- Condições de imagem estão configurados de modo que a aquisição de dois canais ao vivo mostra uma visualização ao vivo dos canais de TCR e GFP que ajuda na busca de células transfectadas. TIRF iluminação é confirmada por concentrando-se o plano de vidro para detectar qualquer coloração da membrana lateral. Se membranas laterais não entram em foco, em seguida, o alinhamento TIRF é bom. As células transfectadas são então trabalhada uma vez ou em uma sequência de imagens para produzir videos.
4. Processamento de Imagens e de Análise de Dados
O software exibe imagens das duas câmeras em um único frame. A saída de cada câmara é de 512 x 512 pixels, daí, a imagem de saída é de 1024 x 512 pixels de tamanho. As imagens primeiro precisam de ser dividida em quadros individuais correspondentes às duas câmaras. As imagens de resolução de sub-grânulos a partir dos dois canais são sobrepostas e os parâmetros de alinhamento são determinados. Uma característica específica para o nosso sistema é uma rotação grau 1 de um canal com respeito ao outro (ver Figura 3). A fonte mais provável dessa rotação são os suportes da câmara. Após esta rotação adicional relativa transformação linear em X e Y têm de ser realizados para alinhar perfeitamente os grânulos com respeito ao outro. Imagens das pérolas são obtidos em cada experiência para determinar os parâmetros precisos para alinhar os dois canais. Estas transformações lineares e rotacional são, então, aplicado a todos osimagens e, em seguida, eles estão sujeitos aos algoritmos de segmentação e análise de co-localização.
5. Os resultados representativos
O sistema aqui descrito pode ser adaptado para estudar a jusante de sinalização de qualquer receptor na membrana plasmática. É particularmente informativa no estudo TCR sinalização porque sinalização ocorre em aglomerados opticamente resolvidos chamados TCR microclusters ou em endosomas como descrito recentemente 18. Se sinalização ocorreu em resolução sub-grupos de receptores ou em un-cluster receptores experimentos adicionais teria que ser feito para interpretar os dados. Como mencionado antes, os CD3 cadeias do complexo TCR sofrer fosforilação por Src quinase família Lck sobre envolvimento do TCR pelo péptido-MHC complexos. A cadeia CD3ζ fosforilada então recruta o quinase família Syk ZAP70. Visualizando o recrutamento de Zap70, assim relata o estado de fosforilação da cadeia CD3ζ. A força de estimulação TCR pode ser alterada utilizando peptídeos mutantes com os resíduos de contacto de TCR. Tais péptidos são chamados alterados ligandos peptídicos (APL). Uma experiência representativa é mostrado na Figura 4, onde os péptidos agonistas robustamente recruta ZAP70 para microclusters TCR enquanto o menor potência péptido T102L falhar para recrutar ZAP70 para TCR microclusters, demonstrando que a cadeia CD3ζ não é fosforilada no presente caso. Nós poderíamos chegar a essa conclusão porque tivemos que apenas observar o recrutamento de ZAP70 a clusters de TCR, que foram facilmente detectados pelo TIRFM. Um algoritmo de segmentação automatizado foi usado para contar o número de ZAP70 microclusters por célula. Também é mostrado no filme complementar é a gravação simultânea de TCR e associados ZAP70 fluorescência. Observe a natureza dinâmica da lamelipodium.
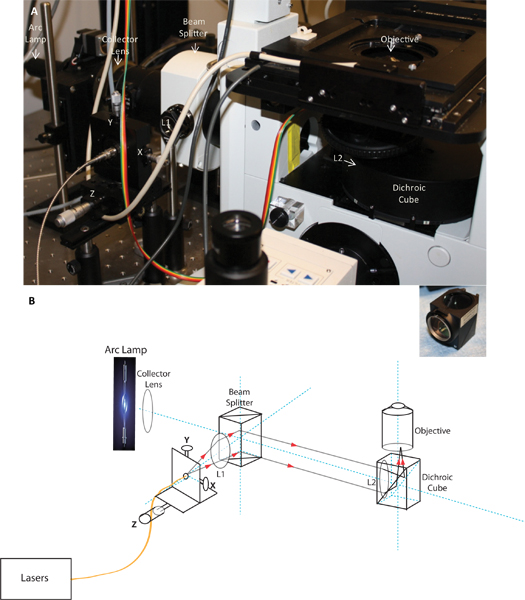
Figura 1. Imagem e esquemática do lançamento TIRF personalizado. O painel A mostra a fotografia do lançamento TIRF como instalado no microscópio, e Painel B é um diagrama esquemático do lançamento TIRF demonstrando o caminho da luz. O esquemática e imagem mostrando a posição da lente colector, o divisor de feixe, o lançamento de fibra com X, Y e Z ajustamentos, a L1 lentes de colimação, ea focagem da lente L2 instalado no cubo dicróico e com o objectivo TIRF. A inserção mostra a lente de focagem instalado no cubo dicróico.
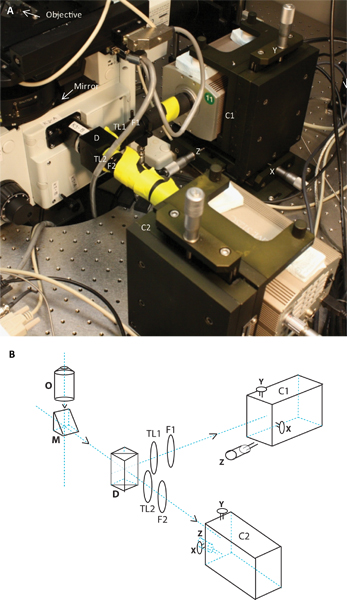
Figura 2. Imagem e esquemática do sistema de câmara de dois. O painel A mostra a fotografia do sistema de câmara de duas como acoplada ao microscópio, e Painel B é um diagrama esquemático do mesmo. O esquema e imagem mostram a posição do objetivo, espelho, dicróica, TL1 e TL2 tubo lentes, filtros de emissão de F1 e F2, as duas câmeras C1 e C2 em therdeiro está com o seu respectivo X, Y e Z ajustes.
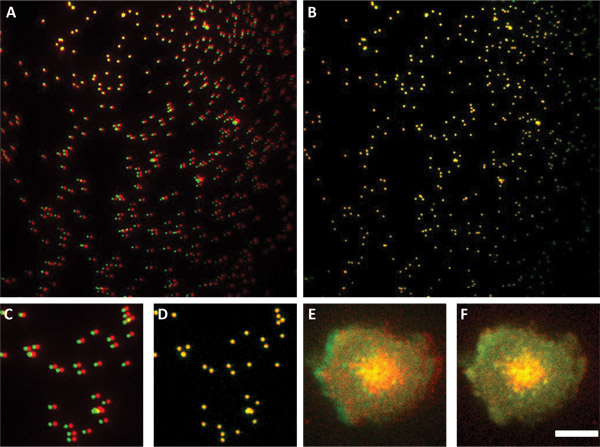
Figura 3. Uso de sub-resolução pérolas para alinhar os dois canais. Painéis A e alinhamento sobreposições mostram B de resolução sub-esferas antes (Painel A) e após (Painel B). O painel A mostra que um dos canais é rodado em relação ao outro. Painéis C e D são uma sub-seção de imagens de A e B. Painel E mostra uma imagem de sobreposição de dois canais de células sem tratamento. Parâmetros de alinhamento utilizados no painel D foram aplicados à imagem mostrada no painel de Aviso F. como o lamelipodium a partir dos dois canais está perfeitamente alinhado em F do painel e não no painel de barra de escala E. 4 mm para todos os painéis.
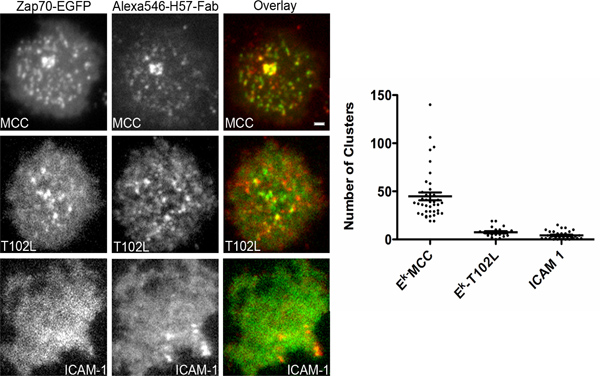
Figura 4. ZAP70 recrutamento para TCR microclusters em resposta a APLs diferentes. In-vitro activado e T As células foram transfectadas com um plasmídeo que codifica ZAP70 fundido a GFP e foram incubadas em vidro apoiados Ni-NTA bicamadas contendo 6 moléculas / iM de péptido carregado 2 His-tag-IE k, 100 moléculas / iM de 2 Alexa647 conjugado His-tag ICAM- 1 e 100 moléculas / mM 2 de GPI-ancorada CD80. Os péptidos carregados estão indicados na inserção. ICAM-1 refere-se a células em bicamadas contendo 100 moléculas / iM de 2 Alexa-647 conjugado His-tag ICAM-1. De canal duplo simultâneo aquisição microscopia TIRF foi realizada na presença contínua de Alexa546 conjugado fragmento Fab H57 (não bloquear) para corar o TCR. As células foram visualizados até uma hora após o contacto inicial com a bicamada. Números de ZAP70 clusters por célula foram analisadas utilizando um software de contagem automatizada cluster. Pelo menos 40 células foram analisadas em cada caso. Barra de escala 2 mm para todas as imagens.
92/3892fig5.jpg "/>
Figura 5. Alinhamento do sistema de câmara dois utilizando dois tipos diferentes de câmaras. Este painel mostra a sobreposição alinhada de resolução sub-contas fotografada usando o sistema de câmara dois, onde as duas câmeras possuem tamanhos de pixel diferentes (verde: 6,45 mM tamanho do pixel fotografado em binning 2x2 e Vermelho: tamanho do pixel 16 mM fotografada sem binning). Inserção mostra a vista ampliada da caixa quadrada no centro do campo. Barra de escala 5 mm.
Filme Suplementar. ZAP70 recrutamento para microclusters TCR em resposta ao peptídeo agonista. In-vitro e as células T activadas foram transfectadas com um plasmídeo que codifica ZAP70 fundido a GFP e foram incubadas em vidro apoiados Ni-NTA bicamadas contendo 6 moléculas / iM de péptido carregado 2 His-tag-IE k, 100 moléculas / iM de 2 Alexa647 conjugado His-tag ICAM-1 e 100 moléculas / 2 mM de GPI-ancorada CD80. Canal duplo simultâneo umaQUISIÇÃO microscopia TIRF foi realizada na presença contínua de Alexa546 conjugado fragmento Fab H57 (não bloquear) para corar o TCR. Um campo de células transfectadas foi repetidamente fotografada 40 vezes com uma resolução de tempo de 10 segundos por quadro. Barra de escala 2 micra. Clique aqui para ver filme suplementar .
Discussão
Descrevemos aqui um sistema para estudar a sinalização em antigénio específicos células de ratinho primárias T utilizando TIRFM e bicamadas lipídicas de vidro com suporte como APCs artificiais. A técnica baseia-se com sucesso expressando as proteínas GFP etiquetados nestas células. Transfecção de células T é sempre uma tarefa desafiadora. Tipicamente, electroporação ou gene de entrega utilizando retrovírus ou lentivírus são utilizados. Uma técnica não é superior em relação ao outro, ambos têm as suas limitações e vantagens. Descobrimos que a electroporação tem as seguintes vantagens: 1) Não é necessário que as células se dividem activamente como é exigido para os retrovírus, mas não lentivírus, e 2) O nível de expressão pode ser controlada através da variação do tempo as células são incubadas antes de imagem. A maior desvantagem é que achamos muito difícil expressar as proteínas que são de tamanho grande em células primárias. Nós apresentamos um método que nos dá a viabilidade celular muito bom após a eletroporação. Como um resultado disso, é applicable a usá-la para alcançar siRNA silenciamento gênico mediado.
Também descrevemos aqui uma personalização de dois canais microscópio TIRF simultânea aquisição que é cromaticamente corrigida e não requer alinhamento separado para comprimentos de onda diferentes. Estas capacidades, no entanto, estão disponíveis comercialmente. Nosso sistema é baseado em princípios de microscopia TIRF publicado por especialistas na área e é mais barato do que as alternativas comerciais. Uma possível crítica de nosso projeto é que nós estamos usando o mesmo ângulo de incidência para os comprimentos de onda de excitação diferentes. Isto resultaria em uma profundidade de penetração diferente TIRF para comprimentos de onda de excitação diferentes. Pensamos que estes efeitos são pequenas, porque a onda evanescente é um campo exponencialmente em decomposição e da profundidade do campo TIRF é uma função linear de comprimento de onda. Fluoróforos próximas da superfície de vidro irá experimentar qualquer diferença na intensidade do laser entre os dois comprimentos de onda. Para fluoróforos perto do PEprofundidade netration, a diferença de intensidade será 1,3 vezes maior para os dois comprimentos de onda 488 e 640 nm e 1,15 de dobragem para os dois comprimentos de onda 488 e 561 nm. O mesmo sistema pode ser utilizado em conjunto com técnicas de resolução super que fazem uso de iluminação TIRF.
Nós também descrito um sistema de câmara de duas, que é construído personalizado. Tal como o sistema TIRF, aparelhos semelhantes estão também comercialmente disponíveis. Nosso sistema oferece flexibilidade para mudar os filtros e dicróicas, permitindo sua utilização com diferentes combinações de comprimentos de onda. O inconveniente do nosso sistema é o grau de rotação um necessária para alinhar as duas imagens. Este problema pode ser resolvido usando suportes da câmara que são acionados piezo e oferecem graus de rotação do alinhamento. Também implementada com sucesso um sistema de câmara de dois sobre este microscópio, no qual as câmaras são diferentes e têm tamanhos de pixel diferentes. Um sistema como este seria útil se uma necessária sensibilidade em um canal e um grande dyfaixa dinâmica em outro, ambos os quais são raramente disponível na mesma câmara. Utilizou-se o Photometrics câmara HQ-2 a binning 2x2 nos dando um tamanho de pixel eficaz de 12,9 uM com a câmara de Photometrics Quant-EM que tem um tamanho de pixel de 16 uM. Um tubo de 180 milímetros lente de distância focal foi utilizado para a Quant-EM e uma lente 145 milímetros de comprimento focal tubo foi utilizado para a câmara HQ-2. A HQ-2 foi controlado usando Metamorph software eo Quant-EM foi controlada através do gestor de micro-software em um computador separado no modo de disparo externo. O gatilho externo foi fornecido com a câmera Quant-EM utilizando uma saída digital da placa DAC medição de informática, que foi implementado como um obturador adicional nas configurações configure iluminação de Metamorph. A razão entre as distâncias focais das lentes de tubo corresponde a razão entre os tamanhos de pixel eficazes. Um alinhamento representativo é mostrado na Figura 5.
Divulgações
Todos os animais utilizados nos experimentos foram mantidos em um ambiente livre de patógenos específicos, e os experimentos foram aprovadas pelos Institutos Nacionais de Saúde Animal e Uso Comissão.
Agradecimentos
Esta pesquisa foi apoiada pela Divisão de Investigação intramuros do Instituto Nacional de Alergia e Doenças Infecciosas, National Institutes of Health. Somos gratos a Johannes Huppa e Mark Davis para fornecer-nos com o scFv de H57 usada nesses estudos. RV gostaria de agradecer Keir Neumann para discussão útil durante o desenvolvimento desta técnica.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| Amaxa Nucleofector Kit para células T rato | Lonza Inc. | VPA-1006 | |
| PureLink HiPure Plasmid Midiprep Kit | Life Technologies | K2100-05 | Para preps endotoxinas livres de DNA |
| Amaxa dispositivo Nucleofector | Lonza Inc. | AAD-1001 | |
| Recombinante IL-2 murina | Peprotech Inc. | 212-12 | |
| Fluoresbrite Multifluorescent Microesferas 0.20μm | Polysciences, Inc. | 24050-5 | |
| Lab-Tek II 8-bem câmaras lamela | Thermo Scientific, Nunc | 155409 |
Referências
- Call, M. E., Wucherpfennig, K. W. The T cell receptor: critical role of the membrane environment in receptor assembly and function. Annu. Rev. Immunol. 23, 101-125 (2005).
- Smith-Garvin, J. E., Koretzky, G. A., Jordan, M. S.T cell activation. Annu. Rev. Immunol. 27, 591-619 (2009).
- Bezbradica, J. S., Medzhitov, R. Integration of cytokine and heterologous receptor signaling pathways. Nat. Immunol. 10, 333-339 (2009).
- Fraser, I. D., Germain, R. N. Navigating the network: signaling cross-talk in hematopoietic cells. Nat. Immunol. 10, 327-331 (2009).
- Dustin, M. L., Starr, T., Varma, R., Thomas, V. K. Supported planar bilayers for study of the immunological synapse. Curr. Protoc. Immunol. Chapter 18, 13-13 (2007).
- Vardhana, S., Dustin, M. Supported Planar Bilayers for the Formation of Study of Immunological Synapses and Kinapse. J. Vis. Exp. (19), e947-e947 (2008).
- Varma, R., Campi, G., Yokosuka, T., Saito, T., Dustin, M. L. T cell receptor-proximal signals are sustained in peripheral microclusters and terminated in the central supramolecular activation cluster. Immunity. 25, 117-127 (2006).
- Yokosuka, T. Newly generated T cell receptor microclusters initiate and sustain T cell activation by recruitment of Zap70 and SLP-76. Nat. Immunol. 6, 1253-1262 (2005).
- Huppa, J. B. TCR-peptide-MHC interactions in situ show accelerated kinetics and increased affinity. Nature. 463, 963-967 (2010).
- Tolar, P., Pierce, S. K. A conformation-induced oligomerization model for B cell receptor microclustering and signaling. Curr. Top. Microbiol. Immunol. 340, 155-169 (2010).
- Treanor, B. The membrane skeleton controls diffusion dynamics and signaling through the B cell receptor. Immunity. 32, 187-199 (2010).
- Treanor, B., Batista, F. D. Mechanistic insight into lymphocyte activation through quantitative imaging and theoretical modelling. Curr. Opin. Immunol. 19, 476-483 (2007).
- Sylvain, N. R., Nguyen, K., Bunnell, S. C. Vav1-mediated scaffolding interactions stabilize SLP-76 microclusters and contribute to antigen-dependent T cell responses. Sci. Signal. 4, ra14-ra14 (2011).
- Purbhoo, M. A. Dynamics of subsynaptic vesicles and surface microclusters at the immunological synapse. Sci. Signal. 3, ra36-ra36 (2010).
- Lasserre, R. Ezrin tunes T-cell activation by controlling Dlg1 and microtubule positioning at the immunological synapse. EMBO. J. 29, 2301-2314 (2010).
- Balagopalan, L., Sherman, E., Barr, V. A., Samelson, L. E. Imaging techniques for assaying lymphocyte activation in action. Nat. Rev. Immunol. 11, 21-33 (2011).
- Axelrod, D. Chapter 7: Total internal reflection fluorescence microscopy. Methods Cell. Biol. 89, 169-221 (2008).
- Williamson, D. J. Pre-existing clusters of the adaptor Lat do not participate in early T cell signaling events. Nat. Immunol. 12, 655-662 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados