Method Article
Una tecnica di microscopia TIRF per Real-time, Imaging simultanea del TCR e delle sue proteine associate segnalazione
In questo articolo
Riepilogo
La compartimentazione delle proteine sia all'interno della membrana plasmatica o in sedi intracellulari è un meccanismo di regolazione che possono influenzare notevolmente i risultati di segnalazione, di conseguenza, per comprendere la segnalazione è importante studiare il comportamento spaziale e temporale delle proteine coinvolte. Descriviamo qui un sistema basato TIRF microscopia a studiare trasduzione del segnale in cellule T, ma è ampiamente applicabile.
Abstract
Signaling viene avviata tramite il recettore delle cellule T (TCR), quando è impegnato da frammenti di peptidi antigenici legati da Complesso Maggiore di Istocompatibilità (pMHC) proteine espresse sulla superficie delle cellule presentanti l'antigene (APC). Il complesso TCR è composto dell'eterodimero TCRαβ legame ligando che associa non covalentemente con CD3 dimeri (e le εδ εγ eterodimeri e la ζζ omodimero) 1. Dopo l'impegno del recettore, le catene zeta CD3 sono fosforilate chinasi della famiglia Src, Lck. Questo porta al reclutamento della famiglia Syk chinasi, ZAP70, che viene poi fosforilato e attivato da Lck. Dopo di che, l'adattatore ZAP70 fosforila proteine LAT e SLP76, avviare la formazione del complesso prossimale segnalazione contenente un gran numero di differenti molecole di segnalazione 2.
La formazione di questo complesso alla fine i risultati di calcio e di Ras-dipendente TRAnscription attivazione del fattore e la conseguente apertura di una complessa serie di programmi di espressione genica che danno luogo a differenziazione delle cellule T 2. Segnali TCR (e la conseguente stato di differenziazione) sono modulati da molti fattori, compresi potenza antigene e diafonia con co-stimulatory/co-inhibitory, chemochine, recettori di citochine e 3-4. Studiare l'organizzazione spaziale e temporale del complesso di segnalazione prossimale in condizioni di stimolazione è, quindi, la chiave per comprendere il percorso di TCR di segnalazione così come la sua regolamentazione attraverso vie di segnalazione di altri.
Un sistema modello molto utile per studiare segnalazione avviata dal TCR a livello della membrana plasmatica di cellule T è vetro supportate bistrati lipidici, come descritto in precedenza 5-6. Possono essere utilizzati per presentare pMHC complessi antigenici, adesione, e molecole co-stimolatorie di cellule T-serve come APC artificiali. Con imaging delle cellule T interagendo con labistrato lipidico totale utilizzando microscopia a fluorescenza riflessione interna (TIRFM), possiamo limitare la eccitazione di 100 nm all'interno dello spazio tra il vetro e la superficie cellulare 7-8. Questo ci permette di immagine in primo luogo gli eventi di segnalazione che si verificano a livello della membrana plasmatica. Come ci interessa nell'imaging l'assunzione di proteine di segnalazione al complesso TCR, descriviamo due telecamera TIRF sistema di imaging in cui il TCR, marcato con Fab fluorescente (frammento legante l'antigene) frammenti di anticorpo H57 (purificato da ibridoma H57-597 , ATCC, ATCC Number: HB-218) che è specifico per TCRβ, e proteine di segnalazione, etichettato con GFP, può essere ripreso simultaneamente e in tempo reale. Questa strategia è necessaria a causa della natura altamente dinamica sia delle cellule T e degli eventi di segnalazione che si verificano al TCR. Questa modalità di imaging ha permesso ai ricercatori di ligandi di immagini singole 9-11, nonché all'assunzione di molecole di segnalazione ai recettori attivati eè un sistema eccellente per studiare biochimica in-situ 12-16.
Protocollo
Procedura sperimentale:
1. Trasfezione con Amaxa mouse cellule T Nucleofector Kit
Descriviamo qui la transfezione di ingenui o attivate in vitro le cellule T primarie con plasmidi di espressione che codificano molecole di segnalazione taggati con GFP, utilizzando il mouse Amaxa cellule T Nucleofector Kit. In vitro attivazione delle cellule T mediante APC spleniche peptide-caricati viene eseguita come descritto prima 7. Tutte le cellule T utilizzati nei nostri studi esprimere la E TCR che riconosce MCC peptide (88-103) legato alla molecola MHC IE k. Trasfezione viene effettuata essenzialmente secondo le raccomandazioni del fabbricante, tuttavia, si presentano alcuni suggerimenti che promuovono la vitalità delle cellule T dopo la trasfezione. Sia la redditività e l'efficienza di trasfezione è molto più alta in vitro in cellule T attivate che in cellule naive.
- Prima di iniziare, preparare 2 ml di mezzo di cellule T integrato con l'aggiunta di 20 microlitri dientrambi i componenti del mezzo A e B e 100 pl di siero fetale bovino (5%) per ogni trasfezione da eseguire. Equilibrare il mezzo a 12-pozzetti in un umidificata 37 ° C, 5% CO 2 incubatore per almeno un'ora. In alternativa, aliquote di mezzo supplementato con 5% di siero e Componente A possono essere preparati e conservati congelati. Se questo è il caso, scongelare il numero richiesto di aliquote, aggiungere il componente B, ed eseguire l'equilibratura.
- Pre-equilibratura del mezzo è molto importante, in quanto morte cellulare risulteranno dalla risospensione delle cellule dopo la trasfezione in mezzo che è freddo o ha un pH diverso da 7,2.
- Quindi, preparare la miscela di trasfezione. Combinare 82 microlitri di soluzione di topo Nucleofector cellula T con 18 pl di Supplemento 2 e 5μg di DNA plasmidico (concentrazione non inferiore a 0,5 mg / pl). Mescolare accuratamente toccando il tubo o con l'uso di micro pipetta.
-È importante eseguire una titolazione della dose di plasmide per un numero fisso di celle per ogni costrutto da utilizzare per la trasfezione. Per alcuni costrutti più grandi, più DNA può essere richiesto. DNA dovrebbe essere libero di endotossine.
- Trasferire 5 a 10 milioni di cellule in una provetta da centrifuga da 15 mL. Centrifugare a 600 rpm per 5 minuti a temperatura ambiente. Rimuovere tanto del supernatante possibile con un aspiratore.
- I produttori consiglia di girare le cellule a meno di 90x g; nel nostro caso, questa velocità corrisponde a 75x g. La scelta forza centrifuga relativa (RCF) per ottenere un pellet di cellule è forse la variabile più importante, come centrifugazione a bassa RCF impedisce danno alla membrana cellulare.
- Risospendere il pellet cellulare nella miscela di trasfezione delicatamente e lentamente Pipette il pellet di cellule fino a quando non ci sono grandi ammassi di cellule rimanenti. In genere, può essere compiuta da aspirare completamente le cellule Tttraverso la punta della pipetta non più di 3-4 volte.
- Utilizzando una pipetta, trasferire accuratamente la sospensione in una cuvetta Amaxa certificata, assicurando che non ci siano bolle presenti. Tappare la cuvetta.
- Selezionare il programma Nucleofector X-001 sul dispositivo Nucleofector. Inserire la cuvetta nel porta cuvette Nucleofector e applicare il programma selezionato.
- Dopo electroporating le cellule, è meglio trasferire immediatamente al pre-equilibrata terreno di coltura. I produttori raccomandano che le cellule essere tenuti in mezzo nucleofector per non più di 15 minuti.
- Portare la cuvetta e pre-equilibrata, terreno di coltura completamente integrata nella cappa. Utilizzando la pipetta di plastica forniti, aggiungere circa 500 pl di mezzo per la cuvetta e trasferire la sospensione cellulare per mezzo della piastra goccia a goccia.
- Incubare le cellule a 37 ° C per 4 ore prima di imaging. Cellule naïve sono incubated a 30 ° C invece di 37 ° C. Questo aiuta a mantenere la loro reattività all'antigene.
- Da tre a quattro ore di incubazione sono generalmente sufficienti per cellule T di esprimere proteine rivelabile la GFP etichetta. Abbiamo scelto di celle di immagine a questo punto del perché il livello di espressione è basso e manufatti di sovra-espressione dovrebbe essere ridotto al minimo. Se sovra-espressione di una proteina particolare si desidera, tuttavia, le cellule T deve essere rimosso dal mezzo Amaxa dopo 4 ore. Questa operazione viene eseguita nuovamente pellettatura le cellule a RCF bassa e risospensione in pre-equilibrata mezzo di coltura cellulare T supplementato con 50 U / ml di interleuchina 2 (IL-2) ad una densità di 2 milioni di cellule / ml.
2. TIRF Microscopia
Descrizione del microscopio TIRF:
- Generali di configurazione: componenti ottici sono stati costruiti intorno ad un microscopio a fluorescenza Olympus IX71 che era originariamente dotata di un modulo Olympus TIRF. Presto discovered effetti cromatici in questo modulo quando uno stato singolo allineamento non ha raggiunto la collimazione coincidente per le linee laser di diverse lunghezze d'onda. Questo modulo, dunque, non consentirebbe l'imaging simultaneo TIRF utilizzando diverse lunghezze d'onda di eccitazione, e abbiamo deciso di costruire un apparato personalizzato TIRF che riduce al minimo gli effetti cromatici nel sistema, come vedremo in dettaglio nelle sezioni seguenti. Il metodo qui descritto è stato sviluppato per l'ingrandimento 150 X, 1,45 apertura numerica (NA) TIRFM obiettivo (Olympus), ma funziona altrettanto bene per l'ingrandimento 60 X, versioni 1.45 NA. Per illuminazione a campo ampio, il microscopio è collegato ad un alogenuro di metallo Lambda-XL sorgente di luce e una ruota filtro di eccitazione (Sutter Instruments) con filtri di eccitazione. Per il posizionamento automatico della fase campione rispetto all'obiettivo, il microscopio è dotato di una traduzione motorizzato XY, piezo-Z controllato stadio (ASI). Le immagini sono state acquisite utilizzando due identiche QuAntem elettroni moltiplicatore (EM) telecamere CCD (fotometrici). La dimensione dei pixel di queste telecamere abbina molto bene con l'ingrandimento offerto dall'obiettivo 150 X TIRF, dando una risoluzione finale di 0,1 micron per pixel. Le telecamere EMCCD offrono anche la sensibilità di visualizzare trasfettanti GFP con basso livello di espressione.
- Laser: Per TIRF illuminazione, un sistema di 5 laser fornire un totale di 6 linee laser (405 nm, 440, 488, 514, 561 e 640) è accoppiata in una fibra monomodale (Solamere Technologies). Il laser ad argon e 561 nm diodo pompato stato solido (DPSS) laser (linee 488, 514 e 561) vengono instradati attraverso un acusto-ottico sintonizzabile fibra (AOTF) per il controllo di intensità, mentre l'intensità del laser a diodi (405, 440 e 640 nm) è controllata modulando la tensione di loro alimentatori. La tensione sul AOTF e le alimentazioni dei laser a diodi è controllata attraverso una scheda di acquisizione dati (DAC) a bordo (Measurement Computing) con MetaMorph software (molecolarelar Devices). L'efficienza di accoppiamento per i laser a diodi è del 30% mentre quelli dei laser DPSS Argon e sono circa il 60% a causa dei loro diametri travi più piccole.
- TIRF modulo: Per simultaneamente l'immagine del TCR e le molecole di segnalazione associati, come descritto in precedenza, era essenziale per avere l'illuminazione TIRF che è stato cromaticamente corretto e che non richiederebbe riallineamento o di riorientamento in caso di acquisizioni delle emissioni di fluorescenza di diverse lunghezze d'onda. TIRFM è stata ottenuta utilizzando passante per l'obiettivo di illuminazione, come descritto in precedenza 17. Il cavo in fibra ottica che produce luce laser al microscopio è stato fissato in una fibra di lancio munito di un detentore delle fibre XY montata su di un micrometro-driven guida ottica per la regolazione Z (Thorlabs). Per ottenere TIRF illuminazione, il fascio laser è focalizzato in corrispondenza del piano focale posteriore dell'obiettivo utilizzando un sistema di due lenti (L1 (lente di collimazione) e L2 (lente di focalizzazione) in figura 1), in modo che la bEAM emerge collimato fuori l'obiettivo. Ciò garantisce che tutti i raggi di luce proveniente dell'obiettivo avrà lo stesso angolo rispetto al piano del campione. La punta della fibra ottica è posizionata al fuoco della lente di collimazione. Spostando il micrometro lungo la rotaia ottico rispetto alla lente di collimazione (direzione Z) è possibile regolare il fuoco del fascio e spostare la trave in posizione X e Y per mezzo delle manopole di regolazione XY. A causa della apertura posteriore molto piccola dell'obiettivo 150 X, abbiamo scoperto che per raggiungere TIRF, la dimensione del punto laser in corrispondenza del piano focale posteriore bisogno di essere piccolo. Per ottenere la focalizzazione necessaria per produrre la piccola macchia, lenti di corta lunghezza focale erano necessarie (Keir Neumann, comunicazione personale), di conseguenza, abbiamo utilizzato una lente con una lunghezza focale di circa 108 mm che è stato posto nel cubo dicroico direttamente precede l'obiettivo. La lente collimatrice è posizionato più vicino alla divisore di fascio che ha portato nel TIRF illuminazione. Per ridurre al minimo indesiderati effetti cromatici, abbiamo usato doppietti acromatici (lente cromaticamente corretto mediante fusione di due elementi ottici realizzati con materiali di diversi indici di rifrazione) con rivestimenti antiriflesso sia per la collimazione e la focalizzazione lenti (JML Optical). Il fuoco può essere verificato controllando che il fascio collimato emerge dalla obiettivo quando posizionato al centro della apertura posteriore e dovrebbe formare un punto stretto sul soffitto della camera. La posizione del punto concentrata viene quindi spostata verso il bordo dell'apertura (nella direzione Y) che provoca il fascio di convergere sul piano immagine a grandi angoli, e infine, l'angolo critico per la riflessione interna totale si ottiene. L'utilizzo di questi componenti ottici, abbiamo raggiunto collimazione simultanea per tutte le lunghezze d'onda da 442 a 640 Nm a lo stesso allineamento. Un allineamento singola ottica, quindi, ci ha permesso di eseguire simultaneamente in tutta TIRFM lunghezze d'onda multiple.
- Camera doppia apparrecchi: acquisizione simultanea delle emissioni distinto fluorescenza derivante da due differenti fluorofori è stato realizzato mediante la costruzione di un sistema a doppia camera che divide l'emissione in due lunghezze d'onda attraverso l'uso di specchi dicroici (Figura 2). La luce emessa è riflessa dall'obiettivo dal porto lato destro del microscopio. Poiché la maggior parte obiettivi sono corretto all'infinito (con immagini formate all'infinito), una lente del tubo è necessario mettere a fuoco l'immagine. Per manipolare due lunghezze d'onda simultaneamente, la lente tubo sulla porta destra è stato rimosso. Una scheda personalizzata contenente c-mount discussioni (Sutter Instruments) è stato attaccato e collegato a un supporto specchio dicroico (Edmund Optics). Specchi dicroici che separano l'emissione GFP da quella di coloranti organici usati per marcare il TCR (Alexa546, Alexa647, ecc) sono stati installati in questo supporto. Questo modulo è stato seguito in entrambi i percorsi di luce da titolare lente del tubo, titolare di un filtro per le emissioni e tubi di prolunga. Illenti tubo (TL1 e TL2 (figura 2)) hanno una lunghezza focale di 180 mm. Le lacune nei tubi di prolunga che portano alle camere CCD EM sono stati coperti con carta nera per proteggere le telecamere dalla luce diffusa. Per consentire l'allineamento dei due canali, le telecamere sono state montate su due stand personalizzati traduzione XYZ (Prodotti Holmarc).
Di seguito viene descritto come allineare il microscopio per l'imaging due canali simultanei. I passaggi necessari sono allineando l'illuminazione TIRF, regolando la potenza del laser, e allineando le due immagini in uscita con risoluzione sub-microsfere.
- Prima di iniziare, tutti i componenti del microscopio sono accesi. Il computer e software vengono poi avviati e si garantisce che tutti i componenti hardware sono riconosciuti dal software.
- Per l'uso come co-localizzazione standard, diluire 1 pl di sub-risoluzione (0,2 micron) Fluoresbrite Multi-microsfere fluorescenti in 2 ml di PBS e aggiungere 0,25 ml della microsferasospensione ciascun pozzetto di una Lab-Tek II 8-ben chambered sistema coverglass (Nunc). Posizionare il sistema di scorrimento della camera sulla piattaforma sopra l'obiettivo. Perline che sono adsorbiti al vetro vengono quindi messo a fuoco.
- Utilizzando l'allineamento Y del lancio fibra XYZ, la posizione del raggio laser viene spostato verso il centro dell'apertura posteriore dell'obiettivo. È importante fare questo quando l'obiettivo è nella posizione in cui il piano in vetro è a fuoco. Se il fascio non è collimata, il micrometro Z è regolata per ottenere collimazione piena. Collimazione viene collaudato singolarmente per tutte le linee laser da utilizzare nell'esperimento. In questa fase, la potenza del laser per ogni linea viene misurata utilizzando un misuratore di potenza laser e viene regolata a 20-30 μW in ogni canale.
T-cellule sono estremamente sensibili alle radiazioni laser, e l'illuminazione è limitata ad un totale di 50 μW.
- Le immagini delle perline vengono acquisite utilizzando TIRF Illuminationi in modalità live. Ruotare il Y-allineamento micrometro il lancio fibra in modo che il fascio inizia a muoversi lontano dal soffitto fino suo angolo rispetto ad approcci asse ottico obiettivo di 90 gradi. Infine, quando l'angolo critico per TIR viene raggiunto, la luce laser non sarà più uscire obiettivo. Allineamento TIRF viene poi giudicato, concentrandosi su e giù attraverso il piano del vetrino coprioggetti. Se perline che galleggiano in soluzione può essere visualizzata, quindi l'angolo del fascio rispetto alle normali esigenze oggettive essere ulteriormente aumentato. Al corretto allineamento TIRF, solo piano ottico sarà a fuoco (cioè, solo le perle che sono adsorbiti al vetrino). Un test più fine di allineamento TIRF può essere attuato quando l'imaging cellule T da un colorante superficie che hanno diffuso su una superficie. (Cfr. punto 3.4).
- Dopo illuminazione TIRF è stato raggiunto, i due canali possono essere allineate con uno rispetto all'altro. Concentrati sulle perline e confrontare la posizione di identifiable caratteristiche delle immagini prodotte in entrambi i canali. Utilizzo X e Y viti micrometriche su un supporto della videocamera, portare la posizione relativa delle perline in quanto vicino possibile. Salvare le immagini da entrambi i canali. Queste immagini saranno utilizzati come co-localizzazione standard per allineare i due canali.
- Le telecamere devono anche essere allineato in modo che siano parafocale rispetto all'altro. Ciò è ottenuto mediante l'acquisizione di una serie di immagini delle perle sub-risoluzione che sono separati l'uno dall'altro nella direzione Z di 100 nm. Se il pixel intensità massima per le perline giace nello stesso piano per entrambe le telecamere, allora sono parafocale.
3. Imaging
Una volta che il microscopio è costituito, il passo successivo è quello di preparare camere di flusso di vetro contenenti doppi strati lipidici supportati molecole che presentano l'antigene e l'adesione come descritto in precedenza 6 e quindi eseguire TIRFM delle cellule T trasfettate che interagiscono con ilsubstrato. Il protocollo pubblicato bistrato 5-6 è stata seguita come pubblicato eccetto che i liposomi non sono state incubate per 20 minuti sulla coverglass dopo la camera di flusso è stato assemblato. Invece, HBS-BSA tampone è stato fatto fluire nella cella di flusso immediatamente dopo l'assemblaggio.
- Assemblare una camera di flusso con doppi strati lipidici planari formate sulla bolla di vetro contenente Ni-NTA lipidi. Molecole di adesione, ICAM-1 (100 molecole / um 2), e peptide-MHC complessi, MCC caricato IE k (molecole di 5-10 um / 2), sono incorporati nel bistrato via loro tag istidina. La molecola di costimolazione CD80 viene aggiunta usando un GPI (100 molecole / um 2).
- Posizionare la cella di flusso sulla scena del microscopio e stabilizzarlo. Attaccare il produttore fornito elemento riscaldante sulla cella di flusso per controllare la temperatura. Posizionare l'obiettivo su un doppio strato e portare l'aereo in doppio strato a fuoco. Abilita riscaldamento l'obiettivo e la portatacella per stabilizzare la temperatura della camera a 37 ° C.
- Trasfettate cellule T sono pellettizzate dopo 4 ore di incubazione a una velocità di 80x g e risospese in tampone HBS-BSA e integrato con 10 pg / mL di H57 Fab o 20 pg / mL di H57 singolo frammento variabile a catena (scFv) a etichettare la TCR. Le cellule sono poi iniettato nelle celle di flusso. Imaging viene fatto in presenza continua di Fab o scFv grazie alla loro elevata costante di dissociazione. La loro presenza nel mezzo non aumenta significativamente il fondo come il campo TIRF è molto sottile.
- Condizioni di ripresa sono impostati in modo che l'acquisizione di due canali in diretta mostra un'anteprima in tempo reale dei canali TCR e GFP che aiuta nella ricerca di cellule trasfettate. TIRF illuminazione è confermato dal concentrando il piano di vetro per rilevare alcuna colorazione laterale membrana. Se le membrane laterali non arrivano a fuoco quindi l'allineamento TIRF è buona. Le cellule trasfettate sono poi fotografato una volta o in una sequenza di immagini per produrre videos.
4. Image Processing e Analisi dei Dati
Il software genera le immagini delle due telecamere in un singolo fotogramma. L'uscita di ogni telecamera è di 512 x 512 pixel, di conseguenza, l'immagine in uscita è di 1024 x 512 pixel. Le prime immagini devono essere suddiviso in singoli fotogrammi corrispondenti alle due telecamere. Le immagini di sub-risoluzione perle dei due canali sono sovrapposti e sono determinati parametri di allineamento. Una caratteristica specifica per il nostro sistema è un 1 ° di rotazione di un canale rispetto all'altro (vedere Figura 3). La fonte più probabile di questa rotazione è sugli spalti della fotocamera. Dopo tale rotazione trasformazioni lineari ulteriori relativi in X e Y devono essere svolte per allineare perfettamente le microsfere con uno rispetto all'altro. Le immagini di perle vengono ottenuti in ogni esperimento per determinare i parametri precisi per allineare i due canali. Queste trasformazioni lineari e di rotazione vengono poi applicati a tuttiimmagini e quindi essi sono soggetti ad algoritmi di segmentazione e di co-localizzazione di analisi.
5. Risultati rappresentativi
Il sistema qui descritto può essere adattato per studiare valle segnalazione di qualsiasi recettore alla membrana plasmatica. E 'particolarmente informativo in studio TCR di segnalazione in quanto la segnalazione avviene in gruppi otticamente risolti chiamate TCR microcluster o in endosomi, come recentemente descritto 18. Se la segnalazione si è verificato in risoluzione sub-gruppi di recettori o in un-cluster recettori ulteriori esperimenti avrebbe dovuto essere fatto per interpretare i dati. Come già accennato, le catene CD3 del complesso TCR subiscono fosforilazione di Lck chinasi della famiglia Src sulla impegno del TCR di peptide-MHC complessi. La catena CD3ζ fosforilato recluta la chinasi della famiglia Syk ZAP70. Visualizzare il reclutamento di Zap70 riferisce quindi sullo stato di fosforilazione di catena CD3ζ. La forza di stimolazione TCR possono essere modificati mediante peptidi mutati a livello dei residui di contatto dei TCR. Tali peptidi sono chiamati ligandi peptidici alterati (APL). Un esperimento rappresentativo è mostrato in Figura 4, dove i peptidi agonisti recluta robusta ZAP70 ai microcluster TCR, mentre minore è la potenza peptide T102L non riesce a reclutare ZAP70 al TCR microcluster, dimostrando che la catena CD3ζ non è fosforilata in questo caso. Potremmo trarre questa conclusione, perché abbiamo dovuto osservare solo il reclutamento di ZAP70 ai cluster TCR che sono stati facilmente individuati dai TIRFM. Un algoritmo di segmentazione automatica è stato utilizzato per contare il numero di ZAP70 microcluster per cella. Anche mostrato nel film supplementare è l'imaging simultanea di TCR e ZAP70 fluorescenza associata. Si noti la natura dinamica del lamelipodium.
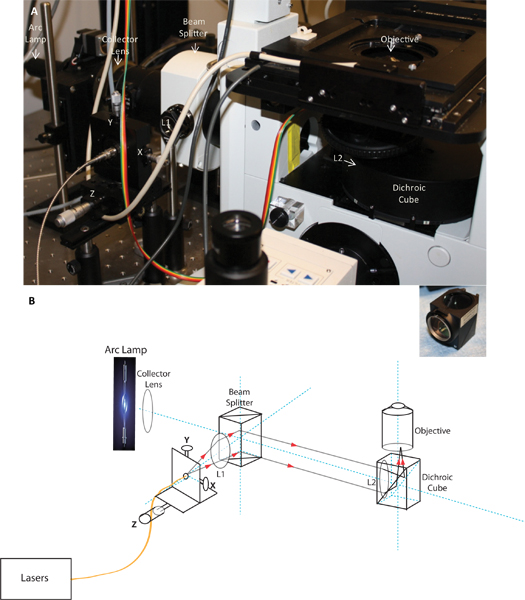
Figura 1. immagine e schematica del lancio TIRF personalizzato. Pannello A mostra la fotografia del lancio TIRF come installato sul microscopio, e pannello B è uno schema di lancio TIRF dimostrare il percorso della luce. L'immagine e schematica che mostra la posizione della lente collettore, il divisore di fascio, il lancio fibra con X, Y e Z regolazioni, la lente collimatrice L1, e la lente di focalizzazione L2 installato nel cubo dicroico e l'obiettivo TIRF. L'inserto mostra la lente di focalizzazione installato nel cubo dicroico.
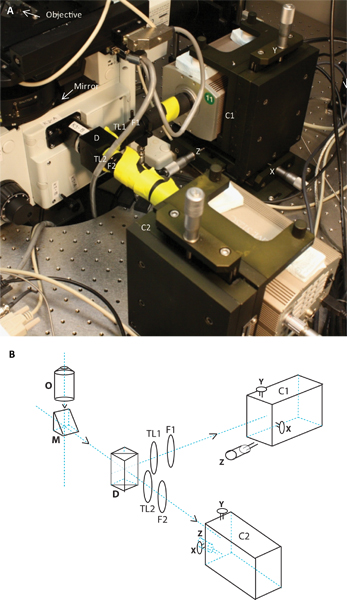
Figura 2. Immagine e schematica del sistema di due telecamere. Pannello A mostra la fotografia del sistema a due fotocamera collegata al microscopio, e pannello B è una vista schematica dello stesso. L'immagine e schematica mostrano la posizione dell'obiettivo, specchio, dicroico, lenti tubo TL1 e TL2, emissione filtri F1 e F2, le due telecamere C1 e C2 su terede sta con loro rispettivi X, Y e Z regolazioni.
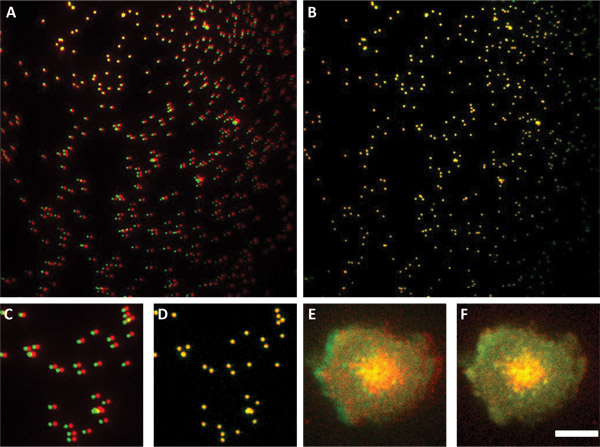
Figura 3. Uso della sub-risoluzione perline per allineare i due canali. Pannelli A e B mostrano sovrapposizioni di sub-risoluzione perline prima (pannello A) e dopo (Panel B) allineamento. Pannello A mostra che uno dei canali è ruotato rispetto all'altro. I pannelli C e D sono una sezione sub delle immagini in A e B. E Panel mostra una sovrapposizione di immagini due canali delle cellule senza trattamento. Parametri utilizzati in allineamento pannello D sono stati applicati all'immagine mostrata in comunicazione pannello F. come il lamelipodium dai due canali è perfettamente allineato in F pannello e non in bar pannello E. Scala 4 um per tutti i pannelli.
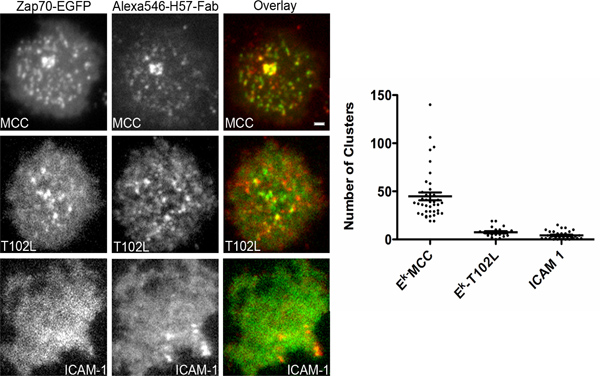
Figura 4. ZAP70 reclutamento TCR microcluster in risposta a diversi APL. In-vitro attivato e T cellule sono state trasfettate con un plasmide codificante ZAP70 fuso GFP e sono stati incubati su vetro supportate Ni-NTA bistrati contenente 6 molecole / 2 micron di peptide caricato His-tag k-IE, 100 molecole / 2 micron di Alexa647 coniugato His-tagged ICAM- 1 e 100 molecole / um 2 di GPI-ancorata CD80. I peptidi caricati sono indicati nel cestello. ICAM-1 si riferisce a cellule di bistrati contenenti 100 molecole / um 2 di Alexa-647 coniugato His-tag ICAM-1. Doppio canale di acquisizione simultanea di microscopia TIRF è stata eseguita in presenza continua di Alexa546 coniugato H57 frammento Fab (non-blocking) per colorare il TCR. Le cellule sono state ripreso fino ad un'ora dopo il contatto iniziale con il doppio strato. Numero di ZAP70 cluster per cella sono stati analizzati utilizzando un sistema automatico di conteggio software cluster. Almeno 40 cellule sono state analizzate in ciascun caso. Scala grafica 2 pm per tutte le immagini.
92/3892fig5.jpg "/>
Figura 5. Allineamento del sistema a due telecamera utilizzando due diversi tipi di telecamere. Questo pannello mostra la sovrapposizione allineata di sub-risoluzione perline fotografato utilizzando il sistema a due camera in cui le due fotocamere hanno dimensioni di pixel differenti (Verde: 6,45 micron ripreso dimensione del pixel a binning 2x2 e Rosso: 16 micron pixel size ripreso senza binning). Inserto mostra la vista ingrandita della scatola quadrato al centro del campo. Scala grafica 5 micron.
Movie supplementare. ZAP70 reclutamento microcluster TCR in risposta al peptide agonista. In-vitro attivate e le cellule T sono state trasfettate con un plasmide codificante ZAP70 fuso GFP e sono stati incubati su vetro supportate Ni-NTA bistrati contenente 6 molecole / 2 micron di peptide caricato His-tag k-IE, 100 molecole / 2 micron di Alexa647 coniugato His-tagged molecole ICAM-1 e 100/2 micron di GPI-ancorata CD80. Dual channel simultanea unocquisition microscopia TIRF è stata eseguita in presenza continua di Alexa546 coniugato H57 frammento Fab (non-blocking) per colorare il TCR. Un campo di cellule trasfettate è stato ripreso più volte 40 volte con una risoluzione temporale di 10 secondi per fotogramma. Scala grafica 2 micron. Clicca qui per vedere il film supplementari .
Discussione
Descriviamo qui un sistema di segnalazione per studiare in antigene-specifiche cellule T primarie topo usando TIRFM e bistrati lipidici vetro supportate come APC artificiali. La tecnica si basa sul successo esprimenti proteine GFP etichetta in queste cellule. Trasfezione delle cellule T è sempre un compito impegnativo. Tipicamente, consegna elettroporazione o gene usando retrovirus o lentivirus vengono utilizzati. Una tecnica non è superiore rispetto all'altra, entrambi hanno i loro limiti e vantaggi. Troviamo che elettroporazione presenta i seguenti vantaggi: 1) non è necessario che le cellule attivamente divisione come è richiesto per i retrovirus ma non lentivirus, e 2) Il livello di espressione può essere controllato variando il tempo di incubazione le cellule sono prima di imaging. Il più grande svantaggio è che ci trovano molto difficile esprimere proteine che sono di grandi dimensioni in cellule primarie. Abbiamo presentato un metodo che ci dà la vitalità cellulare molto buono dopo l'elettroporazione. Come risultato è applicable di usarlo per raggiungere mediata silenziamento del gene siRNA.
Noi descriviamo anche qui una personalizzata due canali simultanei microscopio TIRF acquisizione che è cromaticamente corretto e non richiede l'allineamento separato per diverse lunghezze d'onda. Queste funzionalità, tuttavia, sono disponibili commercialmente. Il nostro sistema si basa su principi di microscopia TIRF pubblicato da esperti del settore ed è più economica rispetto alle alternative commerciali. Una possibile critica del nostro progetto è che stiamo usando lo stesso angolo di incidenza per le diverse lunghezze d'onda di eccitazione. Ciò comporterebbe una diversa profondità di penetrazione TIRF per lunghezze d'onda di eccitazione differenti. Riteniamo che questi effetti sono piccole perché l'onda evanescente è un campo di decadimento esponenziale e la profondità del campo TIRF è una funzione lineare di lunghezza d'onda. Fluorofori vicino alla superficie di vetro non si è avuta differenza di intensità tra i laser due lunghezze d'onda. Per fluorofori vicino al peprofondità netration, la differenza di intensità sarà 1,3 volte per le due lunghezze d'onda 488 e 640 nm e 1,15 volte per il due lunghezze d'onda 488 e 561 nm. Lo stesso sistema può essere utilizzato in combinazione con tecniche di risoluzione super che fanno uso di illuminazione TIRF.
Abbiamo anche descritto un sistema a due fotocamera che è costruito su misura. Come il sistema TIRF, apparati simili sono anche commercialmente disponibili. Il nostro sistema offre flessibilità per cambiare i filtri ei dicroici, consentendone l'uso con diverse combinazioni di lunghezze d'onda. L'inconveniente del nostro sistema è un grado di rotazione necessaria per allineare le due immagini. Questo problema può essere risolto utilizzando stand della fotocamera che vengono guidati piezo e offrono gradi di rotazione di allineamento. Abbiamo anche implementato con successo un sistema a due telecamera sul microscopio, in cui le telecamere sono diversi e hanno dimensioni differenti pixel. Un sistema come questo sarebbe utile se quella necessaria sensibilità in un canale ed un grande dygamma dinamica in un altro, entrambi i quali sono raramente disponibili nella camera stessa. Abbiamo usato la HQ-2 Photometrics fotocamera binning 2x2 darci una dimensione in pixel effettivi di 12,9 micron con la Quant-EM camera Photometrics che ha una dimensione del pixel di 16 micron. A mm 180 focale lunghezza del tubo è stato utilizzato per la Quant-EM e 145 millimetri di lunghezza focale lente del tubo è stato utilizzato per la HQ-fotocamera da 2. La HQ-2 è stato controllato tramite Metamorph software e la Quant-EM è stato controllato tramite micro-manager software su un computer separato in modalità trigger esterno. Il trigger esterno è stato fornito al Quant-EM fotocamera utilizzando un uscita digitale della scheda DAC misura di calcolo, che è stato implementato come uno scatto aggiuntivo di configurazione delle impostazioni di illuminazione di Metamorph. Il rapporto tra le lunghezze focali delle lenti tubo corrisponde al rapporto tra le dimensioni effettive pixel. Un allineamento rappresentante è illustrato nella figura 5.
Divulgazioni
Tutti gli animali utilizzati in questi esperimenti sono state mantenute in uno specifico ambiente privo di agenti patogeni, e gli esperimenti sono stati approvati dal National Institutes of Health Care degli animali e del Comitato uso.
Riconoscimenti
Questa ricerca è stata sostenuta dalla Divisione di Ricerca intramurale dell'Istituto Nazionale per le allergie e malattie infettive, National Institutes of Health. Siamo grati a Johannes Huppa e Mark Davis per averci fornito con il scFv di H57 utilizzato in questi studi. RV ringrazia Keir Neumann di discussione utile durante lo sviluppo di questa tecnica.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| Amaxa Nucleofector Kit per mouse cellule T | Lonza Inc. | VPA-1006 | |
| PureLink HiPure plasmide Midiprep Kit | Life Technologies | K2100-05 | Per le preparazioni di DNA endotossine gratis |
| Amaxa Nucleofector dispositivo | Lonza Inc. | AAD-1001 | |
| Ricombinante murino IL-2 | Peprotech Inc. | 212-12 | |
| Fluoresbrite Multifluorescent Microsfere 0.20μm | Polysciences, Inc. | 24050-5 | |
| Lab-Tek II 8-ben chambered coverglass | Thermo Scientific, Nunc | 155409 |
Riferimenti
- Call, M. E., Wucherpfennig, K. W. The T cell receptor: critical role of the membrane environment in receptor assembly and function. Annu. Rev. Immunol. 23, 101-125 (2005).
- Smith-Garvin, J. E., Koretzky, G. A., Jordan, M. S.T cell activation. Annu. Rev. Immunol. 27, 591-619 (2009).
- Bezbradica, J. S., Medzhitov, R. Integration of cytokine and heterologous receptor signaling pathways. Nat. Immunol. 10, 333-339 (2009).
- Fraser, I. D., Germain, R. N. Navigating the network: signaling cross-talk in hematopoietic cells. Nat. Immunol. 10, 327-331 (2009).
- Dustin, M. L., Starr, T., Varma, R., Thomas, V. K. Supported planar bilayers for study of the immunological synapse. Curr. Protoc. Immunol. Chapter 18, 13-13 (2007).
- Vardhana, S., Dustin, M. Supported Planar Bilayers for the Formation of Study of Immunological Synapses and Kinapse. J. Vis. Exp. (19), e947-e947 (2008).
- Varma, R., Campi, G., Yokosuka, T., Saito, T., Dustin, M. L. T cell receptor-proximal signals are sustained in peripheral microclusters and terminated in the central supramolecular activation cluster. Immunity. 25, 117-127 (2006).
- Yokosuka, T. Newly generated T cell receptor microclusters initiate and sustain T cell activation by recruitment of Zap70 and SLP-76. Nat. Immunol. 6, 1253-1262 (2005).
- Huppa, J. B. TCR-peptide-MHC interactions in situ show accelerated kinetics and increased affinity. Nature. 463, 963-967 (2010).
- Tolar, P., Pierce, S. K. A conformation-induced oligomerization model for B cell receptor microclustering and signaling. Curr. Top. Microbiol. Immunol. 340, 155-169 (2010).
- Treanor, B. The membrane skeleton controls diffusion dynamics and signaling through the B cell receptor. Immunity. 32, 187-199 (2010).
- Treanor, B., Batista, F. D. Mechanistic insight into lymphocyte activation through quantitative imaging and theoretical modelling. Curr. Opin. Immunol. 19, 476-483 (2007).
- Sylvain, N. R., Nguyen, K., Bunnell, S. C. Vav1-mediated scaffolding interactions stabilize SLP-76 microclusters and contribute to antigen-dependent T cell responses. Sci. Signal. 4, ra14-ra14 (2011).
- Purbhoo, M. A. Dynamics of subsynaptic vesicles and surface microclusters at the immunological synapse. Sci. Signal. 3, ra36-ra36 (2010).
- Lasserre, R. Ezrin tunes T-cell activation by controlling Dlg1 and microtubule positioning at the immunological synapse. EMBO. J. 29, 2301-2314 (2010).
- Balagopalan, L., Sherman, E., Barr, V. A., Samelson, L. E. Imaging techniques for assaying lymphocyte activation in action. Nat. Rev. Immunol. 11, 21-33 (2011).
- Axelrod, D. Chapter 7: Total internal reflection fluorescence microscopy. Methods Cell. Biol. 89, 169-221 (2008).
- Williamson, D. J. Pre-existing clusters of the adaptor Lat do not participate in early T cell signaling events. Nat. Immunol. 12, 655-662 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon