É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Photopatterning proteínas e células em Aqueous Ambiente Usando TiO
Neste Artigo
Resumo
We describe a protocol for modifying cell affinity of a scaffold surface in aqueous environment. The method takes advantage of titanium dioxide photocatalysis to decompose organic film in the photo-irradiated region. We show that it can be used to create microdomains of scaffolding proteins, both ex situ and in situ.
Resumo
Os contaminantes orgânicos adsorvidos sobre a superfície de dióxido de titânio (TiO 2) pode ser decomposto por fotocatálise sob luz ultravioleta (UV). Descrevemos aqui um novo protocolo empregando o TiO 2 fotocatálise para alterar localmente afinidade de células da superfície do substrato. Para esta experiência, uma fina película de TiO2 era de uma lamela de vidro revestido por borrifamento, e a superfície de TiO2 foi posteriormente modificado com um derivado de organo-silano monocamada octadecyltrichlorosilane (OTS), que inibe a adesão celular. A amostra foi imersa em um meio de cultura celular, e focada luz UV foi irradiada para uma região octogonal. Quando uma linha celular de células PC12 neuronais foram plaqueadas sobre a amostra, as células aderiram apenas na área de irradiação com UV. Mostramos ainda que esta modificação da superfície pode também ser realizada in situ, ou seja, mesmo quando as células estão em crescimento sobre o substrato. Modificação adequada da superfície necessária uma matriz extracelular protein colagénio de estar presente no meio no momento da irradiação UV. A técnica aqui apresentada pode, potencialmente, ser empregues em vários tipos de células para a construção de sistemas de padronização ou co-cultura para manipular células arbitrariamente sob cultura.
Introdução
Processos de litografia de semicondutores e seus derivados, tais como 1,2 - fotolitografia, litografia por feixe de electrões 3-6, e microcontact impressão 7-10 - tornaram-se agora uma ferramenta estabelecida na biologia das células para crescer as células vivas numa posição definida e geometria. O método baseia-se na modelação da utilização de substratos microfabricados, que consiste em micro-ilha de revestimento celular permissiva em um fundo não permissiva. Tal substrato serve como um modelo padrão para as células. Essas tecnologias nos forneceu os novos métodos para manipular células e sua função em um nível simples e multi-celular, para extrair as propriedades intrínsecas das células, e para aumentar a taxa de transferência com base em células de triagem de drogas 11.
O grau de liberdade na modelação de células aumentariam grandemente, se a geometria do modelo padrão pode ser alterada in situ, ou seja, enquanto que as células são cultivadas sobre o Sseu rosto. Os métodos convencionais para a formação de padrão não pode ser directamente aplicados aqui, uma vez que eles processam amostras em atmosfera ou em vácuo. Por isso têm sido propostos várias novas técnicas de modificação de superfície, que se baseiam, por exemplo, em compostos fotorreactivos 12,13 ou ablação a laser 5,14, apenas para citar alguns. Os métodos propostos foram bem avaliada pelo Robertus et al. 15, e mais recentemente por Choi et al. 16 e 17 por Nakanishi.
Aqui neste artigo, descreve-se um novo protocolo de modificação in situ superfície, o que tira proveito da decomposição fotocatalítica de moléculas orgânicas num dióxido de titânio (TiO2) de superfície 18,19. Neste método, é inserida uma película de TiO2 entre o substrato de vidro e o filme orgânico que faz interface das células, e o filme orgânico é decomposto in situ por localmente irradiação ultravioleta (UV)luz de uma região de interesse (λ <388 nm). Mostra-se que o novo protocolo pode ser utilizado para criar micropadrões de proteínas da matriz extracelular e células vivas tanto ex situ e in situ. TiO 2 é biocompatível, quimicamente estável e opticamente transparentes, recursos de que o torna amigável para introduzir em experimentos de cultura celular. Este protocolo fornece uma alternativa ciência dos materiais à base de andaimes para a modificação de cultura celular no ambiente de cultura de células.
Protocolo
1. Preparação de TiO 2 -Revestido vidro Lamela
- Número as lamelas usando um riscador de diamante. Isso ajuda não só para manter o controle de cada lamelas, mas também para garantir que o lado correto da amostra está virada para cima. Limpar as lamelas, pela primeira vez sob a executar ddH2O, seguida por imersão em solução de piranha (H 2 SO 4: H 2 O 2 = 4: 1). Após 10 minutos, enxaguar as lamelas completamente, 8 vezes em ddH 2 O. Secam-se as lamelas em fluxo de N2.
- Definir TiO2 alvo no sistema de pulverização de rádio-frequência (RF). Anexar as lamelas em um suporte de amostra do dispositivo de pulverização utilizando uma fita de poliimida. Coloque o suporte de amostras na câmara de pulverização. Evacuar a câmara até a pressão atingir 2,0 x 10 -4 Pa.
- Introduzir Ar gasoso para dentro da câmara e ajustar a pressão de deposição a 4,0 mTorr. Enquanto mantém o obturador fechado, aumentar gradualmentea energia RF a 70 W.
- Abrir o obturador e por pulverização catódica durante 15 minutos para se obter uma película com uma espessura de 120-150 nm (Figura 1: Passo A). A velocidade de crescimento do filme necessita de ser derivada para cada máquina.
2. Revestimento de Superfícies com Cell-repelente Film
- Hidrofilizar a superfície de TiO 2, tratando a amostra do plasma com O 2, seguindo as instruções fornecidas pelo fabricante do reactor de plasma. Nós tratar a amostra durante 5 minutos a 200 W com O2 fluxo de 100 sccm. Mergulhe a amostra em DDH 2 O e confirmar que a superfície é superhydrophilic. Secar completamente a superfície sob fluxo de N2.
- Preparar a solução 1 mM octadecyltrichlorosilane (OTS) por adição de 39,6 ul de OTS 100 ml de tolueno. Imergir a amostra na solução durante 1 h à TA. Realizar este passo dentro de um saco de luvas -filled 2 N (Figura 1: Etapa B).
- Para remover moléculas physisorbed, sonicate a amostra em tolueno, acetona, etanol, e ddH2O durante 5 min cada, por essa ordem. Lavar a amostra quatro vezes em fresco ddH2O e secar a superfície sob um fluxo de N2. A superfície deve ser hidrofóbico com um ângulo de contacto de 100-110 °.
3. Ex-Situ Superfície Patterning
- Trabalhando em fluxo laminar capô, desenhe várias marcas de arranhões com um riscador de diamantes na superfície. As marcas de ajudar a manter o controle das regiões processados e também trazendo microscópios em foco. Esteriliza-se o TiO 2 OTS-revestidos por imersão da amostra em etanol a 70% durante 5 min. Em seguida, enxaguar a amostra duas vezes em esterilizado DDH 2 O.
- Colocar a amostra em um prato de 35 mm, e adicionar 2 ml de meio de crescimento PC12 (ver Passo 4.2). Incubar durante mais 3 horas num incubador de CO2 (37 ° C). Este procedimento destina-se a deixar albuminas do soro absorvem na superfície. Albuminas adsorvido inibir a adsorção posterior de outros proteins e células (Figura 1: o passo c).
- Enquanto espera, configurar o microscópio de fluorescência invertido.
- Ligue a lâmpada de arco, insira o cubo filtro UV, e definir a lente objetiva de 20X.
- Medir a intensidade da luz I (W cm-2) utilizando um medidor de intensidade de UV, e calcular o tempo de irradiação t (em segundos) para uma dose de d (em J cm-2) como: t = d / I.
Por exemplo, para irradiar a uma dose de 200 J cm-2 utilizando uma fonte de luz de 600 mW cm de -2, é necessária a irradiação de 333 seg. - Usar um micrómetro de plataforma e fechar o diafragma de campo para definir o tamanho da região a ser irradiada, por exemplo, 200 um.
- Após a incubação de 3 horas, completar o meio com 200 mL de 3,0 mg ml -1 de colágeno tipo IV (Col-IV; finais concentração de 300 ug ml -1).
- Transferir a 35 mm de prato de platina do microscópio. Encontre o zero marca, focar o microscópio sobre a superfície da amostra, e irradiar luz UV, numa dose de 200 J cm-2 (Figura 1: Passo D). A área de irradiação UV pode ser alterado quer através do ajuste da abertura do diafragma de campo ou por substituição do diafragma de campo com uma máscara metálica de geometria de arbitragem.
- Substituir o meio com um meio de crescimento fresco (sem Col-IV), e colocar a amostra de volta na incubadora.
4. Cultura celular
- Cultura de rotina de células PC12 é levada a cabo em um prato de plástico revestidas com colagénio.
- Para revestimento de 60 mm de cultura de tecidos prato com Col-IV, primeiro molhar a superfície com DDH 2 O e aspirar tudo DDH 2 O.
- Preparar 300 ng mL -1 Col-IV por diluição da solução original (3 mg ml-1) 10x com ddH 2 O.
- Adicionar 200 ul de 300 ng mL -1 Col-IV por 60 mm de prato. Deixe a solução se espalhar através da superfície.
- Seco tele prato em uma câmara de fluxo laminar durante aproximadamente 1 h.
- Lavar a superfície duas vezes com 4 ml de solução salina tamponada com fosfato de Dulbecco (D-PBS). Quando não utilizar a placa revestida imediatamente, lavar o prato uma vez com 4 ml de DDH 2 O. Após aspiração DDH 2 O, armazenar o prato na incubadora. Tente não para mantê-lo guardado por mais de duas semanas.
- As células PC12 são cultivadas num meio de crescimento, que consiste em: meio (baixo teor de glucose) de Eagle modificado por Dulbecco solução de penicilina-estreptomicina + 1% de soro + 10% de soro fetal de bovino a 5% + cavalo. Incubar as células em uma incubadora de CO 2 (37 ° C, 5% de CO 2), e substituir metade do meio todos os outros dias. Células de passagem antes que eles atinjam confluência.
- Aspirar o meio de crescimento, e adicionar 2 ml de PBS + (D-PBS + 10 mg mL -1 de albumina de soro bovino (BSA) + EDTA a 10 mM) pré-aquecido a 37 ° C. Incubar durante 5 min a 37 ° C. Bata levemente o prato para retirar todo cells.
- Recolher as células num tubo cónico de 15 ml. Lavar a placa com 3 ml de D-PBS fresco, pré-aquecido a 37 ° C.
- Centrifuga-se o tubo a 150 × g durante 4 min.
- Aspirar o sobrenadante e adicionar 1 ml de meio de crescimento.
- Para cultura de rotina, dividir as células 1: 3 a 1: 5.
- Ao prato células na OTS / TiO 2 amostra modificada-UV, contar densidade de células e adicionar 3,0 × 10 5 células em um prato de 35 mm (Figura 1: Passo E). Incubar a placa na incubadora humidificada (37 ° C, 5% CO2) durante 1-2 dias. Tanto o factor de crescimento ingénuos e do nervo (NGF), as células PC12 -differentiated pode ser usado para as experiências de modelação. Para a diferenciação de NGF, 100 ng ml -1 de 7S-NGF é adicionado ao meio o crescimento de vários dias antes do plaqueamento das células na amostra. NGF-diferenciação parece aumentar a adhesibility das células PC12 e torna o manuseamento mais fácil, especialmente no in situ e padronizaçãoxperiments.
5. In Situ superfície de modelação
- Após 1-2 dias de cultura na superfície do ex-situ modificado, confirmar que as células estão a crescer e anexar apenas na região do irradiadas com UV. Defina-se o microscópio, conforme descrito no Passo 3.4.
- Transferir a amostra para um novo 35-mm de placa de cultura de tecidos contendo o meio de crescimento, 100 ng ml -1 de NGF (no caso da utilização de células diferenciadas por NGF), e 100? G ml -1 Col-IV.
- Coloque o prato no palco microscópio. Localizar a posição adequada e irradiar luz UV, numa dose de 200 J cm-2 (Figura 1: Passo F, G). A região permissiva recém-criado é indicado com um asterisco na Figura 1: Passo G.
- Tente completar o processamento de uma única amostra no prazo de 30 min. Após a irradiação, transferir a amostra de volta para o meio sem Col-IV.
- Tenha muito cuidado ao transportar o prato com cucélulas ltured e transferir a amostra do prato de prato para evitar desanexação das células estampados.
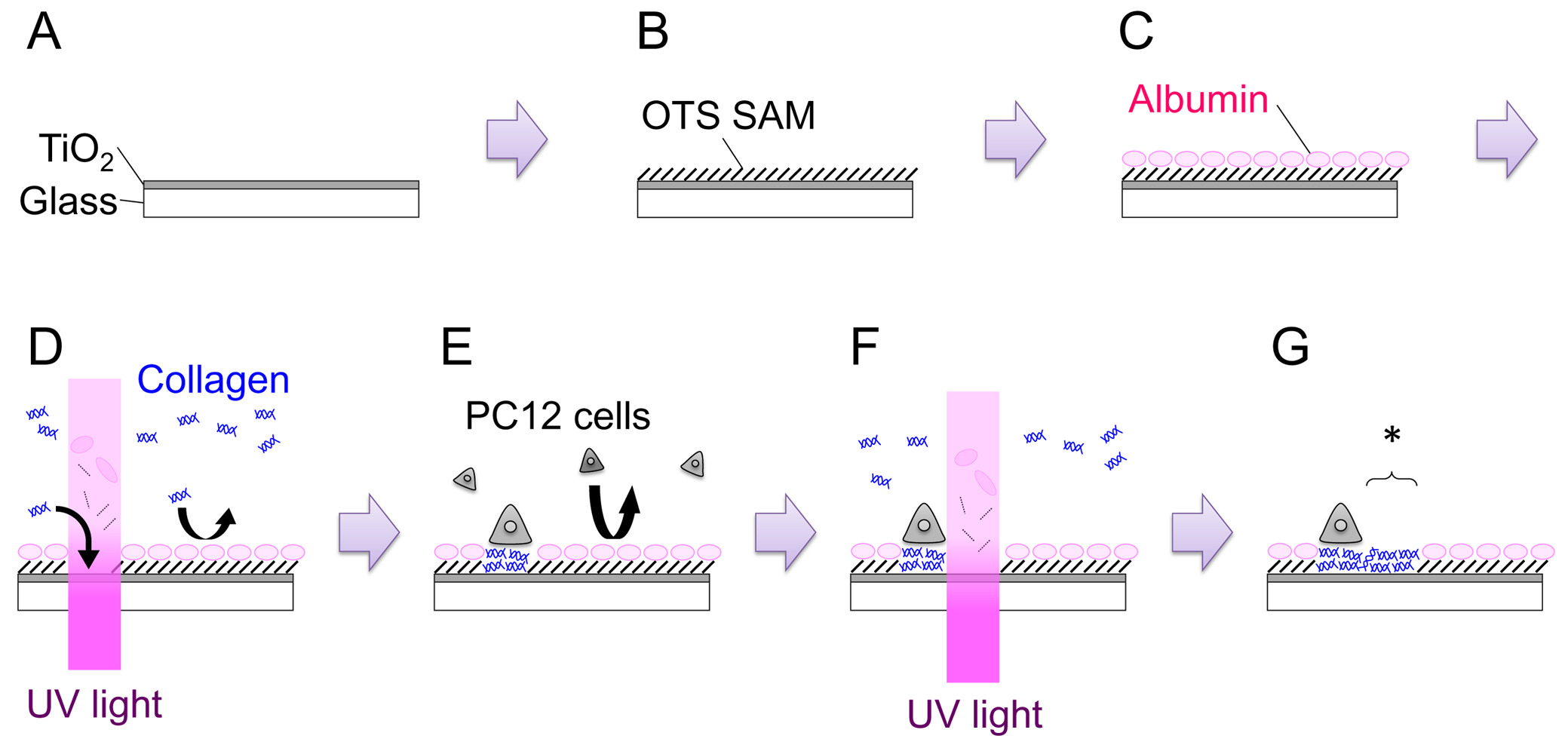
Figura 1. Ilustração esquemática do processo global. Veja o texto para maiores detalhes. Por favor clique aqui para ver uma versão maior desta figura.
Resultados
A Figura 2A apresenta uma imagem de microscopia electrónica de varrimento do corte transversal (SEM) do TiO 2 filme depositado por borrifamento. A partir da observação, da espessura do filme foi estimada como sendo de aproximadamente 150 nm. Perceptível aqui é a planura do filme depositado de TiO 2. Uma análise mais aprofundada por microscopia de força atômica (AFM) revelou que a rugosidade root mean square (RMS) da superfície foi de 0,2 nm (Figura 2B).
Discussão
Em nosso protocolo atual, TiO 2 filme foi formado por RF-pulverização catódica. Somos a favor deste método de deposição, uma vez que nos permite preparar de forma reprodutível um fotocatalítico TiO 2 filme com uma rugosidade sub-nm. Embora os processos de deposição por pulverização catódica são familiares aos cientistas de materiais e engenheiros eletrônicos, não pode ser bastante acessível para os biólogos. Nesse caso, o TiO 2 película revestida por rotação seria um...
Divulgações
The authors have nothing to disclose.
Agradecimentos
Authors thank Mr. Kotaro Okubo for the kind assistance with SEM imaging. This work was supported by the Japan Society for the Promotion of Science Grant-in-Aid for Basic Research (B) (20310069), Grant-in-Aid for Research Activity Start-up (25880021), and by research grants from the Kurata Memorial Hitachi Science and Technology Foundation and the Nippon Sheet Glass Foundation for Materials Science and Engineering.
Materiais
| Name | Company | Catalog Number | Comments |
| Glass coverslip | Warner Instruments | CS-15R15 | 15 mm diameter, #1.5 thickness |
| Diamond scriber | Ogura Jewel Industry | D-Point Pen | |
| RF sputtering system | ANELVA | SPC350 | |
| TiO2 sputtering target | Kojundo Chemical Lab | Titanium (IV) oxide, target | Purity, 99.9% |
| Plasma reactor | Yamato | PR301 | |
| n-octadecyltrichlorosilane (OTS) | Aldrich | 104817 | |
| Toluene | Wako | 204-01866 | |
| Tissue-culture dish (35 mm) | Greiner | 627160 | |
| Tissue-culture dish (60 mm) | BD Falcon | 353002 | |
| Type-IV collagen | Nitta Gelatin | Cellmatrix Type IV | |
| D-PBS | Gibco | 14190-144 | |
| Dulbecco's modified Eagle's medium (DMEM) | Gibco | 11885-084 | |
| Fetal bovine serum | Gibco | 12483-020 | Heat-inactivate and pass through a 0.22 μm filter before use |
| Horse serum | Gibco | 26050-088 | Pass through a 0.22 μm filter before use |
| Penicillin-streptomycin (100x) | Nacalai tesque | 26253-84 | |
| 7S nerve growth factor (NGF) | Alomone Labs | N-130 | |
| Bovine serum albumin (BSA) | Sigma | A2153 | |
| EDTA | Dojindo | N001 | Stock solution in 0.5 M |
| TiO2 nanoparticle | Tayca | TKD-701 |
Referências
- Hughes, M. A., Brennan, P. M., Bunting, A. S., Shipston, M. J., Murray, A. F. Cell Patterning on Photolithographically Defined Parylene-C: SiO2 Substrates. J. Vis. Exp. (85), e50929 (2014).
- Kleinfeld, D., Kahler, K. H., Hockberger, P. E. Controlled Outgrowth of Dissociated Neurons on Patterned Substrates. J. Neurosci. 8, 4098-4120 (1988).
- Pensen, D., Heinz, W. F., Werbin, J. L., Hoh, J. H., Haviland, D. B. Electron Beam Patterning of Fibronectin Nanodots that Support Focal Adhesion Formation. Soft Matter. 3, 1280-1284 (2007).
- Tanii, T., et al. Application of Organosilane Monolayer Template to Quantitative Evaluation of Cancer Cell Adhesive Ability. Jpn. J. Appl. Phys. 50, 06GL01 (2011).
- Yamamoto, H., et al. In-Situ Guidance of Individual Neuronal Processes by Wet Femtosecond Laser Processing of Self-Assembled Monolayers. Appl. Phys. Lett. 99, 163701-1610 (2011).
- Yamamoto, H., et al. Differential Neurite Outgrowth is Required for Axon Specification by Cultured Hippocampal Neurons. J. Neurochem. 123, 904-910 (2012).
- Shen, K., Qi, J., Kam, L. C. Microcontact Printing of Proteins for Cell Biology. J. Vis. Exp. (22), e1065 (2008).
- Johnson, D. M., LaFranzo, N. A., Maurer, J. A. Creating Two-Dimensional Patterned Substrates for Protein and Cell Confinement. J. Vis. Exp. (55), e3164 (2011).
- Singhvi, R., et al. Engineering Cell Shape and Function. Science. 264, 696-698 (1126).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric Control of Cell Life and Death. Science. 276, 1425-1428 (1997).
- Degot, S., et al. Improved Visualization and Quantitative Analysis of Drug Effects using Micropatterned Cells. J. Vis. Exp. (46), e2514 (2010).
- Nakanishi, J., et al. Photoactivation of a Substrate for Cell Adhesion under Standard Fluorescence Microscopes. J. Am. Chem. Soc. 126, 16314-16315 (2004).
- Kim, M., et al. Addressable Micropatterning of Multiple Proteins and Cells by Microscope Projection Photolithography Based on a Protein Friendly Photoresist. Langmuir. 26, 12112-12118 (2010).
- Deka, G., Okano, K., Kao, F. -. J. Dynamic Photopatterning of Cells In Situ by Q-Switched Neodymium-Doped Yttrium Ortho-Vanadate. Laser. J. Biomed. Opt. 19, 011012 (2014).
- Robertus, J., Browne, W. R., Feringa, B. L. Dynamic Control over Cell Adhesive Properties using Molecular-Based Surface Engineering Strategies. Chem. Soc. Rev. 39, 354-378 (2010).
- Choi, I., Yeo, W. -. S. Self-Assembled Monolayers with Dynamicity Stemming from (Bio)chemical Conversions: From Construction to Application. ChemPhysChem. 14, 55-69 (2013).
- Nakanishi, J. Switchable Substrates for Analyzing and Engineering Cellular Functions. Chem. Asian J. 9, 406-417 (2014).
- Yamamoto, H., et al. In Situ Modification of Cell-Culture Scaffolds by Photocatalytic Decomposition of Organosilane Monolayers. Biofabrication. 6, 035021 (2014).
- Sekine, K., Yamamoto, H., Kono, S., Ikeda, T., Kuroda, A., Tanii, T. Surface Modification of Cell Scaffold in Aqueous Solution using TiO2 Photocatalysis and Linker Protein L2 for Patterning Primary Neurons. e-J. Surf. Sci. Nanotech. 13, 213-218 (2015).
- Arima, Y., Iwata, H. Effects of Surface Functional Groups on Protein Adsorption and Subsequent Cell Adhesion using Self-Assembled Monolayers. J. Mater. Chem. 17, 4079-4087 (2007).
- Fujishima, A., Zhang, X., Tryk, D. A. TiO2 Photocatalysis and Related Surface Phenomena. Surf. Sci. Rep. 63, 515-582 (2008).
- Sigal, G. B., Mrksich, M., Whitesides, G. M. Effect of Surface Wettability on the Adsorption of Proteins and Detergents. J. Am. Chem. Soc. 120, 3464-3473 (1998).
- Zhang, X., et al. A Transparent and Photo-Patternable Superhydrophobic Film. Chem. Commun. 2007, 4949-4951 (1039).
- Kaech, S., Banker, G. Culturing Hippocampal Neurons. Nat. Protoc. 1, 2406-2415 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados