È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Photopatterning proteine e cellule in acquosa ambiente utilizzando TiO
In questo articolo
Riepilogo
We describe a protocol for modifying cell affinity of a scaffold surface in aqueous environment. The method takes advantage of titanium dioxide photocatalysis to decompose organic film in the photo-irradiated region. We show that it can be used to create microdomains of scaffolding proteins, both ex situ and in situ.
Abstract
Contaminanti organici adsorbiti sulla superficie del biossido di titanio (TiO 2) possono essere decomposti da fotocatalisi sotto ultravioletta (UV). Qui si descrive l'impiego di TiO 2 fotocatalisi alterare localmente affinità cellule della superficie del substrato un nuovo protocollo. Per questo esperimento, un sottile TiO 2 film era sputtering rivestite su un vetrino di vetro, e la superficie TiO 2 è stato successivamente modificato con un monostrato silano organico derivato da octadecyltrichlorosilane (OTS), che inibisce l'adesione cellulare. Il campione è stato immerso in un mezzo di coltura cellulare, e concentrato luce UV è stata irradiata ad una regione ottagonale. Quando un neuronali PC12 linea cellulare sono state piastrate sul campione, cellule rispettati solo sulla zona UV-irradiati. Mostriamo inoltre che tale modificazione superficiale può essere eseguita anche in situ, cioè, anche quando le cellule crescono sul substrato. Modifica corretta della superficie necessaria una matrice extracellulare protein collagene di essere presente nel mezzo al momento della irradiazione UV. La tecnica qui presentata può potenzialmente essere impiegato in tipi di cellule patterning più per la costruzione di sistemi di co-coltura o di manipolare arbitrariamente cellule in coltura.
Introduzione
Processi litografia di semiconduttori e suoi derivati - come fotolitografia 1,2, litografia a fascio elettronico 3-6, 7-10 e stampare microcontact - sono diventati uno strumento stabilito in biologia cellulare crescere cellule viventi in una posizione e geometria definita. Il metodo patterning si basa sull'utilizzo di substrati microfabbricati, costituito da micro-isola di cellule permissive rivestimento in uno sfondo non permissive. Tale substrato serve come modello per modello cellule. Queste tecnologie ci hanno fornito i nuovi metodi per progettare cellule e la loro funzione a livello singolo e multi-cellulari, per estrarre le proprietà intrinseche delle cellule, e per aumentare la produttività di base cellulare di screening farmaco 11.
Il grado di libertà in patterning cellula sarebbe aumentare notevolmente se la geometria motivo template potrebbe essere modificato in situ, cioè, mentre le cellule sono coltivate sulle surface. I metodi convenzionali per la formazione di pattern non possono essere applicati direttamente qui, dato che elaborano campioni in atmosfera o in vuoto. Sono stati proposti quindi varie nuove tecniche di modificazione superficiale, che si basano, per esempio, su composti fotoreattivi 12,13 o ablazione laser 5,14, solo per citarne alcuni. I metodi proposti sono stati ben recensito da Robertus et al. 15, e più recentemente da Choi et al. 16 e 17 Nakanishi.
Qui, in questo articolo, si descrive un nuovo protocollo di in-situ modifica della superficie, che si avvale di decomposizione fotocatalitica di molecole organiche su un biossido di titanio (TiO2) Superficie 18,19. In questo metodo, un film TiO 2 è inserito tra il substrato di vetro e la pellicola organica che interfaccia le cellule, e la pellicola organica viene decomposta in situ irradiando localmente ultravioletta (UV)luce per una regione di interesse (λ <388 nm). Abbiamo dimostrato che il nuovo protocollo può essere utilizzato per creare microdisegni di proteine della matrice extracellulare e le cellule viventi sia ex situ e in situ. TiO 2 è biocompatibile, chimicamente stabile, e otticamente trasparente, cui caratteristiche rende adatto ad introdurre in esperimenti di coltura cellulare. Questo protocollo fornisce una scienza dei materiali a base alternativa per modificare ponteggi coltura cellulare in ambiente coltura cellulare.
Protocollo
1. Preparazione di TiO2 Rivestiti vetro Coverslip
- Numero i coprioggetti usando una punta per tracciare diamante. Questo non solo aiuta a tenere traccia di ogni coprioggetto ma anche per garantire che il lato corretto del campione è rivolta verso l'alto. Pulire i coprioggetti, prima sotto un getto DDH 2 O, poi immergendole in una soluzione piranha (H 2 SO 4: H 2 O 2 = 4: 1). Dopo 10 minuti, risciacquare accuratamente i coprioggetti, 8 volte in DDH 2 O. Asciugare il coprioggetto sotto N 2 flusso.
- Impostare TiO 2 destinazione nel sistema sputtering a radiofrequenza (RF). Fissare i coprioggetti su un supporto campione del materiale sputtering utilizzando un nastro poliimmide. Collocare il supporto del campione nella camera di sputtering. Evacuare la camera fino a quando la pressione raggiunge 2.0 × 10 -4 Pa.
- Introdurre gas Ar nella camera e impostare la pressione di deposizione a 4,0 mTorr. Pur mantenendo l'otturatore chiuso, aumentare gradualmentela potenza RF a 70 W.
- Aprire l'otturatore e sputter per 15 minuti per ottenere una pellicola con uno spessore di 120-150 nm (Figura 1: Fase A). Tasso di crescita del film deve essere derivato per ciascuna macchina.
2. Surface Coating con cellulare repellente Film
- Hydrophilize superficie TiO 2 trattando il campione con O 2 al plasma, seguendo le istruzioni fornite dal produttore del reattore al plasma. Trattiamo il campione per 5 minuti a 200 W con O 2 flusso di 100 sccm. Immergere il campione in DDH 2 O e confermare che la superficie è superidrofilia. Asciugare accuratamente la superficie sotto N 2 flusso.
- Preparare la soluzione 1 octadecyltrichlorosilane mm (OTS) con l'aggiunta di 39,6 OTS microlitri per 100 ml di toluene. Immergere il campione nella soluzione per 1 ora a RT. Condurre questo passaggio all'interno di un sacchetto 2 guanto -filled N (Figura 1: Fase B).
- Per rimuovere molecole physisorbed, sonicate il campione in toluene, acetone, etanolo, e DDH 2 O per 5 minuti ciascuno, in questo ordine. Sciacquare il campione quattro volte in acqua dolce DDH 2 O e asciugare la superficie sotto N 2 flusso. La superficie deve essere idrofobica con un angolo di contatto di 100-110 °.
3. Ex-Situ Patterning Surface
- Lavorare in una cappa a flusso laminare, disegnare diversi segni di graffi con una punta per tracciare diamante sulla superficie. I segni aiutano a tenere traccia delle regioni elaborati e anche portare i microscopi a fuoco. Sterilizzare il TiO OTS rivestite 2 immergendo il campione in etanolo al 70% per 5 min. Risciacquare il campione due volte nel sterile DDH 2 O.
- Trasferire il campione in un piatto da 35 mm, e aggiungere 2 ml di terreno di coltura PC12 (vedi punto 4.2). Incubare per oltre 3 ore in un incubatore CO 2 (37 ° C). Questa procedura è destinata a far albumine sieriche assorbono sulla superficie. Albumine adsorbite inibiscono conseguente assorbimento di altri proteins e cellule (Figura 1: fase c).
- Durante l'attesa, impostare il microscopio a fluorescenza invertito.
- Accendere la lampada ad arco, inserire il cubo di filtro UV, e impostare la lente dell'obiettivo a 20X.
- Misurare l'intensità della luce I (W cm -2) usando un misuratore UV intensità, e calcolare il tempo di irradiazione t (in secondi) per una dose di d (in cm -2 J) come: t = d / I.
Ad esempio, per irradiare alla dose di 200 J cm -2 utilizzando una sorgente luminosa di 600 mW cm -2, è richiesta 333 sec di irradiazione. - Utilizzare un micrometro e chiudere il diaframma di campo per impostare la dimensione della regione da irradiare, ad esempio, 200 micron.
- Dopo l'incubazione 3 ore, integrare la media con 200 ml di 3,0 mg ml -1 di collagene di tipo-IV (Col-IV; finali concentrazione di 300 ug ml -1).
- Trasferire il piatto 35 mm al palco microscopio. Trova il graffio marca fuoco il microscopio sulla superficie del campione, e irradiare luce UV con una dose di 200 J cm -2 (Figura 1: Fase D). La zona di irradiazione UV può essere modificata sia regolando l'apertura del diaframma di campo o sostituendo il diaframma di campo con una maschera metallica della geometria arbitrale.
- Sostituire la media con un mezzo di coltura fresco (senza Col-IV), e posizionare il campione torna in incubatrice.
Cultura 4. cellulare
- Routine coltura di cellule PC12 è effettuata su un piatto di plastica rivestita di collagene.
- Per rivestire un piatto di tessuto-cultura di 60 mm, con Col-IV, prima bagnare la superficie con DDH 2 O e aspirare tutto DDH 2 O.
- Preparare 300 mg ml -1 Col-IV diluendo la soluzione originale (3 mg ml -1) 10x con DDH 2 O.
- Aggiungere 200 ml di 300 mcg ml -1 Col-IV per 60-mm piatto. Lasciate che la soluzione di diffondersi attraverso la superficie.
- Dry tha piatto in una cappa a flusso laminare per circa 1 ora.
- Lavare la superficie due volte con 4 ml di tampone fosfato salino di Dulbecco (D-PBS). Quando non si usa subito il piatto rivestito, lavare il piatto una volta con 4 ml di DDH 2 O. Dopo aspirazione DDH 2 O, riporre il piatto in incubatrice. Cercate di non conserverà per più di due settimane.
- Cellule PC12 sono coltivate in un terreno di coltura costituito da: medio (glucosio basso) modificato Eagle Dulbecco + 10% siero fetale bovino + 5% soluzione cavallo penicillina streptomicina siero + 1%. Incubare le cellule in un incubatore CO 2 (37 ° C, 5% CO 2), e sostituire la metà del mezzo ogni altro giorno. Passage cellule prima che raggiungano confluenza.
- Aspirare il terreno di coltura, e aggiungere 2 ml di PBS + (D-PBS + 10 mg ml -1 albumina di siero bovino (BSA) + EDTA 10 mM) preriscaldata a 37 ° C. Incubare per 5 min a 37 ° C. Battere delicatamente il piatto di staccare tutti cells.
- Raccogliere le cellule in un tubo da 15 ml. Sciacquare il piatto con 3 ml di fresco D-PBS, preriscaldata a 37 ° C.
- Centrifugare la provetta a 150 g per 4 min.
- Aspirare il surnatante e aggiungere 1 ml di mezzo di crescita.
- Per la cultura di routine, dividere le celle 1: 3 a 1: 5.
- Per piastra cellule sul campione modificato UV OTS / TiO 2, contare densità cellulare e aggiungere 3,0 x 10 5 cellule in un piatto da 35 mm (Figura 1: Stadio E). Incubare il piatto in incubatrice umidificata (37 ° C, 5% CO 2) per 1-2 giorni. Entrambi fattore di crescita ingenuo e nervoso (NGF), le cellule PC12 -differentiated possono essere utilizzati per gli esperimenti patterning. Per NGF differenziazione, 100 ng ml -1 di 7S-NGF viene aggiunto alla crescita media diversi giorni prima placcatura le cellule sul campione. NGF-differenziazione sembra aumentare la adhesibility delle cellule PC12 e facilita la manipolazione, specialmente nella in-situ e patterningxperiments.
5. In-Situ Patterning Surface
- Dopo 1-2 giorni di coltura sulla superficie ex situ modificato, confermano che le cellule sono Collegamento e cresce solo sulla regione UV irradiata. Impostare il microscopio, come descritto al punto 3.4.
- Trasferire il campione in un piatto di coltura di tessuti da 35 mm contenente il mezzo di crescita, 100 ng ml -1 NGF (nel caso di utilizzo di cellule NGF dissociati), e 100 mg ml -1 Col-IV.
- Porre la capsula sul palco microscopio. Trovare la posizione appropriata, e irradiare luce UV con una dose di 200 J cm -2 (Figura 1: Fase F, G). La regione permissiva appena creato viene indicato con un asterisco nella figura 1: Punto G.
- Prova a completare l'elaborazione di un singolo campione entro 30 minuti. Dopo l'irraggiamento, trasferire il campione di nuovo nel mezzo senza Col-IV.
- Fare molta attenzione quando si trasporta il piatto con cucellule ltured e trasferimento del campione dal piatto per piatto per evitare il distacco delle cellule fantasia.
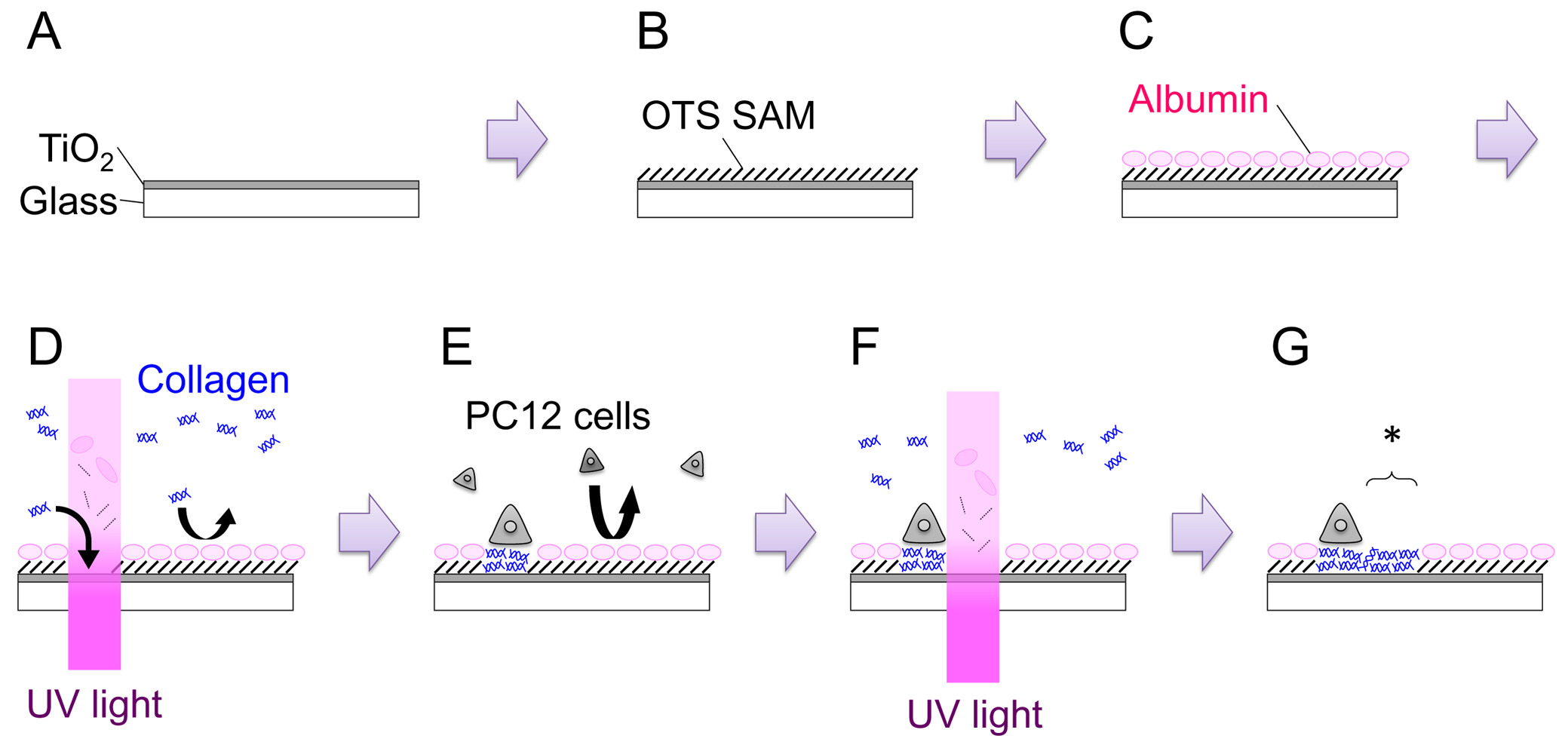
Figura 1. Schema di processo globale. Vedere il testo per i dettagli. Cliccate qui per vedere una versione più grande di questa figura.
Risultati
Figura 2A mostra un'immagine in sezione microscopia elettronica a scansione (SEM) della TiO 2 film di sputtering depositato. Dall'osservazione, spessore del film è stata stimata essere di circa 150 nm. Notevole ecco la planarità del depositato TiO 2 film. Ulteriori analisi mediante microscopia a forza atomica (AFM) ha rivelato che la radice quadrata media (RMS) rugosità della superficie era di 0,2 nm (Figura 2B).
Quando la su...
Discussione
Nel nostro attuale protocollo, TiO 2 film è stato formato da RF-magnetron sputtering. Favoriamo questo metodo di deposito in quanto ci permette di preparare in modo riproducibile un fotocatalitico TiO 2 film con una rugosità sub-nm. Anche se i processi di deposizione sputtering sono familiari a scienziati dei materiali e ingegneri elettronici, potrebbe non essere abbastanza accessibile per i biologi. In tal caso, TiO 2 film di spin-rivestite sarebbe una scelta alternativa 23.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
Authors thank Mr. Kotaro Okubo for the kind assistance with SEM imaging. This work was supported by the Japan Society for the Promotion of Science Grant-in-Aid for Basic Research (B) (20310069), Grant-in-Aid for Research Activity Start-up (25880021), and by research grants from the Kurata Memorial Hitachi Science and Technology Foundation and the Nippon Sheet Glass Foundation for Materials Science and Engineering.
Materiali
| Name | Company | Catalog Number | Comments |
| Glass coverslip | Warner Instruments | CS-15R15 | 15 mm diameter, #1.5 thickness |
| Diamond scriber | Ogura Jewel Industry | D-Point Pen | |
| RF sputtering system | ANELVA | SPC350 | |
| TiO2 sputtering target | Kojundo Chemical Lab | Titanium (IV) oxide, target | Purity, 99.9% |
| Plasma reactor | Yamato | PR301 | |
| n-octadecyltrichlorosilane (OTS) | Aldrich | 104817 | |
| Toluene | Wako | 204-01866 | |
| Tissue-culture dish (35 mm) | Greiner | 627160 | |
| Tissue-culture dish (60 mm) | BD Falcon | 353002 | |
| Type-IV collagen | Nitta Gelatin | Cellmatrix Type IV | |
| D-PBS | Gibco | 14190-144 | |
| Dulbecco's modified Eagle's medium (DMEM) | Gibco | 11885-084 | |
| Fetal bovine serum | Gibco | 12483-020 | Heat-inactivate and pass through a 0.22 μm filter before use |
| Horse serum | Gibco | 26050-088 | Pass through a 0.22 μm filter before use |
| Penicillin-streptomycin (100x) | Nacalai tesque | 26253-84 | |
| 7S nerve growth factor (NGF) | Alomone Labs | N-130 | |
| Bovine serum albumin (BSA) | Sigma | A2153 | |
| EDTA | Dojindo | N001 | Stock solution in 0.5 M |
| TiO2 nanoparticle | Tayca | TKD-701 |
Riferimenti
- Hughes, M. A., Brennan, P. M., Bunting, A. S., Shipston, M. J., Murray, A. F. Cell Patterning on Photolithographically Defined Parylene-C: SiO2 Substrates. J. Vis. Exp. (85), e50929 (2014).
- Kleinfeld, D., Kahler, K. H., Hockberger, P. E. Controlled Outgrowth of Dissociated Neurons on Patterned Substrates. J. Neurosci. 8, 4098-4120 (1988).
- Pensen, D., Heinz, W. F., Werbin, J. L., Hoh, J. H., Haviland, D. B. Electron Beam Patterning of Fibronectin Nanodots that Support Focal Adhesion Formation. Soft Matter. 3, 1280-1284 (2007).
- Tanii, T., et al. Application of Organosilane Monolayer Template to Quantitative Evaluation of Cancer Cell Adhesive Ability. Jpn. J. Appl. Phys. 50, 06GL01 (2011).
- Yamamoto, H., et al. In-Situ Guidance of Individual Neuronal Processes by Wet Femtosecond Laser Processing of Self-Assembled Monolayers. Appl. Phys. Lett. 99, 163701-1610 (2011).
- Yamamoto, H., et al. Differential Neurite Outgrowth is Required for Axon Specification by Cultured Hippocampal Neurons. J. Neurochem. 123, 904-910 (2012).
- Shen, K., Qi, J., Kam, L. C. Microcontact Printing of Proteins for Cell Biology. J. Vis. Exp. (22), e1065 (2008).
- Johnson, D. M., LaFranzo, N. A., Maurer, J. A. Creating Two-Dimensional Patterned Substrates for Protein and Cell Confinement. J. Vis. Exp. (55), e3164 (2011).
- Singhvi, R., et al. Engineering Cell Shape and Function. Science. 264, 696-698 (1126).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric Control of Cell Life and Death. Science. 276, 1425-1428 (1997).
- Degot, S., et al. Improved Visualization and Quantitative Analysis of Drug Effects using Micropatterned Cells. J. Vis. Exp. (46), e2514 (2010).
- Nakanishi, J., et al. Photoactivation of a Substrate for Cell Adhesion under Standard Fluorescence Microscopes. J. Am. Chem. Soc. 126, 16314-16315 (2004).
- Kim, M., et al. Addressable Micropatterning of Multiple Proteins and Cells by Microscope Projection Photolithography Based on a Protein Friendly Photoresist. Langmuir. 26, 12112-12118 (2010).
- Deka, G., Okano, K., Kao, F. -. J. Dynamic Photopatterning of Cells In Situ by Q-Switched Neodymium-Doped Yttrium Ortho-Vanadate. Laser. J. Biomed. Opt. 19, 011012 (2014).
- Robertus, J., Browne, W. R., Feringa, B. L. Dynamic Control over Cell Adhesive Properties using Molecular-Based Surface Engineering Strategies. Chem. Soc. Rev. 39, 354-378 (2010).
- Choi, I., Yeo, W. -. S. Self-Assembled Monolayers with Dynamicity Stemming from (Bio)chemical Conversions: From Construction to Application. ChemPhysChem. 14, 55-69 (2013).
- Nakanishi, J. Switchable Substrates for Analyzing and Engineering Cellular Functions. Chem. Asian J. 9, 406-417 (2014).
- Yamamoto, H., et al. In Situ Modification of Cell-Culture Scaffolds by Photocatalytic Decomposition of Organosilane Monolayers. Biofabrication. 6, 035021 (2014).
- Sekine, K., Yamamoto, H., Kono, S., Ikeda, T., Kuroda, A., Tanii, T. Surface Modification of Cell Scaffold in Aqueous Solution using TiO2 Photocatalysis and Linker Protein L2 for Patterning Primary Neurons. e-J. Surf. Sci. Nanotech. 13, 213-218 (2015).
- Arima, Y., Iwata, H. Effects of Surface Functional Groups on Protein Adsorption and Subsequent Cell Adhesion using Self-Assembled Monolayers. J. Mater. Chem. 17, 4079-4087 (2007).
- Fujishima, A., Zhang, X., Tryk, D. A. TiO2 Photocatalysis and Related Surface Phenomena. Surf. Sci. Rep. 63, 515-582 (2008).
- Sigal, G. B., Mrksich, M., Whitesides, G. M. Effect of Surface Wettability on the Adsorption of Proteins and Detergents. J. Am. Chem. Soc. 120, 3464-3473 (1998).
- Zhang, X., et al. A Transparent and Photo-Patternable Superhydrophobic Film. Chem. Commun. 2007, 4949-4951 (1039).
- Kaech, S., Banker, G. Culturing Hippocampal Neurons. Nat. Protoc. 1, 2406-2415 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon