É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Conformações GPCR G Protein selectivos medida usando FRET Sensores em um Fluorometer Ensaio de células vivas de suspensão
Neste Artigo
Resumo
Simple methods to detect the selective activation of G proteins by G protein-coupled receptors remain an outstanding challenge in cell signaling. Here, Fӧrster resonance energy transfer (FRET) biosensors have been developed by pairwise tethering a GPCR to G protein peptides to probe conformational changes at controlled concentrations in live cells.
Resumo
Fӧrster transferência de energia de ressonância (FRET) à base estudos têm se tornado cada vez mais comum na investigação de sinalização de GPCR. Nosso grupo de pesquisa desenvolveu um sensor de FRET intra-molecular para detectar a interacção entre subunidades e GPCRs em células vivas após a estimulação agonista Gu. Aqui, nós detalhe o protocolo para a detecção de alterações na TERF entre o receptor adrenérgico β 2 e o péptido C-terminal Gαs mediante tratamento com cloridrato de isoproterenol 100 uM como previamente caracterizados 1. O nosso sensor de FRET é um polipéptido único que consiste em série de um GPCR de comprimento completo, um fluoróforo FRET aceitador (mCitrine), um ER / K ESPASMO (afinidade de proteína sistemática modulação força) ligante, um fluoróforo FRET dador (mCerulean), e uma Gu C peptídeo-terminal. Este protocolo irá preparação celular detalhe, condições de transfecção, configuração de equipamentos, execução de ensaio e análise de dados. Este projeto experimental detecta pequena chAnges em FRET indicativa de interacções proteína-proteína, e também pode ser utilizado para comparar a força da interacção entre ligandos de GPCR e emparelhamentos de proteína-G. Para melhorar o sinal-ruído nas nossas medições, este protocolo exige precisão elevado em todas as etapas, e é aqui apresentada para permitir a execução reprodutível.
Introdução
receptores acoplados à proteína G (GPCRs) são receptores transmembranares de sete. O genoma humano sozinho contém aproximadamente 800 genes que codificam para os GPCRs, que são activadas por uma variedade de ligandos incluindo a luz, odorantes, hormonas, péptidos, fármacos e outras moléculas pequenas. Quase 30% de todos os produtos farmacêuticos atualmente no GPCRs alvo do mercado porque eles desempenham um papel importante em muitos estados de doença 2. Apesar de décadas de um extenso trabalho feito nesta família de receptores, continua a haver questões pendentes significativas no campo, especialmente no que diz respeito aos mecanismos moleculares que levam interações GPCR-efetoras. Até à data, apenas uma estrutura cristalina de alta resolução foi publicada, fornecendo informações sobre a interacção entre o receptor β 2 -adrenérgico (β 2-Ar) e a proteína Gs 3. Juntamente com uma extensa pesquisa nas últimas três décadas, reitera um componente estrutural específico que é fundamental nesteinteracção: a subunidade Gu C-terminal. Esta estrutura é importante tanto para a activação da proteína G por a selecção de proteínas GPCR 4 e G 5-6. Assim, a Gu C-terminal fornece uma ligação crucial entre a estimulação do ligando de GPCR e a activação de proteína G selectiva.
Investigação na última década sugere que GPCRs preencher uma ampla paisagem conformacional, com ligando de ligação de estabilização subconjuntos de conformações GPCR. Embora várias técnicas, incluindo a cristalografia, RMN e espectroscopia de fluorescência, espectroscopia de massa e estão disponíveis para examinar a paisagem conformacional GPCR, há uma escassez de abordagens para elucidar o seu significado funcional na selecção efectora 7. Aqui, descrevemos uma abordagem baseada Fӧrster transferência de energia de ressonância (FRET) para detectar G conformações GPCR proteína selectivo. FRET baseia-se na proximidade e orientação paralela dos dois fluoróforos com sobreposição de emissão (doador) umND excitação (aceitador) espectros 8. À medida que os fluoróforos dadores e aceitadores aproximar em conjunto, como resultado de qualquer alteração conformacional na proteína ou uma interacção proteína-proteína, a FRET entre eles aumenta, e pode ser medida utilizando uma variedade de métodos de 8. Biossensores baseados em FRET tem sido utilizado extensivamente no campo 9 GPCR. Eles têm sido utilizados para sondar as mudanças de conformação na GPCR através da inserção de dador e aceitador no terceiro laço intracelular e GPCR C-terminal; sensores foram concebidos para sondar GPCR e interações efetoras, rotulando separadamente do GPCR e efetoras (proteína G subunidades / Arrestinas) com um par FRET 10; alguns sensores também detectar alterações conformacionais na proteína G 11. Estes biossensores permitiram o campo para pedir uma série de perguntas pendentes, incluindo mudanças conformacionais na GPCR e efetoras, cinética de interação GPCR-efetoras, e ligantes alostéricos 12. Nosso grupoestava particularmente interessado na criação de um biossensor que poderá detectar G conformações GPCR específicos para a proteína em condições impulsionado-agonistas. Este biosensor se baseia em uma tecnologia desenvolvida recentemente nomeado SPASM (sistemática de afinidade de proteína força modulação) 13. SPASM envolve domínios proteicos amarrar interagir usando um ligante de ER / K, que controla as suas concentrações eficazes. Flanqueando o ligante com um par FRET de fluoróforos cria uma ferramenta que pode reportar o estado da interacção entre proteínas de 12. Anteriormente 1 módulo SPASM foi usada para amarrar o Gu C-terminal a um GPCR e monitorar suas interações com fluoróforos FRET, mCitrine (referido no presente protocolo por sua variante vulgarmente conhecido, Amarelo Proteína Fluorescente (YFP), excitação / emissão de pico em 490/525 nm) e mCerulean (referido no presente protocolo por sua variante comumente conhecido Cyan Fluorescent Protein (PCP), excitação / pico de emissão 430/475 nm). A partir de N- para C-terminal, tseu único polipéptido codificado geneticamente contém: um comprimento total de GPCR, FRET aceitador (mCitrine / YFP), 10 nm ER / ligante K, FRET dador (mCerulean / PCP), e o peptídeo C-terminal Ga. Neste estudo, os sensores são abreviados como GPCR péptido-ligante-Gu comprimento. Todos os componentes são separados por uma desestruturado (Gly-Ser-Gly) 4 ligante que permite a rotação livre de cada domínio. A caracterização detalhada de tais sensores anteriormente foi realizada utilizando dois GPCRs protótipos: β 2-AR e opsina 1.
Este sensor é transientemente transfectado em células HEK-293T e viver experiências com células medida espectros de fluorescência baseada fluorómetro do par de FRET, em unidades arbitrárias de contagens por segundo (CPS), na presença ou ausência de ligando. Estas medições são utilizadas para calcular um rácio de FRET entre os fluoróforos (YFP max / PCP no máximo). Uma alteração na TERF (ΔFRET) é então calculado subtraindo-se a proporção média de FRETde amostras não tratadas a partir da razão de ligando de FRET de amostras tratadas. ΔFRET podem ser comparados entre construções (β 2-AR-10 peptídeo nm-Gαs contra p2-AR-10 nm-no peptídeo). Aqui, nós detalhe o protocolo para expressar esses sensores em células HEK-293T ao vivo, acompanhar a sua expressão, e a configuração, execução e análise da célula viva baseada fluorômetro FRET medida para tratamento contra condições de drogas tratados. Embora este protocolo é específico para o péptido do sensor nm-Gαs β 2-AR-10 tratadas com 100 uM de bitartarato de isoproterenol, pode ser optimizada para diferentes pares de ligandos de GPCR e-Ga.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparação de DNA
- sensor de projeto constrói usando um esquema de clonagem modular. Por favor, refira o projeto do sensor β 2-AR detalhado anteriormente 1.
- Preparar DNA de acordo com o protocolo do kit miniprep comercial e elui-se em solução 2 mM de Tris-HCl, pH 8, com ≥ concentração de 750 ng / ul, A260 / A280 de 1,7-1,9, A 260 / A 230 de 2,0 - 2.29.
2. Cultura de Células Preparação
- células de cultura de células HEK-293T-Flp-n em DMEM contendo 4,5 g / L de D-glucose, suplementado com 10% de FBS (inactivado pelo calor) (v / v), 1% de suplemento de L-glutamina, HEPES 20 mM, pH 7,5 a 37 ° C em atmosfera humidificada em 5% de CO 2. Lidar com células em capô de segurança biológica para as etapas subsequentes.
- Permitir que as células cresçam a uma monocamada confluente antes passaging em placas de seis poços. Tempo para atingir confluência depende densidade de revestimento inicial. Utilizar placas quevir até à confluência dentro de 1 - 2 dias de plaqueamento para seis poços chapeamento. Um prato tratados com cultura confluente tecido 10 centímetros tem densidade celular de cerca de 4 x 10 6 culas / ml. Ver Figura 1 para uma imagem de crescimento de cultura de células.
- Lave as células com 10 ml de PBS, e trypsinize com 0,25% de tripsina (ver discussão, parágrafo 2). Placa de 8 x 10 5 células / cavidade em 2 ml de meio de cultura de tecidos tratados com seis pratos e permitem assim a aderir durante 16 - 20 h.
3. Condições de transfecção
- Escalonar para transfecções construções que podem exigir diferentes quantidades de tempo para conseguir uma expressão óptima (entre 20 - 36 h). Sincronizar condições para um tempo de experiência unificada. Também tem um controlo não transfectadas bem à densidade celular equivalente a ser usado para subtracção do ruído de fundo e de espalhamento durante a análise.
- Traga reagentes de transfecção à temperatura ambiente: meio de soro reduzido, DNA, reagentes de transfecção.
- Em umcobertura de segurança biológica combinar os reagentes num tubo de microcentrífuga estéril, na seguinte ordem: misturar 2 ug de ADN com 100 ul de meio de soro reduzido. Pico de 6 ul de reagente de transfecção na mistura meio / ADN, sem tocar na superfície de mistura ou o lado do tubo. Configure uma reacção de transfecção por poço. condições de transfecção pode ser optimizado (1-4 ug de ADN, 3 - 6 ul de reagente de transfecção) para alcançar níveis de expressão consistentes. Ver Tabela 1 para os rácios mais optimizado.
- Incubar mistura à temperatura ambiente na capa de segurança biológica para 15-30 min. Não use reação se deixado a incubar durante mais de 30 min.
- Adicionar reação a células de uma maneira gota a gota através de bem e agite de seis poços para assegurar uma mistura completa. Adicione uma reacção por poço.
- Após 20 horas de expressão, monitor de fluorescência usando cultura de tecidos microscópio de fluorescência. Avaliar a expressão da população com 10X localização objectivo e proteína numa célula em 40X. Observe para a expressão da proteína na membrana plasmática (PM). Se internalização substancial é observado, monitorar transfecção até expressão significativa é detectada a PM.
4. Reagente e equipamento para preparação
- Preparar stocks de 100 mm e armazenar a -80 ° C: bitartarato de isoproterenol (100 mM em dH 2 O contendo ácido ascórbico a 300 mM). Faça em gelo / em câmara fria, e flash-congelar imediatamente. As aliquotas podem ser feitas e usadas até um ano.
- Preparar tampão celular (~ 2 ml / condição) e armazenar em um banho de água C 37 °. Faça fresco todos os dias. Tabela de Referência 2 para Célula de Armazenamento constituintes.
- Preparar tampão de Drogas (10 ml) e armazenar em temperatura ambiente. Tabela de Referência 2 para constituintes do tampão de Drogas.
- Ácido lavar cuvetes utilizando HCl concentrado. Neutralizar com uma base fraca (1 M KOH), e lave cuvetes com dH 2 O.
- Prepare estação de trabalho em torno de fluorímetro com diversoscaixas de 10, 200 e 1.000 dicas ul pipeta, um temporizador definido com um 10 min contagem regressiva, uma linha de vácuo acessível com dicas para limpeza de cuvetes, toalhetes tarefa delicada, e garrafa de esguicho com ultrapura H 2 O.
- Aqueça banho de água externo para fluorômetro e calor bloco para 37 ° C.
- Ligue fluorômetro; definir programa de coleta de fluorescência para a coleta PCP à excitação 430 nm, passa-banda 8 nm; faixa de emissão de 450 nm - 600 nm, passagem de banda de 4 nm. Para a coleta de YFP apenas como controle de sensor (ver Discussão) Conjunto de excitação a 490 nm, de banda 8 nm, faixa de emissão de 500-600 nm, passa-banda 4 nm. configurações de coleta da PCP serão usados para adquirir um espectro FRET neste experimento.
- Coloque doze tubos de 1,5 ml de microcentrífuga em bloco de aquecimento, como mostrado na Figura 2, abaixo. Esses tubos são suportes para tubos de alíquota de células (microtubos 500 mL). Coloque um pequeno pedaço de tecido em suportes 1 e 7 para amortecer as cuvetes colocados aqui.
Nota: Use cuvetes separadas para a condição não tratada e da condição de drogas para evitar a contaminação cruzada.

Figura 2. Micro-tubo Configurar e posição de referência no calor do bloco Cuvette para amostras não tratadas está na posição. 1; tubos de alíquota de células estão em posições 2 - 6. Cuvette para amostras tratadas com droga é na posição 7; tubos de alíquota de células estão em posições 8 - 12. Por favor clique aqui para ver uma versão maior desta figura.
- Encha detentores 2 - 6 e 8 - 12 com ~ 750 mL de água para criar uma mini-37 ° C Banho de água.
- Coloque dez 500 microtubos ul de alíquotas de células em banhos mini-água (detentores 2 - 6, 8 - 12). Cada tubo será uma individual de repetição do quadro (5 untreated, 5 de drogas tratados).
- células para a expressão Monitor (veja o passo 3.6).
5. Experiência e Recolha de Dados
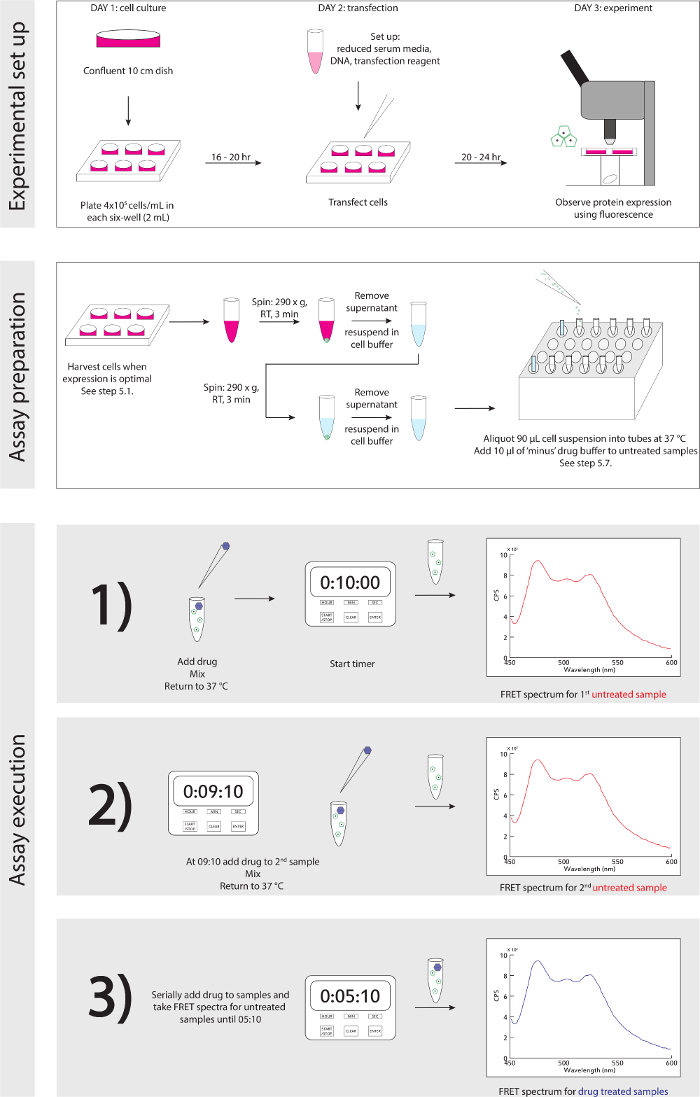
Figura 3. Experimental esquemático. Um guia detalhado passo a passo para a montagem experimental e execução. Por favor clique aqui para ver uma versão maior desta figura.
- Referência Figura 3 para o esquema experimental. Quando as células estão prontas para serem colhidas, com base na expressão de proteínas detectadas com microscópio de fluorescência (ver discussão, parágrafo 4): na capa de segurança biológica, remover suavemente ~ 1 ml de mídia, Ressuspender as células em sua cultura com uma P1,000 e transferir ressuspensão para um tubo de microcentrífuga de 1,5 ml.
Nota: Evitar utilizando tripsina, uma vez que podem digerir o N-terminal e /ou no bolso do sensor GPCR vinculativo. - Contar as células para assegurar a densidade de células apropriada na ressuspensão. Melhorar o volume de ressuspensão para 4 x 10 6 culas / ml.
- Girar em células centrífuga basculante à temperatura ambiente, 290 x g durante 3 min. Remover o sobrenadante, após a centrifugação.
- GENTLY Ressuspender as células em 1 ml de tampão celular (armazenada a 37 ° C) e repetir o passo 5.3. Durante a segunda rodada, reunir 100 mM estoque de drogas alíquota de -80 ° C. Faça diluição 1: 100 em tampão de Drogas imagens trabalhando 1 mM e mantê-RT.
- Após a segunda centrifugação, remover o sobrenadante e as células suavemente ressuspender em 1 ml de tampão celular (4 x 10 6 culas / ml). Medida OD600 da amostra no espectrofotómetro usando 1 ml de células e 1 ml de tampão celular como um branco. Dispense células em uma tina de plástico descartável e transferir de volta para um tubo de microcentrífuga imediatamente após espectrofotometria.
- Para um cel não transfectada controlel espectro de condições, delicadamente ressuspender as células não transfectadas em Tampão 1 ml celular com P1,000 pipeta, adicionar 90 ul de células de Cuvette e adquirir espectro FRET na excitação 430 nm, passa-banda 8 nm, emissão 450-600 nm, passa-banda 4 nm. Colete 3 - 5 espectros de repetição com frescos 90 ul de células. Manter o estoque de células a 37 ° C entre especificações, ressuspender suavemente com P1,000 entre cada aliquota de amostra, e enxaguar tina com ultrapura H2O entre as amostras.
- Para as condições experimentais, alíquotas de 90 ul de células transfectadas para cada um dos 500 tubos ul em suportes 2 - 6, 8 - 12 no bloco de calor. Volte a suspender suavemente estoque de células com uma pipeta P1,000 entre cada alíquota.
- Depois as células são aliquotadas, adicionar 10 ul de tampão de drogas aos tubos 2-6 para as amostras não tratadas de condição.
- Comece a experiência, adicionando 10 ml de solução de droga 1 mM no tubo 8, iniciar o temporizador de contagem regressiva de 10 min, e misture delicadamente tubo com P200 pipeta. Fechar tubo e regresso a 37° bloco de calor C.
- imediatamente pegar tubo 2, misture delicadamente com P200 (usar uma nova ponta), adicionar 90 ul de suspensão de células a condição cuvete não tratada, e coloque em fluorímetro.
- Adquirir espectro de FRET em excitação 430 nm, passagem de banda 8 nm, emissão a 450-600 nm, passagem de banda de 4 nm.
- Aos 9 min - 10 seg, spike tubo 9 com 10 ml de solução de droga 1 mM, misture delicadamente com uma P200 (use nova ponta) e tubo para aquecer bloco retornar.
- Repita os passos 5,10-5,11 com o tubo 3 e 5,12 com tubo de 10.
- Repita os passos de 5,10-5,13 em intervalos de 1 min (08:10, 07:10, etc.) até espectros são recolhidos para todas as amostras não tratadas de condição, e de drogas foi adicionado a todas as amostras condição de drogas. Utilizar uma ponteira nova para cada etapa pipeta para evitar a contaminação cruzada.
- Aos 5 min - 10 seg, começar a misturar tubo 8 (condição de drogas) delicadamente com P200 pipeta, adicionar 90 ul de suspensão de células a cuvete separado para amostra da droga tratada e coloque em fluorímetro.
- adquirir Fespectro de RET (veja o passo 5.11 para configurações).
- Repita os passos de 5,15 - 5,16 em intervalos de 1 min (04:10, 03:10, etc.) para restantes amostras condição de drogas (tubos 9 - 12).
- Depois de experiência termina, salvar arquivos de projeto, lave cuvetes com ultrapura H 2 O, e re-stock tubos para o próximo estado. Tome cuidado para evitar a contaminação cruzada na etapa de lavagem. Mudar a ponta sobre a garrafa H2O, bem como na linha de vácuo entre lavagens.
Análise 6. Os dados
- Salve e arquivos de dados de exportação em SPC formato a ser usado para a análise. programas de análise estão disponíveis para download a partir do site de publicação do Sivaramakrishnan Lab.
- Criar arquivos de caminho para o software de análise que incluem os programas de análise (v9, v15), arquivos de amostras untransfected (veja o passo 5.6 para a coleta não transfectada espectro celular), arquivo de dados de saída e arquivos de valores separados por vírgulas (CSV) para entrada de dados.
- Digite seguintes informaçõesem arquivo CSV (ver exemplo na Tabela 3) e designar respectivas condições para cada amostra, incluindo:
O nome do arquivo - arquivos de gráficos SPC individuais
Receptor - designar quais construção GPCR foi testado (por exemplo, Β2)
Binder - designar quais péptido variante da construção foi testada (por exemplo, S)
Agonista - designar sem tratamento (N) ou condições tratadas com droga (D)
Directory - a pasta do caminho em que os arquivos são salvos SPC, geralmente organizados por data
OD - registrou densidade óptica da amostra do espectrofotômetro - Digite nomes de arquivo para amostras untransfected (passo 5.6) para subtrair tampão e do ruído espalhando a partir de amostras.
- Entre as condições em programa de análise.
- executar programas para analisar amostras em condições individuais (v9) e em diferentes condições (v15).
- Excluir arquivos de amostra que são aparentes outliers no conjunto de dados, ou ajustar para subtração, aumentando ou diminuindo o valor OD de indiviarquivos duais.
- Exportar dados para o arquivo de saída para o acesso à calculada FRET proporções (525 nm / 475 nm).
- Calcule ΔFRET subtraindo-se o rácio médio FRET para a condição não tratada a partir dos rácios de FRET individuais para condições tratados (de drogas).
Access restricted. Please log in or start a trial to view this content.
Resultados
Um esquema generalizado de a configuração da experiência e execução é detalhado na Figura 3.
A fim de detectar uma mudança FRET na faixa dinâmica estreita do sensor, é fundamental respeitar as nuances do sistema ser respeitados. Qualidade celular é imperativo para a expressão da proteína, assim como a consistência na amostragem. A Figura 1 apresenta imagens de células cultivadas crescem em uma monocamada consistente (10X) que é óptima para se...
Access restricted. Please log in or start a trial to view this content.
Discussão
A gama dinâmica apertada de medições FRET neste sistema reforça a necessidade de um controle sensível de qualidade em cada etapa deste protocolo. Os passos mais importantes para assegurar uma experiência de FRET são bem sucedida 1) cultura das células, 2) transfecção 3) a expressão da proteína e 4) oportuna, uma coordenação precisa durante a execução do ensaio.
a saúde das células e qualidade de manutenção / chapeamento pode ter um impacto significativo sobre o ruído sin...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram que não têm interesses conflitantes.
Agradecimentos
RUM foi financiado pela Fellowship da American Heart Association Pré-doutoramento (14PRE18560010). A pesquisa foi financiada pelo Desenvolvimento Scientist Grant American Heart Association (13SDG14270009) e do NIH (1DP2 CA186752-01 & 1-R01-GM-105646-01-A1) para SS
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| B2-AR-10 nm-Gas peptide sensor | Addgene | 47438 | https://www.addgene.org/Sivaraj_Sivaramakrishnan/ |
| GeneJET Plasmid Miniprep Kit | Fermentas/Fisher Sci | FERK0503 | Elute in 2 mM Tris elution buffer |
| HEK-293T-Flp-n cells | Life Technologies | R78007 | |
| Trypsin (0.25%) | Life Technologies | 25200056 | |
| DMEM- high glucose | Life Technologies | 11960-044 | Warm in 37 °C water bath before use |
| FBS, certified, Heat inactivated, US origin | Life Technologies | 10082147 | |
| Glutamax I 100x | Life Technologies | 35050061 | |
| HEPES | Corning | MT25060CL | |
| Opti-MEM | Life Technologies | 31985-070 | Reduced serum media; Bring to RT before use |
| XtremeGene HP transfection reagenet | Roche | 6366236001 | Highly recommended for its consistency. Bring to RT before use |
| FluoroMax 4 | Horiba | Use with FluorEssence V3.8 software | |
| 3-mm path length quartz cuvette | Starna | NC9729944(16.45F-Q-3/z8.5) | May require cuvette holder/adaptor for use in Fluorometer, available from Starna |
| Sc100-S3 Heated Circulating water bath pump | Fisher Scientific | 13-874-826 | Warm to 37 °C before use |
| Thermomixer Heat Block | Eppendorf | 22670000 | Warm to 37 °C before use |
| Ultrapure DNA/RNAse free water | Life Technologies | 10977015 | Use at RT |
| D(+)-glucose, anhydrous | Sigma | G5767 | |
| aprotinin from bovine lung | Sigma | A1153 | |
| leupeptin hemisulfate | EMD | 10-897 | |
| L-ascorbic acid, reagent grade | Sigma | A0278 | |
| (-)-isoproterenol (+)-bitartrate | Sigma | I2760 | Use fresh aliquot each experiment |
Referências
- Malik, R. U., et al. Detection of G Protein-selective G Protein-coupled Receptor (GPCR) Conformations in Live Cells. J. Biol. Chem. 288, 17167-17178 (2013).
- Oldham, W. M., Hamm, H. E. Heterotrimeric G protein activation by G-protein-coupled receptors. Nat. Rev. Mol. Cell Bio. 9, 60-71 (2008).
- Rasmussen, S. G., et al. Crystal structure of the β2 adrenergic receptor-Gs protein complex. Nature. 477, 549-555 (2011).
- Alexander, N. S., et al. Energetic analysis of the rhodopsin-G-protein complex links the α5 helix to GDP release. Nat. Struct. Mol. Biol. 21 (1), 56-63 (2014).
- Conklin, B. R., Farfel, Z., Lustig, K. D., Julius, D., Bourne, H. R. Substitution of three amino acids switches receptor specificity of Gqα to that of Giα. Nature. 363, 274-276 (1993).
- Conklin, B. R., et al. Carboxyl-Terminal Mutations of Gqα and Gsα That Alter the Fidelity of Receptor Activation. Mol. Pharmacol. 50, 855-890 (1996).
- Onaran, H. O., Costa, T. Where have all the active receptor states gone. Nat. Chem. Bio. 8, 674-677 (2012).
- Jares-Erijman, E. A., Jovin, T. M. FRET imaging. Nat Biotechnol. 21 (11), 1387-1395 (2003).
- Lohse, M. J., Nuber, S., Hoffman, C. Fluorescence/bioluminescence resonance energy transfer techniques to study G-protein-coupled receptor activation and signaling. Pharmacol Rev. 64 (2), 299-336 (2012).
- Vilardaga, J. P., Bünemann, M., Krasel, C., Castro, M., Lohse, M. J. Measurement of the millisecond activation switch of G protein-coupled receptors in living cells. Nat. Biotechnol. 21, 807-812 (2003).
- Bünemann, M., Frank, M., Lohse, M. J. Gi protein activation in intact cells involves subunit rearrangement rather than dissociation. Proc. Natl. Acad. Sci. USA. 100 (26), 16077-16082 (2003).
- Stumpf, A. D., Hoffman, C. Optical probes based on G protein-coupled receptors - added work or added value. Brit. J. Pharmacol. 173, 255-266 (2016).
- Sivaramakrishnan, S., Spudich, J. A. Systemic control of protein interaction using a modular ER/K α-helix linker. Proc. Natl. Acad. Sci. USA. 108, 20467-20472 (2011).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat. Methods. 2 (12), 905-909 (2005).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados