Method Article
Utilizando tecnologia de impressão 3D para mesclar MRI com Histologia: Um protocolo para o Cérebro Seccionamento
Neste Artigo
Resumo
O objetivo geral deste protocolo é o de alinhar com precisão ressonância (MRI) Os volumes de imagem magnética com cortes histológicos através da criação de suportes cerebrais impresso-3D personalizados e caixas slicer.
Resumo
Magnetic resonance imaging (MRI) allows for the delineation between normal and abnormal tissue on a macroscopic scale, sampling an entire tissue volume three-dimensionally. While MRI is an extremely sensitive tool for detecting tissue abnormalities, association of signal changes with an underlying pathological process is usually not straightforward. In the central nervous system, for example, inflammation, demyelination, axonal damage, gliosis, and neuronal death may all induce similar findings on MRI. As such, interpretation of MRI scans depends on the context, and radiological-histopathological correlation is therefore of the utmost importance. Unfortunately, traditional pathological sectioning of brain tissue is often imprecise and inconsistent, thus complicating the comparison between histology sections and MRI. This article presents novel methodology for accurately sectioning primate brain tissues and thus allowing precise matching between histology and MRI. The detailed protocol described in this article will assist investigators in applying this method, which relies on the creation of 3D printed brain slicers. Slightly modified, it can be easily implemented for brains of other species, including humans.
Introdução
In vivo MRI provides a noninvasive and sensitive measure of tissue integrity at the macroscopic level. Changes in MRI signal intensity seen in vivo are outcome measures in many ongoing clinical trials.1 While the intensity changes seen via MRI can identify areas of abnormality in the context of the whole brain, they are often not sufficiently specific to differentiate pathological processes. This is especially true of dynamic processes involving multiple pathologies. For example, in multiple sclerosis (MS) or its animal model, experimental autoimmune encephalomyelitis (EAE), inflammation, edema, myelin degradation, axonal destruction, gliosis, and neuronal death overlap. 2, 3 To obtain the necessary specificity regarding the underlying pathology, context must be taken into account, together with knowledge of the histology of the MRI-identified abnormal tissues.
However, even in well-controlled animal experiments, matching histology with in vivo MRI is fundamentally challenging for various reasons. First, the difference in dimensional scales between histology sections and MRI is of several orders of magnitude.4 Second, for proper comparison, the orientation of MRI slice plane must match the sectioning plane of the brain tissue when cut. Due to the shape of the brain, it is very difficult to make consistently straight and accurate cuts when the brain is sitting on a flat surface. Third, the large size of the brain relative to a potentially small area of interest (lesion, tumor, etc.) creates a "needle-in-a-haystack" scenario for the pathologist processing the tissue. Fourth, even when the target tissue is found, it is commonly processed in such a way as to render virtually impossible an association with the original MRI data. Finally, traditional pathological sectioning of brain tissue is often imprecise and inconsistent, further complicating the comparison between histology sections and MRI images.
Previous attempts to overcome these challenges relied on the use of deformational algorithms to coregister the data and/or placement of fiducial markers within or around the tissue as a reference.5, 6, 7, 8 The former approach requires complex computational models that are particularly susceptible to complications due to data formatting, imaging artifacts, and changes caused by tissue processing.4 On the other hand, the latter approach introduces the possibility of contaminating or otherwise harming the tissue itself.9
The approach described here improves the transition between modalities through the use of postmortem MRI to bridge the gap between in vivo MRI and histology. Postmortem MRI provides three-dimensional (3D) images of the brain at higher resolution than can be achieved in vivo and furthermore provides the data needed for producing a morphologically accurate model of the brain surface. This digital model can then be used to create a 3D-printed custom holder for the brain. With careful positioning, the brain holder allows for precise, MRI-oriented brain sectioning, reducing the need for complex mathematical algorithms, and enables a focus on specific regions for targeted sampling.
Our laboratory recently introduced new methods for creating custom brain holders and slicers using postmortem MRI and 3D-printing technology for human10 and marmoset brains.4 The two methods allow for a more accurate correlation between MRI and histology in a research setting, and ultimately allow a deeper understanding of the specific pathology underlying MRI abnormalities. Carefully designed experiments, in which the brain is sampled repeatedly over time in vivo, can provide context for interpretation of the pathology, which in turn can add specificity to interpretation of the MRI. Here, we present a modified protocol in a unified framework that can be applied to any brain tissue, whether it derive from nonhuman primates, rodents, or humans. We provide detailed instructions, and a corresponding video, for the marmoset sectioning. Although the overall protocol applies to any type of brain, due to differences in MRI acquisition and tissue size, as well as the challenges encountered when dealing with specific brain types, there are some differences in the approach depending on the type of brain being processed. In this presentation, sections with "human" will denote differences in protocol specific to the human brain.
Protocolo
Todo o manuseio e procedimentos aqui descritos animais foram realizados de acordo com um protocolo aprovado pelo Instituto Nacional de Distúrbios Neurológicos e Derrame Animal Care e do Comitê Use. Cérebros foram coletadas de Callithrix jacchus (Callithrix jacchus) induzidos a desenvolver EAE. 11 Os cérebros foram armazenados em formalina a 10% para entre 3 semanas e um ano após a eutanásia através de perfusão transcardial de paraformaldeído a 4%.
1. Postmortem MRI Preparação e Aquisição
- Marmoset Cérebro
- Prepara-se uma estação de trabalho com gaze de algodão, um tubo de centrífuga de 50 ml, pequenas espátulas, ~ 30 ml de um óleo fluorado, parafina, e o cérebro de saguim.
- Encha o tubo com o óleo fluorado e gaze para a marca de 20 ml. Comprime-se a gaze para remover as bolhas de ar ao longo do caminho.
- formalina gentilmente seca a partir da superfície do cérebro com uma toalha de papel. Inserir o cérebro com o pólo frontal para a parte inferioro tubo. Cuidadosamente proteger o cérebro no tubo usando mais de gaze em torno dos lados para fixar a sua posição. Consulte a seção complementar 11 para um método de criação de um berço cérebro MRI para a configuração de exames de ressonância magnética adicionais.
- Encher o resto do tubo com gaze e óleo fluorado. Remova cuidadosamente as bolhas de ar ao longo do caminho. Fixar a tampa e selar o tubo com parafina.
- Marque a tampa de acordo com a fissura inter-hemisférica. Enrole o tubo em uma toalha de papel e inseri-lo na bobina com top-centro da marca.
- Adquirir 2D girar eco T2. Os parâmetros são dados na Tabela 1.
- Abra os 10 anatômicas 150 micron aquisições weigted em T2 em Mipav e se registrar para a aquisição 6º.
NOTA: O registro é um algoritmo 3.5D registro automático otimizado com 9 graus de liberdade, em janela de interpolação de sincronismo, função de custo correlação cruzada normalizada, com a chamada algoritmo de busca do Brent do Powell. Rotações foram amostrados de 106; a -10 ° com rotações grosseiros incrementando 5 graus e rotações finas incrementando 1 °. Em seguida, calcular a média das imagens registradas: Utilities, Imagem imagens calculadora-Bulk, média.
- Cérebro humano
- Separa-se o cérebro anterior a partir do tronco cerebral usando um corte ao nível do mesencéfalo. 10 Os hemisférios também pode ser separada com um corte abaixo da linha média.
- Posicionar o cérebro anterior em um tubo cilíndrico com uma cúpula hemisférica numa das extremidades e um bocal na outra extremidade.
- Encha o tubo com um óleo fluorado através do bico. Remover bolhas de ar usando sucção suave para ~ 30 min através do bico.
- Adquirir 3D T1-MPRAGE. Os parâmetros são dados na Tabela 1.
2. Extração de superfície do cérebro: Mipav 7,2
- Abra o MRI na orientação coronal.
- Selecione algoritmos, ferramentas de transformação, Transform, Resample. Selecione o tamanho definido pelo usuário e fazer uma nova amostra para ISOTvoxels ropic: 0,1 x 0,1 x 0,1 mm. Salve o MRI resampled como Brain_MRI_Resampled. Humano: Resample para isotrópico voxels de 0,3 x 0,3 x 0,3 mm.
- Selecione Algoritmos, Filtros (espacial), a redução de ruído não-linear. Use as configurações padrão, clique em OK.
- Selecione Displays tabela de pesquisa e clique no botão dupla limiar. Arraste o controle deslizante no gráfico para cobrir todo o cérebro.
- Selecione Algoritmos, Segmentação, Threshold, Threshold usando min / max. Insira o valor localizado no canto inferior esquerdo do gráfico de intensidade (logo abaixo da escala) na caixa "limite inferior". Selecione "binário" no para o tipo de imagem de saída e desmarque "limiar invertido".
- Selecione Algoritmos, morfológica, preencher buracos. Marque a opção "Processo em 2.5D".
- Porque esta é uma ressonância magnética do cérebro completo, há algum espaço vazio entre a parte posterior do cérebro e do córtex que precisa ser preenchido. Cérebro humano: pular esta etapa.
- Usando linha de VOI, desenhar em uma conexãoentre a parte posterior do cérebro e do córtex em ambos os lados do cérebro, no ponto mais lateral. Continue este através do cérebro.
- Selecione conversão VOI, VOI, Tudo a máscara binária. Selecione Utilitários, calculadora Imagem. Selecionar ou a partir do menu do operador e selecione a máscara cérebro. Selecione "Promover destino tipo de imagem"
- Selecione Algoritmos, morfológica, preencher buracos. Marque a opção "Processo em 2.5D"
- Salvar a máscara binária como Brain_Model.nii.
- Selecione Algoritmos, superfície Extract (cubos de marcha). Selecionar imagem máscara, Salvar como Brain_Model.ply.
3. Seleção de Locais de fatia: Mipav 7,2
- Identificar tecido de interesse ou posição inicial. Calcula-se a espessura da placa que se destina. No cérebro sagüi, contando 30 fatias de ressonância magnética por seção, e 5 fatias de ressonância magnética por diferença de lâmina, cria seções 3 mm com lacunas lâmina 0,5 mm, resultando em ~ lajes de 3,5 mm. Cérebro humano: 20 fatias de ressonância magnética por seção, e 4 MRI slices por diferença de lâmina, cria seções 6 mm com lacunas de lâmina de 1,2 mm, resultando em ~ lajes de 7,2 mm.
- No local da primeira lacuna lâmina, clique em "caixa de VOI" e, em seguida, desenhe uma caixa sobre o cérebro. Clique dentro da caixa para selecioná-lo. Copie e cole o contorno para cada fatia MRI correspondente à diferença lâmina.
- Ir à frente pelo número de fatias de ressonância magnética correspondente à espessura de corte e copie e cole o contorno correspondente à diferença lâmina seguinte. Repita este processo através do cérebro.
- Selecione VOI, VOI conversão, Tudo a máscara binária. Salvar como Blade_Gaps.nii.
- Selecione Algoritmos, Surface Extract (cubos de marcha), imagem Máscara, Blade_Gaps.ply.
4. Criação de ressonância magnética Lâmina Mapa: Mipav 7,2
- Abra o Brain_MRI_Resampled e as imagens Blade_Gaps.nii.
- Com a imagem Blade_Gaps.nii selecionado, selecione Utilities, Imagem Math. Selecione Promover tipo de imagem e Multiply.Digite 10.000 como o valor.
- Selecione Utilitários, Image Calculator. Selecione Adicionar e selecione a imagem Brain_MRI_Resampled da caixa de imagem suspenso. Selecione Promover destino tipo de imagem.
- Guardar a imagem como Brain_BladeMap.nii.
- Ao clicar na vista triplanar, os locais em que o cérebro vai ser cortado pode ser visto em três vistas ortogonais.
5. Importação Cérebro e Blade Gap Surfaces: Netfabb Professional
- Selecione Part, Adicionar parte. Escolha os arquivos Brain_Model.ply e Blade_Gaps.ply
- Selecione o cérebro e clique em modo de reparo. No modo de reparação:
- Clique no botão de seleção de shell, e depois clique sobre o cérebro.
- Clique no botão de seleção de alternância para selecionar as outras malhas. Clique em Remover para excluir as outras malhas.
- Clique em Aplicar Reparar e remover a parte antiga.
- Botão direito do mouse sobre o cérebro. Escolha um movimento. Clique noPara botão de origem. Grave os parâmetros XYZ que aparecem. Estes parâmetros de tradução são necessários para manter o posicionamento da lâmina criada em Mipav. Clique em Traduzir. Em seguida, feche a janela. (Não clique traduzir mais de uma vez. Ele irá traduzir novamente usando os mesmos parâmetros.)
- Escolha o modelo Blade_Gaps. clique direito sobre a parte e selecione Mover. Digite os valores XYZ gravadas anteriormente nos parâmetros caixas XYZ. Agora clique em Traduzir e fechar a janela.
- Escolha o modelo Brain_Model e clique em modo de reparação. No modo de reparação:
- Clique no botão de seleção de shell, e depois clique sobre o cérebro.
- Botão direito do mouse e selecione Triângulos suave. Digite 4-5 iterações. Verifique Prevent encolhimento volume. Cérebro humano: 1-2 iterações.
- Clique direito, selecione Reduzir Triângulos. Digite 200000 na contagem triângulo destino e clique em Executar.
- Clique no reparo automático, reparação padrão. Em seguida, clique em Aplicar Repair
- Botão direito do mouse, Renomear. Renomeie o cérebro alisou como Smoothed_Brain_Model.
- Selecione Smoothed_Brain_Model. Botão direito do mouse, Exportação, STL.
6. Edição Cérebro Contours: Meshmixer
- Importar o Smoothed_Brain_Model em Meshmixer.
- Utilize as ferramentas de escultura e selecção a fazer ajustes na malha. As edições incluem:
- Use ferramentas de escultura, robusto lisas. Alise a superfície correspondente a uma linha de VOIs. Cérebro humano: pular esta etapa.
- Alise a superfície do córtex que serão viradas para baixo na caixa.
- Suavizar pequenas divots que poderiam ter sido criados no processo de entrosamento e edição.
- Escolha Análise, Inspector, AutoRepair todos.
- Exportar o Smoothed_Brain_Model como Smoothed_Edited_Brain_Model.
7. Criar o Slicer Box Cérebro: Netfabb Professional
- Selecione Part, Adicionar Part. Escolha o file Smoothed_Edited_Brain_Model.
- Selecione Part, Adicionar Part. Em seguida, selecione o cérebro arquivo STL Slicer Parts_Marmoset e clique em Abrir. Cérebro humano: Brain Slicer Parts_Human (arquivos de código Suplementar).
- clique direito sobre a parte e selecione estendidos, Shells para as partes. Escolha cada peça individualmente e clique direito de renomeá-los. (Clicando no olho ao lado de um objeto esconde-lo ou torna visível.) Renomear o grande caixa principal, a pequena caixa Sub, e a caixa de corte Box_Cutout, a forma hexágono Blade_Holder_Main, a pequena caixa plana Microtome Blade, eo meia tubo de objeto Cradle. Cérebro humano: não Blade_Holder_Main, Microtome Blade, ou berço.
- Esconder Principal, Sub, Blade, Blade_Holder_Main, Microtome Blade, e berço e usar shift-select para selecionar todos os seis e os Box_Cutout. Apenas o Box_Cutout e o Smoothed_Edited_Brain_Model deve ser visível, mas o Smoothed_Edited_Brain_Model não deve ser selecionada.
- Clique e arraste as peças selecionadas para ajustar a posição da caixa em relação ao cérebro.
- Posicionar o cérebro no centro da caixa. Postion o cérebro a uma profundidade suficiente na caixa para ser agarrados, mas não muito profundo para criar saliências que impedem a colocação adequada.
- Uma vez posicionado, o contorno caixa de cérebro podem ser testados quanto à saliências. Selecione o Box_Cutout eo Smoothed_Edited_Brain_Model. Selecione booleano operação.
- Clique na Smoothed_Edited_Brain_Model para transformá-lo vermelho.
- Selecione booleana Subtração, e aplicar os cálculos.
- Marque a caixa do cérebro para saliências que impediriam a tecido cerebral de ser colocado com segurança na caixa. Se estas saliências estão presentes, ajustar o cérebro por isso é menos profunda no interior da caixa. Se o cérebro é na profundidade desejada e saliênciasestão presentes, consulte a secção complementar 10 para uma solução para remover as saliências.
- Escolha o modelo Blade_Gaps. Botão direito do mouse, selecione Mover. Grave o valor Z posição, em seguida, fechar a janela. Esta será a posição da lâmina lacuna mais posterior.
- Selecione o STL lâmina que veio dos Slicer partes do cérebro. Botão direito do mouse, selecione Mover.
- Selecione tradução Absoluto. Insira o valor de Z a partir do 7.8. Para os valores de X e Y, insira o valor correspondente a partir das caixas de parâmetros posição atual
- Selecione o STL Blade. Direito clique, clique em Duplicar.
- Verifique Organizar partes. Digite o número total de lâminas na contagem total. Introduza o mesmo número na caixa de contagem Z. Digite a espessura da laje na abertura Z. Clique em Duplicar. Se a espessura da laje foi variada, as lâminas terão de ser posicionada individualmente. Duplicar cada nova lâmina do anterior (movimento de posterior para anterior)com a abertura z igual à espessura de corte para essa seção.
- Posicionar as peças Microtome lâmina em exatamente os mesmos intervalos que as lâminas do cortador de repetindo o passo 7,9 e 7,10 para o Microtome Blade. Cérebro humano: pular esta etapa.
- Selecione Microtome lâmina junto com a parte Blade_Holder_Main e selecione Operação booleana.
- Selecione todas as lâminas em Microtome lâmina para destacá-los vermelho. Clique em booleana Subtração, e aplicar os cálculos.
- Escolha um modo de reparação. Executar um reparo Estendida. Selecione aplicar o reparo e remova a parte antiga.
- Mudar o nome do Titular parte Blade. Exportar a peça como STL.
- Shift-select Smoothed_Edited_Brain_Model, todos os modelos de lâmina criadas na etapa anterior, eo Sub e Principal. Clique na operação booleana.
- Faça todas as peças, exceto vermelha principal por selecting-os e clicando na seta na caixa verde para movê-los para vermelho.
- Selecione booleana subtração e selecione aplicar cálculos.
- Clique em modo de reparo. No modo de reparação:
- Marque a caixa de saliências e pontos afiados. Estes podem ser suavizados em Netfabb ou Meshmixer.
- Clique reparo automático, reparação Estendida. Em seguida, aplique a reparação e remover a parte antiga
- Botão direito do mouse na caixa do cérebro reparado e renomeá-lo Cérebro Slicer Box. Exportar como um STL.
8. Imprimir o Slicer Box Cérebro na Ultimaker 2
- Marmoset Cérebro: Cura
- Importar o Slicer Box cérebro em Cura.
- Selecione Girar e arraste o círculo para girar a caixa por isso é plana sobre a cama.
- Ajuste as configurações de impressão: resolução camada de 0,1 mm, 50% de densidade de preenchimento, Raft.
- Selecione Caminho Salvar Ferramenta para cartão SD. (Print vez ~ 12 h.)
- Import porta-lâmina ema Cura e gire-o para os slots estão nas laterais e na face do hexágono está no XZ ou plano YZ.
- Duplicar o objeto.
- Ajuste as configurações de impressão: resolução camada de 0,2 mm, de 20% de densidade de preenchimento, borda.
- Selecione Caminho Salvar Ferramenta para cartão SD. (O tempo de impressão ~ 3 h)
- Cérebro humano: Cura
- Importar o Slicer Box cérebro em Cura e girar como em 8.1.2.
- Ajuste as configurações de impressão: resolução 0,2 milímetros de camada, 30-35% de densidade de preenchimento, Raft.
- Selecione Salvar caminho de ferramenta para o cartão SD. (O tempo de impressão ~ 70 h para a caixa hemisfério única.)
- No Ultimaker 2
- Aplique uma fina camada de cola bastão de cola para construir placa.
- Insira o cartão SD. Escolha de impressão e selecionar a parte.
9. Corte do Cérebro
- Marmoset Cérebro
- Prepara-se uma estação de trabalho com o cérebro fixo, o cortador de cérebro, dois suportes de lâmina, as lâminas do micrótomo, 1 ml de óleo fluorado, fpinças lat, luvas de protecção, e cassetes incorporação.
- Coloque novas lâminas do micrótomo nas ranhuras dos suportes de lâmina. Verifique se a borda chanfrada de cada lâmina está voltada para a mesma direção. Usar luvas de protecção ao manusear lâminas do micrótomo.
- Remover o cérebro de formalina e gentilmente secá-la.
- Coloque o cérebro para o cortador. Algumas gotas de óleo fluorado pode ser aplicado para o cérebro e slicer para permitir a fácil posicionamento. Certifique-se de que o cérebro é firmemente no lugar.
- Posicione os suportes de lâmina com as lâminas nos slots de blade correspondentes.
- Empurre os suportes de lâmina com firmeza e aplicar pressão equilíbrio lento para cortar através do cérebro.
- Remover cada laje, um de cada vez, a partir da parte frontal do cérebro. Isso ajuda a remover a lâmina micrótomo em frente de uma laje antes de remover a própria laje. Preste muita atenção à orientação anterior / posterior de cada laje.
- Tire fotos da uma anteriord superfície posterior de cada laje. As lajes posteriores provavelmente irá conter peças separadas, então preste atenção à orientação das peças para a incorporação. Coloque cada pedaço numa cassete de incorporação e colocá-los todos em uma solução de formol a 10%.
- Cérebro humano
- Cuidadosamente testar o ajuste do cérebro na caixa.
- Fatiar o cérebro a partir de uma extremidade usando um corte em ângulo, devagar mas com firmeza de cortar. Corte o cérebro através de cada abertura da lâmina.
- Remova cada laje de um de cada vez, prestando muita atenção ao número e à anterior orientação / posterior de cada laje.
- Tire fotos da superfície anterior e posterior de cada laje. Coloque placas em seladas 10% sacos de formalina. Cortes de tecido vão ser cortados a partir das placas e colocadas em cassetes para a incorporação.
10. Saliências Remoção na caixa Brain (Seção Suplementar)
- Retirando fatias: Meshmixer
- Importar o Smoothed_Edited_Brain_Model.
- Selecione Editar, faça fatias. Em fatias Marca:
- Selecione empilhado 3D, Z, digite a espessura de 1-2 mm. Clique Compute. Quando as fatias de carregar, clique em Aceitar.
- Selecione 1 ou 2 fatias com grandes perímetros perto da parte inferior do córtex. Estas fatias devem estar abaixo do nível da caixa de Sub.
- Exportar cada uma dessas fatias como Brain_Slice_ #.
- Estendendo as fatias fim de remover saliências: Netfabb Professional
- Importe os Brain_Slice_ # fatias.
- Duplicar cada Brain_Slice_ # (desmarcar organizar as peças se verificado).
- Clique direito em uma cópia de cada Brain_Slice_ # e selecione Escala.
- Desmarque proporção da escala fixa. Em seguida, dimensionar-se a fatia de cérebro na direcção de Y de modo a que irá atingir o nível da parte inferior da caixa do Sub.
- Renomear essas fatias Brain_Slice_Big_ #.
- Verifique a Y posição of o Brain_Slice_ originais # clicando com o botão direito sobre a peça e selecionar movimento. Registra a posição Y para cada uma das fatias originais Brain_Slice_ #.
- Executar o cálculo: Brain_Slice_ # [y posição] - (Brain_Slice_Big_ # [size y] - Brain_Slice _ # [size y])
- Selecione cada um dos Brain_Slice_Big_ # individualmente, clique direito e selecione movimento.
- Digite o valor calculado a partir 10.2.6 na caixa parâmetro de translação da Y. Para os parâmetros X e Z de tradução, introduza os valores localizados nas caixas de parâmetro posição atual. Selecione Absolute Tradução. Clique em Traduzir e fechar a janela.
NOTA: O # fatias Brain_Slice_Big_ será subtraído juntamente com o cérebro e lâminas ao fazer a caixa.
- Digite o valor calculado a partir 10.2.6 na caixa parâmetro de translação da Y. Para os parâmetros X e Z de tradução, introduza os valores localizados nas caixas de parâmetro posição atual. Selecione Absolute Tradução. Clique em Traduzir e fechar a janela.
11. Marmoset Brain MRI Cradle para digitalização adicionais
- Criando o berço do cérebro de MRI
- Certifique-se de que a superfície de topo do CrADLE é na mesma altura que o Box_Cutout. A profundidade ea posição do cérebro no berço deve ser configurado assim como é para o cortador.
- Shift-selecção para seleccionar o Smoothed_Edited_Brain_Model e Berço.
- Selecione operação booleana. Selecione o cérebro para destacá-lo vermelho, em seguida, selecione a subtração booleana. Em seguida, aplique os cálculos. (Além disso, selecione o # fatias Brain_Slice_Big_ se aplicável).
- Entre no modo de reparo para remover quaisquer pontas afiadas no contorno suporte, como feito anteriormente para o cortador. Selecione reparação Estendida. Aplique o reparo e remova a parte antiga.
- Botão direito do mouse para renomear a parte do cérebro de MRI Cradle. Selecione Export, STL.
- Imprimindo o berço: Cura
- Importar o MRI do cérebro Cradle em Cura e gire-a para que a parte plana, com o recorte cérebro está voltada para cima.
- Ajuste as configurações de impressão: resolução camada de 0,1 mm, de 100% de densidade de preenchimento, Raft.
- Selecione Salvar caminho de ferramenta para o cartão SD. (O tempo de impressão ~ 10 h)
- Imprimir no Ultimaker 2, conforme descrito em 8.3.
- Aquisição de alta resolução T2 * MRI usando a base
- formalina gentilmente seca a partir da superfície do cérebro com uma toalha de papel.
- Posicionar o cérebro no suporte, como descrito para o cortador.
- Deslize o cérebro e berço para o tubo de centrífuga de 50 ml. Preenchê-lo até a borda com óleo fluorado.
- Suavemente apertar o tubo para permitir que bolhas de ar se escape a partir do cérebro. Insira o Cap Insert dentro da margem interna da tampa do tubo para evitar bolhas de ar formando lá. Fixar a tampa, e selar o tubo com parafina.
- Colocar o tubo em serpentina como anteriormente descrito. 3D parâmetros T2 * são dadas na Tabela 1.
- Abra as 18 anatômicas 100 micron T2 aquisições * ponderadas em Mipav e se registrar para a aquisição de 10 th. os parâmetros de registo são os mesmos que em 1.1.7. Em média, as imagens registradas: Utilities, Imagem imagens calculadora-Bulk, Média.
Resultados
O fluxo de trabalho deste método é resumido na Figura 1. Uma vez que o cérebro está cortado, uma comparação visual entre as imagens de RM e imagens das superfícies superficiais das lajes mostra um bom jogo orientação através de múltiplas placas (Figura 2). Depois de as placas são embebidos em parafina, seccionados que estão num micrótomo e coradas. Uma comparação mais aprofundada entre a alta resolução post-mortem de ressonância magnética e os cortes histológicos corados demonstra uma correspondência precisa e consistente entre todas as estruturas do cérebro de saguim (Figura 3).
Neste modelo animal de MS, os animais desenvolvem lesões de substância branca espalhados por toda a substância branca cerebral. Estas lesões podem ser detectados de forma não invasiva através da realização de ressonância magnética. A Figura 4 demonstra a capacidade desta técnica para elucidar o substrato patológico dos resultados de MRI. Pequenas lesões detectadas na ressonância magnética in vivo podeser rastreados em ambos postmortem ressonância magnética e histologia. Tal como mostrado nas inserções, desmielinização no interior das lesões é um dos principais componentes que promovem a mudança do sinal de RM (hiperintensidade comparação com o tecido circundante). A histologia e postmortem MRI também pode mostrar lesões perdidas em in vivo ressonância magnética (Figura 4).
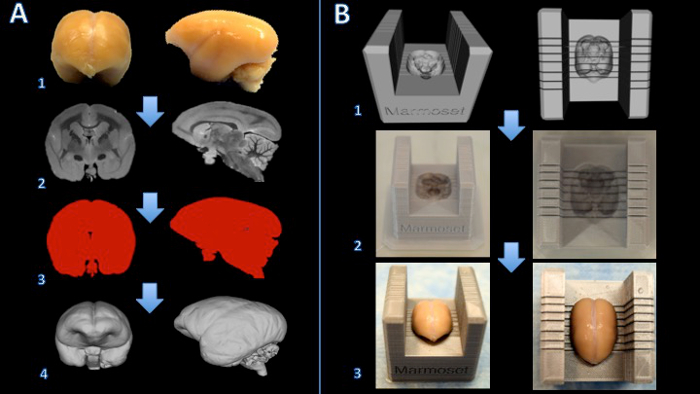
Figura 1. Fluxo de trabalho para a criação de uma caixa de cortador de sagüi cérebro. O cérebro é fixado com formalina (A1) e uma ressonância magnética ponderadas em T2 é adquirido com voxels isotrópicos de 150 um por aresta (A2). As imagens são processadas e thresholded para criar uma máscara binária (A3). A superfície é então processado em software de modelagem 3D (A4). A subtração booleana entre um modelo de cortador eo modelo de cérebro cria um modelo digital do cortador de cérebro (B1). A caixa de cortador de cérebro é impresso em uma impressora 3D (B2). O cérebro é então colocado firmemente na caixa de cortador decorte (B3). Por favor clique aqui para ver uma versão maior desta figura.
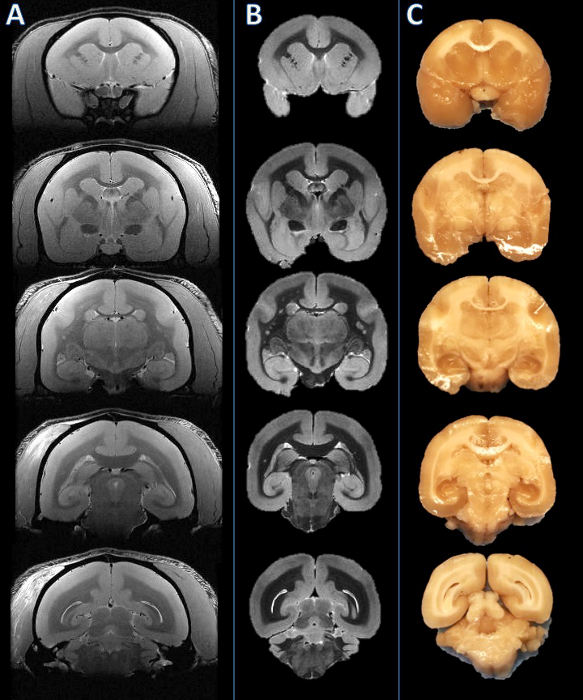
Figura 2. Da esquerda para a direita: In vivo ressonância magnética, postmortem MRI, ea fotografia de placas de tecido. Cortando aviões foram estabelecidas com base na autópsia ressonância magnética (B) e visualmente em comparação com o correspondente ressonância magnética in vivo fatia (A). O cérebro foi então cortado, e as placas resultantes foram encontrados para ser consistente (C). Por favor clique aqui para ver uma versão maior desta figura.
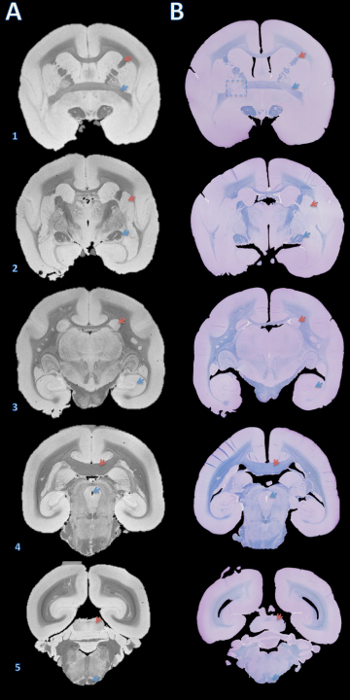
Figura 3. De alta resolução MRI postmortem e histologiaseção correspondente. Placas foram embebidas em parafina, cortadas com um micrótomo para 4 uM secções e coradas com violeta rápido de cresilo e azul (B). As secções foram então visualmente combinado com T2 100 uM * ressonância magnética ponderadas com base em estruturas cerebrais (A). Detalhes para adquirir esta imagem estão na seção complementar do protocolo e Tabela 1. As estruturas cerebrais: (1) seta vermelha = cápsula interna, seta azul = comissura anterior; (2) seta vermelha = putâmen, seta azul = trato óptico; (3) seta vermelha = caudado, seta azul = hipocampo; (4) seta vermelha = corpo caloso, seta azul = aqueduto cerebral; (5) seta vermelha = colículo inferior, seta azul = trato piramidal. A caixa a tracejado na B1 indica uma fatia sempre que, durante o corte do cérebro ou inclusão em parafina, um erro causou uma ligeira rotação em torno do eixo Y, levando a incompatibilidade da comissura anterior do lado esquerdo. Por favor clique aqui para ver umaversão maior desta figura.
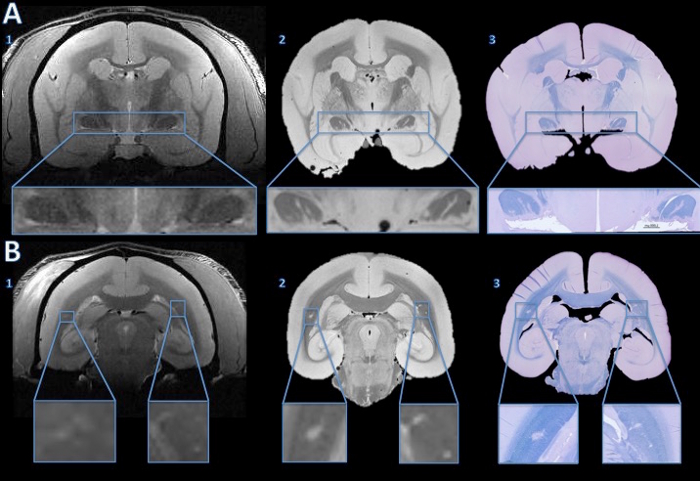
Figura 4. Seguimento lesões de MRI in vivo para a seção de histologia. A in vivo MRI não mostraram nenhuma evidência convincente de hipersinal anormal para sugerir lesões em qualquer trato óptico (A1). No entanto, a alta MRI resolução postmortem mostra claros intensos linhas hiper em ambas as vias ópticas (A2). A mancha azul violeta / cresyl rápida de uma seção histologia 4 mm mostra que as áreas hiperintensas visto na ex vivo MRI são desmielinização (A3). Na substância branca cerebral, in vivo MRI mostra hiperintensidade sutil bilateralmente (B1, ampliado nas inserções). As áreas hipertensas são mais evidentes no postmortem MRI (B2) de alta resolução. A mancha LFB de uma seção histologia 4 mm mostra que estas áreas são de desmielinização (B3). Após a comparação com a linha de base in vivo ressonância magnética e uma mancha hemotoxilina-and-eosina, o lado direito foi determinado para ser uma anormalidade anatômica, não uma lesão demyelinated. Por favor clique aqui para ver uma versão maior desta figura.

Arquivos de código suplementar. Brain_Slicer_Parts_Marmoset.stl: Por favor, clique aqui para fazer o download deste arquivo. Brain_Slicer_Parts_Human.stl: Por favor, clique aqui para fazer o download deste arquivo. Cap_Insert.stl: Por favor, clique aqui para fazer o download deste arquivo.
Discussão
O protocolo descrito aqui permite uma comparação precisa entre a RM e cortes histológicos. O protocolo é apresentada num formato unificado que pode ser aplicado aos cérebros de seres humanos ou animais pequenos, tais como saguis ou roedores. Diferenças específicas para grande (humano) e pequenas (primatas não humanos e roedores) cérebros são destacadas, e no vídeo e figuras de acompanhamento, demonstramos a aplicação no sagüi. Embora a abordagem é simples, o método requer muitos passos, bem como a utilização de vários tipos de software. Além disso, várias questões que possam afectar a precisão deste método é importante mencionar.
A qualidade de imagem da ressonância magnética in vivo é um fator importante. Para minimizar a disparidade na resolução da imagem entre a RM e imagens histológicas digitalizadas, deve ser usado o menor tamanho possível MRI voxel. Este conceito também se aplica à qualidade de imagem da ressonância magnética pós-morte. Embora o aumentotempo de aquisição em MRI postmortem permite muito maior resolução de imagem, a preparação pode introduzir artefatos de imagem, tais como quedas de sinal focais relacionadas com bolhas de ar. Estes artefactos podem obscurecer as áreas do tecido, bem como afectar o seu contorno. Além disso, as dimensões do tecido sobre a TMA post mortem não são susceptíveis de ser afectadas pelo processo de fixação e a duração. Enquanto a in vivo para ex vivo MRI jogo pode estar intimamente aproximada utilizando marcos anatômicos em configuração geometria fatia durante a aquisição, o registro não-linear ainda seria necessário para chegar a um maior grau de precisão em combinar essas duas imagens de ressonância magnética.
O desenho do suporte do cérebro e slicer é também um passo crucial. Na criação do modelo digital de cérebro, um algoritmo de suavização é aplicado que aumenta ligeiramente do modelo em relação ao cérebro fixo. Isso permite a fácil inserção do cérebro em seu suporte e slicer e reduz bordas afiadas no titular &# 39; s de contorno. No entanto, se o modelo é muito grande (por exemplo, por mais de 5%), o cérebro pode mover-se durante a MRI pós-morte e / ou o corte. Outro ponto importante é o de adaptar a concepção do modelo de cérebro de modo a que o cerebelo é adequadamente colocada no interior do objecto 3D impressa. Isto pode ser particularmente desafiador quando o cerebelo foi danificado durante a extração cerebral na autópsia.
Ao imprimir o cortador de cérebro e titular, o tipo de impressora 3D também deve ser escolhida com cuidado. Algumas impressoras multi-jet necessitam de pós-processamento utilizando um forno para remover o material de apoio. Embora estas impressoras podem produzir objectos que são à prova de água e relativamente mais durável do que a área de trabalho fundido modelagem por deposição em impressoras (FDM), o processo de aquecimento para remover suportes podem deformar ligeiramente a caixa, criando intervalos de lâminas que não são perfeitamente perpendicular ao contorno cérebro.
O processo de corte do cérebro é mais um passo crucial. Antes th cortee cérebro inteiro em lajes, é importante ter a certeza que o cérebro está sentado firmemente dentro do cortador de cérebro: não deve haver movimento quando ligeira pressão é aplicada sobre o cérebro. Isto tornará possível para as lâminas para cortar o cérebro na localização precisa definir pelos investigadores. Uma pressão contínua, equilibrada deve ser aplicada a ambos os suportes de lâmina durante o corte. Dependendo da nitidez das lâminas e a rigidez do tecido, um ligeiro movimento de corte transversal pode ser vantajosa para a manutenção da superfície de corte plana.
O processo de incorporação de parafina pode também ser uma fonte de desalinhamento entre RM e histologia. Se a laje de tecido não está sentado plana contra a caixa durante o processo de incorporação, haverá uma inclinação entre o plano de corte do micrótomo e o local da superfície da laje. Isto irá exigir o corte secções inutilizáveis para encontrar uma superfície plana em que todo o tecido é exposto. Uma maneira de corrigir a inclinaçãoé mudando o ângulo do plano de visualização na MRI postmortem high-isotrópico-resolução. No entanto, isso é quase impossível de executar no in vivo ressonância magnética que é geralmente adquirida com a resolução anisotrópica (tipicamente grossas cortes coronais).
Finalmente, o tecido pode experimentar alguma deformação durante o período de formalina e embebidos em parafina de fixação (encolhimento), bem como durante a preparação das lâminas (dobráveis, rachaduras, rugas). Algumas destas deformações pode ser corrigida com a colocação das secções de 4-5 ^ M, num banho de água antes de transferir em lâminas. Outras deformações pode ser parcialmente resolvido através da realização de coregistration imagem deformável das imagens digitalizadas histológicas para as imagens de ressonância magnética pós-morte. No entanto, minimizando as deformações com a prática cuidadosa e hábil é a abordagem mais eficaz para combinar volumes de ressonância magnética para histologia seções.
Em conclusão, a metodologia aqui apresentada permite investigators para avaliar com precisão a patologia subjacente dos resultados de MRI. Em termos mais gerais, é uma abordagem promissora para a identificação e / ou validação de novos biomarcadores de ressonância magnética para estudos de investigação que visam processos patológicos específicos, tais como inflamação ou remielinização.
Divulgações
The authors declare that they have no competing financial interests.
Agradecimentos
The Intramural Research Program of NINDS supported this study. We thank the NIH Functional Magnetic Resonance Imaging Facility. We thank Jennifer Lefeuvre and Cecil Chern-Chyi Yen for assistance with postmortem MRI acquisition. We thank John Ostuni and the Section on Instrumentation Core Facility for assistance with 3D printing. Figure 1 of this work used snapshots from MeshLab, a tool developed with the support of the 3D-CoForm project.
Materiais
| Name | Company | Catalog Number | Comments |
| 7T/30cm USR AVIII Bruker MRI | Bruker Biospin | ||
| 38 mm Bruker Biospin volume coil | Bruker Biospin | ||
| Fomblin | Solvay Solexis | ||
| 50 ml Falcon Centrifuge Tubes, Polypropylene, Sterile | Corning | 21008-951 | |
| Fisherbrand Gauze Sponges | Fisher Scientrific | 13-761-52 | |
| Parafilm M All-Purpose Laboratory Film | Bemis | ||
| Leica RM2235 rotary microtome | Leica | ||
| Leica Disposable Blades, low profile (819) | Leica | ||
| Cresyl Violet Acetate, 0.1% Aqueous | Electron Microscopy Sciences | 26089-01 | |
| Luxol Fast Blue, 0.1% in 95% Alcohol | Electron Microscopy Sciences | 26056-15 | |
| ETOH | |||
| Ultimaker 2 Extended | Ultimaker | ||
| .75 kg Official Ultimaker Branded PLA Filament, 2.85 mm, Silver Metallic | Ultimaker | ||
| Axio Observer.Z1 | Zeiss | ||
| Zen 2 (Blue Edition) | Zeiss | ||
| Netfabb Professional 5.0.1 | Netfabb | http://www.netfabb.com/professional.php | |
| Meshmixer 10.9.332 | Autodesk | http://www.meshmixer.com/download.html | |
| Mipav 7.2 | NIH CIT | http://mipav.cit.nih.edu | |
| Cura | Ultimaker | https://ultimaker.com/en/products/cura-software |
Referências
- Evans, A. C., Frank, J. A., Antel, J., Miller, D. H. The role of MRI in clinical trials of multiple sclerosis: comparison of image processing techniques. Ann Neurol. 41 (1), 125-132 (1997).
- 't Hart, B. A., van Kooyk, Y., Geurts, J. J. G., Gran, B. The primate autoimmune encephalomyelitis model; a bridge between mouse and man. Ann Clin Transl Neurol. 2 (5), 581-593 (2015).
- Ontaneda, D., Hyland, M., Cohen, J. A. Multiple sclerosis: new insights in pathogenesis and novel therapeutics. Annu Rev Med. 63, 389-404 (2012).
- Guy, J. R., Sati, P., Leibovitch, E., Jacobson, S., Silva, A. C., Reich, D. S. Custom fit 3D-printed brain holders for comparison of histology with MRI in marmosets. J Neurosci Methods. 257, 55-63 (2016).
- Breen, M. S., Lazebnik, R. S., Wilson, D. L. Three-dimensional registration of magnetic resonance image data to histological sections with model-based evaluation. Ann Biomed Eng. 33 (8), 1100-1112 (2005).
- Dauguet, J., et al. Three-dimensional reconstruction of stained histological slices and 3D non-linear registration with in-vivo MRI for whole baboon brain. J Neurosci Methods. 164 (1), 191-204 (2007).
- McGrath, D. M., Vlad, R. M., Foltz, W. D., Brock, K. K. Technical note: fiducial markers for correlation of whole-specimen histopathology with MR imaging at 7 tesla. Med Phys. 37, 2321-2328 (2010).
- Schormann, T., Zilles, K. Three-Dimensional linear and nonlinear transformations: An integration of light microscopical and MRI data. Hum Brain Mapp. 6, 339-347 (1998).
- Jiang, L., et al. Combined MR, fluorescence and histology imaging strategy in a human breast tumor xenograft model. NMR Biomed. 26 (3), 285-298 (2013).
- Absinta, M., et al. Postmortem magnetic resonance imaging to guide the pathologic cut: individualized, 3-dimensionally printed cutting boxes for fixed brains. J Neuropathol Exp Neurol. 73 (8), 780-788 (2014).
- Gaitán, M. I., et al. Perivenular brain lesions in a primate multiple sclerosis model at 7-tesla magnetic resonance imaging. Mult Scler. 20 (1), 64-71 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados