Method Article
Identificação de mediadores do Receptor de células T através da triagem de bibliotecas de inibidor químico de sinalização
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este papel utiliza um ensaio de fluxo cytometry-baseado para bibliotecas de tela de inibidores químicos para a identificação de inibidores e os alvos que influenciam a sinalização do receptor de células T. Os métodos descritos aqui também podem ser expandidos para as sessões de alta produtividade.
Resumo
O receptor de células T (TCR) sinalizando o caminho é composto por uma infinidade de mediadores que transmitem sinais após a ativação da TCR. Diferentes estratégias têm sido propostas e implementadas para a identificação de novos mediadores de TCR sinalização, que melhoraria a compreensão dos processos de células T, incluindo a ativação e a seleção do timo. Descrevemos um ensaio de triagem que permite a identificação de moléculas que influenciam a TCR sinalização baseado na ativação do desenvolvimento de timócitos. Sinais de forte TCR causam timócitos em desenvolvimento ativar a maquinaria de apoptose em um processo conhecido como seleção negativa. Através da aplicação de inibidores da quinase, aqueles com alvos que afetam TCR sinalização são capazes de substituir o processo de seleção negativa. O método detalhado neste artigo pode ser usado para identificar inibidores de quinases canônicos com funções estabelecidas nas vias de sinalização do TCR e também inibidores de quinases novos ainda a estabelecer-se em vias de sinalização do TCR. A estratégia de rastreio aqui pode ser aplicada aos ecrãs de maior rendimento para a identificação de novos alvos druggable na TCR sinalização.
Introdução
As células T são de uma linhagem de linfócitos que desempenham um papel crucial na manutenção da imunidade adaptativa. Eles expressam o TCR, que lhes permite reconhecer seus ligantes, complexos consistindo de uma molécula de complexo principal de histocompatibilidade (MHC) com um peptídeo ligado, que são encontradas nas superfícies das apresentadoras células (APCs). O acionamento do TCR sinalização via através da interação de TCR/MHC é crucial para a ativação de células T e desenvolvimento1.
No desenvolvimento de células T, osso medula-derivado de células-tronco hematopoiéticas (linfócitos) migram para o timo, onde sofrem diferenciação e passar pelas etapas de linhagem de células T progressão2. Timócitos (DP) de duplo-positivo, expressando o CD4 e CD8 coreceptors, envolver-se com autopeptídeo / MHC sobre as APCs. Timócitos com uma moderada afinidade com seus ligantes autopeptídeo / MHC amadurecem para se tornar timócitos (SP) CD4 ou CD8 de single-positivo, um processo denominado como selecção positiva. Por outro lado, timócitos que recebem estimulação excessiva de TCR através do autopeptídeo / MHCs sofrem apoptose através de seleção negativa3,4. Esse processo de apoptose induzida por estimulação, caspase dependente pode ser imitado em vitro , estimulando os timócitos, por exemplo, com grânulos de revestido de anticorpo anti-CD3/285. Células T maduras que passa o processo de seleção são ativadas por ligantes não-autopeptídeo / MHC de APCs na periferia. Autopeptídeo / MHCs ainda são relevantes para as células T periféricas, no contexto da tônica para a sobrevivência e proliferação homeostática, a diferenciação de células T de auxiliar e o reforço de respostas de células T para não-autopeptídeo / MHCs através de sinalização coagonism6,7,8,9. Vinculação de TCR de alta afinidade para o ligante de peptídeo/MHC ativa várias vias de sinalização a jusante, que envolvem muitas moléculas de sinalização, formando um complexo TCR sinalização rede10. As vias de sinalização de TCR têm sido estudadas por várias décadas, e ainda a descoberta de novos mediadores da via não mostra sinais de diminuir11,12. A modulação de vias de sinalização TCR tem relevância clínica e pode envolver potenciando respostas de células T para aplicações imunoterapia ou a inibição de respostas de células T para o controle de auto-imunidade13. Estratégias para a modulação de respostas de células T dependem, principalmente, o rompimento da quinase ou fosfatase atividade14,15,16.
Descrevemos um pedido de um ensaio de fluxo cytometry-baseado para o rastreio de pequenos compostos químicos de sua capacidade de modular a TCR sinalização e ativação de células T17. O ensaio baseia-se sobre o fenômeno de timócitos ativando a via de apoptose quando exposto a fortes sinais TCR. O ensaio é suficientemente sensível para identificar as alterações da força de estimulação; Incubar timócitos expressando TCR transgênico com tetrâmeros de peptídeo/MHC com o aumento da afinidade resultou em um aumento correspondente na caspase ativação-usado como uma medida da resposta apoptotic5. Para a tela, usamos uma biblioteca de inibidores da quinase e avaliar sua capacidade de modular a resposta de timócitos para sinais fortes de TCR.
Várias estratégias de fluxo cytometry-baseado ou fluorescência-repórter têm sido descritas para o rastreio da elevado-produção de uma variedade de fenótipos de ativação periférica em vários subconjuntos de células T. Tais estratégias incluem a utilização de genéticas repórteres fluorescentes para avaliar o momento e a magnitude da ativação de células T18, o uso de degranulação como uma leitura de células T citotóxicas atividade19,20e a análise do fosforilação de várias proteínas envolvidas na sinalização21de celular.
O ensaio de triagem aqui apresentado é capaz de identificar com sucesso compostos que inibem a moléculas canônicas da TCR sinalização de via, bem como potenciais, novos compostos com efeitos inibitórios na TCR sinalização. Por exemplo, identificamos inibidores da GSK3β e Hsp90 como novos compostos que afetam respostas de células T17. O ensaio é capaz de distinguir os inibidores que interferem com a transdução de sinal, devido a uma redução na resposta apoptótica, dos efeitos TCR-independente dos inibidores na toxicidade celular. Além da indução da apoptose, também medimos o upregulation de CD69 e TCR downregulation como marcadores de ativação. Como TCR sinalização redes são complexas, o uso de várias leituras pode aumentar as chances de descobrir moléculas com efeitos específicos em um único caminho. Aqui, apresentamos também a utilização de um protocolo de centrifugação independente como uma alternativa de alto rendimento para o protocolo original durante a coloração das células em preparação para a análise do fluxo cytometric. O ensaio descrito neste artigo usa uma pequena biblioteca composta de inibidores da quinase, mas, em princípio, pode ser usada para rastreio de taxa de transferência maior. A biblioteca de escolha também pode incorporar uma variedade de outras moléculas ou inibidores.
Protocolo
Neste estudo, utilizaram-se ratos de C57Bl/6 de masculino e femininos de 6 a 8 semanas de idade. Os ratos foram criados na instalação de animais na Universidade Nacional de Cingapura (Singapore). A Universidade Nacional de Cingapura institucional cuidados com animais e uso Comité (IACUC) aprovaram todas as experimentações com animais.
1. preparação da suspensão de timócitos
- Eutanásia nos ratos em uma câmara de2 CO.
- Execute as etapas subsequentes em uma capa de tecido-cultura para evitar qualquer contaminação das culturas celulares. Proteger a carcaça do mouse à placa de dissecação, usando pinos e pulverizar o mouse com etanol a 70%.
- Usando um par de tesouras, faça uma incisão vertical no lado ventral, a partir do abdômen em direção a mandíbula. Fazer outras incisões ao longo de cada uma das patas. Estique a pele para expor a caixa torácica e fixá-lo.
- Corte o diafragma e ambos os lados da caixa torácica da extremidade posterior com um par de tesouras. Levantar a caixa torácica e fixá-lo para baixo para expor o timo. Separe os tecidos conjuntivos anexados para o Timo e extrair o Timo usando um par de pinças curvas.
- Lugar do timo em um poço de uma placa de 6 contendo 5 mL de mídia RPMI completa.
Nota: Considere a adição de 10% carvão-despojado fetal de soro bovino (FBS) aos meios de comunicação para melhorar a viabilidade de timócitos se um longo tempo de espera é esperado entre a dissecção e o ensaio de estimulação. - Delicadamente, Triture o timo, usando o fim brusco de uma seringa e passar as células através de um filtro de célula de 70 µM. Alternativamente, para coletar timócitos em uma condição mais saudável, considere o uso de dois pares de pinças para espremer o Timo e coletar os timócitos que o fluxo do epitélio tímico.
- Proceda à contagem, de células usando um hemocytometer ou qualquer célula automatizada contando o instrumento.
2. titulação de inibidores da quinase de concentrações não tóxica
Nota: Esta seção se concentra na preparação os inibidores para o uso nas telas de ativação de células T. Inibidores usados em concentrações elevadas podem causar morte celular, que é uma leitura das telas de ativação de células T. A série de diluições dos inibidores visa determinar a concentração final dos inibidores individuais que não deve induzir apoptose independente da estimulação de TCR. A biblioteca de inibidores da quinase utilizado neste estudo foi adquirida de um fornecedor externo. Lista de inibidores é incluída na Tabela de materiais.
-
Preparação de placas de inibidores da quinase em concentrações inferiores
- Para preparar um prato de inibidores em 1mm, adicione 10 µ l de inibidores a 90 µ l de Dimetilsulfóxido (DMSO) para todos os inibidores.
Nota: Os inibidores da biblioteca da pequeno-molécula utilizada neste estudo vêm em uma concentração das ações de 10 mM. Se os inibidores são em forma de pelota, siga os passos recomendados reconstituição dos fornecedores. Se os inibidores não são fornecidos em 10mm, preparar as placas de inibidor em concentrações adequadas alternativas em vez disso e preparar diluições em série separadas dos inibidores com um factor de diluição adequado.
Atenção: Em caso de inibidores de tóxicos, siga as instruções do fabricante sobre a manipulação segura e eliminação. - Para preparar um prato de inibidores a 0,1 mM, adicione 10 µ l de inibidores da placa da inibidores de 1 mM a 90 µ l de DMSO.
- Para preparar um prato de inibidores a 0,01 mM, adicione 10 µ l de inibidores da placa da inibidores de 0,1 mM a 90 µ l de DMSO.
- Para preparar um prato de inibidores em 1mm, adicione 10 µ l de inibidores a 90 µ l de Dimetilsulfóxido (DMSO) para todos os inibidores.
-
Tratamento de timócitos com inibidores da quinase
- Prepare uma suspensão de timócitos conforme seção 1.
- Dilua timócitos em RPMI completo para obter uma suspensão de timócitos de 5 x 106 células/mL.
- Adicione 200 µ l de suspensão de timócitos a todos os poços de uma placa de 96 poços, com uma pipeta multicanal.
- Para cada poço, adicione 2 µ l de inibidores da correspondente bem da placa contendo inibidores de 1 mM (a concentração final dos inibidores é 10 µM).
- No mesmo prato, prepare quatro poços de controlos, quatro poços de 5 µM tratados com dexametasona controlos positivos e quatro poços de controles negativos tratados com veículo, adicionando-se 2 µ l de DMSO.
- Incubar os timócitos em um 37 ° C, 5% CO2 incubadora para 17-20 h (ou durante a noite).
-
Determinação das concentrações adequadas de inibidores individuais
- Girar a placa de timócitos a 300 x g e 4 ° C, durante 5 min. Ressuspender as células em 250 µ l de tampão de lavagem FACS.
- Executar uma fluxo cytometric análise das amostras e analisar os resultados com um programa de análise de citometria de fluxo.
- Determine a porcentagem de células vivas, com base na retenção de FSC-SSC. A estratégia associada é mostrada na figura 1B.
- Calcule uma média da porcentagem de células vivas, com base nos controles de DMSO-tratados, que são normalizados em 100%. Defina uma janela arbitrária de morte celular aceitável (e.g.,20%). Inibidores que resultaram em uma porcentagem de células vivas, abaixo dessa janela (i.., abaixo de 80% dos controles tratados com DMSO) estão a ser testado novamente em concentrações inferiores.
- Para os inibidores que não passe os critérios de viabilidade na etapa 2.3.4, repita os passos de passos 2.2.1 - 2.3.4, mas uso a placa de inibidores em 0,1 mM para etapa 2.2.4 em vez da placa que contém os inibidores de 1 mM. A concentração final de inibidores usado aqui é 1 µM.
- Para os inibidores que ainda produzem altos níveis de morte celular em 1 µM, testar os inibidores em 0.1 µM. Repita os passos 2.2.1 - 2.3.4, mas usar a placa de inibidores em 0,01 milímetros na etapa 2.2.4. A concentração final de inibidores usado aqui é 0.1 µM.
-
Preparação de um prato de estoque de inibidores da quinase
- Para inibidores ser usado em 10 µM, adicione 10 µ l de inibidores de 10 mM a 10 µ l de DMSO.
- Para inibidores ser usado em 1 µM, adicione 1 µ l de inibidores de 10 mM a 19 µ l de DMSO.
- Para inibidores ser usado em 0.1 µM, adicione 1 µ l de inibidores de 10 mM, para µ l 199 de DMSO (Figura 1).
Nota: A placa de estoque preparada de inibidores é 500 x concentração da concentração final pretendida quando adicionado para as suspensões de timócitos. A placa de estoque de inibidores pode ser preparada em tiras PCR ou em placas de 96 poços. - A placa de estoque de inibidores da quinase pode ser aplicada a timócitos para triagem em um sistema convencional dependente de centrifugação (seção 3; ver figura 1A, métodos 1 e 2) ou em um sistema independente de centrifugação alternativo (secção 4; ver Figura 1A, método 3).
3. Biblioteca de inibidor de quinase rastreio (teste de centrífuga-baseado convencional)
-
Tratamento de timócitos com inibidores da quinase
- Prepare uma suspensão de timócitos conforme seção 1.
- Dilua timócitos em RPMI completo para obter uma suspensão de timócitos de 5 x 106 células/mL.
- Adicione 200 µ l de timócitos em cada poço de uma placa de 96 poços, com uma pipeta multicanal. Coloque a placa no gelo.
- Adicione 0,5 µ l de inibidores para a placa de 96 poços, dos poços de correspondente da placa de estoque inibidor preparado na secção 2.4.
- Prepare oito poços de controles não tratados. Prepare quatro poços de controles tratados com veículo, acrescentando 0,5 µ l de DMSO. Prepare quatro poços de 5 controles de tratados com dexametasona µM (Figura 2).
-
Estimulação de timócitos usando anti-CD3/CD28 grânulos
- Tomar 1 mL de grânulos e lave os grânulos com 2 mL de PBS. Separar as contas usando uma base magnética e aspirar a solução. Resuspenda as contas em 5 mL de RPMI completa.
Nota: A proporção de grânulos de células é de 1 a 2,5. Ajuste a quantidade de grânulos de tomar, dependendo do número de poços para estimular e o número de timócitos usado. - Adicione 50 µ l de grânulos para cada amostra tratada com inibidor, quatro amostras tratadas DMSO e quatro das oito amostras não tratadas. Adicione 50 µ l de RPMI completa aos restantes quatro poços não tratados. A Figura 2 mostra o layout geral da placa.
- Misture o conteúdo dos poços com uma pipeta multicanal.
- Incubar os timócitos em um 37 ° C, 5% CO2 incubadora para 17-20 h (ou durante a noite).
- Tomar 1 mL de grânulos e lave os grânulos com 2 mL de PBS. Separar as contas usando uma base magnética e aspirar a solução. Resuspenda as contas em 5 mL de RPMI completa.
-
Coloração de antígenos de superfície
- Prepare uma mistura de coloração de anticorpo que contém anti-TCRβ, anti-CD4, anti-CD8 e anticorpos anti-CD69. Dilua a anticorpos em tampão de lavagem FACS (PBS suplementado com 0,5% albumina de soro bovino [BSA]) na proporção de 1: 200 (v/v).
Nota: Considere otimizando os anticorpos usados para a coloração, em vez de usar diluições de anticorpo fixa, para minimizar a variação de coloração, através de diferentes experiências e melhorar a relação sinal-ruído. - Gire a placa no x g e 4 ° C, por 5 min.
- Agite a placa para descartar a solução.
- Neste ponto, o protocolo pode seguir o protocolo dependente da centrífuga convencional (prossiga para a etapa 3.3.5; Ver figura 1A, método 1) ou o protocolo independente de centrifugação alternativo (prossiga para a etapa 4.4.4; ver figura 1A, método 2).
- Ressuspender as células em 75 µ l da mistura de coloração anticorpo preparada na etapa 3.3.1.
- Misturar as amostras usando uma pipeta multicanal e incube-os em gelo por 30 min.
- Prepare uma mistura de coloração de anticorpo que contém anti-TCRβ, anti-CD4, anti-CD8 e anticorpos anti-CD69. Dilua a anticorpos em tampão de lavagem FACS (PBS suplementado com 0,5% albumina de soro bovino [BSA]) na proporção de 1: 200 (v/v).
-
Fixação de células
- Lavar os poços com 200 µ l de tampão de lavagem FACS e girar a placa em 300 x g e 4 ° C, por 5 min.
- Agite a placa para descartar a solução.
- Adicionar o buffer de fixação/permeabilização (vem com o kit de apoptose de caspase-3 ativo; mesmo com o tampão de lavagem/perm de 10x mencionado na etapa 3.5.1 e o anticorpo anti-caspase-3 etapa 3.5.2) em 200 µ l por bem.
- Incube no gelo por 30 min.
-
Intracelular de coloração para ativo caspase 3
- Prepare 1 tampão de perm/lavagem x diluindo-se 5 mL de tampão de perm/lavagem x 10 em 45 mL de água ultrapura.
- Prepare-se mancha de caspase activa intracelular adicionando 1,3 mL de anticorpo anti-caspase-3 a 6,5 mL de tampão de lavagem de perm/1x. A proporção de anticorpos de perm/tampão de lavagem é 1:5.
- Girar a placa no x g e 4 ° C, durante 5 min. Flick a placa para descartar a solução. Lave a placa com 200 µ l de tampão de perm/lavagem x 1.
- Repita a etapa 3.5.3.
- Girar a placa no x g e 4 ° C, durante 5 min. Flick a placa para descartar a solução. Adicione 75 µ l de caspase intracelular mancha preparada na etapa 3.5.2 para todos os poços.
- Misturar as amostras usando uma pipeta multicanal e incubar no gelo por 1h.
- Lavar as amostras com 200 µ l de tampão de perm/lavagem x 1 e girar a placa no x g e 4 ° C, por 5 min.
- Agite a placa para descartar a solução. Lave a placa com 200 µ l de tampão de perm/lavagem x 1. Gire a placa no x g e 4 ° C, por 5 min.
- Agite a placa para descartar a solução e Resuspenda as amostras em 200 µ l de tampão de lavagem FACS.
- Executar uma fluxo cytometric análise das amostras e analisar os resultados com um programa de análise de FACS.
- Usando um enredo de CD4 e CD8, portão na população de timócitos DP com uma expressão positiva de CD4 e CD8 (Figura 2, metade inferior). Dentro do portão de timócitos DP, determine a percentagem de células com registrados caspase-3, usando a amostra unstimulated como o controlo negativo e dexametasona como controlo positivo. Para a análise da expressão de CD69 no portão de timócitos DP, use a amostra unstimulated como controlo negativo e a amostra estimulada como controlo positivo.
Nota: Quando a retenção sobre os timócitos DP, verifique se que a população de timócitos DP é fechada corretamente para amostras individuais. Células estimuladas downregulate coreceptors superfície e uma exclusão involuntária de eventos podem ocorrer se um apertado portão DP é usado.
4. Biblioteca de inibidor de quinase rastreio (teste de centrífuga-independente)
-
Tratamento de timócitos com inibidores da quinase
- Prepare uma suspensão de timócitos conforme seção 1.
- Dilua os timócitos em RPMI completo para obter uma suspensão de timócitos de 25 x 106 células/mL.
- Adicione 40 µ l de timócitos em cada poço de uma placa de pequeno volume, usando uma pipeta multicanal. Coloque a placa no gelo.
- Dilua os inibidores da placa de estoque, DMSO e dexametasona em RPMI completa em uma proporção de quatro partes de RPMI completa para uma parte do inibidor/DMSO/dexametasona (factor de diluição de 5).
Nota: Como os volumes utilizados na placa deste pequeno volume 5 x menor do que no método convencional, os inibidores e os reagentes de controle são diluídos cinco vezes antes de adicioná-los para os timócitos na placa. - Adicione 0,5 µ l de inibidores para a placa de 96 poços dos poços da placa de inibidor, preparado na etapa 4.1.4 correspondentes.
- Prepare oito poços de controles não tratados. Prepare quatro poços de controles tratados com veículo, acrescentando 0,5 µ l do DMSO preparado na etapa 4.1.4. Prepare quatro poços de 5 µM tratados com dexametasona controles, usando a dexametasona diluída preparada na etapa 4.1.4 (Figura 2).
-
Estimulação de timócitos usando anti-CD3/CD28 grânulos
- Certifique-se que os grânulos são uniformemente resuspended. Tomar 1 mL de grânulos e lave-os com 2 mL de PBS. Separar as contas usando uma base magnética e aspirar a solução. Resuspenda as contas em 1 mL de RPMI completa.
Nota: A proporção de grânulos de células é de 1 a 2,5. Ajuste a quantidade de grânulos, dependendo do número de poços para estimular e o número de timócitos usado. - Adicione 10 µ l de suspensão de cordão para cada amostra tratada com inibidor, quatro amostras tratadas DMSO e quatro das oito amostras não tratadas. Adicione 10 µ l de RPMI completa nos restantes quatro poços não tratados. A Figura 2 mostra o layout geral da placa.
Nota: O volume final dos poços é de 50 µ l, que está dentro da capacidade máxima dos poços. É importante ter cautela e segurar as placas na posição vertical, para evitar derramamento de cruz-bem. - Para misturar, agite a placa usando um agitador orbital de microplacas. Alternativamente, misture o conteúdo dos poços com uma pipeta multicanal.
- Incubar os timócitos em um 37 ° C, 5% CO2 incubadora para 17-20 h (ou durante a noite) com uma tampa de antievaporação.
- Certifique-se que os grânulos são uniformemente resuspended. Tomar 1 mL de grânulos e lave-os com 2 mL de PBS. Separar as contas usando uma base magnética e aspirar a solução. Resuspenda as contas em 1 mL de RPMI completa.
-
Instalação da máquina de lavar prato
Nota: As instruções para configurar a máquina de lavar prato são fornecidas pelo fabricante. As etapas são mencionadas em breve. Aproximadamente 150 mL de solução é necessário para cada etapa de escorva.- Encha o sistema de lavagem com etanol a 70% contendo 1% Tween 20.
- Encha o sistema de lavagem com água desionizada, contendo 1% Tween 20.
- Encha o sistema de lavagem com tampão de lavagem FACS.
-
Coloração de antígenos de superfície
- Prepare uma mistura de coloração de anticorpo que contém anti-TCRβ, anti-CD4, anti-CD8 e anticorpos anti-CD69. Dilua os anticorpos em tampão de lavagem FACS na proporção de 1: 100 (v/v).
- Lave a placa 9 x, usando 55 µ l de tampão de lavagem FACS por lavagem, usando o fluxo laminar automatizado, sistema de lavagem.
Nota: No final das lavagens, haverá 25 µ l de volume residual em cada poço. - Ressuspender as células em 25 µ l da mistura de coloração anticorpo preparada na etapa 4.4.1.
- Se as amostras são transferidas de uma placa de 96 poços (da etapa 3.3.4), Ressuspender as células em 50 µ l de mistura de anticorpo preparada na etapa 3.3.1 e transferir as amostras para a placa de pequeno volume. Esta etapa corresponde ao número de método 2, conforme representado na figura 1A.
- Para misturar, agitar a placa com um agitador orbital de microplacas ou misturar as amostras usando uma pipeta multicanal e incubar no gelo por 30 min.
-
Fixação de células
- Lave a placa 9 x, usando 55 µ l de tampão de lavagem FACS por lavagem, usando o fluxo laminar automatizado, sistema de lavagem.
- Adicionar o buffer de fixação/permeabilização (vem com o kit de apoptose de caspase-3 ativo; mesmo com o tampão de lavagem/perm de 10x mencionado na etapa 4.6.1 e o anticorpo anti-caspase-3 etapa 4.6.2) em 50 µ l por bem.
- Incube no gelo por 30 min.
-
Intracelular de coloração para ativo caspase 3
- Prepare 1 tampão de perm/lavagem x diluindo-se 25 mL de tampão de perm/lavagem x 10 em 225 mL de água ultrapura.
- Prepare o caspase activa intracelular mancha, adicionando 1 mL de anticorpo anti-caspase-3 a 2 mL de tampão de lavagem de perm/1x. A proporção de anticorpos de perm/tampão de lavagem é 1:2.
- Encha o sistema de lavagem com 1x tampão de lavagem/perm.
- Lavar a placa 9 x com 1 x reserva permanente por lavagem, em 55 µ l para cada lavagem.
- Adicione 25 µ l da mancha caspase intracelular preparada na etapa 4.6.2 para todos os poços.
- Para misturar, agitar a placa com um agitador orbital de microplacas ou misturar as amostras usando uma pipeta multicanal e incubar no gelo por 1h.
- Lavar a placa 9 x com 1 x reserva permanente por lavagem, em 55 µ l para cada lavagem.
- Adicione 25 µ l de tampão de lavagem FACS a todos os poços.
- Transferi as amostras para tubos da microplaca após adequada mistura através de pipetagem.
- Adicionar outro 50 µ l de tampão de lavagem FACS nos poços vazios e repita o passo 4.6.9.
- Repita as etapas 4.6.9 e 4.6.10 2x até 200 µ l das amostras são coletadas em tubos de microtitulação.
Nota: O propósito dos procedimentos descritos nos passos 4.6.10 e 4.6.11 é garantir uma máxima recuperação das células da placa de pequeno volume. Se os números de celular não é uma preocupação, depois passo 4.6.10, simplesmente encher a microtiter tubos de 200 µ l de tampão de lavagem FACS. - Executar uma fluxo cytometric análise das amostras e analisar os resultados com um programa de análise de FACS, conforme passo 3.5.11. Ativação de caspase-3 e expressão CD69 são analisados no portão contendo CD4+CD8+DP timócitos.
Resultados
A abordagem para o ensaio de triagem está resumida na figura 1A. Os inibidores da quinase primeiro foram selecionados por seus efeitos latentes na viabilidade de timócitos. Como um controle positivo de apoptosis, dexametasona foi usada como um agente proapoptotic. O canal para a população de células vivas... foi determinado com base nos controlos negativos e os controles positivos tratados com dexametasona (figura 1B). Primeiro, os inibidores foram testados em 10 µM em timócitos, e a percentagem de células viáveis foi medida após incubação por 18 h. Uma janela de 20% para a morte celular foi escolhida tal que os compostos que induziu uma maior do que 20% perda de células no portão de células vivas, em comparação com as amostras de DMSO-tratados, foram testados em concentrações inferiores (figura 1B). Parcelas de FACS representativas das amostras selecionadas tratados com inibidor são mostradas para ilustrar o ensaio da viabilidade. LY294002 (2-(4-morpholinyl)-8-phenyl-4H-1-benzopyran-4-one; CAS 154447-36-6), um inibidor de PI3K22, não aumentar grandemente a morte celular em 10 µM e o inibidor foi usado em 10 µM para os ensaios subsequentes. CAY10626 (N-[2-(dimethylamino)ethyl]-N-methyl-4-[[[4-[4-(4-morpholinyl)-7-(2,2,2-trifluoroethyl)-7H-pyrrolo[2,3-d]pyrimidin-2-yl]phenyl]amino]carbonyl]amino]-benzamide; CAS 1202884-94-3), um duplo inibidor da PI3Kα/mTOR23, induzido por altos níveis de morte celular em 10 µM e 1 µM mas não com 0.1 µM e 0.1 µM foi determinada como sendo a concentração adequada para aplicação em ensaios a jusante. Staurosporine (2,3,10,11,12,13-hexahydro-10R-methoxy-9S-methyl-11R-methylamino-9S,13R-epoxy-1H,9H-diindolo[1,2,3-gh;3',2',1'-lm]pyrrolo[3,4-j][1,7]benzodiazonin-1-one; CAS 62996-74-1), um inibidor de quinase C panproteína com uma capacidade estabelecida de induzir apoptose24, induzido por morte celular significativa em todas as concentrações testadas, mesmo em 0.1 µM. Foi usado como um controle positivo adicional em ensaios subsequentes em 0.1 µM.
As concentrações finais dos inibidores foram selecionadas com base em maiores concentrações em que não amplificam morte celular por mais de 20% das amostras tratadas DMSO. Com as concentrações finais dos inibidores determinados, uma placa de estoque dos inibidores foi preparada tal que todos os inibidores foram 500 vezes a concentração, quando aplicado às células. A Figura 1 ilustra o layout da placa da placa de estoque, com as concentrações finais dos inibidores. No protocolo alternativo de incubação das células diretamente nas placas de pequeno volume para o fluxo laminar do ensaio de lavagem, o uso de pequenos volumes necessitou uma diluição adicional dos inibidores. Para garantir que o conteúdo de DMSO das culturas após adição do inibidor não seria demasiado elevada para as células, os inibidores foram ainda mais diluídos em RPMI completa, por um fator de diluição de 5, tais que eram 100 vezes a concentração pretendido quando aplicado para a células.
Os inibidores, diluídos-se a concentrações não tóxica, foram utilizados no ensaio de TCR estimulação-induzida apoptose em timócitos5,17. A estimulação foi efectuada com anti-CD3/CD28 grânulos para 18 h, e as células foram posteriormente coradas para ativação de caspase-3 no CD4+ e CD8+ DP população timócitos (Figura 2). Aumento da ativação de caspase-3 e expressão CD69 e também uma downregulation TCR, observaram-se na anti-CD3/CD28-estimulada e as amostras anti-CD3/28-estimulada DMSO-simulação-tratada, em comparação com as amostras nonstimulated. As amostras tratadas dexametasona mostraram um aumento na ativação de caspase-3 independente de CD69 upregulation, que se espera do efeito de indução de apoptose sendo independente da estimulação de TCR.
Figura 3A resume os resultados da biblioteca de ensaio para selecionado inibidores de triagem. Tanto a ativação de caspase-3 e CD69 podem ser usado para identificar potenciais inibidores de interesse devido a supressão da expressão. Como esperado, inibidores de mediadores canônicos de TCR sinalização apareceram como positivo bate nas telas. Tais inibidores, que exibiram diversos graus de potência inibitória, incluíam inibidores de largo espectro que se destinam a quinases múltiplas e, também, mais inibidores específicos. Alguns inibidores foram capazes de suprimir tanto a ativação de caspase-3 e upregulation de CD69 (Figura 3B, linha superior, painéis à esquerda). Um tal inibidor é bisindolylmaleimide II (3-(1H-Indol-3-yl)-4-[1-[2-(1-methyl-2-pyrrolidinyl)ethyl]-1H-indol-3-yl]-1H-pyrrole-2,5-dione; CAS 137592-45-1), que inibe a todos de proteína quinase C isoformas, além de proteína da e PDK125,26,27. Outro inibidor nesta categoria é CAY10657 (3-[(aminocarbonyl)amino]-5-[4-(4-morpholinylmethyl)phenyl]-2-thiophenecarboxamide; CAS 494772-86-0), um inibidor da proposta de IKK228.
Foram compostos que inibiu upregulation de CD69 mas não prejudicar a ativação de caspase-3 (Figura 3B, linha superior, painéis certo). CAY10626, um inibidor da PI3Kα e mTOR23e U-0126 (2,3-bis [amino [(2-aminophenyl) thio] metileno]-butanedinitrile; CAS 109511-58-2), um inibidor MEK29, foram alguns dos inibidores identificados. Os resultados mostram que diferentes inibidores como alvo diferentes quinases de ramos específicos da via de sinalização do TCR, especialmente os alvos quinases do tarde-estágio, podem ocasionar a deficiência seletiva de fenômenos de ativação de células T.
Havia também inibidores que não suprimir tanto upregulation de CD69 e ativação de caspase-3 (Figura 3B, linha inferior, painéis à esquerda). Paclitaxel (βS-(benzoylamino) - particular - hidroxi-ácido benzenepropanoic, (2aR,4S,4aS,6R,9S,11S,12S,12aR,12bS)-6,12b-bis(acetyloxy)-12-(benzoyloxy)-2a,3,4,4a,5,6,9,10,11,12,12a,12b-dodecahydro-4,11-dihydroxy-4a,8,13,13-tetramethyl-5-oxo-7 , 11-methano-1H-cyclodeca[3,4]benz[1,2-b]oxet-9-yl éster; CAS 33069-62-4), um disruptor do microtubule dinâmica30e necrostatin-5 (2-[[3,4,5,6,7,8-hexahydro-3-(4-methoxyphenyl)-4-oxo[1]benzothieno[2,3-d]pyrimidin-2-yl]thio]-acetonitrile; CAS 337349-54-9), um inibidor de quinase de RIP131, são dois inibidores identificados para estar nesta categoria. Nesses casos onde a upregulation de CD69 e ativação de caspase-3 não eram deficientes, isto pode ser devido os inibidores não visando a uma quinase relevante da TCR sinalização via.
Como mencionado anteriormente, o staurosporine foi usado nas telas, numa concentração que ainda induziu apoptose em timócitos os. Como esperado, a amostra tratada com staurosporine mostrou altos níveis de ativação de caspase-3 (Figura 3B, linha inferior, coluna da direita). Os baixos níveis de expressão CD69 podem ser atribuídos para a inibição mediada por staurosporine da PKC, como bisindolylmaleimide II, outro inibidor de pan-PKC, também suprimida a expressão de CD69. Alternativamente, staurosporine induzida apoptose nas células antes que eles foram capazes de upregulate a expressão CD69.
Para aumentar a produtividade e automação do protocolo, foram elaborados protocolos paralelos que envolveram o uso de uma placa automatizado sistema através do fluxo laminar de lavagem. Dois protocolos separados usando este dispositivo de lavagem de placa automatizado foram julgado e em comparação com o método convencional de cultivo de células em placas de 96 poços e manchando as células em um protocolo de centrifugação-dependente. Um método envolveu o cultivo das células em placas de 96 poços, conforme procedimento padrão, e em seguida, transferir as células para placas compatíveis com a máquina de lavar prato automatizado para a coloração passos (Figura 4, as amostras DA lavagem). O outro método envolveu cultivo as células diretamente nas placas de chapa-máquina de lavar-compatível e continuando com o protocolo de coloração no mesmo prato (Figura 4, as amostras DA cultura). Os protocolos de centrifugação independente não dão muitas diferenças perceptíveis em ativo caspase-3, CD69, ou TCRβ coloração através de diferentes amostras testadas, em comparação com o protocolo dependente de centrifugação convencional (Figura 4). Diferenças na intensidade da coloração podem ser atribuídas ao uso de anticorpos em concentrações ligeiramente diferentes durante as etapas de coloração.
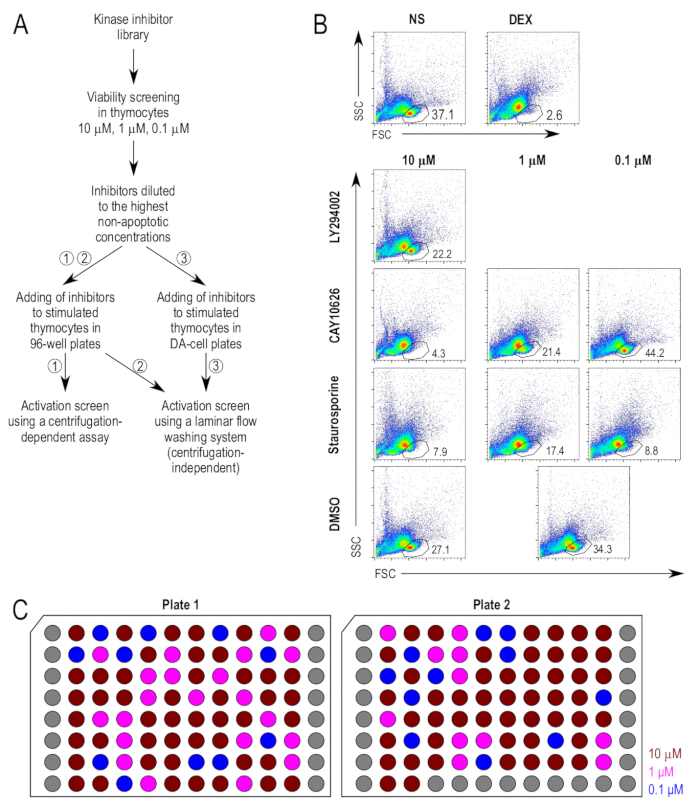
Figura 1 : Viabilidade de timócitos após o tratamento com inibidores da. (A) Experimental contorno das etapas principais no ensaio de triagem. Existem três métodos propostos para a estimulação e a coloração dos timócitos utilizados no ensaio de ativação, ou seja, (1) o cultivo de timócitos em padrão placas de 96 poços, seguidos de coloração usando um protocolo de baseado em centrifugação convencional, (2) a cultivo de timócitos em padrão placas de 96 poços, seguidos de coloração usando um protocolo independente de centrifugação lavagem e (3) o cultivo de timócitos em placas de pequeno volume, seguido de coloração nas placas de mesmo usando um Protocolo de lavagem independente de centrifugação. (B) Gating estratégias utilizadas em ensaios de viabilidade. O portão de célula viva foi derivado da dispersão para a frente (FSC) e dispersão lateral (SSC) plota, como descrito anteriormente,17. Inibidores que foram considerados muito tóxico para as concentrações testadas foram sujeitos a ensaios de viabilidade mais 10 vezes concentrações inferiores. Amostras representativas de inibidor-tratados são mostradas. Note o controle comum (DMSO-tratados [DMSO]) usado para a 1 µM e 0,1 µM de amostras. (C) layout de placa de inibidores diluídos. Uma representação esquemática das placas de inibidores diluídos em DMSO a uma concentração de 500 x concentração final pretendida. Cada um representa bem um único inibidor; os cinzas poços estão vazios. As concentrações mostradas são a concentração final quando adicionado para as culturas de células, ou seja 10 µM (vermelho escuro), 1 µM (fúcsia) e 0.1 µM (azul). Clique aqui para ver uma versão maior desta figura.
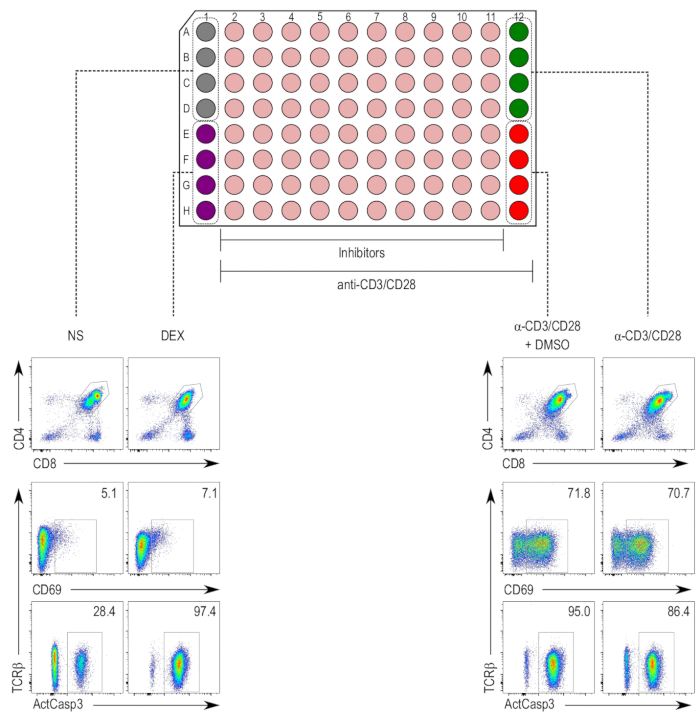
Figura 2 : Layout de placa de ensaio de ativação de timócitos. (Topo) Colunas 1 e 12 são reservadas para controles, enquanto as colunas 2 a 11 são tratados com inibidor de amostras (bege). O controlo negativo (nonstimulated [NS]; cinzento) ocupa poços de A1 a D1, e o controle positivo para morte celular (tratada com dexametasona [DEX]; roxo) ocupa poços E1 para H1. Colunas de 2 a 12 contêm timócitos estimulada com anti-CD3/CD28 grânulos. O controle positivo para ativação de timócitos (amostras estimuladas [α-CD3/CD28]; verde) ocupa poços A12 D12, e o controle do veículo (estimulada e tratada com DMSO [α-CD3/CD28 + DMSO]; vermelho) ocupa poços E12 para H12. (Parte inferior) Fluxo cytometry parcelas do ativo caspase-3 (ActCasp3), CD69 e TCRβ coloração de timócitos condomínio fechados dentro do portão (DP) de duplo-positivo. Parcelas representativas dos diferentes controles são mostradas. NS = nonstimulated; DEX = tratados com dexametasona amostras; Α-CD3/CD28 + DMSO = amostras estimuladas com grânulos CD3/CD28-revestido e tratado com DMSO; Α-CD3/CD28 = amostras estimuladas com grânulos CD3/CD28-revestido. Clique aqui para ver uma versão maior desta figura.
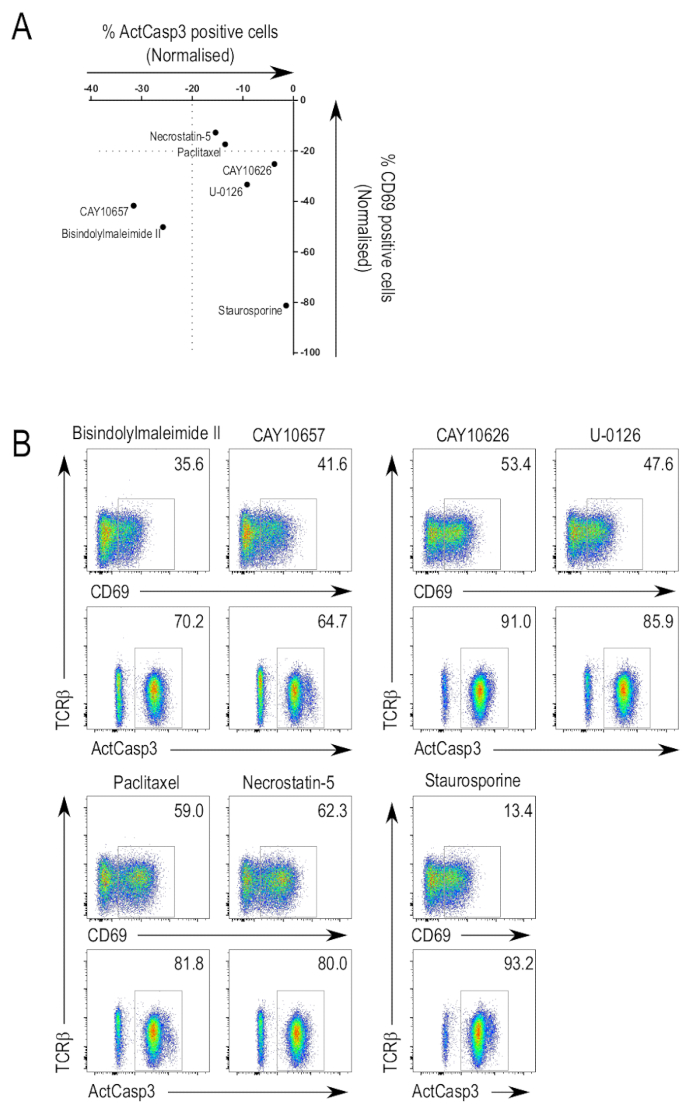
Figura 3 : Seleção da biblioteca inibidor na ativação de timócitos. (A) resumiu os dados do ensaio da ativação. Estes são os resultados de um experimento representativo, mostrando os valores normalizados de células com registrados caspase-3 e expressão CD69 para inibidores selecionados. Normalização foi feita comparando a percentagem de células no ativo-caspase-3-positivo ou portão de CD69-positivo para o valor do Controlarar de DMSO-tratados, que é definido como um valor relativo de 0 no gráfico. (B) selecionado FACS parcelas. Parcelas de citometria de fluxo de inibidores que oprimiu tanto a ativação de caspase-3 e upregulation de CD69 (canto superior esquerdo), suprimida apenas upregulation de CD69 (superior direito), ou não teve efeito sobre a ativação de caspase-3 e upregulation de CD69 (inferior esquerdo). Parcelas da amostra tratada com staurosporine são mostradas para ilustrar os efeitos do uso de um inibidor em concentrações tóxicas (canto inferior direito). Clique aqui para ver uma versão maior desta figura.
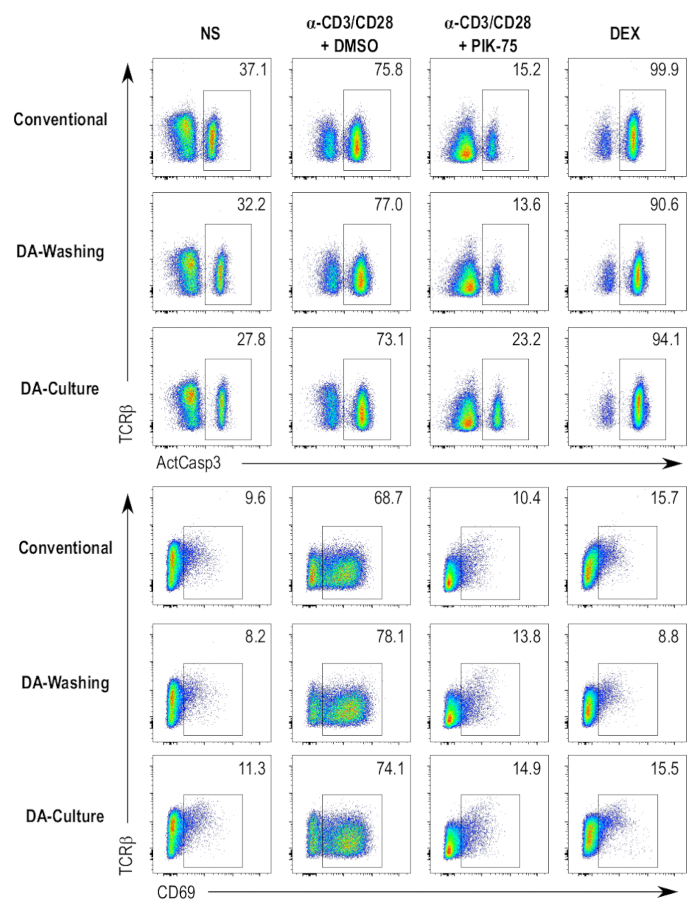
Figura 4 : Comparação entre os protocolos de ensaio diferente. Flow cytometry parcelas do ativo caspase-3 (ActCasp3), CD69, e TCRβ coloração de timócitos DP seguindo os três diferentes protocolos de ensaios. Quatro condições diferentes são testadas, ou seja, o controle negativo (nonstimulated [NS]), o controle positivo para morte celular (tratada com dexametasona [DEX]), o controle do veículo (estimulada e tratada com DMSO [α-CD3/CD28 + DMSO]) e tratados com um inibidor da amostra (estimulada e PIK-75-tratados [α-CD3/CD28 + PIK-75]). Convencional = o cultivo de timócitos em placas de 96 poços padrão e coloração com um protocolo de baseado em centrifugação convencional; DA lavagem = o cultivo de timócitos em placas de 96 poços padrão e coloração usando um fluxo laminar lavagem protocolo; DA cultura = o cultivo de timócitos em placas de pequeno volume e coloração nas placas de mesmo usando um fluxo laminar protocolo de lavagem. Clique aqui para ver uma versão maior desta figura.
Discussão
A estratégia de rastreio aqui proposta avalia a capacidade dos inibidores da pequeno-molécula para suprimir os efeitos de apoptose em timócitos após estimulação, além de marcadores mais convencionais do upregulation de CD69-ativação de células T e TCR downregulation . Marcadores adicionais também podem ser incluídos para permitir a análise de timócitos diferentes subconjuntos32. Um aspecto interessante do ensaio atual reside no fato de que inibidores que impedem a TCR sinalização também poderia umedecer a indução da apoptose, destacando ainda mais a distinção de efeitos de TCR-independente, que os inibidores podem ter na indução de morte celular. Além disso, um ensaio de citometria de fluxo-base permite a utilização de múltiplas leituras como marcadores de ativação distintas, o que poderiam relatar os efeitos dos inibidores em ramos individuais separados de TCR sinalização. No caso apresentado aqui, havia inibidores que mostraram uma diferencial inibição da ativação de caspase-3 e upregulation de CD69. Porque alguns compostos podem afetar funções de limpeza tais como a síntese de proteínas ou tráfico vesicular, não é surpresa observar efeitos sobre o upregulation de marcadores de novo sintetizado (por exemplo, CD69) mas não no posttranslational modificações (por exemplo, a ativação proteolítica de caspase-3).
Como o ensaio apresentado aqui mede apoptose como uma leitura, é imperativo que os efeitos tóxicos latentes dos inibidores não obscurecem os resultados. Por exemplo, na tela, não diluir staurosporine além de 1 nM, apesar de ainda ser tóxico para as células àquela concentração. Os resultados representativos estão de acordo com staurosporine, sendo um inibidor de quinase promíscuo e um indutor de apoptose33. Sem uma suficiente diluição dos compostos testados para concentrações não tóxica, é possível ignorar potenciais hits.
A estratégia de triagem detalhada aqui seria difícil aplicar aos seres humanos devido as complicações associadas à obtenção de um número suficiente de timócitos para seleção da elevado-produção. No entanto, é possível obter amostras do Timo humano de biópsias cardíacas pediátricas34,35 ou de fetos36,37. Apesar de tudo, como TCR sinalização de caminhos e as sequências de aminoácidos de proteínas de sinalização em grande parte são conservadas entre os ratos e os seres humanos, o ensaio de timócitos fornece uma estratégia útil triagem preliminar, e quaisquer resultados obtidos com este ensaio usando o mouse timócitos podem, então, ser verificados em linfócitos humanos primários.
Uma limitação do protocolo dependente de centrifugação convencional refere-se à perspectiva de perda de célula, que pode ser atribuída à natureza várias etapas do processo, que envolve etapas como a permeabilização de célula e centrifugação. Cada centrifugação e ressuspensão passo inevitavelmente resultam na perda de células. Enquanto tais perdas não podem ser críticas para estudos que envolvem um número limitado de amostras, isso poderia causar problemas quando aplicado no rastreio de uma taxa de transferência, em particular no decorrer do ensaio formato de 96 - a 384 - de 1536-bem. Uma maneira de contornar este problema é através do uso de célula-permeável caspase fluorescente sensores38 que permitem a detecção de ativação de caspase, evitando as complicações de permeabilização celular e múltiplas lavagens5. Alternativamente, empregar um método independente de centrifugação de células de lavagem por fluxo laminar também é possível para minimizar a perda de células. Com uma placa automatizada estação em conjunto com uma placa de parede-menos de lavagem, as células são lavadas pelo fluxo laminar sem o uso de uma centrífuga. A exponencial diluição dos reagentes permite a lavagem completa e eficiente de células em menos de 3 min, o que representa uma diluição equivalente a duas rodadas de lavagem centrífuga. Sem agressões externas devido a centrifugação, as células são mais viáveis e perdas de célula são minimizadas.
Exploramos também a possibilidade de utilizar a placa automatizada estação de lavagem após cultivo os timócitos em 96 poços de fundo U placas e, também, o cultivo de células diretamente na parede sem placas compatíveis com a placa automatizada estação de lavagem. O cultivo de células nas placas de parede-menos habilitado a eliminação de todas as etapas de centrifugação e minimizou a perda de células, eliminando a necessidade de uma transferência de amostra através de placas. Geralmente, os três diferentes protocolos são comparáveis em eficiência de estimulação e coloração. A estação de lavagem automatizada oferece o benefício de automação, velocidade e eficiência, o que torna mais fácil para a análise de uma taxa de transferência. Além disso, com a crescente automação, as etapas de lavagem podem ser realizadas mais rapidamente, e há uma maior coerência entre as experiências ou experimentadores. No entanto, a estação de lavagem tem certas desvantagens: grandes volumes de lavagem buffers são necessários para arruela escorva (150 mL por alteração de reserva, dos quais 50 mL é usado para lavar); cuidado extra é necessária ao manusear a placa para evitar qualquer contaminação dos poços devido ao particionamento limitada entre os poços da placa de pequeno volume; buffer de residual de 25 µ l dos poços após lavagem exige a utilização de reagentes preparados no maior do que a concentração de 1 x. Para resolver os problemas de volume residual e capacidade de volume limitado da placa, um acessório para expandir o volume de incubação de 70 µ l a 150 µ l pode ser adicionado, facilitando a adopção de protocolos convencionais. Enquanto placa automatizado de sistemas de tratamento estão disponíveis atualmente, eles têm uma pegada significativa em comparação com o sistema de lavagem laminar, que é uma pequena unidade de ~ 1 pé cúbico (~0.028 m3). Além disso, a integração de centrifugação em placa automatizada, sistemas de tratamento é um desafio, limitando a sua utilização na lavagem da célula. Não há atualmente nenhuma outra célula independente de centrífuga lavar instrumentos disponíveis, tanto quanto sabemos.
A estratégia de seleção apresentada aqui é capaz de identificar moléculas pequenas e suas quinases suposta alvo, que afetam a TCR sinalização e ativação de células T. A biblioteca usada aqui é composto principalmente de pequenas-molécula inibidores de quinases e foi capaz de gerar um número de potencialmente interessantes sucessos. O protocolo também pode ser aplicado facilmente bibliotecas de inibidor de outras classes de enzimas ou de outros tipos de pequenas moléculas, bem como bibliotecas de outros compostos (por exemplo, várias macromoléculas). O protocolo também pode ser usado para outros tipos de células, tais como periféricos linfócitos T ou células imortalizadas, incluindo aqueles expressando TCRs transgénicos ou carregando sistemas repórter da tela. Identificar e caracterizar novos mediadores de células T sinalização pode melhorar nosso conhecimento sobre a via de sinalização e também ajuda no desenvolvimento da terapia-alvo em doenças imunes13,14,15, 16. ao todo, este estudo acrescenta à gama de opções disponíveis para a detecção de mediadores de células T sinalização através de rastreio com throughput alto ensaios.
Divulgações
O autor Chyan Ying Ke é um empregado de Curiox Biosystems, que produz a célula DA lavadora e placas DA célula usadas neste artigo.
Agradecimentos
Este trabalho foi apoiado por concessões do Conselho Nacional de pesquisa médica do Ministério da saúde a Singapura, NMRC CBRG15may017 e o Ministério da educação, 2014 de Singapura-T2-1-136 (para N.R.J.G.).
Materiais
| Name | Company | Catalog Number | Comments |
| RPMI | HyClone | SH30027FS | |
| FBS | HyClone | SH3007103 | |
| L-Glutamine | HyClone | SH3003401 | |
| Sodium pyruvate | HyClone | SH3023901 | |
| Penicillin/Streptomycin | HyClone | SV30010 | |
| b-mercaptoethanol | Sigma Aldrich | 516732 | |
| 10X PBS | Vivantis | PB0344 – 1L | |
| Kinase Screening Library (96-Well) | Cayman Chemical | 10505 | Exact contents of the library may vary |
| DMSO | Sigma Aldrich | D2650 | |
| Dexamethasone | Sigma Aldrich | D4902 | |
| anti-CD3/CD28 beads | Thermo Fisher Scientific | 11452D | |
| FITC Active Caspase-3 Apoptosis Kit | BD Pharmingen | 550480 | Contains Fixation/Permeabilisation buffer, 10X Perm/Wash buffer and anti-caspase 3 antibody |
| DA-Cell Washer | CURIOX | HT1000 | |
| 96-well DA-Cell Plate | CURIOX | 96-DC-CL-05 | |
| Antibodies | |||
| CD3e | BioLegend | 100236 | |
| TCRb | BD Biosciences | 553174 | |
| CD4 | BD Biosciences | 740007 | |
| CD8 | BD Biosciences | 563786 | |
| CD69 | eBioscience | 25-0699-42 | |
| Inhibitors | |||
| TG003 | Cayman Chemical | - | From the Kinase Screening Library |
| PKC 412 | Cayman Chemical | - | From the Kinase Screening Library |
| Doramapimod | Cayman Chemical | - | From the Kinase Screening Library |
| Paclitaxel | Cayman Chemical | - | From the Kinase Screening Library |
| Erlotinib | Cayman Chemical | - | From the Kinase Screening Library |
| Necrostatin-5 | Cayman Chemical | - | From the Kinase Screening Library |
| NVP-BEZ235 | Cayman Chemical | - | From the Kinase Screening Library |
| Phthalazinone pyrazole | Cayman Chemical | - | From the Kinase Screening Library |
| AG-879 | Cayman Chemical | - | From the Kinase Screening Library |
| 1-NA-PP1 | Cayman Chemical | - | From the Kinase Screening Library |
| Torin 1 | Cayman Chemical | - | From the Kinase Screening Library |
| Bisindolylmaleimide II | Cayman Chemical | - | From the Kinase Screening Library |
| BIBF 1120 | Cayman Chemical | - | From the Kinase Screening Library |
| SMI-4a | Cayman Chemical | - | From the Kinase Screening Library |
| Bisindolylmaleimide XI (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10657 | Cayman Chemical | - | From the Kinase Screening Library |
| AS-703026 | Cayman Chemical | - | From the Kinase Screening Library |
| Chelerythrine chloride | Cayman Chemical | - | From the Kinase Screening Library |
| Tunicamycin | Cayman Chemical | - | From the Kinase Screening Library |
| GSK 1059615 | Cayman Chemical | - | From the Kinase Screening Library |
| Ruxolitinib | Cayman Chemical | - | From the Kinase Screening Library |
| Necrostatin-1 | Cayman Chemical | - | From the Kinase Screening Library |
| SB 505124 | Cayman Chemical | - | From the Kinase Screening Library |
| INK128 | Cayman Chemical | - | From the Kinase Screening Library |
| Canertinib (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| SB 431542 | Cayman Chemical | - | From the Kinase Screening Library |
| PD 173074 | Cayman Chemical | - | From the Kinase Screening Library |
| Valproic Acid (sodium salt) | Cayman Chemical | - | From the Kinase Screening Library |
| PD 0325901 | Cayman Chemical | - | From the Kinase Screening Library |
| SB 203580 | Cayman Chemical | - | From the Kinase Screening Library |
| VX-702 | Cayman Chemical | - | From the Kinase Screening Library |
| Emodin | Cayman Chemical | - | From the Kinase Screening Library |
| CHIR99021 | Cayman Chemical | - | From the Kinase Screening Library |
| BIO | Cayman Chemical | - | From the Kinase Screening Library |
| Imatinib (mesylate) | Cayman Chemical | - | From the Kinase Screening Library |
| Sunitinib Malate | Cayman Chemical | - | From the Kinase Screening Library |
| Gefitinib | Cayman Chemical | - | From the Kinase Screening Library |
| PP2 | Cayman Chemical | - | From the Kinase Screening Library |
| 3-Methyladenine | Cayman Chemical | - | From the Kinase Screening Library |
| Bisindolylmaleimide I | Cayman Chemical | - | From the Kinase Screening Library |
| Bisindolylmaleimide IV | Cayman Chemical | - | From the Kinase Screening Library |
| Bisindolylmaleimide V | Cayman Chemical | - | From the Kinase Screening Library |
| NSC 663284 | Cayman Chemical | - | From the Kinase Screening Library |
| D 4476 | Cayman Chemical | - | From the Kinase Screening Library |
| NU 7026 | Cayman Chemical | - | From the Kinase Screening Library |
| H-9 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| Indirubin-3'-monoxime | Cayman Chemical | - | From the Kinase Screening Library |
| KN-62 | Cayman Chemical | - | From the Kinase Screening Library |
| KN-93 | Cayman Chemical | - | From the Kinase Screening Library |
| CGP 57380 | Cayman Chemical | - | From the Kinase Screening Library |
| Iso-Olomoucine | Cayman Chemical | - | From the Kinase Screening Library |
| (S)-Glycyl-H-1152 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| Bisindolylmaleimide VIII (acetate) | Cayman Chemical | - | From the Kinase Screening Library |
| ST638 | Cayman Chemical | - | From the Kinase Screening Library |
| SU 6656 | Cayman Chemical | - | From the Kinase Screening Library |
| LY364947 | Cayman Chemical | - | From the Kinase Screening Library |
| SB 203580 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10621 | Cayman Chemical | - | From the Kinase Screening Library |
| YM-201636 | Cayman Chemical | - | From the Kinase Screening Library |
| ZM 447439 | Cayman Chemical | - | From the Kinase Screening Library |
| AS-041164 | Cayman Chemical | - | From the Kinase Screening Library |
| NVP-AEW541 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| PP242 | Cayman Chemical | - | From the Kinase Screening Library |
| ABT-869 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10622 | Cayman Chemical | - | From the Kinase Screening Library |
| 17β-hydroxy Wortmannin | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10626 | Cayman Chemical | - | From the Kinase Screening Library |
| SU 6668 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10572 | Cayman Chemical | - | From the Kinase Screening Library |
| N,N-Dimethylsphingosine | Cayman Chemical | - | From the Kinase Screening Library |
| LY294002 | Cayman Chemical | - | From the Kinase Screening Library |
| U-0126 | Cayman Chemical | - | From the Kinase Screening Library |
| Staurosporine | Cayman Chemical | - | From the Kinase Screening Library |
| KN-92 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| AS-605240 (potassium salt) | Cayman Chemical | - | From the Kinase Screening Library |
| O-1918 | Cayman Chemical | - | From the Kinase Screening Library |
| Y-27632 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| Leelamine | Cayman Chemical | - | From the Kinase Screening Library |
| PD 98059 | Cayman Chemical | - | From the Kinase Screening Library |
| PD 169316 | Cayman Chemical | - | From the Kinase Screening Library |
| TGX-221 | Cayman Chemical | - | From the Kinase Screening Library |
| (S)-H-1152 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| AS-605240 | Cayman Chemical | - | From the Kinase Screening Library |
| D-erythro-Sphingosine C-18 | Cayman Chemical | - | From the Kinase Screening Library |
| OSU03012 | Cayman Chemical | - | From the Kinase Screening Library |
| JNJ-10198409 | Cayman Chemical | - | From the Kinase Screening Library |
| Leelamine (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| Arachidonic Acid Leelamide | Cayman Chemical | - | From the Kinase Screening Library |
| Lauric Acid Leelamide | Cayman Chemical | - | From the Kinase Screening Library |
| AS-252424 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10505 | Cayman Chemical | - | From the Kinase Screening Library |
| PI-103 | Cayman Chemical | - | From the Kinase Screening Library |
| PIK-75 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| Sphingosine Kinase Inhibitor 2 | Cayman Chemical | - | From the Kinase Screening Library |
| Piceatannol | Cayman Chemical | - | From the Kinase Screening Library |
| SC-1 | Cayman Chemical | - | From the Kinase Screening Library |
| (R)-Roscovitine | Cayman Chemical | - | From the Kinase Screening Library |
| BAY-43-9006 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10561 | Cayman Chemical | - | From the Kinase Screening Library |
| AS-604850 | Cayman Chemical | - | From the Kinase Screening Library |
| PI3-Kinase α Inhibitor 2 | Cayman Chemical | - | From the Kinase Screening Library |
| ML-9 | Cayman Chemical | - | From the Kinase Screening Library |
| Triciribine | Cayman Chemical | - | From the Kinase Screening Library |
| Erbstatin Analog | Cayman Chemical | - | From the Kinase Screening Library |
| Kenpaullone | Cayman Chemical | - | From the Kinase Screening Library |
| Olomoucine | Cayman Chemical | - | From the Kinase Screening Library |
| AG-494 | Cayman Chemical | - | From the Kinase Screening Library |
| AG-825 | Cayman Chemical | - | From the Kinase Screening Library |
| AG-1478 | Cayman Chemical | - | From the Kinase Screening Library |
| SB 216763 | Cayman Chemical | - | From the Kinase Screening Library |
| SB 415286 | Cayman Chemical | - | From the Kinase Screening Library |
| AG-17 | Cayman Chemical | - | From the Kinase Screening Library |
| H-8 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| LFM-A13 | Cayman Chemical | - | From the Kinase Screening Library |
| SC-514 | Cayman Chemical | - | From the Kinase Screening Library |
| Apigenin | Cayman Chemical | - | From the Kinase Screening Library |
| AG-18 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10554 | Cayman Chemical | - | From the Kinase Screening Library |
| DRB | Cayman Chemical | - | From the Kinase Screening Library |
| RG-13022 | Cayman Chemical | - | From the Kinase Screening Library |
| RG-14620 | Cayman Chemical | - | From the Kinase Screening Library |
| AG-490 | Cayman Chemical | - | From the Kinase Screening Library |
| AG-82 | Cayman Chemical | - | From the Kinase Screening Library |
| AG-99 | Cayman Chemical | - | From the Kinase Screening Library |
| AG-213 | Cayman Chemical | - | From the Kinase Screening Library |
| AG-183 | Cayman Chemical | - | From the Kinase Screening Library |
| Lavendustin C | Cayman Chemical | - | From the Kinase Screening Library |
| ZM 336372 | Cayman Chemical | - | From the Kinase Screening Library |
| 5-Iodotubercidin | Cayman Chemical | - | From the Kinase Screening Library |
| SB 202190 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10571 | Cayman Chemical | - | From the Kinase Screening Library |
| Nilotinib | Cayman Chemical | - | From the Kinase Screening Library |
| SP 600125 | Cayman Chemical | - | From the Kinase Screening Library |
| L-threo-Sphingosine C-18 | Cayman Chemical | - | From the Kinase Screening Library |
| H-89 | Cayman Chemical | - | From the Kinase Screening Library |
| HA-1077 (hydrochloride) | Cayman Chemical | - | From the Kinase Screening Library |
| AG-370 | Cayman Chemical | - | From the Kinase Screening Library |
| Wortmannin | Cayman Chemical | - | From the Kinase Screening Library |
| AG-1296 | Cayman Chemical | - | From the Kinase Screening Library |
| KT 5823 | Cayman Chemical | - | From the Kinase Screening Library |
| Janex 1 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10574 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10575 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10576 | Cayman Chemical | - | From the Kinase Screening Library |
| NH125 | Cayman Chemical | - | From the Kinase Screening Library |
| TWS119 | Cayman Chemical | - | From the Kinase Screening Library |
| NSC 210902 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10577 | Cayman Chemical | - | From the Kinase Screening Library |
| CAY10578 | Cayman Chemical | - | From the Kinase Screening Library |
| PD 184161 | Cayman Chemical | - | From the Kinase Screening Library |
| CCT018159 | Cayman Chemical | - | From the Kinase Screening Library |
| Myricetin | Cayman Chemical | - | From the Kinase Screening Library |
Referências
- Gascoigne, N. R., Rybakin, V., Acuto, O., Brzostek, J. TCR Signal Strength and T Cell Development. Annual Review of Cell and Developmental Biology. 32, 327-348 (2016).
- Rothenberg, E. V., Moore, J. E., Yui, M. A. Launching the T-cell-lineage developmental programme. Nature Reviews Immunology. 8 (1), 9-21 (2008).
- Klein, L., Hinterberger, M., Wirnsberger, G., Kyewski, B. Antigen presentation in the thymus for positive selection and central tolerance induction. Nature Reviews Immunology. 9 (12), 833-844 (2009).
- Starr, T. K., Jameson, S. C., Hogquist, K. A. Positive and negative selection of T cells. Annual Review of Immunology. 21, 139-176 (2003).
- Rybakin, V., Gascoigne, N. R. Negative selection assay based on stimulation of T cell receptor transgenic thymocytes with peptide-MHC tetramers. PLoS One. 7 (8), e43191 (2012).
- Krogsgaard, M., Juang, J., Davis, M. M. A role for "self" in T-cell activation. Seminars in Immunology. 19 (4), 236-244 (2007).
- Nakayama, T., Yamashita, M. The TCR-mediated signaling pathways that control the direction of helper T cell differentiation. Seminars in Immunology. 22 (5), 303-309 (2010).
- Hoerter, J. A., et al. Coreceptor affinity for MHC defines peptide specificity requirements for TCR interaction with coagonist peptide-MHC. The Journal of Experimental Medicine. 210 (9), 1807-1821 (2013).
- Zhao, X., et al. Nonstimulatory peptide-MHC enhances human T-cell antigen-specific responses by amplifying proximal TCR signaling. Nature Communications. 9 (1), 2716 (2018).
- Fu, G., et al. Fine-tuning T cell receptor signaling to control T cell development. Trends in Immunology. 35 (7), 311-318 (2014).
- Wang, D., et al. Tespa1 is involved in late thymocyte development through the regulation of TCR-mediated signaling. Nature Immunology. 13 (6), 560-568 (2012).
- Fu, G., et al. Themis sets the signal threshold for positive and negative selection in T-cell development. Nature. 504 (7480), 441-445 (2013).
- Rosenblum, M. D., Gratz, I. K., Paw, J. S., Abbas, A. K. Treating human autoimmunity: current practice and future prospects. Science Translational Medicine. 4 (125), (2012).
- Hebeisen, M., et al. SHP-1 phosphatase activity counteracts increased T cell receptor affinity. The Journal of Clinical Investigation. 123 (3), 1044-1056 (2013).
- Wang, R. E., et al. An immunosuppressive antibody-drug conjugate. Journal of the American Chemical Society. 137 (9), 3229-3232 (2015).
- Borroto, A., et al. First-in-class inhibitor of the T cell receptor for the treatment of autoimmune diseases. Science Translational Medicine. 8 (370), (2016).
- Chen, E. W., Brzostek, J., Gascoigne, N. R. J., Rybakin, V. Development of a screening strategy for new modulators of T cell receptor signaling and T cell activation. Scientific Reports. 8 (1), 10046 (2018).
- Fouda, A., Tahsini, M., Khodayarian, F., Al-Nafisah, F., Rafei, M. A Fluorescence-based Lymphocyte Assay Suitable for High-throughput Screening of Small Molecules. Journal of Visualized Experiments. (121), e55199 (2017).
- Zhao, Z., et al. A high-throughput phenotypic screen of cytotoxic T lymphocyte lytic granule exocytosis reveals candidate immunosuppressants. Journal of Biomolecular Screening. 20 (3), 359-371 (2015).
- Florian, A. E., et al. Flow cytometry enables a high-throughput homogeneous fluorescent antibody-binding assay for cytotoxic T cell lytic granule exocytosis. Journal of Biomolecular Screening. 18 (4), 420-429 (2013).
- Krutzik, P. O., Crane, J. M., Clutter, M. R., Nolan, G. P. High-content single-cell drug screening with phosphospecific flow cytometry. Nature Chemical Biology. 4 (2), 132-142 (2008).
- Vlahos, C. J., Matter, W. F., Hui, K. Y., Brown, R. F. A specific inhibitor of phosphatidylinositol 3-kinase, 2-(4-morpholinyl)-8-phenyl-4H-1-benzopyran-4-one (LY294002). The Journal of Biological Chemistry. 269 (7), 5241-5248 (1994).
- Chen, Z., et al. Synthesis and SAR of novel 4-morpholinopyrrolopyrimidine derivatives as potent phosphatidylinositol 3-kinase inhibitors. Journal of Medicinal Chemistry. 53 (8), 3169-3182 (2010).
- Ruegg, U. T., Burgess, G. M. Staurosporine, K-252 and UCN-01: potent but nonspecific inhibitors of protein kinases. Trends in Pharmacological Sciences. 10 (6), 218-220 (1989).
- Davis, P. D., et al. Inhibitors of protein kinase C. 1. 2,3-Bisarylmaleimides. Journal of Medicinal Chemistry. 35 (1), 177-184 (1992).
- Komander, D., et al. Interactions of LY333531 and other bisindolyl maleimide inhibitors with PDK1. Structure (London, England: 1993). 12 (2), 215-226 (2004).
- Gassel, M., et al. The protein kinase C inhibitor bisindolyl maleimide 2 binds with reversed orientations to different conformations of protein kinase A. The Journal of Biological Chemistry. 279 (22), 23679-23690 (2004).
- Faull, A., Johnstone, C., Morley, A., et al. . Novel compounds. , (2003).
- Favata, M. F., et al. Identification of a novel inhibitor of mitogen-activated protein kinase kinase. The Journal of Biological Chemistry. 273 (29), 18623-18632 (1998).
- Woods, C. M., Zhu, J., McQueney, P. A., Bollag, D., Lazarides, E. Taxol-induced mitotic block triggers rapid onset of a p53-independent apoptotic pathway. Molecular Medicine (Cambridge, MA). 1 (5), 506-526 (1995).
- Teng, X., et al. Structure-activity relationship study of novel necroptosis inhibitors. Bioorganic & Medicinal Chemistry Letters. 15 (22), 5039-5044 (2005).
- Saini, M., et al. Regulation of Zap70 expression during thymocyte development enables temporal separation of CD4 and CD8 repertoire selection at different signaling thresholds. Science Signaling. 3 (114), ra23 (2010).
- Chae, H. J., et al. Molecular mechanism of staurosporine-induced apoptosis in osteoblasts. Pharmacological Research. 42 (4), 373-381 (2000).
- Varas, A., et al. Analysis of the human neonatal thymus: evidence for a transient thymic involution. Journal of Immunology (Baltimore, MD:1950). 164 (12), 6260-6267 (2000).
- Verstichel, G., et al. The checkpoint for agonist selection precedes conventional selection in human thymus. Science Immunology. 2 (8), (2017).
- Yamaguchi, E., de Vries, J., Yssel, H. Differentiation of human single-positive fetal thymocytes in vitro into IL-4- and/or IFN-gamma-producing CD4+ and CD8+ T cells. International Immunology. 11 (4), 593-603 (1999).
- Farley, A. M., et al. Dynamics of thymus organogenesis and colonization in early human development. Development (Cambridge, UK). 140 (9), 2015-2026 (2013).
- Cali, J. J., et al. Bioluminescent assays for ADMET. Expert Opinion on Drug Metabolism & Toxicology. 4 (1), 103-120 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados