Method Article
Um método versátil para a montagem de Arabidopsis deixa para Intravital imagem de lapso de tempo
Neste Artigo
Resumo
Nós relatamos um método simples e versátil para executar ao vivo-imagem fluorescente de Arabidopsis thaliana folhas durante um período prolongado de tempo. Nós usamos uma Arabidopsis de plantas transgênicas expressando um gene repórter fluorescente sob o controle de um promotor de imunidade-relacionados como um exemplo para ganhar spatiotemporal compreensão das respostas imunes de planta.
Resumo
A resposta imune de planta associada com uma reprogramação transcriptional todo o genoma é iniciada no local da infecção. Assim, a resposta imune é regulada espacial e temporalmente. A utilização de um gene fluorescente sob o controle de um promotor de imunidade-relacionados em combinação com uma microscopia de fluorescência automatizada é uma maneira simples de entender spatiotemporal regulação da imunidade da planta. Em contraste com os tecidos de raiz que foram usados para um número de várias experiências de geração de imagens fluorescentes intravital, existem alguns exemplos de imagens ao vivo fluorescentes para os tecidos de folha que encontro uma matriz de infecções microbianas no ar. Portanto, nós desenvolvemos um método simples para montar as folhas das plantas de Arabidopsis thaliana para imagem latente da viver-pilha durante um período prolongado de tempo. Usamos plantas transgênicas de Arabidopsis expressando a proteína fluorescente amarela (YFP) genefused para o sinal de localização nuclear (NLS) sob o controle do promotor de um gene marcador relacionados com defesa, ( Pathogenesis-Related-1 PR1). Nós infiltrados uma folha transgénica com Pseudomonas syringae pv. tomate DC3000 (avrRpt2) Coe (Pst_a2) e realizado em vivo imagens lapso de tempo do sinal YFP para um total de 40 h, usando um microscópio de fluorescência automatizada. Esse método pode ser utilizado não só para estudos de respostas imunes de planta, mas também para as análises de vários eventos no desenvolvimento e respostas ambientais ocorrem nos tecidos da folha.
Introdução
Resposta imune de planta envolve uma dinâmica transcriptional reprogramação regulada pela transcrição múltiplos fatores bem como fitohormônios1. O acúmulo de dados transcriptome fornece oportunidades para recolher informações sobre o sistema imunológico de planta: por exemplo, a estrutura da rede de sinalização cascatas2. No entanto, nosso conhecimento do dinamismo espacial e temporal da imunidade da planta continua limitado3,4,5.
Em estudos anteriores, spatiotemporal regulação da expressão genética relacionados com defesa tem sido principalmente analisada utilizando hibridização in situ e um repórter do β-glucuronidase (GUS) ensaio6,7,8. Estes métodos permitem-nos Visualizar a ativação transcricional de vários genes de interesse no local. No entanto, estes procedimentos requerem fixação química de espécimes e, portanto, resultam na perda de toda a informação temporal. Eventos biológicos, tais como imunidade, progresso ao longo do tempo. O uso de luciferase como repórter permitiu a captura da dinâmica temporal do promotor de interesse3. No entanto, ensaio baseado em luciferase requer um substrato caro e altamente sensíveis detectores. Para aumentar a nossa compreensão dos aspectos spatiotemporal da resposta imune de planta usando um procedimento simples, geramos transgénicos Arabidopsis thaliana plantas expressando a proteína fluorescente amarela (YFP) gene fundidos para a sinal de localização nuclear (NLS-YFP) sob o controle do promotor do gene marcador de defesa-relacionadas, Pathogenesis-Related 1 (PR1)9. Usamos a clorofila autofluorescência, um marcador de células vivas, para capturar o processo de morte celular programada (PCD), que muitas vezes ocorre durante a imunidade acionadas effector (ETI), uma forma de imunidade planta induzida por determinado patógeno infecção9 , 10. monitoramento a dinâmica temporal da intensidade do sinal de fluorescência em movimentando-se livremente objetos, tais como folhas de Arabidopsis a viver, requer uma complexa software processamento de imagem construído internamente ou disponível comercialmente. Alternativamente, impedindo que espécimes vá é um método simples para resolver a questão. Aqui, nós desenvolvemos um método simples e versátil para montagem vivendo folhas de uma planta de Arabidopsis transgênica para observação de longo prazo sob um microscópio de fluorescência automatizada. O método nos permite capturar o promotor deixa de dinâmica no solo cultivado planta intacta durante alguns dias.
Protocolo
Nota: É importante impedir o sono movimento de vida folha amostras durante a imagem latente de lapso de tempo. Para minimizar o estresse mecânico nas folhas, fixação suave de folhas é necessária. Apenas amostras de folha devidamente preparado produzem imagens lapso de tempo apropriadas para várias análises de imagem. Um protocolo utilizando plantas transgênicas de Arabidopsis expressando YFP-NLS fusão sob o controle do promotor do gene de PR1 (plantas pPR1-YFP-NLS) e Pseudomonas syringae pv. tomate DC3000 estirpe (avrRpt2) (Pst_a2) é descrito abaixo como um exemplo.
1. preparação de plantas e patógenos
- Encha uma bandeja de plugue plástico celular (Tabela de materiais) com o solo autoclavado.
- Semear uma semente de Arabidopsis transgênica por célula (Figura 1A).
- A bandeja de transferência para uma sala de crescimento, a 23 ° C e crescer as plantas sob luz branca contínua durante 2-3 semanas.
- Dois dias antes da inoculação do patógeno, raia Pseudomonas syringae pv. tomate DC3000 avrRpt2 Coe (Pst_a2) de um estoque de glicerol para NYG médio (peptona 5g/L, 3 g/L de extrato de levedura, glicerol 20 mL/L, ágar bacteriológico de 15 g/L, pH 7,0) contendo 100 mg/L rifampicina e 50 mg/L canamicina e incubar a 28 ° C, por 48 h.
- Colheita das células bacterianas que aparecem na superfície do meio usando pontas de plástico, transfira para um tubo de plástico contendo 10 mM MgCl2e ressuspender. Medir a densidade óptica (OD) da solução em 600 nm (OD600). Ajustar a concentração celular final de células bacterianas para 108 unidades formação de Colônia (UFC) / mL, que corresponde normalmente ao OD600 = 0,211.
2. inoculação do patógeno
- Corte com cuidado uma ficha de célula que contém uma planta de 2-3-semanas sem danificar a planta. Defina a célula em uma bandeja de plug de célula vazia (2 x 2 células são suficientes) para manter um bom equilíbrio (Figura 1B).
- Selecione uma folha visivelmente saudável para inoculação. Geralmente, o terceiro, quarto e quinto deixa (#3, #4 e #5, respectivamente, na Figura 1C) da parte inferior da planta são fáceis de manusear. Uso deixa na mesma posição em um conjunto de experiências para melhor reprodutibilidade. Água no solo a planta antes da inoculação para a imagem latente de lapso de tempo a longo prazo.
- Opcionalmente, no caso de análise de stress-responsivo promotores como pPR1, para garantir que as plantas não são naturalmente estressadas, examine as folhas sob um microscópio de fluorescência, antes da inoculação do patógeno para verificar a ausência de sinal YFP. Excluir deixa apresentando YFP sinal do experimento.
- Use luvas de látex descartáveis antes de infiltração para evitar o contacto directo com o patógeno. Utilizando uma seringa de 1 mL no plástico, cuidadosamente infiltrar o lado abaxial da folha com a suspensão bacteriana (108 UFC/mL)11 (Figura 1D). Inoculação de uma pequena porção na metade da folha permite uma boa visualização de atividade pPR1 ; a área infiltrada torna-se visível como cor em comparação com a restante folha verde mais escuro. Tenha muito cuidado para não causar danos mecânicos à folha durante a infiltração.
Nota: Certifique-se de que todos os espaços intercelulares na área infiltrada são completamente cumprida (verticalmente) com a suspensão do patógeno; caso contrário, o domínio PCD será difícil Visualizar sob o microscópio fluorescente. Isso simplesmente pode ser confirmado pela conclusão do greening escuro na área infiltrada. - Absorva o excesso de suspensão bacteriana da área circundante a seção infiltrada da folha infiltrada com uma toalha de papel macio.
3. montagem da folha inoculada
- Imediatamente após a inoculação, corrigi uma lâmina de vidro sobre a bandeja plástica usando fita cirúrgica (Tabela de materiais), tal que a folha infiltrada está localizada no centro da lâmina de vidro. Certifique-se de que a lâmina de folha inoculada é completamente montada dentro da lâmina de vidro(Figura 2).
- Preparar duas camadas de fita plástica (no caso da fita grossa de 0,2 mm, consulte Tabela de materiais) e corte-o em duas partes (Figura 2B; partes 1 e 2) para caber os espaços ao longo do pecíolo da folha infiltrado. Organize o comprimento dessas duas peças, indicado com uma seta com duas pontas na Figura 2B, para se encaixar o comprimento da seta de duas pontas, mostrado na Figura 2.
Nota: Cortar um canto de cada uma das duas peças ajuda a evitar danos à lâmina da folha (Figura 2B, setas; ver também passo 3.4 abaixo). Qualquer tipo de fita plástica com espessura similar é apropriado para fazer uma ponte sobre o pecíolo. A rigidez da fita plástica é importante para fácil manuseio durante o procedimento descrito abaixo. - Usando um par de pinças bem, fica pedaços de fita 1 e 2 de cada lado do pecíolo espinhoso, tal que os cantos cortados de cada peça alinharem com a base da lâmina da folha (Figura 2C). Certifique-se de que os pedaços de fita não toque a lâmina pecíolo ou folha.
Nota: Estas peças de duas camadas de fita plástica na base da lâmina da folha agem como espaçadores e evitar estresse físico sobre o pecíolo durante a montagem. - Preparar um dispositivo adicional de duas camadas de fita plástica (Figura 2B, parte 3) para ajustar o tamanho da seta de duas pontas em Figura 2B e colá-la em cima os pedaços de fita 1 e 2 para formar uma ponte sobre o pecíolo ( Figura 2D, E). Tenha muito cuidado para não pegar a lâmina pecíolo e folha diretamente entre os pedaços de fita em posições indicadas com setas na Figura 2D, E.
- Delicadamente enfiar um pequeno pedaço de fita cirúrgica (Figura 2-F, peça 4) sobre a lâmina de vidro acima da ponta da lâmina da folha para que a lâmina da folha é fixada muito suavemente sobre a lâmina de vidro. Apenas Pressione firmemente para baixo a parte da fita cirúrgica diretamente tocando a lâmina de vidro (Figura 2F, área delineada com uma linha tracejada vermelha), não a outra parte sobrejacente a folha.
- Delicadamente enfiar outro pequeno pedaço de fita cirúrgica (Figura 2G, peça 5) na borda do pecíolo espinhoso e pedaços de fita plástica (1, 2 e 3) para que o pecíolo é muito suavemente fixado a lâmina de vidro e o plástico fita peças. Só firmemente fixar a parte da fita cirúrgica diretamente tocando a lâmina de vidro para a lâmina de vidro e pedaços de fita plástica (Figura 2G, área delineada com uma linha tracejada vermelha), não a outra parte sobrejacente do pecíolo.
- Evitar que folhas vizinhas movendo-se para o campo de visão do microscópio usando pontas de pipetas 200 µ l (Figura 2H). Insira as pontas de pipetas no solo para segurar suavemente as folhas vizinhas longe da folha infiltrada. Tenha cuidado para não inserir as pontas muito profundo no solo para evitar dano possível raiz.
Nota: A planta preparada agora está pronta para a imagem latente de microscópio de fluorescência.
4. observação microscópica de lapso de tempo
- Ligue o microscópio fluorescente.
Nota: Aqui, um estereomicroscópio automatizado equipado com um altamente sensível monocromático de 1,4 megapixels câmera digital no modo de 12 bits é usada (Tabela de materiais). O microscópio deve ser colocado em um quarto escuro, equipado com uma unidade de ar condicionado ou em um armário escuro com ventilação adequada para manter a temperatura ambiente a 23 ° C durante a imagem latente de lapso de tempo. - Defina a planta no espaço acima sob a lente objetiva do microscópio estereoscópico para a imagem latente.
- Configurar os parâmetros para a imagem latente de lapso de tempo (veja exemplos na tabela 1). Certifique-se de etapas de programa para a exposição à luz durante o período de intervalo de imagens lapso de tempo, desde que a luz tem um impacto importante na planta imunidade12.
- Use o filtro YFP convencional (excitação 500-520 nm; emissão 540-580 nm) para visualizar o sinal YFP.
- Use o filtro vermelho Texas (TXR) (excitação 540-580 nm; passe longo de emissão 610 nm) para visualizar a autofluorescência de clorofila, para que o domínio PCD é visível como uma área escura (sem autofluorescência) rodeada por YFP-positivo de camadas de célula9 (Figura 3 ).
- Use a configuração convencional de campo epi-brilhante para obter etapas adicionais exposição à luz.
Nota: Antes do experimento, medir o poder da fonte de luz brilhante-epi e ajustá-lo ao nível da condição de crescimento de planta própria.
- Execute o programa de geração de imagens lapso de tempo. Para observação de longo prazo durante vários dias, considere-se de molhar a planta adequadamente, por exemplo, durante as etapas de exposição à luz.
- Após a aquisição da imagem, omita canais extras usados para exposição à luz nos intervalos (correspondendo a 3-8 de canaletas na tabela 1) do conjunto de dados. Analise dados com vários métodos, como análise de região de interesse (ROI), usando o software de análise de imagem diferente como Fiji9.
Resultados
Aqui, usamos Pst_a2-induzido ETI como exemplo para a imagem latente de lapso de tempo. Obtiveram-se dados de lapso de tempo como uma série de imagens, alguns dos quais são mostrados na Figura 3Be como um filme lapso de tempo (Supplemental filme 1)9. Em experiências bem sucedidas usando pPR1-YFP-NLS durante Pst_a2-induzido ETI, ativação transiente de pPR1 observou-se, como é evidente de YFP expressando focos, em várias camadas de células em torno do domínio PCD (Figura 3B) 9. a ativação da pPR1 nas células em torno do domínio PCD geralmente começa em aproximadamente 5 horas post inoculação (hpi), picos em aproximadamente 12 hpi e dura até 40 hpi (Figura 3B)9. Desde imagens foram adquiridas utilizando um sistema de epi-fluorescente, os sinais YFP obtidos aqui foram gerados de várias camadas de células adaxial, incluindo as células epidérmicas, bem como superior do mesofilo.

Figura 1 : Método utilizado para a infiltração de patógeno bacteriano para o Arabidopsis thaliana folha. (A) de dois a três-semanas Arabidopsis plantas foram cultivadas em uma bandeja de plug de célula. (B) uma célula ficha contendo a planta selecionada deve ser usado para inoculação e observação foi cortada e colocada na bandeja do plug de célula vazia para manter o equilíbrio. (C) O terceiro, quarto e quinto folhas (#3, #4 e #5, respectivamente) são adequados para análise de imagem. (D) infiltração de suspensão de patógeno de uma folha seleccionada utilizando uma seringa de auto-injeções. A área infiltrada pode ser reconhecida com base em sua cor verde mais escura do que as folhas restantes. Clique aqui para ver uma versão maior desta figura.

Figura 2 : Método utilizado para a montagem dos infiltrados Arabidopsis folha para a imagem latente de lapso de tempo. (A) fotografia da planta Arabidopsis com uma lâmina de vidro fixada sob a folha inoculada. A folha infiltrada foi posicionada no centro do slide vidro. A seta dupla indica o comprimento dos espaçadores plásticos fita. (B) preparação de espaçadores de plástico fita e ponte. Dupla - fita plástica em camadas foi cortado em duas partes (1 e 2) para os espaçadores e uma outra parte (3, delineados com uma linha tracejada vermelha) foi cortada para preparar uma ponte. Os comprimentos de duas pontas de setas são quase idênticos ao comprimento da seta de duas pontas no (A). Um dos quatro cantos das partes 1 e 2, indicadas com flechas, foram cortados. (C), dois pedaços de fita plástica de duas camadas (numeradas 1 e 2) foram cuidadosamente colados ao vidro no slide ao longo do pecíolo, usando um par de pinças bem, sem fazer contato direto com a lâmina do pecíolo e folhas. (D) uma peça adicional de duas camadas fita plástica (numerada como 3), que foi preparada a partir de (B), foi colocado em ambos os espaçadores basais (1 e 2) para formar uma ponte sobre o pecíolo, garantindo para não pegar o pecíolo entre 1 e 2 em fitas posições indicadas com setas. (E) fotografia mostrando o pecíolo gravado. É importante não pegar o tecido de planta diretamente entre os pedaços de fita plástica em posições indicadas com setas. (F) uma pequeno pedaço de fita cirúrgica (numerado como 4) foi colado suavemente a lâmina de vidro ao redor da ponta da lâmina da folha. Somente a área descrita pela linha tracejada vermelha foi pressionada firmemente a lâmina de vidro. (G), um pequeno pedaço de fita cirúrgica (numerado como 5) delicadamente foi colado na fronteira do pecíolo e pedaços de fita plástica. Somente a área descrita pela linha tracejada vermelha foi pressionada para fixando o pecíolo na corrediça de vidro e pedaços de fita plástica. Pontas de pipeta descartável (H), dois foram usadas para evitar que folhas vizinhas movendo-se para o campo de visão do microscópio. As pontas de pipeta foram inseridas diretamente no solo em posições adequadas. Clique aqui para ver uma versão maior desta figura.
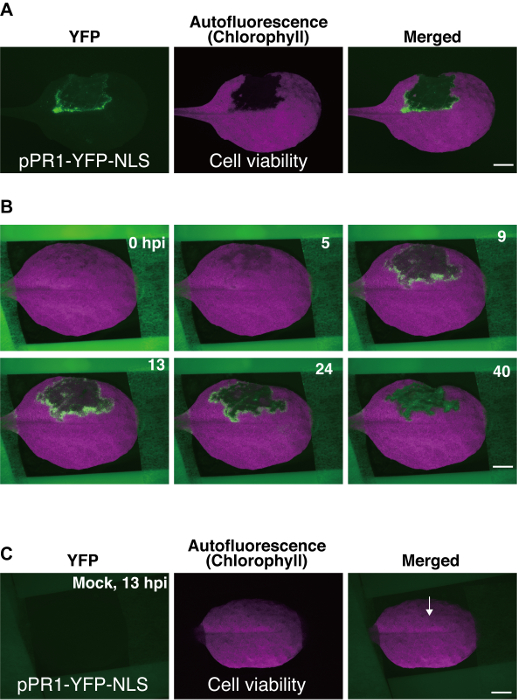
Figura 3 : Representante resultados usando o Arabidopsis método de montagem de folha. (A) fluorescente stereomicroscopic imagens de uma folha de Arabidopsis transgênica (planta pPR1-YFP-NLS). Uma porção da folha foi infiltrada com Pst_a2 (108 UFC/mL) em sua superfície abaxial, e imagens foram capturadas em 22 horas post inoculação (hpi). Núcleos em que o promotor pPR1 foi ativado foram detectados usando o filtro YFP e morte celular programada (PCD) foi detectado com base na perda de clorofila autofluorescência usando o filtro vermelho Texas (TXR). Barra de escala = 2,5 mm. (B) de algumas imagens de lapso de tempo selecionadas de uma coleção de 800 imagens obtidas utilizando o protocolo descrito aqui. Estes dados fornecem uma visão na vivo geral da dinâmica spatiotemporal pPR1 atividade para 40 hpi. São mostradas imagens mescladas de imagens YFP e TXR. Barra de escala = Mock infiltração de 2,5 mm. (C) de uma folha de transgénica (planta pPR1-YFP-NLS). Em infiltração simulada, 10 mM MgCl2 foi infiltrada no lado abaxial da folha, seguido por imagens de lapso de tempo. Tratamento simulado não causa gravidez ectópica pPR1 atividade e não interferiu com autofluorescência de clorofila em 13 hpi. Uma seta indica a posição de infiltração trocista. Barra de escala = 2,5 mm. Estas imagens foram modificadas de uma anterior de estudo9. Clique aqui para ver uma versão maior desta figura.
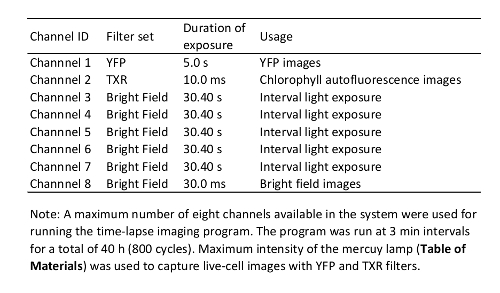
Tabela 1: Time-Lapse de imagem programa utilizado neste estudo.
Suplementar 1 filme: filme lapso de tempo, mostrando a dinâmica spatiotemporal de pPR1 ativação em um transgênico Arabidopsis folha seguinte imunidade acionadas effector (ETI) induzida por Pst_a2 inoculação. Uma parte do lado abaxial da folha transgênica carregando a construção pPR1-YFP-NLS foi infiltrada com Pst_a2 (108 UFC/mL). Imagens foram adquiridas em intervalos de 3 min para um total de 40 h/a. O carimbo de hora mostrado é o formato dd:hh:mm:ss.sss. Este filme foi modificado de um anterior de estudo9. Por favor clique aqui para ver este vídeo. (Botão direito do mouse para fazer o download.)
Discussão
Aqui, nós relatamos um método simples para montar uma vida Arabidopsis folha expressa um gene repórter fluorescente sob o controle de um promotor de interesse para a observação a longo prazo usando um microscópio fluorescente automatizado. Imagem de lapso de tempo de um repórter fluorescente foi frequentemente realizada em tecidos de raiz; no entanto, poucos estudos semelhantes foram realizados em tecidos da folha. Isto é mais provável porque as folhas são capazes de mover-se livremente no espaço, Considerando que as raízes são muitas vezes enterradas e fixadas em meio de ágar sólido.
Neste relatório, enfocamos a spatiotemporal dinâmica da atividade pPR1 durante ETI induzida por Pst_a2. Além da fixação suave da folha detalhada acima, é importante Visualizar claramente a spatiotemporal dinâmica de eventos celulares, como ativação de promotor e PCD. Se a distinção entre células mostrando atividade pPR1 e PCD não é afiada, certifique-se de que todos os espaços intercelulares na área infiltrada são completamente preenchidos com a suspensão do patógeno (consulte a etapa 2.4). Isto é crítico quando usando duas oculares de campo amplo da fluorescência, desde que estes microscópios capturar todos os sinais detectáveis ao longo da mesma posição vertical da amostra. Autofluorescência de clorofila de células sobreviventes acima ou abaixo das células no domínio do PCD máscaras facilmente as células mortas, exibindo sem autofluorescência. Isto também é verdadeiro para sinal YFP.
Condições para a imagem latente de lapso de tempo precisam ser estabelecida cuidadosamente por meio de vários experimentos preliminares sob diferentes condições experimentais. Parâmetros para a imagem latente de lapso de tempo dependem de vários fatores tais como o sistema microscópico, plantas transgênicas e patógenos. Para obter esses parâmetros, primeiro foram analisados vários tempos de exposição para a intensidade de sinal YFP na folha infiltrada no hpi 7, que quase coincide com a ativação inicial da pPR1. Determinou-se uma exposição s 5 conforme apropriado para a captura de sinal YFP com o microscópio utilizado neste estudo. Realizou-se um teste semelhante para clorofila autofluorescência de imagem. Exposição da amostra à luz entre intervalos de 3 min foi programada para o programa da imagem latente de lapso de tempo como imagem de campo brilhante normal com tempo máximo de exposição. Nosso sistema (Tabela de materiais) nos permitiu ter 2,5 min além YFP, TXR e imagem latente de campo brilhante. Essa restrição foi a principal razão para escolher um intervalo de 3 min. Em seguida, nós confirmamos que essa condição de lapso de tempo não causado nenhum dano aparente para as amostras de plantas e não induz a gravidez ectópica acionamento do refletor forç-relacionados da pPR1 (Figura 3B, C). Isto levou ao desenvolvimento do programa utilizado neste estudo. Assim, intervalos de 3 min da imagem latente de fluorescência foram considerados suficientes para capturar pPR1 dinâmica durante Pst_a2-mediada ETI9.
Construções de promotor-repórter, especialmente com o repórter fluorescente fundido para o NLS, têm sido utilizadas por muitos grupos e estão facilmente disponível a partir da comunidade de investigação; usamos a construção publicada pelo Kubo et al . 13. assim, o protocolo descrito aqui pode ser usado em qualquer estudo de biologia de planta examinando tecidos da folha, se plantas transgênicas apropriadas estão disponíveis. Nosso protocolo simples e fácil proporciona uma grande oportunidade para os pesquisadores que pretendem analisar a dinâmica spatiotemporal de qualquer evento biológico, ocorrendo em folhas, como a resposta imune. É plausível que o nosso método usando pedaços de fita induz um ligeiro stress físico sobre as amostras. No entanto, esta questão pode ser controlada, incluindo adequados controlos positivos e negativos, tais como tratamentos simulados, nos experimentos (Figura 3C). As condições experimentais podem ser modificadas e otimizadas, analisando esses controles sob diferentes condições ainda mais.
Nos últimos anos, o rápido desenvolvimento de instrumentos e técnicas de imagem estimulou o interesse dos pesquisadores nos aspectos spatiotemporal complexos de eventos biológicos. Em qualquer análise de imagem, adequada montagem e fixação dos espécimes estão entre as questões mais importantes. O método simples e versátil de montagem viver que deixa de Arabidopsis desenvolvido neste estudo pode ser aplicado e otimizado para várias experiências de imagens.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi financiado pela Agência de tecnologia e ciência de Japão [PRESTO117665 para S.B., ERATOJPMJER1502 de N.N.] e pela sociedade para a promoção da ciência (Grants-in-Aid para start-up de atividade de pesquisa [22880008 para S.B.] e para jovens cientistas (B) [Japão 23780040 para S.B.]). Agradecemos a r. Senzaki, Y. Suzuki, Y. Sugisawa e E. Betsuyaku pela excelente assistência técnica, J. Parker para fornecer a tensão de Pst_a2 e T. Demura para fornecer o vetor de pBGYN.
Materiais
| Name | Company | Catalog Number | Comments |
| Bacto Agar | BD biosciences | 214010 | |
| Bacto Protease Peptone No. 3 | BD biosciences | 211693 | |
| Bacto Yeast Extract | BD biosciences | 212750 | |
| BM2 soil | Berger | ||
| Glycerol | nacalai tesque | 17017-35 | |
| Kanamycin sulfate | Wako | 113-00343 | |
| Leica M205FA with DFC365FX, LED_MCI and Leica EL6000 (Las X software-regulated) | Leica Microsystems | Yellow fluorescent protein (YFP) and Texas Red (TXR) filters installed | |
| Magnesium Chloride Hexahydrate | Wako | 135-00165 | |
| Micro Slide Glass (Size: 76 mm × 26 mm, Thickness: 1.0–1.2 mm) | MATSUNAMI | S1112 | |
| Micropore Surgical Tape (1.25 cm × 9 m) | 3M | 1530-0 | |
| Needleless 1 ml plastic syringe | Terumo | SS-01T | |
| Plastic cell plug tray (cell size: 44 mm × 44mm × 44 mm) | Tanaka sangyo, Japan | htray-bk72 | Any tray with similar sized cells can be used |
| Rifampicin | nacalai tesque | 30259-81 | |
| Plastic Tape (0.2 mm thick × 19 mm wide × 10 m long) | Yamato | No0200-19-1 | Any plastic/vinyl tape with similar thickness can be used |
Referências
- Tsuda, K., Somssich, I. E. Transcriptional networks in plant immunity. New Phytologist. 206 (3), 932-947 (2015).
- Mine, A., Sato, M., Tsuda, K. Toward a systems understanding of plant-microbe interactions. Frontiers in Plant Science. 5, (2014).
- Murray, S. L., Thomson, C., Chini, A., Read, N. D., Loake, G. J. Characterization of a novel, defense-related Arabidopsis mutant, cir1, isolated by luciferase imaging. Molecular Plant-Microbe Interactions. 15 (6), 557-566 (2002).
- Spoel, S. H., Johnson, J. S., Dong, X. Regulation of tradeoffs between plant defenses against pathogens with different lifestyles. Proceedings of the National Academy of Sciences of the United States of America. 104 (47), 18842-18847 (2007).
- Asai, S., Shirasu, K. Plant cells under siege: plant immune system versus pathogen effectors. Current Opinion in Plant Biology. 28, 1-8 (2015).
- Schmelzer, E., Kruger-Lebus, S., Hahlbrock, K. Temporal and Spatial Patterns of Gene Expression around Sites of Attempted Fungal Infection in Parsley Leaves. The Plant Cell. 1 (10), 993-1001 (1989).
- Ohshima, M., Itoh, H., Matsuoka, M., Murakami, T., Ohashi, Y. Analysis of stress-induced or salicylic acid-induced expression of the pathogenesis-related 1a protein gene in transgenic tobacco. The Plant Cell. 2 (2), 95-106 (1990).
- Rushton, P. J., Reinstädler, A., Lipka, V., Lippok, B., Somssich, I. E. Synthetic plant promoters containing defined regulatory elements provide novel insights into pathogen- and wound-induced signaling. The Plant Cell. 14 (4), 749-762 (2002).
- Betsuyaku, S., et al. Salicylic Acid and Jasmonic Acid Pathways are Activated in Spatially Different Domains Around the Infection Site During Effector-Triggered Immunity in Arabidopsis thaliana. Plant & Cell Physiology. 59 (1), 8-16 (2018).
- Dodds, P. N., Rathjen, J. P. Plant immunity: towards an integrated view of plant-pathogen interactions. Nature Reviews Genetics. 11 (8), 539-548 (2010).
- Katagiri, F., Thilmony, R. The Arabidopsis thaliana-Pseudomonas syringae interaction. Arabidopsis Book. , (2002).
- Zeier, J., Pink, B., Mueller, M. J., Berger, S. Light conditions influence specific defence responses in incompatible plant-pathogen interactions: uncoupling systemic resistance from salicylic acid and PR-1 accumulation. Planta. 219 (4), 673-683 (2004).
- Kubo, M., et al. Transcription switches for protoxylem and metaxylem vessel formation. Genes & Development. 19 (16), 1855-1860 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados