Method Article
Следующее поколение Секвенирование для обнаружения осуществимых Мутации в твердых и жидких опухолей
В этой статье
Резюме
This manuscript describes clinical protocols for two next-generation sequencing panels. One panel interrogates hematologic malignancies while the other panel targets genes commonly mutated in solid tumors. Molecular classification of driver mutations in human malignancies offers valuable prognostic and predictive information.
Аннотация
As our understanding of the driver mutations necessary for initiation and progression of cancers improves, we gain critical information on how specific molecular profiles of a tumor may predict responsiveness to therapeutic agents or provide knowledge about prognosis. At our institution a tumor genotyping program was established as part of routine clinical care, screening both hematologic and solid tumors for a wide spectrum of mutations using two next-generation sequencing (NGS) panels: a custom, 33 gene hematological malignancies panel for use with peripheral blood and bone marrow, and a commercially produced solid tumor panel for use with formalin-fixed paraffin-embedded tissue that targets 47 genes commonly mutated in cancer. Our workflow includes a pathologist review of the biopsy to ensure there is adequate amount of tumor for the assay followed by customized DNA extraction is performed on the specimen. Quality control of the specimen includes steps for quantity, quality and integrity and only after the extracted DNA passes these metrics an amplicon library is generated and sequenced. The resulting data is analyzed through an in-house bioinformatics pipeline and the variants are reviewed and interpreted for pathogenicity. Here we provide a snapshot of the utility of each panel using two clinical cases to provide insight into how a well-designed NGS workflow can contribute to optimizing clinical outcomes.
Введение
секвенирования следующего поколения (NGS) клинических образцов онкологическими становится все более широкое распространение в течение последних нескольких лет, как рост научных точек литературы о важности выявления генетических изменений наведения и прогнозирующие / прогностических молекулярных маркеров. Multi-ген панели анализирует и целые секвенирование экзома исследования в обоих эпителиальных 1,2 и гематологические 3 злокачественных опухолей укрепили концепцию опухоли неоднородности и клоновых эволюции , как болезнь прогрессирует и рецидивы. Кроме того, в отличие от конкурирующих технологий , таких как полимеразная цепная реакция (ПЦР) или Sanger секвенирования, НГС может обнаружить большинство геномных изменений во всех клинически значимых генов рака в одном анализе 4.
Центр диагностики Персонализированные первоначально запущен с двумя клиническими NGS панелями, сделанными на заказ Гематологический Panel (Гем-NGS панель) и выключенного готовые панели рака для образцов FFPE (Solid-NGS панели) (см Рисунок 1). Эти панели покрывают клинически значимые или высокие процентные участки выбранных генов; не все гены или экзоны полностью покрыты. Ампликоны генерируются путем гибридизации зонда с последующим расширением и перевязки. Целевые регионы дополнительно усиливаются с помощью ПЦР с универсальными двойственных индексированные праймеров, что позволяет до 96 образцов должны быть объединены для секвенирования.
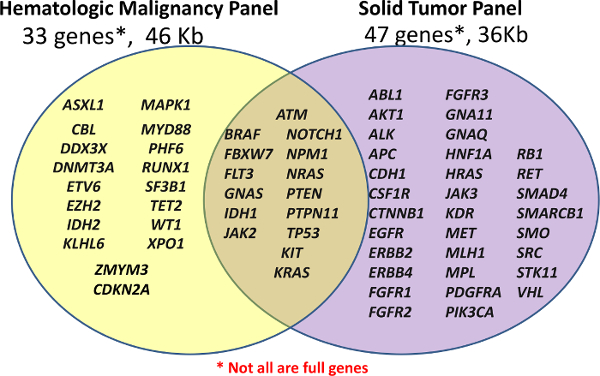
Рисунок 1:. Список Гены , охваченного в панели подготовки библиотеки осуществляется с использованием либо пользовательских Гематологический Panel (Панель Гем-NGS) из 33 генов или вне-полки Amplicon Рак панели (Solid-NGS) из 47 генов. Не все гены или экзонов покрыты в полном объеме, так как некоторые ампликонов может охватывать только определенные точки доступа. Пожалуйста , нажмите здесь , чтобы посмотреть большую версию этого рисунка. </ А>
Содержание для Гем-NGS панель была получена из нескольких источников, но сосредотачивается вокруг 16 генов мутировал в острый миелоидный лейкоз (ОМЛ) , описанной ранее , как демонстрируют высокий уровень клинической полезности 5. Solid-NGS Панель серийно выпускаемых с целевых регионах на основе широко мутировавших генов рака , как сообщалось в каталоге соматические мутации в раковых заболеваний (COSMIC) базы данных 6.
Несколько ключевых шагов характеризуют общий рабочий процесс для клинических NGS. После того, как врач приказывает испытание, патологоанатом определяет адекватность образца для анализа следующих процентах опухолей и объема образца. В нашем учреждении, мы требуем, по крайней мере, 10% опухоли из-за скорости фон секвенирования ошибки ( "шум") технологии и эффективности целевого подхода. Если ткань достаточно для тестирования, геномную ДНК экстрагируют. Эту ДНК затем подвергают многократному контролю качества (QC) шаги. Если ДНК проходит контроль качества, библиотека ампликона генерируется и секвенировали. Полученные данные анализируются через трубопровод биоинформатики в доме. После анализа биоинформатики, варианты вручную рассмотрены и интерпретированы для патогенности перед введением в клинический отчет. Ниже мы опишем два случая, которые прошли через этот строгий рабочий процесс и в конечном итоге привело к изменениям в клиническом лечении.
Случай 1 - острый миелоидный лейкоз
Биопсия костного мозга у пациента был для диагностики ОМЛ без созревания. Цитогенетические исследования были направлены на образец костного мозга и показали нормальную женскую кариотип. Были 95% циркулирующего Взрывы представляют, поэтому периферийную образец крови был послан для персонализированного диагностического тестирования на Гем-NGS панели.
Острый миелоидный лейкоз (ОМЛ) является гематологической злокачественной миелоидного происхождения белых кровяных клеток. обнаружениегенных мутаций в борьбе с отмыванием денег приобретает все большее значение для прогноза и лечения, с рецидивирующим генные мутации признаны важными в патогенезе и прогнозе 7. Мутации в NPM1 и CEBPA были связаны с благоприятным прогностическим риском, в то время как внутренние тандемные дупликации (СДМТ) в FLT3 были связаны с менее благоприятным исходом 8. Все больше доказательств поддерживает патогенную роль для этих и других мутаций в борьбе с отмыванием денег 9.
Случай 2 - легких Аденокарцинома
Биопсия левой надключичной массы от пациента B продемонстрировали легочной аденокарциномы. Биопсийного материала из залитой в парафин (FFPE) масса лимфатических узлов, фиксированных формалином был отправлен для геномного тестирования (панель Полупроводниковый NGS) в виде рулонов / завитки с более чем 50% опухолей, чтобы определить, была ли мутация присутствует для целенаправленного терапевтического вмешательства.
Lung ОТМэр является ведущей причиной смертности, связанной с раком в Соединенных Штатах и делится на два основных типа, немелкоклеточного рака легкого (НМРЛ) и рака легких мелкоклеточный (МРЛ). НМКРЛ может быть дополнительно определен как или аденокарцинома или плоскоклеточный рак, на основании гистологического исследования пораженного участка. Lung аденокарциномы является наиболее распространенным подтипом рака легких, видел как у курящих и некурящих, и является наиболее распространенной формой рака легких для некурящих 10. Молекулярные исследования аденокарциномы легких выявили мутации в нескольких онкогенов 11. Наиболее распространенные мутации драйверов , выявленные у курильщиков являются мутации в KRAS и BRAF. Наиболее распространенные мутации в некурящие являются мутации в EGFR и перегруппировки с участием генов ALK, RET и ROS1. Опухоли легких были описаны с экзон 20 вставки в рамке считывания в гене ERBB2 (HER2 / NEU). Наиболее распространенной аномалией в HER2 / Neu происходит усиление этого локуса при раке молочной железы , для которых целевая терапия доступна (трастузумаб: гуманизированное моноклональное антитело против HER2 / Neu). HER2 / Neu экзон 20 вставки , что наблюдается в 2 - 4% от легкого adenocarcimomas 12 показал частичный ответ на комбинированной терапии с HER2 / Neu и ингибиторы МРМ (neratinib и Темсиролимус, соответственно) 13.
протокол
Этот протокол содержит существенные шаги двух проверенных лабораторных испытаний разработаны для геномного профилирования твердых и жидких опухолей, соответственно. Испытания проводились в лаборатории осуществляется в соответствии с требованиями клинических изменений Лабораторные улучшения (CLIA) 1988 года.
1. ДНК Извлечение из периферической крови или костного мозга
- Определите , сколько крови или костного мозга , чтобы взять на основании таблицы 1.
| Образец / WBC | Сумма, подлежащая обработке, как 1 мл крови |
| Костный мозг | 250 мкл |
| Кровь WBC 12000 - 50000 | 1 мл |
| Кровь WBC 50000 - 100000 | 500 мкл |
| Кровь WBC 100000 - 200000 | 200 мкл |
| Кровь WBC> 200000 | 100 мкл |
| * Для получения крови WBC <12000, берут 2 мл крови | |
Таблица 1:. Кровь / костного мозга Объем Использовать таблицу Поскольку количество лейкоцитов будет отличаться от образца к образцу, то трудно определить конкретный объем крови для использования. Таким образом, количество крови, которые необходимо использовать для анализа должно быть определено, глядя на количество лейкоцитов (WBC) перед началом анализа. Несмотря на то, что меньше крови используется, он по-прежнему следует рассматривать, как будто ее 1 мл поскольку объем крови, используемой снижается, так как количество клеток, присутствующих больше, чем обычно.
- Следовал протоколу коммерчески доступного набора для выделения геномной ДНК.
2. ДНК Извлечение из фиксированных формалином залитых парафином (FFPE) ткани
- на основанииобласть опухоли патологоанатом облет на слайде H & E, выстраиваются в линию неокрашенных слайды с направляющей H & E горкой и наметить аналогичную площадку для добычи. Для макро-рассечение, процесс только один экземпляр / набор пациента слайдов одновременно.
- Нагреть скользит по C тепла блока 45 °, чтобы слегка расплавить парафин. Тщательно очистить ткань в пределах линий, которые отмечены на слайде, используя новый скальпель для каждого образца должны быть извлечены. Поместите воск соскобы в соответствующей маркировкой 1,5 мл трубки. Будьте осторожны, потому что соскабливают воск очень электростатической и может выскочить из трубы.
- Добавьте 320 мкл Депарафинирование раствора на каждые пять-шесть 5 мкм секций (25 - 30 мкм общего объема). Например, если в пробирку, содержащую 3 секции рулона 10 мкм / ротор будет обрабатываться, а затем использовать 320 мкл, но, если были получены 5 секций при той же толщине затем используют 640 мкл.
- Вортексе в течение по крайней мере 10 секунд и выполнять воднuick спина в микроцентрифуге для удаления ткани / воск с боков и кепке и в раствор. Инкубируют при 56 ° С в течение 3 мин, а затем инкубируют при комнатной температуре в течение 5 - 10 мин..
- После инкубации RT, добавляют 180 мкл буфера ATL для каждого 320 мкл раствора добавляют Депарафинирование. Фарш ткани в десять раз с использованием стерильного мини-пестик с использованием нового пестик для каждого образца. Убедитесь, что нет ткань прилипла к пестиком, так как это может быть очень липким. Vortex суспензию энергично в течение 3 сек, затем центрифуге при максимальной скорости в течение 1 мин.
- Добавляют 10 мкл протеиназы К в нижнюю прозрачную фазу. Осторожно перемешать с помощью пипетки вверх и вниз, чтобы убедиться, что ткань ресуспендировали. Не вихрь. Инкубировать при 56 ° С в течение ночи при встряхивании при 400 - 500 оборотов в минуту.
- На следующее утро, проверьте, если ткань полностью растворяется (это произошло, если нижний раствор прозрачен). Если нижнее решение не ясно, а затем вихрь энергично в течение 3 сек и центрифуге при максимальной скорости Foг 1 мин. Добавить дополнительный 5 - 10 мкл протеиназы К и инкубировать при 56 ° С в течение еще 30 - 60 мин.
- Инкубируют при 90 ° С в течение 1 часа, чтобы помочь реверсировать формальдегида поперечную сшивку. Разрешить образцы охлаждают до комнатной температуры в течение 5 - 10 мин, а затем кратковременно центрифугировать каждую пробирку, чтобы закрепить жидкость.
- Перенести нижнюю прозрачную фазу в меченым 1,5 мл пробирку. Если имеется несколько трубок из того же образца пациента (например, в случае, если используются несколько рулонов), рекомбинируют нижние фазы в одну трубу в данный момент. Примечание: Передача небольших количеств Депарафинирование решения не должны мешать процедуры очистки, но есть риск, если большое количество переносится.
- Добавьте 2 мкл раствора РНКазы A. Вихревой нежно или инвертировать в 25 раз и быстрое вращение в микроцентрифуге. Инкубируют при комнатной температуре в течение 5 мин.
- Добавить 200 мкл белка осадков раствора. Если делать один или два рулона, используйте 200 мкл. Если делать три рулонаы сразу, а затем использовать 400 мкл. Вортексе на высокой скорости в течение 30 секунд, чтобы равномерно смешать лизис буферы. Инкубируют на льду в течение 5 мин или образцы могут остаться на льду в течение до одного часа.
- Центрифуга при 5000 х г в течение 5 мин. Осажденный белок должен образовывать плотный, белый осадок. Налейте надосадочную жидкость в меченым 1,5 мл пробирку, а затем инкубируют образцы на льду в течение не менее 3 мин. Центрифуга при 5000 мкг в течение 3 мин.
- Добавить 200 мкл 2-пропанола (изопропанол) на каждые 180 мкл буфера АТЛ добавлены ранее к меченым 1,5 мл трубки (это может быть необходимо использовать 2 мл пробирку). Например, если делать три кассеты добавить 600 мкл изопропанола. Добавляют 1 мкл гликогена для каждого 180 мкл буфера ATL добавляют ранее в изопропаноле и Переверните пробирку несколько раз перемешать.
- Осторожно добавьте супернатант от стадии Протеин осаждением в смеси изопропанола. Смешайте трубку, переворачивая осторожно, по меньшей мере в 50 раз.
- Центрифуга при максимальной зрПЕД в течение 3 мин. ДНК будет выражаться в виде небольшого белого осадку в нижней части трубы.
- Налейте или аспирата надосадочную в соответствующую пробирку отходов. Хранить в отдельную пробирку 1,5 мл отходов для каждого образца в случае осадок приходит смещаются, поэтому он не будет потерян или смешивать с отходами других образцов. Слить трубку на бумажное полотенце и обеспечить большинство Изопропанол удаляется.
- Добавьте 300 мкл свежеприготовленного 70% этанола. Переверните пробирку осторожно несколько раз, чтобы вымыть осадок. Постарайтесь обеспечить осадок приходит смещаться, чтобы обеспечить более тщательную очистку.
- Центрифуга на максимальной скорости в течение 5 мин. а затем осторожно удалить этанол. Осадок может быть свободным, поэтому выливать или аспирата медленно и смотреть осадок. Удалить избыток этанола с внутренней стороны трубки, не касаясь гранул. Дайте образцы высохнуть на воздухе в течение 5 - 15 мин, соблюдая осторожность, чтобы не более сухой образец.
- Добавить между 25 - 100 мкл ДНК-раствора в ГИДРАТАЦИИ еОбразец ACH на основе размера гранул ДНК и исходного количества ткани. Вихревые трубки энергично и кратко спина в микроцентрифуге. Инкубировать в течение 1 часа при 65 ° С, чтобы полностью регидратации ДНК.
3. Контроль качества ДНК Геномная
Примечание: Существуют три самостоятельные шаги для контроля качества ДНК (QC). Таблицу 2 для дальнейшего объяснения того , почему выполняется каждый шаг QC.
| инструмент | результат | индикация | Идеальный диапазон |
| DropSense96 | A260 / A230 | Определение химических загрязнителей (например, этанол) | 1,50 - 2,2 |
| DropSense96 | A260 / A280 | Идентификация белковых примесей | 1,60 - 2,2 |
| DropSense96 | концентрация | количественное ДНК | > 1 нг / мкл |
| TapeStation | ДНК мазок | Определение целостности ДНК (например, деградация / фрагментация ДНК , извлеченной) | 50%> 1000 п.о. |
| Кубит 2.0 | концентрация | Более точная количественная оценка ДНК | > 1 нг / мкл |
Таблица 2:. ДНК QC Ожидаемые результаты Все эти значения принимаются во внимание перед запуском позволяя образец , чтобы перейти к стадии подготовки библиотеки.
- Следуя протоколу производителя, запустить 2 мкл экстрагированной ДНК на флуорометре, чтобы получить рабочую концентрацию (нг / мкл) образца.
- Выполнить 1 - 2 мкл каждого образца на UV / VIS спектрофотометр, чтобы проверить качество образца (A260 / A230 и A260 / A280 крысуИОС) в соответствии с инструкциями изготовителя.
- Для FFPE образцов: В соответствии с инструкциями производителя, работать по 1 мкл каждого образца на микрожидком системы гель-электрофореза для оценки деградации / фрагментации ДНК. На рисунке 2 примерах.
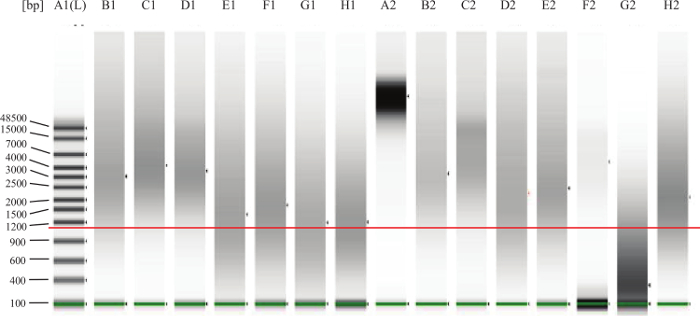
Рис . 2: геномную ДНК QC Gel Пример Зеленая линия над нижними полосами, чтобы указать нижний маркер. Добавленная в красной линией, чтобы указать, приблизительно 1000 пар оснований. Lane A2 представляет собой полностью неповрежденной ДНК, как можно было бы ожидать от свежего ТИС Сю (например, периферической крови или костного мозга). Дорожки B1, C1, D1, B2, C2, E2 являются примерами хорошей, неповрежденной ДНК FFPE. ДНК в дорожке F2, как представляется, без изменений, но при слишком низкой концентрации. ДНК в полосной G2 деградирует или фрагментарной и не будет работать в анализе. Дорожки E1, F1, G1, H1, D2,H2 представляют собой ДНК , которая попадает в «серой зоне» , что означает , что анализ может хорошо работать, но некоторые из ДНК может быть слишком поврежден или сшитый и , таким образом , он не будет хорошо работать в анализе. Пожалуйста , нажмите здесь , чтобы увидеть увеличенную версия этой фигуры.
4. Amplicon Библиотека Подготовка
- Гибридизация Oligo бассейн и разгибания Лигирование сшиваемых олигомеров
- В комнате Pre-PCR, добавьте необходимый объем Low EDTA TE, 5 мкл Oligo Tube Группы (например, Solid-NGS панели или Гем-NGS панели), и 100 - 250 нг геномной ДНК в каждую соответствующую лунку полу-огибала 96 хорошо пластины помечены как (Гибр) пластины Гибридизация.
- Добавьте 40 мкл олиго гибридизация для секвенирования Реагент 1 (OHS1) к каждому образцу в пластине Гибр. Аккуратно пипеткой вверх и вниз, по крайней мере 5 - 6 раз перемешать. Изменить советы после того, как каждый столбец, чтобы избежатьперекрестное загрязнение.
- Уплотнение пластины Гибр липкой алюминиевой фольгой и центрифуге при 1000 мкг в течение 30 сек.
- Инкубируйте планшет Гибр в гибридизация инкубаторе предварительно нагревают при 95 ° С в течение 1 мин. Установите температуру гибридизация инкубатора до 40 ° С. Продолжить инкубирование до тех пор, как он принимает инкубатор для уменьшения от 95 ° C до 40 ° C (~ 90 мин). Примечание: Это постепенное охлаждение имеет решающее значение для правильной гибридизации.
- В течение последних 15 - 20 мин инкубации гибридизации, предварительно промывать фильтр табличек (FPU). Приготовьте только скважины , которые будут использоваться в текущем тесте, то есть, только не использовать свежие / неиспользованные лунки ранее открытой фильтровальной пластины, но никогда не использовать повторно скважин , которые были использованы. Примечание: Это должно быть ясно, на основе номер комплекта на фильтровальной пластине, отметками на пластину фильтра и герметика, используемого по всей пластине.
- С помощью пипетки многоканальный, добавьте 45 мкл Строгие Wash 1 (SW1) в каждую лунку.
ВНИМАНИЕ: Содержит формамида. Накройте и центрифуга ФПУ на 2,250 мкг в течение 3 мин при 20 ° С. Проверьте каждую скважину для остаточной жидкости (> 15 мкл / лунку). - Если остаточная жидкость существует, вращать ФПУ на 180 ° и повторить шаг центрифуга снова в течение еще 3 мин. Если остаточная жидкость вращается через второй раз, а затем перейти к следующему шагу, если нет, то может быть дефект в пластину фильтра и ток пластины необходимо будет заменить.
- С помощью пипетки многоканальный, добавьте 45 мкл Строгие Wash 1 (SW1) в каждую лунку.
- После того, как гибридизация инкубатор охлаждают до 40 & deg; C, центрифугировать планшет при 1000 х г в течение по меньшей мере 30 с при 20 ° С для сбора конденсата. Перенести весь объем каждого образца от пластины Гибр на центр соответствующих предварительно промытой скважин ПДП. Изменение советы после каждого столбца, чтобы избежать перекрестного загрязнения. Накройте FPU и центрифуге при 2250 мкг в течение 3 мин при 20 ° С.
- Добавьте 45 мкл SW1 и центрифуге при 2250 х г в течение 3 мин при 20 ° C, Повторить в общей сложности двух стирок. Поворот на 180 ° FPU и центрифуга снова на 2,250 мкг в течение 3 мин, чтобы полностью удалить все SW1.
- Лукавить ПДП. Выбросьте все проточные (содержащий формамид) в соответствующую емкость для отходов. Сборку FPU с использованием другого MIDI сбора отходов плиты. Добавьте 45 мкл буфера 1 Универсальной (UB1) в каждую лунку для проб и центрифуге при 2250 х г в течение 3 мин при температуре 20 ° С. ВНИМАНИЕ: Содержит формамида.
- Добавить 45 мкл Extension лигирования Mix 3 (ELM3) к каждому образцу и на FPU пластине и пипетки вверх и вниз 3 раза перемешать. Примечание: Реакци удлинени-Лигирование происходит на мембране фильтра пластины.
- Уплотнение пластины FPU липкой алюминиевой фольгой и инкубировать всю сборку FPU в предварительно нагретом 37 ° C инкубаторе в течение 45 мин.
- Индексация и ПЦР - амплификация
- Аликвотировать индексы используются для соответствующих скважин в индексированных Amplification Plели (IAP) путем размещения праймеров в индексе плиты Крепеж, расположенных следующим образом: трубы i5 праймер (белые заглавные буквы, прозрачный раствор) в вертикальном положении, выровненные с рядами от А до Н, трубки i7 праймеров (оранжевые колпачки, желтый раствор) по горизонтали , выровнены с колоннами с 1 по 12. с помощью p10 многоканальную пипетку, добавьте 4 мкл праймеров (i7 желтый раствор) каждой строки IAP и добавить 4 мкл праймеров (i5 прозрачный раствор) в каждой колонке IAP.
- На льду или блок охлаждения, подготовить PCR Master Mix, добавляя 0,5 мкл ДНК-полимеразы 1 (TDP1) к 25 мкл ПЦР-Master Mix 2 (PMM2) на образец. Инверсия, быстро вихрь, и кратко Центрифуга PCR Master Mix для смешивания.
- Добавить 22 мкл PCR Master Mix в каждую лунку ИАП и пипеткой вверх и вниз 3 раза перемешать. Изменение советы между скважинами. Держите IAP при 4 ° С.
- После 45 мин Extension-лигирования реакции (этап 4.1.10), удалите FPU из инкубатора и осторожно удалите альuminum уплотнение из фольги. Накройте крышкой и центрифуге при 2250 мкг в течение 3 мин при 20 ° С.
- Добавить 25 мкл 50 мМ NaOH в каждую лунку для проб на FPU. Пипетировать вверх и вниз, по крайней мере, в 6 раз; обеспечить пипеток вступают в контакт с мембраной. Изменить советы после каждого столбца. Инкубируют при комнатной температуре в течение 5 мин.
- Образцы передачи элюировали из FPU к IAP следующим образом:
- Использование p100 пипетки многоканальный набор до 20 мкл, пипетки NaOH на FPU пластины вверх и вниз по меньшей мере, в 6 раз. Слегка наклоните FPU пластину, чтобы обеспечить полное устремление от пластины.
- Передача 20 мкл из FPU в соответствующем столбце ИАП. Аккуратно пипеткой вверх и вниз, по крайней мере 6 раз, чтобы тщательно совместить ДНК с PCR Master Mix. Уплотнение IAP с клейкой пленкой и центрифуге при 1000 х г в течение 1 мин при температуре 20 ° С.
- Принесите пластину ПЦР в пост-ПЦР номер и загрузите пластину на амплификатор. Запуск ПЦР-рrogram состоящий из тепла шаг при 95 ° С в течение 3 мин денатурации; с последующими 25 циклами 95 ° С в течение 30 сек, 62 ° C в течение 30 сек, 72 ° С в течение 60 сек; с последующим конечным удлинением 72 ° С в течение 5 мин; заканчивая выдерживания при 10 ° С. Если не переходить к следующему этапу после завершения ПЦР, пластина может оставаться на термоциклеру в течение ночи, или его можно хранить при температуре от 2 до 8 ° C до двух дней.
- ПЦР Очистка и бисера основе Нормализация
- Удалите магнитные гранулы для очистки, буфера для элюции (EBT), а также гель-электрофореза реагенты от 4 ° C холодильником и месте при комнатной температуре, по крайней мере за 20 мин до следующей стадии.
- После того, как ПЦР завершена, центрифуге при 1000 х г в течение 1 мин при температуре 20 ° С для сбора конденсата. Передача 1 мкл каждой ПЦР-реакции раздеться трубы / лунки планшета, содержащие 4 мкл воды для разбавления проб 1/5. Пипетировать вверх и вниз, перемешать.
- Добавьте 2 мклразбавленных проб PCRed до 2 мкл буфера Микрожидкостных-гель. Печать полосок / планшет. Взболтать при 1800 оборотах в минуту в течение не менее 30 с и центрифуге при 1000 мкг в течение 30 сек. Следуя протоколу производителя, запустите смесь на микрожидком гель для оценки , если препарат библиотека дала приемлемую библиотеку (смотри рисунок 3).
- Vortex магнитные шарики для очистки до тех пор, пока они хорошо приостанавливалось и цвет кажется однородным. Добавьте 45 мкл шариков для каждого образца.
- Уплотнение пластины с прозрачной клейкой пленкой и встряхните планшет при 1800 оборотах в минуту в течение 2 мин. Инкубируют при комнатной температуре без встряхивания в течение 10 мин.
- Поместите пластину на магнитную подставку. После того, как супернатант освобождалась, тщательно удалить и уничтожить супернатант. Если какие-либо гранулы неосторожно засасывают кончиков, дозировать шарики обратно на тарелку и дайте пластины остальное на магнит в течение 2 мин и убедитесь, что супернатант освобождалась.
- С плиты на тон магнитный стенд, добавить 200 мкл свежеприготовленного 80% этанола в каждую лунку с образцом. Переместите пластину назад и вперед несколько раз. Инкубируйте планшет на магнитной подставке в течение 30 сек. Осторожно удалите и отбросить супернатант. Повторить в общей сложности двух стирок.
- Удалить избыток этанола путем кратковременного центрифугировани планшета при 1000 XG, чтобы снизить любой этанол по бокам трубки, помещая пластину обратно на магнитную подставку, и с помощью пипетки P10 многоканальный установлен на 10 мкл для удаления этанола. Дайте бусинки высохнуть на воздухе в течение 5 - 8 мин.
- С помощью p100 пипетку многоканальный, добавить 30 мкл EBT в каждую лунку. Пипетировать вверх и вниз несколько раз, чтобы гарантировать, что шарики отрываются сторону трубки. Уплотнение пластины с прозрачной клейкой пленкой и встряхните планшет при 1800 оборотах в минуту в течение 2 мин.
- Инкубируют при комнатной температуре без встряхивания в течение 3 мин. Если есть образцы, в которых гранулы не полностью ресуспендировали, осторожно пипеткой вверх и вниз для ресуспендирования бусин,
- Поместите пластину на магнитной подставке. Использование p100 пипетки многоканальный, передача 20 мкл супернатанта к целой новой пластинкой под названием Тарелка Библиотека нормировки (ЛНФ). Передача оставшихся ~ 10 мкл отдельных библиотек секвенирования в отдельную тарелку под названием Оставшийся Начищенное Library Plate (RCLP). Хранить эту пластину вместе с окончательной библиотеки приготовительные, так как он может быть использован в качестве резерва для второго секвенирования перспективе, если это необходимо. Если не переходить к следующему этапу, ЛНФ и RCLP можно хранить при температуре от -15 до -25 ° C.
- Энергично вихрь и ресуспендируют Библиотека Нормализация шариков 1 (LNB1). Очень важно, чтобы полностью ресуспендируют LNB1 осадком из гранул в нижней части трубы. Приготовьте Нормализация Mix, путем смешивания 8 мкл LNB1 с 44 мкл библиотеки Нормализация добавок 1 (LNA1) на образец. Энергично вихрь Нормализация перемешать в течение 10 - 20 сек.
ВНИМАНИЕ: LNA1 содержит формамид. - С прерывистым инверсии ай встряхивая Нормировочная Mix, добавить 45 мкл для каждого образца ЛНП. Уплотнение пластины с прозрачной клейкой пленкой и встряхивают планшет при 1800 оборотах в минуту в течение 30 мин. В этом 30-минутной инкубации имеет решающее значение для правильной библиотеки нормализации как инкубацию больше или меньше, чем 30 мин, может повлиять на библиотечную представление и плотность кластеров.
- В течение 30-минутной инкубации, подготовить реагенты для секвенирования таянием картриджа с реагентом и Гибр Buffer (HT1) [ВНИМАНИЕ: Оба содержат формамид]. Кроме того, получить лед на более поздней стадии и обеспечить нагревательный блок, подходящий для 1,5 мл центрифужные пробирки, устанавливается на 96 ° C.
- Когда стадия смешения 30 мин завершения, поместите LNP на магнитной подставке. После того, как супернатант освобождалась, использовать пипетку многоканальную тщательно удалить и уничтожить супернатант в соответствующий контейнер для отходов.
- Удалите LNP из магнитного стенда и мыть шарики с библиотекой Нормализация Wash 1 (LNW1) следующим образом:
- Добавить 45мкл LNW1 для каждого образца скважины.
ВНИМАНИЕ: Содержит формамида. Уплотнение пластины с прозрачной клейкой пленкой и встряхните планшет при 1800 оборотах в минуту в течение 5 мин. Повторить в общей сложности двух стирок. Убедитесь в том, чтобы удалить все LNW1 после второй стирки.
- Добавить 45мкл LNW1 для каждого образца скважины.
- Удалите LNP из магнитного стенда и, используя пипетку многоканальный, добавить 30 мкл 0,1 N NaOH (менее чем за неделю старой) в каждую лунку для элюирования образца. Уплотнение пластины с прозрачной клейкой пленкой и встряхните планшет при 1800 оборотах в минуту в течение 5 мин.
- В течение 5 мин элюирования, добавляют 30 мкл библиотеки Нормализация хранения буфера 1 (LNS1) в каждую лунку, чтобы использовать в новой пластинке под названием Запоминающее устройство пластина (ПСР).
- Поместите LNP на магнитной подставке. После того, как супернатант освобождалась, перенесите 30 мкл вымывание к LNS1 в ПМГ. Изменение советы между образцами, чтобы избежать перекрестного загрязнения.
- Уплотнение пластины с клейкой пленкой и центрифуге при 1000 мкг в течение не менее 30 сек.
- Добавить 51; л каждого образца, чтобы быть секвенировали с меченым Pooled ампликоне Library (PAL) 1,5 мл трубки. Вихре PAL смешивать и кратко спина в микроцентрифуге.
- В зависимости от того, что секвенирование химии, используемой (V2 или V3) добавить 4 - 10 мкл PAL до 590 - 596 мкл HT1. Как правило, добавляют 5,8 мкл PAL 595 мкл HT1 для химии V2 и 8,5 мкл PAL до 592 мкл HT1 для химии V3. Добавьте эту трубку как разбавленный Amplicon библиотека (ДАЛ) трубки.
- Вихре DAL и кратко спина в микроцентрифуге. Выдержите DAL при 96 ° С в течение 2 мин. Инвертировать Dal трубки 3 раза и поставить DAL на льду в течение не менее 5 минут, при подготовке секвенсор для секвенирования. После 5 мин ДАЛ готова к загрузке.
- Если закончил с ПМГ, запечатать пластину с пленкой клейкой алюминиевой фольгой и пометьте его с датой и пластины ID. Храните запечатанный ПМГ и PAL при температуре от -15 до -25 ° C.

Рис . 3: Библиотека Prep QC Гель Пример Зеленая линия над нижними полосами, чтобы указать нижний маркер и фиолетовую линию над верхними полос , чтобы указать более высокий маркер. Все работало хорошо для полос движения H1, A2, B2, C2, E2 и G2. Библиотека Prep не работает оптимальным образом, для полос движения D2, F2 и H2, но результаты все равно будут получены, они просто не могут иметь достаточный охват. Для A3, библиотека Prep едва работала, и, скорее всего, этот образец ДНК не была адекватной для анализа. Нижние полосы выше нижнего маркера являются неиспользованные праймеры, потому что аликвоту берется непосредственно из PCRed хорошо. NTC образец должен иметь только неиспользованную полосу праймера, и ничего больше. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
5. Секвенирование
- Обеспечить правильное SampleSheet.csv было сделано для бега. См Дополнительный рисунок 1 для примера.
- Тщательно промыть и высушить проточную кювету и добавляют 600 мкл ДАЛ в картридж талой реагента.
- Подготовьте секвенсор для секвенирования, следуя инструкциям на экране.
Анализ 6. Данные
- Выполнение трубопровода биоинформатики. Использовать в доме, специально созданных биоинформационного трубопровода для выявления мутаций, вставки, делеции и амплификациях 18.
- После того, как трубопровод будет завершен, проверьте файлы журналов на наличие ошибок / предупреждения, так как каких-либо значительных ошибок / предупреждений помощи в КК обработки трубопровода.
- Анализ статистики прогона (таблица 3) , чтобы обеспечить упорядоченному библиотеку прошло в лаборатории определяется QC метрик (Рисунок 4). Вручную просмотреть каждый вариант при просмотре .bam файлов в средстве просмотра геномных данных (например, интегративной Genomics ViКувшин 16 (ВНА)).
Примечание: только варианты в пределах одобренного диапазона аллель частот и выше минимальной глубины охвата после фильтрации качества сообщаются (с использованием HGVS) номенклатура (Human Genome вариацию общества). Для окончательной отчетности, каждый exonic вариант был разделить на: патогенные, вероятно заболевания, связанного, вариант неизвестного значения (ВУС), вероятно, доброкачественная, и доброкачественной. Все варианты выше критериев отчетности 5% частоты аллели, за исключением тех считаются доброкачественных вариантов и синонимичных изменений, не сообщалось.
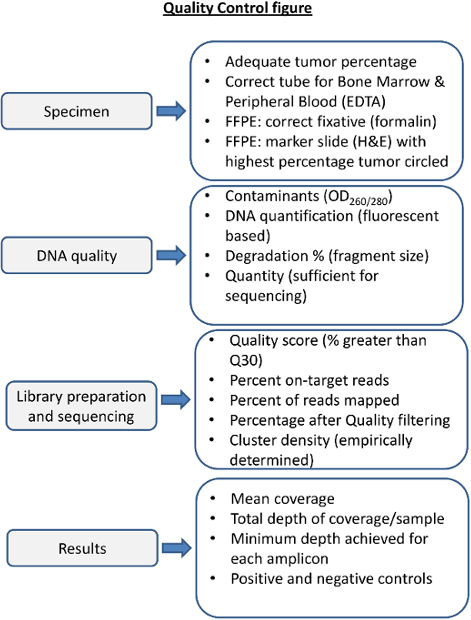
Рисунок 4. Обзор качества шагов управления для NGS. Контроль качества каждого шага в процессе необходимо для обеспечения последовательности будет давать результаты такие , что до и после метрики секвенирования рассматриваются. Надлежащее лечение образца имеет важное значение для высокого качества ДНК. Кровь икостный мозг в ненадлежащих фиксаторов может дать низкое качество ДНК. Неуместный фиксация твердых образцов опухоли может привести к снижению ДНК (например, фиксация в B5). Качество ДНК должны быть оценены на белок и загрязнения РНК с помощью спектрофотометрических средств, и точно оцениваются на предмет количества и целостности ДНК. Метрики секвенирования должны быть определены эмпирически в лаборатории секвенирования и последующим для каждой реакции секвенирования и каждого образца. До представления результатов секвенирования для каждого образца должны быть оценены на предмет охвата, глубины и адекватного выполнения положительных и отрицательных контролей. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
Результаты
Случай 1 - Гем-NGS панель
ДНК, выделенную из лейкозных периферической крови было достаточного количества и качества (176 нг / мкл) для панели Гем-NGS. Общая средняя глубина охвата была 4,933x (выше минимальной средней глубины охвата 1,000x). Дополнительные статистические данные прогона в таблице 3. Из 8 областей ниже покрытия 250x, 3 были из - за неправильной обрезке праймеров (т.е. последовательность праймера не был должным образом удален из - за ошибок последовательности), 1 был известным артефактом анализа, а остальные 4 были частичные области экзонов различных генов, не имеющих отчетными вариантов. Хотя наш клинический протокол включает только отчетность варианты охвачены по меньшей мере 250 считывает все данные по крайней мере 100 чтений будут импортированы в базу данных для обзора.
Обработка данных Fром трубопровода биоинформатики обнаружены три отчетных, связанных с заболеванием мутации; миссенс мутация в FLT3, мутация Миссенс в IDH2, и мутации в рамки считывания NPM1. В exonic варианты с их аллельных частот приведены в таблице 4. Общей мутации в FLT3 является -Internal тандемной дупликации FLT3 (ОИТ) , который не вызывается автоматически нашим трубопроводом и требует визуального осмотра экзон 14. Визуальный осмотр экзона 14 из FLT3 не показали вставки или дублирования в представленных образцов. FLT3 I836del был на 1% частоты аллели и не была включена в окончательный доклад , потому что он упал ниже минимальной частоты одобренного аллеля 5%. Эта мутация не на той же молекуле ДНК, что и изменение FLT3 D835Y (то есть, наблюдается в том же регионе ампликонов, но не в "цис" в любом из последовательности чтения) и наблюдалось только созданный вручную .bam Fiле при проверке изменения p.D835Y. Нижние частоты аллели двух Flt3 мутаций предлагает эти мутации могут представлять гетерогенность и / или клональной эволюции; однако, разница может быть из-за проведения анализа для того ампликона или одного нуклеотидного полиморфизма (SNP) вблизи или перекрывающихся последовательность праймера, что повлияло на усиление этого аллеля.
Результаты панели Гем-NGS для случая 1 с ОМЛ выявлены мутации в FLT3, IDH2 и NPM1, три часто мутировавшие гены в ОМЛ. Мутации в FLT3 наблюдаются в ~ 25% взрослых пациентов с ОМЛ (КОСМИК базы данных 17) и являются либо внутренний тандем дупликации (СДМТ) или миссенс мутации в домене тирозинкиназы. В FLT3-СДМТ являются более распространенной мутацией и связаны с плохим ответом на стандартной химиотерапии, в то время как прогностическое значение FLT3 киназы Domain точечные мутации, как показано в этом пациента AML, имеет неопределенное влияние на прогноз 14. Изоцитратдегидрогеназа 2 (НАДФ +), митохондриальные (IDH2) представляет собой ген , который кодирует эпигенетическую модификатор , который обычно мутировавший в AML. Мутации в эпигенетических модификаторов являются довольно распространенным явлением в борьбе с отмыванием денег, с мутациями в IDH1 и Dnmt3a , представляющих другие мутации в этом классе генов , которые приводят к нарушению регуляции генов. Мутации в гене нуклеофозмина (NPM1) являются одним из наиболее распространенных приобретенных мутаций в борьбе с отмыванием денег и , как правило , считается хорошим прогностическим фактором (в отсутствие FLT3 - ОИТ).
Со-мутации NPM1 и IDH2 были описаны в литературе как благоприятный прогностический индикатор 5, с 3-летней общей выживаемости 89%. Это представляет собой существенное увеличение выживаемости по сравнению с общей 3-летней выживаемости вдикого типа NPM1 и IDH2 на 31%. Например, стандартной практики ухода включает в себя анализ мутаций для НПМ 1 и FLT3-ITD мутации. В этом случае, обнаружение только мутации NPM1 бы не в состоянии надлежащим образом наслаиваются пациента к риску, так как вторичные мутации могут быть благоприятны (например, IDH2) или неблагоприятное (например, TET2), что снижает доверие к облегчению трансплантации костного мозга.
Случай 2 - Твердо-NGS панель
ДНК, выделенную из ткани FFPE было достаточного количества и качества для твердого тела NGS панели, с концентрацией 252 нг / мкл и только 4% ДНК ниже 1000 пар оснований (п.о.). После анализа данных средняя глубина охвата была 9167 читает (значительно выше нашей минимальной глубины среза 1000 прочтений) без каких-либо областей ниже 250 глубины чтения. Дополнительные QC метрики шошп в таблице 3.
Данные , обработанные по трубопроводу биоинформатики обнаружены две болезни мутации , связанные с : введение в рамке считывания в экзоне 20 ERBB2 (HER2 / Neu) и миссенс - мутации в TP53. Все exonic варианты с их аллельных частот представлены в таблице 5. Вставка в ERBB2 на самом деле представляет собой тандем дублированный (HER2 / NEU) последовательности, как это отражено в номенклатуре. Идентификация и подтверждение требуемой вставки в рамке ручной обзор секвенирования читает через ВНА. Обнаружение мутаций частот более чем на 50%, как видно, как TP53 и HER2 / Neu мутации наводит на мысль о потере гетерозиготности (LOH) события (обсуждение см).
Твердокристаллическую NGS результаты панель для случая 2 с аденокарциномы легких обнаруженных мутаций яп ERBB2 (HER2 / Neu) и TP53, два гена обычно не тестировали на как часть текущего стандарта оказания медицинской помощи для больных раком легкого. HER2 / Neu кодирует тирозинкиназы рецептора подобную другому часто видоизмененный ген при раке легкого, EGFR , Активация HER2 / Neu экзон 20 Вставки наблюдаются в 2 - 4% от аденокарциномы легких, составляют большинство HER2 / Neu мутаций , наблюдаемых при раке легкого, и , как правило , рассматривается в опухолях без мутаций в других генах драйверов , таких как EGFR и ALK 12 , Есть несколько линий доказательств , свидетельствующих о возможности для различных вариантов лечения для пациентов с активацией HER2 / Neu вставки, в том числе частичный ответ на комбинированной терапии с HER2 / Neu и ингибиторы MTOR 13 и значительного контроля заболеваний с моноклональное антитело трастузумаб в сочетании с химиотерапией 15. Обнаружив изменение TP53 не жуткоMMON при раке, но в настоящее время нет выполнимые терапии.
| Дело 1 | Случай 2 | |
| Общее Запуск двигателя Читает | 2215926 | 2129110 |
| Процент Считывает Mapping | 98,42% | 98,29% |
| Процент Считывает на Target | 99,01% | 97,29% |
| Процент Читает на цель после фильтра | 97,60% | 95.45% |
| Процент Годно Читает | 94,87% | 91,79% |
| Процент Основ выше 250x Покрытие | 98,40% | 100% |
| Процент базамиБове 1000X Покрытие | 95,90% | 99,70% |
| Покрытие Ниже 250X - Amplicon Номер | 8 | 0 |
Таблица 3:. Секвенирование Run QC Метрики Это краткое изложение наиболее важных статистических данных пробега, не считая среднего покрытия, которые используются для анализа данных , чтобы определить , является ли образец библиотеки Prep прошло QC. Весь этот процесс является успешным, если все проценты указаны выше 90%, но это возможно, с выносом SW1 или UB1 на стадии ФПУ промывки или праймера перекрестных помех, чтобы иметь более низкую 'Проценты по Target' в диапазоне 80 - 90%. Если 'Процент Подключенные' слишком низко, что указывало бы на заражение бактериями или другой ДНК, так как все образцы должны привести к человеку. Когда любой из этих спецификаций ниже 80%, то образец попадает дальнейшего обзора, чтобы помочь определить, как рroceed и улучшить процесс.
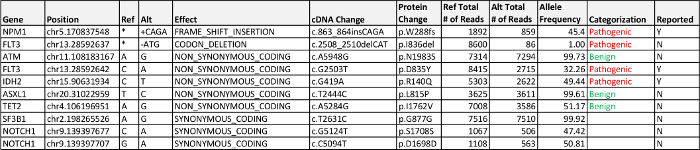
Таблица 4:.. Случай 1 Результаты Определен патогенные, заболевание , связанное, вариант неизвестного значения (ВУС), и , вероятно , доброкачественные exonic варианты выше критериев отчетности 5% частоты аллеля перечислены Пожалуйста , нажмите здесь , чтобы загрузить эту таблицу.
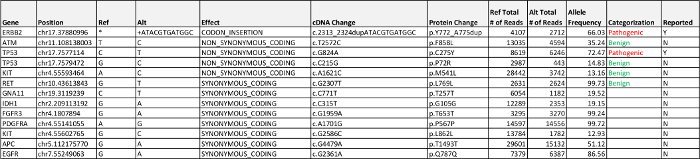
Таблица 5:.. Случай 2 Результаты Определен патогенные, заболевание , связанное, вариант неизвестного значения (ВУС), и , вероятно , доброкачественные exonic варианты выше критериев отчетности 5% частоты аллеля перечислены Пожалуйста , нажмите здесь , чтобы загрузить эту таблицу,
Справочная Рисунок 1: пример ампликона на основе SampleSheet.csv. Этот лист передает секвенсера , что химия для запуска (в данном случае ампликона), какой рабочий процесс (например, GenerateFastq), то , что применение и анализ (например, FastqOnly), сколько оснований (или читает) к последовательности (в данном случае 186 б.п. х 186 пар оснований), и, наконец, что образцы связаны с определенными индексами (в данном случае двойной индексации). Части, которые выделены желтым цветом может быть изменен на то , что экспериментатор хочет, но в этом случае лаборатория использует эти параметры. Пожалуйста , нажмите здесь , чтобы загрузить эту цифру.
Обсуждение
Поскольку два NGS испытания, описанные в этой рукописи предлагаются клинически, наиболее важным практическим фактором является контроль качества. В частности, близко внимание должно быть уделено качеству и количеству извлеченной ДНК. Это особенно важно для образцов FFPE, которые часто сильно деградировали с выходом переменной ДНК. Метод осаждения изопропанолом был разработан с целью максимального выхода ДНК из образцов FFPE, как были найдены способы столбцов на основе иногда приводят к стрижке ДНК с ограниченными объемами элюирования. Таким образом, большую часть времени, когда образец дает слишком низкую концентрацию или слишком деградировали для анализа, то, скорее всего, из-за размера ткани, типу или фиксации, а не процесс экстракции. Для получения образцов крови / костного мозга, если есть сбой в добычу полезных ископаемых, как правило , из - за образце hemodilute (т.е., не имея достаточного количества белых кровяных или опухолевых клеток в этом розыгрыше) или химио абляции.
. Нт "> Во время проверки, отсечек на приемлемость качества и количества ДНК должны быть установлены Рекомендуемый вход 100 - 250 нг часто используется в анализе, однако, если качество ДНК хорошо, то более низкие входные суммы могут быть успешными. Кроме того, если качество ДНК бедна (то есть, количество укрупнения ДНК составляет менее 100 - 250 нг) , то более высокие количества входных может улучшить качество результатов секвенирования (так как количество укрупнения ДНК достигнет заданной входной) . Метрики для качества и количества ДНК должны быть применены к каждому образцу , прежде чем перейти ДНК в подготовке к библиотеке. Эти образцы в "серой зоне" (смотри рисунок 2) должны выполняться по усмотрению директора лаборатории или исполняющему. в настоящее время лучший способ предсказать, если ДНК не будет хорошо работать во время последовательности является выполнение КПЦР на основе анализа на который позволяет для количественной оценки и оценки качества входных ДНК. Этот подход устраняет bioavailability разного размера фрагментов в образце, путем усиления различных размеров фрагментов (например, 100 б.п., 150 б.п., 200 б.п. и 300 б.п.) и сравнения доходности.В настоящее время подготовка библиотека включает в себя большое количество ручных шагов, где неверный шаг в одном из нескольких стыках может привести к библиотеке либо выходят из строя или быть низкого качества. Анализ Микрожидкостных гель является единственным шагом QC для проверки вопроса библиотека приготовительную перед тем секвенирования. Соответственно, существует несколько важных шагов, где дополнительные осознанность могут увеличить вероятность успешной реакции. Крайне важно, чтобы гарантировать правильный образец и олигонуклеотидный пула используются для каждого образца. Обеспечение и правильной записи, что каждый образец содержит один из 96 уникальных комбинаций пар двойственных индексированные праймеров ПЦР уменьшает шанс перепутать образца. Кроме того, важно обеспечить фильтрационную планшету (FPU) истощает правильно; если он не сливает воду должным образом, это может привести к EXTEnsion-лигирование этап подготовки библиотеки для выполнения неоптимально и привести к ухудшению качества данных секвенирования. После того, как библиотеки QC, важно, чтобы гарантировать, что LNB1 гранулы полностью ресуспендировали и что раствор LNB1 / LNA1 хорошо перемешиваются, прежде чем добавить к образцам, как концентрация этой смеси используется для определения молярность библиотеки. И, наконец, если этап шарик элюирование приводит к неоптимальным количеством библиотеки элюирование от бусинки это уменьшит плотность кластеров и, возможно, привести к тому, чтобы библиотека не получить адекватную средний охват. С другой стороны, избыток библиотеки приведет к более низкое качество чтения. Поэтому, важно быть последовательным в бисерной основе нормализации шага для обеспечения оптимальной скапливание и кластеризацию библиотек на секвенсор.
В дополнение к подготовке библиотеки, она имеет решающее значение для проверки биоинформатики трубопровода, который будет производить точные вызовы мутации из сырья, де-уплотненный fastq файлов. Выборпользовательское решение может занять много времени, поскольку есть много с открытым исходным кодом и коммерчески доступные каппы, вариант вызывающих абонентов, а также программные пакеты NGS, которые можно было бы просеивать. Пользовательские алгоритмы должны быть разработаны, чтобы извлечь необходимые статистические данные по эффективности, выявить уникальные повторяющиеся мутации, которые ускользали инструменты наиболее открытым исходным кодом, а также определить статус числа копий по каждому из локусов. Во время процесса валидации биоинформатики трубопровода, важно определить отчетных отсечек для вариантов , которые соответствуют или превосходят как минимальную глубину покрытия после фильтрации качества (например, минимум 250 прочтений) и частоту минимального аллель (например, 4 %). С этого мультиплексной анализа ампликонов на основе, важно , чтобы определить , минимальная средняя глубина охвата (например, 1,000x) , что библиотека должна достичь , чтобы иметь возможность получить самую низкую выполняя ампликона на минимальную глубину читает. Кроме того, мультиплексированный характер анализа делает CAUSE от целевых эффектов и эти «артефакты» должны быть открыты и полностью проверены перед запуском. Другим важным ограничением для описанного анализа потребность в образцах, чтобы содержать более чем 10% опухолей с целью достижения проверенную минимальной частоты аллеля.
Обнаружение низкой частоты, 1%, Flt3 вставках доказательства того, что руководство обзор по - прежнему желательно в этом процессе. Даже при частоте аллеля отсечке 5%, некоторые важные мутации, возможно, пропустили и, таким образом, ручной обзор будет иметь важное значение для выяснения этих вариантов. Для FLT3-СДМТ, визуальный осмотр экзона 14 выполняется для всех больных ОМЛ , чтобы обеспечить низкий уровень или большой вставки / дублирования не остается незамеченным. Кроме того, к HER2 экзон 20 Вставки, которые обычно рядом с часть последовательности праймера, требуют вмешательства. Несмотря на наличие надежного трубопровода биоинформатики, некоторые варианты могли бы пойти упускать из виду что только природа с трудом вырезатьвыкл для большинства статистика упоминалось выше. Лучше биоинформатики было бы необходимо, чтобы помочь решить эту проблему, как будет лучше библиотека методологии подготовки и / или секвенирования, потому что это более выгодно иметь хорошие качественные данные даже при более низких обрезаний, которые содержат меньше артефактов и ложных срабатываний.
Обнаружение и интерпретация частот аллелей может быть затруднено из-за трудностей в определении процентного опухоли и смещения усиления некоторых областей генома. Кроме того, частота аллели более 50% могут быть обнаружены, как это наблюдается в случае 2. Это интерпретируется как потеря гетерозиготности (LOH) событий, либо из-за потери нормального аллеля, что приводит к очевидному увеличению мутанта читает, А усиление мутантной аллели (например, 2 мутанта и одна нормальная копия) или другие механизмы. Эти механизмы могут быть выяснены путем использования массива сравнительную геномную гибридизацию (aCGH 19) и / или SNP массив генотипирования. 20.
Существующие методики целевого обогащения полагаются на полный рабочий день процедур либо неэффективного гибридного захвата или уплотненных методов ПЦР, которые приводят к необходимости более секвенирования охвата одного образца и более от целевой последовательности читает. Дополнительные приложения для NGS молекулярной онкологии ожидается в ближайшем будущем будет включать в себя более простые методы библиотеки подготовки , которые могут быть полностью автоматизированы и быть в состоянии обрабатывать образцы с очень низким количеством входных ДНК (т.е. менее 1 нг), а также образцы с сильно деградировали ДНК. Для решения этих проблем, большинство методов предположительно будет на основе ПЦР, либо будучи многоступенчатым подходом ПЦР или массово-параллельной singleplex ПЦР подход. Кроме того, молекулярная Barcoding отдельных ампликонов было показано, значительно уменьшить фоновый шум и секвенирования позволит испытания образцов с более низкой долей опухолевых клеток для достижения более низких частот аллеля и двигаться в направлении захвата circulatИНГ опухолевых клеток.
Обнаружение связанных с заболеванием мутации в образцах рака был стандартом лечения в течение многих десятилетий. Исторически сложилось так, гены часто испытания последовательно, один ген / экзон в то время, с идентификацией мутации, приводящей к концу тестовой последовательности. Появление NGS позволило менее предвзятый подход к секвенирования нескольких генов, связанных с большим количеством раковых образований параллельно ведущих к идентификации нескольких мутаций, которые связаны с неоплазии. Клиническая полезность NGS для выявления соматических мутаций при раке становится все более очевидным. Действительно, NGS на основе анализа образцов опухолей представляет собой новую парадигму, которая бросает вызов традиционной тестирования отдельных генов, но клиническая полезность очень ясна. Клинические лаборатории сегодня имеют прекрасную возможность Wed тщательной проверки метода и интерпретации тестового с применением этой мощной технологии.
Раскрытие информации
Авторы не имеют ничего раскрывать.
Благодарности
Авторы хотели бы поблагодарить за помощь Дэниела Wild для чтения рукописи и помощь в производстве.
Материалы
| Name | Company | Catalog Number | Comments |
| Genomic DNA ScreenTape | Agilent Technology | 5067-5365 | |
| Genomic DNA Reagents | Agilent Technology | 5067-5366 | |
| High Sensitivity D1000 ScreenTape | Agilent Technology | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent Technology | 5067-5585 | |
| TapeStation 2200 | Agilent Technology | G2965A | |
| TapeStation Analysis Software | Agilent Technology | A.01.04 or higher | |
| 96-well Tube Storage Racks | Any Vendor | ||
| 15/50 ml Tube Rack | Any Vendor | ||
| 96-well Plate Rack | Any Vendor | ||
| Pipette, single-channel, 0.5–2.5 μl | Any Vendor | ||
| Pipette, single-channel, 1–10 μl | Any Vendor | ||
| Pipette, single-channel, 2–20 μl | Any Vendor | ||
| Pipette, single-channel, 10–100 μl | Any Vendor | ||
| Pipette, single-channel, 20–200 μl | Any Vendor | ||
| Pipette, single-channel, 100–1,000 μl | Any Vendor | ||
| Serological Pipettor | Any Vendor | ||
| Vortexer | Any Vendor | ||
| Ice bucket | Any Vendor | ||
| Microcentrifuge (for tubes and strip tubes) | Any Vendor | ||
| Freezer, -20 °C | Any Vendor | ||
| 4 °C Refrigerator | Any Vendor | ||
| Water or Bead Bath | Any Vendor | ||
| Incubator (37 °C) | Any Vendor | ||
| Serological Pipettes, 1 ml | Any Vendor | ||
| Serological Pipettes, 5 ml | Any Vendor | ||
| Serological Pipettes, 10 ml | Any Vendor | ||
| Serological Pipettes, 25 ml | Any Vendor | ||
| Gloves | Any Vendor | ||
| Razor Blades/Scaples | Any Vendor | ||
| KimWipes | Any Vendor | ||
| 15 ml Conical Tube | Any Vendor | ||
| 50 ml Conical Tube | Any Vendor | ||
| Paper Towels | Any Vendor | ||
| 200 proof Ethanol | Any Vendor | Store in Flammable Cabinet | |
| 2-Propanol (Isopropanol) | Any Vendor | Store in Flammable Cabinet | |
| 25 ml Reservoirs | Any Vendor | ||
| 10 N NaOH | Any Vendor | ||
| Pipette, 8-channel, 1 – 10 μl | Any Vendor | ||
| Pipette, 8-channel, 10 – 100 μl | Any Vendor | ||
| Pipette, 8-channel, 20 – 300 μl | Any Vendor | ||
| Ice Bucket | Any Vendor | ||
| Water Squirt Bottle | Any Vendor | ||
| Alcohol Squirt Bottle | Any Vendor | ||
| Lens Cleaning Paper | Any Vendor | ||
| Plates, 96-well PCR, Semi-Skirted | Any Vendor | ||
| Tube strips, 8-well, 0.2 ml | Any Vendor | ||
| Agencourt AMPure XP Beads | Beckman Coulter | A63881 | |
| BioShake IQ or 3000-T elm | Bulldog Bio/Q.Instruments | 1808-0506/ 1808-0517 | |
| DropPlate96 S - LabChipDS | Caliper | 128876 | |
| DropPlate96 D - LabChipDS | Caliper | 132848 | |
| DropSense96 | Caliper (Trinean) | ||
| DropQuant Software | Caliper (Trinean) | ||
| Plate Sealing Film | Denville | B1212-5S | |
| Aluminum Seal Foil | Denville | B1212-6S | |
| Nuclease-Free, Pure Water System | EMD Millipore | ||
| 5424 centrifuge | Eppendorf | 22621408 | |
| 5804R centrifuge | Eppendorf | 22623508 | Both 15 ml tube and plate rotators, preferably a centrifuge that can go up to 2,500 x g. |
| Safe-Lock Tube 1.5 ml, Natural | Eppendorf | 22431021 | |
| 5 ml Tube, DNA LoBind Tube | Eppendorf | 30108310 | |
| 5430R Centrifuge | Eppendorf | 022620645 | Any plate rotator centrifuge will work |
| Hybex Microsample Incubator | Fisher Scientific | 1057-30-0 | |
| Hybex 0.2 ml Tube Block | Fisher Scientific | 1057-31-0 | |
| TruSeq Amplicon – Cancer Panel | Illumina | FC-130-1008 | 96 reactions |
| TruSeq Custom Amplicon | Illumina | PE-940-1011 | 96 reactions |
| TruSeq Custom Amplicon Index Kit | Illumina | FC-130-1003 | 96 Indices, 384 Samples |
| MiSeq Reagent Kit v3, 500 Cycles | Illumina | MS-102-3003 | |
| MiSeq Reagent Kit v2, 300 Cycles | Illumina | MS-102-2002 | |
| MiSeq Reagent Kit v2, 500 Cycles | Illumina | MS-102-2003 | |
| Experiment Manager | Illumina | 1.3 or higher | |
| MiSeq Reporter | Illumina | 2.0 or higher | |
| Sequencing Analysis Viewer | Illumina | 1.8 or higher | |
| TruSeq Index Plate Fixture and Collar Kit | Illumina | FC-130-1007 | |
| MiSeq v2 | Illumina | SY-410-1003 | |
| TruSeq Custom Amplicon Filter Plate | Illumina | FC-130-1006 | |
| Index Adapter Replacement Caps | Illumina | 11294657 | |
| Qubit 2.0 | Invitrogen | Q32866 | |
| Qubit 0.5 ml Tubes | Invitrogen | Q32856 | |
| Qubit dsDNA Broad Range Assay Kit | Invitrogen | Q32853 | |
| DynaMa6-96 Magnetic Stand, Side Skirted | Invitrogen | 120.27 | |
| GeneAmp PCR System 9700 (gold/silver block) | Life Technologies | N8050200 | |
| Gentra Puregene Blood Kit | Qiagen | 158489 | |
| Deparaffinization Solution (16 ml) | Qiagen | 19093 | |
| Buffer ATL (4 x 50ml) | Qiagen | 939011 | |
| Protein Precipitation Solution (50 ml) | Qiagen | 158910 | |
| DNA Hydration Solution (100 ml) | Qiagen | 158914 | |
| Glycogen Solution (500 μl) | Qiagen | 158930 | |
| Qiagen Proteinase K | Qiagen | 19133 | |
| Rnase (5 ml) | Qiagen | 158924 | |
| Nuclease-Free Water (10 x 50 ml) | Qiagen | 129114 | |
| Pestles | USA Scientific | 1415-5390 | |
| TipOne RPT 10 µl elongated filter pipet tips in sterilized racks, 10 racks of 96 tips (960 tips). | USA Scientific | 1180-3810 | |
| TipOne RPT 100 µl natural, beveled filter pipet tips in sterilized racks, 10 racks of 96 tips (960 tips) | USA Scientific | 1180-1840 | |
| TipOne RPT 200 μl natural, beveled filter pipet tips in racks, sterilized racks, 10 racks of 96 tips (960 tips) | USA Scientific | 1180-8810 | |
| TipOne RPT 20 μl natural, beveled filter pipet tips in racks, sterilized racks, 10 racks of 96 tips (960 tips) | USA Scientific | 1180-1810 | |
| TipOne RPT 1,000 μl natural, graduated XL filter pipet tips in | USA Scientific | 1182-1830 |
Ссылки
- Gerlinger, M., et al. Intratumor heterogeneity and branched evolution revealed by multiregion sequencing. N Engl J Med. 366 (10), 883-892 (2012).
- Campbell, P. J., et al. The patterns and dynamics of genomic instability in metastatic pancreatic cancer. Nature. 467 (7319), 1109-1113 (2010).
- Ding, L., et al. Clonal evolution in relapsed acute myeloid leukaemia revealed by whole-genome sequencing. Nature. 481 (7382), 506-509 (2012).
- Frampton, G. M., et al. Development and validation of a clinical cancer genomic profiling test based on massively parallel DNA sequencing. Nature Biotechnol. 31 (11), 1023-1031 (2013).
- Patel, J. P., et al. Prognostic relevance of integrated genetic profiling in acute myeloid leukemia. N Engl J Med. 366 (12), 1079-1089 (2012).
- Forbes, S. A., et al. COSMIC (the Catalogue of Somatic Mutations in Cancer ): a resource to investigate acquired mutations in human cancer. Nucleic Acids Res. 38 (Database Issue), 652-657 (2010).
- Shih, A. H., Abdel-wahab, O., Patel, J. P., Levine, R. L. The role of mutations in epigenetic regulators in myeloid malignancies. Nat Rev Cancer. 12 (9), 599-612 (2012).
- Liersch, R., Müller-Tidow, C., Berdel, W. E., Krug, U. Prognostic factors for acute myeloid leukaemia in adults - biological significance and clinical use. Br J Haematol. 165 (1), 17-38 (2014).
- Bacher, U., Schnittger, S., Haferlach, T. Molecular genetics in acute myeloid leukemia. Curr Opin Oncol. 22 (6), 646-655 (2010).
- Subramanian, J., Govindan, R. Lung cancer in "Never-smokers": a unique entity. Oncology (Williston Park). 24 (1), 29-35 (2010).
- Sakashita, S., Sakashita, M., Tsao, M. S. Genes and pathology of non-small cell lung carcinoma. Semin Oncol. 41 (1), 28-39 (2014).
- Arcila, M. E., Chaft, J. E., Nafa, K. Prevalence clinicopathologic associations, and molecular spectrum of ERBB2 (HER2) tyrosine kinase mutations in lung adenocarcinomas. Clin Cancer Res. 18 (18), (2012).
- Gandhi, L., et al. Phase I study of neratinib in combination with temsirolimus in patients with human epidermal growth factor receptor 2-dependent and other solid tumors. J Clin Oncol. 32 (2), 68-75 (2014).
- Sheikhha, M. H., Awan, A., Tobal, K., Liu Yin, J. A. Prognostic significance of FLT3 ITD and D835 mutations in AML patients. Hematol J. 4 (1), 41-46 (2003).
- Mazières, J., et al. Lung cancer that harbors an HER2 mutation epidemiologic characteristics and therapeutic perspectives. J Clin Oncol. 31 (16), 1-8 (2014).
- Robinson, J. T., et al. Integrative Genomics Viewer. Nat Biotechnol. 29 (1), 495-500 (2011).
- Forbes, S. A., et al. COSMIC: exploring the world's knowledge of somatic mutations in human cancer. Nucleic Acids Res. 43 (Database issue), D805-D811 (2014).
- Daber, R., Sukhadia, S., Morrissette, J. J. Understanding the limitations of next generation sequencing informatics, an approach to clinical pipeline validation using artificial data sets. Cancer Genetics. 206 (12), 441-448 (2013).
- Haraksingh, R. R., et al. Genome-Wide Mapping of Copy Number Variation in Humans: Comparative Analysis of High Resolution Array Platforms. PLoS ONE. 6 (11), e27859(2011).
- de Leeuw, N., et al. SNP Array Analysis in Constitutional and Cancer Genome Diagnostics - Copy Number Variants, Genotyping and Quality Control. Cytogenet Genome Res. 135 (3-4), 212-221 (2011).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены