Method Article
고체 및 액체 종양에서 실행 가능한 돌연변이의 검출을위한 차세대 시퀀싱
요약
This manuscript describes clinical protocols for two next-generation sequencing panels. One panel interrogates hematologic malignancies while the other panel targets genes commonly mutated in solid tumors. Molecular classification of driver mutations in human malignancies offers valuable prognostic and predictive information.
초록
As our understanding of the driver mutations necessary for initiation and progression of cancers improves, we gain critical information on how specific molecular profiles of a tumor may predict responsiveness to therapeutic agents or provide knowledge about prognosis. At our institution a tumor genotyping program was established as part of routine clinical care, screening both hematologic and solid tumors for a wide spectrum of mutations using two next-generation sequencing (NGS) panels: a custom, 33 gene hematological malignancies panel for use with peripheral blood and bone marrow, and a commercially produced solid tumor panel for use with formalin-fixed paraffin-embedded tissue that targets 47 genes commonly mutated in cancer. Our workflow includes a pathologist review of the biopsy to ensure there is adequate amount of tumor for the assay followed by customized DNA extraction is performed on the specimen. Quality control of the specimen includes steps for quantity, quality and integrity and only after the extracted DNA passes these metrics an amplicon library is generated and sequenced. The resulting data is analyzed through an in-house bioinformatics pipeline and the variants are reviewed and interpreted for pathogenicity. Here we provide a snapshot of the utility of each panel using two clinical cases to provide insight into how a well-designed NGS workflow can contribute to optimizing clinical outcomes.
서문
임상 종양학 표본의 차세대 시퀀싱 (NGS)는 타겟팅 유전 적 변화와 예측 / 예후 분자 마커를 식별의 중요성에 과학 문헌 포인트를 성장으로 더 널리 사용할 수있는 지난 몇 년 동안이되고있다. 다중 유전자 패널 분석과 질병의 진행 및 재발로 두 상피 1, 2, 혈액 3 악성 종양의 전체 엑솜 시퀀싱 연구는 종양 이질성의 개념과 클론 진화를 응고있다. 또한, 이러한 중합 효소 연쇄 반응 (PCR) 또는 생거 시퀀싱 같은 경쟁 기술과 달리, NGS 번의 시험 4의 모든 임상 적 암 유전자의 대부분의 게놈 변화를 검출 할 수있다.
처음 두 개의 임상 NGS 패널, 사용자 정의 혈액 학적 패널 (헴-NGS 패널)와 기성 FFPE 표본에 대한 암 패널 (솔리드 NGS 패널)를 출시 맞춤 진단을위한 센터 (참조 그림 1). 이 패널은 선택된 유전자의 임상 적 또는 높은 관심 영역을 커버; 모든 유전자 또는 엑손 완전히 덮여 없습니다. 증폭은 확장 및 결찰 다음 프로브 하이브리드 화에 의해 생성됩니다. 대상 지역은 추가 시퀀싱 풀링 할 96 샘플까지 허용, 보편적 인 듀얼 인덱스 프라이머와 PCR을 이용하여 증폭된다.
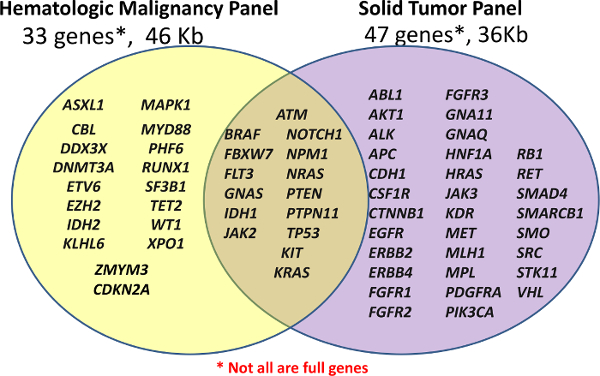
그림 1 :. 패널에서 해당 유전자의 목록 라이브러리 준비 사용하여 수행하거나 사용자 정의 혈액 학적 패널 (33) 유전자 또는 기성 앰플 리콘 암 패널 (47)의 유전자 (솔리드 NGS)의 (헴-NGS 패널). 유전자 또는 엑손의 모든 일부 증폭가 특정 핫스팟을 커버 할 수있다. 전체에 적용되지는 않습니다 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오. <가 />
헴-NGS 패널에 대한 내용은 여러 소스에서 유래하지만, 이전에 임상 적 유용성 5의 높은 수준을 보여주는 설명 급성 골수성 백혈병 (AML)에 돌연변이 16 유전자를 중심 하였다. 고체 NGS 패널은 상업적으로 (COSMIC) 데이터베이스 (6) 암에 체세포 돌연변이의 카탈로그에보고 된 암에서 일반적으로 돌연변이 유전자에 따라 대상 지역으로 생산된다.
몇 가지 주요 단계는 임상 NGS에 대한 전반적인 워크 플로우를 특징. 임상 테스트를 정렬 한 후, 병리학 종양 비율 및 샘플 볼륨에 대한 분석에 따라 시험편의 적절성을 결정한다. 우리 기관에서는 기술의 배경 시퀀싱 에러 레이트 ( "잡음")과 목표 접근의 효율성으로 인해 10 % 이상의 종양을 필요로한다. 조직 테스트에 적합한 경우, 게놈 DNA를 추출한다. 이 DNA 후 (복수의 품질 관리를 실시한다QC) 단계. DNA가 QC를 통과 할 경우, 앰플 리콘 라이브러리가 생성 순서가된다. 얻어진 데이터는 사내 정보학 파이프 라인을 통해 분석된다. 생물 정보학 분석에 따라, 변형 수동으로 검토하고 임상 보고서로 통합하기 전에 병원성에 대한 해석했다. 우리가 설명 아래 궁극적으로이 엄격한 워크 플로우를 통해 가서 두 가지 경우 임상 관리의 변화로 이끌어 냈다.
사례 1 - 급성 골수성 백혈병
환자 A로부터 골수 조직 검사는 성숙하지 않고, AML 진단했다. 유전 학적 연구는 골수 시편상에서 전송 및 정상 핵형 여성을 입증 하였다. 이 현재의 95 % 순환 폭발이었다, 그래서 말초 혈액 표본은 헴-NGS 패널의 맞춤 진단 테스트에 보내졌다.
급성 골수성 백혈병 (AML)은 백혈구의 골수 계통의 혈액 악성 종양이다. 검출재발 유전자 돌연변이 기전과 예후 7에서 중요하게 인식과 AML에서 유전자 변이의 예후 및 치료에 점점 더 중요 해지고있다. FLT3 내부 탠덤 중복 (ITDs에이) 덜 유리한 결과 8과 관련이있는 반면 NPM1과 CEBPA 돌연변이는 양호한 예후 위험과 관련이있다. 증거의 성장 몸은 AML 9 이들과 다른 돌연변이에 대한 병원성 역할을 지원합니다.
사례 2 - 폐 선암
환자 B에서 왼쪽 쇄골 대량의 조직 검사는 폐 선암을 보여주었습니다. 50 % 이상 종양 롤 / 컬은, 돌연변이가 대상으로 치료 적 개입의 존재 여부를 확인하기로 포르말린 고정 파라핀 포함 (FFPE) 림프절 질량에서 생검 재료는 게놈 테스트 (솔리드 NGS 패널)을 위해 보내졌다.
폐 cancER은 미국의 암 관련 사망의 주요 원인이며, 두 가지 유형의 비 - 소세포 폐암 (NSCLC) 및 소세포 폐암 (SCLC)로 분할된다. NSCLC는 또한 병변의 조직학에 기초 선암 또는 편평 세포 암종로 정의 될 수있다. 폐 선암종 흡연자와 비 흡연자 모두에서 가장 흔한 폐암 서브 타입이며, 비 흡연자 10 폐암의 가장 흔한 형태이다. 폐 선암의 분자 연구는 여러 종양 유전자 (11)에 돌연변이를 확인했다. 흡연자에서 식별 된 가장 일반적인 드라이버 돌연변이 KRAS 및 BRAF 돌연변이이다. 비 흡연자에서 가장 일반적인 돌연변이 EGFR의 돌연변이와 유전자 ALK, RET와 ROS1을 포함하는 재 배열입니다. 폐 종양 유전자 ERBB2에서의 프레임 삽입 엑손 20 (HER2 / NEU)으로 설명되었다. HE에서 가장 일반적인 이상R2 / NEU는 표적 치료 (: HER2 / NEU에 대한 인간화 단클론 항체 트라 스투 주맙)이 사용할 수있는 유방암에서이 궤적의 증폭이다. 이 관찰되는 HER2 / neu의 엑손 20 삽입 - 폐의 4 %는 12 (각각 네 라티 닙과 temsirolimus) 13 HER2 / neu의과 mTOR의 억제제와 병용 요법에 부분적 반응을 보였다 adenocarcimomas.
프로토콜
이 프로토콜은, 각각, 고체 및 액체 종양 유전자 프로파일 두 검증 기관 발전 시험의 돌출 단계를 포함한다. 실험실에서 수행 된 테스트는 1988의 임상 실험실 개선 개정 (CLIA)의 규정에 따라 행해진 다.
말초 혈액 또는 골수에서 1. DNA 추출
- 표 1을 기준으로 적용하려면 얼마나 많은 혈액이나 골수를 결정합니다.
| 샘플 / WBC | 양 혈액 1 ㎖로 처리 될 |
| 골수 | 250 μL |
| 혈액 WBC 12,000 - 50,000 | 1 ml의 |
| 혈액 WBC 50,000 - 100,000 | 500 μL |
| 혈액 WBC 100,000 - 200,000 | 200 μL |
| 혈액 WBC> 20 | 100 μL |
| * 혈액 WBC <12000은 혈액 2 ㎖을 위해 | |
표 1. 백혈구 수가 샘플들마다 다를 수 있기 때문에 혈액 / 골수 볼륨 차트를 사용하기 위해서는, 사용할 수있는 혈액의 체적을 지정하는 것이 곤란하다. 따라서, 분석에 사용하는 혈액의 양이 사전 분석을 시작으로 백혈구 (WBC)보고 결정되어야한다. 이하 혈액을 이용하지만, 여전히 본 셀의 수가 정상보다 크기 때문에 사용할 혈액의 부피는 1 ml의 사람이 감소 된 경우로 간주한다.
- 게놈 DNA를 분리하기 위해 상업적으로 이용 가능한 키트의 프로토콜을 따르십시오.
포르말린 고정 파라핀 포함 (FFPE) 조직 2. DNA 추출
- 에 기반병리학은 H & E 슬라이드에 동그라미 종양 영역은 가이드 H & E 슬라이드로 흠없는 슬라이드를 정렬 및 추출을위한 유사한 영역을 설명합니다. 매크로 절개를 들어, 프로세스를 하나의 표본 / 한 번에 슬라이드 환자의 집합입니다.
- 45 ° C 열 블록에 슬라이드 파라핀을 약간 녹아 가열한다. 조심스럽게 각각의 표본을 추출하는 새로운 메스를 사용하여 슬라이드에 표시되는 라인 내에서 조직을 긁어. 적절하게 표시 1.5 ML 튜브에 왁스 부스러기를 놓습니다. 긁어 왁스가 매우 정전과 튜브에서 이동할 수 있기 때문에주의해야합니다.
- (- 30 μm의 총 25) 매 5 μm의 5 여섯 부분에 대한 탈 파라핀 용액 320 μl를 추가합니다. 예를 들어, 10 μm의 롤 / 컬 3 부분을 포함하는 튜브를 처리하는 경우는 다음 320 μl를 사용하지만, 동일한 두께 5 섹션 얻었다이라면 640 μl를 사용한다.
- 적극적으로 최소 10 초 동안 수성을 수행 소용돌이미세 원심에 요 약 스핀 측면과 캡에서와 용액에 조직 / 왁스를 제거합니다. . 3 분 56 ° C에서 품어, 다음 5 RT 부화 - 10 분.
- 역전사 배양 후, 추가 탈 파라핀 솔루션의 모든 320 ㎕를 위해 ATL 버퍼 180 μl를 추가합니다. 각 시편에 대한 새로운 유봉을 사용하여 무균 미니 유봉을 사용하여 조직 10 번을 말하다. 매우 끈적 수있는 바와 같이, 유봉을 고집 어떠한 조직이없는 확인. 소용돌이 현탁액을 적극적으로 3 초 후 1 분 동안 최대 속도로 원심 분리기.
- 낮은 명확한 단계에 테 K의 10 μl를 추가합니다. 조직이 재현 탁 확인하기 위해 아래 피펫 팅에 의해 부드럽게 섞는다. 소용돌이하지 않습니다. 500 rpm으로 - 밤새 400에서 진탕 56 ° C에서 품어.
- 조직이 완전히 용해되면 다음 날 아침, (낮은 솔루션이 분명하면 이런 일이 발생했습니다) 확인합니다. 낮은 솔루션은 FO 최대 속도에서 3 초 원심 분리기에 대한 적극적 다음 소용돌이, 명확하지 않은 경우R 1 분. 테 K 10 μL 및 추가 30 56 ° C에서 부화 - - 추가 5 추가 60 분.
- 포름 알데히드 가교 역방향 있도록, 1 시간 동안 90 ℃에서 인큐베이션. 10 분 후, 간단히 액체를 통합하고, 각 튜브를 원심 분리 - 샘플 5, 실온으로 냉각시킨다.
- 레이블 1.5 ML 튜브로 낮은 명확한 상을 전송합니다. (다중 롤이 사용되는 경우와 같은 경우) 동일한 환자의 시료로부터 복수 튜브가있는 경우,이 시점에서 하나의 관으로 낮은 위상 재결합. 주 : 정제 과정을 방해하지 않아야 탈 파라핀 용액을 소량 전송하지만 다량 전달되면 위험이있다.
- 의 RNase 솔루션 2 μl를 추가합니다. 소용돌이 부드럽게 또는 마이크로 원심에서 25 배 빠른 스핀을 반전. 5 분 동안 실온에서 인큐베이션.
- 단백질 강수량 솔루션의 200 μl를 추가합니다. 하나 또는 두 개의 롤을 수행하는 경우, 200 μl를 사용합니다. 세 롤을 수행하는 경우한 번에, 다음 400 μl를 사용하는 s의. 적극적으로 높은 속도에서 30 초 동안 소용돌이 균일하게 용해 버퍼를 혼합합니다. 5 분 동안 얼음에 품어 또는 샘플은 시간까지 얼음에 머물 수 있습니다.
- 5 분 동안 5,000 XG에 원심 분리기. 침전 된 단백질은 꽉, 흰색 펠렛을 형성한다. 레이블 1.5 ML 튜브에 뜨는을 붓고 한 다음 최소 3 분 동안 얼음에 샘플을 품어. 3 분 동안 5,000 XG에 원심 분리기.
- 버퍼 ATL마다 180 ㎕의 2 프로판올 (이소프로판올) 200 μL 이전 표지 1.5 ㎖의 튜브에 첨가 추가 (2 ㎖의 튜브를 사용할 필요가있을 수 있음). 예를 들어, 일을하면 세 가지 롤 이소프로판올 600 μl를 추가합니다. 이전 이소프로판올에 추가 버퍼 ATL의 모든 180 ㎕를 위해 글리코겐 1 μl를 추가하고 혼합 튜브를 여러 번 반전.
- 조심스럽게 아이소 프로필 알코올 혼합물로 단백질 침전 단계에서 뜨는을 추가합니다. 부드럽게 적어도 50 회 반전 튜브를 섞는다.
- 최대 SP 원심 분리기3 분 EED. DNA를 튜브의 하단에 작은 흰색 펠렛으로 표시됩니다.
- 부어 또는 적합한 폐기물 관에 뜨는을 대기음. 분실 또는 다른 시료의 폐기물과 혼합되지 않도록 펠렛이 빠질 오는 경우 각 시험편에 대해 별도의 1.5 ml의 폐기물 튜브를 유지합니다. 종이 타월에 튜브를 드레인과 이소프로판올의 대부분이 제거되도록.
- 갓 70 % 에탄올 300 μl를 추가합니다. 펠렛을 씻어 부드럽게 여러 번 튜브를 반전. 보다 철저한 청소를 위해 빠질 제공되는 펠렛을 확인하려고합니다.
- 5 분 동안 최대 속도로 원심 분리기. 그리고 조심스럽게 에탄올을 제거합니다. 펠렛은 느슨하게, 천천히 붓거나 흡인와 펠렛을 볼 수 있습니다. 펠렛을 건드리지 않고, 관의 내부로부터 과량의 에탄올을 제거한다. 이상의 샘플을 건조하지 않도록주의하면서 15 분 - 5 건조 공기 샘플을 허용합니다.
- 전자에 DNA 수화 용액 100 μL - 25 사이에 추가DNA의 펠릿의 크기 및 조직의 개시 량에 기초 ACH 샘플. 소용돌이는 튜브를 적극적으로하고 잠깐의 microcentrifuge에 스핀. 완전히 DNA를 재수 65 ° C에서 1 시간 동안 인큐베이션.
3. 게놈 DNA 품질 관리
주 : DNA 품질 관리 (QC)를위한 3 개의 독립적 인 단계가 있습니다. 각 QC 단계가 수행되는 이유 자세한 설명은 표 2를 참조하십시오.
| 악기 | 결과 | 표시 | 이상적인 범위 |
| DropSense96 | A260 / A230 | 화학 오염 물질의 식별 (예 : 에탄올) | 1.50-2.2 |
| DropSense96 | A260 / A280 | 단백질 오염 물질의 식별 | 1.60-2.2 |
| DropSense96 | 집중 | DNA 정량 | > 1 NG / μL |
| TapeStation | DNA 얼룩 | DNA의 무결성의 결정 (예를 들어, 분해 / 추출 된 DNA의 단편화) | > 1,000 bp의 50 % |
| 큐 비트 2.0 | 집중 | 보다 정확한 DNA 정량 | > 1 NG / μL |
표 2 :. DNA QC 예상 결과는이 값의 모든 라이브러리 준비 단계로 진행하기 위해 샘플을 허용 실행하기 전에 고려됩니다.
- 제조사의 프로토콜에 따라, 작업 농도 검체 (NG / μL)을 얻었다 형광 분석기에서 추출한 DNA의 2 μL를 실행.
- 상기 샘플의 품질 (A260 / A230 및 A260 / A280 쥐를 확인하기 위해 UV / VIS 분광 광도계에 각 샘플의 2 μL - 1을 실행IOS) 제조자의 지시에 따라.
- FFPE 샘플의 경우 : 제조업체의 지침에 따라, DNA의 분해 / 분열을 평가하기 위해 미세 유체 겔 전기 영동 시스템에서 각 샘플 1 μl를 실행합니다. 예를 그림 2를 참조하십시오.
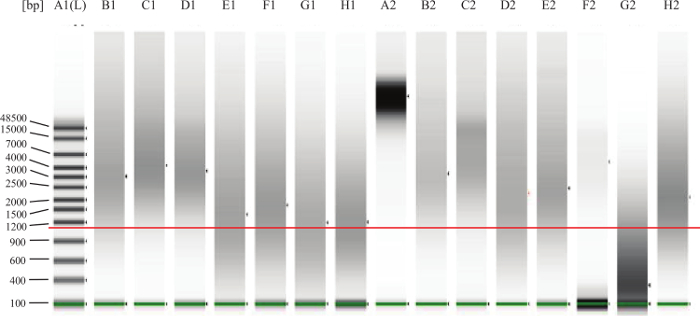
그림 2 :. 게놈 DNA QC 젤 예는 낮은 대역을 통해 녹색 선이 낮은 마커를 표시하는 것입니다. 추가 된-에서 빨간색 선은 약 1,000 염기쌍을 표시하는 것입니다. 하나의 신선한 TIS의 고소 (예를 들어, 말초 혈액 또는 골수)에서 기대하는 것처럼 레인 A2는 완전히 손상 DNA를 나타냅니다. 레인의 B1, C1, D1, B2, C2, E2 좋은, 그대로 FFPE의 DNA의 예입니다. 차선 F2의 DNA는 그대로,하지만 너무 낮은 농도로 나타납니다. 차선 G2의 DNA는 분해 또는 조각하고 분석에서 작동하지 않습니다. 레인 E1, F1, G1, H1, D2,H2는 분석이 잘 작동 할 수 있음을 의미하는 '회색 지대'에 빠진다 DNA를 표현하지만, DNA의 일부도 손상 될 수 있습니다 또는 크로스 링크 따라서이 분석에서 잘 수행하지 않습니다. 더 큰 보려면 여기를 클릭하십시오 이 그림의 버전입니다.
4. 앰플 리콘 라이브러리 준비
- 바운드 올리고의 올리고 풀과의 교잡 확장-내고
- 각각에 잘 대응하는 게놈 DNA 250 ng의 - 사전-PCR 룸에 필요한 낮은 EDTA TE의 볼륨 패널의 올리고 튜브 (예를 들어, 솔리드 NGS 패널 또는 헴-NGS 패널)의 5 μL, 100 추가 혼성화 (HYB) 판으로 표시 반 스커트 96 웰 플레이트.
- HYB 플레이트의 각 샘플 시퀀싱 시약 1 올리고 하이브리드 (OHS1)의 40 μl를 추가합니다. 혼합 6 회 - 부드럽게 아래로 최소 5를 피펫합니다. 각 열은 피하기 후 변경 팁교차 오염.
- 30 초 동안 1,000 XG에 접착 알루미늄 호일과 원심 분리기와 HYB 판을 밀봉합니다.
- 1 분 동안 95 ℃로 예열 된 혼성화 배양기에서 HYB 플레이트를 인큐베이션. 40 ° C에 하이브리드 인큐베이터의 온도를 설정합니다. 오래는 95 ° C에서 40 ° C (~ 90 분)로 감소 인큐베이터를 걸립니다 배양을 계속합니다. 참고 :이 점진적 냉각이 적절한 하이브리드 중요합니다.
- 지난 15 동안 - 하이브리드 배양 20 분, 필터 플레이트 장치 (FPU)를 미리 씻는다. 준비 만 웰 즉, 단지 이전에 열린 필터 판의 새로운 / 사용하지 않은 웰을 사용하지 않지만 결코 사용 된 웰 재사용 현재 분석에 사용한다. 주 :이 필터 판의 키트 번호, 필터 판의 표시, 및 상기 플레이트에 걸쳐 사용되는 밀봉재에 기초하여 명백하다.
- 멀티 채널 피펫을 사용하여, 각 웰에 엄격한 세척 1 (SW1)의 45 μl를 추가합니다.
주의 : 포름 아미드가 포함되어 있습니다. 커버와 원심 분리기 20 ° C에서 3 분 동안 2,250 XG에서 FPU. 잔류 액에 대해 하나 하나 잘 확인 (> 15 μL / 웰). - 잔류 액체가 존재하는 경우, FPU (180)도 회전하고 추가 3 분 동안 다시 원심 분리 단계를 반복한다. 잔류 액체가 제 시간을 통해 회전한다면이 필터 판에 결함이있을 수 있고, 정류 판을 교체 할 필요가 아니라면, 다음 단계로 진행.
- 멀티 채널 피펫을 사용하여, 각 웰에 엄격한 세척 1 (SW1)의 45 μl를 추가합니다.
- 혼성화 배양기는 40 ° C로 냉각되면 결로를 수집하는 20 ° C에서 적어도 30 초 동안 1,000 XG에 플레이트를 원심 분리. FPU에의 대응하는 사전 세척 우물의 중심 상에 HYB 판에서 각 시료의 전체 볼륨을 전송합니다. 교차 오염을 방지하기 위해 각 열 후 팁을 변경합니다. 20 ° C에서 3 분 동안 2,250 XG에서 FPU와 원심 분리기를 커버.
- 20 ° C에서 3 분 동안 2,250 XG에 SW1과 원심 분리기 45 μl를 추가. 이 세척의 총 반복합니다. 완전히 모든 SW1을 제거하기 위해 3 분 동안 2,250 XG에서 다시 FPU 180 ° 원심 분리기를 돌립니다.
- FPU를 감추고. 적절한 폐기물 용기에 모든 흐름을 통해을 (포름 아미드를 포함) 폐기하십시오. 다른 MIDI 폐기물 수집 플레이트를 사용하여 FPU를 재 조립. 20 ° C에서 3 분 동안 2,250 XG에서 각각의 샘플 잘 원심 분리기에 범용 버퍼 1 (UB1)의 45 μl를 추가합니다. 주의 : 포름 아미드가 포함되어 있습니다.
- FPU에 판과 피펫 위로 섞어 아래로 3 번에서 잘 각 샘플에 확장 내고 믹스 3 (ELM3)의 45 μl를 추가합니다. 주 : 확장 - 연결 반응이 필터 플레이트 막에 발생한다.
- 접착제, 알루미늄 호일로 FPU 접시를 밀봉하고 45 분 동안 예비 가열 37 ° C의 배양기에서 전체 FPU 조립체를 배양한다.
- 인덱싱 및 PCR 증폭
- 인덱스는 인덱스 증폭 경기 수에 해당 우물에 사용되는 나누어지는I5 프라이머 튜브 (흰색 모자, 맑은 용액) 수직, H를 통해 행 A를 정렬, i7의 프라이머 튜브 (오렌지 모자, 황색 용액) 수평을 다음 방식으로 배열 색인 플레이트기구의 프라이머를 배치하여 (IAP)을 먹었다 하는 P10 멀티 채널 피펫을 사용하여 (12)를 통해 열 1과 정렬의 IAP의 각 행에 i7의 프라이머 (황색 용액)의 4 μl를 추가하고 IAP의 각 컬럼의 i5 프라이머의 4 μL (맑은 용액)를 추가합니다.
- 얼음 또는 냉각 블록에서 샘플 당 PCR 마스터 믹스 2 (PMM2)의 25 μL에 DNA 중합 효소 1 (TDP1)의 0.5 μl를 추가하여 PCR 마스터 믹스를 준비합니다. 간단히 반전 빠르게 소용돌이, 그리고 혼합 할 수있는 PCR 마스터 믹스 원심 분리기.
- 각 IAP의 잘 피펫 위로 섞어 아래로 3 번에 PCR 마스터 믹스 22 μl를 추가합니다. 우물 사이의 조언을 변경합니다. 4 ° C에서 IAP를 유지합니다.
- 45 분 확장 - 연결 반응 (단계 4.1.10) 후, 알 제거 조심스럽게 인큐베이터에서 FPU를 제거하고호일 씰을 uminum. 20 ° C에서 3 분 동안 2,250 XG에 뚜껑을 원심 분리로 커버.
- FPU에 잘 각 샘플의 50 mM NaOH를 25 μl를 추가합니다. 아래로 최소 6 시간을 피펫과; 막과 접촉 피펫 팁을 보장합니다. 각 열 후 변경 팁. 실온에서 5 분간 인큐베이션.
- 다음과 같이 전송 샘플은 IAP에 FPU에서 용출 :
- 20 μL로 설정 P100 멀티 채널 피펫을 사용하여, 상하 FPU 플레이트 상에 적어도 6 회 NaOH를 피펫. 약간 플레이트 오프 완료 열망을 보장하기 위해 FPU 플레이트를 기울이십시오.
- 전송합니다 IAP의 해당 컬럼에 FPU 20 μL. 부드럽게 피펫 아래로 최소 6 시간은 철저하게 PCR 마스터 믹스로 DNA를 결합 할 수 있습니다. 20 ° C에서 1 분 동안 1,000 XG에 접착 필름과 원심 분리기와 IAP를 밀봉합니다.
- 포스트-PCR 룸에 PCR 플레이트를 가져와 열 자전거 타는 사람에 접시를로드합니다. PCR을 피를 실행3 분 95 ° C에서 단계를 변성 열로 구성된 rogram; 30 초, 30 초 동안 62 ° C, 60 초, 72 ° C, 95 ° C의 25주기 다음에; 5 분, 72 ℃에서 최종 연장 하였다; 10 ° C에서 보류로 마무리. PCR의 완료에 따라 다음 단계로 진행하지 않는 경우, 플레이트 밤새 열 순환기에 남아있을 수 있거나, 두 가지 일 8 ° C 2에 저장 될 수있다.
- PCR 정제 및 비드 기반 정규화
- 적어도 20 분 다음 단계 전에 실온에서 4 ° C 냉장고와 장소에서 자기 정화 구슬, 용출 버퍼 (EBT), 및 겔 전기 영동 시약을 제거합니다.
- PCR을가 완료되면, 20 ° C에서 1 분 1,000 XG에 원심 분리기는 결로를 수집합니다. 각 PCR 반응의 양도 1 μL은 샘플 1/5 희석 물을 4 μl를 포함하는 튜브 / 판 우물을 제거합니다. 최대 피펫 아래로 혼합.
- 의 2 μl를 추가미세 겔 완충액 2 μL를 PCRed 샘플을 희석시켰다. 스트립 / 플레이트를 밀봉합니다. 30 초 동안 1,000 XG에서 최소 30 초 원심 분리기에 대한 1,800 rpm으로 흔들어. 제조사의 프로토콜에 따라, 라이브러리의 제조가 허용 가능한 라이브러리 (도 3 참조)를 수득하는 경우 평가 마이크로 유체 겔 혼합물을 실행.
- 그들은 잘 현탁하고 색상이 균일 나타날 때까지 자기 정화 비드 소용돌이. 각 샘플에 구슬의 45 μl를 추가합니다.
- 명확한 접착 필름과 판을 밀봉하고 2 분 동안 1,800 rpm으로 접시를 흔들. 10 분간 진탕없이 실온에서 인큐베이션.
- 자기 스탠드에 접시를 놓습니다. 상층 액이 해제되면, 조심스럽게 제거하고 상층 액을 버린다. 어떤 구슬이 실수로 팁으로 흡입하는 경우, 다시 판에 구슬을 분배하고 보자 자석에 접시 받침대를 2 분 동안 상층 액이 해제되었는지 확인합니다.
- t에 접시와그는 자기 스탠드는 물론 각 샘플에 새로 제조 된 80 % 에탄올 200 μl를 추가합니다. 앞뒤로 몇 번 플레이트를 이동합니다. 30 초 동안 자기 스탠드에 접시를 품어. 조심스럽게 제거하고 상층 액을 버린다. 이 세척의 총 반복합니다.
- 간단히, 튜브 측에 어떤 에탄올을 절감하기 위해 1000 XG에서 접시를 원심 분리 자기 스탠드에 다시 접시를 배치하고, 에탄올을 제거하기 위해 10 μL로 설정 P10의 멀티 채널 피펫을 사용하여 여분의 에탄올을 제거합니다. 8 분 - 5 자연 건조 구슬을 허용합니다.
- P100 멀티 채널 피펫을 사용하여, 각 웰에 세전 이익의 30 μl를 추가합니다. 최대 피펫과 몇 번 아래 구슬은 튜브의 측면에서 올 수 있도록. 명확한 접착 필름과 판을 밀봉하고 2 분 동안 1,800 rpm으로 접시를 흔들.
- 3 분 동안 흔들림없이 실온에서 품어. 비드가 완전히 재현 탁하고 있지 않은 시료가있을 경우 부드럽게 비드를 재현 탁 위아래로 피펫.
- 자기 스탠드에 접시를 놓습니다. P100 멀티 채널 피펫을 사용하여 완전히 새로운 판에 뜨는 20 μl를 전송 도서관 정규화 플레이트 (LNP)라고합니다. 별도의 플레이트에 각각의 염기 서열 라이브러리의 ~ 나머지 10 μl를 전송은 남은 청소 업 도서관 플레이트 (RCLP)라고합니다. 필요한 경우이 두 번째 순서 실행을 위해 예비로 사용할 수있는, 최종 라이브러리 준비와 함께이 판을 보관하십시오. 다음 단계로 진행하지 않는 경우, 및 LNP RCLP -15 -25 ℃에서 저장 될 수있다.
- 적극적으로 와동 및 라이브러리 정규화 구슬 1 (LNB1)을 재현 탁. 완전히 튜브의 하단에 LNB1 비드 펠렛을 재현 탁 중요하다. 샘플 당 도서관 정상화 첨가제 1 (LNA1)의 44 μL와 LNB1의 8 μl를 혼합하여 정규화 믹스를 준비합니다. 20 초 - 적극적 (10)의 정규화 믹스 소용돌이.
주의 : LNA1는 포름 아미드가 포함되어 있습니다. - 간헐 반전 A의차 정상화 믹스의 텍싱는 LNP의 각 시료 45 μl를 추가합니다. 명확한 접착 필름과 판을 밀봉하고 30 분 동안 1,800 rpm으로 접시를 흔들. 이는 30 분 인큐베이션 라이브러리 표현 클러스터의 밀도에 영향을 미칠 수있는 큰 미만 또는 30 분의 배양과 같은 적절한 라이브러리 정규화 중요하다.
- 30 분 동안 배양 시약 카트리지를 해동 HYB 완충액 (HT1)에 의해 시퀀싱 시약을 제조 [주의 : 모두 포름 아미드를 포함]. 또한, 이후 단계 얼음을 얻고 96 ° C로 설정되어있는 150 mL의 원심 분리 튜브에 적합한 열 블록을 보장한다.
- 30 분간 혼합 단계가 완료되면, 자기 LNP 스탠드의 장소. 상층 액이 해제되면, 조심스럽게 제거하고, 적절한 폐기 용기에 뜨는을 폐기 멀티 채널 피펫을 사용합니다.
- 다음과 같이 라이브러리 정규화 워시 1 (LNW1)와 구슬을 자기 스탠드에서 LNP를 제거하고 세척 :
- 45 추가물론 각 샘플에 LNW1의 μL.
주의 : 포름 아미드가 포함되어 있습니다. 명확한 접착 필름과 판을 밀봉하고 5 분 동안 1,800 rpm으로 접시를 흔들. 이 세척의 총 반복합니다. 두 번째 세척 후 모든 LNW1을 제거해야합니다.
- 45 추가물론 각 샘플에 LNW1의 μL.
- 자기 스탠드에서 LNP를 제거하고, 멀티 채널 피펫을 사용하여 샘플을 용리 각 웰에 (보다 오래된 주 이하), 0.1 N NaOH로 30 μL를 추가한다. 명확한 접착 필름과 판을 밀봉하고 5 분 동안 1,800 rpm으로 접시를 흔들.
- 5 분 용출 동안 잘 보관 플레이트 (SGP)를라는 새 판에 사용되는 각 도서관 정규화 저장 버퍼 1 (LNS1)의 30 μl를 추가합니다.
- 자기 스탠드에 LNP를 놓습니다. 상등액 클리어되면, SGP에 LNS1에 30 ㎕의 용출 옮긴다. 교차 오염을 방지하기 위해 샘플과 팁을 변경합니다.
- 적어도 30 초 동안 1,000 XG에 접착 필름 및 원심 분리기와 함께 접시를 밀봉합니다.
- 5 추가(1) 각 시료의 리터가 표시 풀링 앰플 리콘 라이브러리 (PAL) 1.5 ML 튜브에 서열합니다. 혼합 잠깐의 microcentrifuge에서 회전을 멈추도록 PAL을 소용돌이.
- HT1의 596 μL - 590에 PAL 10 μL - 사용되는 어떤 순서 화학 (V2 또는 V3)에 따라 4를 추가 할 수 있습니다. 일반적으로, V2 화학에 대한 HT1의 595 μL와 V3 화학에 대한 HT1의 592 μL에 PAL의 8.5 μL에 PAL의 5.8 μl를 추가합니다. 희석 된 앰플 리콘 라이브러리 (DAL) 튜브 등이 튜브 라벨.
- DAL 와동과 잠깐의 microcentrifuge에 스핀 다운. 2 분 동안 96 ° C에서 DAL을 품어. DAL 관 3 회 전환하고 시퀀싱을위한 시퀀서를 준비하는 동안, 적어도 5 분 동안 얼음에 DAL했습니다. 5 분 후, DAL로드 될 준비가되어있다.
- SGP 완료되면, 접착 알루미늄 호일 필름과 판을 밀봉하고, 날짜와 판 ID로 레이블을 붙입니다. -15 -25 ° C에서 밀봉 SGP와 PAL을 저장합니다.

그림 3 :. 도서관 준비 QC 젤 예는 낮은 대역을 통해 녹색 선이 낮은 마커 및 상부 밴드 위에 보라색 라인을 표시하는 것입니다 높은 마커를 표시하는 것입니다. 모든 차선 H1, A2, B2, C2, E2, 및 G2 잘했다. 라이브러리 준비 차선 D2, F2, 및 H2를 들어, 최적으로 작동하지 않았다,하지만 결과는 여전히 그들은 단지 적절한 보상을하지 않을 수 있습니다 얻을 수 있습니다. A3의 경우, 라이브러리 준비는 거의 근무하고 대부분이 DNA 샘플은 분석에 적합하지 않았다. 표본이 잘 PCRed에서 직접 수행되기 때문에 하부 마커 상기 하부 대역은 사용되지 않는 프라이머이다. NTC에 샘플 만 사용하지 않은 프라이머 밴드, 그리고 아무것도 있어야합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
5. 시퀀싱
- 올바른 SampleSheet.csv가 실행을 위해 만들어졌다 확인합니다. 예는 보충 그림 1을 참조하십시오.
- 헹구고 흐름 셀을 건조 및 해동 시약 카트리지에 DAL 600 μl를 추가합니다.
- 화면의 지시에 따라 순서에 대한 시퀀서를 준비합니다.
6. 데이터 분석
- 생물 정보학 파이프 라인을 실행합니다. 돌연변이, 삽입, 삭제 및 증폭 (18)를 식별하기 위해 사내, 사용자 정의 디자인 생물 정보학 파이프 라인을 활용합니다.
- 파이프 라인이 완료되면 파이프 라인 처리의 품질 관리에 중대한 오류 / 경고 원조로, 오류 / 경고에 대한 로그 파일을 확인하십시오.
- 서열 라이브러리를 확인하기 위해 실행 통계 (표 3)을 분석 통과 한 실험실 품질 관리 측정 (그림 4) 결정. 수동 (게놈 데이터 뷰어에서 예를 들어, 통합적인 게놈 바이올렛을 .bam 파일을 확인하여 각 변형을 검토물병 16 (IGV)).
참고 : 검증 된 대립 유전자 주파수 범위 및 품질 필터링 후 범위의 최소 깊이 이상 만 변종은 (인간 게놈 변이 사회의 (HGVS) 명칭 사용)보고됩니다. 가능성 양성, 양성 병원성 관련, 아마 질환, 알 수없는 의미 (VUS)의 변형 : 최종보고, 각 엑손 변종으로 분류되었다. 그 것으로 간주 양성 변형과 동의어 변경을 제외하고 5 % 대립 유전자 주파수의보고 기준, 위의 모든 변종이보고되었다.
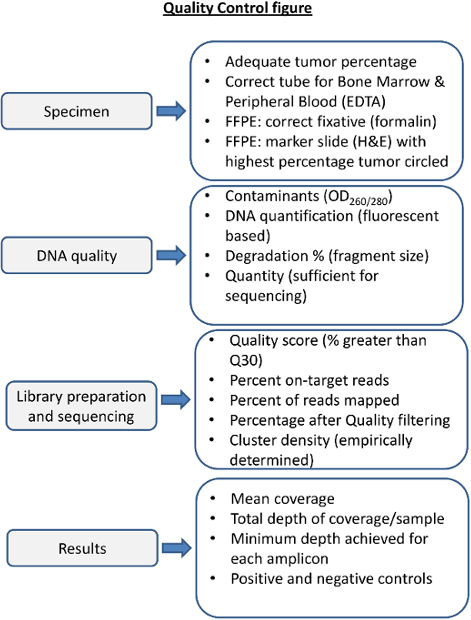
NGS에 대한 품질 관리 단계의 그림 4. 개요. 그 과정에서 각 단계의 품질 관리는 전후 순서 측정이 고려되도록 결과를 얻을 것입니다 시퀀싱을 보장 할 필요가있다. 해당 시료 처리 고품질 DNA 필수적이다. 혈액과부적절한 고정 제의 골수 저품질 DNA를 얻을 수있다. 고형 종양 표본의 부적절한 고정은 DNA (B5에서 예를 들어, 고정을)이 저하 될 수 있습니다. DNA 품질은 분광 수단에 의해 단백질 및 RNA의 오염으로 평가하고, 정확한 양 및 DNA의 무결성을 평가해야한다. 시퀀싱 통계는 경험적으로 시퀀싱 실험실에서 결정하고 각 시퀀싱 반응과 모든 시료에 대해 따라야 할 필요가있다. 각 시편에 대한 시퀀싱 결과를보고하기 전에이 범위, 깊이, 그리고 긍정적이고 부정적인 컨트롤의 적절한 성능을 평가해야한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
사례 1 - 헴-NGS 패널
백혈병 말초 혈액에서 추출한 DNA는 헴-NGS 패널에 대한 충분한 품질과 수량 (176 NG / μL)이었다. 범위의 전체 평균 깊이 (1000 배의 범위의 최소 평균 깊이 위) 4,933x이었다. 추가 실행 통계를 표 3에있다. 250x 범위 아래 팔 영역 3 의한 프라이머의 부적절한 트리밍 (즉, 프라이머 서열이 제대로 인해 순서 오류로 제거되지 않은) 1 분석 알려진 아티팩트 하였다이었다 다른 4없이보고 변형에 다른 유전자의 엑손 부분 영역이었다. 임상 프로토콜은 적어도 250 읽 적용 변종을보고 포함하지만, 적어도 100 모든 데이터는 검토를 위해 데이터베이스로 가져 읽습니다.
데이터 처리부 (F)롬 생물 정보학 파이프 라인은 세보고, 질병 관련 돌연변이를 검출; FLT3의 과오 돌연변이, IDH2의 과오 돌연변이와 NPM1의 틀 이동 돌연변이. 그들의 대립 유전자 주파수와 엑손 변형은 표 4에 설명되어 있습니다. FLT3에서 일반적인 돌연변이가 자동으로 우리의 파이프 라인에 의해 호출과 엑손 (14)의 엑손 (14) 육안 검사의 육안 검사를 필요로하지 않는 FLT3 - 내부 탠덤 중복 (ITD)입니다 FLT3의 제출 된 시료에는 삽입 또는 중복을 보이지 않았다. FLT3 I836del 1 % 대립 유전자의 빈도에 있었다과 5 %의 검증 최소 대립 유전자 주파수를 하회하기 때문에 최종 보고서에 포함되지 않았습니다. 이 돌연변이는 FLT3의 D835Y 변화와 같은 DNA 분자에 있지 않은 (즉, 동일한 앰플 리콘 영역에서 관찰 아니라 "CIS"서열 중 판독에서의)하고 만 .bam 인터넷의 수동 검토으로 관찰레는 p.D835Y 변화를 확인할 때. 두 FLT3 돌연변이의 낮은 대립 유전자 주파수는 이러한 돌연변이가 이질성 및 / 또는 클론 진화를 나타낼 수 제안; 그러나, 차이점은 앰플 리콘 또는 근처에서 단일 염기 다형성 (SNP) 또는 이러한 대립 유전자의 증폭에 영향을 프라이머 서열을위한 중첩 분석 성능에 기인 될 수있다.
FLT3, IDH2 및 NPM1의 AML 확인 된 돌연변이 사례 1 헴-NGS 패널 결과, FLT3에서 AML. 돌연변이 세 일반적으로 돌연변이 유전자는 AML (COSMIC 데이터베이스 17)와 성인 환자의 ~ 25 %에서 관찰과 하나되어 있습니다 티로신 키나아제 도메인에서의 내부 직렬 중복 (ITD) 또는 과오 돌연변이. FLT3-ITDs에는 일반적인 돌연변이이며, 표준 화학 요법에 가난한 응답과 관련된 동안 FLT3 키나제 (D)의 예후 중요성omain 점 돌연변이,이 AML 환자에서 볼 수 있듯이, 예후 (14)에 분명 영향을 미친다. 이소 시트 레이트 탈수소 효소 2 (NADP +), 미토콘드리아 (IDH2)는 일반적으로 AML에 돌연변이 된 후생 유전 학적 수정을 코딩하는 유전자이다. 후생 유전 학적 수정의 돌연변이는 유전자 조절 곤란으로 이어질 유전자의이 클래스의 다른 변이를 나타내는 IDH1 및 DNMT3A 돌연변이와 AML에서 비교적 일반적이다. nucleophosmin (NPM1) 유전자에서의 돌연변이는 AML에서 가장 흔한 돌연변이 획득 하나, 일반적으로 (a FLT3의 부재 - ITD) 좋은 예후 인자 인 것으로 간주된다.
NPM1 및 IDH2 공동 돌연변이는 89 %의 3 년 생존율과 함께 양호한 예후 표시기 5로 문헌에 기재되어있다. 전체적인 3 년 생존율과 비교했을 때 상당한 생존 이점을 나타낸다야생형 NPM1 31 %의 IDH2. 예를 들어, 표준의 치료 방법은 NPM 1 돌연변이 분석 및 FLT3-ITD 변이를 포함한다. 이차 돌연변이가 유리한 수있는 바와 같이,이 시나리오에서는 오직 NPM1 돌연변이의 검출 위험 적절 계층화 환자에게 실패 (예 IDH2) (예 TET2) 또는 불리한, 골수 이식을 경감을위한 신뢰를 감소시킨다.
사례 2 - 솔리드 NGS 패널
FFPE 조직에서 추출한 DNA는 252 NG / μL의 농도와 1,000 염기쌍 (bp의) 아래의 DNA의 4 %로, 고체 NGS 패널에 대한 충분한 질과 양이었다. 데이터 분석 후 범위의 평균 깊이는 9167 250 읽기 깊이 아래에없는 지역으로 (물론 1000 우리의 최소 깊이 차단 읽고 위) 읽습니다. 추가 QC 측정은 웃음입니다표 3 WN.
ERBB2 (HER2 / NEU)의 엑손 (20)에서의 프레임 삽입 및 TP53의 과오 돌연변이 : 생물 정보학 파이프 라인을 통해 처리 된 데이터는 두 질병 관련 돌연변이를 발견했습니다. 그들의 대립 유전자 주파수와 모든 엑손 변형은 표 5에 표시됩니다. 명명법에 반영된 ERBB2에 삽입 실제로 중복 탠덤 (HER2 / NEU) 순서를 나타냅니다. 식별 및 시퀀싱의에 프레임 삽입이 필요 수동 검토의 확인은 IGV를 읽습니다. TP53 및 HER2 모두 바와 같이 50 % 초과의 주파수 변이의 검출 / NEU 돌연변이 이형 (LOH) 이벤트 (논의를 참조)의 손실을 암시한다.
폐 선암 검출 돌연변이 사례 2의 솔리드 NGS 패널 결과 전N ERBB2 (HER2 / NEU) 및 TP53, 두 유전자는 일반적. HER2가 / NEU 폐암, EGFR 다른 통상적 돌연변이 유전자 유사 티로신 키나제 수용체를 코딩 폐암 환자의 현재 표준 진료의 일부로서 테스트되지 . 20 삽입 2에서 관찰되는 HER2 / NEU 엑손 활성화 - 폐 선암의 4 %, 폐암에서 관찰 HER2 / NEU 돌연변이의 대부분을 설명하고, 일반적으로 EGFR 및 ALK (12)와 같은 다른 드라이버 유전자 변이없이 종양에서 볼 수 있습니다 . HER2 / NEU과는 mTOR 억제제 (13)와 화학 요법 (15)과 함께 단일 클론 항체 트라 스투 주 마브으로 중요한 질병 관리와 병용 요법에 부분 반응을 포함 HER2 / NEU 삽입을 활성화 환자를위한 다양한 치료 옵션에 대한 증거를 보여주는 가능성의 몇 줄이 있습니다. TP53 변화를 발견하는 낯선되지 않습니다암 MMON하지만,이 시간에 더 실행 가능한 치료법이 없습니다.
| 사례 1 | 사례 2 | |
| 총 시작 읽고 | 2215926 | 2129110 |
| 의 비율 매핑을 읽 | 98.42 % | 98.29 % |
| 의 비율은 대상에 읽어 | 99.01 % | 97.29 % |
| 의 비율은 필터 후 대상에 읽어 | 97.60 % | 95.45 % |
| 사용 가능한 비율 읽고 | 94.87 % | 91.79 % |
| 250x의 범위 상기베이스의 백분율 | 98.40 % | 100 % |
| 베이스 (a)의 비율보브 1000X 범위 | 95.90 % | 99.70 % |
| 250x 아래 범위 - 앰플 리콘 번호 | 8 | 0 |
표 3. 시퀀싱 실행 QC 메트릭이 라이브러리 준비 QC 샘플이 경과되었는지를 결정하기 위해 데이터를 검토에 사용되는 평균 범위를 포함하는 가장 중요한 실행 통계에 대한 요약이 아니다. 모든 백분율이 90 % 이상일 경우, 전체 프로세스는 성공적이지만, FPU 세정 단계 또는 프라이머 누화에서 SW1의 이월 또는 UB1 80의 범위에서 '대상 비율'하부를 갖도록하여, 수 - 90 %. '비율 매핑'이 너무 낮 으면 모든 샘플은 인간에 정렬해야합니다으로, 즉, 세균이나 다른 DNA의 오염을 나타냅니다. 이러한 사양의 80 % 이하가되면, 샘플 방법 페이지를 결정하는 데 도움이 추가 검토 네요roceed 프로세스를 향상시킬 수 있습니다.
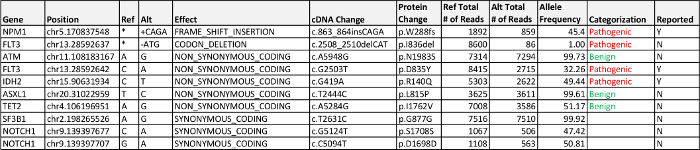
표 4 :.. 사례 1 결과 검출 된 병원성, 관련 질환, 알 수없는 의미의 변형 (VUS), 그리고 나와 5 % 대립 유전자 주파수의보고 기준 이상 가능성 양성 엑손 변형 이 표를 다운로드하려면 여기를 클릭하십시오.
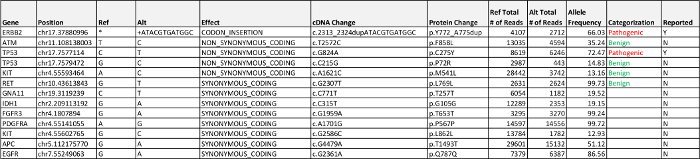
표 5 :.. 사례 2 결과 검출 관련된 병원성, 질병, 알 수없는 의미의 변형 (VUS), 그리고 나와 5 % 대립 유전자 주파수의보고 기준 이상 가능성 양성 엑손 변형 이 표를 다운로드하려면 여기를 클릭하십시오.
보충 그림 1 : 앰플 리콘 기반 SampleSheet.csv의 예. 이 시트는, 어떤 워크 플로우 (예를 들어, GenerateFastq), 어떤 응용 프로그램 및 분석 (예를 들어, FastqOnly), 얼마나 많은 순서 염기 (또는 읽기) (이 경우 186 bp의에서 (이 경우의 증폭에) 실행하는 데 어떤 화학 시퀀서에 전달 X 186 BP), 및 샘플)이 경우 이중 색인에 (특정 인덱스와 관련된 마지막으로 무엇. 노란색으로 강조하는 부분은 실험이 원하는대로 변경,하지만 실험실이 매개 변수를 사용하여이 경우에. 할 수 있습니다 이 그림을 다운로드하려면 여기를 클릭하십시오.
토론
이 논문에서 설명 된 두 NGS 시험은 임상 적으로 제공되는 바와 같이 가장 중요한 실제적인 고려 사항은 품질 관리된다. 특히, 가까운 고려 사항은 품질과 추출 된 DNA의 양에 지불해야합니다. 이것은 종종 변수 DNA 수율이 매우 저하되어 FFPE 샘플에 특히 중요하다. 이소프로판올 침전 법 컬럼 - 기반 방법은 때때로 제한 용출 부피와 DNA 전단 이어질 것으로 나타났다로 FFPE 시료로부터 DNA 수율을 최대화하기 위해 개발되었다. 따라서, 검체가 너무 낮은 농도를 산출하거나 분석 너무 저하되는 대부분의 시간 인한 조직의 크기, 형태 또는 고정이 아닌 추출 처리에 가장 쉽다. 추출 오류가있는 경우 혈액 / 골수 시험편 들어, 인해 샘플 인 hemodilute 통상 (즉, 백혈구 또는 종양이 연신 세포의 충분한 수가 갖지 않는) 절제 또는 항암 치료.
. 검증 동안 NT "는> DNA의 질과 양의 수용성에 대한 단절을 확립한다 (100)의 추천 입력 - 250 NG들은 상기 분석에 사용되는 상기 DNA의 품질이 좋은 경우에는 다음 낮은 입력 양이 성공적 일 수있다. (증폭 DNA의 양이 권장 된 입력에 도달 할 것이기 때문에)을 높은 입력 양 시퀀싱 결과의 품질을 향상시킬 수있다 - DNA의 품질이 나쁘면 또한 (NG (250) 즉, 증폭 DNA 양은 적은 100 초과) . DNA의 품질과 수량에 대한 메트릭 라이브러리 준비에 DNA를 진행하기 전에 각 시료에 적용해야합니다. 그 샘플에 "회색 지대"(그림 2 참조)는 연구소 책임자 또는 지정 인의 재량에 따라 실행해야합니다. 현재 가장 DNA가 서열 중에 잘 수행하지 않을 경우 예측하는 방법은 입력 된 DNA의 정량, 품질 평가를 허용하는 qPCR에 기반 분석을 수행하는 것이다.이 방식은 bioavailab을 해결다른 크기의 조각 (예를 들어, 100 bp의 150 bp의 200 bp의 300 bp의)와 비교 수율의 증폭을 통해 표본의 크기가 다른 조각의 ility.현재 라이브러리 준비는 몇 가지 고비 중 하나에서 실족 라이브러리에 오류가 있거나 품질이 좋지 질 수 있습니다 수동 단계의 큰 숫자를 포함한다. 마이크로 유체 겔 분석은 시퀀싱 전에 라이브러리 준비 문제를 확인하는 유일한 QC 공정이다. 따라서, 여분의 마음 챙김은 성공적인 반응의 확률을 높일 수있는 몇 가지 중요한 단계가 있습니다. 올바른 샘플 올리고 뉴클레오티드 풀 각 시험편에 사용되는 보장 필수적이다. 보장과 제대로 각 샘플은 듀얼 인덱스 PCR 프라이머 쌍 중 하나 (96)의 고유 한 조합을 포함 기록은 샘플 믹스 업에 대한 가능성을 줄일 수 있습니다. 또한, 필터 판 (FPU)를 올바르게 배수되도록하는 것이 중요하다; 이 적절하게 배출하지 않을 경우이는 exte을 일으킬 수 있습니다라이브러리 준비 nsion 결찰 단계는 suboptimally 수행하고 품질이 좋지 시퀀싱 데이터로 이어질 수 있습니다. 라이브러리 QC 후에는 LNB1 비드가 완전히 현탁되도록하는 것이 중요하고 LNB1 / LNA1 용액 혼합물의 농도는 라이브러리의 몰 농도를 결정하는 데 사용되는 샘플에 첨가하기 전에 잘 혼합된다. 비드 용출 단계는 비드를 용출 라이브러리의 최적 량 시킨다면 마지막으로 클러스터링 밀도를 감소 할 가능성이 라이브러리는 평균 충분한 커버리지를 획득하지 않는 원인이된다. 반대로, 도서관의 과잉은 품질이 좋지 읽기에 이어질 것입니다. 따라서, 시퀀서 라이브러리 최적 풀링 및 클러스터링을 할 수 있도록 비드 기반 정규화 단계에서 일치하는 것이 중요하다.
라이브러리의 제조뿐만 아니라, 그 원료, 역 다중화 fastq 파일의 정확한 돌연변이 호출을 생성하는 생물 정보학 파이프 라인을 확인하는 것이 중요하다. 선택하나를 통해 선별해야 할 것이다 많은 오픈 소스와 시판 얼 라이너, 변형 발신자 및 NGS 소프트웨어 패키지 있기 때문에 사용자 정의 솔루션은 많은 시간이 소요될 수 있습니다. 맞춤 알고리즘은 필수적 성능 통계를 추출 대부분 오픈 소스 도구를 회피 고유 반복 돌연변이를 식별하고, 각각의 궤적 위에 카피 수의 상태를 결정하도록 설계 될 필요가있다. 생물 정보학 파이프 라인의 검증 프로세스 동안, 충족 또는 품질 여과 후 따르면 최소 깊이 (예, (250)의 최소 판독) 및 최소 대립 유전자 빈도를 모두 초과 변이체에 대한보고 단절을 결정하는 것이 중요하다 (예를 들어, 4 %). 이 다중화 된 앰플 리콘 기반 분석하기 때문에, 그 최소 범위의 평균 깊이를 결정하는 것이 중요하다 (예를 들면, 1000 배) 라이브러리의 읽기의 최소 깊이까지 최저 수행 앰플 리콘을 얻을 수있게 달성 할 필요가있다. 또한, 분석의 다중 특성은 C를 수행ause 오프 대상 효과와이 '유물'발견 완전히 실행하기 전에 심사해야합니다. 기재된 분석 또 다른 중요한 한계는 유효 최소 대립 유전자 빈도를 얻기 위해서는 10 % 이상의 종양을 포함하는 샘플이 필요하다.
저주파 1 %, FLT3 삽입의 검출은 수동 검토를 여전히이 과정에서 바람직하다고 증거이다. 심지어 5 %의 대립 유전자 주파수 차단과 함께, 몇 가지 중요한 돌연변이는 아마 놓친 때문에 수동 검토는 이러한 변형을 확인하는 것이 필수적 일 것이다. FLT3-ITDs에 들어, 엑손 (14)의 육안 검사가 주목하지 않는 로우 레벨 혹은 큰 삽입 / 복제를 위해 모든 AML 환자에 대해 수행된다. 또한, 일반적으로 프라이머 서열 옆에있는 HER2 엑손 (20) 삽입은 수동 개입이 필요합니다. 강력한 생물 정보학 파이프 라인을 가지고에도 불구하고, 일부 변종은 하드 컷을 갖는 단지 성격 인 간과 갈 수있다대부분의 통계는 위에서 언급 한 오프. 더 나은 생물 정보학은이 문제를 완화하기 위해 필요한 것 같은 것보다 도서관 준비 및 / 또는 시퀀싱 방법, 그것은 더 적은 유물과 오탐 (false positive)를 포함하는 더 낮은 컷오프에서 양질의 데이터가 더 유리하기 때문이다.
탐지 및 대립 유전자 주파수 해석에 의한 종양의 백분율을 결정하고, 게놈의 일부 영역의 증폭 바이어스 곤란하기 어려울 수있다. 이는 이형 (LOH) 이벤트의 손실로 해석되는 케이스 (2)의 관찰뿐만 아니라, 50 % 이상의 대립 유전자 빈도가 돌연변이의 명백한 증가로 이어지는 어느 인해 정상 대립 유전자의 손실을 검출 할 수는 읽는 돌연변이 대립 유전자 (예를 들어,이 돌연변이와 하나의 정상 사본) 또는 다른 메커니즘의 이득. 이러한 메커니즘은 배열 비교 게놈 혼성화 (aCGH 19) 및 / 또는 SNP 유전자형 어레이를 이용하여 설명 될 수있다. (20).
현재 대상 농축 방법은 더 시퀀싱 단일 샘플의 범위와 대상 시퀀싱 읽기 오프 더의 필요성이 발생할 비효율적 인 하이브리드 캡처 또는 다중 PCR 기법 중 하나의 전일 절차에 의존하고 있습니다. 가까운 장래에 기대 NGS 분자 종양학 추가 애플리케이션이 완전히 오토메이션하고 입력 DNA의 매우 낮은 양의 샘플을 처리 할 수있다 쉽게 라이브러리 제조 방법을 포함한다 (즉, 1 NG 미만)과 샘플 높은 분해로 DNA. 이러한 문제를 해결하기 위해, 대부분의 방법은 아마도, PCR 기반이 될 것 중 하나 다단계 PCR 방법 또는 대규모 병렬 싱글 브레이크 PCR 방법을 주도했습니다. 또한, 각각의 증폭 분자 바코드 대폭 배경 시퀀싱 소음의 저감을 도시 한 하부 대립 유전자 빈도를 달성 circulat 캡처를 향해 이동하는 종양 세포의 낮은 비율로 샘플을 테스트 할 수있게종양 세포를 보내고.
암 표본에서 질병 관련 돌연변이의 검출은 수십 년 동안 치료의 표준이되어왔다. 역사적으로, 유전자는 종종 테스트 시퀀스의 끝 선도 돌연변이의 식별로, 한 번에 연속적으로 한 유전자 / 엑손을 시험 하였다. NGS의 출현은 종양과 관련된 다수의 돌연변이의 식별에 이르는 병렬로 많은 암과 관련된 여러 유전자 시퀀싱에 덜 편향된 접근 허용했다. 암 체세포 돌연변이 검출 용 NGS의 임상 적 유용성이 더욱 명백하다. 사실상, 종양 샘플 NGS 기반 분석은 기존의 단일 유전자 테스트 도전 새로운 패러다임을 나타내고 있지만 임상 적 유용성은 매우 명백하다. 임상 실험실 오늘이 강력한 기술의 응용 프로그램과주의 방법 검증 및 시험 해석을 결혼 할 수있는 흥미로운 기회를 가질 수있다.
공개
저자는 공개 아무것도 없어.
감사의 말
저자는 생산에 원고와 도움이 읽어 다니엘 야생의 도움을 인정하고 싶습니다.
자료
| Name | Company | Catalog Number | Comments |
| Genomic DNA ScreenTape | Agilent Technology | 5067-5365 | |
| Genomic DNA Reagents | Agilent Technology | 5067-5366 | |
| High Sensitivity D1000 ScreenTape | Agilent Technology | 5067-5584 | |
| High Sensitivity D1000 Reagents | Agilent Technology | 5067-5585 | |
| TapeStation 2200 | Agilent Technology | G2965A | |
| TapeStation Analysis Software | Agilent Technology | A.01.04 or higher | |
| 96-well Tube Storage Racks | Any Vendor | ||
| 15/50 ml Tube Rack | Any Vendor | ||
| 96-well Plate Rack | Any Vendor | ||
| Pipette, single-channel, 0.5–2.5 μl | Any Vendor | ||
| Pipette, single-channel, 1–10 μl | Any Vendor | ||
| Pipette, single-channel, 2–20 μl | Any Vendor | ||
| Pipette, single-channel, 10–100 μl | Any Vendor | ||
| Pipette, single-channel, 20–200 μl | Any Vendor | ||
| Pipette, single-channel, 100–1,000 μl | Any Vendor | ||
| Serological Pipettor | Any Vendor | ||
| Vortexer | Any Vendor | ||
| Ice bucket | Any Vendor | ||
| Microcentrifuge (for tubes and strip tubes) | Any Vendor | ||
| Freezer, -20 °C | Any Vendor | ||
| 4 °C Refrigerator | Any Vendor | ||
| Water or Bead Bath | Any Vendor | ||
| Incubator (37 °C) | Any Vendor | ||
| Serological Pipettes, 1 ml | Any Vendor | ||
| Serological Pipettes, 5 ml | Any Vendor | ||
| Serological Pipettes, 10 ml | Any Vendor | ||
| Serological Pipettes, 25 ml | Any Vendor | ||
| Gloves | Any Vendor | ||
| Razor Blades/Scaples | Any Vendor | ||
| KimWipes | Any Vendor | ||
| 15 ml Conical Tube | Any Vendor | ||
| 50 ml Conical Tube | Any Vendor | ||
| Paper Towels | Any Vendor | ||
| 200 proof Ethanol | Any Vendor | Store in Flammable Cabinet | |
| 2-Propanol (Isopropanol) | Any Vendor | Store in Flammable Cabinet | |
| 25 ml Reservoirs | Any Vendor | ||
| 10 N NaOH | Any Vendor | ||
| Pipette, 8-channel, 1 – 10 μl | Any Vendor | ||
| Pipette, 8-channel, 10 – 100 μl | Any Vendor | ||
| Pipette, 8-channel, 20 – 300 μl | Any Vendor | ||
| Ice Bucket | Any Vendor | ||
| Water Squirt Bottle | Any Vendor | ||
| Alcohol Squirt Bottle | Any Vendor | ||
| Lens Cleaning Paper | Any Vendor | ||
| Plates, 96-well PCR, Semi-Skirted | Any Vendor | ||
| Tube strips, 8-well, 0.2 ml | Any Vendor | ||
| Agencourt AMPure XP Beads | Beckman Coulter | A63881 | |
| BioShake IQ or 3000-T elm | Bulldog Bio/Q.Instruments | 1808-0506/ 1808-0517 | |
| DropPlate96 S - LabChipDS | Caliper | 128876 | |
| DropPlate96 D - LabChipDS | Caliper | 132848 | |
| DropSense96 | Caliper (Trinean) | ||
| DropQuant Software | Caliper (Trinean) | ||
| Plate Sealing Film | Denville | B1212-5S | |
| Aluminum Seal Foil | Denville | B1212-6S | |
| Nuclease-Free, Pure Water System | EMD Millipore | ||
| 5424 centrifuge | Eppendorf | 22621408 | |
| 5804R centrifuge | Eppendorf | 22623508 | Both 15 ml tube and plate rotators, preferably a centrifuge that can go up to 2,500 x g. |
| Safe-Lock Tube 1.5 ml, Natural | Eppendorf | 22431021 | |
| 5 ml Tube, DNA LoBind Tube | Eppendorf | 30108310 | |
| 5430R Centrifuge | Eppendorf | 022620645 | Any plate rotator centrifuge will work |
| Hybex Microsample Incubator | Fisher Scientific | 1057-30-0 | |
| Hybex 0.2 ml Tube Block | Fisher Scientific | 1057-31-0 | |
| TruSeq Amplicon – Cancer Panel | Illumina | FC-130-1008 | 96 reactions |
| TruSeq Custom Amplicon | Illumina | PE-940-1011 | 96 reactions |
| TruSeq Custom Amplicon Index Kit | Illumina | FC-130-1003 | 96 Indices, 384 Samples |
| MiSeq Reagent Kit v3, 500 Cycles | Illumina | MS-102-3003 | |
| MiSeq Reagent Kit v2, 300 Cycles | Illumina | MS-102-2002 | |
| MiSeq Reagent Kit v2, 500 Cycles | Illumina | MS-102-2003 | |
| Experiment Manager | Illumina | 1.3 or higher | |
| MiSeq Reporter | Illumina | 2.0 or higher | |
| Sequencing Analysis Viewer | Illumina | 1.8 or higher | |
| TruSeq Index Plate Fixture and Collar Kit | Illumina | FC-130-1007 | |
| MiSeq v2 | Illumina | SY-410-1003 | |
| TruSeq Custom Amplicon Filter Plate | Illumina | FC-130-1006 | |
| Index Adapter Replacement Caps | Illumina | 11294657 | |
| Qubit 2.0 | Invitrogen | Q32866 | |
| Qubit 0.5 ml Tubes | Invitrogen | Q32856 | |
| Qubit dsDNA Broad Range Assay Kit | Invitrogen | Q32853 | |
| DynaMa6-96 Magnetic Stand, Side Skirted | Invitrogen | 120.27 | |
| GeneAmp PCR System 9700 (gold/silver block) | Life Technologies | N8050200 | |
| Gentra Puregene Blood Kit | Qiagen | 158489 | |
| Deparaffinization Solution (16 ml) | Qiagen | 19093 | |
| Buffer ATL (4 x 50ml) | Qiagen | 939011 | |
| Protein Precipitation Solution (50 ml) | Qiagen | 158910 | |
| DNA Hydration Solution (100 ml) | Qiagen | 158914 | |
| Glycogen Solution (500 μl) | Qiagen | 158930 | |
| Qiagen Proteinase K | Qiagen | 19133 | |
| Rnase (5 ml) | Qiagen | 158924 | |
| Nuclease-Free Water (10 x 50 ml) | Qiagen | 129114 | |
| Pestles | USA Scientific | 1415-5390 | |
| TipOne RPT 10 µl elongated filter pipet tips in sterilized racks, 10 racks of 96 tips (960 tips). | USA Scientific | 1180-3810 | |
| TipOne RPT 100 µl natural, beveled filter pipet tips in sterilized racks, 10 racks of 96 tips (960 tips) | USA Scientific | 1180-1840 | |
| TipOne RPT 200 μl natural, beveled filter pipet tips in racks, sterilized racks, 10 racks of 96 tips (960 tips) | USA Scientific | 1180-8810 | |
| TipOne RPT 20 μl natural, beveled filter pipet tips in racks, sterilized racks, 10 racks of 96 tips (960 tips) | USA Scientific | 1180-1810 | |
| TipOne RPT 1,000 μl natural, graduated XL filter pipet tips in | USA Scientific | 1182-1830 |
참고문헌
- Gerlinger, M., et al. Intratumor heterogeneity and branched evolution revealed by multiregion sequencing. N Engl J Med. 366 (10), 883-892 (2012).
- Campbell, P. J., et al. The patterns and dynamics of genomic instability in metastatic pancreatic cancer. Nature. 467 (7319), 1109-1113 (2010).
- Ding, L., et al. Clonal evolution in relapsed acute myeloid leukaemia revealed by whole-genome sequencing. Nature. 481 (7382), 506-509 (2012).
- Frampton, G. M., et al. Development and validation of a clinical cancer genomic profiling test based on massively parallel DNA sequencing. Nature Biotechnol. 31 (11), 1023-1031 (2013).
- Patel, J. P., et al. Prognostic relevance of integrated genetic profiling in acute myeloid leukemia. N Engl J Med. 366 (12), 1079-1089 (2012).
- Forbes, S. A., et al. COSMIC (the Catalogue of Somatic Mutations in Cancer ): a resource to investigate acquired mutations in human cancer. Nucleic Acids Res. 38 (Database Issue), 652-657 (2010).
- Shih, A. H., Abdel-wahab, O., Patel, J. P., Levine, R. L. The role of mutations in epigenetic regulators in myeloid malignancies. Nat Rev Cancer. 12 (9), 599-612 (2012).
- Liersch, R., Müller-Tidow, C., Berdel, W. E., Krug, U. Prognostic factors for acute myeloid leukaemia in adults - biological significance and clinical use. Br J Haematol. 165 (1), 17-38 (2014).
- Bacher, U., Schnittger, S., Haferlach, T. Molecular genetics in acute myeloid leukemia. Curr Opin Oncol. 22 (6), 646-655 (2010).
- Subramanian, J., Govindan, R. Lung cancer in "Never-smokers": a unique entity. Oncology (Williston Park). 24 (1), 29-35 (2010).
- Sakashita, S., Sakashita, M., Tsao, M. S. Genes and pathology of non-small cell lung carcinoma. Semin Oncol. 41 (1), 28-39 (2014).
- Arcila, M. E., Chaft, J. E., Nafa, K. Prevalence clinicopathologic associations, and molecular spectrum of ERBB2 (HER2) tyrosine kinase mutations in lung adenocarcinomas. Clin Cancer Res. 18 (18), (2012).
- Gandhi, L., et al. Phase I study of neratinib in combination with temsirolimus in patients with human epidermal growth factor receptor 2-dependent and other solid tumors. J Clin Oncol. 32 (2), 68-75 (2014).
- Sheikhha, M. H., Awan, A., Tobal, K., Liu Yin, J. A. Prognostic significance of FLT3 ITD and D835 mutations in AML patients. Hematol J. 4 (1), 41-46 (2003).
- Mazières, J., et al. Lung cancer that harbors an HER2 mutation epidemiologic characteristics and therapeutic perspectives. J Clin Oncol. 31 (16), 1-8 (2014).
- Robinson, J. T., et al. Integrative Genomics Viewer. Nat Biotechnol. 29 (1), 495-500 (2011).
- Forbes, S. A., et al. COSMIC: exploring the world's knowledge of somatic mutations in human cancer. Nucleic Acids Res. 43 (Database issue), D805-D811 (2014).
- Daber, R., Sukhadia, S., Morrissette, J. J. Understanding the limitations of next generation sequencing informatics, an approach to clinical pipeline validation using artificial data sets. Cancer Genetics. 206 (12), 441-448 (2013).
- Haraksingh, R. R., et al. Genome-Wide Mapping of Copy Number Variation in Humans: Comparative Analysis of High Resolution Array Platforms. PLoS ONE. 6 (11), e27859(2011).
- de Leeuw, N., et al. SNP Array Analysis in Constitutional and Cancer Genome Diagnostics - Copy Number Variants, Genotyping and Quality Control. Cytogenet Genome Res. 135 (3-4), 212-221 (2011).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유