Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Использование одной молекулы флуоресцентные в гибридизации Situ (SM-рыбы) для Quantify и Localize mRNAs в мышиных ооцитов
В этой статье
Резюме
Можно воспроизвести подсчитать количество мРНК в отдельных ооцитов, одной молекулы РНК флуоресцирования на месте гибридизации (РНК-рыбы) был оптимизирован для non сторонник клеток. Ооциты были собраны, гибридизированных конкретных датчиками Стенограмма и количественно с помощью программного обеспечения количественной оценки изображения.
Аннотация
Нынешние методы, обычно используемые для количественного определения мРНК в яйцеклеток и эмбрионов включают цифровые реверс транскрипция полимеразной цепной реакции (dPCR), количественные, реальном времени RT-PCR (RT-ПЦР) и РНК последовательности. Когда эти методы выполняются с помощью одной яйцеклетки или эмбриона, низкий копия mRNAs надежно не обнаруживаются. Для преодоления этой проблемы, яйцеклеток или эмбрионов могут быть объединены вместе для анализа; Однако это часто приводит к высокой изменчивости среди образцов. В этом протоколе мы описывают использование флюоресценции в гибридизации situ (рыба) с помощью разветвленной ДНК химия. Этот метод определяет пространственное распределение mRNAs в отдельных клетках. Когда техника связан с местом нахождения и отслеживания программного обеспечения, обилие mRNAs в ячейке также может быть определена количественно. Используя эту технику, есть снижение изменчивости в экспериментальной группе и меньше яйцеклеток и эмбрионов обязаны обнаружить существенные различия между экспериментальной группы. Коммерчески доступные разветвленной ДНК SM-рыба наборы были оптимизированы для выявления mRNAs в секционного тканей или сторонником клетки на слайдах. Однако ооциты не эффективно придерживаться слайды и некоторые реагенты из комплекта были слишком тяжелыми, результате лизис ооцитов. Для предотвращения этого лизиса, некоторые изменения были внесены комплектом рыбу. В частности ооцитов permeabilization и мыть буферы предназначены для иммунофлюоресценции яйцеклеток и эмбрионов вместо несвободных буферов. Permeabilization, моет и инкубаций с датчики и усилители выполнены в 6-ну пластины и ооциты были размещены на слайды в конце протокола, с помощью установки средств массовой информации. Эти изменения были в состоянии преодолеть ограничения, коммерчески доступных комплект, в частности, лизис ооцитов. Для герметизации и точно подсчитать количество мРНК в отдельных ооцитов, компьютерного программного обеспечения был использован. Вместе этот протокол представляет собой альтернативу ПЦР и последовательности для сравнения выражения конкретных протоколов в одиночных клетках.
Введение
Обратной транскриптазы полимеразной цепной реакции (ПЦР) был золотым стандартом для мРНК quantitation. В настоящее время используются два анализов, цифровой ПЦР (dPCR)1 и количественных, реальное время ПЦР (ПЦР)2 . Из двух методов ПЦР dPCR имеет большую чувствительность, чем ПЦР предполагая, что он может использоваться для измерения mRNA изобилия в одиночных клетках. Однако в наших руках, dPCR анализ низкого залегания mRNAs в бассейнах 5 до 10 яйцеклеток за каждый экспериментальный образец выпустила данных с низкой воспроизводимостью и высокий вариант3. Это, вероятно, из-за экспериментальной ошибки, связанной с РНК добыча и эффективности обратной транскрипции. РНК последовательности также была выполнена с помощью мыши и человеческих яйцеклеток4,5. Этот метод требует cDNA амплификации шаги, необходимые для библиотеки поколения, которое вероятно увеличивает изменчивость в экспериментальной группе. Кроме того низкие изобилии стенограммы не может быть обнаружено. Хотя последовательности цены снизились за последние несколько лет, это может быть дорого из-за высокой стоимости биоинформатики анализов. Наконец мРНК Локализация — это динамичный процесс с пространственных изменений, способствующих белков функции6. Таким образом мы намереваемся принять метод, который будет производить точные и воспроизводимые количественные показатели и локализации индивидуальных mRNAs в одно ооцитов.
Разветвленный ДНК, в сочетании с флуоресценции в гибридизации situ усиливает сигнал флуоресценции, вместо того, чтобы усилительных благоприятных обнаружение РНК/кДНК одной мРНК в отдельных клетках 7,8,9. Генотипирования через серию гибридизации, амплификация (с помощью разветвленной ДНК) и флуоресценции маркировки шаги для того, чтобы усилить флуоресценции сигнала7. Этот метод начинается с привязки 18 - 25-base олигонуклеотида зонд пар, которые дополняют конкретные мРНК3,8,10. Пятнадцати до двадцати зонд пары предназначены для обеспечение специфику каждого Стенограмма для целевого транскрипт. МРНК конкретных гибридизации следуют предварительный усилитель и усилитель зондами, которые формируют разветвленную конфигурации. Приблизительно 400 лейбл флуорофоров привязку каждого усилителя, что привело к увеличению 8000-fold в флуоресцировании, позволяя обнаружения индивидуальных mRNAs (рис. 1)11.
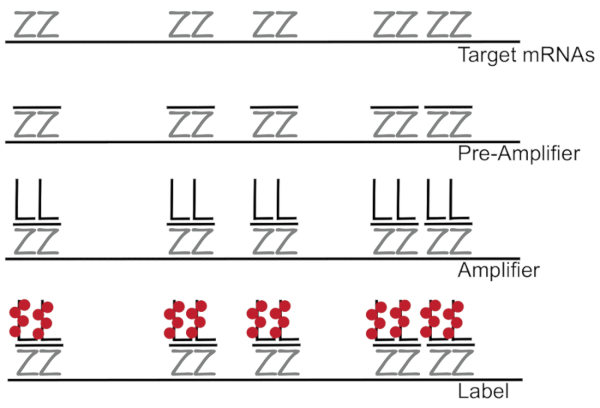
Рисунок 1: Схема протокола SM-рыба. Последовательные гибридизации Стенограмма конкретного ПЭП, разветвленной ДНК усилитель и Флюорофор к цели, которую показана mRNA. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Предыдущие исследования с использованием одной молекулы флуоресценции в situ гибридизация (SM-рыбы) локализованные β-актина mRNAs в12 отдельных нейронов и ДНК вируса папилломы человека в рак шейки матки клеток линии7. Компьютерное программное обеспечение, месте нахождения и отслеживания Программа идентифицирует отдельных пунктата флуоресцентного сигнала и успешно используется для количественного определения количество мРНК в каждой ячейке3,13.
Основываясь на результаты обнаружения мРНК в нейроны12, мы предположили, что SM-рыб окажется полезным инструментом для quantitate Стенограмма уровней в мышиных яйцеклеток и эмбрионов, включая низкие изобилии мРНК. Однако техника оптимизирована для использования с фиксированной адэрентных клеток и формальдегида фиксированной парафин встроенных разделов ткани (FFPE). Ооциты не может присоединиться к слайду, даже когда они покрыты с поли-L-лизин. Кроме того они являются более хрупкие, чем соматических клетках и результате лизис клеток когда подвергаются некоторые несвободные буферов в коммерчески доступные комплекты3разделах ткани. Для преодоления этих проблем, ооциты фиксировали и вручную переведен между капли буферов. Кроме того permeabilization и мыть буферов в наборы были заменены уменьшить lysis клетки. Встроенные датчики приобретаются вместе с комплектом рыбу или конкретных протоколов могут запрашиваться. Каждого собственности зонда набор доступен в одном из трех каналов флуоресцирования (C1, C2 и C3) для мультиплексирования. В ходе текущего эксперимента мышиных ооциты были двойной окрашенных и количественных, используя зонд Nanog C2 и C3 Pou5f1 зонда. Эти зонды были отобраны на основе сообщенных выражения Nanog и Pou5f1 яйцеклеток и эмбрионов. В заключение гибридизации шаги ооциты были помещены в капли анти затухания монтажа СМИ для приложения гистологические слайды. Конфокальный изображения были использованы для количественного определения количество пунктата флуоресцентные сигналов, которые представляют индивидуальных mRNAs. Помимо количественного мРНК, изображений также показал пространственное распределение конкретные мРНК в ячейке, какие другие методы количественного определения РНК не в состоянии достичь. Этот метод оказался низким изменчивости в пределах экспериментальной группы, что позволяет использовать меньшее количество яйцеклеток в каждой экспериментальной группе выявить существенные различия между экспериментальной группы3.
Access restricted. Please log in or start a trial to view this content.
протокол
Животных процедуры были рассмотрены и одобрены институциональный уход животных и использования Комитетом в университете штата Небраска-Линкольн и все методы были исполнены в соответствии с соответствующих руководящих принципов и правил. Для этого исследования, Беспородные CD-1 мышей были ad libitum доступ к нормальной грызунов Чоу и воды; они были сохранены в 12:12 темно: свет цикла.
1. Подготовка необходимых средств массовой информации
- Для базового СМИ (OMM) добавьте 100 мм NaCl, 5 мм KCl, 0.5 мм х2PO4и 1,7 мм CaCl2-2 H2O 100 мл стерильной воды.
Примечание: OMM среднего может храниться до 1 месяца. - Для полной СМИ (OMOPS) добавьте 20 мм 3-morpholinopropane-1-сульфокатиониты кислота (МОПЫ), 1,2 мм MgSO4-7 H2O, глюкоза 0,5 мм, 6 мм L-лактат, Ала gln 1 мм, 0,1 мм таурина, 1 x несущественные аминокислот (NEAA), 0,01 мм Этилендиаминтетрауксусная кислота ( ЭДТА), Альфа-липоевая кислота 10 мкм, 10 мкг/мл неразбавленном виде гентамицина, 21 мм 1 M NaOH, 5 мм NaHCO3, 0,2 мм пируват, 0.5 мм цитрат, 4 мг/мл FAF BSA до 1:10 разрежения ОММ в стерильной воде для суммарный объем 100 мл. Стерилизуйте среды с 0,22 мкм фильтром.
Примечание: OMOPS может храниться до 1 недели. - Для проведения среднесрочной (HM) добавьте плода бычьим сывороточным 5% OMOPS. Сделайте 2 мл HM в мышь.
- Гиалуронидаза решения добавьте 0,1 мг/мл гиалуронидазы, производный от семенников крупного рогатого скота, на 1 мл HM.
- Для фиксации буфера объединить параформальдегида 4% 10 мл ПБС наряду с 0,1% эмбрионов класс поливинилпирролидона (ПВП)14.
- Для приготовления 50 мл мыть буфера (ВБ), добавьте неионных ПАВ 0,1% и 0,1% PVP 1 x PBS14.
- Чтобы подготовить 10 мл permeabilization буфер добавьте 1% не - ионных ПАВ PBS 1 x14.
Примечание: Мыть и permeabilization буферов, описанные выше заменить проприетарные буферов в коммерчески доступные наборы.
2. сбор овуляцией ооциты от самок мышей
- Подготовка:
- Стимулировать самок мышей в возрасте 5-8 недель внутрибрюшинной инъекции (IP) лошадей хорионического гонадотропина 5 МЕ (ЭКГ) следуют 5 МЕ хорионического гонадотропина человека (ХГЧ) 44-48 h позднее15,16.
- Держите Петри 35 мм, содержащие 2 мл HM на 37 ° C потепления тарелку. Пипетка 1, 100 мкл капля HM, содержащих разреженных гиалуронидазы, следуют три капли 50 мкл HM без гиалуронидазы в чашке Петри 60 мм. Поместите пластины, содержащий капли на 37 ° C потепления пластины перед использованием.
Примечание: Гиалуронидаза капли должны быть сделаны только до рассечение каждой пары маточных труб для предотвращения испарения и концентрации компонентов HM с или без гиалуронидазы.
- Усыпить мышей, 16 ч после инъекции ХГЧ IP, используя изофлюрановая передозировки следуют шейки матки дислокации.
- Почистите мышь с помощью 70% этиловом спирте. Разоблачить брюшной полости и визуализировать женского репродуктивного тракта. Держите яичника с щипцами и удалить маточных связок и избыток жировой ткани из вокруг яичника. Вырезать маточных труб из матки и пара яичников-маточных труб в теплой HM в блюдо 35 мм.
- Удаление яичников и любой окружающие жировой ткани. Рвать опухшие ампула маточных труб, с помощью иглы 27-калибруйте ½ дюйма. Нажмите маточных труб на месте разрыва и кучевые клеток ооцитов комплексы (КОК) будет исключена. Передача овуляцией ооцитов, которые считаются в метафазы II (MII) мейоз, падение 100 мкл, содержащие HM СМИ с гиалуронидазы, используя рот пипеткой (рис. 2).
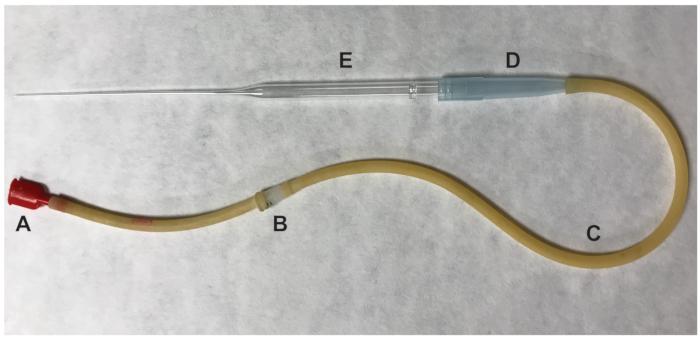
Рисунок 2 : Части рот дозаторов, используемых для переноса ооцитов. (A) рот кусок (B) 0,22 мкм, 4 мм фильтр (C) Аспиратор труб (D) 1000 мкл накапайте наконечник (E) 9" Pasteur накапайте. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
- Пипетка MII яйцеклетки кучевые клеток комплексы вверх и вниз в гиалуронидазы, содержащие HM с рот пипеткой выбить кучевые клеток. Перевод каждого ооцитов, после того, как они лишены кучевые клетки к мыть падение содержащие HM, используя только рот пипеткой. Повторите это действие для каждой капли мыть. Не передавать фрагментирован или прозрачный ооциты15.
Примечание: Важно передать яйцеклеток из каждой капли в 35 мм блюдо с как мало HM как можно скорее. Это верно для каждой передачи в протоколе. MII ооциты не должны оставаться в гиалуронидазы, содержащие HM среднего для более чем одной минуты.
3. SM-FISH окрашивания яйцеклеток
- Исправьте яйцеклеток в индивидуальных хорошо 6-ну пластины, содержащий 500 мкл буфера фиксации. Потопить 20 яйцеклеток или менее в колодец. Проинкубируйте втечение 20 мин при комнатной температуре.
Примечание: Каждый шаг SM-рыба окрашивание происходит в течение отдельных хорошо в 6-ну конические пластине. Обеспечить полностью погруженным в буферы и не плавающей поверх буфера ооцитов. Каждый шаг следует выполнять с 20 яйцеклеток или меньше, в каждой скважине. - Передавать фиксированной ооциты 500 мкл буфера мытья (ВБ, описанный в шаге 1.6) за 10 мин. Повторите 2 раза.
- Инкубируйте яйцеклеток в буфер permeabilization за 30 мин при комнатной температуре.
Примечание: В буфер permeabilization, описанный в шаге 1.7 заменяет буфер permeabilization приличия.- Соберите наборы зонд и быстро вращаться их вниз в microcentrifuge. Теплый каждый зонд для 10 мин в водяной бане 40 ° C или инкубатора. Охладите до комнатной температуры.
Примечание: Этот шаг должен выполняться во время инкубации permeabilization
- Соберите наборы зонд и быстро вращаться их вниз в microcentrifuge. Теплый каждый зонд для 10 мин в водяной бане 40 ° C или инкубатора. Охладите до комнатной температуры.
- Вымойте яйцеклеток в 500 мкл ВБ 10 мин при комнатной температуре.
- Ооциты передать 80 мкл протеазы III буфера (доступно из комплекта), что разбавленный 1:8 в 1 X PBS, за 30 мин при комнатной температуре.
Примечание: Объем 80 мкл адекватно охватывает в нижней части отдельных хорошо в пластине 6-хорошо. - Вымойте яйцеклеток в 500 мкл ВБ 10 мин при комнатной температуре.
- Разбавить утепленные зонд наборы для Nanog, Pou5f1 и DapB (отрицательный контроль ген), 1:50 в зонд разбавителя. Инкубировать яйцеклеток в 80 мкл Стенограмма конкретной зонда для 2 часов при 40° C.
Примечание: Каждый собственности зонда набор доступен в одном из трех каналов флуоресцирования (C1, C2 и C3). Nanog и Pou5f1 зонды были помечены с C2 и C3, соответственно. - Теплый проприетарная, усилитель 1 (AMP 1), 2 (AMP2), усилитель усилитель 3 (AMP3) и усилитель 4-флуоресценции (AMP 4-ФЗ) при комнатной температуре.
Примечание: Этот шаг должен выполняться во время Стенограмма конкретных зонд 2-часовой инкубации. - Передать 500 мкл ВБ яйцеклеток и проинкубируйте втечение 10 мин при комнатной температуре.
- Инкубируйте ооциты последовательно в буферах амплификации.
- Инкубируйте яйцеклеток в 80 мкл AMP1 за 30 мин при 40° C. переноса ооцитов до 500 мкл ВБ 10 мин при комнатной температуре.
- Инкубировать яйцеклеток в 80 мкл AMP2 15 мин при 40 ° C. Переноса ооцитов 500 мкл ВБ 10 мин при комнатной температуре.
- Инкубировать яйцеклеток в 80 мкл AMP3 за 30 мин при 40 ° C. Переноса ооцитов 500 мкл ВБ 10 мин при комнатной температуре.
Примечание: Остальная часть протокола осуществляется в темноте, потому что AMP-ФЗ содержит Флюорофор. При работе под микроскопом рассечения, сократить как можно больше света. - Добавьте ооциты 80 мкл AMP4-FL 15 мин при 40° C.
Примечание: AMP4-FL предоставляется в качестве альтернативы буфера A (Alt-A), Alt-B или Alt-C. Выберите AMP4-FL буфера зависимые на выбросов желаемого волны.
- Вымойте яйцеклеток в 500 мкл ВБ 10 мин при комнатной температуре. Инкубируйте яйцеклеток в 80 мкл DAPI 20 мин при комнатной температуре. Вымойте яйцеклеток в 500 мкл ВБ на 5 мин при комнатной температуре.
- Пипетка 12 мкл СМИ против выцветания монтажа на центр слайд без добавления реагента пузыри. Передача яйцеклеток с как мало ВБ максимально в СМИ монтажа и применить coverslip.
- Coverslip с углом наклона и медленно и осторожно поместите жидкость на слайде. Избегайте нажатия coverslip слишком сложно, чтобы предотвратить искажение ооцитов и введение пузырей.
- Храните слайды в темной коробке для высыхания на ночь при комнатной температуре. Герб края слайды в ясно Лак для уплотнения coverslip.
- Используйте стандартный Микроскоп найти яйцеклеток на слайд и круг с постоянным маркером.
Примечание: Этот шаг не является обязательным, но улучшает поиск яйцеклеток на слайде. Для достижения наилучших результатов слайды изображений в течение 1-5 дней как флуоресцентные сигнал начнут исчезать.
4. обработка изображений
- Изображение 3-мерной ооцитов, используя z шаг confocal микроскопии.
Примечание: Для точного анализа изображения, каждый шаг z должна быть 1.0 мкм/срез. - Сохраните конфокальный изображения, как сжатый nd2 или индивидуального. TIFF-файлы для каждого ооцитов. Оба изображения типы совместимы с открытым исходным кодом изображения программа обработки, Фиджи.
- Скачать и установить программное обеспечение открытого доступа Фиджи (https://imagej.net/Fiji/Downloads).
- Перетащите файлы nd2 в Фиджи и выберите hyperstack. Если конфокальный изображения были сохранены как. Файлы TIFF перейдите к шагу 4.4.
Примечание: При падении nd2 файл в Фиджи раскрывающемся списке hyperstack должен появиться автоматически. - Перейдите на вкладку изображение , выберите Цвети нажмите кнопку Разделить каналы для разделения флуоресцентные каналы nd2 файла.
- Создание индивидуальных. TIFF-файлы для каждого z ломтик яйцеклетки в каждом канале, флуоресцентные. Перейдите на вкладку изображение , выберите стекии нажмите Стек изображений. Перейдите на вкладку изображение , выберите типи нажмите кнопку Цвет RGB для преобразования каждый фрагмент z в отдельное изображение цвета RGB.
Примечание: Цвет RGB является искусственным и может быть выбран как желаемого для каждой длины волны выбросов. - Сохраните каждое преобразованное изображение как. TIFF-файл. Место изображения из одной яйцеклетки для каждого флуоресцентные канала в новую папку, чтобы избежать путаницы во время шитья (шаг 4.3).
- Перетащите файлы nd2 в Фиджи и выберите hyperstack. Если конфокальный изображения были сохранены как. Файлы TIFF перейдите к шагу 4.4.
- Нормализовать каждый. Изображения в формате TIFF для Pou5f1 и Nanog с использованием отрицательного контроля изображения (DapB).
Примечание: Нормализация выполняется с помощью программы для редактирования фотографий. Убедитесь в том удалить тот же уровень фона флуоресценции из изображения каждого элемента управления. - Открытый каждый нормированный. TIFF-файл в Фиджи, чтобы сшить все z фрагменты для каждого яйцеклетки в каждой длины волны.
- Щелкните вкладку плагинов , колющиеи нажать на Коллекцию сетки (рис. 3A). Последовательные изображения выберите из раскрывающегося меню и нажмите кнопку ОК (рис. 3B).
- Просмотрите каталог и выберите папку, содержащую все z фрагмент изображения для отдельных яйцеклетки в одной длины волны (см. шаг 4.3.4). Нажмите кнопку ОК.
- Переместите ползунок в нижней части сшитые изображение в соответствующей цветовой канал для волны используется и создать сшитые изображение RGB, щелкнув изображение, выбрав типи нажмите кнопку Цвет RGB.
Примечание: Этот образ будет использоваться для флуоресценции количественный, описанный в шаге 4.6 ниже.
- Преобразуйте сшитые изображение 32-разрядных максимального прогнозируемого картинку. Нажмите на изображение, выберите типи нажмите кнопку 32-бит (рис. 3 c). Сохраните этот образ как новый. TIFF-файл.
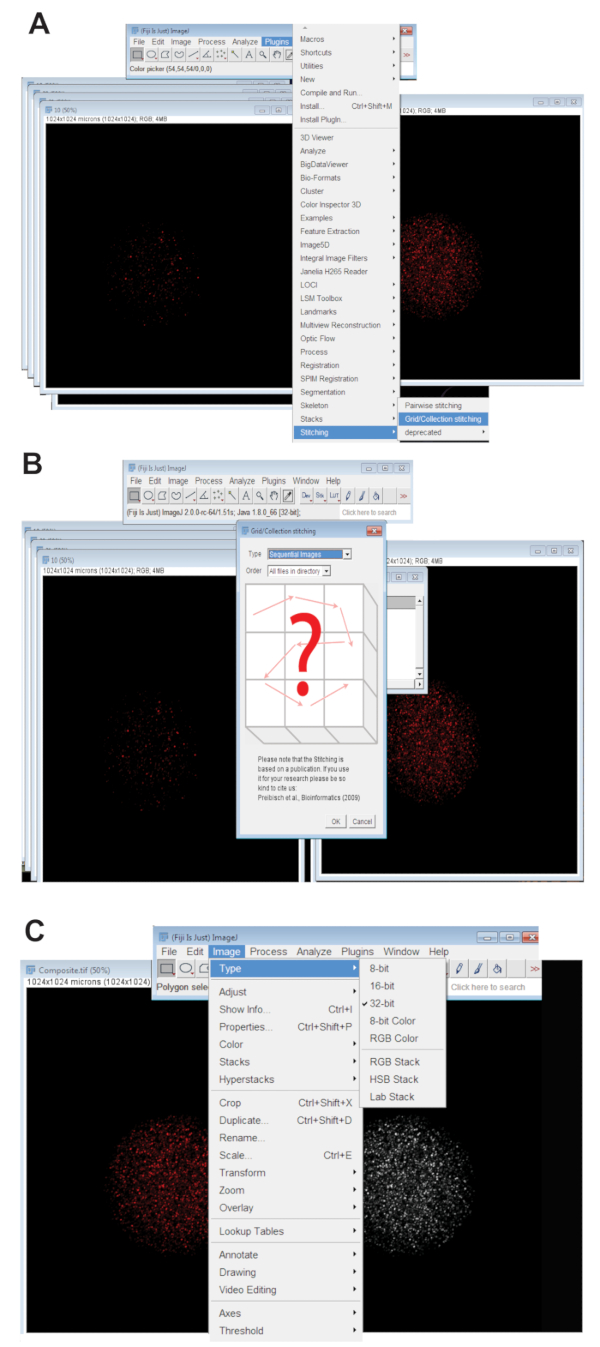
Рисунок 3 : Сшивая конфокальный z серии изображений яйцеклеток. (A) скриншот с указанием инструментом Plug-in сетки/коллекции в Фиджи, которая использовалась для создания композитных изображений ооцитов. (B) последовательных изображений использует флуоресценции дублирования между последовательным. TIFF-файлы для создания составного изображения. (C) составное изображение был сохранен как 32-разрядные. TIFF-файл. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
- Скачать и установить место нахождения и отслеживания программы13, который доступен на веб-сайте для д.р. Ларсон, следователь национальных институтов из здравоохранения Национального института рака (https://ccr.cancer.gov/Laboratory-of-Receptor-Biology-and-Gene-Expression/daniel-r-larson). Загрузить и установить виртуальную машину открытого доступа для интерактивных данных языка (IDL) операционной системы, которая требуется для запуска местом поиска и отслеживания программа (http://www.spacewx.com/pdf/idlvm.pdf).
- Откройте 32-бит, сшитые изображение, который был создан в шаге 4.6 (рис. 4A), в месте нахождения и отслеживания программы. Выберите Localize раскрывающегося списка и нажмите Localize (рис. 4B), которая будет вычислять число пятен, найденный в образе.
Примечание: Каждое пятно насчитал представляет индивидуальный мРНК. Группа пасс и Фотон порог параметры показаны на рисунке (рис. 4В). Для этого протокола используется по умолчанию для каждого параметра порог. Представитель позитивные и пятен фоновый показаны (рис. 4 c).

Рисунок 4 : Количественная оценка мРНК, используя Finder пятно и отслеживания. (A) отдельных z серии изображений были сшиты вместе, как описано в рисунке 3 и сохранены как прогнозируемые максимум 32-разрядных. TIFF-файл. (B) составное изображение был открыт в месте Finder и отслеживания. Локализация была использована для подсчета флуоресцентные пятна (красный прямоугольник). Группа пасс и Фотон порог обозначается синей коробке. (C) голубая стрелка указывает на позитивный сигнал (выше порога). Белая стрелка показывает флуоресцентные пятна ниже порога и, следовательно, не учитываются. Пожалуйста, нажмите здесь, чтобы посмотреть большую версию этой фигуры.
Access restricted. Please log in or start a trial to view this content.
Результаты
После завершения протокола, результат будет отдельных изображений из конфокальный z серии (рис. 4а и 5), склеенные изображения (рис. 4 c), и рассчитывает мРНК (рис. 4B). Когда выполняе...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Ряд мелких шагов во время протокол обеспечит успешное флуоресценции и точное количество мРНК. Во-первых протокол должны быть выполнены сразу после сбора и фиксации яйцеклеток. Обратите внимание, что PVP добавляется в буфер фиксации параформальдегида 4% для предотвращения ооциты от прил?...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторы не имеют ничего объявить
Благодарности
Мы благодарим д-р Даниэль р. Ларсон за его щедрую помощь с установки и использования местом поиска и отслеживания программы 13 и технической поддержке Университета Небраска Линкольн микроскопии Core для воображения confocal микроскопии. Это исследование представляет собой вклад Университета Небраска сельскохозяйственных исследований отдела, Линкольн, штат Небраска и была поддержана UNL Люк фондов (NEB-26-206/присоединения номер-232435 и НЭБ-26-231/присоединения номер-1013511).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
Ссылки
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930(2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , https://www.biotek.com/resources/application-notes/multiplexed-detection-of-cytokine-cancer-biomarkers-using-fluorescence-rna-in-situ-hybridization-and-cellular-imaging/ 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4(2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459(2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, Elsevier Inc. (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855(2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15(2003).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены