JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Murine Oocytes에 계량 및 방화 mRNAs 단일 분자 형광에서 제자리 교 잡 (SM-물고기)의 사용
요약
Reproducibly 개별 oocytes, 단일 분자 RNA 형광 제자리에서 mRNAs의 숫자를 카운트를 교 잡 (RNA-물고기) 비 부착 한 세포에 대 한 최적화 되었다. Oocytes 수집 했다, 대 본 특정 프로브 때 교배 하 고 이미지 정량화 소프트웨어를 사용 하 여 계량.
초록
정기적으로 oocytes에서 배아 mRNA를 측정 하는 데 사용 하는 현재 방법 디지털 역 전사 연쇄 반응 (dPCR), 양적, 실시간 RT-PCR (RT-정량) 및 RNA 시퀀싱 포함 됩니다. 이러한 기술은 단일 oocyte 또는 배아를 사용 하 여 수행 됩니다, 낮은 복사 mRNAs 안정적으로 검색 되지 않습니다. 이 문제를 해결 하려면 oocytes 또는 배아 수 수 풀링된 함께 분석; 그러나,이 종종 샘플 중 높은 가변성에 이어집니다. 이 프로토콜에서 우리 형광 제자리 교 잡 (물고기) 분기 DNA 화학을 사용 하 여의 사용을 설명 합니다. 이 기술은 개별 셀에서 mRNAs의 공간적 패턴을 식별합니다. 기술은 자리 찾아서 추적 컴퓨터 소프트웨어와 결합 되어 때 셀에서 mRNAs의 풍부도 측정할 수 수 있습니다. 이 기술을 사용 하 여, 실험적인 그룹에서 감소 변화 이며 적은 oocytes 및 배아 실험 그룹 간의 중요 한 차이 검출 해야. 상업적으로 사용 가능한 분기 DNA SM-물고기 키트 sectioned 조직 또는 슬라이드에 부착 세포 mRNAs를 검색에 최적화 되었습니다. 그러나, oocytes 효과적으로 슬라이드에 고착 하지 않는다 고 일부 시 약 키트에 oocyte 세포의 결과로 너무 가혹 했다. 이 세포의 용이 해를 방지 하기 위해, 물고기 키트 여러 수정 되었다. 특히, oocyte permeabilization 및 워시 버퍼를 oocytes와 태아의 면역 형광을 위한 독점 버퍼를 교체 했습니다. Permeabilization, 세척, 그리고 프로브와 앰프 외피 6 잘 플레이트에서 수행한 고 oocytes 슬라이드 설치 미디어를 사용 하 여 프로토콜의 끝에 배치 했다. 이러한 수정 상용 키트, 특히, oocyte 세포의 한계를 극복할 수 있었다. 정확 하 게 그리고 reproducibly 개별 oocytes에서 mRNAs의 수를 계산, 컴퓨터 소프트웨어 사용 되었다. 함께,이 프로토콜 PCR 및 시퀀싱을 비교할 단일 셀에 특정 성적 표현 하는 대안을 나타냅니다.
서문
역전사 중 합 효소 연쇄 반응 (PCR) mRNA 정량에 대 한 황금 표준 되었습니다. 2 분석 실험, 디지털 PCR (dPCR)1 과 양적, 리얼 타임 PCR (정량)2 현재 사용 됩니다. 두 개의 PCR 기법, dPCR 그것은 단일 세포에서 mRNA 풍부를 측정 하는 데 사용할 수 제안 하는 정량 보다 더 큰 감도 있다. 그러나, 우리 손에 각 실험 샘플 당 5 ~ 10 oocytes의 수영장에서 낮은 풍부 mRNAs의 dPCR 분석은 생산 데이터 낮은 재현성 및 높은 변화3. 이 RNA 추출 및 반전 녹음 방송 효율성과 관련 된 실험적인 오류로 인해 높습니다. RNA 시퀀싱도 하나의 마우스와 인간의 oocytes4,5를 사용 하 여 수행 되었습니다. 이 기술은 가능성이 실험적인 그룹 내의 다양성을 증가 하는 라이브러리 생성에 필요한 cDNA 증폭 단계를 해야 합니다. 또한, 낮은 풍부 성적표 감지 하지 않을 수 있습니다. 시퀀싱 가격은 지난 몇 년 동안 추락, 비록 그것은 여전히 비용 생물 정보학 분석의 높은 비용 때문에 금지 수 있습니다. 마지막으로, mRNA 지역화 단백질 기능6에 기여 하는 공간 변경 동적 과정 이다. 따라서, 우리는 정확 하 고 재현 가능한 정량적 측정 및 단일 oocytes에서 개별 mRNAs의 지역화 생성 하는 기술 채택에 밖으로 설정.
분기 DNA 형광 제자리 교 잡에서에 결합 된 개별 셀 7,,89단일 mRNAs의 증폭 RNA/cDNA 활성화 탐지 보다는 형광 신호를 증폭 한다. 분석 결과 교 잡, 증폭 (분기 DNA를 사용 하 여), 및 형광 형광 신호7증폭 하기 위하여 단계를 라벨의 시리즈를 통해 수행 됩니다. 기술은 특정 mRNA3,,810를 보완 하는 18-25 베이스 oligonucleotide 조사 쌍의 바인딩으로 시작 합니다. 15 ~ 20 프로브 쌍 대상 성적에 대 한 각 사본 보장 특이성에 대 한 설계 되었습니다. MRNA 전용 하이브리드 프리 앰프 및 증폭기 프로브 분기 구성 구성 하 옵니다. 약, 400 라벨 fluorophores 개별 mRNAs (그림 1)11의 탐지를 허용 하는 형광에 8000-fold 증가 결과로 각 증폭기에 바인딩합니다.
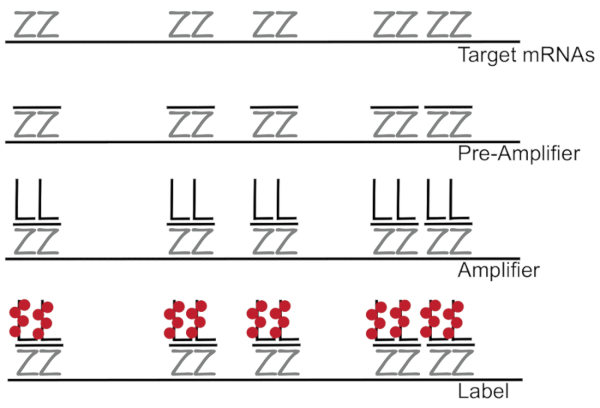
그림 1: SM 물고기 프로토콜의 도식. 대 본 특정 조사의 순차적 교 잡 DNA 증폭기 및 mRNA 표시 대상에 fluorophore 분기. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
개별 뉴런12 에 인간 유 두 종 바이러스 DNA에 자 궁 경부 암 제자리 교 잡 (SM-물고기) 지역화 된 β-말라 mRNAs에 있는 단일 분자 형광을 사용 하 여 이전 연구 라인7세포. 자리 찾기는 컴퓨터 소프트웨어 및 추적 프로그램 개별 punctate 형광 신호를 식별 하 고 각 셀3,13mRNAs의 수 척도를 성공적으로 사용 되었습니다.
신경12mRNA 검색의 결과 바탕으로, 우리 SM 물고기 quantitate murine oocytes에서 배아 낮은 풍부 mRNAs를 포함 한 성적 수준에 유용한 도구를 증명할 것을 가정 했다. 그러나, 기술은 부착 고정된 셀과 함께 사용 하기 위해 최적화와 포름알데히드 파라핀 고정 임베디드 (FFPE) 조직 단면도. Oocytes 그들은 폴 리-L-리 신을 코팅 하는 경우에 슬라이드를 준수 수 없습니다. 또한, 그들은 체세포와 직물 단면도 세포 세포의 용 해 상용 키트3독점 버퍼의 일부를 받게 될 때의 결과 보다 더 약 합니다. 이러한 과제를 극복 하기 위해 oocytes는 고정 되었고 수동으로 버퍼의 방울 사이 전송. 또한, 키트에 permeabilization 및 워시 버퍼 세포 세포의 용 해를 감소 시키기 위하여 대체 되었다. 미리 디자인 된 프로브 물고기 키트와 함께 구입 또는 특정 성적 증명서를 요청할 수 있습니다. 각 독자적인 프로브 세트는 3 개의 형광 채널 (C1, C2, 및 C3) 다중화 허용 중 하나에. 현재 실험에 murine oocytes 듀얼 스테인드과 C2 Nanog 프로브와 C3 Pou5f1 프로브를 사용 하 여 계량 했다. 이 프로브 oocytes에서 배아 Nanog 와 Pou5f1 의 보고 된 식에 따라 선정 됐다. 교 잡 단계의 결론에 oocytes 조직학 슬라이드에 응용 프로그램에 대 한 방지 페이드 설치 미디어의 방울에 배치 했다. 공초점 이미지 개별 mRNAs를 대표 하는 punctate 형광 신호 수 계량에 사용 되었다. 영상 또한 셀에 특정 mRNA의 공간적 분포를 보였다는 mRNAs를 측정 이외에 다른 RNA 정량화 방법 달성할 수 없습니다. 이 기술은 실험 그룹3사이의 중요 한 차이점을 식별 하기 위해 각 실험 그룹에 oocytes의 작은 숫자의 사용을 허용 하는 실험적인 그룹 내에서 낮은 다양성을 입증 했다.
프로토콜
동물 절차 검토 하 고 기관 동물 관리 및 사용 위원회 네브래스카 링컨의 대학에 의해 승인 했다 그리고 모든 방법을 관련 지침 및 규정에 따라 수행 했다. 이 연구에 대 한 CD-1 outbred 쥐 정상적 설치류 차 우와 물; ad libitum 접근 했다 그들은 어두운 12시 12분에 유지 되었다: 빛을 주기.
1입니다. 필요한 미디어의 준비
- 기본 미디어 (OMM), 살 균 물 100 mL를 100 mM NaCl, KCl 5mm, 0.5 m m KH2포4및 1.7 m m CaCl2-2 H2O를 추가 합니다.
참고: OMM 매체는 1 개월까지 저장할 수 있습니다. - 완전 한 미디어 (OMOPS)에 대 한 추가 20 m m 3-morpholinopropane-1-sulfonic 산 (MOPS), 1.2 m m MgSO4-7 H2O, 포도 당 0.5 m m, 6mm L-젖 산, 1mm ala-gln, 황소자리, 0.1 m m 1 x 비 본질적인 아미노산 (NEAA), 0.01 m m ethylenediaminetetraacetic 산 ( EDTA), 10 µ M 알파 리 포 산, 10 µ g/mL undiluted gentamicin, 21 m m 1 M NaOH, 5mm NaHCO3, 0.2 m m Pyruvate, 0.5 m m 구 연산, 1시 10분을 4 mg/mL FAF BSA OMM 100 mL의 전체 볼륨에 대 한 살 균 물에 희석. 0.22 μ M 필터 매체를 소독.
참고: OMOPS 최대 1 주일까지 저장할 수 있습니다. - 보유 매체 (HM)에 대 한 OMOPS를 5% 태아 둔감 한 혈 청을 추가 합니다. 마우스 당 2 mL HM을 확인 합니다.
- Hyaluronidase 솔루션 1 ml 흠 소 고환에서 파생 된 hyaluronidase의 0.1 mg/mL를 추가 합니다.
- 고정 버퍼에 대 한 4% paraformaldehyde 0.1% 태아 학년 polyvinylpyrrolidone (PVP)14함께 1 x PBS의 10 ml에서을 결합 합니다.
- 50ml 워시 버퍼 (WB)를 준비 하려면 0.1% 비 이온 계면 활성 제 및 0.1% 추가 PBS14x 1로 PVP.
- 준비 10 mL permeabilization 버퍼에 1 x PBS141% 비 이온 계면 활성 제를 추가 합니다.
참고: 위에서 설명한 세척 및 permeabilization 버퍼 상용 키트에서 독점 버퍼를 교체 합니다.
2. 여성 쥐에서 ovulated oocytes의 수집
- 준비:
- 나이의 5-8 주에 5 IU 말 chorionic 생식 샘 자극 호르몬 (eCG) 5 IU 인간의 융 생식 샘 자극 호르몬 (hCG) 44-48 h 뒤의 복 (IP) 주입에 의해 자극 하는 여성 쥐 나중15,16.
- 35 mm 접시 포함 하는 37 ° C 온난 화 접시에 HM의 2 개 mL를 유지. 하나 플라스틱, 흠 있는 100 µ L 방울 희석 hyaluronidase 3, 60 mm 페 트리 접시에 hyaluronidase 없이 HM의 50 µ L 상품 이어서. 37 ° C 온난 화 포함 상품 사용 전에 접시는 접시를 놓습니다.
참고: hyaluronidase 방울 여야 한다 각 수 란 관 쌍의 해 부 직전 증발 및 또는 hyaluronidase 없이 HM의 구성의 집중을 방지 하기 위해.
- 마우스, hCG의 IP 주사 후 16 h를 안락사, 자 궁 경부 전위 뒤 isoflurane 과량을 사용 하 여.
- 70% 에탄올을 사용 하 여 마우스를 청소 하십시오. 복 부 구멍을 노출 하 고 여성 생식 관을 시각화. 집게와 난소를 누른 자 궁 인 대 및 난소 주위에서 과잉 지방 조직을 제거 합니다. 자 궁에서 수 란 관 고 35 mm 접시에 따뜻한 HM에 난소 수 란 관 쌍을 놓습니다.
- 난소와 모든 주변의 지방 조직을 제거 합니다. ½ 인치 27 게이지 주사 바늘을 사용 하 여 수 란 관의 부 ampulla 눈물. 눈물의 사이트에는 수 란 관을 밀어 하 고 적 운 세포 oocyte 단지 (COCs) 퇴 학 될 것입니다. II (MII) HM 미디어 hyaluronidase 입 피 펫 (그림 2)를 사용 하 여 포함 하는 100 μ를 감수 분열의 분열에서 것으로 추정 되는 ovulated oocytes를 전송 합니다.
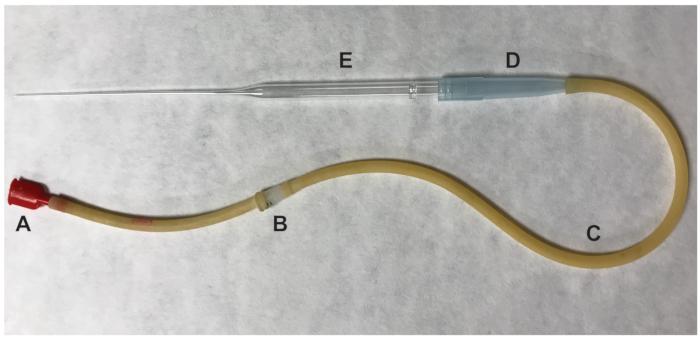
그림 2 : Oocytes를 전송 하는 데 사용 하는 입 pipettor의 부분. (A) 입 음, (B) 0.22 조각 4 m m 필터 (C) 흡 인기 튜브 (D) 1000 μ 피펫으로 팁 (E) 9"파스퇴르 피펫으로. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
- 적 운 세포를 꺼내 려 입 피 펫과 HM를 포함 hyaluronidase에서 MII oocyte-적 운 세포 복합물을 아래로 플라스틱. 각 oocyte 전송, 일단 그들은 세척을 적 운 세포 없는 드롭 포함 HM만 입 피 펫을 사용 하 여. 각 세척 물방울에 대해 이것을 반복 합니다. 조각난 또는 투명 한 oocytes15를 전송 하지 않습니다.
참고: 그것은 가능한 작은 흠으로 함께 35 mm 접시에 각 드롭에서는 oocytes를 전송 하는 것이 중요입니다. 이것은 모든 전송 프로토콜에 대 한 사실입니다. MII oocytes hyaluronidase HM 매체를 포함 하는 1 분 이상에 남아 있지 해야 합니다.
3. SM-물고기 Oocytes의 얼룩
- 고정 버퍼의 500 µ L을 포함 하는 6 잘 플레이트의 개별 잘에서 oocytes를 수정. 20 oocytes 잠수함 또는 우물에서 더 적은. 실 온에서 20 분 동안 품 어.
참고: 각 SM 물고기 착 단계 6-잘 원뿔 접시에 개별 잘 내에서 발생합니다. Oocytes 버퍼에 완전히 침수 및 버퍼 위에 하지 부동 인지 확인 합니다. 각 단계는 20 oocytes 함께 이하의 각 잘에서 해야 합니다. - 워시 버퍼 (WB 단계 1.6에서에서 설명)의 500 μ 하 고정된 oocytes를 전송 10 분. 2 번 더 반복 합니다.
- 실 온에서 30 분 동안 permeabilization 버퍼에 oocytes를 품 어.
참고: 단계 1.7에서에서 설명한 permeabilization 버퍼 예 permeabilization 버퍼를 대체 합니다.- 프로브 세트를 수집 하 고 신속 하 게는 microcentrifuge에서 그들을 아래로 회전. 각 프로브는 40 ° C 물 목욕 이나 인큐베이터에 10 분 동안 설정 따뜻한. 실내 온도에 냉각 하십시오.
참고:이 단계는 permeabilization 외피 동안에 수행 해야
- 프로브 세트를 수집 하 고 신속 하 게는 microcentrifuge에서 그들을 아래로 회전. 각 프로브는 40 ° C 물 목욕 이나 인큐베이터에 10 분 동안 설정 따뜻한. 실내 온도에 냉각 하십시오.
- 실 온에서 10 분에 대 한 WB의 500 µ L에 oocytes를 씻어.
- 프로 테아 제 III 버퍼 (키트에서 사용 가능)를 실 온에서 30 분 동안 1 X PBS에 희석된 1: 8의 80 μ에 oocytes를 전송.
참고: 80 µ L 볼륨 6 잘 플레이트에는 개별의 하단에 적절 하 게 다루고 있습니다. - 실 온에서 10 분에 대 한 WB의 500 µ L에 oocytes를 씻어.
- 따뜻하게 프로브 세트 Nanog, Pou5f1, 및 DapB (부정적인 제어 유전자)에 대 한 희석, 프로브 희석제에 1시 50분. 40 ° c.에서 2 시간 동안 대 본 특정 조사의 80 μ에 oocytes를 품 어
참고: 각 독점 프로브 세트는 3 개의 형광 채널 (C1, C2, C3) 중 하나에. Nanog 및 Pou5f1 프로브 C2 및 C3, 각각 태그 했다. - 독점, 앰프 1 (앰프 1), 증폭기 2 (AMP2), 증폭기 3 (AMP3)와 증폭기 4-형광 (앰프 4-플로리다) 실내 온도 따뜻하게.
참고:이 단계는 2-시간 대 본 특정 조사 인큐베이션 기간 동안 수행 되어야 합니다. - WB의 500 μ 하 여 oocytes를 전송 하 고 실 온에서 10 분 동안 품 어.
- 증폭 버퍼에 순차적으로 oocytes를 품 어.
- 80 µ L AMP1의 실 온에서 10 분에 대 한 WB의 500 µ L에 40 ° C. 전송 oocytes에서 30 분 동안에 oocytes를 품 어.
- 40 ° c.에서 15 분 동안 AMP2의 80 µ L에 oocytes를 품 어 실 온에서 10 분에 대 한 WB의 500 µ L에 oocytes를 전송.
- 40 ° c.에 30 분 동안 AMP3의 80 µ L에 oocytes를 품 어 실 온에서 10 분에 대 한 WB의 500 µ L에 oocytes를 전송.
참고: 프로토콜의 나머지 부분에서에서 수행 됩니다 어둠 앰프-플로리다에 fluorophore를 포함 하기 때문에. 작업할 때 해 현미경, 빛을 최대한으로 줄일 수 있습니다. - 40 ° c.에서 15 분의 AMP4 80 μ에 oocytes를 추가
참고: AMP4 층 버퍼 A 대체로 제공 됩니다 (Alt-A), Alt-B 또는 Alt-C 파장은 원하는 어떤 방출에 따라 AMP4 층 버퍼를 선택 합니다.
- 실 온에서 10 분에 대 한 WB의 500 µ L에 oocytes를 씻어. 80 µ L DAPI의 실 온에서 20 분 동안에 oocytes를 품 어. 실 온에서 5 분에 대 한 WB의 500 µ L에 oocytes를 씻어.
- 시에 거품을 추가 하지 않고도 슬라이드의 중심에 안티-페이딩 설치 미디어의 12 µ L 플라스틱. 설치 미디어에 가능한 작은 WB로와 oocytes를 전송 하 고는 coverslip 적용.
- 기울기 각도에 coverslip와 천천히 그리고 부드럽게 슬라이드에 액체 위에 놓습니다. 너무 열심히 oocytes 그리고 거품의 소개의 왜곡을 방지 하는 coverslip 눌러 하지 마십시오.
- 실 온에서 건조 하룻밤 어두운 상자에 슬라이드를 저장 합니다. Coverslip 인감 투명 매니큐어의 슬라이드의 가장자리 코트.
- 표준 현미경을 사용 하 여 영구 표시와 함께 슬라이드에 동그라미 oocytes를 찾을.
참고:이 단계는 필요 하지 않습니다 하지만 슬라이드 찾기 oocytes를 향상 시킵니다. 최상의 결과 얻으려면 이미지 슬라이드 형광 신호 1 ~ 5 일 이내 페이드 시작 됩니다.
4. 이미지 처리
- 3 차원 oocytes, z 단계 confocal 현미경 검사 법을 사용 하 여 이미지.
참고: 이미지를 정확 하 게 분석 하려면 각 z 단계는 1.0 µ m/조각 이어야 한다. - 압축된 nd2 또는 개별 confocal 이미지를 저장 합니다. 각 oocyte의 TIFF 파일입니다. 두 이미지 유형 오픈 소스 이미지 처리 프로그램, 피지와 호환 됩니다.
- 다운로드 및 오픈 액세스 피지 소프트웨어 (https://imagej.net/Fiji/Downloads)를 설치.
- 피지에 nd2 파일을 드래그 하 고 hyperstack을 선택 합니다. 만약 confocal 이미지로 저장 했다. TIFF 파일 4.4 단계로 건너뜁니다.
참고: 피지에 nd2 파일은 삭제 하는 때 hyperstack 드롭다운 표시 됩니다 자동으로. - 이미지 탭을 클릭 하 고 색상을 선택한 분할 채널 nd2 파일의 형광 채널 분리를 클릭 합니다.
- 개별 생성 합니다. 각 z 슬라이스 각 형광 채널에서 oocyte의에 대 한 TIFF 파일. 이미지 탭을 클릭 하 고 선택한 스택, 이미지 스택을클릭 합니다. 이미지 탭을 클릭 하 고 선택한 형식, 각 z 슬라이스 개별 RGB 컬러 이미지를 변환 하려면 RGB 색상 을 클릭 합니다.
참고: RGB 색상 인공 이며 각 방출 파장에 대 한 선택으로 원하는 될 수 있습니다. - 각 변환 된 이미지를 저장 합니다. TIFF 파일입니다. 단계 바느질 (4.3) 혼동을 피하기 위해 새 폴더에 각 형광 채널에 대 한 단일 oocyte에서 이미지를 배치 합니다.
- 피지에 nd2 파일을 드래그 하 고 hyperstack을 선택 합니다. 만약 confocal 이미지로 저장 했다. TIFF 파일 4.4 단계로 건너뜁니다.
- 각 정규화. Pou5f1 및 Nanog 부정적인 제어 이미지 (DapB)를 사용 하 여 TIFF 이미지.
참고: 정규화는 사진 편집 프로그램을 사용 하 여 수행 됩니다. 각 컨트롤 이미지에서 배경 형광의 동일한 수준을 제거 되었는지 확인 합니다. - 각 정규화 오픈. 함께 각 oocyte 각 파장에 대 한 모든 z 슬라이스를 피지에 TIFF 파일.
- 플러그인 탭을 클릭 하 고 선택한 바느질, 그리드/컬렉션 (그림 3A) 클릭. 드롭 다운 메뉴에서 확인을 클릭 합니다 (그림 3B) 순차적인 이미지 를 선택 합니다.
- 디렉터리를 검색 하 고 포함 하는 하나의 파장에서 개별 oocyte의 z 슬라이스 이미지의 모든 폴더를 선택 (단계 4.3.4 참조). 확인을 클릭 합니다.
- 사용 파장에 대 한 적절 한 색상 채널에 맞춘된 이미지의 하단에서 슬라이더를 이동 하 고 최종 RGB 맞춘된 이미지 이미지, 종류, 선택을 클릭 하 여 만들고 RGB 색상을 클릭 합니다.
참고:이 이미지는 4.6 아래 단계에서 설명 하는 형광 정량에 사용 됩니다.
- 32 비트 최대 예상된 그림에 맞춘된 이미지를 변환 합니다. 이미지를 클릭, 종류, 선택 하 고 32 비트 (그림 3C)을 클릭 합니다. 새로운로이 이미지를 저장 합니다. TIFF 파일입니다.
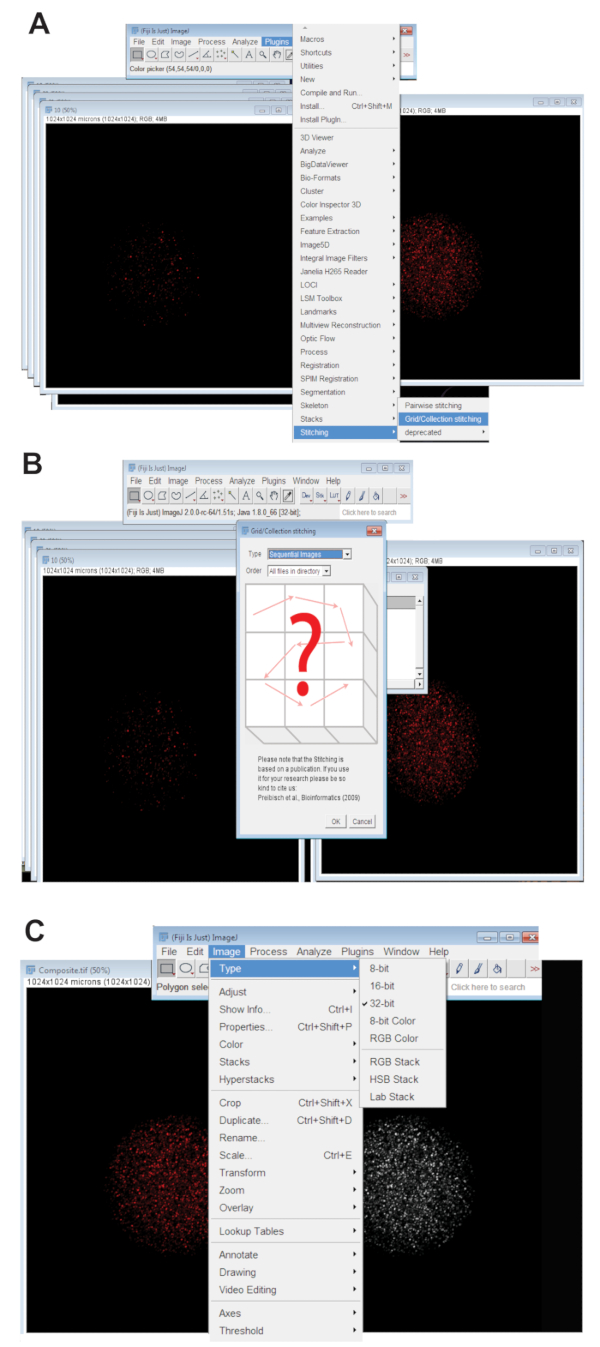
그림 3 : 함께 oocytes의 confocal z-시리즈 이미지의 바느질. (A) 는 oocyte의 합성 이미지를 생산 하는 데 사용 된 피지에 플러그 인 그리드/컬렉션 도구를 보여주는 스크린샷. (B) 연속 이미지 형광 겹치는 순차를 사용합니다. 합성 이미지를 생성 하기 위해 TIFF 파일입니다. (C) 복합 이미지 32 비트로 저장 되었습니다. TIFF 파일입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
- 다운로드 및 설치는 자리 찾기 및 추적 프로그램13, 라 슨 박사, 국립 연구소의 건강 국립 암 연구소 (https://ccr.cancer.gov/Laboratory-of-Receptor-Biology-and-Gene-Expression/daniel-r-larson)에 수 사관에 대 한 웹 사이트에서 사용할 수 있는. 다운로드 및 설치는 자리 찾기 및 추적 프로그램 (http://www.spacewx.com/pdf/idlvm.pdf)를 실행 하는 데 필요한 대화형 데이터 언어 (IDL) 운영 체제에 대 한 오픈 액세스 가상 컴퓨터.
- 자리 찾기 및 추적 프로그램에서 단계 4.6 (그림 4A)에서 생성 된 32 비트, 물 렸 다 이미지를 엽니다. Localize 드롭다운 을 선택 하 고 지 방화 (그림 4B), 이미지에 숫자 명소 계산을 클릭 합니다.
참고: 계산 각 자리는 개별 mRNA를 나타냅니다. 밴드 패스와 광자 임계값 설정 화면 (그림 4B)에 표시 됩니다. 이 프로토콜에 대 한 각 임계값 설정에 대 한 기본 사용 되었다. 긍정적인 대표 및 배경 명소 (그림 4C) 표시 됩니다.

그림 4 : 자리 Finder를 사용 하 여 mRNAs의 정량화 및 추적. (A) 개별 z-시리즈 이미지 그림 3 에 설명 된 대로 함께 봉합 되었고 예상 32 비트 최대로 저장. TIFF 파일입니다. (B) 합성 이미지 스팟 파인더와 추적에 오픈 했습니다. 지역화 형광 명소 (빨간색 상자)을 계산 하는 데 사용 했다. 밴드 패스와 광자 임계값 파란색 상자에 의해 표시 됩니다. (C) 파란색 화살표 (임계값) 긍정적인 신호를 가리킵니다. 흰색 화살표는 형광 임계값 아래 자리 표시 하 고, 따라서 계산 되지. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
결과
프로토콜 완료 되 면 결과 (그림 4A , 그림 5), 맞춘된 이미지 (그림 4C), confocal z 시리즈에서 개별 이미지를 있을 것입니다 그리고 mRNA 계산 (그림 4B). 다중화를 수행 하는 경우 또한 병합 된 이미지를 보여주는 두 개의 다른 mRNAs (
토론
일련의 작은 단계 프로토콜 동안 mRNAs의 성공적인 형광 및 정확한 보장 합니다. 첫째, 프로토콜 수집과 oocytes의 고정 후에 즉시 수행 되어야 합니다. Note PVP oocytes 서로에 집착 하지 않도록 4 %paraformaldehyde 고정 버퍼에 추가 됩니다. 우리 컬렉션은 oocytes의 고정 후 즉시 실험을 수행 하는 데 필요한 발견. 어떤 지연 결과 훨씬 낮은 형광 신호를 녹취 록의 undercounting 귀 착될 것입니다. 이것은 때문에 RNA ...
공개
저자는 아무것도 선언 하
감사의 말
설치 및 사용 자리 찾아서 추적 프로그램 13 의 confocal 현미경 이미징에 대 한 대학 네브라스카 링컨 현미경 코어의 기술 지원 그의 관대 한 도움에 감사 박사 다니엘 R. 라 슨 하 고. 이 연구는 네브라스카 농업 연구 부문, 링컨, 네브라스카 대학에의 기여도 나타냅니다과 UNL 해치 자금 (코-26-206/가입 번호-232435 및 코-26-231/승인 번호-1013511)에 의해 지원 되었다.
자료
| Name | Company | Catalog Number | Comments |
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
참고문헌
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유