Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La reprogramación de células somáticas humanas en células madre pluripotentes inducidas (iPSCs) utilizando el vector retroviral con las buenas prácticas agrarias
En este artículo
Resumen
Un método para generar células madre pluripotentes inducidas (iPSCs) a través de retrovirus mediada por la expresión ectópica de Oct4, Sox2, Klf4 y MYC se describe. Una forma práctica para identificar las colonias de humanos IPSC basadas en la expresión de GFP también se discute.
Resumen
Las células madre embrionarias (hESCs) son pluripotentes y una fuente inestimable para celulares en el modelado de la enfermedad in vitro y la medicina regenerativa 1. Se ha demostrado previamente que las células somáticas humanas pueden ser reprogramadas para pluripotencia por la expresión ectópica de cuatro factores de transcripción (Oct4, Sox2, Klf4 y Myc) y se convierten en células madre pluripotentes inducidas (iPSCs) 2-4. Al igual que hESCs, iPSCs humanos son pluripotentes y una fuente potencial de células autólogas. A continuación se describe el protocolo de reprogramar las células de fibroblastos humanos con los cuatro factores de reprogramación clonados en las buenas prácticas agrarias que contienen columna vertebral de retrovirales 4. Utilizando el protocolo siguiente, generamos iPSCs humanos en 3-4 semanas bajo condiciones de la cultura humana ESC. Humanos colonias IPSC se parecen mucho a hESCs en la morfología y mostrar la pérdida de la fluorescencia de GFP como resultado de silenciamiento de transgenes retrovirales. colonias aisladas IPSC mecánicamente bajo una fluorescencia microscópicaPE se comportan de una manera similar como hESCs. En estas células, se detecta la expresión de múltiples genes pluripotencia y marcadores de superficie.
Protocolo
1. Reprogramación por retrovirus Expresando factores de reprogramación
- Fibroblastos humanos se cultivaron en medio de fibroblastos (10% de FBS en DMEM con Pen / Strep).
- Un día antes de la infección, la placa de 1x10 5 fibroblastos humanos en un pocillo de una placa de 6 pocillos.
- Aspirar el medio para eliminar las células muertas y añadir 2 ml de medio de fibroblastos fresco. Añadir sulfato de protamina a una concentración final de 5 mg / ml.
- Añadir cuidadosamente la cantidad apropiada de cada virus que expresan GFP correspondiente a una multiplicidad de infección (MOI) 5 5.
- Un día después de la infección, separar el sobrenadante viral, lavar tres veces con 2 ml de PBS, a continuación, agregar 2 ml de medio de fibroblastos.
- Tres días después de la infección, consulte la fluorescencia de GFP y reponer el bien con 2 ml de medio de fibroblastos.
- Cuatro días después de la infección, la placa de 1x10 4 / cm 2 de fibroblastos de embriones de ratón irradiados (MEFs) células alimentadoras en medio de fibroblastos sobre una10-cm placa de Petri revestidas con gelatina al 0,1%. Incubar a 37 ° C durante la noche.
- Cinco días después de la infección, separar los fibroblastos humanos infectados con 1 ml de 0,05% typsin / EDTA durante 5 minutos a 37 ° C, y se centrifuga durante 5 minutos a 200 g. Aspirar el medio y resuspender las células con 10 ml de medio de fibroblastos. Transferir las células en un pre-recubiertas 10-cm placa.
- Después de 24 horas, vuelva a colocar el medio con un medio de hESC la cultura (20% Knock-Out reemplazo de suero, DMEM/F12, 0,1 ml no aminoácidos esenciales, de 4 ng / ml de bFGF, Pen / Strep / glutamato, el beta-merceptoethanol). Cambiar el medio día. ESC-como colonias comenzarán a aparecer durante el día 20-27 después de la infección.
2. El aislamiento y la expansión de iPSCs
- Bajo un microscopio de fluorescencia, comprobar la ausencia de la fluorescencia de GFP en una colonia que muestra una morfología similar a hESCs.
- Con una pipeta 10 l, escoja colonias individuales IPSC y colocarlos en un pocillo de una gelatina y el MEF-CoATed de 12 pocillos y se completará con medio de hESC. Cambiar medio diario.
- Para los pases, se lava la placa con 1 ml de DMEM/F12, a continuación, agregar 0,5 ml de colagenasa, y se incuba durante 10 minutos a 37 ° C.
- Se lavan las células dos veces con DMEM/F12.
- Añadir 2 ml de medio de hESC fresco. El uso de un elevador de la célula, romper las colonias en pequeños pedazos y separar las células restantes de la placa.
- Transferir los trozos de colonias resuspendidas en un pocillo de una gelatina y MEF recubierto-6-así placa.
3. El análisis de inmunofluorescencia de marcadores pluripotentes
- Se lavan las células tres veces con PBS y fijar con paraformaldehído al 4% durante 20 min a temperatura ambiente.
- Lave suavemente las células tres veces con PBS y permeabilizar con 0,2% de Triton X-100 en PBS durante 30 min.
- Bloque no específicos de unión mediante la incubación de células con 3% de BSA en PBS durante dos horas.
- Se incuban las células con el anticuerpo primario durante la noche a 4 ° C.
- Se lavan las células tres veces con PBS y se incuban las células con anticuerpo secundario específico durante una hora a temperatura ambiente, protegiendo de la luz.
- Se lavan las células tres veces con PBS y DAPI complemento durante el último lavado, seguido de incubación a temperatura ambiente durante 5 minutos.
- Detectar la tinción con un microscopio de fluorescencia.
4. Cuantitativo en tiempo real PCR para los marcadores pluripotentes
- Aislar ARN total de las iPSCs humanos derivados de fibroblastos humanos utilizando RNeasy kit de Qiagen.
- Sintetizar el cDNA primer capítulo de la transcriptasa reversa utilizando SuperScript II.
- Realizar qPCR para la detección de genes usando cebadores pluripotencia se informó anteriormente. 6
5. Los resultados representativos
- El cambio morfológico durante la reprogramación
Estamos infectados fibroblastos humanos BJ1 y Detroit 551 con un cóctel de los retrovirus que llevan Oct4, Sox2, Klf4 y MYC,y fueron capaces de detectar cambios morfológicos durante la reprogramación (Figura 1). Veintiún días después de la infección, reconocemos pequeñas colonias de IPSC por su hESC-como la morfología. Además, reconocemos iPSCs por la fluorescencia de GFP. Las células madre pluripotentes, como los CES e IPSCs, expresan la maquinaria molecular para reprimir la expresión del gen proviral 7-9. Nuestro único vector retroviral expresa GFP junto con los genes reprogramación por LTR retroviral. Así, las células que expresan GFP de forma continua se consideran para expresar transgenes, sin silenciamiento génico proviral. Fielmente reprogramadas colonias IPSC que adquieren la red molecular pluripotencia muestran la ausencia de expresión de las buenas prácticas agrarias (Figura 2) 10. - Caracterización de la pluripotencia de iPSCs humanos
Se analizaron colonias derivadas de Detroit-551 fibroblastos a través de inmunohistoquímica con Tra-1-81, Tra-1-60, SSEA-4, 3-SSEA, Oct4 y anticuerpos NANOG ( Tabla 2). IPSCs con éxito reprogramadas expresar todos estos marcadores (Figura 3). También se analizó la expresión génica a través de RT-PCR cuantitativa análisis. Hemos observado que la expresión de Oct4, Sox2, Klf4, MYC y NANOG se incrementó significativamente en comparación con las células de fibroblastos de los padres, sino a la de las H9 hESCs (Figura 3B).
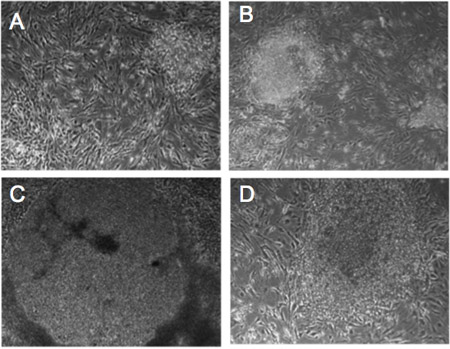
Figura 1. Los cambios morfológicos de los infectados por retrovirus fibroblastos humanos. (AD), el cambio progresivo morfológica en las colonias de Detroit-551 fibroblastos infectados con factores de reprogramación. Día 5 (A), el día 10 (B), el día 14 (C), el día 21 (D). Las células muestran la morfología hESC-al igual que después de 21 días.
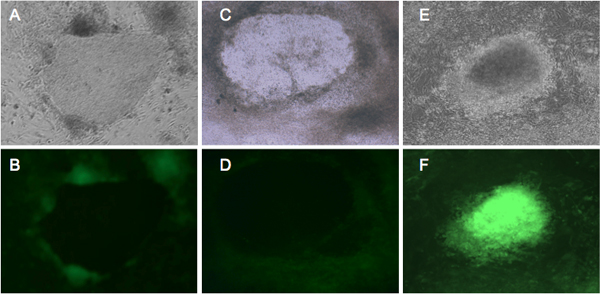
Figura 2. Representante de las buenas prácticas agrarias fluorescente de expresión en las células sometidas a una reprogramación. BJ y Detroit 551 fibroblastos fueron infectadas con retrovirus que expresa cuatro factores de reprogramación 4, y se incubaron en medio hCME durante cuatro semanas. Fibroblastos BJ (A, B) y Detroit 551 (C, D) muestran similares morfológica. Desde el día 21, las colonias de las buenas prácticas agrarias negativos comienzan a formarse, que representan las iPSCs de buena fe 10. (E, F) muestran transformado Detroit 551 células que no se han sometido a una reprogramación adecuada. (A, C, E) colonias bajo punto de vista de contraste de fase. (B, D) adecuadamente reprogramado las células que muestran el silenciamiento de las buenas prácticas agrarias. (F) la expresión de GFP luminosa de la colonia transformada.
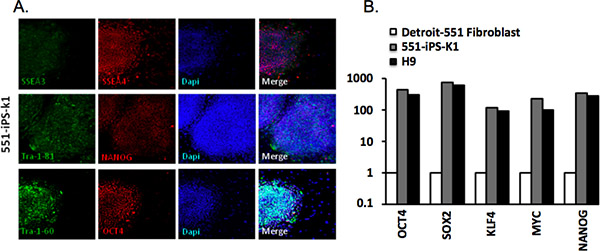
Figura 3. Caracterización de las células madre pluripotentes inducidas (A). Humanos 551-iPS-K1 colonias de células expresan marcadores comunes a las células pluripotentes. DAPI indica el contenido total de células por campo. (B) cuantitativa en tiempo real PCR (RT-qPCR) para la expresión de Oct4, Sox2, Klf4, MYC en f los padresibroblast, 551-IPS-K1 iPSCs y H9 células madre embrionarias humanas (hESCs). Los datos se normalizaron contra limpieza de genes β-actina y trazan en relación con el nivel de expresión en las células de fibroblastos parentales 4.
Discusión
Expresión de los cuatro factores de transcripción reprograma fibroblastos humanos a iPSCs. Muchos intentos se hicieron para generar iPSCs humanos utilizando los enfoques no integrar o no genéticos para generar iPSCs clínicamente seguros. Hasta ahora, estos métodos presentan una eficiencia extremadamente baja y requieren una mayor optimización para mejorar la reproducibilidad 11-14. Métodos de retro-o lentiviral son fácilmente utilizados para obtener y aplicar iPSCs para personas en modelos de...
Divulgaciones
No tenemos nada que revelar.
Agradecimientos
Este trabajo fue financiado por la Escuela de Medicina de Yale y el Premio de Investigación en Salud Infantil de la Fundación Charles Hood.
Materiales
| Name | Company | Catalog Number | Comments |
| DMEM/F12 | Invitrogen | 11330057 | 80% |
| Knockout Serum Replacer | Invitrogen | 10828-028 | 20% |
| L-Glutamine (200 mM) | Invitrogen | 25030081 | 2 mM |
| Nonessential Amino Acids (10 mM) | Invitrogen | 11140050 | 0.1 mM |
| β-Mercapt–thanol (14.3 M) or MTG | Invitrogen | M-6250 | 0.1 mM |
| bFGF-2 10 μg/ml | GIBCO, by Life Technologies | GF003AF | 4 ng/ml |
| Penicillin/Streptomycin | EMD Millipore | 15140-122 | 1% |
| DMEM | Invitrogen | 11965118 | 90% |
| FBS | Invitrogen | 10407028 | 10% |
| Penicillin/Streptomycin | EMD Millipore | 15140-122 | 1% |
| Table 1. Culture Medium | |||
| OCT4 | Abcam | Ab19857 | 1:500 |
| SSEA3 | EMD Millipore | MAB4303 | 1:100 |
| SSEA4 | BD Biosciences | BD560218 | 1:100 |
| Tra-1-81 | BD Biosciences | BD560173 | 1:100 |
| Tra-1-60 | BD Biosciences | BD560174 | 1:100 |
| NANOG | Abcam | Ab21624 | 1:500 |
| Alexa-Flur 488 | Invitrogen | A11008 | 1:1000 |
| Alexa-Flur 555 | Invitrogen | A21422 | 1:1000 |
| DAPI | Invitrogen | D1306 | 1:5000 |
| pMIG-OCT4 | Addgene | 17225 | |
| pMIG-SOX2 | Addgene | 17226 | |
| pMIG-KLF4 | Addgene | 17227 | |
| pMIG-MYC | Addgene | 18119 | |
| Collagenase type IV | Invitrogen | 17104019 | 1mg/ml |
| Gelatin, Porcine | Sigma-Aldrich | G 1890 | 0.1% |
| Triton | Sigma-Aldrich | X100-500ML | 0.2% |
| Paraformaldehyde | Sigma-Aldrich | 47608 | 4% |
| BSA | American Bioanalytical | AB01800 | 3% |
| MEF feeder cells | EMD Millipore | PMEF-N | |
| Cell Lifter | Corning | 3008 | |
| Fluorescent microscopy: inverted microscope with GFP filter | |||
| Table 2. Reagents and equipment | |||
Referencias
- Murry, C. E., Keller, G. Differentiation of embryonic stem cells to clinically relevant populations: lessons from embryonic development. Cell. 132, 661-680 (2008).
- Takahashi, K., Tanabe, K., Ohnuki, M., Narita, M., Ichisaka, T., Tomoda, K., Yamanaka, S. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131, 861-872 (2007).
- Yu, J., Vodyanik, M. A., Smuga-Otto, K., Antosiewicz-Bourget, J., Frane, J. L., Tian, S., Nie, J., Jonsdottir, G. A., Ruotti, V., Stewart, R. Induced pluripotent stem cell lines derived from human somatic cells. Science. 318, 1917-1920 (2007).
- Park, I. H., Zhao, R., West, J. A., Yabuuchi, A., Huo, H., Ince, T. A., Lerou, P. H., Lensch, M. W., Daley, G. Q. Reprogramming of human somatic cells to pluripotency with defined factors. Nature. 451, 141-146 (2008).
- Park, I. H., Lerou, P. H., Zhao, R., Huo, H., Daley, G. Q. Generation of human-induced pluripotent stem cells. Nature Protocols. 3, 1180-1186 (2008).
- Park, I. H., Zhao, R., West, J. A., Yabuuchi, A., Huo, H., Ince, T. A., Lerou, P. H., Lensch, M. W., Daley, G. Q. Reprogramming of human somatic cells to pluripotency with defined factors. Nature. 451, 141-146 (2008).
- Hotta, A., Ellis, J. Retroviral vector silencing during iPS cell induction: an epigenetic beacon that signals distinct pluripotent states. Journal of Cellular Biochemistry. 105, 940-948 (2008).
- Matsui, T., Leung, D., Miyashita, H., Maksakova, I. A., Miyachi, H., Kimura, H., Tachibana, M., Lorincz, M. C., Shinkai, Y. Proviral silencing in embryonic stem cells requires the histone methyltransferase ESET. Nature. 464, 927-931 (2010).
- Wolf, D., Goff, S. P. Embryonic stem cells use ZFP809 to silence retroviral DNAs. Nature. 458, 1201-1204 (2009).
- Chan, E. M., Ratanasirintrawoot, S., Park, I. H., Manos, P. D., Loh, Y. H., Huo, H., Miller, J. D., Hartung, O., Rho, J., Ince, T. A. Live cell imaging distinguishes bona fide human iPS cells from partially reprogrammed cells. Nat. Biotechnol. 27, 1033-1037 (2009).
- Yu, J., Hu, K., Smuga-Otto, K., Tian, S., Stewart, R., Slukvin, ., Thomson, J. A. Human induced pluripotent stem cells free of vector and transgene sequences. Science. 324, 797-801 (2009).
- Kim, D., Kim, C. H., Moon, J. I., Chung, Y. G., Chang, M. Y., Han, B. S., Ko, S., Yang, E., Cha, K. Y., Lanza, R. Generation of human induced pluripotent stem cells by direct delivery of reprogramming proteins. Cell Stem Cell. 4, 472-476 (2009).
- Warren, L., Manos, P. D., Ahfeldt, T., Loh, Y. H., Li, H., Lau, F., Ebina, W., Mandal, P. K., Smith, Z. D., Meissner, A. Highly efficient reprogramming to pluripotency and directed differentiation of human cells with synthetic modified mRNA. Cell Stem Cell. 7, 618-630 (2010).
- Ban, H., Nishishita, N., Fusaki, N., Tabata, T., Saeki, K., Shikamura, M., Takada, N., Inoue, M., Hasegawa, M., Kawamata, S. Efficient generation of transgene-free human induced pluripotent stem cells (iPSCs) by temperature-sensitive Sendai virus vectors. Proceedings of the National Academy of Sciences of the United States of America. 108, 14234-14239 (2011).
- Wolf, D., Goff, S. P. TRIM28 mediates primer binding site-targeted silencing of murine leukemia virus in embryonic cells. Cell. 131, 46-57 (2007).
- Park, I. H., Arora, N., Huo, H., Maherali, N., Ahfeldt, T., Shimamura, A., Lensch, M. W., Cowan, C., Hochedlinger, K., Daley, G. Q. Disease-specific induced pluripotent stem cells. Cell. 134, 877-886 (2008).
- Kim, K. Y., Hysolli, E., Park, I. H. Neuronal maturation defect in induced pluripotent stem cells from patients with Rett syndrome. Proceedings of the National Academy of Sciences of the United States of America. 108, 14169-14174 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados