Method Article
Mapeo de la Organización Espacial Emergente de Células De mamíferos usando Micropatrones e Imágenes Cuantitativas
En este artículo
Resumen
El método presentado aquí utiliza micropatrones junto con imágenes cuantitativas para revelar la organización espacial dentro de los cultivos de mamíferos. La técnica es fácil de establecer en un laboratorio de biología celular estándar y ofrece un sistema manejable para estudiar el patrón in vitro.
Resumen
Un objetivo fundamental en biología es entender cómo surgen los patrones durante el desarrollo. Varios grupos han demostrado que el patrón puede lograrse in vitro cuando las células madre se limitan espacialmente a micropatrones, estableciendo así modelos experimentales que ofrezcan oportunidades únicas para identificar, in vitro, los principios fundamentales de la Organización.
Aquí describimos nuestra propia implementación de la metodología. Adaptamos una técnica de fotopatrones para reducir la necesidad de equipos especializados para facilitar el establecimiento del método en un laboratorio estándar de biología celular. También desarrollamos un marco de análisis de imágenes gratuito, de código abierto y fácil de instalar con el fin de medir con precisión el posicionamiento preferencial de las subpoblaciones de células dentro de colonias de formas y tamaños estándar. Este método permite revelar la existencia de eventos de patrones incluso en poblaciones aparentemente desorganizadas de células. La técnica proporciona información cuantitativa y se puede utilizar para desacoplar las influencias del entorno (por ejemplo, señales físicas o señalización endógena), en un proceso de patrón determinado.
Introducción
En los sistemas de mamíferos, el patrón es una propiedad emergente del comportamiento colectivo de las células y por lo tanto, los patrones pueden formarse in vitro si se proporcionan señales apropiadas a las células1,2,3,4, 5 , 6. Una manera de revelar la capacidad intrínseca de las células para autoorganizarse in vitro es forzar a las células a formar grupos / colonias de una forma y tamaños definidos7,8,9,10 . Una técnica que permite esto es el micropatrón11. El micropatrón permite definir con precisión la ubicación donde se depositan moléculas de matriz extracelular (ECM) en una superficie. Esto, a su vez, dicta dónde se pueden adherir las celdas y, por lo tanto, controla cómo se organizan espacialmente las celdas.
El micropatrón es una técnica con numerosas aplicaciones, por ejemplo, el micropatrón permite la estandarización de las condiciones iniciales antes de la diferenciación12. Es importante destacar que el micropatrón permite controlar fácilmente el tamaño, la forma y el espaciado de las colonias celulares y esta propiedad se puede utilizar para idear experimentos destinados a interrogar la respuesta colectiva de las células al mortogeno o a las señales físicas7 , 8 , 10 , 13 , 14 , 15 , 16 , 17.
Se han desarrollado varios métodos de micropatrones11. Las técnicas de fotopatrones son quizás los métodos más fáciles de establecer18. Estos enfoques también tienen la ventaja de la precisión, ya que se pueden utilizar para controlar la forma de las células individuales18,19,20. Sin embargo, también requieren costosos equipos especializados, incluyendo un revestimiento de espín, una cámara de plasma y un limpiador UVO (UV-Ozone) que generalmente no están disponibles en laboratorios de biología estándar. Para facilitar la adopción de la técnica, adaptamos el protocolo para requerir sólo la lámpara UVO. Partimos de portaobjetos de plástico disponibles comercialmente que se pueden cortar con tijeras o con un punzón de agujero al formato deseado.
Una utilidad importante de los micropatrones es la capacidad de estandarizar colonias con el fin de comparar colonias individuales a través de múltiples réplicas. Esto permite preguntar en qué medida la formación de patrones dentro de estas colonias es reproducible, y explorar factores que influyen en la robustez del proceso de patrón. Es importante destacar que la cuantificación de patrones "promediados" en múltiples colonias estandarizadas también puede revelar procesos de patrones que de otro modo no serían evidentes. La ventaja de ser capaz de cuantificar el patrón en colonias estandarizadas depende de ser capaz de medir con precisión la expresión de proteínas, idealmente a nivel de una sola célula. Sin embargo, las células de los micropatrones a menudo están apretadas, lo que hace que sean difíciles de segmentar con alta precisión. Las celdas también se organizan a menudo en tres en lugar de dos dimensiones, y puede ser difícil detectar y preservar la información tridimensional (3D) durante la segmentación. Una vez que las celdas se han segmentado correctamente, se necesitan métodos informáticos para extraer información de patrones de los conjuntos de datos resultantes.
Hemos desarrollado herramientas de segmentación y análisis de imágenes para ayudar a superar estos problemas. Este método de análisis sólo utiliza software libre y de código abierto y no requiere conocimiento de la línea de comandos o la programación para implementar. Para ilustrar el método aquí, utilizamos células madre embrionarias de ratón (mES) que expresan espontáneamente un marcador de brachyury de diferenciación temprana (Tbra)21,22. Aunque ninguna disposición espacial aparente es visualmente detectable, el método permite la creación de un mapa del posicionamiento preferencial de las células T+ en colonias. También mostramos que el patrón de Tbra contrasta con la ausencia de una localización preferente de las células que expresan Id1, una lectura directa de la vía23de la proteína morfogenética ósea (BMP). También discutimos las limitaciones actuales del método y cómo esta técnica puede adaptarse a otros sistemas experimentales.
Protocolo
NOTA: En la Figura 1se proporciona información general sobre el método.
1. Diseño de máscara
- Diseñar la fotomascarilla de acuerdo con las directrices descritas en Azioune et al.18. Consulte la Tabla de materiales para obtener una referencia al fabricante de software y máscara utilizado para este estudio.
NOTA: Se pueden crear varias geometrías, tamaños o espaciado entre formas en una máscara. La lámpara UV puede adaptarse a una fotomáscara de 15 cm que puede contener hasta 49 diseños diferentes (suponiendo chips de 2 cm x 2 cm).
2. Procedimiento de fabricación de micropatrones
- Preparar los materiales necesarios.
- Preparar una solución de poloxámero 407 al 0,1% (10 mg para 10 ml) en solución salina tamponada de fosfato (PBS) y dejar en una coctelera a temperatura ambiente. El poloxámero 407 tardará unos 20 minutos en disolverse.
- Poner película de laboratorio (ver la Tabla de Materiales) en la parte inferior de un plato Petri cuadrado de 10 cm. Esto se utilizará como la cámara para la deposición de la matriz.
- Limpiar la superficie de la fotomáscara, primero con 100% acetona, luego con 100% isopropanol y finalmente con ddH2O. Si es posible, seque al aire la mascarilla o, de lo contrario, seque la máscara con una toalla de papel limpia.
- Preparar una pieza cuadrada, rígida y opaca de plástico con el mismo tamaño exacto que la fotomáscara (más tarde denominada "holder").
NOTA: Esto se utilizará para mantener las cubiertas de plástico en contacto con la fotomáscara durante el paso de iluminación. - Encienda la lámpara UVO y ejecute una iluminación de calentamiento durante 10 minutos.
- Crea fichas fotopatrónadas.
NOTA: Es posible adaptar el procedimiento para crear virutas de cualquier tamaño deseado. Para simplificar, aquí describimos el procedimiento para generar un chip micropatrón redondo de 12 mm.- Usando un punzón de orificio de 12 mm, corte los portaobjetos de plástico hidrófobo para crear tapas redondas de 12 mm y colóquelos en una nueva y limpia placa Petri.
ADVERTENCIA: Utilice guantes en todo momento para evitar el contacto de la piel con la superficie del plástico, ya que esto puede dañar el tratamiento superficial. - Retire con cuidado la película protectora de los labios de las cubiertas con pinzas.
NOTA: Evite dañar la superficie de plástico, ya que esto puede influir en la colocación de las células en el chip durante el procedimiento de sembración (sección 3). - Coloque la fotomáscara sobre una superficie limpia y estable (por ejemplo, caja de fotomáscara), lado cromado hacia arriba y agregue una gota de 2 l de ddH2O en la posición del diseño de viruta deseado.
- Coloque un cubreobjetos sobre la gota de ddH2O y presione suavemente.
NOTA: Asegúrese de que el lado de plástico que da a la fotomáscara es el lado que estaba protegido por la película que se eliminó en el paso anterior. - Coloque el soporte encima de las guías de plástico y fije cuidadosamente este sándwich con abrazaderas para mantener las piezas de plástico en contacto con la fotomáscara.
NOTA: Coloque las abrazaderas lo más cerca posible de la ubicación de las guías de plástico para asegurarse de que las guías de plástico se mantienen perfectamente en contacto con la superficie de la fotomáscara. - Coloque el conjunto en la lámpara UVO a aproximadamente 2 cm de la fuente de luz e ilumine durante 10 min.
NOTA: Se estima que la potencia de la luz es de 6 mW/cm2 a 254 nm de longitud de onda cuando el chip se coloca a una distancia de 2 cm de la fuente. - Sostenga el sándwich con la fotomáscara en la parte inferior y retire cuidadosamente las abrazaderas mientras mantiene la presión con una mano para evitar que las diapositivas se muevan mientras desmonta el sándwich. Retire el soporte, asegurándose de que todas las piezas de plástico todavía están en la máscara y no pegadas al soporte.
- Agregue ddH2O encima de las fichas y separe suavemente las virutas de la fotomáscara.
NOTA: Si el chip de plástico está pegado a la fotomáscara, desenganche el chip con una punta de pipeta de plástico para empujar el chip mientras sostiene las pinzas ligeramente por encima del chip en caso de que el chip se seque repentinamente. - Finalmente, coloque las virutas fotoplaspatrónadas dentro de la cámara de deposición de matriz.
NOTA: Asegúrese de que el lado iluminado del chip esté orientado hacia arriba.
- Usando un punzón de orificio de 12 mm, corte los portaobjetos de plástico hidrófobo para crear tapas redondas de 12 mm y colóquelos en una nueva y limpia placa Petri.
- Deposite la matriz.
NOTA: Todo el procedimiento de esta sección debe realizarse en una campana de cultivo de tejido.- Filtrar la solución de poloxámero 407 a través de un filtro de poliétersulfone (PES) de 0,22 m.
- Preparar la solución de recubrimiento ECM mezclando 500 g/ml de poloxámero filtrado estéril 407 y 1 mg/ml de gelatina.
NOTA: Véase también la Tabla 1 para obtener información adicional sobre otras posibles moléculas de ECM. - Añadir 200 l de la solución de recubrimiento a cada chip iluminado. La película de laboratorio evitará que la caída caiga fuera del chip.
- Añadir un plato Petri de 3 cm lleno de ddH2O con el fin de limitar la evaporación y colocarlo con el chip a 4 oC durante la noche.
3. Procedimiento de sembrado
NOTA: Los pasos descritos a continuación se han optimizado para células madre embrionarias de ratón CGR8 (mESC)24 utilizando el medio mESC estándar (véase también la Tabla de Materiales). Sin embargo, en principio es posible adaptar el procedimiento para cualquier tipo de célula. Tenga en cuenta también que el cultivo celular convencional de células madre embrionarias de ratón no se describe aquí, ya que se puede encontrar una amplia documentación en otros lugares25.
- Aspirar la solución de recubrimiento e incubar las virutas dos veces durante al menos 5 minutos con PBS estéril.
- Mientras tanto, prepare una suspensión celular de 5,5 x 105 células/ml en medio cálido.
- Pipet 200 l de suspensión celular en cada viruta (100.000 celdas/cm2).
- Cierre la cámara de sembración y deje que las células se adhieran durante 1 h en la incubadora.
- Después de 1 h, llene los pocillos de una placa multipocilla (placa de 4 pocillos o 24 pocillos dependiendo del número de virutas) con 500 ol/pozo de medio caliente y transfiera las virutas a la placa con pinzas estériles.
- Agitar la placa vigorosamente para separar las células no adherentes. Aspirar el medio e inmediatamente reemplazar con un medio caliente fresco. Compruebe bajo el microscopio si el patrón es visible (Figura2a).
NOTA: Es posible que sea necesario optimizar el tiempo de adhesión cuando se utilizan líneas celulares distintas de mESC u otras proteínas de matriz (véase también la Tabla 2). - Repita este paso hasta que los patrones sean claramente visibles como se muestra en la Figura 2a.
NOTA: Este paso es crítico y determina el éxito del procedimiento. Un procedimiento de lavado que es demasiado intenso puede separar las células, en contraste lavado insuficiente puede resultar en células que permanecen unidas entre los patrones (Figura2c,d).
4. Fijación
NOTA: Después de 48 h en cultivo, las células deben formar colonias densas que sigan rigurosamente la forma de los patrones (como se muestra en la Figura 2b).
- Dejando las virutas en la placa, retire el 90% del medio, dejando el medio justo para evitar que las virutas se sequen.
NOTA: Es importante que el chip nunca se seque para evitar manchas artefactos y para evitar el desprendimiento celular de la superficie. Debido a la hidrofobicidad de la superficie de la viruta entre los patrones de adhesivo, el chip puede tener una tendencia a des-wet. En esta etapa, el fijador puede hacer que las grandes colonias abovedadas se desprendan del chip. Los lavados deben ser muy suaves, idealmente realizados por el líquido pipeteado en el lado del pozo y no directamente en el chip. - Añadir al menos 500 l de solución de fijación basada en paraformaldehído (PFA) por pozo e incubar durante 10 min.
NOTA: Si las colonias parecen particularmente gruesas (más de 5 capas celulares), puede ser necesario ajustar el tiempo de fijación a 20 min. - Después de la fijación, lavar 3 veces con la solución de lavado (PBS con 0,01% poloxámero 407). Un lavado adicional con 50 mM NH4Cl diluido en solución de lavado puede intercalarse con la actividad de reticulación de PFA residual.
- Incubar las muestras durante al menos 30 minutos en solución de bloqueo.
NOTA: En esta etapa, las muestras pueden almacenarse a 4 oC durante aproximadamente una semana antes de la tinción. Si es así, selle la placa con película de laboratorio para evitar la evaporación.
5. Inmunomanchación
- Preparar una cámara de tinción colocando una hoja de película de laboratorio en la parte inferior de un plato Petri cuadrado de 10 cm.
- Preparar soluciones de anticuerpos (ver Tabla 3 para obtener una lista de anticuerpos y diluciones utilizadas en este artículo).
- Coloque el chip en la cámara de tinción con el lado que sostiene las células hacia arriba y agregue inmediatamente 100 sl de solución de anticuerpos primarios en el chip.
ADVERTENCIA: En esta etapa, las virutas no deben desmojarse fácilmente como en el paso 4.1. Sin embargo, se debe tener cuidado porque es importante que las virutas no se sequen. Si se tienen que procesar varios chips, aplique el paso 5.3 a cada chip secuencialmente. - Incubar durante 1 h en una plataforma giratoria a temperatura ambiente.
NOTA: Si las células han formado grandes estructuras 3D, puede ser necesario un tiempo de incubación más largo para permitir una tinción uniforme de la muestra. El tiempo de incubación puede aumentarse hasta 24 h. Sin embargo, la cámara de tinción debe contener un plato de 3 cm lleno de agua y la cámara de tinción debe estar sellada con película de laboratorio para evitar la evaporación. - Transfiera las virutas a un plato multipocillo fresco y lave 3 veces con la solución de lavado.
- Realizar incubación con anticuerpos secundarios como se describe en los pasos 5.3 y 5.4.
- Monte el chip en una corredera de microscopía utilizando 20 s de cualquier medio de montaje estándar (por ejemplo, Mowiol).
6. Imágenes
NOTA: Las imágenes se pueden realizar en un microscopio confocal estándar. Aquí sólo proporcionamos recomendaciones para garantizar una calidad de imagen que será suficiente para el análisis cuantitativo posterior.
ADVERTENCIA: Para evitar cualquier sesgo del operador, las colonias a la imagen sólo deben ser elegidas usando la señal de sobre nuclear (para ver si una colonia sigue correctamente la forma del patrón). Evite comprobar la señal de los marcadores de interés excepto al ajustar la configuración del microscopio.
- Asegúrese de que la profundidad de bits de adquisición sea de 12 o 16 bits.
- Identifique los ajustes adecuados para maximizar el rango dinámico para cada canal de imagen. En particular, evite el recorte de imágenes.
- Incluya un canal para crear una imagen de la autofluorescencia del micropatrón (consulte la figura 3).
- Ajuste el tamaño de la imagen y el factor de zoom para obtener tamaños de vóxeles que oscilan entre 0,1 y 0,6 m en los ejes X e Y y que oscilan entre 0,2 y 2 m en el eje z.
NOTA: Por ejemplo, en este estudio, utilizamos un microscopio confocal de escaneo invertido con un objetivo de 40x (apertura numérica igual a 1.3), un tamaño de imagen de 1024 x 1024 píxeles sin zoom digital y un tamaño de paso z de 0,5 m. Esto dio lugar a un tamaño de voxel de 0,38 ám x 0,38 ám x 0,5 m. - Para cada colonia, defina la posición mínima y máxima a lo largo del eje z para asegurarse de que se adquiere toda la colonia. Al menos un avión con señal baja o sin señal debe incluirse por debajo y por encima de la colonia.
- Asegúrese de que las orientaciones de la pila z se adquieren de forma coherente (ya sea siempre de arriba a abajo o siempre de abajo a arriba)
- Ajuste la velocidad de escaneado, la resolución de la imagen, el promedio de fotogramas y las ganancias del detector para identificar un tiempo óptimo entre la calidad de imagen y el tiempo de imagen. Como indicación, el tiempo de creación de imágenes para una colonia como se muestra en la Figura 3 fue de aproximadamente 2-3 min. Realizar la adquisición de la imagen.
ADVERTENCIA: Todas las imágenes, para ser comparables, deben adquirirse en el mismo microscopio con el mismo objetivo y ajustes de adquisición. - Al final de la adquisición, guarde todas las imágenes y asigne una convención de nomenclatura única para identificar la condición experimental que representa cada imagen.
NOTA: Vea la figura 4 como ejemplo, esta convención se utilizará más adelante durante el procedimiento de análisis. Tenga en cuenta que las imágenes se pueden guardar en cualquier formato compatible con BioFormats 26. Si las colonias son más grandes que el campo de visión, el plugin de costura de ImageJ se puede utilizar27. Tenga en cuenta también que en caso de costura, la corrección del rollo de iluminación podría ser necesaria.
7. Análisis de imágenes
NOTA: Las especificaciones recomendadas del equipo para este procedimiento son: 16 GB de RAM, una CPU multinúcleo de 3,33 GHz y al menos 50 GB de espacio en disco (o más dependiendo del número de chips que se han generado). El software ha sido probado en Linux, Windows y MacOS. PickCells es una aplicación de análisis de imágenes multiplataforma con una interfaz gráfica de usuario dedicada al análisis de la organización colectiva de las células en imágenes multidimensionales complejas (Blin et al, en preparación). Tenga en cuenta que se puede encontrar más información sobre PickCells, así como documentación para los módulos específicos mencionados aquí: https://pickcellslab.frama.io/docs/. Tenga en cuenta también que la interfaz está sujeta a cambios a medida que seguimos mejorando el software. Si la interfaz difiere de lo que se muestra en la figura o el vídeo, consulte el manual en línea.
- Instale y ejecute PickCells siguiendo la documentación disponible en línea.
- Importe imágenes y verifique la exactitud de la información proporcionada (Figura4-1).
- Documente el nombre de cada canal.
- Núcleos de segmento basados en la señal de envolvente nuclear utilizando el módulo Nessys28 (Figura4-2) y proporcionan un prefijo ("núcleos" por ejemplo) que se utilizará para nombrar las imágenes segmentadas generadas.
NOTA: Las documentación sobre el uso y los ajustes de parámetros se pueden encontrar en https://framagit.org/pickcellslab/nessys. - Inspeccione y edite segmentaciones si es necesario, utilizando el módulo del editor de segmentación (Figura4-3)
NOTA: Si por alguna razón el proceso de segmentación no proporcionó resultados satisfactorios, elimine manualmente las imágenes en la carpeta de la base de datos y también elimine el nodo 'resultado de segmentación' en la vista MetaModel. A continuación, repita los pasos 7.4 y 7.5. Si la segmentación de sólo un pequeño subconjunto de imágenes no proporcionó resultados satisfactorios, utilice la aplicación independiente de Nessys (consulte el enlace en 7.4), intente la segmentación en las 'imágenes defectuosas' y reemplace el archivo correspondiente en la carpeta de la base de datos). - Segmente la señal de autofluorescencia del patrón utilizando el módulo de segmentación básica (Figura4-4)
- Proporcione un prefijo ("patrón", por ejemplo) que se utilizará para nombrar las imágenes segmentadas generadas.
- Seleccione el canal que contiene la señal de autofluorescencia.
- Aplicar reducción de ruido; generalmente el uso de un filtro gaussiano con un tamaño de núcleo de 10 x 10 x 0,5 vóxeles da resultados satisfactorios.
- Establezca el umbral inferior para que el fondo aparezca en azul mientras el primer plano aparece en blanco. Establezca también el umbral superior en su valor máximo para evitar que se excluyan las intensidades altas del resultado final (áreas rojas).
- Seleccione Omitir para el último paso.
- Haga clic en Finalizar y espere hasta que se procesen todas las imágenes.
- En cuanto a los núcleos, los resultados de segmentación ahora se pueden inspeccionar visualmente y corregir si es necesario utilizando el módulo del editor de segmentación (Figura4-5).
- Cree objetos de núcleos y calcule las entidades básicas de objetos.
- Inicie el módulo de características intrínsecas desde la barra de tareas a la izquierda de la interfaz principal (Figura4-6).
- Cierre los paneles Ellipsoid Fitter y Surface Extractor para mantener abierto solo el panel Características básicas.
- Elija Núcleo como tipo de objeto y elija el prefijo que aparece en el paso 7.4 para "imágenes segmentadas".
- Pulse Calcular y espere hasta que se hayan procesado todas las imágenes.
NOTA: Después de este paso, no será posible editar las segmentaciones de núcleos de nuevo.
- Cree objetos de matriz y calcule las entidades básicas de objetos. Repita los pasos 7.8.1 a 7.8.4, solo esta vez elija Tipo personalizado como tipo de objeto y el prefijo indicado en el paso 7.6.1 para imágenes segmentadas.
NOTA: Después de este paso, no será posible editar las segmentaciones de patrones de nuevo. - Almacene el nombre de la imagen a la que pertenece cada núcleo como atributo de núcleo.
- Haga clic en Datos > Nuevo atributo y seleccione Núcleo en el cuadro de diálogo emergente y haga clic en Aceptar.
- Seleccione Recopilar datos de otros objetos conectados al nodo y haga clic en Siguiente.
- En el panel izquierdo, seleccione Imagen y, a continuación, haga doble clic en la marca de interrogación debajo de la marca Finalizar en el panel Definición de ruta para establecer el nodo de imagen como destino de la ruta.
- Expanda el panel Atributos disponibles en el panel izquierdo y seleccione el atributo name.
- Expanda el panel Operación de reducción y seleccione Obtener unoy, a continuación, haga clic en el botón Cambiar y haga clic en Siguiente.
- Escriba "Nombre de imagen", pulse la tecla de tabulación y haga clic en Aceptar.
- Cree un atributo "coordenada normalizada" en objetos nuclei (Figura4-7).
- Adapte los pasos 7.10.1 a 7.10.6 para almacenar las coordenadas del centroide de patrón como un atributo de núcleo. Asigne a este nuevo atributo el nombre "Coordenada de patrón".
- A continuación, haga clic en Datos > Nuevo atributo, seleccione Núcleo y haga clic en Aceptar.
- Seleccione Definir una función entre vectores espaciales o direccionales del nodo y haga clic en Siguiente.
- En Tipo de función, seleccione Operación de matriz, en V1, seleccione Vector de elemento y, a continuación, Centroidey, a continuación, V2, seleccione Vector de elemento y, a continuación, Coordinación de patróne.
- Haga clic en Siguiente, Escriba "Coordenada normalizada" en el campo Nombre y haga clic en Finalizar.
- Exporte los datos a un archivo de valores separados por tabulaciones.
8. Análisis R
- Descargue e instale Rstudio.
NOTA: La información del software y los enlaces de descarga están disponibles en https://www.rstudio.com/. - Descargue los scripts de R necesarios para este análisis.
NOTA: Los scripts se pueden descargar desde el repositorio de GitLab: https://framagit.org/pickcellslab/hexmapr. - Abra Rstudio.
NOTA: Si ejecuta los scripts por primera vez, instale los paquetes de R necesarios (ggplot2 y escalas). - En Rstudio, abra binnedmap_template. Script R.
- Establezca el directorio de trabajo en la ubicación del archivo de origen.
- Siga las instrucciones proporcionadas en el script para adaptar el script a cualquier conjunto de datos dado con el fin de obtener mapas espaciales como se muestra en la figura5.
- Ejecute el script para generar mapas de densidad.
Resultados
El método de fotopatrones descrito aquí permite organizar con precisión las células cultivadas en colonias de formas y tamaños definidos. El éxito de este procedimiento debe ser claramente evidente inmediatamente después del procedimiento de sembrado celular (paso 3.7) como las células adheridos se agruparán de acuerdo con el diseño de la fotomáscara como se muestra en la Figura 2a. A 1 h después de la sembración celular, los patrones individuales pueden no ser completamente confluentes (sólo unas pocas células por patrón), sin embargo, a medida que las células proliferan con el tiempo, los patrones se colonizarán completamente con muy pocas células fuera de las superficies adhesivas (Figura2b). La apariencia exacta de la referencia cultural dependerá de la línea celular. Por ejemplo, mESC forman colonias en forma de cúpula10. Un chip donde el patrón no es claro de 1 a 2 h después de la sembración de celdas indica el fracaso del procedimiento (Figura2c,d).
Las colonias grandes y gruesas a veces pueden ser difíciles de manchar homogéneamente. Sugerimos fijar y permeabilizar las células en un solo paso (sección 4) ya que esto puede mejorar la penetración de anticuerpos29. Esta es la razón por la que la solución fijativa elegida contiene un detergente. La Figura 3 muestra la señal de fluorescencia que se espera después de la inmunomancha. Observe que las células positivas I1 brillantes se encuentran dentro de regiones densas (regiones NPC brillantes) de las colonias (Figura3A). Sugerencias como esta son útiles para evaluar la calidad del procedimiento de tinción de anticuerpos. Observe también que los micropatrones creados con la técnica actual son autofluorescentes. Esta señal (Figura3A,B a la izquierda la mayoría de las imágenes) es útil durante la etapa de análisis para registrar espacialmente las colonias entre sí y crear los resultados mostrados en la Figura5. La señal de autofluorescencia es generalmente la más brillante cuando la muestra se excita con un láser de 405 nm y este canal debe dejarse sin manchar para este propósito. La Figura 3 también muestra cómo las celdas están restringidas con precisión en patrones de diferentes formas.
El análisis de los datos de imagen se realiza en PickCells, un software libre y de código abierto desarrollado en nuestro laboratorio (Blin et al., en preparación). Este software incluye los módulos de análisis de imágenes para leer y ordenar imágenes confocales (Figura4-1), segmentar (Figura4-2,4-4) y seleccionar objetos segmentados (Figura4-3,4-5), para calcular objetos de cálculo tales como coordenadas o intensidad media (Figura4-6) y para exportar los datos (Figura4-7,4-8). Es importante destacar que desarrollamos un método de segmentación nuclear robusto llamado Nessys28 que es particularmente adecuado para poblaciones densas y heterogéneas de células como las células cultivadas en micropatrones (Figura3). Figura 4 -2 muestra una salida representativa del módulo Nessys donde cada celda individual recibe con precisión una identidad de color única. Sólo debe ser necesaria una edición mínima, sin embargo la edición es factible si el usuario así lo decide (Figura 4-3). Finalmente PickCells proporciona una serie de módulos de visualización para visualizar los datos. Un ejemplo se da en la Figura 4-6:una colonia en forma de anillo se representa en 3D donde los núcleos están codificados por colores de acuerdo con su posición a lo largo del eje z. Una vez que el análisis se valida en PickCells, los datos se pueden exportar para crear los mapas espaciales en R utilizando los scripts disponibles en (https://framagit.org/pickcellslab/hexmapr) como se muestra en la Figura 530.
Hemos demostrado recientemente que el confinamiento espacial de mESC en micropatrones de disco o elipse pequeños (30.000 m2) guía el patrón de una subpoblación de células que expresa niforique el marcador mesodérmico Tbra10. Por lo tanto, para ilustrar nuestro método aquí, preguntamos si el patrón de Tbra puede ser influenciado por la señalización BMP en colonias más grandes (90.000 m2). La Figura 5A muestra que cuando el mESC se cultiva en micropatrones de disco grandes, las células Tbra+ se limitan preferentemente a la periferia del patrón (mapa de densidad Tbra+), donde la densidad celular local es la más baja (ver el mapa azul a la izquierda de la Figura 5A ). Este patrón de Tbra es confirmado por el mapa de intensidad media de Tbra.
Estos datos demuestran que el método puede revelar información subvisual. De hecho, a partir de la Figura3, la inspección visual de una colonia no es suficiente para identificar cualquier forma de organización espacial en la expresión Tbra. Esto se explica notablemente por la importante variedad de coloniaa a colonia que se cuantifica y se muestra en el panel más a la derecha de la Figura 5A.
La técnica también muestra que no existe ningún patrón detectable para el Id1 (un objetivo de señalización BMP) que puede indicar que el patrón T no es impulsado por la señalización BMP en este contexto.
El micropatrón permite forzar a las colonias a adoptar casi cualquier geometría deseada. Esto es particularmente útil para interrogar cómo el sistema responde a varias geometrías. Por ejemplo, podemos razonar que si un gradiente de morógeno se acumula en el centro de la colonia, crear un agujero en la colonia interrumpiría este gradiente. Curiosamente todavía observamos el patrón en un micropatrón de anillo aunque de una manera menos robusta (Figura 5B).
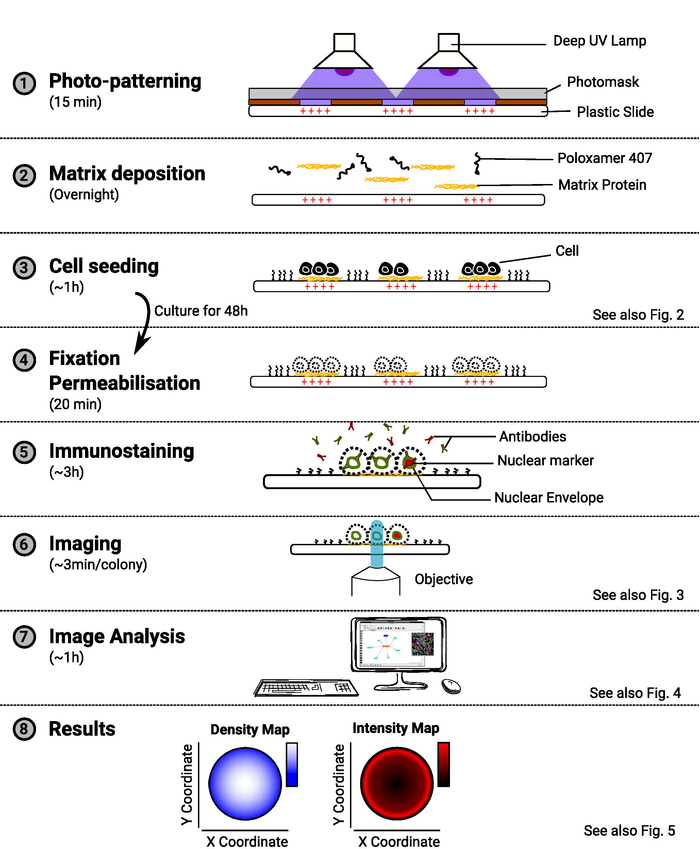
Figura 1: Descripción general del método. El diagrama que muestra los pasos principales del método. Para cada paso, la cantidad estimada de tiempo se indica bajo el nombre de la tarea y un esquema ilustra el propósito del procedimiento. También se proporciona una referencia a una figura relevante cuando está disponible. Haga clic aquí para ver una versión más grande de esta figura.
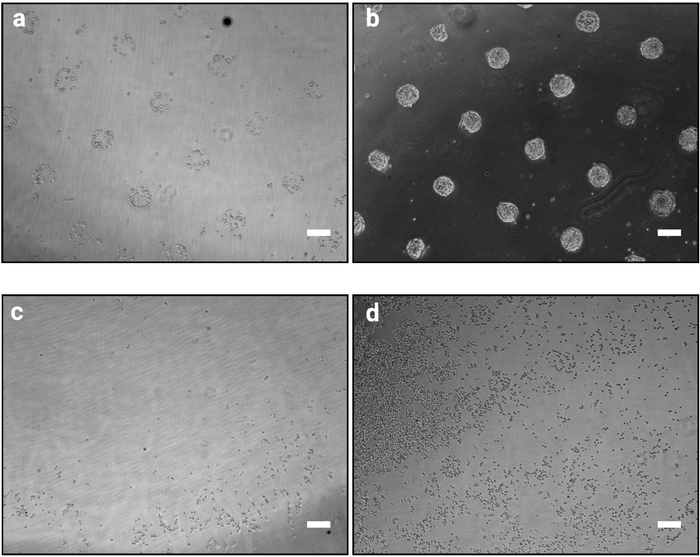
Figura 2: Apariencia del cultivo 1 h y 48 h después de sembrar las células en micropatrones. Imágenes de Brightfield de mESC sembradas sobre micropatrones. (a) Organización celular esperada 1 h después de la sembración, los patrones deben ser claramente identificables. (b) Resultado esperado después de 48 h de cultivos. mESC han proliferado y todavía están estrictamente confinados a las formas de patrón. (c–d) Posibles resultados no óptimos, ya sea muy pocas células se adhieren al plástico excepto en la periferia de la diapositiva (c) o las células se adhieren entre los patrones (d). Consulte la Tabla 2 para obtener una guía de solución de problemas. Haga clic aquí para ver una versión más grande de esta figura.
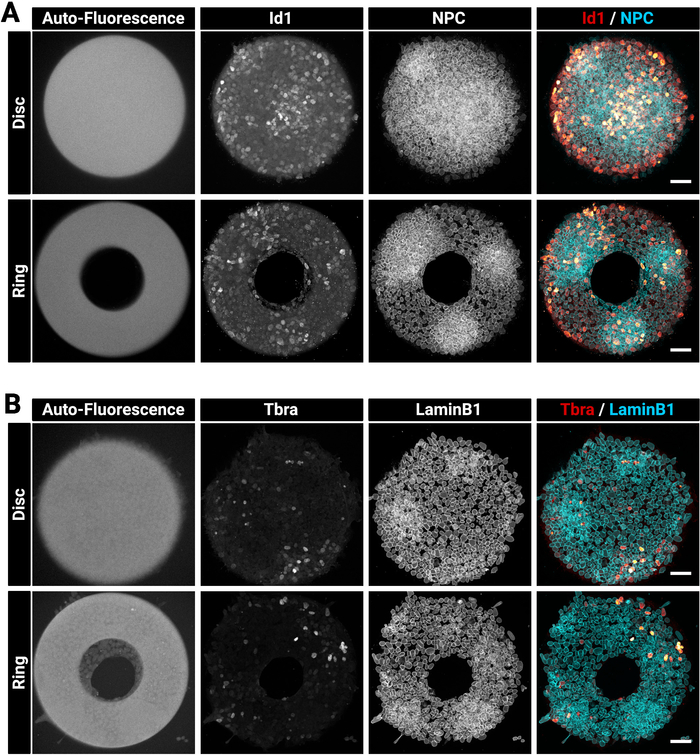
Figura 3: Imágenes confocales representativas de colonias inmunomanchadas cultivadas con micropatrones.
Colonias representativas de mESC después de la inmunofluorescencia para (A) Id1 y Nuclear Pore Complex o (B) Tbra y LaminB1. Para cada tinción, se muestra una colonia cultivada en un micropatrón de disco y una colonia cultivada en un micropatrón de anillo. Los canales individuales se proporcionan como imágenes en escala de grises. Observe la señal de autofluorescencia clara del micropatrón (excitación láser de 405 nm). La barra de escala representa 50 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.
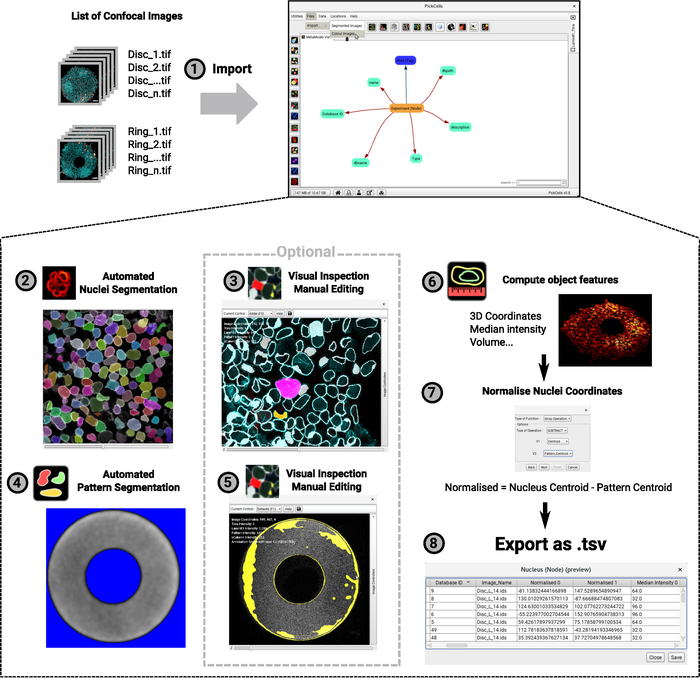
Figura 4: Gráfico de flujo del procedimiento de análisis de imágenes. Se importa una lista de imágenes confocales 3D en PickCells para su análisis (1). En este ejemplo se muestra un experimento con dos formas distintas (discos y anillos como en la Figura2). La convención de nomenclatura de imagen se muestra a la izquierda y la interfaz PickCells a la derecha. A continuación, el módulo Nessys se utiliza para segmentar automáticamente los núcleos (2). En la captura de pantalla, a cada núcleo individual se le da un color único que indica una segmentación precisa. La autofluorescencia del patrón también se segmenta, esta vez, utilizando el módulo de "segmentación básica" (4). El fondo aparece en azul y la señal blanca se definirá como la forma del patrón. Las formas segmentadas se inspeccionan visualmente para garantizar una segmentación precisa y se editan si es necesario utilizando el módulo del editor de segmentación (3–5). Las capturas de pantalla muestran el contorno de las formas detectadas. Las formas rosa y amarilla han sido editadas. Por último, las entidades de objetos se calculan y exportan a un archivo para procesarlas posteriormente en R (6–7). Se proporciona una captura de pantalla de una colonia representada como una vista 3D (6). Para los pasos 2 a 6, los iconos que se encuentran en la interfaz PickCells en el momento de escribir este artículo se indican junto al índice de pasos. Haga clic aquí para ver una versión más grande de esta figura.
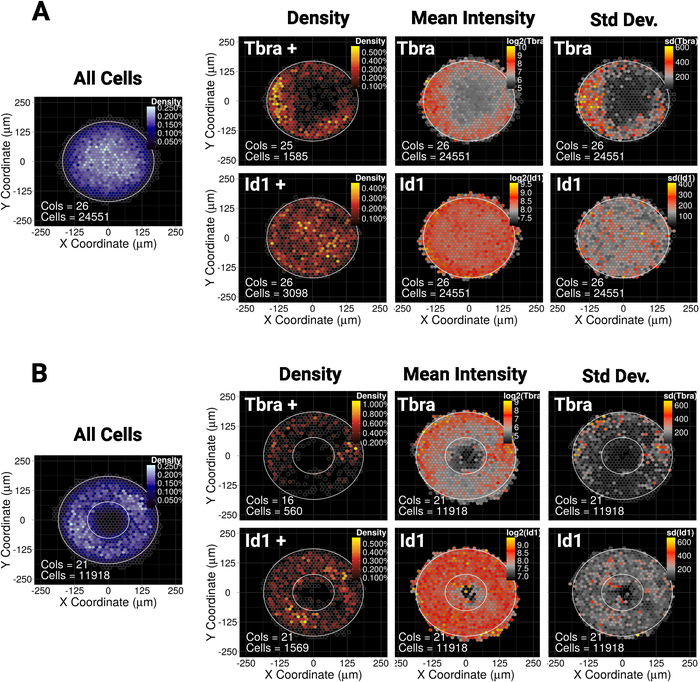
Figura 5: Resultados representativos de dos factores de transcripción diferentes y formas micropatrones
Mapa espacial trenzado para mESC cultivado durante48 h en (A) micropatrones en forma de disco o (B ) micropatrones en forma de anillo. Para cada forma de micropatrón, el mapa de densidad de celda, independientemente del fenotipo celular, se muestra a la izquierda con una escala de color azul. Luego, para cada marcador (Tbra en la fila superior e Id1 en la fila inferior), se proporcionan tres mapas distintos, de izquierda a derecha: mapa de densidad de celda de marcador que expresa solo celdas (análisis basado en umbrales), el mapa de la intensidad media del marcador (log2) y el mapa del mapa del desviación estándar de la intensidad del marcador. Las intensidades se dan como unidades arbitrarias de fluorescencia. Para cada mapa, la forma de micropatrón se da como un contorno blanco. Haga clic aquí para ver una versión más grande de esta figura.
| Tipo ECM | Gelatina | Fibronectina | Matriz de membrana de sótano | |
| Concentración | Concentración de ECM | 1 mg/ml | 20 g/ml | 200 g/ml |
| Concentración de poloxámero 407 | 500 g/ml | 400 g/ml | 1 mg/ml | |
| Probado con | mESC | Sí | Sí | Sí |
| mepiSC | No | Sí | Sí | |
| Medio libre de suero | No | Sí | Sí |
Tabla 1: Concentraciones probadas de poloxámero 407 y ECM. Esta tabla proporciona una visión general de las concentraciones de ECM y poloxámero 407 que probamos en nuestro laboratorio. Para cada combinación ECM/poloxamer 407, se muestra el tipo de celda para el que se logró el patrón con éxito, así como si el cultivo contenía suero o no. mESC - célula madre embrionaria de ratón, mEpiSC - célula madre de epiblasto de ratón.
| Procedimiento | Observación | Posible problema | Solución |
| Micropatrón | Acoplamiento celular bajo | relación de concentración inapropiada ECM/poloxamero 307 | Aumentar la relación de concentración ECM/poloxamer 307 |
| Tiempo de fijación celular demasiado corto | Aumentar el tiempo de incubación para dar suficiente tiempo a las células para adherirse adecuadamente a los patrones (paso 3.4). Para optimizar este paso, comprobar las células bajo un microscopio puede ayudar a detectar un cambio en la morfología celular que indica que las células han comenzado a adherirse. | ||
| Lavados demasiado intensos (paso 3.6) | Cuando reemplace el medio, evite el medio de pipeteo directamente sobre las virutas. En su lugar, en su lugar, entuba rinde suavemente el medio en las paredes del pozo en su lugar | ||
| Las células se adhieren entre los patrones | relación de concentración inapropiada ECM/poloxamero 307 | Disminuir la relación de concentración ECM/poloxamero 407 | |
| Tiempo de fijación celular demasiado largo | Disminuir el tiempo de incubación (paso 3.4). | ||
| Lavados ineficaces (paso 3.6) | Agitar la placa vigorosamente suele ser suficiente para separar las células en exceso. Para los tipos de celda que tienden a adherirse fuertemente a la viruta, pipetear directamente en el chip puede mejorar el resultado. Aumentar el número de lavados también puede ayudar, en particular para asegurarse de que ninguna célula permanezca flotando en el medio después de este paso. | ||
| Las celdas no siguen estrictamente la forma del patrón | Tipo de celda 'incompatible' y geometría de patrón | Planifique/diseñe varias geometrías/tamaños que se agregarán a la máscara de fotomáscara para poder probar e identificar el tamaño de patrón óptimo para una forma de patrón y un tipo de celda determinados. Por favor, vea la sección "Limitaciones" en la discusión | |
| Foto-patrón no óptimo, esto se puede diagnosticar observando la nitidez de la señal de autofluorescencia. Los límites del patrón deben verse nítidos como en la Fig. 2. Si los bordes de los patrones aparecen borrosos, es necesario mejorar el paso de fotopatrones. | Los bordes borrosos del patrón indican que la diapositiva de plástico no estaba lo suficientemente cerca de la superficie de la máscara durante el paso de iluminación. Asegúrese de que las piezas que sujetan las diapositivas a la fotomáscara estén uniformes y de que se aplique una presión constante y suficiente al ensamblaje durante el procedimiento de iluminación. | ||
| Tinción | Tinción no homogénea | tiempo de incubación de anticuerpos demasiado corto | Aumentar el tiempo de incubación de anticuerpos (hasta 24h a temperatura ambiente) |
| colonias aplanadas durante el procedimiento de montaje | Monte diapositivas de micropatrón en una cámara como chamlide o cámaras cytoo para realizar tanto inmunomanchas como imágenes sin necesidad de montar las células. Esto preservará mejor la estructura 3D de las colonias. | ||
| Separar colonias durante el procedimiento de tinción | Deswetting del chip | Deje suficiente medio o utilice 2 pipetas, una para extraer el medio y la otra para añadir la solución fresca | |
| Las colonias aparecen cizalladas bajo el microscopio | colonias fueron cizadas mientras se montaba el chip en la diapositiva de microscopía | Sea muy suave al montar las virutas. Alternativamente, monte micropatrones se desliza en una cámara como chamlide o citocámaras para realizar tanto inmunomanchas como imágenes sin necesidad de montar las células. Esto también preserva la ultraestructura de la colonia. |
Tabla 2: Guía de solución de problemas. Esta tabla proporciona una visión general de los posibles resultados subóptimos. Las posibles fuentes de los problemas también se enumeran junto con las soluciones recomendadas.
Discusión
Aquí describimos un método para analizar patrones emergentes en cultivos de células. Un enfoque simplificado de micropatrones se utiliza para estandarizar la forma y el tamaño de las colonias celulares, y presentamos herramientas de análisis de imágenes y scripts de R que permiten la detección y cuantificación de patrones dentro de estas colonias.
El oleoducto que proponemos es similar en cierta medida con un método publicado anteriormente31 donde los autores se centran en las condiciones de cultivo, utilizando micropatrones disponibles comercialmente, para obtener la formación de capas germinales reproducibles en las colonias de ESC para el estudio de los primeros eventos de gastrulación in vitro. Nuestro objetivo está más centrado en proporcionar una tubería generalizable para el descubrimiento de la formación de patrones in vitro donde la organización colectiva de las células sólo puede hacerse evidente después del análisis estadístico. Por esta razón, proporcionamos un sólido flujo de trabajo de análisis de imágenes que permite la identificación y el análisis precisos de la posición nuclear en el espacio 3D en múltiples colonias (véase también la sección "Ventajas y limitaciones del método de detección de patrones" de este discusión). También decidimos desarrollar un enfoque de micropatrones interno que ofrezca una alternativa más flexible y barata a las soluciones disponibles comercialmente a largo plazo, lo que esperamos sea útil para la comunidad.
Por último, observamos que durante la revisión de este manuscrito, se ha publicado un nuevo paquete para el análisis de patrones in vitro similares a nuestros scripts R32. Este nuevo paquete acepta tablas de entidades de celda como entrada que se pueden obtener de plataformas de imágenes de alto rendimiento. Creemos que la tabla de características de núcleos generada en el paso 7 de nuestro protocolo podría servir en principio como una entrada a este nuevo paquete, aunque no hemos probado esta posibilidad nosotros mismos.
Adaptabilidad del método a otros tipos de células y geometrías de colonias
Presentamos este enfoque en el contexto del estudio de la aparición de factores de transcripción mesodérmica en cultivos de células pluripotentes en presencia de suero. Sin embargo, el método es fácilmente adaptable a otros tipos de células y a cultivos libres de suero, aunque puede ser necesario optimizar las concentraciones de ECM/poloxamer 407 (consulte la Tabla 1 para las concentraciones probadas y la Tabla 2 para obtener una guía de solución de problemas). El método también se puede adaptar a tamaños más grandes o más pequeños de micropatrones y a una amplia gama de formas según las necesidades del usuario. Sin embargo, al establecer el método, es importante tener en cuenta que no todas las combinaciones de forma/tipo de celda son óptimas. Por ejemplo, mESC expresa altos niveles de E-cadherin33,34 permitiendo que estas células formen estructuras colectivas que abarquen áreas que están desprovistas de ECM. Estas celdas no siguen estrictamente geometrías con ángulos agudos o que incluyen pequeños agujeros en el patrón. Observe por ejemplo que en el anillo de la Figura 3B,las células están en el proceso de colonizar el área central. En nuestras manos una zona central más pequeña no obligó a mESC a formar colonias en forma de anillo. Por lo tanto, es muy recomendable incluir una diversidad de geometrías mientras se diseña la fotomáscara para poder probar e identificar los tamaños y curvaturas óptimos que serán adecuados para el tipo de celda de elección.
Otro factor importante a tener en cuenta es la duración del experimento y la tasa de proliferación de las células. Para algunos tipos de células que proliferan rápidamente (incluyendo células pluripotentes) puede ser difícil mantener las células en micropatrones durante muchos días (Para mESC tres días es un máximo). Además, la sembración de células en micropatrones no siempre ocurre de manera óptima para cada colonia, por lo que es aconsejable sembrar un exceso de colonias para tener repuestos.
Ventajas y limitaciones del método de detección de patrones
Una ventaja particular del método es la capacidad de detectar patrones "promediados" mediante la combinación de resultados de análisis de imágenes de varias colonias de réplica (Figura5). Esto puede revelar eventos de patrones que no son evidentes a partir de la inspección de colonias individuales. Una desventaja de este enfoque de "promediación" es que puede pasar por alto ciertos tipos de patrones repetitivos, por ejemplo pequeñas manchas o rayas estrechas. Sin embargo, estos tipos de patrón pueden revelarseen su lugar con una combinación de tamaños de patrón cuidadosamente elegidos 8. Además, la canalización de análisis de imágenes descrita aquí proporciona datos cuantitativosen la resolución de una sola célula y colonia que ofrece la posibilidad de investigar el nivel de variabilidad entre colonias (Figura 5) o realizar análisis de vecinos en múltiples escalas10.
Otra ventaja importante del método de promediación es que ofrece la oportunidad de mapear la ubicación preferencial de muchos marcadores sin estar limitado por los fluoróforos disponibles de los canales de detección. De hecho, aunque hacemos uso de sólo dos marcadores de diferenciación en el trabajo presentado aquí, la capacidad de estandarizar colonias y extraer patrones "promediados" permite comparar los mapas de distribución de diferentes conjuntos de colonias en orden para revelar las relaciones espaciales generalizadas de los marcadores entre sí.
Además, aunque nos centramos en estudiar los marcadores de diferenciación, el método de análisis puede ampliarse para estudiar otros procesos biológicos para los que se dispone de marcadores nucleares. Por ejemplo, la micropatrón de una línea celular que contiene un indicador35 del ciclo celular de ubiquitinación de fluorescencia (FUCCI) permitiría estudiar cómo la geometría del nivel de colonia puede influir en los eventos del ciclo celular en el grupo.
Direcciones futuras
El método es susceptible a un análisis de imágenes de rendimiento medio, sin embargo, la adquisición de imágenes actualmente no está totalmente automatizada y puede llegar a ser limitante para experimentos muy grandes. Los arreglos regulares de las colonias deben permitir crear rutinas de adquisición totalmente automatizadas similares a las que se han desarrollado para un promedio de células monocelulares de20. Sin embargo, debido a que el tamaño del campo requerido para tomar una imagen de una colonia es grande, posiblemente requiere mosaicos, y debido a que las colonias son tridimensionales, es muy deseable reducir tanto el tamaño del conjunto de datos como el tiempo de adquisición mediante imágenes sólo colonias relevantes. Por lo tanto, los esfuerzos futuros pueden dedicarse a desarrollar un microscopio "inteligente" capaz de identificar colonias relevantes y adaptar las coordenadas de imagen a cada muestra. Esto no sólo reducirá el tiempo y el esfuerzo, sino que también evitará posibles sesgos del operador.
Las canalizaciones de análisis también pueden ser más eficientes al reducir el número de pasos que el usuario debe tomar. Tenemos planes para construir un mecanismo de construcción de tuberías e integrar R directamente en nuestro software (véase también problemas pickcells-api-3 y pickcells-rjava-1 en el rastreador de problemas de nuestros repositorios de código [https://framagit.org/groups/pickcellslab/-/issues]). La automatización completa del procedimiento de análisis reducirá el tiempo y el esfuerzo y limitará los posibles errores de usuario.
Por último, observamos que nuestro método de análisis aún no captura completamente la naturaleza dinámica del patrón celular. Se puede extraer cierta información dinámica limitada examinando una serie temporal de imágenes instantáneas8,10,36. Sin embargo, ser capaz de registrar la historia de la población celular es altamente deseable si queremos entender mejor cómo surge el patrón. Una limitación es que el seguimiento preciso de células individuales en una población de células densas 3D sigue siendo una tarea muy difícil37. Nuestro método de detección celular utiliza la envolvente nuclear y funciona particularmente bien en poblaciones de células densas y superpuestas28. Los reporteros en vivo del sobre nuclear están fácilmente disponibles28,38 y una ventaja de la técnica de micropatrones es que se puede utilizar para evitar que las células se muevan fuera del campo de visión durante la toma de imágenes a largo plazo. En general, estamos seguros de que el seguimiento automatizado de las células será alcanzable utilizando una combinación de herramientas recientemente establecidas28,39,40 y que esto debe aportar nuevos conocimientos en la principios de autoorganización.
Divulgaciones
Los autores no tienen nada que revelar
Agradecimientos
Este trabajo fue financiado por una beca post-doctoral de Sir Henry Wellcome (WT100133 a G.B.), una beca Wellcome Trust Senior (WT103789AIA a S.L.), y una beca de doctorado Wellcome Trust a (108906/Z/15/Z a D.W.). También estamos agradecidos al Dr. Manuel Thery por su consejo para adaptar la técnica del fotopatrón.
Materiales
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Gibco | 31350-010 | handle in fume hood |
| 1× Master quartz anti-reflective chromium photomask | Toppan Photomask | Custom design | |
| 24-well plates | Corning | 3526 | |
| 4-well plates | Nunclon Delta | 176740 | |
| 5% Donkey Serum | Sigma | D9663 | |
| Anti Nuclear pore complex | Abcam | ab24609 | Mouse monoclonal, use 1:1000 |
| Anti-Id1 | Biocheck | 37-2 | Rabbit polyclonal, use 1:200 |
| Anti-Lamin B1 | Abcam | ab16048 | Rabbit polyclonal, use 1:1000 |
| Anti-Tbra | R&D | AF2085 | Goat ployclonal, use 1:400 |
| Blocking Solution | NA | NA | 0.1% TritonX-100 0.01% Pluronic F-127 5% Donkey Serum 0.003% Sodium Azide In PBS |
| CGR8 mESC | NA | NA | Reference: Mountford et al. PNAS, 1994 |
| Fixation solution | NA | NA | 4% PFA diluted in washing solution |
| Foetal bovine serum | Gibco | 10270-106 | Serum batches must be tested to ensure compatibility with ESC maintenance |
| Gelatin | Sigma | G1890 | |
| Glasgow Minimum Essential Medium | Sigma | G5154 | |
| Laboratory Film | VWR | 291-1212 | |
| Layout Editor Software | LayoutEditor | NA | https://layouteditor.com/ |
| Layout Editor Software | Klayout | NA | https://www.klayout.de/ |
| L-glutamine | Gibco | 25030-024 | |
| LIF | Millipore | ESG1107 | |
| MEM NEAA | Gibco | 11140-035 | |
| mESC Culture medium | NA | NA | GMEM 10% FCS 100 U/ml LIF 100 nM 2-mercaptoethanol 1× non-essential amino acids, 2 mM L- glutamine 1 mM sodium pyruvate |
| NH4Cl 50mM | Sigma | 9718 | |
| Paraformaldehyde | Sigma | 158127 | CAUTION: Toxic, handle undiluted stocks in fume hood and wear protective equipment |
| PBS (immunostaining) | Sigma | P4417 | Dilute in 200ml of ddH2O |
| PBS (tissue culture) | Gibco | 11140-035 | |
| Plastic slides | Ibidi | IB-10813 | |
| Poloxamer 407 | Sigma | P2443 | |
| ProLong Gold Antifade Mountant | Molecular Probes | P36930 | |
| Sodium Azide | Sigma | 8591 | CAUTION: Toxic, handle in fume hood and wear protective equipment |
| Sodium pyruvate | Gibco | 11360070 | |
| TritonX-100 | Sigma | T8532 | |
| Trypsin | Gibco | 25200056 | |
| UVO cleaner | Jetlight, USA | 42-220 | CAUTION: Follow manufacturer’s safety recommendations |
| Washing Solution | NA | NA | 0.1% TritonX-100 0.01% Pluronic F-127 In PBS |
Referencias
- Sasai, Y. Cytosystems dynamics in self-organization of tissue architecture. Nature. 493 (7432), 318-326 (2013).
- Turner, D. A., Baillie-Johnson, P., Martinez Arias, A. Organoids and the genetically encoded self-assembly of embryonic stem cells. BioEssays. 38 (2), 181-191 (2016).
- Simunovic, M., Brivanlou, A. H. Embryoids, organoids and gastruloids: new approaches to understanding embryogenesis. Development. 144 (6), 976-985 (2017).
- Tewary, M., Shakiba, N., Zandstra, P. W. Stem cell bioengineering: building from stem cell biology. Nature Reviews. Genetics. 19 (10), 595-614 (2018).
- Shahbazi, M. N., Zernicka-Goetz, M. Deconstructing and reconstructing the mouse and human early embryo. Nature Cell Biology. 20 (8), 878 (2018).
- Laurent, J., et al. Convergence of microengineering and cellular self-organization towards functional tissue manufacturing. Nature Biomedical Engineering. 1 (12), 939 (2017).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Developmental Cell. 6 (4), 483-495 (2004).
- Tewary, M., et al. A stepwise model of Reaction-Diffusion and Positional-Information governs self-organized human peri-gastrulation-like patterning. Development. , (2017).
- Warmflash, A., Sorre, B., Etoc, F., Siggia, E. D., Brivanlou, A. H. A method to recapitulate early embryonic spatial patterning in human embryonic stem cells. Nature Methods. 11 (8), 847-854 (2014).
- Blin, G., Wisniewski, D., Picart, C., Thery, M., Puceat, M., Lowell, S. Geometrical confinement controls the asymmetric patterning of brachyury in cultures of pluripotent cells. Development. 145 (18), (2018).
- . . Micropatterning in cell biology. Pt. A. , (2014).
- Bauwens, C. L., et al. Control of human embryonic stem cell colony and aggregate size heterogeneity influences differentiation trajectories. Stem Cells. 26 (9), 2300-2310 (2008).
- Heemskerk, I., Burt, K., Miller, M., Chabra, S., Guerra, M. C., Warmflash, A. Morphogen dynamics control patterning in a stem cell model of the human embryo. bioRxiv. , 202366 (2017).
- Nemashkalo, A., Ruzo, A., Heemskerk, I., Warmflash, A. Morphogen and community effects determine cell fates in response to BMP4 signaling in human embryonic stem cells. Development. 144 (17), 3042-3053 (2017).
- Peerani, R., Onishi, K., Mahdavi, A., Kumacheva, E., Zandstra, P. W. Manipulation of signaling thresholds in “engineered stem cell niches” identifies design criteria for pluripotent stem cell screens. PloS One. 4 (7), e6438 (2009).
- Peerani, R., et al. Niche-mediated control of human embryonic stem cell self-renewal and differentiation. The EMBO journal. 26 (22), 4744-4755 (2007).
- Etoc, F., et al. A Balance between Secreted Inhibitors and Edge Sensing Controls Gastruloid Self-Organization. Developmental Cell. 39 (3), 302-315 (2016).
- Azioune, A., Carpi, N., Tseng, Q., Théry, M., Piel, M. Protein micropatterns: A direct printing protocol using deep UVs. Methods in Cell Biology. 97, 133-146 (2010).
- Théry, M. Micropatterning as a tool to decipher cell morphogenesis and functions. Journal of Cell Science. 123 (Pt 24), 4201-4213 (2010).
- Degot, S., et al. Improved Visualization and Quantitative Analysis of Drug Effects Using Micropatterned Cells. JoVE (Journal of Visualized Experiments). (46), e2514 (2010).
- Beddington, R. S. P., Rashbass, P., Wilson, V. Brachyury - a gene affecting mouse gastrulation and early organogenesis. Development. 116 (Supplement), 157-165 (1992).
- Wilkinson, D. G., Bhatt, S., Herrmann, B. G. Expression pattern of the mouse T gene and its role in mesoderm formation. Nature. 343 (6259), 657-659 (1990).
- Hollnagel, A., Oehlmann, V., Heymer, J., Rüther, U., Nordheim, A. Id Genes Are Direct Targets of Bone Morphogenetic Protein Induction in Embryonic Stem Cells. Journal of Biological Chemistry. 274 (28), 19838-19845 (1999).
- Mountford, P., et al. Dicistronic targeting constructs: reporters and modifiers of mammalian gene expression. Proceedings of the National Academy of Sciences of the United States of America. 91 (10), 4303-4307 (1994).
- Smith, A. G. Culture and differentiation of embryonic stem cells. Journal of tissue culture methods. 13 (2), 89-94 (1991).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Preibisch, S., Saalfeld, S., Tomancak, P. Globally optimal stitching of tiled 3D microscopic image acquisitions. Bioinformatics. 25 (11), 1463-1465 (2009).
- Blin, G., Sadurska, D., Migueles, R. P., Chen, N., Watson, J. A., Lowell, S. Nessys: a novel method for accurate nuclear segmentation in 3D. bioRxiv. , 502872 (2018).
- Weiswald, L. -. B., Guinebretière, J. -. M., Richon, S., Bellet, D., Saubaméa, B., Dangles-Marie, V. In situ protein expression in tumour spheres: development of an immunostaining protocol for confocal microscopy. BMC cancer. 10, 106 (2010).
- Deglincerti, A., et al. Self-organization of human embryonic stem cells on micropatterns. Nature Protocols. 11 (11), 2223-2232 (2016).
- Ostblom, J., Nazareth, E. J. P., Tewary, M., Zandstra, P. W. Context-explorer: Analysis of spatially organized protein expression in high-throughput screens. PLOS Computational Biology. 15 (1), e1006384 (2019).
- Larue, L., Ohsugi, M., Hirchenhain, J., Kemler, R. E-cadherin null mutant embryos fail to form a trophectoderm epithelium. Proceedings of the National Academy of Sciences. 91 (17), 8263-8267 (1994).
- Pieters, T., van Roy, F. Role of cell-cell adhesion complexes in embryonic stem cell biology. J Cell Sci. 127 (12), 2603-2613 (2014).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Morgani, S. M., Metzger, J. J., Nichols, J., Siggia, E. D., Hadjantonakis, A. -. K. Micropattern differentiation of mouse pluripotent stem cells recapitulates embryo regionalized cell fate patterning. eLife. 7, e32839 (2018).
- Ulman, V., et al. An objective comparison of cell-tracking algorithms. Nature Methods. 14 (12), 1141 (2017).
- Moir, R. D., Yoon, M., Khuon, S., Goldman, R. D. Nuclear Lamins a and B1. The Journal of Cell Biology. 151 (6), 1155-1168 (2000).
- McDole, K., et al. In Toto Imaging and Reconstruction of Post-Implantation Mouse Development at the Single-Cell Level. Cell. 175 (3), 859-876 (2018).
- Stegmaier, J., et al. Real-Time Three-Dimensional Cell Segmentation in Large-Scale Microscopy Data of Developing Embryos. Developmental Cell. 36 (2), 225-240 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados