このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
哺乳類細胞におけるCRISPRベースの遺伝子スクリーンのプール
要約
CRISPR-Cas9技術は、あらゆる細胞型の哺乳類ゲノムを正確に編集する効率的な方法を提供し、ゲノム全体の遺伝スクリーンを実行する新しい手段を表します。プールされたゲノム全体のCRISPR-Cas9スクリーンのパフォーマンスを成功させるには必要な手順を説明する詳細なプロトコルをここで提供します。
要約
CRISPR-Casシステムを用いてゲノム編集を行い、様々な生物のゲノムを精密に編集する能力を大幅に向上させています。哺乳類細胞の文脈では、この技術は、機能的ゲノミクス研究のためのゲノム全体の遺伝的スクリーンを実行するための新しい手段を表す。すべての開いた読み取りフレームを標的とするガイドRNA(sgRNA)のライブラリは、遺伝子機能および細胞プロセスを含む特定の表現型をスクリーニングすることができる細胞の単一プール内の何千もの遺伝的摂動の顔の生成を可能にする公平で体系的な方法。CRISPR-Casスクリーンは、細胞の型示体の遺伝的青写真を明らかにするために、簡単で効率的で安価な方法を研究者に提供します。さらに、様々な細胞株や異なるがんタイプから行われるスクリーンの微分解析により、腫瘍細胞に文脈的に不可欠な遺伝子を同定し、特定の抗癌治療の潜在的な標的を明らかにすることができます。ヒト細胞でゲノム全体のスクリーンを実行することは、数千万個の細胞の処理を伴い、大規模なデータセットの分析を必要とするので、困難な場合があります。セルラインの特性認識、CRISPR ライブラリの考慮事項、解析中の CRISPR テクノロジの制限と機能の理解など、これらの画面の詳細は見落とされることがよくあります。ここでは、プールされたゲノム全体のCRISPR-Cas9ベースの画面の正常なパフォーマンスのための詳細なプロトコルを提供します。
概要
CRISPR-Casは、クラスター化された定期的に間隔をあけた短いパリンドロミックリピートおよびCRISPR関連ヌクレアーゼの略で、合成ガイドRNA(sgRNA)と複合体内の単一のヌクレアーゼタンパク質(例えば、Cas9)から構成される。このリボヌクレオタンパク質複合体は、Cas9酵素を標的とし、特定のゲノム遺伝子座1で二本鎖DNA破断を誘導する。二本鎖の切断は、相同性指向修復(HDR)を介して修復することができ、より一般的には、非相同エンド結合(NHEJ)を介して、遺伝子機能を頻繁に破壊する挿入および/または欠失(INDELS)をもたらすエラーが発生しやすい修復メカニズム1.CRISPRの効率性とシンプルさは、以前のゲノム編集技術(すなわち、亜鉛フィンガーヌクレアーゼ(ZNF)または転写活性化剤様エフェクターヌクレアーゼ()をはるかに上回る、これまで達成不可能なレベルのゲノムターゲティングを可能にします。TALENS)は、いずれも設計の複雑さの増大、トランスフェクション効率の低下、多重遺伝子編集2の限界に苦しんでいる。
CRISPRシングルガイドRNAベースのゲノム編集の基礎研究アプリケーションは、科学者が効率的かつ安価に個々の遺伝子の機能と遺伝的相互作用ネットワークのトポロジーを問うことを可能にしました。機能的ゲノムワイドスクリーンを実行する能力は、特にRNA干渉(RNAi)や遺伝子トラップ変異などの以前の遺伝的摂動技術と比較した場合、CRISPR-Casシステムの使用によって大幅に強化されました。特に、RNAiは高いオフターゲット効果と不完全なノックダウンに苦しんでおり、CRISPR3、4、5に比べて感度と特異性が低く、遺伝子トラップ法はハプロイドでのみ実現可能です。機能喪失画面のセルは、尋問可能なセルモデルの範囲を制限する 6.完全な遺伝子ノックアウトを生成するCRISPRの能力は、低ノイズ、最小限のオフターゲット効果と試薬5全体の一貫した活性で、変異型の現象型を調知するためのより生物学的に堅牢なシステムを提供します。ヒトゲノム全体を標的とするCRISPR-Cas9 sgRNAライブラリーが広く利用可能となり、1回の実験で何千もの遺伝子ノックアウトを同時に生成できる3、7、8、9.
トロントノックアウト(TKO)ライブラリー(Addgeneを通じて入手可能)と呼ばれる独自のCRISPR-Cas9ゲノムワイドsgRNAレンチウイルスライブラリを開発し、高解像度機能ゲノミクススクリーンを容易にするためにコンパクトでシーケンス最適化されています。最新のライブラリTKOv3は、経験データ10を使用して効率を編集するために最適化された71,090ガイドを持つ〜18,000人のヒトタンパク質コード遺伝子を対象としています。さらに、TKOv3は、単一のベクター上でCas9およびsgRNAを発現する1成分ライブラリ(LCV2:TKOv3、Addgene ID#90294)として利用可能であり、安定したCas9発現細胞を生成する必要性を緩和し、幅広いゲノムワイドノックアウトを可能にする。哺乳類細胞型。TKOv3は、Cas9(pLCKO2::TKOv3、アディジーンID#125517)を持たないベクターでも利用可能であり、Cas911を発現する細胞で利用することができる。
ゲノム全体のCRISPR-Cas9編集細胞集団は、次世代シーケンシングによって時間の経過とともに定量化されたsgRNAの豊富さと異なる増殖条件にさらされる可能性があります。摂 動。CRISPRノックアウトライブラリーは、摂動時に細胞フィットネス欠陥を引き起こし、中程度の薬物感受性(例えば、感受性または耐性遺伝子)、タンパク質発現を調節する(例えば、レポーター)、または特定の遺伝子を同定するために利用することができる。経路機能および細胞状態12、13、14。例えば、癌細胞株における差動適合性スクリーンは、腫瘍遺伝子および濃縮の枯渇または減少または腫瘍抑制遺伝子3、14、15の増加の両方を同定することができる。同様に、治療薬の中間用量を使用すると、薬剤耐性および感量遺伝子16、17の両方を明らかにすることができる。
ここでは、ライブラリー生成からデータ解析までの哺乳類細胞におけるトロントノックアウトライブラリー(TKOv1またはv3)を用いてゲノムスケールCRISPR-Cas9の機能喪失スクリーニングのための詳細なスクリーニングプロトコルを提供します。このプロトコルは、トロントノックアウトライブラリを使用してスクリーニング用に最適化されていますが、適用でき、すべてのCRISPR sgRNAプールライブラリにスケーラブルにすることができます。
プロトコル
以下に概説する実験は、研究所の環境保健安全局のガイドラインに従うべきである。
1. プールされたCRISPR sgRNAレンチウイルスライブラリープラスミド増幅
- 既製のCRISPR sgRNAプラスミドDNAライブラリーをTEで50ng/μL(例えば、TKOv3)に希釈します。
- エレクトロコンピテントセルを使用してライブラリを電気ポレートします。以下に説明するように合計4つのエレクトロポレーション反応を設定する。
- 氷上の予め冷やされたキュベット(1.0 mm)に解凍された電気有能な細胞の25 μLに50 ng/μL TKOライブラリの2 μLを追加します。
- 製造元のプロトコルによって提案される最適な設定を使用して電気ポレートします。パルスの10s以内に、975 μLの回収媒体(またはSOC培地)をキュベットに加えます。
- 電ポレートされた細胞を培養管に移し、回収培地の1mLを加える。37°Cで1時間250rpmで揺れるインキュベーターでチューブをインキュベートします。
- ライブラリを盛り上げ、変換効率を推定するために希釈プレートを設定します。
- 回収した細胞の全8mLをプールし、よく混ぜます。プールされた細胞の10 μLを回収媒体の990 μLに移し、800倍の希釈を行い、よく混合します。
- 予め温められた10 cm LB +カルベニシリン(100 μg/L)寒天板に希釈のプレート20 μL。これにより、変換効率の計算に使用される変換体の 40,000 倍の希釈が行われます。
- 各プレート上の回収細胞のプレート400 μLを、合計20個の予温められた15cm LB +カルベニシリン寒天プレートにわたってにわたって行う。30°Cで14-16時間プレートをインキュベートします。
注:この低い温度での成長は長端の反復(LTR)18間の再組み換えを最小にする。 - 変換効率を計算するには、40,000 倍の希釈プレート上のコロニーの数をカウントします (ステップ 1.3.2)。すべてのプレート上のコロニーの合計数を取得するために、40,000 でカウントされたコロニーの数を乗算します。コロニーの合計数が sgRNA あたりの最小 200x コロニーに相当するライブラリー カバレッジを表す場合に続行します (最も最適なのは 500 ~ 1000 倍)。
- 例えば、TKOv3ライブラリー(71,090 sgRNA)の最小コロニー数は1.4 x 107であり、sgRNA当たり200倍のコロニーに相当します。コロニー表現が不十分な場合は、希釈プレート上のコロニー数に基づいてステップ1.2のエレクトロポレーションの数を増やし、最小のライブラリーカバレッジを達成します。
- 以下に説明するようにコロニーを収穫
- 各15cmプレートに、LB+カルベニシリン(100μg/L)培地の7 mLを追加し、細胞拡散機でコロニーを削り取ります。10 mLピペットを使用して、スクローした細胞を無菌1L円錐フラスコまたはボトルに移します。
- もう一度LB +カルベニシリン培地の5 mLでプレートをすすいで、ボトルに溶液を転送します。
- すべてのプレートが20枚のプレートから無菌ボトルに細胞をプールするために繰り返します。
- 集めた細胞を室温(RT)で1時間かき混ぜて混ぜて細胞塊を分解する。細胞を予め計量された遠心ボトルに移し、7,000 x gの遠心分離機をペレット細菌に移し、培体を廃棄する。
- 湿った細胞ペレットを計量し、遠心分離機の重量を減算して、湿ったペレットの最終的な重量を決定します。各カラムが処理できる細菌ペレットの量に応じて、マキシまたはメガスケールのプラスミド精製キットを用いてプラスミドDNAを精製する。
2. 大規模なCRISPR sgRNAライブラリーレンチウイルス産生
注: プロトコルのこのセクションのすべてのステップは、クラス II、タイプ A2 バイオセーフティ キャビネットの BSL2+ 機能で実行されます。
- ウイルスの18 mLは、通常、1つの15センチメートルプレートから収穫される推定に基づいて、ウイルスの生産に必要な15センチメートルプレートの数を計算します。
- 低抗生物質増殖培地(DMEM+10%FBS+オプション:0.1xペン/ストレップ)でHEK-293T包装細胞を20mLの20mLの15cmプレート当たり8 x 106細胞で播種することによりトランスフェクション用の細胞を調製する。細胞を37°C、5%CO2で一晩インキュベートする。めっきされた細胞が70%~80%のコンフルエントであり、トランスフェクションの瞬間に均等に広がっていることを確認します。
- 翌日、表1に概説されているように、15cmプレート用に3つのトランスフェクションプラスミド混合物を調製する。1回のトランスフェクションに必要なプラスミドの量を計算し、プレートの数にプラスミドを混ぜて、トランスフェクションするプラスミドを作ります。
- 表2に概説されているように、トランスフェクション毎に脂質ベースのトランスフェクション試薬を調製する。アリコットは、トランスフェクトされるプレートの数のために、個々の1.5 mLマイクロ遠心管に血清培剤を減らしました。トランスフェクション試薬を追加し、穏やかに混合し、RTで5分間インキュベートします。
- 5分間のインキュベーションに続いて、トランスフェクション試薬に1回のトランスフェクションに必要なDNA量を加え、DNA複合体のトランスフェクション試薬とμgの3:1比を追加します。穏やかに混合し、RTで30分間インキュベートします。
注:その後のトランスフェクションは、時間を最適化し、オーバーインキュベーションを避けるために5分間隔で、5分以下のセットで準備することができます。 - インキュベーションの30分後、慎重に包装細胞の各プレートに各トランスフェクションミックスを転送します。セル単層を妨げることなく、円形のジグザグ運動で1 mLピペットチップを使用してミックス全体を追加します。細胞を37°Cで18時間、CO2でインキュベートする。
- ウイルス収穫媒体を調圧する:500 mLのDMEM培地+32mLのBSAストック(20g/100mL、DMEMに溶解し、0.22μmフィルターで殺菌したフィルター)+100倍ペン/ストレップの5mL。
- 18時間後、培地を取り除きます(処分前に1%次亜塩素酸ナトリウムでインキュベーションなどのレンチウイルス廃棄物を適切に取り扱います)。各プレートに18 mLのウイルス収穫培地をゆっくりと交換してください。細胞を37°Cで18時間、CO2でインキュベートする。
- 24時間後、良好なウイルス産生の指標として、異常および融合形態の包装細胞をチェックしてください。その後、すべての上清を収集し、無菌円錐遠心管に転送することにより、レンチウイルスを収穫します。
- ウイルスを含む培体を300xgで5分間回転させ、包装細胞をペレットにします。 上清をペレットを乱さずに無菌ポリプロピレンチューブに入れます。
- ウイルスを短期間(1週間未満)、または-80°Cで直ちに長期保存してください。Aliquotの大規模なウイルスは、凍結/解凍を避けるために、長期保存のための単一の使用量に準備します。
3. スクリーニング用セルラインの特性
- 目的のセルラインを選択します。
- セルのおおよその倍数を測定および記録します。
- 選択した組織培養容器(例えば、15cm組織培養プレート)で3〜4細胞倍増ごとに細胞を培養するための最適な細胞めっき密度を決定する。
- ピューロマイシン耐性マーカーを含むTKOライブラリーの選択のために所望の細胞株で使用するピューロマイシン濃度を次のように決定します。
- 72時間後に合流に達するために必要な密度で12ウェルプレート中の種子細胞を、次いで一晩インキュベートする(37°C、5%CO2)。
- 翌日、0 μg/mL から 10 μg/mL までのプロマイシン濃度の希釈範囲を含む培地に、0.5 μg/mL 単位で変更します。細胞を48時間インキュベートする。
- 48時間後、細胞計数またはアラマールブルー染色により細胞生存率を測定する。
- 48時間で細胞の100%を殺す最低濃度を決定する この濃度を使用して、ステップ4.6および5.2.6でCRISPRライブラリー変換細胞集団を選択します。
注:より長い倍増時間を持つ細胞株の場合、ピューロマイシンを使用したより長いインキュベーションを許容することができます。このような状況では、<3 細胞の倍増に必要なインキュベーション時間のキルカーブを決定します。スクリーニング開始前に必須遺伝子の脱落を避けるために、選択の時間を最小限に抑えます。
- プロマイシン感受性の測定と同じ方法で用量応答曲線を実行することにより、ブロミドヘキサジメトリン(最大8μg/mL)に対する細胞の感受性を確認してください(ステップ3.2)。ヘキサジメトリン臭化物の<8 μg/mLで毒性が認められる場合は使用しないでください。
4. MOIの決定のためのプールされたCRISPRレンチウイルスライブラリーの機能的滴定
- プールされたCRISPR sgRNAライブラリーレンチウイルス(例えば、LCV2:TKOv3)の新鮮なアリコートを解凍し、氷の上に保ちます。
- 0~ 2 mL の範囲 (0 mL、0.25 mL、0.5 mL、1 mL、および 2 mL) の範囲でテストする一連のウイルス ボリュームを設計します。
- 72時間で合流に達するために必要な密度で15cmプレートで標的細胞および種子細胞を収穫する。
- テストする各ウイルス量に対して、複製プレートを準備します。細胞、ウイルス、ヘキサジメトリン臭化物(8 μg/mL)、および培中を20mLの最終体積に加えます。プレートを十分に混合し、インキュベーターでプレートレベルに座り、24時間(37°C、5%CO2)のインキュベートを行います。
- 24時間後、培地を含むウイルスを除去し、処分する(レンチウイルス廃棄物の取り扱いにバイオセーフティの注意を使用する)。必要に応じて、温かいPBSでプレートを穏やかに洗浄し、無関係なウイルスを除去します。
- 各ウイルス状態について、セクション3で細胞を殺すと判定した濃度を用いてピューロマイシンを含む培地を20mLに置き換え、1つの複製プレートに置き換える。もう一方のプレートに、ピューロマイシンなしで20mLの新鮮な培地を加えます。48時間(37°C、5%CO2)のインキュベートを行います。
- 48時間後、ピューロマイシンで処理されたすべての未感染細胞(0 mLウイルス状態)が死んでいることを確認してください。すべてのプレートを個別に収穫し、穏やかなピペッティングを繰り返すことによって細胞を分散させます。
- すべてのプレートから細胞をカウントし、ピューロマイシンを含まない細胞数(すなわち+/-ピューロマイシン)と細胞数を比較することにより、各ウイルス体積のMOIを計算します。
- グラフの結果は、ピューロマイシンを選択した場合とピューロマイシンを選択した30%~40%の細胞生存につながるウイルス体積を決定する結果である。同じ組織培養条件下で画面中に0.3-0.4のMOIを達成するために、このウイルス量を使用してください。
5. 一次画面感染、選択、細胞通過
- 画面全体で維持する CRISPR sgRNA ライブラリ カバレッジを選択します(推奨される最小値は 200 倍)。
- ライブラリーカバレッジに基づいて、sgRNA当たりのカバレッジを維持するために必要な細胞数と、MOI 0.3(表3)で感染に必要な細胞の数を決定します。
- 感染をセットアップするために必要なプレートの数を決定します (表 4).
- CRISPR ライブラリを使用したセルへの感染
- 細胞を収穫し、各15cmプレートに必要な細胞数を播種する。
- すべてのプレートにヘキサジメトリン臭化(8 μg/mL)を加えます。
- MOI 0.3に必要な体積でウイルスをスクリーニングとコントロール2プレートに追加します。コントロール 1 の場合は、ウイルスを追加せず、そのボリュームをメディアに置き換えます。
- プレートを傾けてよく混ぜます。プレートをインキュベーターに入れ、水平であることを確認します。
注:バッチ感染は、めっき前に懸濁液中の細胞にウイルス、メディア、ヘキサジメトリン臭化物のマスターミックスを組み合わせることによって行うことができます。 - 培中を取り出し、スクリーニングにステップ3.2.4で決定した濃度でピューロマイシンを含む新鮮な培体に置き換え、ウイルス感染後24時間を制御する。コントロール2プレートにピューロマイシンを含む新鮮な媒体を追加します。48時間(37°C、5%CO2)の細胞をインキュベートする。
- ピューロマイシンを加えて48時間後、すべての未感染細胞が死んでいることを確認し(対1)、ピューロマイシン活性を確認し、感染した細胞を収穫する。
- 感染した細胞集団の収穫と細胞通過
- すべてのスクリーニングプレートからピューロマイシン選択細胞を1つの滅菌容器に収穫します。各コントロールプレートからセルを個別に収集します。穏やかな繰り返しピペッティングによって細胞を分散させる。
- プールされたスクリーニングセルから細胞をカウントし、制御1、および制御2を別々にカウントし、1 mLあたりの細胞数を計算する。
- MOI とフォールド カバレッジを次のように計算します。
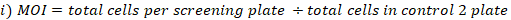
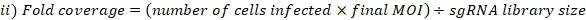
- ゲノムDNA抽出のために選択されたライブラリーカバレッジでプールされた細胞から細胞ペレットの3つの複製を収集します。細胞を500 x gで5分間遠心分離し、PBSで洗います。チューブにラベルを付け、-80 °Cで細胞ペレットを凍結乾燥します(これらはT0リファレンスサンプルです)。
- 感染した細胞のプールを3つの反復群(例えば、反復A、反復B、複製C)に分割し、各反復内のライブラリーカバレッジを維持する。シードセルは、通常、それらを展開するときに使用されるのと同じシード密度でセル。反復プレートごとに同じ数のセルを使用し、反復間のセルの合計数を同じにします。
- 細胞を通過し続け、上記のようにプール感染細胞の各複製から3回の細胞ペレットの複製を、細胞株に応じて3〜8日ごとに、最大15〜20細胞の倍増のために収穫する。各通路において、各複製群内のすべてのプレートから細胞を収穫する(すなわち、複製Aプレートからのすべての細胞が再混合され、複製Bプレートからのすべての細胞が再混合される等)。
- 各ペレットに時間(T)のラベルを付け、指定を複製します。これは、ペレットが収集された T0 後の日数 (T3_A、T3_B、T3_C など) に対応します。
- 陰性選択薬物スクリーンの場合、治療前にT0後に少なくとも1回の経過のために細胞が回復することを可能にする。T3またはT6では、ステップ5.3.5で使用されるのと同じシード密度を使用して、各反復群(A、B、C)から薬物治療および対照集団に細胞を分割する。
- 薬物治療グループ内の各反復のライブラリーカバレッジに必要なセル数を別途プールします。中間濃度で薬剤を添加する(IC20-IC50)。細胞を播種し、次の通路までインキュベート(37°C、5%CO2)を入れる。
- 車両制御グループ内の各反復のライブラリ カバレッジに必要なセルの数を個別にプールします。薬物と同じ容積を使用して車両制御を追加します(<0.5% v/v)。細胞を播種し、次の通路までインキュベート(37°C、5%CO2)を入れる。
- ステップ5.3.5に記載されているように、細胞を通過させ、ゲノムDNAの細胞ペレットを3日ごとに収穫し、各通路で薬物または車両をリフレッシュする。
- 陽性選択または薬剤耐性スクリーンの場合は、ライブラリーカバレッジに必要なセル数に応じて各反復群を分割します。各反復にIC90の薬物濃度を加える。IC90では、細胞の大半が殺されます。耐性集団が成長し、ゲノムDNA抽出のための細胞ペレット(1-2 x 107細胞)を収集できるようにします。
6. CRISPRサンプルの調製とシーケンシング
- ゲノムDNA精製
- 解凍のためにRTで5-10分間凍結細胞ペレットをインキュベートします。
- セルペレットを含む50 mL遠心管に1.4mLのPBSを追加します。細胞を再中断し、1分間休息するために20sのための渦。必要に応じて、P1000を使用してピペット15xを使用して、残りの細胞塊を分解します。15 mLまたは1.5 mLチューブから細胞を転写する場合は、1mLのPBSで細胞を再転写し、50 mLチューブに細胞を移し、400 μLのPBSで元のチューブをすすいでください。
- 再懸濁した細胞に5 mLの核溶解溶液を添加する。10 mLピペットを使用して、上下5xをピペットでサンプルを混合します。
- 核リザートに32 μLのRNase A(20mg/mL;100 μg/mL)を加え、チューブ5xを反転してサンプルを混合します。37°Cで15分間インキュベートし、サンプルがRTで10分間冷却できるようにします。
- 1.67 mLのタンパク質沈殿液を溶解液に加え、20秒間激しく渦を加えると、混合後に小さなタンパク質塊が見える場合があります。
- RTで10分間4,500 x gの遠心分離機。
- 10 mLピペットを使用して、上清を5mLのイソプロパノールを含む50 mL遠心管に移します。DNAが観察されるまで、溶液10xを反転して穏やかに混合する。
注:DNAは、目に見える塊を形成する白い糸状の鎖として観察することができます。 - DNAをペレットするためにRTで5分間4,500 x gで遠心分離機。
- 10 mLピペットを使用して、上清を慎重に取り除き、DNAペレットを外さないようにしてください。RTで70%エタノールの5 mLをDNAに加えます。チューブをゆっくりと回転させて、遠心管のDNAペレットと側面を洗浄します。
- RTで5分間4,500 x gの遠心分離機。
- 10 mLピペットを使用して、70%のエタノールを慎重に除去し、DNAペレットを外さないようにしてください。RTで10分間の空気乾燥ゲノムDNA。
- チューブに400μLのTE溶液を加え、1時間ごとに65°CでインキュベートしてDNAを溶かします。DNAが完全に溶解しない場合は、チューブを15分ごとに穏やかにフリックしながら、さらに1時間65°Cでチューブをインキュベートし、一晩4°Cのままにします。
- RTで1分間4,500 x gの遠心分離機を用いて、ゲノムDNAを1.5mLの低結合チューブに移す。
- 分光光度計(全核酸含有量)と蛍光計(二本鎖DNA含有量の場合)の両方でゲノムDNAの純度を定量し、測定します。
- 必要に応じて、sgRNAの下流PCR増幅に問題がある場合は、以下のようにゲノムDNAを沈殿させる。
- 400 μL ゲノムDNAを1.5mLマイクロ遠心管に移す。
- 5 M NaCl の 18 μL (最終濃度 0.2 M) と 95% エタノールの 900 μL を追加します。
- チューブを完全に混合するまで10xを反転し、RTで10分間16,000 x gで遠心分離します。
- 上清を慎重に取り除き、DNAペレットを外さないようにしてください。70%エタノールの500 μLでDNAペレットを洗浄します。チューブをゆっくりと回転させてDNAペレットを洗浄します。
- RTで5分間16,000 x gの遠心分離機。
- 慎重に上清を取り除き、DNAペレットを外さないようにしてください。RTで10分間の空気乾燥ゲノムDNA。
- 手順6.1.12で説明されているようにDNAを溶解するためにTEの300 μLを追加します。
- ステップ6.1.14に記載されているゲノムDNAの純度を定量し、測定します。
- CRISPR シーケンス ライブラリの準備
- 表5に概説されているように、合計100μgのゲノムDNAを用いてPCR1をセットアップする。50 μL反応あたり3.5 μgのゲノムDNAを追加し、同一の50 μL反応を設定して、所望のカバレッジを達成します。表 6に、LCV2::TKOv3 シーケンス ライブラリの増幅のためのプライマー シーケンスの例を示します。表 7に、pLCKO2::TKOv3 シーケンスライブラリの増幅のためのプライマーシーケンスの例を示します。
- 表8に概説するプログラムを使用して、サーモサイクラー中のPCR 1反応を増幅する。
- 1%のアガロースゲルでPCR製品の2μLを実行してPCR1増幅を確認してください。PCR 1 は 600 bp の積を生成します。
- 各ゲノムDNAサンプルに対するすべての個々の50 μL反応をプールし、渦によって混合する。
- プールされた PCR 1 製品の 5 μL をテンプレートとして使用して、表 9に概説されているように、各サンプルに対して 1 つの PCR 2 反応 (50 μL) を設定します。個々のサンプルごとに固有のインデックス プライマーの組み合わせを使用して、シーケンス ライブラリ サンプルのプールを許可します。
- 表10に概説するプログラムを用いて、サーモサイクラー中のPCR2反応を増幅する。
- ゲルを鋳造する前に0.1 N HClで増幅された製品を10分間精製するためのクリーンアガロースゲル装置。PCR2増幅産物を精製するためのDNA染色を含む2%アガロースゲルを調剤する。
- PCR 2製品を低電圧(1.0~1.5h実行)で2%アガロースゲルで実行します。PCR 2 は 200 bp の製品を生成します。
- 青色光トランジユミレータでPCR製品を可視化します。200 bpバンドを切除し、ゲル抽出キットを使用してアガロースゲルスライスからDNAを精製します。分光光度計と蛍光計の両方でシーケンシングライブラリの純度を定量し、測定します。
注:典型的なゲル精製シーケンシングライブラリ濃度は、5〜10 ng/μLおよび150〜300 ngの総収率の範囲である。
- ハイスループットシーケンス
- 次世代シーケンサーで CRISPR シーケンス ライブラリをシーケンスします。
- 400~ 500 倍のライブラリ カバレッジの読み取り深度が高いシーケンスリファレンス T0 サンプル。ドロップアウト画面のシーケンス実験タイムポイントサンプルを、読み取り深度を 200 倍に抑えます。強い肯定的な選択のスクリーンのために、50倍のカバーの最低の読書の深さは濃縮されたsgRNAの識別のために十分である。
注: T0 サンプルをシーケンスして特定の画面のライブラリー表現を決定し、時間の経過に合った sgRNA フォールドの変化を決定するためのリファレンスとして機能することが重要です。
7. データ分析
- Bowtie などのプログラムを使用してシーケンスを並べ合わせて、次のパラメーターを使用して参照ライブラリにシーケンスの読み取りをマップします。
- 特定のサンプルの sgRNA ごとに一意にマップされた読み取りの数を、サンプルごとに 1,000 万読み取りとして正規化します。
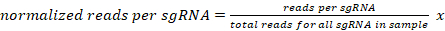 107
107
- T0 サンプル (Tn/T0) と比較して、各反復時 (Tn) ごとに各 sgRNA の log2 倍の変化を計算します。ゼロからの不連続性を防ぐために、すべての読み取りカウントに 0.5 読み取りの疑似カウントを追加します。折りたたみ変更計算およびダウンストリーム解析から、T0 サンプルの <30 未処理読み取りを含む sgRNA を除外します。
- 遺伝子本質のベイズ分析(BAGEL)アルゴリズムと共に折り畳みの変化を分析、遺伝子必須スクリーンのために以前に定義されたコア必須および非必須トレーニングセットを使用して(補足テーブル S1) または DrugZ 薬物スクリーン用 20.
- BF スコアを使用して、画面パフォーマンス評価の精度とリコールを計算します。上記の BF スコア サブセットと共に、Python の Scikit 学習ライブラリの precision_recall_curve 関数の真の正のリストとして、ステップ 7.4 の必須セットを使用します。または、R の PRROC パッケージを使用して同じ操作を実行します。
- 各遺伝子のすべてのガイドの平均折りたたみ変化を計算します。Rまたは同等のソフトウェアで、必須遺伝子および非必須遺伝子の密度プロットを生成します(ステップ7.4参照)。R では、x.ess が必須遺伝子のログフォールド変更値を含むベクターであり、x.nonEss に非必須遺伝子が含まれている場合は、次のコマンドを使用してプロットします。
プロット( 密度( x.ess) 、 xlab="平均 logFC"、col="赤"、lwd=2 )
線 ( 密度 ( x.nonEss ), col="青",lwd=2 )
注:使用されるPythonバージョンの詳細とパッケージについては、scikit-learn v0.19.1:(Pedregosa et al.21によって公開)を参照してください。
結果
ゲノムスケールCRISPRスクリーニングワークフローの概要
図1は、単一の統合イベントと適切なライブラリ表現(通常200~1000)を確保するために、低MOIでCRISPRライブラリーレンチウイルスを持つ標的細胞の感染から始まる、プールされたCRISPRスクリーニング作業フローの概要を示しています。-...
ディスカッション
使用の簡易性および高い準備性のために、CRISPRの技術は精密なゲノム編集のための選択の用具として広く採用されている。プールされたCRISPRスクリーニングは、単一の実験で何千もの遺伝的摂動を尋問する方法を提供する。プールされたスクリーンでは、sgRNAライブラリは分子バーコードとして機能し、各配列は一意であり、標的遺伝子にマッピングされます。細胞集団からゲノムDNAを分離?...
開示事項
著者は、競合する金銭的利益を宣言しません。
謝辞
この研究は、ゲノムカナダ、オンタリオ州研究基金、およびカナダ保健研究所(MOP-142375、PJT-148802)によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 0.22 micron filter | |||
| 30°C plate incubator | |||
| 37°C shaking incubator | |||
| 37°C, 5% CO2 incubator | |||
| 5 M NaCl | Promega | V4221 | |
| 50X TAE buffer | BioShop | TAE222.4 | |
| 6 N Hydrochloric acid solution | BioShop | HCL666.500 | |
| 95% Ethanol | |||
| Alamar blue | ThermoFisher Scientific | DAL1025 | |
| Blue-light transilluminator | ThermoFisher Scientific | G6600 | |
| Bovine Serum Albumin,Heat Shock Isolation, Fraction V. Min. 98%, Biotechnology grade | Bioshop | ALB001.250 | |
| Dulbecco's Modification of Eagles Medium | Life Technologies | 11995-065 | Cel culture media |
| Electroporation cuvettes | BTX | 45-0134 | |
| Electroporator | BTX | 45-0651 | |
| Endura electrocompetent cells | Lucigen | 90293 | |
| Fetal Bovine Serum | GIBCO | 12483-020 | |
| HEK293T packaging cells | ATCC | CRL-3216 | recommend passage number <15 |
| Hexadimethrine Bromide (Polybrene) | Sigma | H9268 | Cationic polymer to enhance transduction efficiency |
| Hexadimethrine Bromide (Polybrene) | |||
| LB agar plates with carbenicillin | |||
| LB medium with carbenicillin | |||
| Low molecular weight DNA ladder | New England Biolabs | N3233S | |
| Nanodrop spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| NEBNext Ultra II Q5 Master Mix | New England Biolabs | M0544L | |
| Opti-MEM | Life Technologies | 31985-070 | Reduced serum media |
| Plasmid maxi purification kit | Qiagen | 12963 | |
| pMD2.G (envelope plasmid) | Addgene | Plasmid #12259 | lentiviral system |
| psPAX2 (packaging plasmid) | Addgene | Plasmid #12260 | lentiviral system |
| Puromycin | Wisent | 400-160-UG | |
| QIAquick gel extraction kit | Qiagen | 28704 | |
| Qubit dsDNA BR assay | ThermoFisher Scientific | Q32853 | |
| Qubit fluorometer | ThermoFisher Scientific | Q33226 | |
| RNAse A | Invitrogen | 12091021 | |
| S.O.C recovery medium | Invitrogen | 15544034 | |
| SYRB Safe DNA gel stain | ThermoFisher Scientific | S33102 | |
| Toronto KnockOut CRIPSR library (TKOv3) - Cas9 included | Addgene | Addgene ID #90203 | Genome-wide CRISPR library , includes Cas9, 71,090 sgRNA |
| Toronto KnockOut CRIPSR library (TKOv3) - non-cas9 | Addgene | Addgene ID #125517 | Genome-wide CRISPR library, non-Cas9, 71,090 sgRNA |
| Tris-EDTA (TE) solution, pH8.0 | |||
| UltraPure agarose | ThermoFisher Scientific | 16500500 | |
| Wizard genomic DNA purification kit | Promega | A1120 | |
| X-tremeGENE 9 DNA transfection reagent | Roche | 06 365 809 001 | Lipid based transfection reagent |
参考文献
- Jiang, F., Doudna, J. A. CRISPR-Cas9 Structures and Mechanisms. Annual Review of Biophysics. 46, 505-529 (2017).
- Baliou, S., et al. CRISPR therapeutic tools for complex genetic disorders and cancer (Review). International Journal of Oncology. 53 (2), 443-468 (2018).
- Hart, T., et al. High-Resolution CRISPR Screens Reveal Fitness Genes and Genotype-Specific Cancer Liabilities. Cell. 163 (6), 1515-1526 (2015).
- Morgens, D. W., Deans, R. M., Li, A., Bassik, M. C. Systematic comparison of CRISPR/Cas9 and RNAi screens for essential genes. Nature Biotechnology. 34 (6), 634-636 (2016).
- Evers, B., et al. CRISPR knock-out screening outperforms shRNA and CRISPRi in identifying essential genes. Nature Biotechnology. 34 (6), 631-633 (2016).
- Miles, L. A., Garippa, R. J., Poirier, J. T. Design, execution, and analysis of pooled in vitro CRISPR/Cas9 screens. The FEBS Journal. 283 (17), 3170-3180 (2016).
- Wang, T., Wei, J. J., Sabatini, D. M., Lander, E. S. Genetic screens in human cells using the CRISPR-Cas9 system. Science. 343 (6166), 80-84 (2014).
- Wang, T., et al. Identification and characterization of essential genes in the human genome. Science. 350 (6264), 1096-1101 (2015).
- Sanson, K. R., et al. Optimized libraries for CRISPR-Cas9 genetic screens with multiple modalities. Nature Communications. 9 (1), 5416 (2018).
- Hart, T., et al. Evaluation and Design of Genome-Wide CRISPR/SpCas9 Knock-out Screens. G3: Genes|Genomes|Genetics. 7 (8), 2719-2727 (2017).
- Mair, B., Tomic, J., et al. Essential gene profiles for human pluripotent stem cells identify uncharacterized genes and substrate dependencies. Cell Reports. 27 (2), 599-615 (2019).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knock-out screening in human cells. Science. 343 (6166), 84-87 (2014).
- Sharma, S., Petsalaki, E. Application of CRISPR-Cas9 Based Genome-Wide Screening Approaches to Study Cellular Signalling Mechanisms. International Journal of Molecular Sciences. 19 (4), (2018).
- Steinhart, Z., et al. Genome-wide CRISPR screens reveal a Wnt-FZD5 signaling circuit as a druggable vulnerability of RNF43-mutant pancreatic tumors. Nature Medicine. 23 (1), 60-68 (2017).
- Wang, T., et al. Gene Essentiality Profiling Reveals Gene Networks and Synthetic Lethal Interactions with Oncogenic Ras. Cell. 168 (5), 890-903 (2017).
- Zimmermann, M., et al. CRISPR screens identify genomic ribonucleotides as a source of PARP-trapping lesions. Nature. 559 (7713), 285-289 (2018).
- Deans, R. M., et al. Parallel shRNA and CRISPR-Cas9 screens enable antiviral drug target identification. Nature Chemical Biology. 12 (5), 361-366 (2016).
- Trinh, T. J. J., Bloom, F., Hirsch, V. STBL2: an Escherichia coli strain for the stable propagation of retroviral clones and direct repeat sequences. Focus. 16, 78-80 (1994).
- Hart, T., Moffat, J. BAGEL: a computational framework for identifying essential genes from pooled library screens. BMC Bioinformatics. 17, 164 (2016).
- Wang, G. Z. M., et al. Identifying drug-gene interactions from CRISPR knock-out screens with drugZ. bioRxiv. , (2017).
- Pedregosa, F. V., G, , et al. Scikit-learn: Machine Learning in Python. Journal of Machine Learning Research. 12, 2825-2830 (2011).
- Ketela, T., et al. A comprehensive platform for highly multiplexed mammalian functional genetic screens. BMC Genomics. 12, 213 (2011).
- Doench, J. G. Am I ready for CRISPR? A user's guide to genetic screens. Nature Review Genetics. 19 (2), 67-80 (2018).
- Hartenian, E., Doench, J. G. Genetic screens and functional genomics using CRISPR/Cas9 technology. FEBS Journal. 282 (8), 1383-1393 (2015).
- Li, W., et al. MAGeCK enables robust identification of essential genes from genome-scale CRISPR/Cas9 knock-out screens. Genome Biology. 15 (12), 554 (2014).
- Sheel, A., Xue, W. Genomic Amplifications Cause False Positives in CRISPR Screens. Cancer Discovery. 6 (8), 824-826 (2016).
- Meyers, R. M., et al. Computational correction of copy number effect improves specificity of CRISPR-Cas9 essentiality screens in cancer cells. Nature Genetics. 49 (12), 1779-1784 (2017).
- Henser-Brownhill, T., Monserrat, J., Scaffidi, P. Generation of an arrayed CRISPR-Cas9 library targeting epigenetic regulators: from high-content screens to in vivo assays. Epigenetics. 12 (12), 1065-1075 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved