Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Una adaptación de semi-alto rendimiento del ensayo ATPase acoplado a NADH para la detección de inhibidores de moléculas pequeñas
En este artículo
Resumen
Un ensayo ATPase acoplado a la nicotinamida adenina dinucleótido (NADH) se ha adaptado al cribado de rendimiento semialto de inhibidores de la miosina de moléculas pequeñas. Este ensayo cinético se ejecuta en un formato de microplaca de 384 pocillos con volúmenes de reacción totales de sólo 20 s por poca. La plataforma debe ser aplicable a prácticamente cualquier enzima productora de ADP.
Resumen
Las enzimas ATPase utilizan la energía libre almacenada en el trifosfato de adenosina para catalizar una amplia variedad de procesos bioquímicos endergónicos in vivo que no ocurrirían espontáneamente. Estas proteínas son cruciales para esencialmente todos los aspectos de la vida celular, incluyendo el metabolismo, división celular, respuestas a los cambios ambientales y movimiento. El protocolo presentado aquí describe un ensayo ATPase acoplado a la nicotinamida adenina dinucleótido (NADH) que se ha adaptado a la detección de rendimiento semi-alto de inhibidores ATPase de moléculas pequeñas. El ensayo se ha aplicado a la miosina cardiaca y esquelética de miosina II, dos ATPases de motor molecular a base de actina, como prueba de principio. La hidrólisis de ATP se acopla a la oxidación de NADH por reacciones enzimáticas en el ensayo. En primer lugar, el ADP generado por el ATPase se regenera a ATP por piruvato quinasa (PK). PK cataliza la transición de fosfoenolpiruvato (PEP) al piruvato en paralelo. Posteriormente, el piruvato se reduce a lactato por lactato deshidrogenasa (LDH), que cataliza la oxidación de NADH en paralelo. Por lo tanto, la disminución de la concentración de ATP está directamente correlacionada con la disminución de la concentración de NADH, que es seguida por el cambio a la fluorescencia intrínseca de NADH. Mientras la PEP esté disponible en el sistema de reacción, la concentración de ADP sigue siendo muy baja, evitando la inhibición de la enzima ATPase por su propio producto. Además, la concentración de ATP se mantiene casi constante, produciendo cursos de tiempo lineales. La fluorescencia se monitorea continuamente, lo que permite una fácil estimación de la calidad de los datos y ayuda a filtrar los artefactos potenciales (por ejemplo, derivados de precipitaciones compuestas o cambios térmicos).
Introducción
Las miosinas son transductores de energía mecanoquímicos que hidrolizan trifosfato de adenosina (ATP) para generar movimientodireccional a lo largo de los filamentos del citoesqueleto de actina en eucariotas 1,2. Se han adaptado tanto estructural como cinéticamente a sus diversas funciones intracelulares, como el transporte deorgánulos, la contracción muscular o la generación de tensión citoesquelética 1,2. La superfamilia de miosina está representada por genes de miosina de 40 euros pertenecientes a 12 clases distintas de miosina en el genoma humano3,4. Los miembros de las clases de miosina desempeñan diversos papeles en un conjunto muy diverso de trastornos, como varios tipos de cáncer, trastornos neurológicos, miopatías esqueléticas y cardiomiopatía hipertrófica5,6. Dado el gran número de funciones fisiológicas y patológicas de estos motores moleculares, no es de extrañar que se estén volviendo cada vez más reconocidos como objetivos farmacológicos para una variedad de condiciones7. Recientemente se han realizado progresos significativos en el descubrimiento de nuevos inhibidores de la miosina8,9,10 y activadores11,y para mejorar las propiedades de los existentes12, 13 , 14 , 15.
El ensayo ATPase acoplado a la nicotinamida adenina dinucleótido (NADH) se ha utilizado durante mucho tiempo para medir la actividad ATPase de varias enzimas, como la bomba sarcoplasmática reticulum Ca2+ ATPase16, la reparación de ADN ATPase Rad5417, la AAA+ ATPase p9718 o la quinesina motora microtúbula19. El ensayo emplea un ciclo de regeneración ATP. El difosfato de adenosina (ADP) generado por la ATPase se regenera a ATP por piruvato quinasa (PK), que transforma una molécula de fosfoenolpiruvato (PEP) a piruvato en paralelo. Posteriormente, el piruvato se reduce a lactato por lactato deshidrogenasa (LDH). Eso, a su vez, oxida una molécula de NADH a NAD. Por lo tanto, la disminución en la concentración de NADH en función del tiempo es igual a la tasa de hidrólisis ATP. El ciclo de regeneración de ATP mantiene la concentración de ATP casi constante y la concentración de ADP baja siempre y cuando PEP esté disponible. Esto da como resultado cursos de tiempo lineales, por lo que es fácil determinar las tasas de reacción inicial y ayuda a evitar la inhibición del producto por ADP19. Aunque el ensayo ATPase acoplado a NADH ya se ha adaptado a un formato de 96 pozos20,los altos volúmenes de reacción (-150 l) lo hacen relativamente caro debido a la alta demanda de reactivos, lo que lo hace menos apto para un cribado rápido de grandes Compuestos. Los métodos alternativos, como el ensayo verde de malaquita19,21, que se basa en la detección del fosfato producido por la enzima ATPase, se demostraron más adecuados para la miniaturización y el cribado de alto rendimiento22 , 23 , 24. Sin embargo, es más probable que un ensayo de punto final se vea afectado por varios artefactos (que se describen a continuación), que pueden permanecer sin descubrir se descubierta en ausencia de cursos de tiempo completo.
Aquí, el ensayo ATPase acoplado a NADH se ha optimizado para la detección de rendimiento semi-alto de inhibidores de moléculas pequeñas. La miosina muscular esquelética y cardíaca II y los inhibidores de la miosina blebbistatin8, para-aminoblebbistatin13 y para-nitroblebbistatin12 se utilizan para demostrar el poder del ensayo, que se basa en NADH fluorescencia como una lectura. Este protocolo es susceptible de cribado de proyectos centrados en cualquier enzima que produzca ADP.
Protocolo
1. Preparación de soluciones de stock y reactivos
- Preparar la solución de material de ditilothreitol (TDT) disolviendo la TDT cristalina en agua destilada a una concentración final de 1000 mM. Ajuste el pH a 7.0 con 1 M de solución NaOH. Aliquot y almacenar a -20oC.
- Prepare la solución de stock ATP disolviendo ATP cristalino en agua destilada a una concentración final de 100 mM. Ajuste el pH a 7.0 con 1 M de solución NaOH. Aliquot y almacenar a -20oC.
- Preparar 10x nadh tampón que contenga 70 mM 3-(N-morpholino)ácido propanesulfónico (MOPS), 10 mM MgCl2, 0,9 mM etilenglicol-bis (éter de aminoetilo)-N,N,N,N,N-tetraacético ácido (EGTA), y 3 mM NaN3. Ajuste el pH a 7.0 con 1 M de solución NaOH. Conservar a 4oC.
- Prepare 1 búfer de miosina que contenga 10 mM MOPS y 0,1 mM EGTA. Ajuste el pH a 7.0 con 1 M de solución NaOH. Conservar a 4oC. Añadir albúmina sérica bovina (BSA) y TDT a una concentración final de 0,1% (p/v%) y 1 mM, respectivamente, antes de su uso.
- Prepare 1 tampón de actina que contenga 4 mM MOPS, 0,1 mM EGTA, 2 mM MgCl2y 3 mM NaN3. Ajuste el pH a 7.0 con 1 M de solución NaOH. Conservar a 4oC. Añadir BSA y TDT a una concentración final de 0,1% (p/v%) y 1 mM, respectivamente, antes de su uso.
- Prepare la solución de stock NADH disolviendo NADH cristalino en un búfer NADH de 10x a una concentración final de 5,5 mM. Aliquot y almacenar a -20oC.
- Prepare la solución de stock PEP disolviendo PEP cristalino en búfer NADH 10x a una concentración final de 50 mM. Aliquot y almacenar a -20oC.
- Preparar la solución de material LDH disolviendo polvo LDH liofilizado en una mezcla de glicerol y tampón NADH 10x (50%:50%) a una concentración final de 2000 U/mL. Centrifugar la solución para eliminar cualquier proteína no disuelta presente (7.197 x g, 20 oC, 10 min). Transfiera el sobrenadante a un tubo de centrífuga limpio con cuidado. Aliquot y almacenar a -20oC.
- Preparar la solución de stock PK mediante la disolución de polvo PK liofilizado en una mezcla de glicerol y 10x buffer NADH (50%:50%) a una concentración final de 10000 U/ml. Centrifugar la solución para eliminar cualquier proteína no disuelta presente (7.197 x g, 20 oC, 10 min). Transfiera el sobrenadante a un tubo de centrífuga limpio con cuidado. Aliquot y almacenar a -20oC.
- Reconstituir las muestras de miosina ii cardiaca y esquelética liofilizada añadiendo 100 l de agua destilada para obtener soluciones de stock de 10 mg/ml correspondientes a concentraciones de miosina (monoméricas) de 37,9 m y 40,8 m (monoméricas), respectivamente. Para obtener más información, consulte las instrucciones del fabricante.
- Preparar F-actin a partir de polvo de acetona muscular conejo como se describe por Pardee y Spudich25.
2. Medición de las actividades de ATPase y los efectos inhibitorios de los inhibidores de moléculas pequeñas
- Prepare la placa compuesta.
- Disolver compuestos de interés en dimetilsulfóxido de alta calidad (DMSO).
- Cree diluciones seriales 1:2 de quince pasos a partir de una concentración compuesta de 10 mM en DMSO.
- Transfiera las muestras a una placa de polipropileno de 384 pocillos en triplicados (12,5 l cada una) utilizando una pipeta multicanal. Utilice dos filas en la placa compuesta para un compuesto (en lugar de tres columnas) para minimizar el número de pozos potencialmente afectados por los efectos de borde. Utilice los últimos tres pozos de la segunda fila para cada compuesto como control negativo (solo DMSO). No utilice la primera y la última fila de la placa para diluciones compuestas.
- Transfiera DMSO puro a los pozos de la primera fila (reservado para la calibración NADH).
- Utilice la última fila para el control positivo.
NOTA: Para-aminoblebbistatin a 4 mM de concentración en DMSO se utilizó aquí.
- Preparar 4500 ml de solución de actina diluida de 20 m para cada placa de ensayo (microplaca de poliestireno de pared negra de 384 pocillos) diluyendo la solución de material de actina en tampón de actina. Mezcle bien la solución pipeteando 30 veces arriba y abajo utilizando una pipeta de 5 ml para reducir la viscosidad y la heterogeneidad rompiendo filamentos de actina. Centrifugar la solución para eliminar cualquier proteína precipitada presente (7.197 x g, 20oC, 10 min). Transfiera cuidadosamente el sobrenadante a un tubo de centrífuga limpio.
- Preparar la mezcla maestra que contiene enzimas LDH y PK ("mezcla de enzimas"). Para cada placa de ensayo, combine 171,4 l de solución de LDH, 171,4 l de solución PK y 3189,3 l o 3252,9 l de tampón de miosina para ensayos que involucren miosina muscular cardiaca o esquelética II, respectivamente, en un tubo centrífugo cónico de 15 ml. No agregue miosina en este punto para evitar la agregación y la precipitación.
- Preparar la mezcla maestra que contiene todos los sustratos ("mezcla de sustratos"). Para cada placa, combine 162,1 l de ATP, 162,1 ml de PEP y 324,1 l de solución de NADH en un tubo centrífugo cónico de 15 ml. No agregue actina en este punto para evitar la agregación y la precipitación.
- Cree diluciones seriales 1:2 de siete pasos de NADH para calibración a partir de 250 m.
- Mezclar 12,3 l de solución de nadh con 257,7 l de tampón de miosina en un tubo de microcentrífuga de 1,5 ml.
- Alícuota 135 l de tampón de miosina en siete tubos de microcentrífuga de 1,5 ml.
- Transfiera 135 ml de solución desde el primer tubo al segundo y mezcle mediante pipeteo. Repita el proceso hasta llegar altubo 7.
- Utilice el último tubo como control no-NADH (solo búfer).
- Mediante una pipeta de 8 canales, transfiera 20 ml de las soluciones de calibración NADH a la primera fila de la placa de ensayo en triplicados.
- Añadir 68 ml de cardiaco o 4,2 ml de miosina muscular esquelética II a la mezcla enzimática. Vórtice brevemente.
- Excepto la primera fila, dispensar 8,4 ml de la mezcla de miosina-enzima preparada en cada pocificador de la placa de ensayo utilizando un dispensador automatizado.
- Transfiera 100 nL de soluciones de la placa compuesta a la placa de ensayo que contiene la mezcla enzimática utilizando un sistema automatizado de manipulación de líquidos equipado con un cabezal de herramienta de pasador de 100 nL.
- Agitar la placa de ensayo durante 1 min a temperatura ambiente a 1200 rpm utilizando una coctelera de microplaca.
- Añadir 4.052 ml de la solución de actina centrifugada a la mezcla de sustrato. Vórtice brevemente.
- Dispensar 11,6 ml de mezcla de actina-sustrato en cada pocelo de la placa de ensayo (excepto primera fila) para iniciar la reacción enzimática utilizando un dispensador automatizado.
- Agitar la placa de ensayo durante 1 min a temperatura ambiente a 1200 rpm utilizando una coctelera de microplaca.
- Centrifugar la placa de ensayo a 101 x g durante 30 s.
- Asegúrese de que la temperatura interior del lector de placas se ha estabilizado a 25 oC. Cargue el plato y agite durante otros 30 s. Este paso de agitación es necesario para que la forma de la superficie líquida sea similar en cada pocil y permite tiempo para que la placa alcance la temperatura de medición.
- Registre la fluorescencia NADH durante 30 minutos escaneando la placa en intervalos de 45 s. Utilice un filtro de excitación de ancho de banda de 380 nm y 10 nm y un filtro de emisión de ancho de banda de 470 nm y 24 nm junto con un espejo dicroico de corte de 425 nm. Ejecute la medición en modo de alta concentración. Optimice el número de destellos, la ganancia del detector, las dimensiones de la placa y la altura de medición antes de ejecutar los ensayos.
NOTA: Las condiciones finales del ensayo son 300 nM cardiaca/20 nM de miosina muscular esquelética II, 10 M de actina, 40 U/ml LDH, 200 U/mL PK, 220 M NADH, 1 mM de PEP, 1 mM de ATP en un tampón que contiene 10 mM MOPS (pH a 7,0), 2 mM MCl2 , 0,15 mM EGTA, 0,1 mg/mL BSA, 0,5% (v/v) DMSO y 1 mM de TDT. El volumen total es de 20 l/bueno. La concentración de compuesto final más alta es de 50 M. 20 m para-aminoblebbistatin en 0.5% DMSO sirve como el control positivo y 0.5% DMSO solo es el control negativo. Todas las mediciones se realizan en triplicados.
3. Análisis de datos
- Trazar la intensidad de fluorescencia observada contra el tiempo para cada pocal.
- Realice una regresión lineal simple para determinar la pendiente y la interceptación de las respuestas de fluorescencia para cada poca. La pendiente es proporcional a la tasa de consumo de ATP (NADH), mientras que la intercepción es proporcional a la concentración de NADH al principio de la medición (t a 0 s).
- Construir una curva de calibración para NADH trazando las interceptaciones obtenidas para la primera fila de la placa contra la concentración de NADH. Asegúrese de que las interceptaciones dependen linealmente de la concentración de NADH.
NOTA: Las interceptaciones estiman las intensidades reales de fluorescencia en t a 0 s con mucha más confianza que la media de la intensidad de fluorescencia bruta que se lee en t a 0 s. - Realice una regresión lineal simple para obtener la pendiente y la interceptación de la línea de calibración NADH.
NOTA: La interceptación describe la señal de fondo de fluorescencia (sin NADH presente), mientras que la pendiente corresponde a la intensidad de fluorescencia extrapolada/teórica de una solución NADH de 1 M en ese experimento en particular. - Divida la pendiente de la respuesta de fluorescencia obtenida para el resto de los pozos por la pendiente de la línea de calibración NADH para convertir los cambios de fluorescencia en tasas de consumo de ATP.
- Trazar las tasas de consumo de ATP contra la concentración del inhibidor.
- Para determinar las constantes inhibitorias, utilice el software estadístico adecuado para ajustar los datos de dosis-respuesta a la siguiente ecuación cuadrática correspondiente a un modelo de equilibrio vinculante simple uno a uno:
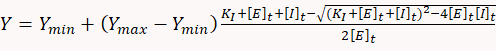
donde Y es la tasa de consumo de ATP, Ymin es la tasa de consumo de ATP int la ausencia de inhibidor, Ymax es la tasa de consumo de ATP teórica en 100% inhibición, KI es la constante inhibitoria , [E]t y [I]t son la concentración total de la enzima (miosina) e inhibidor, respectivamente.
Resultados
El mapa de diseño de placa típico utilizado para los experimentos de cribado se muestra en la Figura1. La primera y la última fila están reservadas para la calibración de NADH y el control positivo (20 m para-aminoblebbistatin, 0,5% DMSO), respectivamente. Las filas restantes (B a O) se utilizan para probar la actividad inhibitoria de los compuestos. Aquí, las diluciones seriales 1:2 de quince pasos a partir de una concentración compuesta de 1...
Discusión
Pasos críticos en el protocolo
Optimice el diseño de la placa ejecutando varias placas con control negativo solamente (reacción ATPase sin inhibidor). Inspeccione los resultados cuidadosamente en busca de patrones en las tasas de reacción. Por ejemplo, estos pueden surgir de efectos de borde y / o imperfecciones en el recubrimiento de superficie hidrófila de placas "no vinculantes". Si se observa un patrón, cambie el tipo de placa y/o el diseño de placa para minimizar l...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por una subvención del Instituto Nacional de Trastornos Neurológicos y Accidentes Cerebrovasculares y el Instituto Nacional sobre Abuso de Drogas NS096833 (CAM).
Materiales
| Name | Company | Catalog Number | Comments |
| 384-well Low Flange Black Flat Bottom Polystyrene NBS Microplate | Corning | 3575 | |
| ATP (Adenosine 5′-triphosphate disodium salt hydrate) | Sigma | A7699 | |
| Aurora FRD-IB Dispenser | Aurora Discovery, Inc. | 00017425 | |
| Biomek NXP Multichannel Laboratory Automation Workstation | Beckman Coulter | A31841 | |
| Blebbistatin | AMRI | N/A | Custom synthesis |
| BSA (Bovine Serum Albumin, Protease-Free) | Akron Biotech | AK1391 | |
| Centrifuge 5430 R, refrigerated, with Rotor FA-35-6-30 | Eppendorf | 022620663 | |
| Centrifuge 5430, non-refrigerated, with Rotor A-2-MTP | Eppendorf | 022620568 | |
| DMSO (Dimethyl sulfoxide) | Sigma | D2650 | |
| DTT (DL-Dithiothreitol) | Sigma | D5545 | |
| E1 ClipTip Multichannel Pipette; 384-format; 8-channel | Thermo Scientific | 4672010 | |
| E1 ClipTip Multichannel Pipette; 96-format; 8-channel | Thermo Scientific | 4672080 | |
| EGTA (Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid) | Sigma | E3889 | |
| EnVision 2104 Multilabel Plate Reader | PerkinElmer | 2104-0010 | |
| Glycerol | Sigma | G2025 | |
| LDH (L-Lactic Dehydrogenase from rabbit muscle) | Sigma | L1254 | |
| MgCl2.6H2O (Magnesium chloride hexahydrate) | Sigma | M2670 | |
| Microplate Shaker | VWR | 12620-926 | |
| Microplate, 384 well, PP, Small Volume, Deep Well, Natural | Greiner Bio-One | 784201 | |
| MOPS (3-(N-Morpholino)propanesulfonic acid) | Sigma | M1254 | |
| Myosin Motor Protein (full length) (Bovine cardiac muscle) | Cytoskeleton | MY03 | |
| Myosin Motor Protein (full length) (Rabbit skeletal muscle) | Cytoskeleton | MY02 | |
| NADH (β-Nicotinamide adenine dinucleotide, reduced disodium salt hydrate) | Sigma | N8129 | |
| NaN3 (Sodium azide) | Sigma | 71289 | |
| NaOH (Sodium hydroxide) | Sigma | S8045 | |
| Optical Filter CFP 470/24nm (Emission) | PerkinElmer | 2100-5850 | Barcode 240 |
| Optical Filter Fura2 380/10nm (Excitation) | PerkinElmer | 2100-5390 | Barcode 112 |
| Optical Module: Beta Lactamase | PerkinElmer | 2100-4270 | Barcode 418 |
| OriginPro 2017 software | OriginLab | N/A | |
| para-Aminoblebbistatin | AMRI | N/A | Custom synthesis |
| para-Nitroblebbistatin | AMRI | N/A | Custom synthesis |
| PEP (Phospho(enol)pyruvic acid monopotassium salt) | Sigma | P7127 | |
| PK (Pyruvate Kinase from rabbit muscle) | Sigma | P9136 | |
| Rabbit Muscle Acetone Powder | Pel Freez Biologicals | 41995-2 |
Referencias
- Heissler, S. M., Sellers, J. R. Kinetic Adaptations of Myosins for Their Diverse Cellular Functions. Traffic. 17 (8), 839-859 (2016).
- Hartman, M. A., Spudich, J. A. The myosin superfamily at a glance. Journal of Cell Science. 125 (Pt 7), 1627-1632 (2012).
- Berg, J. S., Powell, B. C., Cheney, R. E. A millennial myosin census. Molecular Biology of the Cell. 12 (4), 780-794 (2001).
- Sebe-Pedros, A., Grau-Bove, X., Richards, T. A., Ruiz-Trillo, I. Evolution and classification of myosins, a paneukaryotic whole-genome approach. Genome Biology and Evolution. 6 (2), 290-305 (2014).
- Newell-Litwa, K. A., Horwitz, R., Lamers, M. L. Non-muscle myosin II in disease: mechanisms and therapeutic opportunities. Disease Models & Mechanisms. 8 (12), 1495-1515 (2015).
- He, Y. M., Gu, M. M. Research progress of myosin heavy chain genes in human genetic diseases. Yi Chuan. 39 (10), 877-887 (2017).
- Rauscher, A. A., Gyimesi, M., Kovacs, M., Malnasi-Csizmadia, A. Targeting Myosin by Blebbistatin Derivatives: Optimization and Pharmacological Potential. Trends in Biochemical Sciences. 43 (9), 700-713 (2018).
- Straight, A. F., et al. Dissecting temporal and spatial control of cytokinesis with a myosin II Inhibitor. Science. 299 (5613), 1743-1747 (2003).
- Sirigu, S., et al. Highly selective inhibition of myosin motors provides the basis of potential therapeutic application. Proceedings of the National Academy of Sciences of the United States of America. 113 (47), E7448-E7455 (2016).
- Green, E. M., et al. A small-molecule inhibitor of sarcomere contractility suppresses hypertrophic cardiomyopathy in mice. Science. 351 (6273), 617-621 (2016).
- Morgan, B. P., et al. Discovery of omecamtiv mecarbil the first, selective, small molecule activator of cardiac Myosin. ACS Medicinal Chemistry Letters. 1 (9), 472-477 (2010).
- Kepiro, M., et al. para-Nitroblebbistatin, the non-cytotoxic and photostable myosin II inhibitor. Angewandte Chemie International Edition. 53 (31), 8211-8215 (2014).
- Varkuti, B. H., et al. A highly soluble, non-phototoxic, non-fluorescent blebbistatin derivative. Scientific Reports. 6, 26141 (2016).
- Verhasselt, S., et al. Discovery of (S)-3'-hydroxyblebbistatin and (S)-3'-aminoblebbistatin: polar myosin II inhibitors with superior research tool properties. Organic and Biomolecular Chemistry. 15 (9), 2104-2118 (2017).
- Verhasselt, S., Roman, B. I., Bracke, M. E., Stevens, C. V. Improved synthesis and comparative analysis of the tool properties of new and existing D-ring modified (S)-blebbistatin analogs. European Journal of Medicinal Chemistry. 136, 85-103 (2017).
- Warren, G. B., Toon, P. A., Birdsall, N. J., Lee, A. G., Metcalfe, J. C. Reconstitution of a calcium pump using defined membrane components. Proceedings of the National Academy of Sciences of the United States of America. 71 (3), 622-626 (1974).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. Rad54 protein exerts diverse modes of ATPase activity on duplex DNA partially and fully covered with Rad51 protein. Journal of Biological Chemistry. 277 (48), 46205-46215 (2002).
- Hanzelmann, P., Schindelin, H. Structural Basis of ATP Hydrolysis and Intersubunit Signaling in the AAA+ ATPase p97. Structure. 24 (1), 127-139 (2016).
- Hackney, D. D., Jiang, W. Assays for kinesin microtubule-stimulated ATPase activity. Methods in Molecular Biology. 164, 65-71 (2001).
- Kiianitsa, K., Solinger, J. A., Heyer, W. D. NADH-coupled microplate photometric assay for kinetic studies of ATP-hydrolyzing enzymes with low and high specific activities. Analytical Biochemistry. 321 (2), 266-271 (2003).
- Carter, S. G., Karl, D. W. Inorganic phosphate assay with malachite green: an improvement and evaluation. Journal of Biochemical and Biophysical Methods. 7 (1), 7-13 (1982).
- Henkel, R. D., VandeBerg, J. L., Walsh, R. A. A microassay for ATPase. Analytical Biochemistry. 169 (2), 312-318 (1988).
- Rowlands, M. G., et al. High-throughput screening assay for inhibitors of heat-shock protein 90 ATPase activity. Analytical Biochemistry. 327 (2), 176-183 (2004).
- Rule, C. S., Patrick, M., Sandkvist, M. Measuring In Vitro ATPase Activity for Enzymatic Characterization. Journal of Visualized Experiments. (114), 54305 (2016).
- Pardee, J. D., Spudich, J. A. Purification of muscle actin. Methods in Cell Biology. 24, 271-289 (1982).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- Kovacs, M., Toth, J., Hetenyi, C., Malnasi-Csizmadia, A., Sellers, J. R. Mechanism of blebbistatin inhibition of myosin II. Chem Journal of Biological Chemistry. 279 (34), 35557-35563 (2004).
- Allingham, J. S., Smith, R., Rayment, I. The structural basis of blebbistatin inhibition and specificity for myosin II. Nature Structural & Molecular Biology. 12 (4), 378-379 (2005).
- Kettlun, A. M., et al. Purification and Characterization of 2 Isoapyrases from Solanum-Tuberosum Var Ultimus. Phytochemistry. 31 (11), 3691-3696 (1992).
- Hulme, E. C., Trevethick, M. A. Ligand binding assays at equilibrium: validation and interpretation. British Journal of Pharmacology. 161 (6), 1219-1237 (2010).
- Motulsky, H. J., Neubig, R. R. Analyzing binding data. Current Protocols in Neuroscience. 52 (1), 7.5.1-7.5.65 (2010).
- Sehgal, P., Olesen, C., Moller, J. V. ATPase Activity Measurements by an Enzyme-Coupled Spectrophotometric Assay. Methods in Molecular Biology. 1377, 105-109 (2016).
- Solinger, J. A., Lutz, G., Sugiyama, T., Kowalczykowski, S. C., Heyer, W. D. Rad54 protein stimulates heteroduplex DNA formation in the synaptic phase of DNA strand exchange via specific interactions with the presynaptic Rad51 nucleoprotein filament. Journal of Molecular Biology. 307 (5), 1207-1221 (2001).
- Banik, U., Roy, S. A continuous fluorimetric assay for ATPase activity. Biochemistry Journal. 266 (2), 611-614 (1990).
- Xiao, Y. X., Yang, W. X. KIFC1: a promising chemotherapy target for cancer treatment?. Oncotarget. 7 (30), 48656-48670 (2016).
- See, S. K., et al. Cytoplasmic Dynein Antagonists with Improved Potency and Isoform Selectivity. ACS Chemical Biology. 11 (1), 53-60 (2016).
- Datta, A., Brosh, R. M. New Insights Into DNA Helicases as Druggable Targets for Cancer Therapy. Frontiers in Molecular Biosciences. 5, 59 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados