È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Estrazione e purificazione della proteina FAHD1 dal rene suino e dal fegato di topo
In questo articolo
Riepilogo
Questo protocollo descrive come estrarre la proteina 1 contenente dominio della fumarilacetoacetato idrolasi (FAHD1) dal rene suino e dal fegato di topo. I metodi elencati possono essere adattati ad altre proteine di interesse e modificati per altri tessuti.
Abstract
La proteina 1 contenente dominio fumarilacetato idrolasi (FAHD1) è il primo membro identificato della superfamiglia FAH negli eucarioti, che agisce come ossalacetato decarbossilasi nei mitocondri. Questo articolo presenta una serie di metodi per l'estrazione e la purificazione di FAHD1 dal rene suino e dal fegato di topo. I metodi coperti sono la cromatografia a scambio ionico con cromatografia liquida proteica veloce (FPLC), la filtrazione preparativa e analitica del gel con FPLC e gli approcci proteomici. Dopo l'estrazione totale delle proteine, sono state esplorate la precipitazione del solfato di ammonio e la cromatografia a scambio ionico e FAHD1 è stato estratto tramite una strategia sequenziale utilizzando lo scambio ionico e la cromatografia ad esclusione dimensionale. Questo approccio rappresentativo può essere adattato ad altre proteine di interesse (espresse a livelli significativi) e modificato per altri tessuti. Le proteine purificate dai tessuti possono supportare lo sviluppo di anticorpi di alta qualità e/o inibitori farmacologici potenti e specifici.
Introduzione
La proteina 1 contenente dominio FAH eucariotico (FAHD1) agisce come ossalacetato bifunzionale (OAA) decarbossilasi (ODx)1 e idrolasi acilpropurato (ApH)2. È localizzato nei mitocondri2 e appartiene all'ampia superfamiglia FAH degli enzimi 1,2,3,4,5,6. Mentre la sua attività ApH è solo di minore rilevanza, l'attività ODx di FAHD1 è coinvolta nella regolazione del flusso del ciclo TCA 1,7,8,9. L'OAA non è solo richiesto per la reazione centrale della citrato sintasi nel ciclo dell'acido tricarbossilico, ma agisce anche come inibitore competitivo della succinato deidrogenasi come parte del sistema di trasporto degli elettroni e come metabolita cataplerotico. La downregulation dell'espressione genica di FAHD1 nelle cellule endoteliali della vena ombelicale umana (HUVEC) ha determinato una significativa riduzione del tasso di proliferazione cellulare10 e una significativa inibizione del potenziale di membrana mitocondriale, associata a un concomitante passaggio alla glicolisi. Il modello di lavoro si riferisce alla senescenza associata alla disfunzione mitocondriale (MiDAS)11-like fenotipo8, in cui i livelli di OAA mitocondriale sono strettamente regolati dall'attività FAHD1 1,8,9.
La proteina ricombinante è più facile da ottenere attraverso l'espressione e la purificazione dai batteri12 piuttosto che dai tessuti. Tuttavia, una proteina espressa nei batteri può essere influenzata dalla possibile mancanza di modifiche post-traduzionali, o può semplicemente essere problematica (ad esempio, a causa della perdita di plasmidi, risposte allo stress batterico, legami disolfuro distorti / non formati, nessuna o scarsa secrezione, aggregazione proteica, scissione proteolitica, ecc.). Per alcune applicazioni, le proteine devono essere ottenute dal lisato cellulare o dal tessuto, al fine di includere tali modifiche e/o di escludere possibili artefatti. La proteina purificata dai tessuti supporta lo sviluppo di anticorpi di alta qualità e/o inibitori farmacologici potenti e specifici per enzimi selezionati, come per FAHD113.
Questo manoscritto presenta una serie di metodi per l'estrazione e la purificazione di FAHD1 dal rene suino e dal fegato di topo. I metodi descritti richiedono una cromatografia liquida proteica veloce (FPLC), ma per il resto utilizzano apparecchiature di laboratorio comuni. Metodi alternativi possono essere trovati altrove 14,15,16,17. Dopo l'estrazione totale delle proteine, il protocollo proposto prevede una fase di test, in cui vengono discussi i sottoprotocolli per la precipitazione del solfato di ammonio e la cromatografia a scambio ionico (Figura 1). Dopo aver definito questi sotto-protocolli, la proteina di interesse viene estratta attraverso una strategia sequenziale utilizzando lo scambio ionico e la cromatografia di esclusione dimensionale con FPLC. Sulla base di queste linee guida, il protocollo finale può essere adattato individualmente per altre proteine di interesse.
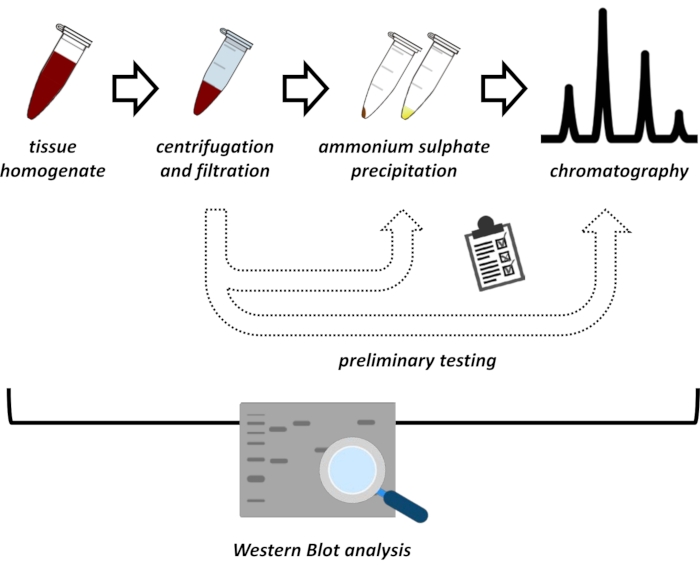
Figura 1: La strategia generale di questo protocollo. Dall'alto verso il basso: le proteine vengono estratte dai tessuti. L'omogeneizzato tissutale viene preparato, centrifugato e filtrato. Per ogni coppia di campioni surnatante e derivati da pellet, è necessario eseguire test per la precipitazione del solfato di ammonio e la cromatografia a scambio ionico (FPLC) per sondare le condizioni ottimali. Dopo aver stabilito questi sotto-protocolli, la proteina può essere estratta tramite una procedura sequenziale di precipitazione del solfato di ammonio, cromatografia a scambio ionico e cromatografia di esclusione di dimensioni ripetitive (FPLC) a diverse concentrazioni di pH e sale. Tutti i passaggi devono essere controllati da western blot. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
Tutti gli esperimenti sono stati eseguiti nel rispetto delle linee guida istituzionali. Il rene suino è stato ottenuto fresco dal supermercato locale. I tessuti epatici sono stati raccolti da topi selvatici C57BL6 mantenuti presso l'Istituto per la ricerca sull'invecchiamento biomedico dell'Università di Innsbruck, Rennweg 10, 6020 Innsbruck, Austria sotto la supervisione di Univ.-Doz. Dr. Pidder Jansen-Dürr, coperto da autorizzazione etica come leader del progetto rilasciata nel 2013 (BMWF-66.008/0007-II/3b/2013). La manutenzione e l'uso dei topi per il progetto sono coperti dal permesso etico n. 2020-0.242.978 dal 5 maggio 2020, rilasciato dal Ministero austriaco per l'istruzione, la scienza e la ricerca (BMBWF).
1. Preparativi
NOTA: Prima dell'inizio del protocollo, è necessario preparare diverse cose, ad esempio il tampone di lisi proteica, il campione di tessuto grezzo e un anticorpo specifico, oltre a sostanze chimiche e materiali generali.
- Preparare 250 mL di tampone di lisi proteica per 100 g di peso netto del tessuto: 250 mL di 1x PBS con 50 mM di NaF, 1 mM di PMSF, 2 μg/mL di aprotinina e 1 mM di orovavanadato attivato (vedere Tabella 1). Filtrare la soluzione utilizzando un'unità filtrante a siringa da 0,22 μm.
NOTA: L'attivazione dell'ortopvanadato è necessaria prima dell'uso per convertirlo in un inibitore più potente della proteina tirosina fosfatasi18. L'orovavanadato attivato può essere ottenuto da fornitori commerciali, ma anche preparato come segue.- Preparare una soluzione madre da 200 mM di ortopvanadato (sodio) in ddH2O. Per preparare 10 mL di soluzione, aggiungere 368 mg di Na3VO4 a 9 mL di acqua e sciogliere mescolando. Una volta disciolto, portare il volume a 10 mL con ddH2O.
NOTA: il pH iniziale della soluzione di ortovanadato di sodio può variare con la fonte del materiale e il pH deve essere regolato a 10 in un approccio ripetitivo come segue. - A seconda del pH iniziale della soluzione, regolare il pH a 10 con NaOH o HCl. A pH > 10, la soluzione avrà un colore giallo. Far bollire la soluzione fino a quando non diventa incolore, raffreddarla a temperatura ambiente e controllare il pH. Se il pH è >10, aggiungere un piccolo volume di HCl per regolare il pH a 10. A questo punto, la soluzione potrebbe ingiallire di nuovo.
- Ripetere l'ebollizione e il raffreddamento fino a quando la soluzione rimane incolore e il pH si stabilizza a 10 (circa 5-7 volte). A questo punto, l'aggiunta di HCl si traduce in un debole aspetto di colore giallo nella soluzione. Conservare l'orovavanadato attivato in aliquote da 1 mL a -20 °C.
- Preparare una soluzione madre da 200 mM di ortopvanadato (sodio) in ddH2O. Per preparare 10 mL di soluzione, aggiungere 368 mg di Na3VO4 a 9 mL di acqua e sciogliere mescolando. Una volta disciolto, portare il volume a 10 mL con ddH2O.
- Preparare i tubi con 2 ml di tampone di lisi per grammo di tessuto e metterli sul ghiaccio.
NOTA: Questo protocollo utilizzava otto tubi da 50 mL, ciascuno riempito con 30 mL di tampone di lisi in totale per un rene suino (circa 100-150 g), e due tubi ciascuno riempito con 40 mL di tampone di lisi per 20 fegati di topo (ciascuno 1-2 g) in totale. - Preparare il tessuto: sezionare il tessuto su una lastra di vetro pre-pulita posta sul ghiaccio in una scatola di polistirene espanso. Tagliare pezzi di tessuto di circa 100 mg ciascuno per essere facilmente trasferiti nei rispettivi tubi per la successiva lisi. Trasferire i pezzi di tessuto nei tubi preparati (fase 1.2).
- Preparare una soluzione di solfato di ammonio saturo: riscaldare 500 mL di ddH2O a 70 °C e, mescolando, aggiungere gradualmente solfato di ammonio in polvere (vedere Tabella dei materiali) fino a quando non si dissolve più solfato di ammonio. Raffreddare questa soluzione (sovra)satura a temperatura ambiente e conservarla a 4 °C durante la notte.
2. Estrazione totale delle proteine
NOTA Dopo aver preparato il campione in un tampone di lisi proteica fredda (vedere il punto 1.3), omogeneizzare il tessuto nel miglior modo possibile tramite sonicazione mediante sonda ad ultrasuoni o utilizzando un omogeneizzatore elettrico come segue.
- Omogeneizzazione dei tessuti
- Nel caso di un rene suino, sonicare la sospensione preferibilmente con una sonda ad ultrasuoni mantenendo il campione sul ghiaccio (10 cicli di impulso di 15 s, con intervalli di 30 s tra gli impulsi per raffreddare il campione su ghiaccio, a media ampiezza con ciclo di lavoro del 50%).
- Nel caso di organi di topo, omogeneizzare la sospensione utilizzando un omogeneizzatore elettrico (iniziando con una forza bassa e accelerando lentamente a forza media) mantenendo il campione sul ghiaccio. Lavare regolarmente l'omogeneizzatore elettrico in PBS per rimuovere qualsiasi materiale organico che ostruisce il dispositivo.
- Prelevare 20 μL dai campioni e verificare al microscopio se le cellule del tessuto omogeneizzato sono correttamente distrutte; altrimenti, ripetere l'omogeneizzazione.
- Centrifugare i tubi in una centrifuga da tavolo a 10.000 x g per 30 min a 4 °C.
NOTA: Facoltativamente, centrifugare il surnatante una seconda volta a 20.000 x g per 30 minuti a 4 °C per eliminare piccole frazioni del pellet iniziale che potrebbero essere state trasferite. Ciò semplificherà la successiva filtrazione nella fase 2.3. - Raccogliere il surnatante in un tubo fresco e metterlo sul ghiaccio. Filtrare sequenzialmente il surnatante utilizzando unità filtranti a siringa da 0,45 μm e 0,22 μm. Aliquotare il surnatante in lotti da 10 ml e congelarli a -20 °C per la conservazione a breve termine o a -80 °C per una conservazione più lunga.
NOTA: il prefiltraggio con 0,45 μm rimuove la maggior parte delle particelle prima che una seconda fase di filtraggio con 0,22 μm rimuova le particelle più fini. L'utilizzo diretto del filtro da 0,22 μm può causare il rischio di intasare i filtri. - Preparare un campione da 50 μL per l'analisi SDS-PAGE/western blot aggiungendo 10 μL di tampone campione SDS 5x (vedere Tabella 1) a 40 μL del surnatante e quindi bollendo a 95 °C per 10 minuti.
- Facoltativamente, risospendere circa 100 μL di pellet ottenuto nella fase 2.2 in 900 μL di ddH2O e preparare un campione per l'analisi SDS-PAGE/western blot come descritto sopra.
NOTA: l'inclusione di campioni derivati da pellet nell'analisi western blot, oltre al controllo positivo, indicherà se l'espressione della proteina è bassa o se l'anticorpo è problematico.
- Facoltativamente, risospendere circa 100 μL di pellet ottenuto nella fase 2.2 in 900 μL di ddH2O e preparare un campione per l'analisi SDS-PAGE/western blot come descritto sopra.
3. Analisi SDS-PAGE e western blot
NOTA: l'analisi Western blot è necessaria per verificare la solubilità delle proteine. Di seguito viene descritto un protocollo per l'elettroblotting, utilizzando un sistema di wet/tank blotting (vedi Tabella dei materiali). Un protocollo alternativo per SDS-PAGE può essere trovato altrove19.
- Preparare un gel discontinuo al 12,5% di poliacrilammide SDS-PAGE secondo le istruzioni del produttore (ad esempio, un gel impilabile sopra un gel risolutivo; vedere Tabella 1). Eseguire gli esempi preparati in precedenza durante il passaggio 2 (simile ai passaggi 4, 5 e 6; vedere di seguito).
- Caricare una scala marcatore proteica nel primo pozzo (vedi Tabella dei materiali). Carico 5 ng della proteina ricombinante hFAHD1 (ottenuta dai batteri12; vedi Tabella 1) come controllo positivo nel secondo pozzo.
- Successivamente, caricare 20 μL del campione da analizzare e riempire tutti i pozzetti rimanenti con 20 μL di tampone campione SDS-PAGE 1x preparato (cioè 5x tampone campione diluito con ddH2O). Eseguire i gel SDS-PAGE a 125 V utilizzando il buffer in esecuzione SDS (vedere Tabella 1).
- Una volta completato SDS-PAGE, eseguire un'analisi western blot e sondare le membrane utilizzando l'anticorpo disponibile sollevato contro FAHD1 (vedere Tabella 1).
NOTA: Poiché i campioni vengono prelevati da tessuto omogeneizzato grezzo, di solito la qualità della SDS-PAGE e l'analisi western blot a questo punto sono compromesse; tuttavia, è importante verificare se la proteina da estrarre è solubile nel surnatante. Il seguente protocollo è stato testato per il rene suino e diversi organi di topo, tra cui fegato, cuore, cervello e rene.- Preparare il buffer di trasferimento western blot 10x (vedere Tabella 1). Preparare il buffer di trasferimento 1x western blot (vedere Tabella 1) e raffreddarlo a 4 °C.
- Attivare una membrana PVDF per 2 minuti in metanolo. Lavare la membrana in ddH2O per 2 min. Equilibrare la membrana per 15 minuti in 1x tampone di trasferimento western blot.
- Lavare il gel SDS con 1x PBS per 10 minuti mentre si agita per rimuovere il buffer in esecuzione SDS, quindi incubare il gel in 1x western blot transfer buffer per 10 min per l'equilibrio. Assemblare la cassetta di elettroblotting (cioè combinando la membrana PVDF attivata e i gel) secondo le istruzioni del produttore.
- Eseguire il blot tramite elettroblotting a 300 mA per 1 ora in una scatola di polistirene espanso riempita di ghiaccio o nella cella frigorifera (4 °C). Trasferire la membrana PVDF in un tubo da 50 ml con il lato esposto rivolto verso il lato interno del tubo. Incubare la membrana in 20 mL di tampone di blocco western blot (vedere Tabella 1) durante la notte a 4 °C mentre si rotola su un rullo tubolare (vedere Tabella dei materiali).
- Il giorno successivo, lavare la membrana per 5 minuti con 20 ml di tampone di lavaggio Western Blot (PBS con 0,1% (v / v) Tween 20) nello stesso tubo durante la laminazione. Incubare la membrana nello stesso tubo con l'anticorpo primario2 (mirato a FAHD1; vedere Tabella 1) diluito 1:500 nel tampone di blocco western blot per 1 ora a temperatura ambiente durante la laminazione.
- Lavare la membrana nello stesso tubo tre volte per 10 minuti ciascuno con 20 ml di tampone di lavaggio western blot durante la laminazione. Incubare la membrana per 30 minuti a temperatura ambiente con anticorpo secondario coniugato HRP (vedere Tabella dei materiali) diluito 1:3000 in 5 mL di tampone bloccante western blot.
- Lavare la membrana nello stesso tubo tre volte per 10 minuti ciascuno con 20 ml di tampone di lavaggio western blot e due volte per 5 minuti ciascuno con 1x PBS. Asciugare la membrana tenendola con cura con una pinzetta su un bordo e toccando un pezzo di cellulosa o un pezzo di carta Whatman con il bordo opposto (inferiore) della membrana. Mettere la membrana (lato esposto verso l'alto) su una lastra di vetro pulita.
- Coprire con cura l'intera membrana con 1 mL di substrato ECL western blot preparato utilizzando una pipetta, facendo attenzione a non creare bolle d'aria. Lasciare incubare la soluzione ECL per 3 minuti e sviluppare immediatamente la membrana utilizzando un film a raggi X o un sistema di imaging.
NOTA: Se la proteina non è stata rilevata in nessuno dei campioni ma solo nel controllo positivo, ciò può indicare che la proteina è insolubile o non presente in quantità adeguate per essere rilevata dall'anticorpo. Se venissero caricati solo nanogrammi del controllo positivo, il primo scenario è più probabile. Se non è stata rilevata alcuna proteina, controllare la qualità dell'anticorpo e magari passare a un anticorpo policlonale piuttosto che a un anticorpo monoclonale. In rare occasioni, ad esempio per alcune proteine idrofobiche, la proteina può essere rilevabile dopo la centrifugazione, ma non dopo la filtrazione. In tal caso, si consiglia di utilizzare speciali unità filtranti per proteine idrofobiche.
- Facoltativamente, colorare le membrane PVDF dopo western blot per controllare il successo del trasferimento della proteina dal gel SDS-PAGE alla membrana PVDF.
NOTA: la colorazione coomassie è consigliata per la risoluzione dei problemi, lo sviluppo del metodo e la documentazione, ma tieni presente che dopo aver applicato questo protocollo, le membrane vengono perse a causa di ulteriori analisi western blot. La colorazione di Ponceau S dà una colorazione più debole, ma può essere utilizzata se le membrane devono essere nuovamente sondate.- Preparare piccoli vassoi contenenti la colorazione (Coomassie o Ponceau S) e le soluzioni di decolorazione.
- Usando una pinzetta, mettere la membrana nella soluzione di colorazione e agitare delicatamente fino a quando la membrana è ben macchiata (5-10 min).
- Trasferire la membrana nella soluzione di decolorazione e agitare fino a quando la soluzione è satura (5-10 min). Ripetere la fase di detaining fino a quando le bande proteiche possono essere osservate sulla membrana; se non si osserva alcuna banda, ripetere la colorazione con un tempo di incubazione più lungo. Asciugare la membrana posizionandola su una lastra di vetro usando una pinzetta.
4. Test: precipitazione del solfato di ammonio
NOTA: La precipitazione del solfato di ammonio è un metodo di purificazione delle proteine alterando la solubilità della proteina. In un esperimento preliminare, la concentrazione di solfato di ammonio viene aumentata sequenzialmente a un valore che precipita una quantità massima di contaminanti proteici, lasciando FAHD1 in soluzione. La solubilità della proteina viene nuovamente sondata tramite analisi western blot.
- Procedere dal punto 2.3: scongelare un'aliquota del campione o procedere direttamente dopo l'estrazione delle proteine (cioè senza congelare il campione). Filtrare il campione utilizzando un'unità filtrante da 0,22 μm per escludere possibili precipitati dopo lo scongelamento. Preparare sei tubi da 1,5 ml su ghiaccio e trasferire 250 μL di campione in ciascun tubo.
- Preparare una serie di diluizione di 5%, 10%, 15%, 20%, 25% e 30% di solfato di ammonio nei tubi preparati sopra e portare il volume finale a 1000 μL con tampone di lisi proteica. Incubare i campioni a 4 °C durante la notte su un rotatore a tubo (vedere Tabella dei materiali).
- Utilizzando una centrifuga da tavolo, centrifugare a 10.000 x g per 30 minuti a 4 °C e trasferire con cura tutti i supernatanti in tubi separati. Asciugare all'aria i pellet risultanti e risospestarne ciascuno in 1000 μL di ddH2O.
- Per ogni coppia di pellet risospeso e surnatante della fase precedente, mescolare 40 μL con 10 μL di tampone campione SDS 5x e far bollire a 95 °C con coperchi aperti fino a quando la maggior parte del liquido non si è vaporizzata. Quindi, risospesciare il pellet in una miscela di DMSO al 50% in ddH2O.
- Eseguire SDS-PAGE (passaggio 3) ma eseguire i gel a 80 V per 3 ore. Per ogni concentrazione di solfato di ammonio, caricare i campioni derivati dal pellet risospeso e dal surnatante (fase 4.3) in coppia. Eseguire un'analisi western blot (passaggio 3).
- Verificare la massima concentrazione di solfato di ammonio, alla quale la proteina da purificare (ad esempio, FAHD1) rimane nel campione derivato dal surnatante. Sulla base dei risultati, definire un protocollo di precipitazione del solfato di ammonio per la proteina di interesse, da utilizzare in esperimenti futuri.
NOTA: il solfato di ammonio è ben noto per distorcere SDS-PAGE e western blot. Con l'aumentare della concentrazione di solfato di ammonio, la qualità dell'analisi western blot sarà compromessa. Tuttavia, come per la fase 3 precedente, questa analisi viene utilizzata per verificare la solubilità della proteina di interesse a determinate concentrazioni di solfato di ammonio. Questo protocollo ha lo scopo di far precipitare altre proteine, mentre la proteina da purificare deve rimanere solubile.
5. Test: cromatografia a scambio ionico con FPLC
NOTA: Le molecole con gruppi funzionali carichi sono legate a una colonna di particelle di silice per FPLC, consentendo la differenziazione delle proteine in base alla loro carica superficiale. Eseguire questo passaggio due volte, utilizzando la colonna di scambio cationico e la colonna di scambio anionico (vedere Tabella dei materiali). Le fasi del protocollo sono le stesse per la cromatografia a scambio cationico o anionico, ma i buffer da utilizzare sono diversi (vedi Tabella 1); entrambi con condizioni "a basso contenuto di sale" 15 mM NaCl e "ad alto sale" 1 M NaCl. Per le colonne utilizzate si consiglia una portata di 1 mL/min.
- Impostare il sistema FPLC con la colonna di scambio anionico o cationico. Lavare la colonna con 5 volumi di colonna (CV) del 20% etOH (in H2O), seguiti da 5 CV di ddH2O. In alternativa, lavare la colonna con 1 CV di tampone a basso contenuto di sale, tampone ad alto contenuto di sale e di nuovo tampone di sale basso nell'ordine fino a quando non si osservano più picchi nel cromatogramma, ma lavare almeno una volta.
- Dopo aver determinato il protocollo ottimale per la precipitazione del solfato di ammonio su piccola scala (fase 4), applicare il protocollo di precipitazione a 10 ml di omogeneizzato tissutale originale (fase 2). Facoltativamente, dializzare il campione contro il buffer a basso contenuto di sale.
- Applicare il campione sulla colonna (ad esempio, per iniezione o utilizzando una pompa campione) e raccogliere il flusso attraverso. Lavare la colonna con 1 CV del tampone a basso contenuto di sale.
- Impostare un'eluizione a gradiente lineare da tampone salino basso al 100% / tampone salino alto 0% a tampone salino basso a 0% / tampone salino alto al 100% entro 3 CV. Raccogliere continuamente frazioni da 1 ml. Al termine del gradiente, continuare a funzionare con l'alto tampone salino fino a quando non vengono rilevati più picchi associati alle proteine (assorbimento UV a 280/255 nm) nel cromatogramma nell'intervallo di 1 CV.
- Applicare 1 mL di SDS al 25% disciolto in 0,5 M NaOH (in ddH2O) per pulire la colonna. Consecutivamente, lavare la colonna con 3 CV di ddH2O e 3 CV di EtOH al 20% (in ddH2O).
- Raccogliere campioni SDS-PAGE di tutte le frazioni di picco e del flow-through e sondarli tramite western blot per la presenza della proteina di interesse (fase 3). Congelare a scatto le frazioni raccolte in azoto liquido e conservarle a -80 °C.
- Una volta completata l'analisi western blot, scongelare e raggruppare le frazioni contenenti la proteina di interesse e scartare le altre. Ripetere i passaggi 5.1-5.5 con la colonna alternativa (cioè colonna di scambio cationica o anionica).
- Dopo che entrambe le colonne sono state sondate, definire un protocollo FPLC per la proteina di interesse, da utilizzare in esperimenti futuri. Ridurre il volume della soluzione proteica utilizzando unità filtranti ultracentranti (10 kDa, vedi Tabella dei materiali) fino a 2 ml.
NOTA: ci sono due risultati attesi di questa serie di esperimenti. O la proteina di interesse si è attaccata a una delle colonne, e la soluzione proteica è già abbastanza pura dopo l'eluizione, o la proteina è rimasta nel flusso in entrambi i casi. In quest'ultimo scenario, sebbene la proteina sia nel flusso attraverso, l'effetto di pulizia di questo passaggio potrebbe essere ancora significativo. In tal caso, come per FAHD1 nel rene suino e nel fegato di topo, questa fase di scambio ionico verrà comunque eseguita. Se né la colonna di scambio cationico né quella a scambio anionico possono fornire un corretto effetto di pulizia, si può provare a modificare il pH del lisato e del tampone e a dializzare il campione contro il buffer in esecuzione prima dell'applicazione a FPLC.
6. Estrazione di proteine mediante sottoprotocolli definiti per la precipitazione del solfato di ammonio e FPLC
NOTA: Le particelle porose in una colonna di gel di silice per FPLC (Vedi Tabella dei materiali) consentono la differenziazione delle proteine in base al loro raggio idrodinamico. I passaggi descritti devono essere eseguiti con un sistema FPLC, utilizzando la cromatografia di esclusione dimensionale (SEC). Per la colonna SEC utilizzata (vedere Tabella dei materiali), si consiglia una portata di 0,3 ml/min.
- Preparare tutti i materiali necessari (vedere il passaggio 1) ed estrarre la proteina totale dal tessuto (vedere il passaggio 2). Eseguire una precipitazione del solfato di ammonio con tutto l'omogeneizzato tissutale che non è stato utilizzato per il test (vedere il passaggio 4). Per volumi più grandi, concentrare il lisato utilizzando unità filtranti ultracentrifugabili (10 kDa; vedi Tabella dei materiali) fino a un volume inferiore di 50 ml o meno.
- Eseguire una prima fase di purificazione utilizzando la cromatografia a scambio ionico (vedere il passaggio 5).
- Preparare i campioni per Western Blot, come descritto nei passaggi precedenti. Eseguire l'analisi western blot e raggruppare tutte le frazioni contenenti FAHD1 dalla cromatografia a scambio ionico.
- Ridurre il volume della soluzione proteica fino a 2 mL utilizzando unità filtranti ultracentranti (10 kDa). Filtrare sequenzialmente la soluzione con unità filtranti a siringa da 0,45 μm e 0,22 μm per rimuovere eventuali microindurite.
- Equilibrare la colonna SEC con 1 CV di buffer in esecuzione SEC (vedere Tabella 1), contenente 1 mM DTT. Caricare il campione sulla colonna ed eseguire la cromatografia fino a quando tutte le proteine sono eluite (1-2 CV).
- Raccogliere frazioni di 1 mL del flusso passante che corrisponde a picchi significativi nel cromatogramma (assorbimento UV a 280/255 nm) e preparare campioni da 50 μL di ciascuna frazione raccolta per l'analisi SDS-PAGE e western blot, come descritto nei passaggi precedenti. Congelare a scatto tutte le frazioni utilizzando azoto liquido e conservarle a -80 °C.
- Lavare consecutivamente la colonna SEC con 1 CV di ddH2O e 1 CV di 20% EtOH (in ddH2O). Eseguire l'analisi western-Blot e raggruppare tutte le frazioni contenenti FAHD1. Ridurre il volume della soluzione proteica fino a 2 ml utilizzando unità filtranti ultracentranti (10 kDa, vedi Tabella dei materiali).
- Valutare la concentrazione proteica utilizzando un kit di analisi BCA commerciale (vedere Tabella dei materiali).
NOTA: Il pH e il contenuto salino della fase mobile possono influenzare il profilo di eluizione delle proteine globulari20. Le condizioni acide o basiche possono comportare picchi meno definiti e un aumento delle interazioni proteina-matrice che porta a una parziale ritenzione di proteine sulla colonna20. Questo effetto può essere sfruttato per un'ulteriore purificazione delle proteine. Una ripetizione della fase 6 con diverse portate, pH e concentrazioni di sale può migliorare la purezza della proteina20.
7. Colorazione dell'argento
NOTA: L'analisi della colorazione dell'argento dei gel SDS-PAGE è necessaria per verificare la presenza di contaminazioni proteiche che potrebbero non essere viste con la colorazione coomassie. Il seguente protocollo è una delle tante versioni che si possono trovare nella letteratura21. Eseguire tutte le fasi di incubazione agitando in un vassoio di vetro pulito. Raccogliere tutti i liquidi contenenti argento e formaldeide in un contenitore speciale per rifiuti e scartarli correttamente.
- Incubare i gel SDS-PAGE in soluzione fissante colorante all'argento (vedere Tabella 1) durante la notte nella cella frigorifera. Incubare i gel in soluzione di incubazione contenente argento (vedere Tabella 1) per 3 ore a temperatura ambiente. Facoltativamente, aggiungere glutaraldeide (vedere Tabella 1) per migliorare il rilevamento di bande deboli. Lavare i gel quattro volte in ddH2O per 10 minuti ciascuno.
- Incubare i gel in soluzione di argento colorante argento (vedere Tabella 1) per 1 ora.
NOTA: Attenzione che d'ora in poi tutti i liquidi e il gel stesso contengono argento e formaldeide che sono tossici. - Incubare i gel in una soluzione di sviluppo per la colorazione dell'argento (vedere Tabella 1) con un vigoroso scuotimento fino a quando le bande sono chiaramente visibili. Per arrestare la reazione, scartare la soluzione di sviluppo e incubare immediatamente i gel in soluzione di arresto della colorazione dell'argento (vedere Tabella 1) per un minimo di 10 minuti.
NOTA: le bande macchiate nei passaggi 7.2 e 7.3 diventeranno costantemente più sviluppate. L'aggiunta di più formaldeide alla soluzione di quanto dichiarato può essere necessaria se la colorazione è debole.
Risultati
La proteina FAHD1 è stata estratta dal rene suino e dal fegato di topo utilizzando il protocollo presentato. Per il tessuto di topo, sono necessari più organi per ottenere diversi μg dopo la fase finale di purificazione. Per questo motivo, questo articolo si concentra sull'estrazione di FAHD1 dai reni suini, che è un esperimento molto più esemplare. L'estrazione di FAHD1 dal fegato di topo viene eseguita per presentare le difficoltà e le possibili insidie di questo protocollo. Si raccomanda generalmente di utilizza...
Discussione
Passaggi critici nel protocollo
Seguire le linee guida comuni per la manipolazione delle proteine è essenziale, come lavorare sul ghiaccio e in condizioni di pH e sale moderate. L'uso di inibitori della proteasi è benefico per il metodo, mentre l'uso di inibitori del proteasoma è altamente raccomandato. Il congelamento e lo scongelamento del campione possono sempre comportare la precipitazione proteica (almeno parzialmente), pertanto qualsiasi aliquota scongelata del lisato proteico iniziale (fase ...
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti.
Riconoscimenti
Gli autori sono molto grati per l'assistenza tecnica di Ayse Öztürk ed Eva Albertini. I topi utilizzati per la generazione di tessuto epatico sono stati mantenuti sotto la supervisione di Univ.-Doz. Dr. Pidder Jansen-Dürr (Istituto per la ricerca sull'invecchiamento biomedico presso l'Università di Innsbruck, Rennweg 10, 6020 Innsbruck, Austria).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 µm filter units | MERCK | SLGP033RS | Millex-HP, 0.22 µm, PES 33 mm, not steril |
| 0.45 µm filter units | MERCK | SLHP033NS | Millex-HP, 0.45 µm, PES 33 mm, not steril |
| 15 mL Falcon tubes | VWR | 734-0451 | centrifugal tubes |
| 50 mL Falcon tubes | VWR | 734-0448 | centrifugal tubes |
| 96-Well UV Microplate | Thermo-Fischer | 8404 | UV/VIS transparent flat-bottom 96 well plates |
| Acrylamide/Bis Solution (40%, 29:1 ratio) | BIO-RAD | #1610147 | 40% acrylamide/bis-acrylamide, 29:1 (3.3% crosslinker) solution for casting polyacrylamide gels |
| ÄKTA FPLC system | GE Healthcare Life Sciences / Cytiva | - | using the FPLC system by GE Healthcare; different custom versions exist; this work used the "ÄKTA pure" system |
| Amicon Ultra-15, PLGC Ultracel-PL Membran, 10 kDa | MERCK | UFC901024 | centrifigal filters for protein enrichment; 10 kDa molecular mass filter; 15 mL |
| Amicon Ultra-4, PLGC Ultracel-PL Membran, 10 kDa | MERCK | UFC801024 | centrifigal filters for protein enrichment; 10 kDa molecular mass filter; 4 mL |
| Ammonium sulfate powder | MERCK | A4418 | ammonium sulphate for molecular biology, ≥99.0% |
| Ammoniumpersulfat reagent grade, 98% | MERCK | 215589 | Catalyst for acrylamide gel polymerization. |
| Coomassie Brilliant blue R 250 | MERCK | 1125530025 | Coomassie Brilliant blue R 250 (C.I. 42660) for electrophoresis Trademark of Imperial Chemical Industries PLC. CAS 6104-59-2, pH 6.2 (10 g/l, H2O, 25 °C) |
| Dialysis tubing cellulose membrane | MERCK | D9277 | Cellulose membranes for the exchange of buffers via dialysis. |
| Eppendof tubes 1.5 mL | VWR | 525-1042 | microcentrifugal tubes; autoclaved |
| HiLoad 26/600 Superdex 75 pg | GE Healthcare Life Sciences / Cytiva | 28989334 | HiLoad Superdex 75 pg prepacked columns are for high-resolution size exclusion chromatography of recombinant proteins |
| Immun-Blot PVDF Membrane | BIO-RAD | #1620177 | PVDF membranes are protein blotting membranes optimized for fluorescent and multiplex fluorescent applications. |
| Mini Trans-Blot Electrophoretic Transfer Cell | BIO-RAD | #1703930 | Use the Mini Trans-Blot Cell for rapid blotting of Mini-PROTEAN precast and handcast gels. |
| Mini-PROTEAN Tetra Vertical Electrophoresis Cell for Mini Precast Gels | BIO-RAD | #1658004 | 4-gel vertical electrophoresis system, includes electrode assembly, companion running module, tank, lid with power cables, mini cell buffer dam. |
| Mono Q 10/100 GL | GE Healthcare Life Sciences / Cytiva | 17516701 | Mono Q columns are strong anion exchange chromatography columns for protein analysis or small scale, high resolution polishing of proteins. |
| Mono S 10/100 GL | GE Healthcare Life Sciences / Cytiva | 17516901 | Mono S columns are strong cation exchange chromatography columns for protein analysis or small scale high resolution polishing of proteins. |
| PageRuler Prestained Protein Ladder, 10 to 180 kDa | Thermo-Fischer | 26616 | A mixture of 10 blue-, orange-, and green-stained proteins (10 to 180 kDa) for use as size standards in protein electrophoresis (SDS-PAGE) and western blotting. |
| Pierce BCA Protein Assay Kit | Thermo-Fischer | 23225 | A two-component, high-precision, detergent-compatible protein assay for determination of protein concentration. |
| Sonifier 250; Ultrasonic Cell Disruptor w/ Converter | Branson | - | New models at https://www.emerson.com/documents/automation/brochure-sonifier-sfx250-sfx550-cell-disruptors-homogenizers-branson-en-us-168180.pdf |
| Swine Anti-Rabbit Immunoglobulins/HRP (affinity isolated) | Agilent Dako | P0399 | The antibody used for horseradish peroxidase conjugation reacts with rabbit immunoglobulins of all classes. |
| TEMED, 1,2-Bis(dimethylamino)ethane, TMEDA | MERCK | T9281 | TEMED (N,N,N′,N′-Tetramethylethylenediamine) is molecule which allows rapid polymerization of polyacrylamide gels. |
| Tube Roller | - | - | A general tube rotator roller; e.g. a new model at https://labstac.com/de/Mixer/Roller/c/71 |
| Tube Rotator | - | - | A general tube rotator wheel; e.g. a new model at https://labstac.com/de/Tube-Roller/p/MT123 |
| ULTRA-TURRAX; T 25 digital | IKA | 0003725000 | New models at https://www.ika.com/de/Produkte-Lab-Eq/Dispergierer-Dipergiergeraet-Homogenisierer-Homogenisator-csp-177/T-25-digital-ULTRA-TURRAX-cpdt-3725000/ |
Riferimenti
- Pircher, H., et al. Identification of FAH domain-containing protein 1 (FAHD1) as oxaloacetate decarboxylase. Journal of Biological Chemistry. 290 (11), 6755-6762 (2015).
- Pircher, H., et al. Identification of human Fumarylacetoacetate Hydrolase Domain-containing Protein 1 (FAHD1) as a novel mitochondrial acylpyruvase. Journal of Biological Chemistry. 286 (42), 36500-36508 (2011).
- Kang, T. -. W., et al. Senescence surveillance of pre-malignant hepatocytes limits liver cancer development. Nature. 479 (7374), 547-551 (2011).
- Hong, H., Seo, H., Park, W., Kim, K. K. -. J. Sequence, structure and function-based classification of the broadly conserved FAH superfamily reveals two distinct fumarylpyruvate hydrolase subfamilies. Environmental Microbiology. 22 (1), 270-285 (2020).
- Timm, D. E., Mueller, H. A., Bhanumoorthy, P., Harp, J. M., Bunick, G. J. Crystal structure and mechanism of a carbon-carbon bond hydrolase. Structure. 7 (9), 1023-1033 (1999).
- Bateman, R. L., et al. Mechanistic inferences from the crystal structure of Fumarylacetoacetate Hydrolase with a bound phosphorus-based inhibitor. Journal of Biological Chemistry. 276 (18), 15284-15291 (2001).
- Weiss, A. K. H., et al. Structural basis for the bi-functionality of human oxaloacetate decarboxylase FAHD1. Biochemical Journal. 475 (22), 3561-3576 (2018).
- Etemad, S., et al. Oxaloacetate decarboxylase FAHD1 - a new regulator of mitochondrial function and senescence. Mechanisms of Ageing and Development. 177, 22-29 (2019).
- Weiss, A. K. H., et al. Regulation of cellular senescence by eukaryotic members of the FAH superfamily - A role in calcium homeostasis. Mechanisms of Ageing and Development. 190, 111284 (2020).
- Petit, M., Koziel, R., Etemad, S., Pircher, H., Jansen-Dürr, P. Depletion of oxaloacetate decarboxylase FAHD1 inhibits mitochondrial electron transport and induces cellular senescence in human endothelial cells. Experimental Gerontology. 92, 7-12 (2017).
- Wiley, C. D., et al. Mitochondrial dysfunction induces senescence with a distinct secretory phenotype. Cell Metabolism. 23 (2), 303-314 (2016).
- Weiss, A. K. H., et al. Expression, purification, crystallization, and enzyme assays of Fumarylacetoacetate Hydrolase Domain-containing proteins. Journal of Visualized Experiments: JoVE. (148), e59729 (2019).
- Weiss, A. K. H., et al. Inhibitors of Fumarylacetoacetate Hydrolase Domain Containing Protein 1 (FAHD1). Molcules. 26 (16), 5009 (2021).
- Mizutani, H., Kunishima, N. Purification, crystallization and preliminary X-ray analysis of the fumarylacetoacetase family member TTHA0809 from Thermus thermophilus HB8. Acta Crystallographica Section F Structural Biology and Crystallization Communications. 63 (9), 792-794 (2007).
- Lee, C. H. A simple outline of methods for protein isolation and purification. Endocrinology and Metabolism. 32 (1), 18-22 (2017).
- Amer, H. E. A. Purification of proteins: Between meaning and different methods). Proteomics Technologies and Applications. , (2019).
- Niu, L., Yuan, H., Gong, F., Wu, X., Wang, W. Protein extraction methods shape much of the extracted proteomes. Frontiers in Plant Science. 9, 802 (2018).
- Gordon, J. A. Use of vanadate as protein-phosphotyrosine phosphatase inhibitor. Methods in Enzymology. 201, 477-482 (1991).
- Gallagher, S. R. SDS-polyacrylamide gel electrophoresis (SDS-PAGE). Current Protocols in Essential Laboratory Techniques. , (2012).
- . Effect of pH on Protein Size Exclusion Chromatography Available from: https://www.agilent.com/cs/library/applications/5990-8138EN.pdf (2011)
- Sørensen, B. K., et al. Silver staining of proteins on electroblotting membranes and intensification of silver staining of proteins separated by polyacrylamide gel electrophoresis. Analytical Biochemistry. 304 (1), 33-41 (2002).
- Fagerberg, L., et al. Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics. Molecular & Cellular Proteomics. 13 (2), 397-406 (2014).
- . Cytiva Life Fundamentals of size exclusion chromatography Available from: https://www.cytivalifesciences.com/en/us/solutions/protein-research/knowledge-center/protein-purification-methods/size-exclusion-chromatography (2022)
- Rosano, G. L., Ceccarelli, E. A. Recombinant protein expression in Escherichia coli: advances and challenges. Frontiers in Microbiology. 5, 172 (2014).
- Rosano, G. L., Morales, E. S., Ceccarelli, E. A. New tools for recombinant protein production in Escherichia coli: A 5-year update. Protein Science: A Publication of the Protein Society. 28 (8), 1412-1422 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon