A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בידוד וזיהוי חיידקים עמידים לאנטיביוטיקה במים ואפיון מולקולרי של גני העמידות לאנטיביוטיקה שלהם
In This Article
Summary
במאמר זה אנו מציגים פרוטוקול מפורט לבידוד וזיהוי של חיידקים עמידים לאנטיביוטיקה ממים ולאפיון המולקולרי של גני העמידות לאנטיביוטיקה שלהם (ARGs). השימוש בטכניקות מבוססות תרבית ולא מבוססות תרבית (ניתוח מטגנומי) מספק מידע מלא על מגוון החיידקים הכולל ועל המאגר הכולל של ARGs שונים הנמצאים במים מתוקים ממומבאי, הודו.
Abstract
הפיתוח וההתפשטות של עמידות לאנטיביוטיקה (AR) באמצעות מיקרוביוטה הקשורה לגופי מים מתוקים הוא דאגה בריאותית עולמית מרכזית. במחקר הנוכחי, דגימות מים מתוקים נאספו ונותחו ביחס למגוון החיידקים הכולל ולגנים של AR (ARGs) תוך שימוש הן בטכניקות קונבנציונליות מבוססות תרבית והן בגישה מטגנומית בלתי תלויה בתרבית בתפוקה גבוהה. מאמר זה מציג פרוטוקול שיטתי לספירה של החיידקים הכוללים והעמידים לאנטיביוטיקה מדגימות מים מתוקים ולקביעת עמידות פנוטיפית וגנוטיפית במבודדים הניתנים לתרבות. יתר על כן, אנו מדווחים על שימוש באנליזה מטא-גנומית שלמה של סך הדנ"א המטגנומי המופק מדגימת המים המתוקים לצורך זיהוי מגוון החיידקים הכולל, כולל חיידקים שאינם ניתנים לתרבות, וזיהוי המאגר הכולל של ARGs שונים (התנגדות) בגוף המים. בעקבות הפרוטוקולים המפורטים האלה, ראינו עומס חיידקים גבוה עמיד לאנטיביוטיקה בטווח של 9.6 × 10 5-1.2 × 109 CFU/mL. רוב המבודדים היו עמידים לאנטיביוטיקה הנפוצה שנבדקה, כולל cefotaxime, ampicillin, levofloxacin, chloramphenicol, ceftriaxone, gentamicin, neomycin, trimethoprim, ו ciprofloxacin, עם מספר אינדקסים של עמידות לאנטיביוטיקה (MAR) של ≥0.2, מה שמעיד על רמות גבוהות של עמידות בבידודים. ריצוף 16S rRNA זיהה פתוגנים אנושיים פוטנציאליים, כגון דלקת ריאות קלבסיאלה, וחיידקים אופורטוניסטיים, כגון Comamonas spp., Micrococcus spp., Arthrobacter spp., ו- Aeromonas spp. האפיון המולקולרי של המבודדים הראה נוכחות של ARGs שונים, כגון blaTEM, blaCTX-M (β-לקטאמים), aadA, aac (6')-Ib (אמינוגליקוזידים) ו-dfr1 (טרימתופרימים), אשר אושרה גם על ידי ניתוח הדנ"א המטגנומי כולו. שכיחות גבוהה של קידוד ARGs אחרים עבור משאבות שטף אנטיביוטיקה-mtrA, macB, mdtA, acrD, β-lactamases-SMB-1, VIM-20, ccrA, ampC, blaZ, הגן כלורמפניקול אצטילטרנספראז catB10, ואת גן עמידות rifampicin rphB-זוהה גם בדנ"א המטגנומי. בעזרת הפרוטוקולים שנדונו במחקר זה, אישרנו את נוכחותם של חיידקי MAR הנישאים במים עם תכונות פנוטיפיות וגנוטיפיות מגוונות של AR. לפיכך, ניתוח דנ"א מטגנומי שלם יכול לשמש כטכניקה משלימה לטכניקות קונבנציונליות המבוססות על תרביות כדי לקבוע את מצב ה-AR הכולל של גוף מים.
Introduction
עמידות מיקרוביאלית (AMR) זוהתה כאחת הבעיות העולמיות הדחופות ביותר. ההתפתחות המהירה של AMR והתפשטותו העולמית הם אחד האיומים הגדולים ביותר על בריאות האדם ועל הכלכלה העולמית במונחים של עלויות הבריאות הכרוכות בו1. שימוש יתר ושימוש לרעה באנטיביוטיקה הובילו לעלייה ב-AR. זה הודגש על ידי מגיפת COVID-19, שבמהלכה הטיפול בזיהומים משניים הקשורים, במקרים רבים, נפגע מאוד בגלל AMR בחולים שנפגעו2. מלבד השימוש הישיר/שימוש לרעה באנטיביוטיקה על ידי בני אדם, שימוש יתר ושימוש לרעה באנטיביוטיקה בחקלאות ובגידול בעלי חיים והזרמתן הבלתי הולמת לסביבה, כולל גופי מים, הם דאגה מרכזית3. עלייתן של תכונות עמידות חדשות ועמידות רב-ממדית בחיידקים מדגישה בדחיפות את הצורך בהבנה טובה יותר של הגורמים המובילים להתפתחות AR והפצתו. חיידקים עמידים לאנטיביוטיקה מרובים, שלעתים קרובות נושאים גנים מרובים של AR (ARGs) על אלמנטים גנטיים ניידים כגון פלסמידים, יכולים להעביר את גני העמידות האלה למיקרואורגניזמים שאינם עמידים, כולל פתוגנים אנושיים פוטנציאליים, ובכך להוביל להופעתם של חיידקי-על שאינם ניתנים לריפוי אפילו עם אנטיביוטיקה של מוצא אחרון4. החיידקים העמידים לאנטיביוטיקה המרובים האלה, אם הם נמצאים במערכות אקולוגיות של מים, יכולים להיכנס ישירות למעי האנושי באמצעות צריכת מזונות מזוהמים על בסיס מים, כגון דגים, סרטנים ורכיכות. מחקרים קודמים הראו כי התפשטות חיידקי AR במערכות מים טבעיות יכולה להגיע גם לאספקת מים אחרת, כולל מי שתייה, ובכך יכולה להיכנס לשרשרת המזון האנושית 5,6,7.
מטרת המחקר הנוכחי היא לספק פרוטוקול מקיף תוך שימוש בשילוב של טכניקות מבוססות תרבית ולא מבוססות תרבית (ניתוח מטגנומי שלם) כדי לקבל מידע מלא על מגוון החיידקים הכולל ועל המאגר הכולל של ARGs שונים הנמצאים בגוף מים במומבאי, הודו. באופן קונבנציונלי, נעשה שימוש בטכניקות מבוססות תרבית כדי לחקור את מגוון החיידקים בגופי מים. מכיוון שמיקרואורגניזמים תרבותיים מהווים רק אחוז קטן מכלל המיקרוביוטה בכל נישה, כדי להבין טוב יותר את המצב הכללי של מגוון החיידקים ואת תכונות העמידות השונות הנפוצות בכל דגימה, יש להשתמש בטכניקות שונות המבוססות על תרבית ובלתי תלויות בתרבית במקביל. טכניקה אחת חזקה ואמינה כזו, שאינה תלויה בתרבות, היא ניתוח דנ"א מטגנומי שלם. שיטה זו בעלת תפוקה גבוהה שימשה בהצלחה במחקרים שונים על מגוון חיידקים או על ביאורים פונקציונליים של ARGsשונים 8,9. טכניקה זו משתמשת במטגנום (החומר הגנטי הכולל בדגימה) כחומר המוצא לניתוחים שונים, ולכן היא בלתי תלויה בתרבית. הפרוטוקולים במחקר הנוכחי יכולים לשמש לניתוח דנ"א מטגנומי שלם כדי לקבל מידע על מגוון החיידקים הכולל ועל ARGs (התנגדות) שונים בדגימות מים.
Protocol
1. איסוף ועיבוד דגימות
- איסוף דגימות
- אסוף את הנפח המתאים של דגימת המים במיכלי דגימה סטריליים, וודא שלא יותר מ-3/4 מהמיכל ימולא.
- להעביר את הדגימות למעבדה בתנאים אספטיים בהקדם האפשרי לאחר האיסוף ומיד לעבד אותם.
- עיבוד דוגמאות
- מסננים באופן אספטי את דגימת המים דרך בד מוסלין סטרילי כדי להסיר כל חומר חלקיקי.
- לבצע דילולים סדרתיים מתאימים של המים המסוננים לניתוח נוסף.
2. הערכה של עומס החיידקים הכולל וספירת החיידקים העמידים לאנטיביוטיקה
- קביעת העומס החיידקי הכולל
- יש להשהות 18.12 גרם של R2A Agar, אבקה מעובדת ב-1,000 מ"ל של מים מזוקקים כפולים, ולהמיס את התערובת על ידי חימום. Autoclave את התערובת מומסת ב 121 °C, 15 psi במשך 20 דקות. הכן R2A Agar, צלחות מותאמות על ידי מזיגת הכמות המתאימה של תערובת autoclaved לתוך צלחות פטרי סטריליות (למשל, להוסיף כ 20 מ"ל של מדיום סטרילי autoclaved לצלחת פטרי סטרילית 90 מ"מ).
- מפזרים באופן שווה 100 μL של דילולים מתאימים של דגימת המים המסוננים על R2A Agar, צלחת שונה ברגע שהמדיום מתמצק. בצע את הניסוי בכפילות.
- דגירה של כל הלוחות הנ"ל ב 35-37 מעלות צלזיוס למשך 48 שעות (לשנות את הטמפרטורה ואת זמן הדגירה בהתאם למדיה המשמשת לבידוד).
- בטא את העומס החיידקי הכולל במונחים של יחידות יוצרות מושבה למיליליטר (CFU/mL) באמצעות משוואה (1):
 (1)
(1)
- קביעת ספירת חיידקי AR
- בצע את השלבים 2.1.1-2.1.4. עם זאת, במקום R2A Agar, צלחות מעובדות, השתמש R2A Agar, צלחות מותאמות בתוספת בנפרד עם חמש אנטיביוטיקות שונות, כלומר cefotaxime (3 מיקרוגרם / מ"ל), ciprofloxacin (0.5 מיקרוגרם / מ"ל), אריתרומיצין (20 מיקרוגרם / מ"ל), kanamycin (15 מיקרוגרם / מ"ל) ו vancomycin (3 מיקרוגרם / מ"ל).
- הוסף את האנטיביוטיקה בנפרד לתוך צינורות המכילים 20 מ"ל של R2A Agar מותך סטרילי, שונה (עם הטמפרטורה של R2A Agar מותך, שונה ב ≤40 מעלות צלזיוס) כדי להשיג את הריכוז האנטיביוטי הסופי כאמור בשלב 2.2.1.
- מערבלים לערבוב אחיד ויוצקים על צלחות פטרי סטריליות לפני שהאגר מתמצק. בצע את הניסוי בכפילות.
- דגירה של כל הלוחות הנ"ל ב 35-37 מעלות צלזיוס במשך 48 שעות (אם משתמשים במדיה אחרת, הטמפרטורה וזמן הדגירה עשויים להשתנות).
- לצורך בקרת איכות ובדיקת יעילות האנטיביוטיקה, יש למרוח 100 μL של תרחיפים חיידקיים של זני Escherichia coli ATCC 25922 ו-Staphylococcus aureus ATCC 29213 על גבי R2A Agar המכילים אנטיביוטיקה בהתאמה, צלחות מותאמות (ודא שצפיפות התרבית הטרייה המשמשת לחיסון היא OD = 0.5 ב-600 ננומטר).
- קבע את ספירת החיידקים העמידים לאנטיביוטיקה במונחים של CFU/mL כמתואר בשלב 2.1.4.
- מלאי גליצרול של המבודדים
- בחר מושבות AR מובחנות מבחינה מורפולוגית.
- להשעות מושבה בודדת בודדת ב-2 מ"ל של מרק לוריא-ברטני סטרילי המכיל את האנטיביוטיקה המתאימה (למשל, אם נבחרה מושבה מתוך צלחת המכילה cefotaxime, לחסן את המושבה משלב 2.3.1 במרק לוריא-ברטני סטרילי המכיל cefotaxime בריכוזו המתאים).
- לדגום את הצינורות המחוסנים ב 37 מעלות צלזיוס ב 80 סל"ד עד OD600 מגיע 0.5.
- הכינו מלאי גליצרול של המבודדים על ידי ערבוב 750 μL של מתלי התרבית משלב 2.3.3 ל-250 μL של 100% גליצרול סטרילי בתנאים אספטיים.
- יש לאחסן את מלאי הגליצרול בטמפרטורה של 80°C- עד לניתוח נוסף.
הערה: להחייאת התרביות ממניות הגליצרול, הפשירו את מלאי הגליצרול בטמפרטורה של 4 מעלות צלזיוס. לחסן לולאה של מלאי זה לתוך 2 מ"ל של מרק לוריא-ברטני סטרילי המכיל את האנטיביוטיקה המתאימה ולאפשר לגדול.
3. זיהוי חיידקים הניתנים להתרבות על ידי ריצוף גנים 16S rRNA
- הכנת תבנית דנ"א מהמבודדים ל-PCR
הערה: הפרוטוקול המתואר להכנת תבנית דנ"א עבור PCR לבידוד דנ"א גולמי מהחיידק ניתן על ידי Carlson et al.10.- בעזרת קיסם סטרילי, קחו מושבה אחת, מבודדת וטהורה של המבודד הגדל על צלחת פטרי. להשעות את מושבת החיידקים ב 100 μL של מים סטריליים מזוקקים פעמיים בצינור microcentrifuge סטרילי להרתיח במשך 10 דקות.
- צנטריפוגה את המתלים ב 10,000 × גרם למשך 2 דקות כדי לזרוק את הפסולת, ולהעביר את supernatant לצינור microcentrifuge סטרילי טרי לשימוש כתבנית DNA גולמי.
- הגברה ממוקדת של PCR של אזור V3 בגן 16S rRNA וריצוף
- הכן 40 μL של תערובת התגובה בצינור PCR להגברת PCR, כאמור בטבלה 1.
הערה: הכנת הדנ"א צריכה להתבצע על גוש קרח תוך מזעור הסיכוי לזיהום (ללבוש כפפות בעת הטיפול בריאגנטים, ולנקות את משטח העבודה ביסודיות עם 70% אתנול). - הנח את הצינור בבלוק התרמי, והפעל את התוכנית המתאימה במחזור התרמי PCR. ראו טבלה 2 עבור תנאי מחזור ה-PCR המתוקננים ומידע הפריימר להגברה של אזורי V3 של הגנים 16S rRNA.
- לפתרון אמפליקונים והדמיה, לבצע אלקטרופורזה ג'ל agarose (AGE). יש לערבב 10 μL של מוצר ה-PCR המוגבר ו-2 μL של מאגר העמסת ג'ל 6x (טבלה 3), ולהעמיס תערובת זו לתוך בארות על 1.5% ג'ל אגרוז (להמיס 1.5 גרם של אבקת אגרוז ב-100 מ"ל של 1x TAE buffer [טבלה 3]) המכיל 5 μL של 10 מ"ג/מ"ל אתידיום ברומיד (EtBr) לריכוז סופי של 0.5 מיקרוגרם/מ"ל EtBr ב-100 מ"ל של ג'ל האגרוז.
אזהרה: EtBr הוא חומר מסרטן רב עוצמה. יש ללבוש את הכפפות בכל עת בעת הטיפול ב- EtBr ובג'לים המכילים EtBr. - הוסף סולם DNA להערכת גודל האמפליקונים.
- בצע אלקטרופורזה של הג'ל בחיץ טנק TAE ב 80-100 V.
- לאחר שצבע המעקב פועל 3/4 מהג'ל, עצרו את האלקטרופורזה, ודמיינו את רצועות האמפליקון מתחת לטרנסילומינטור UV.
- השתמש במוצר PCR (amplicon) לריצוף גנים 16S rRNA כדי לזהות את המבודד.
- כימות האמפליקון על ידי הכפפתו לאנליזה ספקטרופוטומטרית באמצעות משוואה (2).
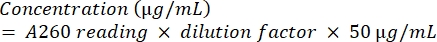 (2)
(2) - כדי לבדוק את טוהר הדנ"א, חשב את היחס בין A260/A280.
הערה: באופן אידיאלי, מספר זה צריך להיות מעל 1.5, ועדיף, בין 1.8 ל 2.0. - כדי לזהות את המבודדים, השווה את הרצפים המתקבלים עם מסדי נתונים של רצפים באמצעות כלי חיפוש יישור מתאים.
- הכן 40 μL של תערובת התגובה בצינור PCR להגברת PCR, כאמור בטבלה 1.
4. איתור עמידות לאנטיביוטיקה במבודדים באמצעות בדיקת רגישות לאנטיביוטיקה
הערה: פרוטוקול זה מתאר את השיטה לבדיקת רגישות לאנטיביוטיקה (AST) על ידי דיפוזיה של דיסק. נעשה שימוש בדיסקי האנטיביוטיקה הבאים: cefotaxime (5 מיקרוגרם), אמפיצילין (10 מיקרוגרם), לבופלוקסצין (5 מיקרוגרם), כלוראמפניקול (30 מיקרוגרם), טיגציקלין (15 מיקרוגרם), צפטריקסון (30 מיקרוגרם), אימיפנם (10 מיקרוגרם), גנטמיצין (10 מיקרוגרם), נאומיצין (10 מיקרוגרם), טרימתופרים (5 מיקרוגרם) וציפרופלוקסצין (5 מיקרוגרם).
- הכנת האינוקולום ל- AST
- לחסן באופן אספטי מושבת AR יחידה, מבודדת ומטוהרת באמצעות לולאה סטרילית ב-2 מ"ל של מדיום סטרילי לא סלקטיבי, כגון מרק לוריא-ברטני (ללא כל אנטיביוטיקה), ולדגור בטמפרטורה של 37 מעלות צלזיוס ב-80 סל"ד למשך הלילה.
- יש להשהות את השימוש ב-100-150 מיקרוליטר של התרבית שגדלה בן לילה (בערך, OD 600 = 1.8-2.0) ב-2 מ"ל של מרק לוריא-ברטני טרי ולא סלקטיבי ולדגור במשך 2-4 שעות (עד שה-OD600 מגיע ל-0.4-0.5).
- לדלל את מתלה התרבית הטרי הזה באמצעות תמיסת מלח סטרילית של 0.85% כך שצפיפות התרבית שווה לתקן מקפרלנד 0.5 (בערך, OD600 = 0.1), אשר מתאים בערך ל 1-2 × 108 תאים / מ"ל.
- ערבבו בעדינות את תרחיף החיידקים לחלוקת תאים אחידה.
- השתמש במתלה הנ"ל תוך 15 דקות מרגע הדילול.
- חיסון צלחות האגר
- הכינו צלחות מולר-הינטון אגר (MHA) לביצוע ה-AST על ידי ערבוב 38 גרם של MHA ב-1,000 מ"ל של מים מזוקקים כפולים, והמיסו את התערובת על ידי חימום. Autoclave את התערובת מומסת ב 121 מעלות צלזיוס, 15 psi במשך 15 דקות.
- ודא כי עומק MHA בצלחות הוא 4 מ"מ (25 מ"ל של בינוני לכל צלחת).
- במקביל, מוציאים את דיסקיות האנטיביוטיקה מהמקפיא ומחממות אותן לטמפרטורת החדר.
הערה: יש להפשיר בהדרגה את הדיסקים האנטיביוטיים על ידי הפשרה ראשונית של הדיסקים בטמפרטורה של 4 מעלות צלזיוס ומאוחר יותר בטמפרטורת החדר כדי להפחית כל סכנה פוטנציאלית של עיבוי על הדיסקים, אשר עלולה להשפיע לאחר מכן על אזור העיכוב (ZOI). - בתנאים אספטיים, טבלו צמר גפן סטרילי לתוך האינוקולום שהוכן בשלב 4.1, והסירו את עודפי ההשעיה כדי למנוע חיסון יתר של הצלחות.
- פזרו את התרבית באופן שווה על הצלחות, החל מהחלק העליון של צלחת ה-MHA והמשיכו קדימה ואחורה מקצה לקצה. סובבו את הצלחת ב-60° תוך כדי התנדנדות.
- יישום הדיסקים האנטיביוטיים
- בעזרת מלקחיים מעוקרים בלהבה, מעבירים באופן אספטי את הדיסקים האנטיביוטיים על לוחות ה-MHA המחוסנים, ולוחצים בעדינות על הדיסקים כדי להבטיח מגע מלא עם האגר.
הערה: הליך זה צריך להיעשות תוך 15 דקות מחיסון התרבית על הצלחות. - מקם את המספר המתאים של דיסקים אנטיביוטיים על צלחת אגר על ידי התחשבות האורגניזם, האנטיביוטיקה בשימוש, ואת גודל הצלחת כדי למנוע חפיפה של אזורי העיכוב.
הערה: ניתן לאכלס ארבעה עד חמישה דיסקים על לוח עגול בקוטר 90 מ"מ.
- בעזרת מלקחיים מעוקרים בלהבה, מעבירים באופן אספטי את הדיסקים האנטיביוטיים על לוחות ה-MHA המחוסנים, ולוחצים בעדינות על הדיסקים כדי להבטיח מגע מלא עם האגר.
- דגירה של הצלחות
- תוך 15 דקות מיישום הדיסקים האנטיביוטיים, הופכים את הצלחות ודוגרים בטמפרטורה של 37 מעלות צלזיוס למשך הלילה.
- פרשנות התוצאות
- מודדים את קוטר ה-ZOI במילימטרים (מ"מ), ומפרשים לפי ערכי נקודת השבירה שניתנו על ידי EUCAST11. ראה את שתי הדוגמאות המובאות להלן.
- נקודת השבירה בקוטר האזור (מ"מ) עבור דיסק אנטיביוטיקה ציפרופלוקסצין (5 מיקרוגרם) עבור Enterobacterales היא S ≥ 25 ו- R < 22, כלומר היא נחשבת רגישה (S) אם ZOI ≥ 25 מ"מ, בעוד שהיא עמידה (R) אם ZOI < 22 מ"מ. אם קוטר ה-ZOI נופל בין 22 ל-25, המבודד נחשב לבינוני (I).
- נקודת השבירה בקוטר האזור (מ"מ) עבור דיסק אנטיביוטי כלורמפניקול (30 מיקרוגרם) עבור סטפילוקוקוס spp. היא S ≥ 18 ו- R < 18, כלומר היא נחשבת רגישה אם ZOI ≥ 18 מ"מ, בעוד שהיא עמידה אם ZOI < 18 מ"מ.
- קבע את מדד עמידות לאנטיביוטיקה מרובה (MAR) על ידי מציאת היחס בין מספר האנטיביוטיקה שאליה עמיד המבודד למספר הכולל של האנטיביוטיקה שאליה נחשף המבודד.
הערה: עבור בקרת איכות, E. coli ATCC 25922 ו - S. אוראוס ATCC 29213 משמשים כזני ייחוס בהתאם לפרוטוקול כמו בשלב 4.
- מודדים את קוטר ה-ZOI במילימטרים (מ"מ), ומפרשים לפי ערכי נקודת השבירה שניתנו על ידי EUCAST11. ראה את שתי הדוגמאות המובאות להלן.
5. זיהוי מבוסס PCR של גנים עמידים לאנטיביוטיקה במבודדים
- השתמש בפרוטוקול PCR סטנדרטי לזיהוי ARGs במבודדים. הכן את תבנית הדנ"א באמצעות הפרוטוקול שניתן בשלב 3.1.
הערה: תנאי מחזור ה-PCR ששימשו במחקר זה היו 94 °C למשך 10 דקות, ולאחר מכן 35 מחזורים של 94 °C למשך 30 שניות, חישול במשך 30 שניות בטמפרטורה המתאימה (כפי שנקבע עבור כל ערכת פריימר), הארכה ב-72 °C למשך 40 שניות, והארכה סופית ב-72 °C למשך 5 דקות. תערובת התגובה מתוארת בטבלה 4. רשימת ה-ARGs, הפריימרים וטמפרטורות החישול מופיעה בטבלה 5. - כדי לפתור, לדמיין ולבדוק את טוהר האמפליקונים, בצע את השלבים 3.2.3-3.2.10.
6. ניתוח דנ"א מטגנומי שלם לזיהוי מגוון החיידקים הכולל וזיהוי ARGs במטגנום
- מיצוי הדנ"א הכולל (מטגנום) מדגימת המים
- חלצו את הדנ"א המטגנומי מדגימות המים המסוננים.
הערה: במחקר הנוכחי, הדנ"א המטגנומי (סך הדנ"א) הופק מדגימות המים המסוננים באמצעות ערכת בידוד הדנ"א המוזכרת בהתאם לפרוטוקול היצרן (ראה טבלת החומרים). - בדוק את איכות הדנ"א על ידי טעינת 3 μL של הדנ"א המטגנומי המופק על ג'ל אגרוז של 0.8%, והפעל את הג'ל ב-80-110 וולט למשך כ-30 דקות.
- בדוק את נוכחותה של רצועה שלמה אחת.
- בדוק את ריכוז הדנ"א באמצעות פלואורומטר.
- חלצו את הדנ"א המטגנומי מדגימות המים המסוננים.
- קביעת מגוון חיידקים וזיהוי ARGs באמצעות ריצוף DNA מטאגנומי שלם
- הכנת ספרייה והגברת PCR:
- הכינו ספריית ריצוף צמודה באמצעות ערכת ההכנה של ספריית הדנ"א שאליה יש הפניה (ראו טבלת חומרים).
- הכן את הדנ"א לקשירת מתאם על ידי לקיחת 200 ננוגרם של דנ"א והטיה מכנית שלו למקטעים קטנים יותר, ולאחר מכן שלב רציף של תיקון קצה שבו "A" מתווסף לקצוות 3 '.
- בהתאם לפלטפורמה המשמשת לריצוף, ליגייט מתאמים ספציפיים לשני הקצוות של מקטעי הדנ"א.
הערה: רצפים חיוניים לקשירת ספריות עם ברקוד כפול לתא זרימה לצורך ריצוף נמצאים במתאמים אלה. זה מאפשר הגברה PCR של שברים קשירה מתאם וקשירת פריימרים ריצוף סטנדרטיים. - כדי לבדוק את האיכות והכמות, נתחו את הספרייה המוגברת באמצעות שבב DNA בעל רגישות גבוהה בהתאם להוראות היצרן.
- יצירת אשכולות וריצוף:
- טען את הספרייה המוגברת על פלטפורמת הריצוף המתאימה ליצירת אשכולות ולריצוף שלאחר מכן.
הערה: מולקולות הספרייה נקשרות לאוליגוס המתאם המשלים בתא הזרימה הזוגי. במהלך הרצף, הגדילים הקדמיים נבקעים באופן סלקטיבי לאחר סינתזה מחדש של הגדיל ההפוך. גדיל הפוך מועתק זה מרוצף מהקצה הנגדי של הקטע.
- טען את הספרייה המוגברת על פלטפורמת הריצוף המתאימה ליצירת אשכולות ולריצוף שלאחר מכן.
- ניתוח ביואינפורמטי:
- צור פיגומים מהנתונים האיכותיים באמצעות הפלטפורמה המתאימה.
- הכפיף פיגומים אלה לניתוח ביואינפורמטיקה לצורך סיווג טקסונומי וזיהוי של ה- ARGs.
הערה: תהליך העבודה של כל ניתוח הדנ"א המטגנומי לזיהוי המגוון הכולל של החיידקים ולזיהוי ה-ARGs במטגנום ניתן באיור 1. גיליון זרימה של המתודולוגיה המלאה המתוארת בכתב היד מובא באיור 2.
- הכנת ספרייה והגברת PCR:
תוצאות
סך כל עומס החיידקים וספירת החיידקים העמידים לאנטיביוטיקה (AR)
הספירה של עומס החיידקים הכולל בוצעה על ידי פיזור 10−4 עד 10−6 דילולים של דגימות המים על R2A Agar, מדיום שונה. לצורך הספירה של ספירת חיידקי ה-AR, 10−3 עד 10−6 דילולים של פי 6 הופצו על לוחות מדיה המכילים אנטי...
Discussion
איסוף הדגימות ועיבודן ממלאים תפקיד משמעותי ועשויים להשפיע על התוצאות והפרשנות של המחקר. לפיכך, כדי לשלול שונות בדגימות, חשוב לבצע דיגום במספר מוקדים של גוף המים המתוקים הנחקרים. שמירה על תנאי סביבה אספטיים נאותים בעת טיפול בדגימות כאלה יכולה למנוע זיהום. יתר על כן, כדי למנוע שינויים בהרכב ...
Disclosures
למחברים אין אינטרסים מנוגדים לחשוף.
Acknowledgements
עבודה זו נתמכה חלקית על ידי מענקים כספיים מהמחלקה למדע וטכנולוגיה - קידום מחקר אוניברסיטאי ומצוינות מדעית (DST-PURSE) של אוניברסיטת מומבאי. דוויקה גדיגאונקר עבדה כעמיתת פרויקט במסגרת התוכנית. העזרה הטכנית שניתנה על ידי Harshali Shinde, עמית מחקר בכיר תחת המחלקה למדע וטכנולוגיה-מדע והנדסה מחקר המועצה למחקר (DST-SERB) פרויקט מספר: CRG/2018/003624, היא מוכרת.
Materials
| Name | Company | Catalog Number | Comments |
| 100 bp DNA ladder | Himedia | MBT049-50LN | For estimation of size of the amplicons |
| 2x PCR Taq mastermix | HiMedia | MBT061-50R | For making PCR reaction mixture |
| 37 °C Incubator | GS-192, Gayatri Scientific | NA | For incubation of bacteria |
| 6x Gel Loading Buffer | HiMedia | ML015-1ML | Loading and Tracking dye which helps to weigh down the DNA sample and track the progress of electrophoresis |
| Agarose powder | Himedia | MB229-50G | For resolving amplicons during Agarose Gel Electrophoresis (AGE) |
| Ampicillin antibiotic disc | HiMedia | SD002 | For performing AST |
| Autoclave | Equitron | NA | Required for sterilization of media, glass plates, test tubes, etc |
| Bioanalyzer 2100 | Agilent Technologies | NA | To check the quality and quantity of the amplified library |
| Bisafety B2 Cabinet | IMSET | IMSET BSC-Class II Type B2 | Used for microbiological work like bacterial culturing, AST etc. |
| Cefotaxime antibiotic disc | HiMedia | SD295E-5VL | For performing AST |
| Cefotaxime antibiotic powder | HiMedia | TC352-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Ceftriaxone antibiotic disc | HiMedia | SD065 | For performing AST |
| Centrifuge Minispin | Eppendorf | Minispin Plus-5453 | Used to pellet the debris during crude DNA preparation |
| Chloramphenicol antibiotic disc | HiMedia | SD006-5x50DS | For performing AST |
| Ciprofloxacin antibiotic disc | HiMedia | SD060-5x50DS | For performing AST |
| Ciprofloxacin antibiotic powder | HiMedia | TC447-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Colorimeter | Quest | NA | For checking the OD of culture suspensions |
| Comprehensive Antibiotic Resistance Database (CARD) database | functional annotation of ARGs; https://card.mcmaster.ca/ | ||
| Cooling Shaker Incubator | BTL41 Allied Scientific | NA | For incubation of media plates for culturing bacteria |
| Deep Freezer (-40 °C) | Haier | DW40L, Haier Biomedicals | For storage of glycerol stocks |
| DNA Library Prep Kit | NEB Next Ultra DNA Library Prep Kit for Illumina | NA | Paired-end sequencing library preparation |
| EDTA | HiMedia | GRM1195-100G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Electrophoresis Apparatus | TechResource | 15 cm gel casting tray | For making the agarose gel and carrying out electrophoresis |
| Electrophoresis Power pack with electrodes | Genei | NA | For running the AGE |
| Erythromycin antibiotic disc | HiMedia | SD222-5VL | For performing AST |
| Erythromycin antibiotic powder | HiMedia | CMS528-1G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Erythromycin antibiotic powder | HiMedia | TC024-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Escherichia coli ATCC 25922 | HiMedia | 0335X-1 | Used as a control while performing AST |
| Ethidium Bromide | HiMedia | MB071-1G | Intercalating agent and visualizaion of DNA after electrophoresis under Gel Documentation System |
| Fluorometer | Qubit 2.0 | NA | For determining concentration of extracted metagenomic DNA |
| Gel Documentation System | BioRad | Used for visualizing PCR amplicons after electrophoresis | |
| Gentamicin antibiotic disc | HiMedia | SD170-5x50DS | For performing AST |
| Glacial Acetic Acid | HiMedia | AS119-500ML | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Glycerol | HiMedia | GRM1027-500ML | For making glycerol stocks |
| Imipenem antibiotic disc | HiMedia | SD073 | For performing AST |
| Kaiju Database | NA | NA | For taxonomical classification of reads; https://kaiju.binf.ku.dk/ |
| Kanamycin antibiotic disc | HiMedia | SD017-5x50DS | For performing AST |
| Kanamycin antibiotic powder | HiMedia | MB105-5G | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Levofloxacin antibiotic disc | HiMedia | SD216-5VL | For performing AST |
| Luria Bertani broth | Himedia | M1245-500G | For enrichment of cultures |
| McFarland Standards | Himedia | R092-1No | To compare density of culture suspension |
| Molecular Biology water | HiMedia | TCL018-500ML | For making PCR reaction mixture |
| Mueller-Hinton Agar (MHA) | HiMedia | M173-500G | For performing Antibiotc Susceptibility Testing (AST) |
| Neomycin antibiotic disc | HiMedia | SD731-5x50DS | For performing AST |
| PCR Gradient Thermal Cycler | Eppendorf | Mastercycler Nexus Gradient 230V/50-60 Hz | Used for performing PCR for amplification of 16S rRNA region and various Antibiotic Resistance genes |
| Primers | Xcelris | NA | For PCR amplication |
| R2A Agar, Modified | HiMedia | M1743 | For preparation of media plates for isolation of total and antibiotic resistant (AR) bacterial load |
| Scaffold generation | CLC Genomics Workbench 6.0 | NA | For generation of scaffolds |
| Sequencer | Illumina platform (2 x 150 bp chemistry) | NA | Sequencing of amplified library |
| Sodium Chloride | HiMedia | TC046-500G | For preparation of 0.85% saline for serially diluting the water sample |
| Soil DNA isolation Kit | Xcelgen | NA | For extraction of whole metagenomic DNA from the filtered water sample |
| Staphylococcus aureus subsp. aureus ATCC 29213 | HiMedia | 0365P | Used as a control while performing AST |
| Taxonomical Classification | Kaiju ioinformatics tool | NA | For classification of reads into different taxonomic groups from phylum to genus level |
| The Comprehensive Antibiotic Resistance Database (CARD) | NA | NA | For functional annotation of ARGs |
| Tigecycline antibiotic disc | HiMedia | SD278 | For performing AST |
| Trimethoprim antibiotic disc | HiMedia | SD039-5x50DS | For performing AST |
| Tris base | HiMedia | TC072-500G | For preparation of Gel running buffer for Agarose Gel Electrophoresis (AGE) |
| Vancomycin antibiotic powder | HiMedia | CMS217 | For preparation of antibiotic stock solution required during isolation of antibiotic resistant bacteria |
| Weighing Balance | Mettler Toledo | ME204 Mettler Toledo | Used for weighing media powders, reagent powders etc. |
| NA - Not Applicable |
References
- Prestinaci, F., Pezzotti, P., Pantosti, A. Antimicrobial resistance: A global multifaceted phenomenon. Pathogens and Global Health. 109 (7), 309-318 (2015).
- Knight, G., et al. Antimicrobial resistance and COVID-19: Intersections and implications. Elife. 10, 64139 (2021).
- Ventola, C. L. The antibiotic resistance crisis: Part 1: Causes and threats. Pharmacy and Therapeutics. 40 (4), 277-283 (2015).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J. R., Rath, A. Metagenomic analysis of total microbial diversity and antibiotic resistance of culturable microorganisms in raw chicken meat and mung sprouts (Phaseolus aureus) sold in retail markets of Mumbai. India. Current Science. 113 (1), 71-79 (2017).
- Naik, O. A., Shashidhar, R., Rath, D., Bandekar, J., Rath, A. Characterization of multiple antibiotic resistance of culturable microorganisms and metagenomic analysis of total microbial diversity of marine fish sold in retail shops in Mumbai, India. Environmental Science and Pollution Research. 25 (7), 6228-6239 (2018).
- Czekalski, N., GascónDíez, E., Bürgmann, H. Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake. The ISME Journal. 8 (7), 1381-1390 (2014).
- Kraemer, S., Ramachandran, A., Perron, G. Antibiotic pollution in the environment: From microbial ecology to public policy. Microorganisms. 7 (6), 180 (2019).
- Edmonds-Wilson, S., Nurinova, N., Zapka, C., Fierer, N., Wilson, M. Review of human hand microbiome research. Journal of Dermatological Science. 80 (1), 3-12 (2015).
- de Abreu, V., Perdigão, J., Almeida, S. Metagenomic approaches to analyze antimicrobial resistance: An overview. Frontiers in Genetics. 11, 575592 (2021).
- Carlson, S., et al. Detection of multiresistant Salmonella typhimurium DT104 using multiplex and fluorogenic PCR. Molecular and Cellular Probes. 13 (3), 213-222 (1999).
- Breakpoint tables for interpretation of MICs and zone diameters, Version 12.0. European Committee on Antimicrobial Susceptibility Testing Available from: https://www.eucast.org/fileadmin/src/media/PDFs/EUCAST_files/Breakpoint_tables/v_12.0_Breakpoint_Tables.pdf (2022)
- Bharti, R., Grimm, D. Current challenges and best-practice protocols for microbiome analysis. Briefings in Bioinformatics. 22 (1), 178-193 (2019).
- Choo, J., Leong, L., Rogers, G. Sample storage conditions significantly influence faecal microbiome profiles. Scientific Reports. 5, 16350 (2015).
- Clinical and Laboratory Standards Institute. . Methods for Dilution Antimicrobial Susceptibility Tests for Bacteria that Grow Aerobically, 11th edition. , (2015).
- Bayot, M., Bragg, B. Antimicrobial Susceptibility Testing. StatPearls. , (2021).
- Joseph, A. A., Odimayo, M. S., Olokoba, L. B., Olokoba, A. B., Popoola, G. O. Multiple antibiotic resistance index of Escherichia coli isolates in a tertiary hospital in South-West Nigeria. Medical Journal of Zambia. 44 (4), 225-232 (2017).
- Lorenz, T. Polymerase chain reaction: Basic protocol plus troubleshooting and optimization strategies. Journal of Visualized Experiments. (63), e3998 (2012).
- Rolin, J. Food and human gut as reservoirs of transferable antibiotic resistance encoding genes. Frontiers in Microbiology. 4, 173 (2013).
- Racewicz, P., et al. Prevalence and characterisation of antimicrobial resistance genes and class 1 and 2 integrons in multiresistant Escherichia coli isolated from poultry production. Scientific Reports. 12, 6062 (2022).
- Gebreyes, W., Thakur, S. Multidrug-resistant Salmonella enterica serovar Muenchen from pigs and humans and potential interserovar transfer of antimicrobial resistance. Antimicrobial Agents and Chemotherapy. 49 (2), 503-511 (2005).
- Li, L., et al. Prevalence and characteristics of extended-spectrum β-lactamase and plasmid-mediated fluoroquinolone resistance genes in Escherichia coli isolated from chickens in Anhui Province, China. PLoS One. 9 (8), 104356 (2014).
- Akers, K., et al. Aminoglycoside resistance and susceptibility testing errors in Acinetobacter baumannii-calcoaceticus complex. Journal Of Clinical Microbiology. 48 (4), 1132-1138 (2010).
- Ciesielczuk, H. . Extra-intestinal pathogenic Escherichia coli in the UK: The importance in bacteraemia versus urinary tract infection, colonisation of widespread clones and specific virulence factors. , (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved