É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Microengenharia 3D Colágeno Hidrogéis com Alinhamento de Fibras de Longo Alcance
Neste Artigo
Resumo
Este protocolo demonstra o uso de um canal microfluídico com geometria variável ao longo da direção do fluxo de fluido para gerar deformação extensional (alongamento) para alinhar as fibras em um hidrogel de colágeno 3D (<250 μm de espessura). O alinhamento resultante se estende por vários milímetros e é influenciado pela taxa de deformação extensional.
Resumo
As fibras alinhadas de colágeno I (COL1) guiam a motilidade das células tumorais, influenciam a morfologia das células endoteliais, controlam a diferenciação das células-tronco e são uma marca registrada dos tecidos cardíaco e musculoesquelético. Para estudar a resposta celular a microambientes alinhados in vitro, vários protocolos foram desenvolvidos para gerar matrizes de COL1 com alinhamento de fibras definido, incluindo métodos magnéticos, mecânicos, baseados em células e microfluídicos. Destas, as abordagens microfluídicas oferecem recursos avançados, como controle preciso sobre os fluxos de fluidos e o microambiente celular. No entanto, as abordagens microfluídicas para gerar matrizes alinhadas de COL1 para plataformas avançadas de cultura in vitro têm sido limitadas a "esteiras" finas (<40 μm de espessura) de fibras de COL1 que se estendem por distâncias inferiores a 500 μm e não são propícias a aplicações de cultura celular 3D. Aqui, apresentamos um protocolo para fabricar matrizes 3D de COL1 (130-250 μm de espessura) com regiões de alinhamento de fibras definidas em escala milimétrica em um dispositivo microfluídico. Esta plataforma fornece recursos avançados de cultura de células para modelar microambientes de tecidos estruturados, fornecendo acesso direto à matriz micro-projetada para cultura de células.
Introdução
As células residem em uma complexa rede fibrosa 3D denominada matriz extracelular (MEC), cuja maior parte é composta pela proteína estrutural colágeno tipo I (COL1)1,2. As propriedades biofísicas da MEC fornecem pistas de orientação às células e, em resposta, as células remodelam a microarquitetura da MEC 3,4,5. Essas interações recíprocas célula-matriz podem dar origem a domínios alinhados das fibras COL16 que promovem angiogênese e invasão celular no ambiente tumoral 7,8,9 e influenciam a morfologia celular 10,11,12, polarização13 e diferenciação 14. As fibras colágenas alinhadas também promovem a cicatrização deferidas15, desempenham papel fundamental no desenvolvimento tecidual 16 e contribuem para a comunicação celular de longo alcance17,18. Portanto, replicar a microarquitetura da fibra COL1 nativa in vitro é um passo importante para o desenvolvimento de modelos estruturados para estudar as respostas celulares a microambientes alinhados.
Sistemas de cultura de células microfluídicas têm se estabelecido como tecnologia preferencial para o desenvolvimento de sistemas microfisiológicos (MPS)19,20,21,22,23. Aproveitando os efeitos favoráveis de escala em microescala, esses sistemas fornecem controle preciso sobre os fluxos de fluidos, suportam a introdução controlada de forças mecânicas e definem o microambiente bioquímico dentro de um microcanal 21,24,25,26,27. As plataformas MPS têm sido utilizadas para modelar microambientes tecido-específicos e estudar interações multi-órgãos28. Simultaneamente, hidrogéis têm sido amplamente explorados para recapitular a mecânica 3D e a influência biológica da MEC que são observadas in vivo29,30. Com uma ênfase crescente na integração da cultura 3D com plataformas microfluídicas, inúmeras abordagens podem combinar hidrogéis de COL1 em dispositivos microfluídicos31,32,33. No entanto, os métodos para alinhar hidrogéis de COL1 em canais microfluídicos têm sido limitados a "esteiras" 2D finas (<40 μm de espessura) em canais <1 mm de largura, oferecendo potencial limitado para modelar respostas celulares em microambientes 3D alinhados31,34,35,36.
Para obter hidrogéis 3D de COL1 alinhados em um sistema microfluídico, foi demonstrado que, quando uma solução de COL1 automontável é exposta a fluxos extensionais locais (mudança de velocidade ao longo da direção do fluxo), os hidrogéis COL1 resultantes exibem um grau de alinhamento da fibra que é diretamente proporcional à magnitude da taxa de deformação extensional que experimentam37, 38º. O design de microcanais neste protocolo é único de duas maneiras; primeiro, o projeto segmentado introduz a tensão extensional local para a solução COL1 e, segundo, sua construção de "duas peças" permite que o usuário alinhe as fibras COL1 e, em seguida, desmonte o canal para acessar diretamente as fibras alinhadas em um formato aberto. Esta abordagem pode ainda ser adotada para o desenvolvimento de plataformas microfluídicas modulares que desenvolvam sistemas microfisiológicos com matrizes de COL1 ordenadas. O protocolo a seguir descreve o processo de fabricação de microcanais segmentados e detalha o uso dos canais para alinhar o ATEL bovino COL1. Este protocolo também fornece instruções para o cultivo de células em COL1 em um formato de poço aberto e discute a adição de funcionalidade à plataforma usando uma camada de base magnética modular.
Protocolo
1. Fabricação do canal de duas peças e da base da plataforma modular
NOTA: O canal microfluídico é construído usando duas partes — o "recorte" do canal microfluídico, que é cortado a partir de uma folha de polidimetil siloxano (PDMS) de espessura definida, e a tampa do canal, que se liga reversivelmente ao recorte e forma o canal. O canal é circundado por uma estrutura de poli(metacrilato de metila) (PMMA) que atuará como um reservatório de meio (Figura 1). O quadro de PMMA também pode ser usado para travar magneticamente módulos especializados para maior funcionalidade.
- Canais de corte de navalha de folhas PDMS:
NOTA: O projeto do canal microfluídico para esta etapa é fornecido na Figura Suplementar 1. O canal é composto por cinco segmentos de comprimento de 5 mm cada e larguras de 10 mm, 5 mm, 2,5 mm, 1,25 mm e 0,75 mm. A velocidade da solução de colágeno, injetada a uma taxa de fluxo constante (50-400 μL·min−1), aumenta localmente ao longo do canal à medida que a largura do canal é diminuída para gerar fluxo extensional.- Monte uma folha de PDMS de 250 μm de espessura em uma folha transportadora de plástico e corte o design microfluídico usando um cortador artesanal a uma profundidade de lâmina de 0,5 mm, velocidade de 1 cm·s−1 e alta força.
- Usando um banho ultrassônico, limpe os recortes do canal microfluídico por 5 min. Enxaguar os canais sonicados, em água deionizada (DI), e secá-los em uma placa de aquecimento limpa a 100 °C por 5 min.
- Guarde os canais em uma placa de Petri limpa e coberta até o uso.
- Fabricação da tampa PDMS e passivação superficial:
NOTA: O recorte de canal da seção 1.1 requer uma tampa que possa ser colocada sobre ela para criar um canal fechado no qual uma solução de COL1 possa ser injetada (Figura 1 suplementar). A tampa pode ser removida após o COL1 ter se auto-montado. Para minimizar as chances de ligação do COL1 no canal à tampa, a cobertura é passivada com albumina de soro bovino (BSA). O projeto do molde para a tampa do PDMS é fornecido. O molde deve ter pelo menos 2 mm de espessura e deve ter uma pegada igual à pegada do recorte do canal microfluídico. Nesse protocolo, a pegada do molde foi de 35 mm x 15 mm.- Para preparar o molde, afixe uma folha de adesivo sensível à pressão (PSA) em uma folha de PMMA de 2,5 mm de espessura e corte a laser na forma desejada do molde.
- Limpe o molde de PMMA cortado a laser com um lenço umedecido e sem fiapos e remova o suporte da película PSA. Fixe o molde a um wafer de silicone de 100 mm de diâmetro pressionando firmemente.
- Prepare o PDMS na proporção de 10:1 (base:crosslinker). Misture vigorosamente por 1 min para garantir a mistura adequada e desgaseifique a mistura em uma câmara de vácuo para remover bolhas de ar.
- Despeje a solução de PDMS desgaseificado no molde de PMMA/silício e cure em uma placa de aquecimento a 100 °C por 20 min. Deixe o molde esfriar, remova o PDMS curado e corte qualquer excesso com uma lâmina de barbear.
NOTA: Certifique-se de que os lados voltados para o wafer de silicone (lados planos) das tampas do PDMS estejam voltados para cima ao longo de todas as etapas a seguir. - Use um punção de biópsia de 1 mm de diâmetro para criar orifícios de entrada e saída que correspondam às extremidades do canal microfluídico. O recorte do canal do passo 1.1 pode ser usado como um molde para orientar a posição dos furos.
- Sonicar a tampa em isopropanol (IPA) por 5 min, enxaguar com água DI, secar com uma fonte de ar comprimido e, em seguida, armazenar em uma placa de Petri limpa e coberta. Coloque a placa de Petri em uma câmara de esterilização UV (descoberta) por 1 min para esterilizar.
- Cobertura e transferência para um gabinete de biossegurança (BSC).
- Pipetar 300 μL de 40 mg•mL-1 de albumina de soro bovino (BSA) em solução salina tamponada com fosfato (PBS) 1x nas tampas do PDMS e espalhar a solução uniformemente usando uma ponta de pipeta. Colocar no frigorífico a 4 °C durante, pelo menos, 4 h antes da utilização.
- Mova as tampas da geladeira para um BSC, lave 5x com 1x PBS e deixe-as secar ao ar por 10 min.
NOTA: As tampas PDMS podem ser armazenadas na geladeira por até 1 semana após o revestimento com BSA.
- Tratamento com glutaraldeído de lamínulas
NOTA: Funcionalizar as lamínulas com glutaraldeído liga covalentemente COL1 à lamínula e evita que o hidrogel se desprenda.- Preparar uma solução de aminopropil trietoxisilano (APTES) a 2% (v/v) num copo de vidro adicionando 1 ml de APTES a 49 ml de acetona.
- Diluir uma solução de glutaraldeído a 25% a 5% em água DI. Completar 2 ml de solução por cada lamínula de 24 mm x 50 mm. Para lamínulas de 24 mm x 24 mm ou 22 mm x 22 mm, fazer 1 ml de solução para cada uma.
- Limpe as tampas em um sonicator de banho por 5 min usando IPA. Enxágue o IPA das lamínulas usando água DI. O IPA é completamente enxaguado quando uma película lisa de água pode ser vista na lamínula.
- Secar as lamínulas numa placa quente durante 5 minutos a 100 °C. Coloque as tampas secas em placas de Petri limpas, garantindo que elas não se sobreponham.
- Usando uma varinha de descarga corona, exponha as lamínulas a uma descarga corona por 1 min cada.
NOTA: Esta etapa deve ser realizada em uma área bem ventilada ou em uma coifa química. - Remova as lamínulas da placa de Petri e, em seguida, mergulhe na solução APTES por 10 s dentro de 5 minutos da exposição corona, garantindo que a lamínula fique submersa.
NOTA: Certifique-se de manter o controle de qual lado foi tratado com plasma e mantê-lo voltado para cima. - Em seguida, mergulhe a tampa em acetona por 10 s e seque com ar comprimido. Coloque a tampa seca de volta na placa de Petri, com o lado tratado virado para cima.
NOTA: Repita os passos 1.3.5-1.3.7 para todas as lamínulas. - Pipetar 1 mL de solução de glutaraldeído para a superfície de cada lamínula. Cubra o máximo de superfície possível sem permitir que a solução se espalhe sobre a borda da lamínula. Mais solução de glutaraldeído pode ser adicionada, se necessário. Certifique-se de não riscar a superfície com a ponta da pipeta.
- Deixe as lamínulas em contato com a solução por 30 min e, em seguida, enxágue com água DI por 20 s. Seque as lamínulas usando ar comprimido e coloque-as de volta nas placas de Petri, tratadas com plasma de lado para cima.
NOTA: As lamínulas podem ser armazenadas por até 1 semana à temperatura ambiente (RT).
- Corte a laser da base magnética modular
NOTA: O projeto da base magnética modular é fornecido na Figura Suplementar 1. A base modular serve como um poço para segurar a mídia e também pode ser usada para fixar magneticamente módulos especializados, como descrito em trabalhos publicados anteriormente 22,37,38,39.- Recorte os projetos da camada de PMMA usando configurações de laser apropriadas, como o número de passadas e a potência.
NOTA: As configurações do laser devem ser ajustadas de modo que os ímãs possam ser pressionados na camada de PMMA. Cada laser é diferente, e os parâmetros de corte devem ser otimizados experimentalmente. Para um laser de 45 W, 100% de velocidade, 100% de potência e 3 passadas são recomendados para cortar PMMA de 2 mm a 2 mm de espessura. - Lave a parte cortada a laser usando água e sabão para remover os detritos do processo de corte a laser.
NOTA: Não utilize solventes para limpar as peças cortadas a laser. Os solventes podem resultar na propagação de microfissuras nas bordas cortadas a laser.
- Recorte os projetos da camada de PMMA usando configurações de laser apropriadas, como o número de passadas e a potência.
- Montagem da plataforma
- Empurre ímãs (3/16 de diâmetro, 1/16 de espessura) manualmente na base cortada a laser. Um martelo macio ou uma extremidade de chave de fenda podem ser usados para ajudar a empurrar o ímã. A espessura dos ímãs deve ser menor do que a espessura da base de PMMA para garantir que os ímãs estejam nivelados com a superfície da base.
NOTA: Os ímãs permitem que o usuário adicione funcionalidade à plataforma anexando módulos adicionais. - Retire o encosto da folha de PSA e prenda a base a uma lamínula tratada com glutaraldeído com o lado funcionalizado voltado para cima.
- Coloque suavemente o recorte do canal PDMS na cavidade definida pelo quadro. Pressione para baixo com pinças de ponta larga para remover bolhas de ar e garantir contato conforme.
- Coloque a tampa do canal tratado com BSA na parte superior do recorte do canal, com o lado BSA voltado para baixo. Verifique se as portas de entrada e saída de fluido estão alinhadas com o canal.
- O dispositivo está pronto para a injeção de COL1.
- Empurre ímãs (3/16 de diâmetro, 1/16 de espessura) manualmente na base cortada a laser. Um martelo macio ou uma extremidade de chave de fenda podem ser usados para ajudar a empurrar o ímã. A espessura dos ímãs deve ser menor do que a espessura da base de PMMA para garantir que os ímãs estejam nivelados com a superfície da base.
2. Injetar a solução de COL1 no microcanal e remover a tampa para aplicações em cultura de células
- Preparando a solução COL1
- Coloque todos os reagentes necessários (solução estoque de COL1 [6 mg·mL−1], água ultrapura, 10x PBS, NaOH 0,1 M) no gelo no gabinete de biossegurança.
- Calcule o volume de reagentes da seguinte forma:

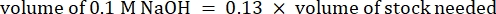
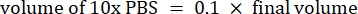
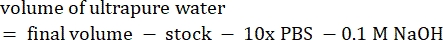
NOTA: Portanto, para fazer 1 mL de colágeno neutralizado 2,5 mg·mL−1 a partir de um estoque de 6 mg·mL−1, adicione 416,67 μL de colágeno, 429,16 μL de água DI, 100 μL de 10x PBS e 54,16 μL de NaOH 0,1 M para um pH final de 7. Adicionar 20 μL de NaOH 0,1 M para atingir um pH de 9,0. - Adicionar os reagentes em um frasco vazio de 5 mL na seguinte ordem: i) estoque de COL1, ii) água DI, iii) 10x PBS, iv) NaOH 0,1 M.
Observação : use uma pipeta de deslocamento para manipular a solução COL1. Pode ocorrer uma perda significativa de amostra se for utilizada uma pipeta regular, levando a flutuações na concentração final da solução. - Elevar o pH da solução para o pH desejado (tipicamente entre 7-9).
- Injetando a solução de COL1
- Coloque uma bomba de seringa, uma seringa estéril resfriada, uma solução de COL1 neutralizada resfriada e uma agulha de bloqueio Luer estéril de 20 G e 90° em um armário de biossegurança. Coloque a solução de COL1 na seringa, tomando cuidado para evitar a introdução de bolhas.
- Ligue a ponta da agulha de 90° 20 G à seringa, carregue a seringa na bomba da seringa, agulha virada para baixo e prima a agulha com a solução de COL1.
- Ajuste a bomba de seringa para a taxa de fluxo necessária, entre 50-2.000 μL/min.
- Coloque os canais PDMS preparados (secção 1) numa tomada de laboratório e nivele com a agulha.
- Insira a agulha na porta de entrada do canal PDMS (lado largo). Injete o canal até que uma gota de ~30 μL de COL1 se colete no lado de saída.
- Abaixe o macaco de laboratório e separe suavemente a agulha do canal recém-preenchido.
- Repita as etapas 2.2.5-2.2.9 até que todos os canais tenham sido preenchidos com a solução COL1.
- Coloque os canais preenchidos nas placas de Petri ao lado de um lenço limpo e sem fiapos saturado com água DI para evitar a desidratação do gel COL1 recém-formado.
- Cubra a placa de Petri e coloque os canais carregados na incubadora (37 °C, 95% de umidade) por 2 h antes da etapa de descascamento.
- Descascamento e equilíbrio dos meios
- Exponha o gel polimerizado de COL1 retirando a tampa do PDMS usando uma pinça. O tratamento BSA impede que o COL1 se fixe à tampa.
- Adicionar 650 μL de EGM ao poço.
- Deixar os dispositivos na incubadora (37 °C, 95% de umidade) por um mínimo de 4 h para equilibrar o gel e o meio. Substitua a mídia antes de semear por células
- Células semeadoras
- Colocar tripsina a 0,25% e meios de cultura aquecidos juntamente com o número necessário de pipetas de 5 mL e 10 mL no gabinete de biossegurança.
- Cultura de células endoteliais da veia umbilical humana (HUVECs) para confluência de 80% em meio de crescimento endotelial (EGM) a 37 °C, em umidade de 95%. Colocar o frasco de cultura de tecidos contendo HUVECs no BSC após verificar a confluência ao microscópio.
- Eliminar o meio no frasco T25 e lavar as células 2x com PBS 1x. Adicionar 1 ml de tripsina ao balão e colocá-lo na incubadora (37 °C, 95% de humidade) durante 3 min.
- Verifique o frasco ao microscópio após 3 min para garantir que as células estão completamente separadas da superfície.
- Adicionar 3 mL de EGM (com soro) ao balão para neutralizar a tripsina. Em seguida, transferir a solução celular usando uma pipeta de 5 mL para um tubo cônico de 15 mL. Centrifugar o tubo cónico contendo células a 150 x g durante 5 min.
- Coloque o tubo cônico no armário de biossegurança e descarte o sobrenadante sem perturbar o pellet celular. Ressuspender as células em 1 mL de meio de cultura fresco.
- Adicionar e misturar 15 μL de azul de tripano e 15 μL da solução celular ressuspensa num tubo cónico de 1 ml.
- Injete azul de tripano e solução de células em ambos os lados de uma lâmina de contagem de células e insira a lâmina em um dispositivo de contagem de células.
- Diluir a solução celular em meio de crescimento endotelial até a concentração necessária com base na concentração celular obtida do dispositivo de contagem celular.
NOTA: Um frasco T25 de HUVECs com 80% de confluência produzirá ~750.000 células·ml−1 se diluído em 1 mL de meio. O volume da suspensão celular a ser semeada é calculado como área da superfície de cultura × densidade celular/concentração de suspensão celular. Exemplo: Para semear 20.000 células·cm−2 no poço de 35 mm x 15 mm, será necessário ~140 μL da suspensão celular. - Aspirar a mídia na matriz COL1 projetada.
- Adicione o volume necessário da solução celular ao substrato COL1 e deixe as células acomodarem-se por um mínimo de 4 h antes da aquisição de imagens.
- Marcação do núcleo celular e citoesqueleto
- Aspirar o meio das células e lavar 3x com 1x PBS, 500 μL em cada lavagem. Cobrir a camada celular com paraformaldeído a 4% por 15 min em TR.
- Aspirar o paraformaldeído e lavar com 1x PBS Tween-20 por 5 min.
- Permeabilizar a membrana celular usando 500 μL de solução de Triton-X a 0,1% em PBS por 15 min. Lave com 1x PBS Tween-20 por 5 min.
- Bloquear os sítios de ligação inespecíficos com 500 μL de BSA a 4% em PBS por 30 min na RT.
- Diluir a solução fluorescente de faloidina-actina em BSA a 4% (1 μL de estoque em 400 μL de BSA).
- Aspirar a solução de BSA, adicionar a solução de faloidina às células e aguardar 30 min na RT.
- Diluir a coloração nuclear em BSA a 4% (1 μL em 500 μL), aspirar a faloidina, adicionar a solução de corante nuclear de trabalho e aguardar 15 min na RT.
- Aspirar a solução de trabalho de coloração nuclear, lavar com PBS Tween-20 3x por 5 min cada, e substituir por 1x PBS antes da aquisição de imagens.
- Imagem utilizando microscópio epifluorescente utilizando o canal FITC (ex 491 nm/em 516 nm) e canal DAPI (ex 360 nm/em 460 nm) com lente de 40x. Obtenha imagens do colágeno usando um confocal de varredura a laser no modo de reflectância usando a linha laser de 488 nm (15% de potência) e objetiva de imersão em água de 40x.
Resultados
Quando uma solução de COL1 automontável flui através de um canal com área de secção transversal decrescente, a velocidade de fluxo (v x) da solução de COL1 aumenta localmente em uma magnitude, ∂v x, ao longo do comprimento da constrição entre os dois segmentos (∂x), resultando em uma taxa de deformação extensional (ε̇) onde ε̇ = ∂v x/∂x. A taxa de deformação extensional pode ser calculada a partir da velocidade do fluido, que é medida por meio da velocimetria p...
Discussão
Protocolos para geração de matrizes de COL1 com fibras alinhadas têm sido descritos utilizando métodos magnéticos, aplicação direta de deformações mecânicas e técnicasmicrofluídicas47. Abordagens microfluídicas são comumente usadas para criar sistemas microfisiológicos devido às suas características de fluxo e transporte bem definidas, que permitem o controle preciso sobre o microambiente bioquímico. Uma vez que as fibras de COL1 alinhadas fornecem pistas instrutivas importantes ...
Divulgações
Todos os autores declaram não haver interesses concorrentes.
Agradecimentos
Este trabalho foi apoiado em parte pelo Instituto Nacional de Saúde sob o número de prêmio R21GM143658 e pela National Science Foundation sob o número de concessão 2150798. O conteúdo é de responsabilidade exclusiva dos autores e não representa, necessariamente, a opinião oficial das agências de fomento.
Materiais
| Name | Company | Catalog Number | Comments |
| (3-Aminopropyl)triethoxysilane, 99% (APTES) | Sigma Aldrich | 440140-100ML | |
| 20 Gauge IT Series Angled Dispensing Tip | Jensen Global | JG-20-1.0-90 | |
| 3/16" dia. x 1/16" thick Nickel Plated Magnet | KJ Magnetics | D31 | |
| 3M (TC) 12X12-6-467MP | DigiKey | 3M9726-ND | |
| ACETONE ACS REAGENT ≥99.5% | Signa Aldrich | 179124-4L | |
| BD-20AC LABORATORY CORONA TREATER | Electro-Technic Products | 12051A | |
| Bovine Serum Albumin (BSA), Fraction V, 98%, Reagent Grade, Alfa Aesar | VWR | AAJ64100-09 | |
| Clear cast acrylic sheet | McMaster-Carr | 8560K181 | |
| Corning 100 mL Trypsin 10x, 2.5% Trypsin in HBSS [-] calcium, magnesium, phenol red, Porcine Parvovirus Tested | VWR | 45000-666 | |
| Countess II Automated Cell Counter | Thermo Fisher Scientific | AMQAX1000 | |
| CT-FIRE software | LOCI - University of Wisconsin | ||
| EGM-2 Endothelial Cell Growth Medium-2 BulletKit, (CC-3156 & CC-4176), Lonza CC-3162, 500 mL | Lonza | CC-3162 | |
| Glutaraldehyde 50% in aqueous solution, Reagent Grade, Packaging=HDPE Bottle, Size=100 mL | VWR | VWRV0875-100ML | |
| Graphtec CELITE-50 | Graphtec | CE LITE-50 | |
| HEPES (1 M) | Thermo Fisher Scientific | 15-630-080 | |
| High-Purity Silicone Rubber .010" Thick, 6" X 8" Sheet, 55A Durometer | McMaster-Carr | 87315K62 | |
| Human Umbilical Vein Endothelial cells | Thermo Fisher Scientific | C0035C | |
| Invitrogen Trypan Blue Stain (0.4%) | Thermo Fisher Scientific | T10282 | |
| Isopropanol | Fisher Scientific | A4154 | |
| Laser cutter | Full Spectrum | 20x12 H-series | |
| Microfluidics Syringe pump | New Era Syringe Pumps | NE-1002X | |
| Microman E Single Channel Pipettor, Gilson, Model M1000E | Gilson | FD10006 | |
| Molecular Probes Alexa Fluor 488 Phalloidin | Thermo Fisher Scientific | A12379 | |
| Molecular Probes Hoechst 33342, Trihydrochloride, Trihydrate | Thermo Fisher Scientific | H3570 | |
| Nutragen Bovine Atelo Collagen | Advanced BioMatrix | 5010-50ML | |
| Pbs (10x), pH 7.4 | VWR | 70011044.00 | |
| PBS pH 7.4 | Thermo Fisher Scientific | 10010049.00 | |
| Phosphate-buffered saline (PBS, 10x), with Triton X-100 | Alfa Aesar | J63521 | |
| Replacement carrier sheet for graphtec craft ROBO CC330L-20 | USCUTTER | GRPCARSHTN | |
| Restek Norm-Ject Plastic Syringe 1 mL Luer Slip | Restek | 22766.00 | |
| Silicon wafer | University wafer | 452 | |
| Sodium Hydroxide, ACS, Packaging=Poly Bottle, Size=500 g | VWR | BDH9292-500G | |
| Sylgard 184 | VWR | 102092-312 | |
| Thermo Scientific Pierce 20x PBS Tween 20 | Thermo Fisher Scientific | 28352.00 |
Referências
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Bosman, F. T., Stamenkovic, I. Functional structure and composition of the extracellular matrix. The Journal of Pathology. 200 (4), 423-428 (2003).
- Cox, T. R., Erler, J. T. Remodeling and homeostasis of the extracellular matrix: Implications for fibrotic diseases and cancer. Disease Models & Mechanisms. 4 (2), 165-178 (2011).
- Cross, V. L., et al. Dense type I collagen matrices that support cellular remodeling and microfabrication for studies of tumor angiogenesis and vasculogenesis in vitro. Biomaterials. 31 (33), 8596-8607 (2010).
- Lu, P., Takai, K., Weaver, V. M., Werb, Z. Extracellular matrix degradation and remodeling in development and disease. Cold Spring Harbor Perspectives in Biology. 3 (12), 005058 (2011).
- Piotrowski-Daspit, A. S., Nerger, B. A., Wolf, A. E., Sundaresan, S., Nelson, C. M. Dynamics of tissue-induced alignment of fibrous extracellular matrix. Biophysical Journal. 113 (3), 702-713 (2017).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4 (1), 38 (2006).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6 (1), 11 (2008).
- Szulczewski, J. M., et al. Directional cues in the tumor microenvironment due to cell contraction against aligned collagen fibers. Acta Biomaterialia. 129, 96-109 (2021).
- Aubin, H., et al. Directed 3D cell alignment and elongation in microengineered hydrogels. Biomaterials. 31 (27), 6941-6951 (2010).
- Gruschwitz, R., et al. Alignment and cell-matrix interactions of human corneal endothelial cells on nanostructured collagen type I matrices. Investigative Ophthalmology & Visual Science. 51 (12), 6303-6310 (2010).
- Wang, W. Y., et al. Extracellular matrix alignment dictates the organization of focal adhesions and directs uniaxial cell migration. APL Bioengineering. 2 (4), 046107 (2018).
- Wang, W. Y., Lin, D., Jarman, E. H., Polacheck, W. J., Baker, B. M. Functional angiogenesis requires microenvironmental cues balancing endothelial cell migration and proliferation. Lab on a Chip. 20 (6), 1153-1166 (2020).
- Lanfer, B. The growth and differentiation of mesenchymal stem and progenitor cells cultured on aligned collagen matrices. Biomaterials. 30 (30), 5950-5958 (2009).
- Brauer, E., et al. Collagen fibrils mechanically contribute to tissue contraction in an in vitro wound healing scenario. Advanced Science. 6 (9), 1801780 (2019).
- Ingber, D. E. From mechanobiology to developmentally inspired engineering. PhilosophicalTransactions of the Royal Society B: Biological Sciences. 373 (1759), 20170323 (2018).
- Wang, H., Abhilash, A. S., Chen, C. S., Wells, R. G., Shenoy, V. B. Long-range force transmission in fibrous matrices enabled by tension-driven alignment of fibers. Biophysical Journal. 107 (11), 2592-2603 (2014).
- Reinhart-King, C. A., Dembo, M., Hammer, D. A. Cell-cell mechanical communication through compliant substrates. Biophysical Journal. 95 (12), 6044-6051 (2008).
- Ahadian, S., et al. Organ-on-a-chip platforms: A convergence of advanced materials, cells, and microscale technologies. Advanced Healthcare Materials. 7 (2), 1700506 (2018).
- Hou, X., et al. Interplay between materials and microfluidics. Nature Reviews Materials. 2 (5), 17016 (2017).
- Abhyankar, V. V., et al. A platform for assessing chemotactic migration within a spatiotemporally defined 3D microenvironment. Lab on a Chip. 8 (9), 1507-1515 (2008).
- Abhyankar, V. V., Wu, M., Koh, C. Y., Hatch, A. V. A reversibly sealed, easy access, modular (SEAM) microfluidic architecture to establish in vitro tissue interfaces. PLoS One. 11 (5), 0156341 (2016).
- Williams, M. J., et al. A low-cost, rapidly integrated debubbler (RID) module for microfluidic cell culture applications. Micromachines. 10 (6), 360 (2019).
- Hsu, M. C., et al. A miniaturized 3D printed pressure regulator (µPR) for microfluidic cell culture applications. Scientific Reports. 12, 10769 (2022).
- Huh, D., Torisawa, Y. S., Hamilton, G. A., Kim, H. J., Ingber, D. E. Microengineered physiological biomimicry: organs-on-chips. Lab on a Chip. 12 (12), 2156-2164 (2012).
- Abhyankar, V. V., Lokuta, M. A., Huttenlocher, A., Beebe, D. J. Characterization of a membrane-based gradient generator for use in cell-signaling studies. Lab on a Chip. 6 (3), 389-393 (2006).
- Hasan, M. R., et al. One-step fabrication of flexible nanotextured PDMS as a substrate for selective cell capture. Biomedical Physics & Engineering Express. 4 (2), 025015 (2018).
- Meyvantsson, I., Beebe, D. J. Cell culture models in microfluidic systems. Annual Review of Physical Chemistry. 1, 423-449 (2008).
- Ma, Y., et al. Viscoelastic cell microenvironment: Hydrogel-based strategy for recapitulating dynamic ECM mechanics. Advanced Functional Materials. 31 (24), 2100848 (2021).
- Ma, Y., et al. 3D spatiotemporal mechanical microenvironment: A hydrogel-based platform for guiding stem cell fate. Advanced Materials. 30 (49), 1705911 (2018).
- Lee, P., Lin, R., Moon, J., Lee, L. P. Microfluidic alignment of collagen fibers for in vitro cell culture. Biomedical Microdevices. 8 (1), 35-41 (2006).
- Del Amo, C., Borau, C., Movilla, N., Asín, J., García-Aznar, J. M. Quantifying 3D chemotaxis in microfluidic-based chips with step gradients of collagen hydrogel concentrations. Integrative Biology. 9 (4), 339-349 (2017).
- Shi, N., et al. A 3D, magnetically actuated, aligned collagen fiber hydrogel platform recapitulates physical microenvironment of myoblasts for enhancing myogenesis. Small Methods. 5 (6), 2100276 (2021).
- Lanfer, B., et al. Aligned fibrillar collagen matrices obtained by shear flow deposition. Biomaterials. 29 (28), 3888-3895 (2008).
- Saeidi, N., Sander, E. A., Ruberti, J. W. Dynamic shear-influenced collagen self-assembly. Biomaterials. 30 (34), 6581-6592 (2009).
- Saeidi, N., Sander, E. A., Zareian, R., Ruberti, J. W. Production of highly aligned collagen lamellae by combining shear force and thin film confinement. Acta Biomaterialia. 7 (6), 2437-2447 (2011).
- Ahmed, A., et al. Microengineered 3D collagen gels with independently tunable fiber anisotropy and directionality. Advanced Materials Technologies. 6 (4), 2001186 (2021).
- Ahmed, A., et al. Local extensional flows promote long-range fiber alignment in 3D collagen hydrogels. Biofabrication. 14 (3), 035019 (2022).
- Mansouri, M., et al. The modular µSiM reconfigured: Integration of microfluidic capabilities to study in vitro barrier tissue models under flow. Advanced Healthcare Materials. , (2022).
- Paten, J. A., et al. Flow-induced crystallization of collagen: a potentially critical mechanism in early tissue formation. ACS Nano. 10 (5), 5027-5040 (2016).
- Liu, Y., Eliceiri, K. W. Quantifying fibrillar collagen organization with curvelet transform-based tools. Journal of Visualized Experiments. (165), e61931 (2020).
- Bredfeldt, J. S., et al. Automated quantification of aligned collagen for human breast carcinoma prognosis. Journal of Pathology Informatics. 5 (1), 28 (2014).
- Bredfeldt, J. S., et al. Computational segmentation of collagen fibers from second-harmonic generation images of breast cancer. Journal of Biomedical Optics. 19 (1), 016007 (2014).
- Carey, S. P., et al. Local extracellular matrix alignment directs cellular protrusion dynamics and migration through Rac1 and FAK. Integrative Biology. 8 (8), 821-835 (2016).
- Carey, S. P., Kraning-Rush, C. M., Williams, R. M., Reinhart-King, C. A. Biophysical control of invasive tumor cell behavior by extracellular matrix microarchitecture. Biomaterials. 33 (16), 4157-4165 (2012).
- Ahmed, A., et al. Engineering fiber anisotropy within natural collagen hydrogels. AmericanJournal of Physiology-Cell Physiology. 320 (6), 1112-1124 (2021).
- Mohammadi, H., Janmey, P. A., McCulloch, C. A. Lateral boundary mechanosensing by adherent cells in a collagen gel system. Biomaterials. 35 (4), 1138-1149 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados