È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Idrogel di collagene 3D di microingegneria con allineamento delle fibre a lungo raggio
In questo articolo
Riepilogo
Questo protocollo dimostra l'uso di un canale microfluidico con geometria variabile lungo la direzione del flusso del fluido per generare tensione estensionale (stretching) per allineare le fibre in un idrogel di collagene 3D (<250 μm di spessore). L'allineamento risultante si estende su diversi millimetri ed è influenzato dalla velocità di deformazione estensionale.
Abstract
Le fibre di collagene I allineato (COL1) guidano la motilità delle cellule tumorali, influenzano la morfologia delle cellule endoteliali, controllano la differenziazione delle cellule staminali e sono un segno distintivo dei tessuti cardiaci e muscoloscheletrici. Per studiare la risposta cellulare a microambienti allineati in vitro, sono stati sviluppati diversi protocolli per generare matrici COL1 con allineamento delle fibre definito, inclusi metodi magnetici, meccanici, cellulari e microfluidici. Di questi, gli approcci microfluidici offrono funzionalità avanzate come un controllo accurato dei flussi di fluidi e del microambiente cellulare. Tuttavia, gli approcci microfluidici per generare matrici COL1 allineate per piattaforme di coltura in vitro avanzate sono stati limitati a sottili "tappetini" (<40 μm di spessore) di fibre COL1 che si estendono su distanze inferiori a 500 μm e non sono favorevoli alle applicazioni di coltura cellulare 3D. Qui, presentiamo un protocollo per fabbricare matrici 3D COL1 (130-250 μm di spessore) con regioni su scala millimetrica di allineamento delle fibre definito in un dispositivo microfluidico. Questa piattaforma fornisce funzionalità avanzate di coltura cellulare per modellare microambienti tissutali strutturati fornendo accesso diretto alla matrice microingegnerizzata per la coltura cellulare.
Introduzione
Le cellule risiedono in una complessa rete fibrosa 3D chiamata matrice extracellulare (ECM), la maggior parte della quale è composta dalla proteina strutturale collagene di tipo I (COL1)1,2. Le proprietà biofisiche dell'ECM forniscono indicazioni alle cellule e, in risposta, le cellule rimodellano la microarchitettura ECM 3,4,5. Queste interazioni reciproche cellula-matrice possono dare origine a domini di fibre COL1 allineati6 che promuovono l'angiogenesi e l'invasione cellulare nell'ambiente tumorale 7,8,9 e influenzano la morfologia cellulare 10,11,12, la polarizzazione 13 e la differenziazione 14. Le fibre di collagene allineate promuovono anche la guarigione delle ferite15, svolgono un ruolo chiave nello sviluppo dei tessuti 16 e contribuiscono alla comunicazione cellulare a lungo raggio17,18. Pertanto, replicare la microarchitettura nativa della fibra COL1 in vitro è un passo importante verso lo sviluppo di modelli strutturati per studiare le risposte cellulari a microambienti allineati.
I sistemi di coltura cellulare microfluidica sono stati stabiliti come tecnologia preferita per sviluppare sistemi microfisiologici (MPS)19,20,21,22,23. Sfruttando effetti di ridimensionamento su microscala favorevoli, questi sistemi forniscono un controllo preciso sui flussi di fluidi, supportano l'introduzione controllata di forze meccaniche e definiscono il microambiente biochimico all'interno di un microcanale 21,24,25,26,27. Le piattaforme MPS sono state utilizzate per modellare microambienti tessuto-specifici e studiare le interazioni multiorgano28. Allo stesso tempo, gli idrogel sono stati ampiamente esplorati per ricapitolare la meccanica 3D e l'influenza biologica della ECM che si osservano in vivo29,30. Con una crescente enfasi sull'integrazione della coltura 3D con piattaforme microfluidiche, numerosi approcci possono combinare idrogel COL1 in dispositivi microfluidici31,32,33. Tuttavia, i metodi per allineare gli idrogel COL1 nei canali microfluidici sono stati limitati a sottili "tappetini" 2D (<40 μm di spessore) in canali larghi <1 mm, offrendo un potenziale limitato per modellare le risposte cellulari in microambienti 3D allineati31,34,35,36.
Per ottenere idrogel COL1 3D allineati in un sistema microfluidico, è stato dimostrato che, quando una soluzione COL1 autoassemblante è esposta a flussi estensionali locali (variazione di velocità lungo la direzione del flusso), gli idrogel COL1 risultanti mostrano un grado di allineamento delle fibre che è direttamente proporzionale all'entità della velocità di deformazione estensionale che sperimentano37, 38. Il design a microcanali in questo protocollo è unico in due modi; in primo luogo, il design segmentato introduce la deformazione estensionale locale nella soluzione COL1 e, in secondo luogo, la sua costruzione "a due pezzi" consente all'utente di allineare le fibre COL1 e quindi smontare il canale per accedere direttamente alle fibre allineate in un formato aperto. Questo approccio può essere ulteriormente adottato per sviluppare piattaforme microfluidiche modulari che sviluppano sistemi microfisiologici con matrici COL1 ordinate. Il seguente protocollo descrive il processo di fabbricazione di microcanali segmentati e descrive in dettaglio l'uso dei canali per allineare l'atelo bovino COL1. Questo protocollo fornisce anche istruzioni per la coltura di cellule su COL1 in un formato a pozzo aperto e discute l'aggiunta di funzionalità alla piattaforma utilizzando uno strato di base magnetico modulare.
Protocollo
1. Fabbricazione del canale a due pezzi e della base della piattaforma modulare
NOTA: Il canale microfluidico è costruito utilizzando due parti: il "cutout" del canale microfluidico, che è tagliato a rasoio da un foglio di polidimetil silossano (PDMS) di spessore definito e il coperchio del canale, che si lega reversibilmente al ritaglio e forma il canale. Il canale è circondato da un telaio in poli(metacrilato di metile) (PMMA) che fungerà da serbatoio di supporti (Figura 1). Il telaio in PMMA può anche essere utilizzato per bloccare magneticamente moduli specializzati per una maggiore funzionalità.
- Canali di taglio rasoio da fogli PDMS:
NOTA: la progettazione del canale microfluidico per questo passaggio è fornita nella Figura supplementare 1. Il canale è costituito da cinque segmenti di lunghezza 5 mm ciascuno e larghezze di 10 mm, 5 mm, 2,5 mm, 1,25 mm e 0,75 mm. La velocità della soluzione di collagene, iniettata ad una portata costante (50-400 μL·min−1), aumenta localmente lungo il canale man mano che la larghezza del canale diminuisce per generare un flusso estensionale.- Montare un foglio PDMS spesso 250 μm su un foglio di supporto di plastica e tagliare il design microfluidico utilizzando una fresa artigianale a una profondità della lama di 0,5 mm, velocità 1 cm·s−1 e forza elevata.
- Utilizzando un bagno ad ultrasuoni, pulire i ritagli del canale microfluidico per 5 minuti. Risciacquare i canali sonicati in acqua deionizzata (DI) e asciugarli su una piastra riscaldante pulita a 100 °C per 5 minuti.
- Conservare i canali in una capsula di Petri pulita e coperta fino all'uso.
- Fabbricazione del coperchio PDMS e della passivazione superficiale:
NOTA: Il ritaglio del canale dalla sezione 1.1 richiede un coperchio che può essere posizionato sopra di esso per creare un canale chiuso in cui può essere iniettata una soluzione COL1 (Figura supplementare 1). Il coperchio può essere rimosso dopo che il COL1 si è auto-assemblato. Per ridurre al minimo le possibilità che il COL1 nel canale si leghi al coperchio, il coperchio viene passivato utilizzando l'albumina sierica bovina (BSA). Viene fornito il design dello stampo per il coperchio PDMS. Lo stampo deve avere uno spessore di almeno 2 mm e deve avere un'impronta uguale all'impronta del foro del canale microfluidico. In questo protocollo, l'impronta dello stampo era di 35 mm x 15 mm.- Per preparare lo stampo, applicare un foglio di adesivo sensibile alla pressione (PSA) su un foglio di PMMA spesso 2,5 mm e tagliare al laser la forma dello stampo desiderata.
- Pulire lo stampo in PMMA tagliato al laser con una salvietta umida e priva di lanugine e rimuovere il supporto dalla pellicola PSA. Fissare lo stampo a un wafer di silicio da 100 mm di diametro che preme saldamente.
- Preparare PDMS in un rapporto di 10:1 (base:crosslinker). Mescolare vigorosamente per 1 minuto per garantire una corretta miscelazione e degasare la miscela in una camera a vuoto per rimuovere le bolle d'aria.
- Versare la soluzione PDMS degassata nello stampo in PMMA/silicone e polimerizzare su una piastra riscaldante a 100 °C per 20 minuti. Lasciare raffreddare lo stampo, rimuovere il PDMS polimerizzato e tagliare l'eccesso con una lametta da barba.
NOTA: assicurarsi che i lati rivolti verso il wafer di silicio (lati piatti) dei coperchi PDMS siano rivolti verso l'alto durante tutti i passaggi successivi. - Utilizzare un punzone bioptico di 1 mm di diametro per creare fori di ingresso e uscita che corrispondono alle estremità del canale microfluidico. Il ritaglio del canale del punto 1.1 può essere utilizzato come modello per guidare la posizione dei fori.
- Sonicare il coperchio in isopropanolo (IPA) per 5 minuti, risciacquare con acqua DI, asciugare con una fonte di aria compressa e quindi conservare in una capsula di Petri pulita e coperta. Posizionare la capsula di Petri in una camera di sterilizzazione UV (scoperta) per 1 minuto per sterilizzare.
- Coprire e trasferire in un armadio di biosicurezza (BSC).
- Pipettare 300 μL di albumina sierica bovina (BSA) da 40 mg•mL-1 in soluzione salina tamponata con fosfato (PBS) 1x sui coperchi PDMS e distribuire uniformemente la soluzione utilizzando una punta di pipetta. Porre in frigorifero a 4 °C per almeno 4 ore prima dell'uso.
- Spostare i coperchi dal frigorifero in un BSC, lavare 5x con 1x PBS e lasciarli asciugare all'aria per 10 minuti.
NOTA: Le coperture PDMS possono essere conservate in frigorifero fino a 1 settimana dopo averle rivestite con BSA.
- Trattamento con glutaraldeide dei coprivetrini
NOTA: La funzionalizzazione dei coprivetrini con glutaraldeide lega covalentemente COL1 al coprivetrino e impedisce il distacco dell'idrogel.- Preparare una soluzione di amminopropiltrietossisilano (APTES) al 2% (v/v) in un becher di vetro aggiungendo 1 mL di APTES a 49 mL di acetone.
- Diluire una soluzione di glutaraldeide al 25% al 5% in acqua DI. Preparare 2 ml di soluzione per ogni coprislip da 24 x 50 mm. Per i coprivetrini da 24 mm x 24 mm o 22 mm x 22 mm, preparare 1 mL di soluzione per ciascuno.
- Pulire i coprivetrini in un sonicatore da bagno per 5 minuti usando IPA. Risciacquare l'alcool isopropilico dai vetrini con acqua DI. L'alcool isopropilico viene completamente risciacquato quando sulla copertina si può vedere una pellicola liscia d'acqua.
- Asciugare i coperchi su una piastra calda per 5 minuti a 100 °C. Posizionare i vetrini di copertura essiccati in piastre di Petri pulite, assicurandosi che non si sovrappongano.
- Utilizzando una bacchetta di scarico corona, esporre i coperchi a una scarica corona per 1 minuto ciascuno.
NOTA: questo passaggio deve essere eseguito in un'area ben ventilata o in una cappa chimica. - Rimuovere i coperchi dalla capsula di Petri e quindi immergerli nella soluzione APTES per 10 secondi entro 5 minuti dall'esposizione alla corona, assicurandosi che il coprislip sia immerso.
NOTA: Assicurarsi di tenere traccia di quale lato è stato trattato con plasma e tenerlo rivolto verso l'alto. - Quindi, immergere il coprislip in acetone per 10 secondi e asciugare con aria compressa. Riporre il coprislip asciutto nella capsula di Petri, con il lato trattato rivolto verso l'alto.
NOTA: ripetere i passaggi 1.3.5-1.3.7 per tutti i vetrini. - Pipettare 1 mL di soluzione di glutaraldeide sulla superficie di ciascun vetrino. Coprire quanta più superficie possibile senza permettere alla soluzione di fuoriuscire oltre il bordo del vetrino. Se necessario, è possibile aggiungere più soluzione di glutaraldeide. Assicurarsi di non graffiare la superficie con la punta della pipetta.
- Lasciare riposare i coperchi a contatto con la soluzione per 30 minuti e quindi risciacquare con acqua DI per 20 s. Asciugare i vetrini di copertura con aria compressa e riporli nelle piastre di Petri, trattate al plasma con il lato rivolto verso l'alto.
NOTA: I coprivetrini possono essere conservati fino a 1 settimana a temperatura ambiente (RT).
- Taglio laser della base magnetica modulare
NOTA: Il design della base magnetica modulare è fornito nella figura supplementare 1. La base modulare funge da pozzo per contenere il supporto e può anche essere utilizzata per collegare magneticamente moduli specializzati, come descritto nei lavori precedentemente pubblicati 22,37,38,39.- Ritaglia i disegni dallo strato di PMMA utilizzando le impostazioni laser appropriate come il numero di passaggi e la potenza.
NOTA: le impostazioni del laser devono essere regolate in modo tale che i magneti possano essere montati nello strato di PMMA. Ogni laser è diverso e i parametri di taglio devono essere ottimizzati sperimentalmente. Per un laser da 45 W, si consiglia il 100% di velocità, il 100% di potenza e 3 passaggi per tagliare PMMA da 2 mm a 2 mm di spessore. - Lavare la parte tagliata al laser con acqua e sapone per rimuovere i detriti dal processo di taglio laser.
NOTA: Non utilizzare solventi per pulire le parti tagliate al laser. I solventi possono provocare la propagazione di microfratture nei bordi tagliati al laser.
- Ritaglia i disegni dallo strato di PMMA utilizzando le impostazioni laser appropriate come il numero di passaggi e la potenza.
- Assemblaggio della piattaforma
- Spingere i magneti (3/16 di diametro, 1/16 di spessore) a mano nella base tagliata al laser. Un martello morbido o un'estremità del cacciavite possono essere utilizzati per aiutare a spingere il magnete. Lo spessore dei magneti deve essere inferiore allo spessore della base in PMMA per garantire che i magneti siano a filo con la superficie della base.
NOTA: I magneti consentono all'utente di aggiungere funzionalità alla piattaforma collegando moduli aggiuntivi. - Staccare il supporto dal foglio PSA e fissare la base a un coprislip trattato con glutaraldeide con il lato funzionalizzato rivolto verso l'alto.
- Posizionare delicatamente il ritaglio del canale PDMS nella cavità definita dal telaio. Premere verso il basso con pinzette a punta larga per rimuovere le bolle d'aria e garantire un contatto conforme.
- Posizionare il coperchio del canale trattato BSA sopra il ritaglio del canale, con il lato BSA rivolto verso il basso. Assicurarsi che le porte di ingresso e uscita del fluido siano allineate con il canale.
- Il dispositivo è pronto per l'iniezione COL1.
- Spingere i magneti (3/16 di diametro, 1/16 di spessore) a mano nella base tagliata al laser. Un martello morbido o un'estremità del cacciavite possono essere utilizzati per aiutare a spingere il magnete. Lo spessore dei magneti deve essere inferiore allo spessore della base in PMMA per garantire che i magneti siano a filo con la superficie della base.
2. Iniezione della soluzione COL1 nel microcanale e rimozione del coperchio per applicazioni di coltura cellulare
- Preparazione della soluzione COL1
- Introdurre tutti i reagenti necessari (soluzione madre COL1 [6 mg·mL−1], acqua ultrapura, 10x PBS, 0,1 M NaOH) su ghiaccio nell'armadio di biosicurezza.
- Calcolare il volume dei reagenti come segue

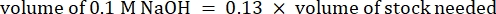
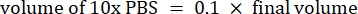
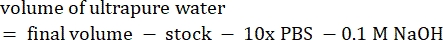
NOTA: Pertanto, per produrre 1 mL di collagene neutralizzato da 2,5 mg·mL−1 da un brodo di 6 mg·mL−1, aggiungere 416,67 μL di collagene, 429,16 μL di acqua DI, 100 μL di 10x PBS e 54,16 μL di 0,1 M NaOH per un pH finale di 7. Aggiungere 20 μL di 0,1 M NaOH per ottenere un pH di 9,0. - Aggiungere i reagenti in un flaconcino vuoto da 5 mL nell'ordine seguente: i) COL1 stock, ii) DI acqua, iii) 10x PBS, iv) 0,1 M NaOH.
NOTA: utilizzare una pipetta di spostamento per maneggiare la soluzione COL1. Se si utilizza una pipetta normale, può verificarsi una perdita significativa del campione, con conseguenti fluttuazioni nella concentrazione della soluzione finale. - Aumentare il pH della soluzione al pH desiderato (tipicamente tra 7-9).
- Iniezione della soluzione COL1
- Inserire una pompa per siringa, una siringa sterile refrigerata, una soluzione COL1 neutralizzata refrigerata e un ago sterile con punta angolare Luer da 20 G a 90° in un armadio di biosicurezza. Caricare la soluzione di COL1 nella siringa, facendo attenzione ad evitare l'introduzione di bolle.
- Collegare la punta dell'ago da 90° 20 G alla siringa, caricare la siringa nella pompa della siringa, nell'ago rivolto verso il basso e innescare l'ago con la soluzione COL1.
- Impostare la pompa della siringa sulla portata richiesta, tra 50-2.000 μL/min.
- Posizionare i canali PDMS preparati (sezione 1) su un jack da laboratorio e livellare con l'ago.
- Inserire l'ago nella porta di ingresso del canale PDMS (lato largo). Iniettare il canale fino a quando una goccia di ~30 μL di COL1 si raccoglie sul lato di uscita.
- Abbassare il jack da laboratorio e separare delicatamente l'ago dal canale appena riempito.
- Ripetere i passaggi 2.2.5-2.2.9 fino a riempire tutti i canali con la soluzione di COL1.
- Caricare i canali riempiti nelle piastre di Petri insieme a una salvietta pulita e priva di lanugine satura di acqua DI per prevenire la disidratazione del gel COL1 appena formato.
- Coprire la piastra di Petri e posizionare i canali caricati nell'incubatore (37 °C, 95% di umidità) per 2 ore prima della fase di peel-off.
- Peel off ed equilibrio dei media
- Esporre il gel polimerizzato COL1 sollevando il coperchio PDMS con una pinzetta. Il trattamento BSA impedisce al COL1 di attaccarsi al coperchio.
- Aggiungere 650 μL di EGM al pozzetto.
- Lasciare i dispositivi nell'incubatore (37 °C, 95% di umidità) per un minimo di 4 ore per equilibrare il gel e il supporto. Sostituire il supporto prima della semina con le celle
- Celle di semina
- Introdurre tripsina calda allo 0,25% e terreni di coltura insieme al numero richiesto di pipette da 5 mL e 10 mL nell'armadio di biosicurezza.
- Coltura di cellule endoteliali della vena ombelicale umana (HUVEC) all'80% di confluenza nei terreni di crescita endoteliale (EGM) a 37 °C, con un'umidità del 95%. Introdurre il matraccio di coltura tissutale contenente HUVEC nella BSC dopo aver controllato la confluenza al microscopio.
- Eliminare il supporto nel pallone T25 e lavare le celle 2x con 1x PBS. Aggiungere 1 mL di tripsina al matraccio e metterlo nell'incubatore (37 °C, 95% umidità) per 3 minuti.
- Controllare il pallone al microscopio dopo 3 minuti per assicurarsi che le cellule siano completamente staccate dalla superficie.
- Aggiungere 3 mL di EGM (con siero) al matraccio per neutralizzare la tripsina. Quindi, trasferire la soluzione cellulare utilizzando una pipetta da 5 mL in un tubo conico da 15 ml. Centrifugare il tubo conico contenente celle a 150 x g per 5 min.
- Posizionare il tubo conico nell'armadio di biosicurezza e scartare il surnatante senza disturbare il pellet cellulare. Risospendere le cellule in 1 mL di terreno di coltura fresco.
- Aggiungere e mescolare 15 μL di blu di tripano e 15 μL di soluzione cellulare risospesa in un tubo conico da 1 mL.
- Iniettare la soluzione di tripano blu e di cellule su entrambi i lati di un vetrino per il conteggio delle cellule e inserire il vetrino in un dispositivo di conteggio delle cellule.
- Diluire la soluzione cellulare nei mezzi di crescita endoteliale alla concentrazione richiesta in base alla concentrazione cellulare ottenuta dal dispositivo di conteggio cellulare.
NOTA: Un matraccio T25 di HUVEC all'80% di confluenza produrrà ~750.000 cellule·ml−1 se diluito in 1 mL di media. Il volume della sospensione cellulare da seminare è calcolato come area della superficie di coltura × densità cellulare/concentrazione della sospensione cellulare. Esempio: per seminare 20.000 cellule·cm−2 nel pozzo di 35 mm x 15 mm, saranno necessari ~140 μL della sospensione cellulare. - Aspirare il supporto sulla matrice ingegnerizzata COL1.
- Aggiungere il volume richiesto della soluzione cellulare sul substrato di COL1 e lasciare che le cellule si depositino per un minimo di 4 ore prima dell'imaging.
- Marcatura del nucleo cellulare e del citoscheletro
- Aspirare il fluido dalle celle e lavare 3 volte con 1x PBS, 500 μL in ogni lavaggio. Coprire lo strato cellulare con paraformaldeide al 4% per 15 minuti a RT.
- Aspirare la paraformaldeide e lavare con 1x PBS Tween-20 per 5 min.
- Permeabilizzare la membrana cellulare utilizzando 500 μL di soluzione Triton-X allo 0,1% in PBS per 15 minuti. Lavare con 1x PBS Tween-20 per 5 min.
- Bloccare i siti di legame non specifici con 500 μL di BSA al 4% in PBS per 30 minuti a RT.
- Diluire la soluzione fluorescente di etichette falloidin-actina in BSA al 4% (1 μL di materiale madre in 400 μL di BSA).
- Aspirare la soluzione BSA, aggiungere la soluzione di falloidina alle cellule e attendere 30 minuti a RT.
- Diluire la macchia nucleare in BSA al 4% (1 μL in 500 μL), aspirare la falloidina, aggiungere la soluzione di colorazione nucleare funzionante e attendere 15 minuti a RT.
- Aspirare la soluzione di lavoro delle macchie nucleari, lavare con PBS Tween-20 3x per 5 minuti ciascuno e sostituire con 1x PBS prima dell'imaging.
- Immagine al microscopio epifluorescente utilizzando il canale FITC (ex 491 nm/em 516 nm) e il canale DAPI (ex 360 nm/em 460 nm) con una lente 40x. Immagine del collagene utilizzando una scansione laser confocale in modalità riflettanza utilizzando la linea laser a 488 nm (potenza 15%) e l'obiettivo di immersione in acqua 40x.
Risultati
Quando una soluzione COL1 autoassemblante scorre attraverso un canale con area di sezione trasversale decrescente, la velocità del flusso (v x) della soluzione di COL1 aumenta localmente di una grandezza, ∂v x, lungo la lunghezza della costrizione tra i due segmenti (∂x), risultando in una velocità di deformazione estensionale (ε̇) dove ε̇ = ∂v x/∂x. La velocità di deformazione estensionale può essere calcolata dalla velocità del fluido, che viene misurata utilizzando la ...
Discussione
I protocolli per generare matrici COL1 con fibre allineate sono stati descritti utilizzando metodi magnetici, l'applicazione diretta di deformazioni meccaniche e tecniche microfluidiche47. Gli approcci microfluidici sono comunemente usati per creare sistemi microfisiologici a causa delle loro caratteristiche di flusso e trasporto ben definite, che consentono un controllo preciso sul microambiente biochimico. Poiché le fibre COL1 allineate forniscono segnali istruttivi chiave durante i processi fi...
Divulgazioni
Tutti gli autori non dichiarano interessi concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto in parte dal National Institute of Health con il numero di premio R21GM143658 e dalla National Science Foundation con il numero di sovvenzione 2150798. Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente il punto di vista ufficiale delle agenzie di finanziamento.
Materiali
| Name | Company | Catalog Number | Comments |
| (3-Aminopropyl)triethoxysilane, 99% (APTES) | Sigma Aldrich | 440140-100ML | |
| 20 Gauge IT Series Angled Dispensing Tip | Jensen Global | JG-20-1.0-90 | |
| 3/16" dia. x 1/16" thick Nickel Plated Magnet | KJ Magnetics | D31 | |
| 3M (TC) 12X12-6-467MP | DigiKey | 3M9726-ND | |
| ACETONE ACS REAGENT ≥99.5% | Signa Aldrich | 179124-4L | |
| BD-20AC LABORATORY CORONA TREATER | Electro-Technic Products | 12051A | |
| Bovine Serum Albumin (BSA), Fraction V, 98%, Reagent Grade, Alfa Aesar | VWR | AAJ64100-09 | |
| Clear cast acrylic sheet | McMaster-Carr | 8560K181 | |
| Corning 100 mL Trypsin 10x, 2.5% Trypsin in HBSS [-] calcium, magnesium, phenol red, Porcine Parvovirus Tested | VWR | 45000-666 | |
| Countess II Automated Cell Counter | Thermo Fisher Scientific | AMQAX1000 | |
| CT-FIRE software | LOCI - University of Wisconsin | ||
| EGM-2 Endothelial Cell Growth Medium-2 BulletKit, (CC-3156 & CC-4176), Lonza CC-3162, 500 mL | Lonza | CC-3162 | |
| Glutaraldehyde 50% in aqueous solution, Reagent Grade, Packaging=HDPE Bottle, Size=100 mL | VWR | VWRV0875-100ML | |
| Graphtec CELITE-50 | Graphtec | CE LITE-50 | |
| HEPES (1 M) | Thermo Fisher Scientific | 15-630-080 | |
| High-Purity Silicone Rubber .010" Thick, 6" X 8" Sheet, 55A Durometer | McMaster-Carr | 87315K62 | |
| Human Umbilical Vein Endothelial cells | Thermo Fisher Scientific | C0035C | |
| Invitrogen Trypan Blue Stain (0.4%) | Thermo Fisher Scientific | T10282 | |
| Isopropanol | Fisher Scientific | A4154 | |
| Laser cutter | Full Spectrum | 20x12 H-series | |
| Microfluidics Syringe pump | New Era Syringe Pumps | NE-1002X | |
| Microman E Single Channel Pipettor, Gilson, Model M1000E | Gilson | FD10006 | |
| Molecular Probes Alexa Fluor 488 Phalloidin | Thermo Fisher Scientific | A12379 | |
| Molecular Probes Hoechst 33342, Trihydrochloride, Trihydrate | Thermo Fisher Scientific | H3570 | |
| Nutragen Bovine Atelo Collagen | Advanced BioMatrix | 5010-50ML | |
| Pbs (10x), pH 7.4 | VWR | 70011044.00 | |
| PBS pH 7.4 | Thermo Fisher Scientific | 10010049.00 | |
| Phosphate-buffered saline (PBS, 10x), with Triton X-100 | Alfa Aesar | J63521 | |
| Replacement carrier sheet for graphtec craft ROBO CC330L-20 | USCUTTER | GRPCARSHTN | |
| Restek Norm-Ject Plastic Syringe 1 mL Luer Slip | Restek | 22766.00 | |
| Silicon wafer | University wafer | 452 | |
| Sodium Hydroxide, ACS, Packaging=Poly Bottle, Size=500 g | VWR | BDH9292-500G | |
| Sylgard 184 | VWR | 102092-312 | |
| Thermo Scientific Pierce 20x PBS Tween 20 | Thermo Fisher Scientific | 28352.00 |
Riferimenti
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Bosman, F. T., Stamenkovic, I. Functional structure and composition of the extracellular matrix. The Journal of Pathology. 200 (4), 423-428 (2003).
- Cox, T. R., Erler, J. T. Remodeling and homeostasis of the extracellular matrix: Implications for fibrotic diseases and cancer. Disease Models & Mechanisms. 4 (2), 165-178 (2011).
- Cross, V. L., et al. Dense type I collagen matrices that support cellular remodeling and microfabrication for studies of tumor angiogenesis and vasculogenesis in vitro. Biomaterials. 31 (33), 8596-8607 (2010).
- Lu, P., Takai, K., Weaver, V. M., Werb, Z. Extracellular matrix degradation and remodeling in development and disease. Cold Spring Harbor Perspectives in Biology. 3 (12), 005058 (2011).
- Piotrowski-Daspit, A. S., Nerger, B. A., Wolf, A. E., Sundaresan, S., Nelson, C. M. Dynamics of tissue-induced alignment of fibrous extracellular matrix. Biophysical Journal. 113 (3), 702-713 (2017).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4 (1), 38 (2006).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6 (1), 11 (2008).
- Szulczewski, J. M., et al. Directional cues in the tumor microenvironment due to cell contraction against aligned collagen fibers. Acta Biomaterialia. 129, 96-109 (2021).
- Aubin, H., et al. Directed 3D cell alignment and elongation in microengineered hydrogels. Biomaterials. 31 (27), 6941-6951 (2010).
- Gruschwitz, R., et al. Alignment and cell-matrix interactions of human corneal endothelial cells on nanostructured collagen type I matrices. Investigative Ophthalmology & Visual Science. 51 (12), 6303-6310 (2010).
- Wang, W. Y., et al. Extracellular matrix alignment dictates the organization of focal adhesions and directs uniaxial cell migration. APL Bioengineering. 2 (4), 046107 (2018).
- Wang, W. Y., Lin, D., Jarman, E. H., Polacheck, W. J., Baker, B. M. Functional angiogenesis requires microenvironmental cues balancing endothelial cell migration and proliferation. Lab on a Chip. 20 (6), 1153-1166 (2020).
- Lanfer, B. The growth and differentiation of mesenchymal stem and progenitor cells cultured on aligned collagen matrices. Biomaterials. 30 (30), 5950-5958 (2009).
- Brauer, E., et al. Collagen fibrils mechanically contribute to tissue contraction in an in vitro wound healing scenario. Advanced Science. 6 (9), 1801780 (2019).
- Ingber, D. E. From mechanobiology to developmentally inspired engineering. PhilosophicalTransactions of the Royal Society B: Biological Sciences. 373 (1759), 20170323 (2018).
- Wang, H., Abhilash, A. S., Chen, C. S., Wells, R. G., Shenoy, V. B. Long-range force transmission in fibrous matrices enabled by tension-driven alignment of fibers. Biophysical Journal. 107 (11), 2592-2603 (2014).
- Reinhart-King, C. A., Dembo, M., Hammer, D. A. Cell-cell mechanical communication through compliant substrates. Biophysical Journal. 95 (12), 6044-6051 (2008).
- Ahadian, S., et al. Organ-on-a-chip platforms: A convergence of advanced materials, cells, and microscale technologies. Advanced Healthcare Materials. 7 (2), 1700506 (2018).
- Hou, X., et al. Interplay between materials and microfluidics. Nature Reviews Materials. 2 (5), 17016 (2017).
- Abhyankar, V. V., et al. A platform for assessing chemotactic migration within a spatiotemporally defined 3D microenvironment. Lab on a Chip. 8 (9), 1507-1515 (2008).
- Abhyankar, V. V., Wu, M., Koh, C. Y., Hatch, A. V. A reversibly sealed, easy access, modular (SEAM) microfluidic architecture to establish in vitro tissue interfaces. PLoS One. 11 (5), 0156341 (2016).
- Williams, M. J., et al. A low-cost, rapidly integrated debubbler (RID) module for microfluidic cell culture applications. Micromachines. 10 (6), 360 (2019).
- Hsu, M. C., et al. A miniaturized 3D printed pressure regulator (µPR) for microfluidic cell culture applications. Scientific Reports. 12, 10769 (2022).
- Huh, D., Torisawa, Y. S., Hamilton, G. A., Kim, H. J., Ingber, D. E. Microengineered physiological biomimicry: organs-on-chips. Lab on a Chip. 12 (12), 2156-2164 (2012).
- Abhyankar, V. V., Lokuta, M. A., Huttenlocher, A., Beebe, D. J. Characterization of a membrane-based gradient generator for use in cell-signaling studies. Lab on a Chip. 6 (3), 389-393 (2006).
- Hasan, M. R., et al. One-step fabrication of flexible nanotextured PDMS as a substrate for selective cell capture. Biomedical Physics & Engineering Express. 4 (2), 025015 (2018).
- Meyvantsson, I., Beebe, D. J. Cell culture models in microfluidic systems. Annual Review of Physical Chemistry. 1, 423-449 (2008).
- Ma, Y., et al. Viscoelastic cell microenvironment: Hydrogel-based strategy for recapitulating dynamic ECM mechanics. Advanced Functional Materials. 31 (24), 2100848 (2021).
- Ma, Y., et al. 3D spatiotemporal mechanical microenvironment: A hydrogel-based platform for guiding stem cell fate. Advanced Materials. 30 (49), 1705911 (2018).
- Lee, P., Lin, R., Moon, J., Lee, L. P. Microfluidic alignment of collagen fibers for in vitro cell culture. Biomedical Microdevices. 8 (1), 35-41 (2006).
- Del Amo, C., Borau, C., Movilla, N., Asín, J., García-Aznar, J. M. Quantifying 3D chemotaxis in microfluidic-based chips with step gradients of collagen hydrogel concentrations. Integrative Biology. 9 (4), 339-349 (2017).
- Shi, N., et al. A 3D, magnetically actuated, aligned collagen fiber hydrogel platform recapitulates physical microenvironment of myoblasts for enhancing myogenesis. Small Methods. 5 (6), 2100276 (2021).
- Lanfer, B., et al. Aligned fibrillar collagen matrices obtained by shear flow deposition. Biomaterials. 29 (28), 3888-3895 (2008).
- Saeidi, N., Sander, E. A., Ruberti, J. W. Dynamic shear-influenced collagen self-assembly. Biomaterials. 30 (34), 6581-6592 (2009).
- Saeidi, N., Sander, E. A., Zareian, R., Ruberti, J. W. Production of highly aligned collagen lamellae by combining shear force and thin film confinement. Acta Biomaterialia. 7 (6), 2437-2447 (2011).
- Ahmed, A., et al. Microengineered 3D collagen gels with independently tunable fiber anisotropy and directionality. Advanced Materials Technologies. 6 (4), 2001186 (2021).
- Ahmed, A., et al. Local extensional flows promote long-range fiber alignment in 3D collagen hydrogels. Biofabrication. 14 (3), 035019 (2022).
- Mansouri, M., et al. The modular µSiM reconfigured: Integration of microfluidic capabilities to study in vitro barrier tissue models under flow. Advanced Healthcare Materials. , (2022).
- Paten, J. A., et al. Flow-induced crystallization of collagen: a potentially critical mechanism in early tissue formation. ACS Nano. 10 (5), 5027-5040 (2016).
- Liu, Y., Eliceiri, K. W. Quantifying fibrillar collagen organization with curvelet transform-based tools. Journal of Visualized Experiments. (165), e61931 (2020).
- Bredfeldt, J. S., et al. Automated quantification of aligned collagen for human breast carcinoma prognosis. Journal of Pathology Informatics. 5 (1), 28 (2014).
- Bredfeldt, J. S., et al. Computational segmentation of collagen fibers from second-harmonic generation images of breast cancer. Journal of Biomedical Optics. 19 (1), 016007 (2014).
- Carey, S. P., et al. Local extracellular matrix alignment directs cellular protrusion dynamics and migration through Rac1 and FAK. Integrative Biology. 8 (8), 821-835 (2016).
- Carey, S. P., Kraning-Rush, C. M., Williams, R. M., Reinhart-King, C. A. Biophysical control of invasive tumor cell behavior by extracellular matrix microarchitecture. Biomaterials. 33 (16), 4157-4165 (2012).
- Ahmed, A., et al. Engineering fiber anisotropy within natural collagen hydrogels. AmericanJournal of Physiology-Cell Physiology. 320 (6), 1112-1124 (2021).
- Mohammadi, H., Janmey, P. A., McCulloch, C. A. Lateral boundary mechanosensing by adherent cells in a collagen gel system. Biomaterials. 35 (4), 1138-1149 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon