Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Применение неконтролируемого мультиомного факторного анализа для выявления закономерностей вариаций и молекулярных процессов, связанных с сердечно-сосудистыми заболеваниями
В этой статье
Резюме
Мы представляем гибкий, расширяемый рабочий процесс на основе Jupyter-lab для неконтролируемого анализа сложных мультиомных наборов данных, который сочетает в себе различные этапы предварительной обработки, оценку модели мультиомного факторного анализа и несколько последующих анализов.
Аннотация
Механизмы заболевания обычно сложны и регулируются взаимодействием нескольких различных молекулярных процессов. Сложные, многомерные наборы данных являются ценным ресурсом для получения более подробной информации об этих процессах, но анализ таких наборов данных может быть сложным из-за высокой размерности, возникающей, например, в результате различных состояний заболевания, временных точек и омиксов, фиксирующих процесс с разным разрешением.
В этой статье мы демонстрируем подход к анализу и исследованию такого сложного мультиомного набора данных без учителя путем применения мультиомического факторного анализа (MOFA) к набору данных, созданному из образцов крови, которые отражают иммунный ответ при острых и хронических коронарных синдромах. Набор данных состоит из нескольких анализов с различным разрешением, включая данные цитокинов на уровне образца, плазмо-протеомику и прайм-секвенирование нейтрофилов, а также данные РНК-секвенирования одиночных клеток (scRNA-seq). Еще больше усложняется тем, что для каждого пациента измеряется несколько разных временных точек, а также несколько подгрупп пациентов.
Рабочий процесс анализа описывает, как интегрировать и анализировать данные в несколько этапов: (1) предварительная обработка и гармонизация данных, (2) оценка модели MOFA, (3) последующий анализ. В шаге 1 описывается, как обрабатывать признаки различных типов данных, отфильтровывать признаки низкого качества и нормализовать их для гармонизации их распределений для дальнейшего анализа. На шаге 2 показано, как применить модель MOFA и изучить основные источники дисперсии в наборе данных по всем омикам и признакам. На шаге 3 представлено несколько стратегий для последующего анализа захваченных паттернов, связывающих их с заболеваниями и потенциальными молекулярными процессами, управляющими этими состояниями.
В целом, мы представляем рабочий процесс для неконтролируемого исследования сложных мультиомиксных наборов данных, чтобы обеспечить идентификацию основных осей вариации, состоящих из различных молекулярных особенностей, которые также могут быть применены к другим контекстам и мультиомиксным наборам данных (включая другие анализы, представленные в примере использования).
Введение
Механизмы заболевания обычно сложны и регулируются взаимодействием нескольких различных молекулярных процессов. Расшифровка сложных молекулярных механизмов, которые приводят к конкретным заболеваниям или управляют эволюцией болезни, является задачей с высокой медицинской значимостью, поскольку она может открыть новые идеи для понимания и лечения заболеваний.
Последние технологические достижения позволяют измерять эти процессы с более высоким разрешением (например, на уровне отдельных клеток) и в то же время на различных биологических уровнях (например, ДНК, мРНК, доступность хроматина, метилирование ДНК, протеомика). Это приводит к увеличению количества больших многомерных наборов биологических данных, которые могут быть совместно проанализированы для получения более глубокого понимания лежащих в их основе процессов. В то же время объединение и анализ различных источников данных биологически значимым образом остается сложной задачей1.
Различные технологические пределы, шумы и диапазоны изменчивости между различными омиксами представляют собой одну проблему. Например, данные секвенирования РНК отдельных клеток (scRNA-seq) очень скудны и часто подвержены влиянию больших технических или пакетных эффектов. Кроме того, пространство признаков часто бывает очень большим, варьируясь от нескольких тысяч измеренных генов или белков, в то время как размеры выборки ограничены. Это еще больше осложняется сложными конструкциями, которые могут включать в себя несколько состояний заболевания, смешанные факторы, временные точки и решения. Например, в представленном сценарии использования различные типы данных были доступны либо на уровне одной ячейки, либо на уровне выборки (объема). Кроме того, данные могут быть неполными, и не все измерения могут быть доступны для всех анализируемых субъектов.
Из-за этих проблем различные омиксы и включенные в них особенности по-прежнему часто анализируются только по отдельности2, хотя выполнение интегрированного анализа не только может дать полную картину процесса, но и биологические и технические шумы от одной омики также могут быть компенсированы другими омиксами 3,4. Для выполнения комплексного анализа мультиомиксных данных было предложено несколько различных методов, включая байесовские методы, сетевые методы 5,6, мультимодальное глубокое обучение7 и методы уменьшения размерности с помощью матричной факторизации 8,9. Что касается последнего, то результаты большого сравнительного исследования10 показали, что метод MOFA9 (мультиомический факторный анализ) является одним из наиболее подходящих инструментов, когда данные должны быть увязаны с клиническими аннотациями.
Особенно в сложных условиях методы факторизации матрицы без учителя являются полезным подходом для снижения сложности и извлечения общих и дополнительных сигналов из различных источников данных и функций. Разложив сложное пространство на латентные представления более низкого ранга, можно быстро изучить основные источники дисперсии в данных и связать их с известными ковариатами. В случае, если один и тот же паттерн вариаций является общим для нескольких признаков (например, генов или белков), это может быть агрегировано до нескольких факторов, в то время как шум уменьшается. Регуляризация может быть использована для увеличения разреженности коэффициентов модели, что делает этот подход хорошо подходящим в условиях, где пространство признаков велико, а количество выборок ограничено9.
Этот протокол представляет собой гибкий рабочий процесс анализа, который использует модель MOFA для демонстрации того, как быстро изучить сложный мультиомный набор данных и выделить основные закономерности вариаций, характеризующие этот набор данных. Рабочий процесс состоит из трех основных этапов. На первом этапе, «Предварительная обработка и гармонизация данных», представлены различные стратегии предварительной обработки данных, основанные на различных типах входных данных (scRNA-seq, протеомика, цитокин, клинические данные). В протоколе подробно описывается, как обрабатывать признаки различных входных наборов данных, отфильтровывать признаки низкого качества и нормализовать их для гармонизации их распределений. Мы также показываем, как эти решения по предварительной обработке могут повлиять на последующие результаты. На втором этапе к данным применяется модель MOFA, и полученная дисперсионная декомпозиция может быть использована для оценки интеграции различных наборов данных. На третьем шаге показано, как связать захваченные факторы с ковариатами и раскрыть молекулярные программы, определяющие эти факторы. С помощью представленного рабочего процесса мы смогли извлечь несколько латентных факторов, связанных с клиническими ковариатами, в наборе данных пациентов, страдающих коронарными синдромами, и идентифицировать потенциальные лежащие в основе многоклеточные иммунные программы изпредыдущего проекта. Здесь мы будем использовать этот набор данных, но протокол можно легко применить к другим контекстам, включая другие омики.
Набор данных состоит из образцов пациентов со стабильными хроническими коронарными синдромами (ХКС), острыми коронарными синдромами (ОКС) и контрольной группы со здоровыми коронарными артериями (без ХКС) (рис. 1). ОКС вызывается разрывом бляшек в уже существующем ССХ, что приводит к острому нарушению притока крови к миокарду и последующему ишемическому повреждению сердца. Это повреждение вызывает воспалительную реакцию иммунной системы, за которой следует репаративная фаза, которая длится в течение нескольких дней после острого события12. Чтобы иметь возможность охарактеризовать этот иммунный ответ у пациентов с ОКС, образцы крови были взяты в четыре разных временных момента: острый (TP1); после реканализации (14 [± 8] ч) (TP2); через 60 [± 12] ч (TP3); до выписки (6,5 [±1,5] дней) (TP4) (рисунок 1A). Для ССК и пациентов со здоровыми коронарными артериями была доступна только одна временная точка - (TP0). Для всех пациентов и временных точек на основе образцов крови были измерены различные анализы: клинические маркеры воспаления (креатин-киназа (КФК), КФК-МБ, тропонин, С-реактивный белок (СРБ)), scRNA-seq мононуклеарных клеток периферической крови (PBMCs), цитокиновый анализ, плазменная протеомика и данные нейтрофилов prime-seq13 .
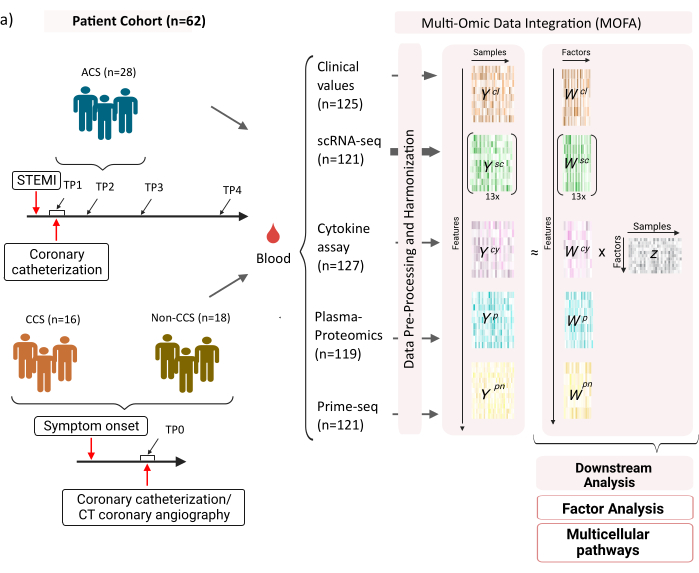
Рисунок 1: Мультиомный входной набор данных по инфаркту миокарда. Входной набор данных: Анализируемые данные включают образцы крови пациентов (n = 62) с острым коронарным синдромом (ОКС), хроническими коронарными синдромами (ХКС) и пациентами со здоровыми коронарными артериями (без ОАС). У пациентов с ОКС образцы крови были включены в четыре разных временных момента (TP1-4), у пациентов с CCS и без CCS в одну временную точку (TP0). Каждый пациент и комбинация временных точек рассматривается как отдельный образец в анализе. На образцах были проведены различные омические анализы: клинические анализы крови (n = 125), scRNA-seq (n = 121), плазма-протеомика (n = 119), цитокиновый анализ (n = 127) и нейтрофильный прайм-секвен (n = 121). Впоследствии описанный протокол был применен для интеграции данных по всем омиксам и их изучения с использованием модели MOFA и дальнейшего анализа (факторный анализ, обогащение путей). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
В качестве входных данных для рабочего процесса, представленного здесь, мы берем исходные подсчеты из данных scRNA-seq после обработки с помощью cellranger и контроля качества (QC), как, например, описано в учебном пособии по предварительной обработке scanpy14 . Для аннотирования ячеек мы использовали автоматизированный конвейер Azimuth15 . Затем подсчеты агрегируются на уровне выборки для каждого типа клеток путем вычисления среднего значения по всем ячейкам для каждой выборки и типа клеток (псевдообъемная агрегация). Плазменная протеомика включена в виде нормализованной и медиан-центрированной интенсивностей, а для нейтрофилов мы берем количество экзонов уникального молекулярного идентификатора UMI (UMI) из прайм-секвенирования. Что касается цитокинов и клинических показателей, предыдущая предварительная обработка не применялась. Более подробная информация о (экспериментальной) генерации данных изложена в соответствующей рукописи11. Поскольку представленные здесь результаты основаны на использовании автоматизированной аннотации азимута для типов клеток в данных scRNA-seq по сравнению со стратегией, основанной на маркерах, которая использовалась в упомянутой публикации, представленные здесь результаты аналогичны, но не полностью совпадают с теми, которые представлены в публикации. В рукописи можно показать, что стратегия аннотации по типам клеток не изменяет основных закономерностей и биологических интерпретаций анализа, но небольшие изменения в точных значениях, полученных в результате модели, могут варьироваться. В целом, входные данные представляли собой сложный многомерный набор данных, включающий различные временные точки и уровни измерения (одиночные клетки против объемных) более чем 10 000 различных признаков (генов, белков, клинических значений). Было показано, что строгая стратегия предварительной обработки и гармонизации данных с последующим анализом MOFA является полезным и быстрым инструментом для изучения данных и извлечения соответствующей иммунной программы. Каждая временная точка и комбинация пациентов рассматриваются как независимая выборка в анализе MOFA. Каждый тип данных и тип ячейки рассматривается как отдельное представление в анализе MOFA.
Этот протокол содержит инструкции по подготовке входных данных для рабочего процесса, выполнению различных этапов рабочего процесса, настройке конфигураций, интерпретации полученных цифр и итеративной корректировке конфигураций на основе интерпретаций. Обзор различных этапов протокола, необходимых входных наборов данных на каждом шаге, а также результирующих рисунков и наборов данных представлен в обзоре технического рабочего процесса (рисунок 2).
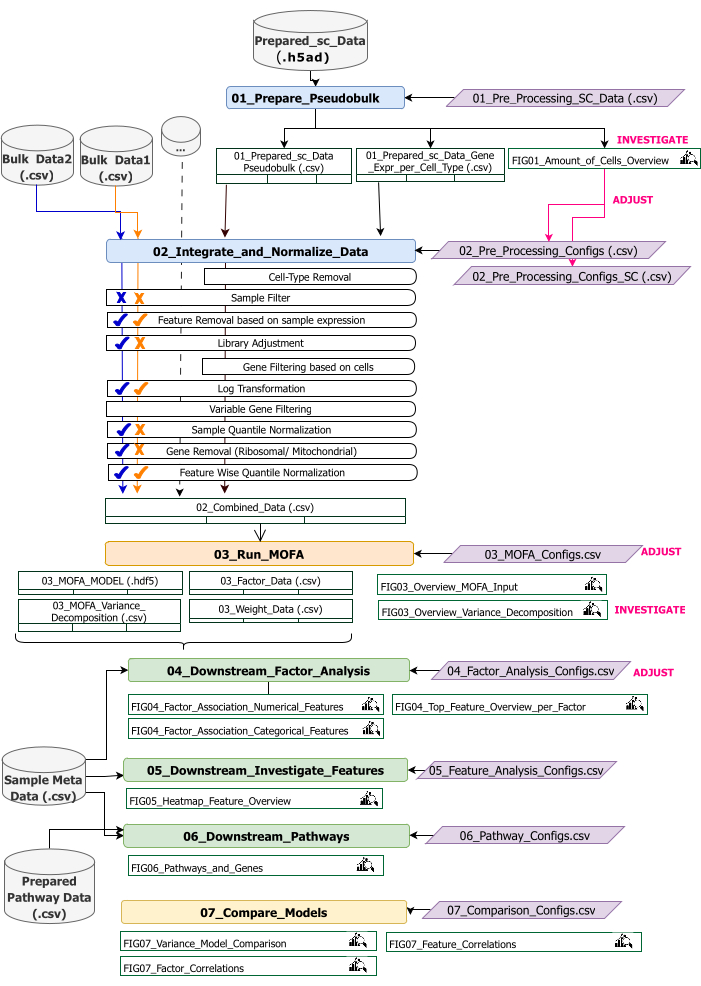
Рисунок 2: Обзор технического рабочего процесса. Схема рабочего процесса для анализа набора мультиомических данных. Разные элементы выделены разными цветами и символами. Записные книжки Jupyter, относящиеся к этапу Предварительная обработка и гармонизация данных (1), окрашены в синий цвет. Блокноты Jupyter, относящиеся к шагу «MOFA Model» (2), окрашены в оранжевый цвет. Записные книжки Jupyter, относящиеся к шагу «Анализ на последующих этапах» (3), окрашены в зеленый цвет. Один из блокнотов Jupyter, который будет использоваться для сравнения результатов, окрашен в желтый цвет. Файлы конфигурации, в которых можно изменить параметры для выполнения расчетной схемы, выделены фиолетовым цветом. Входные наборы данных, необходимые для запуска рабочего процесса, обозначены символом набора данных и выделены серым цветом. Все выходные данные рисунка, которые генерируются во время выполнения рабочего процесса, обозначаются символом лупы. Наборы данных, созданные во время выполнения расчетной схемы, обозначаются как таблицы. В общем случае рабочий процесс выполняется последовательно: (1) Предварительная обработка и гармонизация данных состоит из двух этапов: сначала генерируется псевдообъемная таблица на основе входных данных scRNA-seq (01_Prepare_Pseudobulk) и последующая интеграция и нормализация этих данных вместе со всеми остальными входными данными уровня выборки (объемными) (02_Integrate_and_Normalize_Data). На этом шаге с помощью конфигурационных файлов можно настроить для каждого набора данных отдельно, какой из указанных шагов предварительной обработки и нормализации (например, фильтр выборки) должен быть применен. (2) «MOFA Model»: запускает модель MOFA на сгенерированных входных данных первого шага с конфигурациями, указанными в файле конфигурации (03_MOFA_configs.csv) (3) «Downstream Analysis»: состоит из трех различных блокнотов, которые могут быть запущены независимо друг от друга для получения аналитических сведений о сгенерированных результатах MOFA и связывания их с образцами метаданных (ковариатами), предоставленными в качестве входных данных через файл «Sample Meta Data.csv». (4) «Сравнение моделей»: это небольшой отдельный шаг, который может быть использован для сравнения различных моделей, созданных на шаге 2. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Рабочий процесс состоит из нескольких записных книжек Jupyter, написанных на R и Python (знание языков R и Python не требуется для запуска рабочего процесса, но может быть полезно в случае появления ошибок). На различных этапах протокола изменение параметров осуществляется с помощью конфигурационных файлов (файлы '.csv, содержащие в имени постфикс '_Configs'). В протоколе мы только выделяем параметры, которые необходимо изменить, начиная с конфигурации по умолчанию.
Некоторые другие параметры также могут быть изменены, например, для настройки предварительной обработки. Документация по этим параметрам и пояснения приведены в файле 'Documentation_Config_Parameter', который включен в загруженный репозиторий.
протокол
1. Подготовка: Техническая настройка и монтаж
ПРИМЕЧАНИЕ: Чтобы запустить эту программу, на устройстве должны быть предустановлены wget, git и Apptainer. Инструкция по установке Apptainer на разные системы (Linux, Windows, Mac) приведена здесь: https://apptainer.org/docs/admin/main/installation.html. Информацию об установке git можно найти здесь: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git. В зависимости от размера различных входных наборов данных рекомендуется запускать рабочий процесс на подходящем компьютере (16 ЦП, 64 ГБ памяти). Дымовой тест с предоставленными примерами данных может быть выполнен на локальном компьютере. Инструкции и ожидаемые результаты выполнения протокола на данных примера приведены в дополнительном файле 1. В дополнительном видеофайле 1 приведены важные шаги протокола, которые выполняются на описанном выше наборе данных.
- Откройте консоль и выберите или создайте папку, в которой будет храниться весь код анализа и выходные данные. Перейдите в папку, набрав в терминале команду: cd path_to_folder.
- Скачайте или клонируйте репозиторий кода с Github (https://github.com/heiniglab/mofa_workflow) или набрав git clone https://github.com/heiniglab/mofa_workflow.git в окне терминала.
- Загрузите образ, содержащий все необходимые установки от Zenodo, набрав wget https://zenodo.org/records/11192947/files/mofa_image.sif в окне терминала.
- Сгенерируйте папку, в которой будут храниться все данные о результатах, набрав mkdir results в окне терминала.
- Сгенерируйте папку, в которую будут добавлены все входные данные, которые будут использоваться в анализе, набрав в окне терминала команду mkdir input_data .
- Запустите контейнер, который запустит сессию JupyterLab, введя в терминале следующую команду: apptainer run mofa_image.sif. Скопируйте URL-адрес, возвращенный командой, в браузер, который откроет сеанс Jupyter-lab (более подробную информацию о Jupyter-lab можно найти в документации по программному обеспечению16).
ПРИМЕЧАНИЕ: Когда рабочий процесс выполняется локально на ноутбуке, рекомендуется использовать команду apptainer exec mofa_image.sif jupyter-lab , которая напрямую вернет адрес локального хоста. В случае, если контейнер выполняется в кластерной вычислительной среде, может потребоваться настроить переадресацию портов, что можно сделать через ssh.
2. Инициализация и подготовка данных
- В сеансе Jupyter-Lab используйте меню навигации слева. Перейдите в папку input_data , дважды кликнув по input_data.
- Скопируйте все наборы данных, которые будут использоваться в качестве входных данных для анализа, в директорию input_data с помощью Drag&Drop. Перетащите файл из папки, в которой он находится в данный момент, и поместите его в сеанс Jupyter-lab в область под папкой input_data.
ПРИМЕЧАНИЕ: Все наборы данных должны быть либо в формате .csv , либо в формате .h5ad (в случае данных с одной ячейкой). Все .csv файлы должны содержать соответствующий столбец sample_id (во всех наборах данных должны использоваться идентичные идентификаторы). Все остальные столбцы будут использоваться в качестве характеристик. В файле h5ad аннотация ячейки должна содержать два идентификатора, указывающих sample_id и cluster_id. Они будут использоваться для агрегирования и сопоставления. Наборы данных Omic в других форматах необходимо преобразовать в указанный формат .csv перед использованием (рис. 3). Наборы данных scRNA-seq, предоставленные в формате .h5seurat , могут быть преобразованы в .h5ad , выполняя Jupyter-notebook: 00_Data_Conversion.ipynb. - Перейдите в папку конфигураций , щелкнув символ папки , а затем дважды щелкнув папки mofa_workflow, скрипты и конфигурации. В папке откройте файл Data_configs.csv , дважды кликнув по нему.
- В столбце value добавьте пути к папкам папок input_data (data_path) и results (result_path). Добавьте имя, которое будет добавлено в качестве расширения ко всем сохраненным файлам, в столбце значений для configuration_name (этот протокол использовался MI_v1 [Myocardial Infarction version1]) (рис. 4).
- Сохраните изменения, нажав на «Файл» > «Сохранить файл CSV » в меню вверху.
- Используйте меню навигации слева для перехода в папку скриптов, нажав на скрипты. Откройте записную книжку инициализации, дважды щелкнув по 00_Configuration_Update.ipynb. Выполните скрипт, нажав на кнопку «Перезагрузить ядро и запустить все ячейки» вверху и нажав «Перезапустить» во всплывающем окне (рисунок 5).
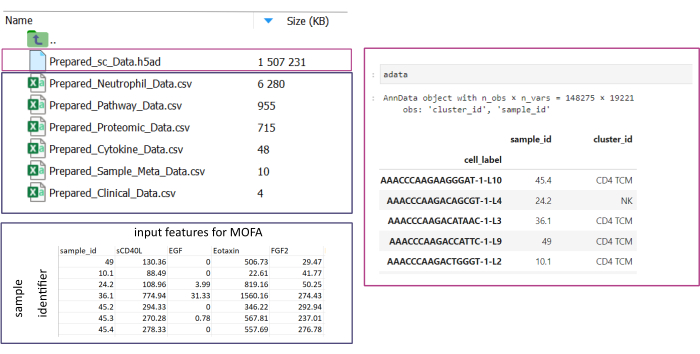
Рисунок 3: Ввод и настройка данных. Для выполнения рабочего процесса все данные должны храниться в указанной папке input_data. Для каждого входного набора данных должен быть предоставлен отдельный файл. Данные по отдельным ячейкам должны быть представлены в виде .h5ad, содержащего аннотацию к ячейке на cluster_id (полученную, например, в результате предыдущих шагов аннотации типа ячейки) и столбец sample_id (однозначно идентифицирующий каждую отдельную выборку, которая должна быть проанализирована). Все остальные входные наборы данных должны быть предоставлены в формате '.csv', включая один столбец, указывающий sample_id (соответствие соответствующему столбцу данных одной ячейки) и признаки, которые будут использоваться при анализе MOFA во всех остальных столбцах. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
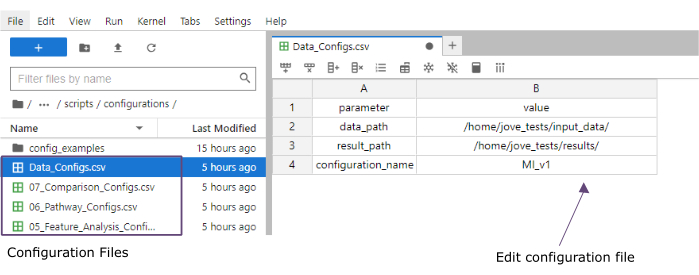
Рисунок 4: Конфигурационные файлы Jupyter-lab. Во время выполнения расчетной схемы изменения параметров (например, настройка параметров фильтрации и т. д.) указываются через файлы конфигурации «.csv». В клонированный репозиторий включаются файлы конфигурации по умолчанию для каждого шага. Они могут быть отредактированы непосредственно в консоли jupyter-lab, аналогично электронной таблице. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Рисунок 5: Скрипты Jupyter-notebooks. Полный рабочий процесс состоит из серии записных книжек Jupyter, которые будут выполняться последовательно после изменения соответствующих файлов конфигурации. При двойном щелчке мыши по записной книжке Jupyter с левой стороны соответствующий файл будет открыт с правой стороны. Полное выполнение файла можно запустить с помощью кнопки, выделенной вверху. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
3. Предварительная обработка и гармонизация данных
- Предварительная обработка - Преобразование данных sc в pseudobulk.
ПРИМЕЧАНИЕ: Этот шаг необходимо выполнять только в том случае, если в анализе используются данные одной ячейки.- Используйте меню навигации слева для перехода в папку с конфигурациями, дважды щелкнув по конфигурации. Откройте файл 01_Preprocessing_SC_Data.csv двойным щелчком мыши. Проверьте автоматически заполненные значения в файле и, при необходимости, настройте значения в столбце data_name в соответствии с именами файлов наборов данных с одной ячейкой в папке input_data , которая будет использоваться для анализа.
ПРИМЕЧАНИЕ: По умолчанию все имена файлов .h5ad в папке входных данных будут добавлены в файл конфигурации в скрипте инициализации. Если некоторые наборы данных не должны использоваться для анализа, их можно удалить здесь. - Сохраните внесенные изменения, нажав на «Файл» > «Сохранить файл CSV » в меню вверху.
- Используйте меню навигации слева для перехода в папку скриптов , нажав на скрипты. Откройте записную книжку 01_Prepare_Pseudobulk.ipynb , дважды кликнув по ней. Выполните скрипт, нажав на кнопку «Перезагрузить ядро и запустить все ячейки » вверху и нажав «Перезапустить » во всплывающем окне.
- Используйте меню навигации слева для перехода в папку с рисунками, дважды щелкнув сначала по рисункам , а затем по 01_figures. Откройте только что сгенерированный FIG01_Amount_of_Cells_overview графика, дважды кликнув по нему.
ПРИМЕЧАНИЕ: Выполнение записной книжки может занять несколько минут. Когда блокнот будет выполнен успешно, появится всплывающее окно, и файл FIG01_Amount_of_Cells_Overview будет либо обновлен блокнотом, либо сгенерирован заново. В столбце «Последнее изменение » можно указать, когда был создан файл, чтобы оценить, является ли он новым или старым. - Исследуйте график для выявления кластеров клеточного типа с очень низким числом клеток в образце. Запишите имена этих cluster_ids чтобы исключить их на последующих шагах (рисунок 6).
- Используйте меню навигации слева, чтобы вернуться в папку с настройками, нажав на ... А затем двойной клик по настройкам. Откройте файл 02_Preprocessing_Configs_SC.csv , дважды кликнув по нему.
- Проверьте значения в столбцах configuration_name и data_name и при необходимости скорректируйте их.
ПРИМЕЧАНИЕ: В сценарии инициализации эти значения предварительно заполняются всеми именами файлов .h5ad в папке входных данных и значением configuration_name , установленным ранее в файле Data_Configs.csv . В случае, если файлы должны быть исключены из анализа или должно быть использовано другое расширение для имен файлов, это можно настроить здесь. - Измените значение в столбце cell_type_exclusion и добавьте все cluster_id, которые были определены для исключения на предыдущем шаге, разделив их знаком ",".
- Сохраните изменения, нажав « Файл» > «Сохранить файл CSV » на панели навигации вверху.
- Используйте меню навигации слева для перехода в папку с конфигурациями, дважды щелкнув по конфигурации. Откройте файл 01_Preprocessing_SC_Data.csv двойным щелчком мыши. Проверьте автоматически заполненные значения в файле и, при необходимости, настройте значения в столбце data_name в соответствии с именами файлов наборов данных с одной ячейкой в папке input_data , которая будет использоваться для анализа.
- Предварительная обработка - Гармонизация и интеграция других источников омиксных данных.
- Откройте файл 02_Preprocessing_Configs.csv , дважды кликнув по нему, и настройте конфигурацию предварительной обработки для каждого из наборов данных, которые будут включены и хранятся в папке data_input (по одной строке на набор данных).
- Проверьте значения в столбцах configuration_name и data_name и при необходимости скорректируйте их.
- Настройте остальные параметры в столбцах соответствующим образом, в зависимости от того, какие шаги предварительной обработки должны быть применены.
ПРИМЕЧАНИЕ: Значения по умолчанию добавляются для каждого набора данных, найденного в папке input_dataset, но не специфичны для отдельных типов данных. Поэтому будут необходимы корректировки. Подробная документация параметров приведена в файле Documentation_Config_Parameter.doc . - Сохраните изменения, нажав « Файл» > «Сохранить файл CSV».
- Используйте меню навигации слева для перехода в папку скриптов , нажав на скрипты. Откройте записную книжку 02_Integrate_and_Normalized_Data_Sources.ipynb , дважды кликнув по ней. Выполните скрипт, нажав на кнопку «Перезагрузить ядро и запустить все ячейки » вверху и нажав «Перезапустить » во всплывающем окне.
- Используйте меню навигации слева для перехода к созданной папке 02_results , нажав на символ папки , а затем дважды щелкнув по результатам и 02_results. Проверьте, есть ли в нем файл 02_Combined_data_'configuration_name'_Integrated.csv , содержащий объединенный входной файл предварительно обработанных данных.
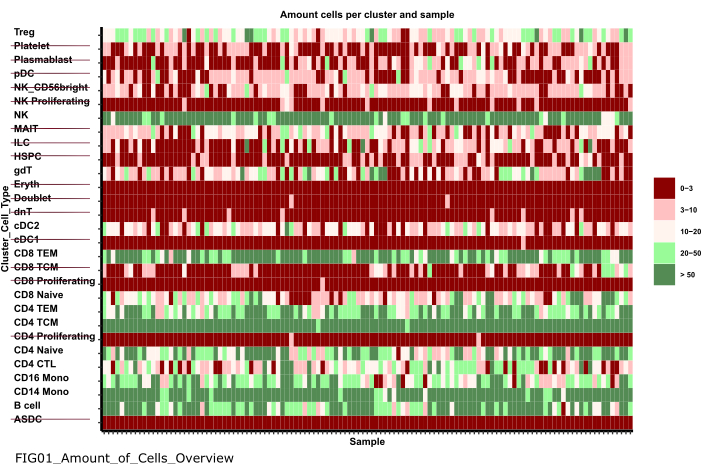
Рисунок 6: Предварительная обработка и гармонизация данных. Одним из выходных данных шага '01_Prepare_Pseudobulk' является график 'Fig01_Amount_of_Cells_Overview'. Здесь для каждого cluster_id (ось Y, указывающая на тип ячейки из предыдущих шагов аннотации типа ячейки) указывается количество ячеек на выборку («sample_id»). В рамках представленных результатов типы клеток с малым количеством клеток в образце исключаются из последующего анализа (обозначаются зачеркиванием). Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
4. Запуск MOFA
- В Jupyter-Lab используйте меню навигации слева для перехода к папке конфигураций, щелкнув по символу папки, а затем дважды кликнув по mofa_workflow, а затем дважды кликнув по скриптам и конфигурациям. Откройте файл 03_MOFA_Configs.csv, дважды кликнув по нему.
- Проверьте записи на наличие столбцов configuration_name и mofa_result_name и скорректируйте записи, если следует использовать альтернативные имена.
ПРИМЕЧАНИЕ: mofa_result_name будет добавлен в качестве расширения ко всем файлам результатов, созданным на основе MOFA. Это значение может отличаться от значения configuration_name , так как разные настройки MOFA могут выполняться с одними и теми же входными данными (этот протокол использует MI_v1_MOFA). - Введите количество факторов, которые должны быть оценены в модели MOFA (amount_of_factors столбец), и определите, следует ли применять взвешивание и масштабирование (столбцы weighting_of_views и scale_views ), скорректировав значения в файле.
- Сохраните изменения, нажав « Файл» > «Сохранить файл CSV».
- Используйте меню навигации слева, чтобы перейти в папку скриптов , нажав на «скрипты». Откройте блокнот 03_Run_MOFA.ipynb , дважды кликнув по файлу. Запустите скрипт, нажав на кнопку «Перезагрузить ядро и запустить все ячейки » вверху и нажав «Перезапустить » во всплывающем окне.
- Перейдите в папку 03_figures , дважды кликнув по цифрам , а затем 03_figures. Откройте сгенерированный график FIG03_Overview_Variance_Decomposition_ mofa_result_name и изучите результат модели (рис. 7A).
- Используйте меню навигации слева для перехода к созданной папке 03_results , нажав на символ папки , а затем дважды щелкнув по результатам и 03_results. Проверьте, включает ли он файл значений коэффициента выборки 03_Factor_Data_'mofa_result_name'.csv и файл веса фактора признака 03_Weight_Data_'mofa_result_name'.csv.
5. Анализ на последующих этапах
- Факторная интерпретация.
- Используйте меню навигации с левой стороны, чтобы перейти к папке input_data , нажав на символ папки , а затем дважды кликнув по input_data.
- Подготовьте файл .csv (Prepared_Sample_Meta_Data.csv), содержащий все метаданные (ковариаты) образцов, которые будут анализироваться в связи с сгенерированными факторами. Скопируйте файл в папку input_data с помощью Drag&Drop , перетащив файл в обзор папки input_data .
ПРИМЕЧАНИЕ: Он должен содержать столбец sample_id для сопоставления его с ранее использованными данными, а также дополнительные столбцы для каждого объекта, который следует анализировать. - В Jupyter-Lab используйте меню навигации слева, чтобы вернуться к папке конфигураций, щелкнув символ папки , а затем дважды щелкнув mofa_workflow, а затем перейдя к скриптам и конфигурации. Откройте файл 04_Factor_Analysis.csv , дважды кликнув по нему.
- Убедитесь, что записи для configuration_name и mofa_result_name содержат названия конфигурации и результаты MOFA, которые будут анализироваться в скрипте, и при необходимости скорректируйте их.
- В столбец numeric_covariates добавьте имена всех числовых столбцов в файле Prepared_Sample_Meta_Data.csv, которые будут исследоваться в отношении факторов MOFA, разделенных запятой (этот протокол использует CRP,CK).
- В столбец categorical_covariates добавьте названия всех категориальных столбцов в файле Prepared_Sample_Meta_Data.csv , которые будут исследоваться по отношению к факторам MOFA через запятую (в этом протоколе используется измерение).
- Сохраните изменения, нажав на «Файл» > «Сохранить файл CSV».
- Используйте меню навигации слева для перехода в папку «скрипты», нажав на «скрипты». Откройте блокнот 04_Downstream_Factor_Analysis.ipynb , дважды кликнув по нему. Запустите скрипт, нажав на кнопку «Перезапустить ядро и запустить все ячейки » вверху и нажав «Перезапустить» во всплывающем окне.
- Используйте меню навигации слева для перехода к папке 04_figures , дважды щелкнув по цифрам , а затем 04_figures. Откройте сгенерированные графики, дважды кликнув по ним, и исследуйте факторы на предмет интересных закономерностей и ассоциаций: FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf (Рисунок 7B). FIG04_Factor_Association_
with_categorical_features_'mofa_result_name.pdf (Рисунок 7C). FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf (рис. 8A).
- Анализ признаков
- Используйте меню навигации слева, чтобы вернуться в папку с настройками, нажав на ... А затем двойной клик по настройкам. Откройте файл 05_Feature_Analysis_Configs.csv , дважды кликнув по нему.
- Убедитесь, что записи для столбцов configuration_name и mofa_result_name соответствуют названиям конфигурации и сгенерированному результату MOFA, которые будут использоваться для последующего анализа, и при необходимости скорректируйте их.
- В столбце коэффициента добавьте коэффициент, для которого будут построены верхние объекты в следующем скрипте.
- В столбце faceting_variable добавьте имя столбца категориального столбца в Prepared_Sample_Meta_Data.csv который будет использоваться для группировки выборок на графике (Этот протокол использует измерение)
- Сохраните изменения, нажав « Файл» > «Сохранить файл CSV».
- Используйте меню навигации слева для перехода в папку скриптов , нажав на скрипты. Откройте записную книжку 05_Downstream_Investigate_Features_Heatmap.ipynb , дважды кликнув по ней. Выполните скрипт, нажав на кнопку «Перезагрузить ядро и запустить все ячейки » вверху и нажав «Перезапустить » во всплывающем окне.
- Используйте меню навигации слева для перехода к папке 05_figures , дважды кликнув сначала по цифрам , а затем по 05_figures. Откройте и исследуйте сгенерированный сюжет FIG05_Heatmap_Feature_Overview__ 'mofa_result_name'.pdf , дважды щелкнув по файлу (Рисунок 8B).
ПРИМЕЧАНИЕ: В зависимости от количества функций, которые будут отображаться на графике, может потребоваться настроить параметры plot_width и plot_height в 05_Feature_Analysis_Configs.csv и повторно запустить скрипт, чтобы убедиться, что все помещается на графике.
- Анализ путей прохождения
- Используйте меню навигации с левой стороны, чтобы перейти к папке input_data , нажав на символ папки , а затем дважды кликнув по input_data.
- Подготовьте файл .csv (Prepared_Pathway_Data.csv), содержащий список путей, которые будут проверены на обогащение. Скопируйте файл в папку input_data с помощью Drag&Drop, перетащив файл в обзор папки input_data .
ПРИМЕЧАНИЕ: Он должен содержать три столбца: ID (уникальный идентификатор пути), gene (гены, обозначенные их именем гена (SYMBOL), принадлежащим пути, по одной строке на ген), pathway_name (название/текстовое описание путей). - В сеансе Jupyter-Lab используйте меню навигации слева для перехода к папке конфигураций, нажав на ... А затем двойной клик по настройкам. Откройте файл 06_Pathway_Configs.csv , дважды кликнув по нему.
- Проверьте запись в столбце mofa_result_name и убедитесь, что она соответствует имени сгенерированного результата MOFA, который будет использоваться для расчета обогащения пути.
- Проверьте запись в столбце types и удалите записи в столбце types , которые не содержат признаков, соответствующих столбцу gene в файле Prepared_Pathway_Data.csv .
ПРИМЕЧАНИЕ: По умолчанию все различные виды, которые использовались в модели MOFA, добавляются в этот файл во время выполнения рабочего процесса. В случае, если есть виды, которые не содержат объектов, соответствующих хотя бы одному пути, их необходимо удалить; В противном случае выполнение завершится ошибкой. Например, файл пути содержит только аннотации для генов, но есть представление, содержащее названия белков. - Сохраните изменения, нажав « Файл» > «Сохранить файл CSV».
- Используйте меню навигации для перехода в папку скриптов , нажав на скрипты. Откройте блокнот 06_Downstream_Pathways.ipynb , дважды кликнув по нему. Выполните скрипт, нажав на кнопку «Перезагрузить ядро и запустить все ячейки » вверху и нажав «Перезапустить » во всплывающем окне.
- Используйте меню навигации слева для перехода к папке 06_figures, сначала дважды кликнув по цифрам, а затем 06_figures. Откройте сгенерированный график FIG06_Pathways_and_Genes_ mofa_result_name, дважды щелкнув по нему, и изучите визуализированные пути (рисунок 8C).
ПРИМЕЧАНИЕ: Способ выбора визуализированных путей может быть сконфигурирован с помощью файла конфигурации. Для получения более подробной информации обратитесь к документации по параметрам. - Используйте меню навигации с левой стороны для перехода к сгенерированной папке 06_results , нажав на символ папки , а затем дважды щелкнув по результатам и 06_results. Проверьте, содержит ли он файл, включающий обогащенные пути 06_Pathway_enrichment__'mofa_result_name'.
6. Сравнение различных конфигураций и версий (Дополнительный рисунок 1, Дополнительный рисунок 2, Дополнительный рисунок 3, Дополнительный рисунок 4)
- Чтобы сравнить эффект от использования различных параметров/конфигураций на протяжении всего рабочего процесса, повторите разделы 3-5, изменив параметры в файлах конфигурации и используя другие идентификаторы configuration_name и mofa_result_name .
ПРИМЕЧАНИЕ: Новые результаты будут сохранены с этими именами, чтобы использовать их для сравнения различных прогонов. - В Jupyter-Lab используйте меню навигации слева для перехода в папку с настройками . Откройте файл 07_Comparison_Configs.csv , дважды кликнув по нему.
- В столбце mofa_result_name добавьте названия всех предыдущих запусков MOFA, которые будут сравниваться (по одной строке на имя/конфигурацию, например, MI_v1_MOFA, MI_v2_MOFA).
- В столбце compare_factors добавьте факторы, которые будут сравниваться между моделями. По умолчанию это Factor1,Factor2,Factor3. (Дополнительный рисунок 2А).
ПРИМЕЧАНИЕ: В этом скрипте значения признаков и факторов различных моделей будут сравниваться путем их корреляции. Это работает только для моделей, основанных на одних и тех же образцах (обозначенных sample_id) и одном и том же наборе функций. В случае, если образцы или функции не совпадают между сравниваемыми версиями, они будут исключены из сравнения. - Сохраните изменения, нажав « Файл» > «Сохранить файл CSV».
- Используйте меню слева для перехода в папку со скриптами , нажав на скрипты. Откройте записную книжку 07_Compare_Models.ipynb , дважды кликнув по ней. Выполните скрипт, нажав на кнопку «Перезагрузить ядро и запустить все ячейки » и нажав «Перезапустить » во всплывающем окне.
- Используйте меню слева для перехода к папке 06_figures , дважды кликнув сначала по цифрам , а затем по 06_figures. Откройте сгенерированные графики, дважды кликнув по файлам, чтобы проанализировать сходство разных версий:
FIG07_Variance_Model_Comparison.pdf (Дополнительный рисунок 2B)
FIG07_Factor_Correlations.pdf
(Дополнительный рисунок 2C)
FIG07_Feature_Correlations.pdf
(Дополнительный рисунок 3C)
7. Расширение рабочего процесса: добавление других параметров и конфигураций
ПРИМЕЧАНИЕ: Помимо параметров, которые в настоящее время можно настроить в файлах конфигурации, могут быть включены и другие изменения в коде или другие параметры. Например, сама модель MOFA предлагает несколько других обучающих параметров17 , которые могут быть либо изменены непосредственно в коде, либо настроены с помощью конфигурационных файлов. В следующем разделе протокола будет приведен пример того, как это сделать для дополнительных параметров обучения модели MOFA. Для этой части требуются знания программирования на R.
- В Jupyter-Lab используйте меню навигации слева для перехода в папку scripts . Откройте записную книжку 03_Run_MOFA.ipynb , дважды кликнув по ней.
- Нажмите на вкладку «Оглавление » слева, а затем перейдите к подразделу 4.3 «Установить параметры обучения MOFA» и запустите обучение модели , щелкнув по нему. Прокрутите вниз, чтобы увидеть печатный вывод модели MOFA с настраиваемыми параметрами в блокноте.
- В цикле R for в коде под заголовком задаются все данные MOFA, модель и параметры обучения. Ниже строки model_opts$num_factors = mofa_configs$amount_of_factors[i] добавьте еще одну строку с кодом ниже
model_opts$problihoods['data_type'] = 'пуассон'.
ПРИМЕЧАНИЕ: Это изменит распределение, которое модель принимает в качестве входных данных для представления, указанного именем data_type для всех прогонов MOFA. При указании пуассона для типа данных модель будет выполняться только в том случае, если признаки для этого типа данных являются целыми числами (например, счетчики прочтений из RNA-seq). Чтобы получить дополнительную информацию о данных MOFA, обучении и вариантах модели, можно также обратиться к учебным пособиям и документации MOFA17. - Сохраните изменения в блокноте, нажав на кнопку «Сохранить » вверху.
- Чтобы передать новые параметры через файлы конфигурации .csv , используйте навигацию слева, чтобы перейти в папку конфигураций , дважды кликнув по конфигурации, и откройте файл 03_MOFA_Configs.csv двойным щелчком мыши.
- Добавьте новую колонку с указанием имени параметра, например, number_iterations и введите значение, например, 1000. Сохраните изменения, нажав « Файл» > «Сохранить файл CSV».
- Используйте меню навигации для навигации по папке скриптов, нажав на скрипты. Откройте записную книжку 03_Run_Mofa.ipynb , дважды кликнув по ней. Нажмите на вкладку «Оглавление » слева, а затем перейдите к подразделу 4.3 «Установить параметры обучения MOFA» и запустите обучение модели , щелкнув по нему.
- Замените строку train_opts$maxiter = 50000 на train_opts$maxiter = mofa_configs$column_name[i] (когда имя добавленного столбца number_of_iterations это train_opts$maxiter = mofa_configs$number_of_iterations[i])).
ПРИМЕЧАНИЕ: Файл конфигурации, 03_MOFA_Configs.csv в этой записной книжке, считывается в начале записной книжки (подраздел: Предварительные требования, конфигурации и параметры) как mofa_config data.frame в сеансе, и поэтому в этой строке кода содержится ссылка на этот объект и соответствующий вновь сгенерированный столбец. Поскольку несколько конфигураций могут быть запущены одновременно, i идентифицирует строку data.frame, поскольку оценка модели выполняется в цикле for для всех различных строк в файле .csv . Принцип чтения в файле конфигурации в начале записной книжки в разделе «Предварительные требования, конфигурации и параметры» одинаков для всех записных книжек, и дальнейшие модификации могут производиться таким образом. - Сохраните изменения в блокноте, нажав на кнопку Сохранить .
Результаты
После успешного выполнения рабочего процесса создается несколько таблиц и рисунков, как показано на рисунке 2. Рисунки помещаются в папку /figures (Рисунок 6, Рисунок 7, Рисунок 8, Дополнительный ...
Обсуждение
С помощью описанного протокола представлен модульный и расширяемый рабочий процесс на основе Jupyter-notebook, который можно использовать для быстрого изучения сложного мультиомного набора данных. Основные части рабочего процесса состоят из предварительной обработки и га...
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов.
Благодарности
C.L. поддерживается Ассоциацией имени Гельмгольца в рамках совместной исследовательской школы "Munich School for Data Science - MUDS".
Материалы
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
Ссылки
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены