Method Article
Kriyo-elektron Tomogramlarında aktin ağlarını segmentlere ayırmak için etkili bir strateji olarak basitleştirilmiş hacimsel modeller
Bu Makalede
Özet
Burada, basitleştirilmiş hacimsel modelleri gürültülü, karmaşık, tomografik 3D hacimlere yerleştirmek için bir protokol sunuyoruz. Bu, aktin filament yoğunluklarının hızlı bir şekilde segmentasyonuna, sistematik filament bükülmesinin ve saç demeti filamentlerindeki boşlukların tespit edilmesine ve ayrıca mesafeler gibi hacimsel model özelliklerinin uygun şekilde ölçülmesine olanak tanır.
Özet
İlgilenilen özelliklerin çıkarılması için etkili yöntemler, kriyo-elektron tomogramlarının yorumlanması için en büyük zorluklardan biri olmaya devam etmektedir. Birçoğu, ilgilenilen özelliklerin kolayca tespit edilebildiği ve birbirinden net bir şekilde ayrıldığı yüksek kontrastlı veri kümeleri için iyi çalışan çeşitli otomatik yaklaşımlar önerilmiştir. İç kulak stereosilia kriyo-elektron tomografik veri kümelerimiz, sıklıkla çapraz bağlanan, altıgen olarak paketlenmiş yoğun bir aktin filamentleri dizisi ile karakterize edilir. Bu özellikler, kriyo-elektron tomografilerinin yüksek gürültülü ortamı ve yoğun şekilde paketlenmiş özelliklerin yüksek karmaşıklığı ile daha da kötüleşen otomatik segmentasyonu çok zorlaştırır. Aktin demeti organizasyonu hakkında önceki bilgileri kullanarak, önce yoğunluk haritasına küresel bir uyum elde etmek için oldukça basitleştirilmiş bir bilye ve çubuk aktin modelinin katmanlarını yerleştirdik, ardından modelin bölgesel ve yerel ayarlamalarını yaptık. Hacimsel model oluşturmanın sadece yüksek karmaşıklıkla başa çıkmamıza izin vermekle kalmayıp, aynı zamanda aktin demeti hakkında kesin ölçümler ve istatistikler sağladığını gösteriyoruz. Hacimsel modeller, aktin-aktin çapraz konektörleri gibi yerel segmentasyon için bağlantı noktaları olarak da işlev görür. Hacimsel model oluşturma, özellikle bilgisayar tabanlı otomatik montaj yaklaşımlarıyla daha da artırıldığında, geleneksel otomatik segmentasyon yaklaşımları başarılı olmadığında güçlü bir alternatif olabilir.
Giriş
Kriyo-elektron tomografisi, tüm organellerin veya hücrelerin ve dokuların parçalarının, daldırmalı dondurma4 veya yüksek basınçlı dondurma ultra hızlı vitrifikasyon5 kullanılarak doğal durumlarınayakın 1,2,3'te nanometre çözünürlüklerde görüntülenmesini sağlar. Kriyo ile korunmuş, boyanmamış, donmuş hidratlı numune tarafından yalnızca sınırlı bir elektron dozu tolere edilebildiğinden, tomografik 3D veriler çok gürültülüdür. Bu gürültü genellikle, doğrusal olmayan anizotropik difüzyon8, iki taraflı filtreleme9 ve özyinelemeli medyan filtreleme 10 dahil olmak üzere çeşitli gürültü filtreleme algoritmaları 6,7 ile önemli ölçüde azaltılabilir.
Ayrıca, mikroskop aşamasının eğim sınırlamaları, eksik bir bilgi kamasına neden olur ve numune kalınlığının yüksek eğim açılarında artması, anizotropik çözünürlüğe sahip 3D rekonstrüksiyonlara yol açar. Bu, Z yönündeki daha düşük çözünürlük nedeniyle yoğunluğun üçüncü boyutta bulaştığı anlamına gelir. Sonuç olarak, makromoleküllerin şekli bozulmuş (yani, üçüncü boyutta daha az iyi tanımlanmış ve uzamış) görünür.
Tomografik verilerin yorumlanmasındaki en büyük zorluklardan biri, segmentasyon11 olarak da bilinen ilgili özelliklerin otomatik olarak çıkarılmasıdır. Yeterli benzersiz şekil özellikleri ve düşük gürültü ile, karmaşık 3D hacimlerdeki makromoleküler makineler, 12,13,14 şablonunu eşleştirerek tanımlanabilir; Bununla birlikte, şablon eşleştirmenin başarısı, Tomogram çözünürlüğüne, uygun bir arama modeline ve özellik hacimlerinin boyut ve şekil özelliklerine bağlıdır. İlgilenilen özellikler yeterince aralıklıysa ve tekrar eden motifler (büyük makromoleküler makineler gibi) kolayca tanımlanabilirse, sinyal-gürültü oranını artırmak ve bireysel parçacık şekli bozulmalarının ortalamasını almak için tomogram alt hacimleri birleştirilebilir. Donmuş hidratlı Dictyostelium discoideum hücrelerinin ince kenarının elektron tomogramlarında bir aktin filament ağının şablon eşleştirme ile otomatik segmentasyonu bildirilmiştir15.
Bununla birlikte, ilgilenilen özellikler yakın aralıklıysa, veri çözünürlüğü anizotropisi, Z yönünde (elektron demetinin yönü boyunca) harita yoğunluklarının dışına çıkmasına neden olabilir, bu da yakın aralıklı makromoleküler makinelerin veya supramoleküler komplekslerin yoğunluk zarfının belirgin bir şekilde birleşmesine neden olabilir. Bu gibi durumlarda, havza16, sınır segmentasyonu17 veya çeşitli makine öğrenimi tabanlı sınıflandırma yaklaşımları18,19 gibi segmentasyon için otomatik yaklaşımlar, ilgilenilen özellikleri tanıyamayabilir veya ilgilenilen bir nesne etrafında doğru bir sınır oluşturamayabilir. Çoğu zaman, ya birkaç çok büyük parça ya da çok fazla parçalanmış bir hacim ile sonuçlanır, burada ilgilenilen özelliğin tamamlandığı algılanana kadar birçok küçük parçayı birleştirmek için çok çaba sarf edilmesi gerekir. Segmentasyon sonuçlarının bu tür manuel küratörlüğü çok emek yoğun olabilir ve hatta ilgilenilen yapı, kısa bağlayıcılar aracılığıyla birbirine bağlanan yakın aralıklı filamentler dizisi olduğunda tamamen başarısız olabilir. Bu devasa filamentli yapılar ağında, kendini yönlendirmek zor olabilir. Bunun nedeni, çözünürlük anizotropisi nedeniyle, yoğunlukların birbirine karışıyor gibi görünmesi ve hem otomatik hem de etkileşimli manuel segmentasyon yaklaşımları için zorlu bir zorluk sunmasıdır. Sonuç olarak, yalnızca küçük bölgeleri görsel olarak incelerken filamentler arasında kolayca "atlayabilir".
Neyse ki, iç kulak kıl hücresi stereosilyasındaki aktin demeti durumunda, genel aktin demeti organizasyonu ve aktin filamentlerininyönlülüğü hakkında bilgiye sahibiz 20,21. Aktin demeti, birbirinden yaklaşık 12-13 nm aralıklı 6-8 nm çapında altıgen, yoğun şekilde paketlenmiş yüzlerce aktin filamentinden oluşur22.
Bu, aktin filamentlerini temsil etmek için basitleştirilmiş top ve çubuk modellerine dayanan segmentasyona oldukça farklı bir yaklaşım benimsememizi sağladı. Strateji, aktin demetinin katman katman 3 boyutlu bir modelini oluşturmak için kriyo-elektron tomografi yoğunluk haritalarının levhalarına aynı anda idealize edilmiş düzenli bir filament modelleri dizisinin yerleştirilmesini içeriyordu. Yoğunluk haritasına yakından uyması için tek tek filament modellerinde veya filament model gruplarında yerel ayarlamalar yapmadan önce modelin yoğunluk haritasına genel bir uyum sağladığından emin olduk. Filament model konumundaki harita yoğunluk değerinin otomatik renk kodlamasıyla, aktin demetindeki görünür boşlukları kolayca tespit edebildik. Hacimsel modeller, aktin filamentleri arasındaki mesafeler gibi hacimsel özelliklerin nicel bir analizine izin verir ve ayrıca genel 3B filamentli ağ organizasyonunun basitleştirilmiş bir görüntüsüne yol açar.
Ek olarak, modeller, aktin-aktin bağlayıcıları gibi ek özelliklerin segmentasyonu için ankraj yapıları olarak da hizmet edebilir, çünkü ayrı filament modelleri (bölümleri) seçilebilir, bu modellerin etrafında inceleme ve daha fazla segmentasyon için uygun yarıçap haritası yoğunluk bölgeleri oluşturulabilir.
Hacimsel model tabanlı segmentasyon yaklaşımımızın, boşluklar ve filamentler arası çapraz bağlantılar içerebilen büyük filamentli yapı ağları için özellikle yararlı olduğuna inanıyoruz. Segmentasyon algoritmaları yerel olarak çalışma eğilimindedir, oysa insan beyni daha geniş alanları dikkate alır ve bu nedenle karmaşık, yüksek gürültülü bir ortamda bile filament yapılarını tanıma konusunda bilgisayarlardan üstündür.
Protokol
Protokol, Güneydoğu Üniversitesi'nin insan araştırmaları etik komitesinin yönergelerini takip eder.
1. Hacimsel model oluşturma için kriyo-elektron tomografisi veri kaynağı
NOT: Hacimsel bir model binası için kullanılan stereocilia kriyo-elektron tomografik rekonstrüksiyonları daha önce yayınlanmıştır22,23 ve Metlagel ve ark.22 tarafından tarif edildiği gibi elde edilmiştir.
Stereocilia modelleme için UCSF Chimera python komut dosyaları , Ek Dosya 1, Ek Kodlama Dosyası 1, Ek Kodlama Dosyası 2, Ek Kodlama Dosyası 3, Ek Kodlama Dosyası 4 ve Ek Kodlama Dosyası 5'te sağlanır.
- Kısaca, fare utrikül duyusal epitelinin apikal yüzeyinden stereosilia'yı bir elektron mikroskobu (EM) ızgarasının dantelli karbon destek filmi üzerine kurulayın. Ardından, 3,5-4,5 μm nominal defokus ile 300 kV'da çalıştırılan bir kriyojenik transmisyon elektron mikroskobu (kriyo-TEM) üzerinde ultra hızlı daldırmalı dondurma vitrifikasyonu ve tek eksenli kriyo-elektron tomografik veri toplama gerçekleştirin, entegrasyon modunda CMOS tipi bir kamera kullanarak, 0,47 ila 0,59 nm piksel boyutuna sahip (bkz.
- Tek eksenli veri toplama için tipik bir doz tipik olarak 80-100 elektron /Å2'dir. Tomogram 3D ciltlerini IMOD24 yazılım paketini kullanarak, ağırlıklı geri projeksiyon veya SIRT yöntemi25,26 ile yeniden oluşturun.
- Priism27'de özyinelemeli medyan veya bilateral filtreleme veya IMOD'da doğrusal olmayan anizotropik difüzyon seçeneğini kullanarak tomogramların gürültüsünü azaltın.
NOT: Etkileşimli görselleştirme, hacimsel model oluşturma ve kantitatif analiz için kullanılan temel yazılım paketi UCSF Chimera yazılımıydı28,29. Kullanılan tüm yazılım paketleri Malzeme Tablosunda listelenmiştir.
2. Hacimsel model oluşturma için kriyo-elektron tomografisi verilerinin hazırlanması
- Tomogram döndürme
NOT: Bu adımın amacı, aktin çekirdeğinin üç eksenden ikisinde (x ve z) hizalandığı "en uygun açıları" bulmaktır, böylece modelleri 3D olarak yerleştirmek, endişelenecek tek bir eksenle yapılabilir. IMOD yazılım paketi içerisinde aşağıdaki adımlar gerçekleştirilir.- 3dmod'u (IMOD) açın ve 3B görüntü yığını dosyasını .mrc dosya biçiminde açın. 3B yığınının 16 bit gri tonlama modunda olduğundan ve görüntü dosyası başlığında doğru X, Y, Z boyutlarını görüntülediğinden emin olun. Gerekirse, alterheader komutunu kullanarak düzeltmeleri yapın: alterheader -d (x_pixelspacing),(y_pixelspacing),(z_pixelspacing) inputfilename.mrc komut isteminde. 3B yığını TIFF dosya biçimindeyse, bir .mrc dosyası oluşturmak için komut isteminde tif2mrc inputfilename.tif outputfilename.mrc komutunu kullanın.
- Tomogramı görsel olarak inceleyin ve aktin filament düzleminin Z düzlemi ile hizalanması için X, Y ve Z boyutlarında en iyi dönme açılarını bulmak için "dilimleyiciyi" açın ("\" ters eğik çizgiye basarak). Aktin filamentlerinin X ve Z düzlemlerinde hizalandığı en uygun açıyı bulmak için X dönüşünü, Y dönüşünü, Z dönüş çubuğunu manipüle edin. Tomogramın görüntülenmesi için ortalama kalınlığın değiştirilmesinin (Kalın: Img), Z düzlemindeki yoğunluğun ideal bir ortalama miktarını bulmaya ve dolayısıyla kontrastı artırmaya yardımcı olabileceğini unutmayın. Klavyedeki sırasıyla + ve - tuşlarını kullanarak tomogramı yakınlaştırın ve uzaklaştırın.
- Döndürme için ideal açılar belirlendikten sonra, IMOD komutunu yazarak tomogramı döndürün rotatevol -a (Z dönüş açıları),(Y dönüş açıları),(X dönüş açıları) -s (x-genişlik),(y-yükseklik),(z-depth) inputfilename.mrc outputfilename.mrc komut istemine. Döndürme sırasında haritanın bazı bölümlerinin kesilmesini önlemek için, haritaya döndürmeye uygun bol miktarda alan bıraktığınızdan emin olun.
NOT: rotatevol komutunun nasıl kullanılacağı hakkında daha fazla bilgi için, yardım menüsündeki seçeneği kullanın veya aşağıdaki URL'yi (https://bio3d.colorado.edu/imod/doc/man/rotatevol.html) ziyaret edin. Bu komut (rotatevol), IMOD penceresinde o anda gösterilen haritayı değiştirmez, bunun yerine döndürülen bir yoğunluk haritasıyla yeni bir harita dosyası oluşturur. - Dilimleyici penceresini kullanarak, trimvol komutunu kullanarak stereosiliumu içeren kırpma bölgesini belirlemek için haritanın iki zıt köşesinin X, Y ve Z koordinatlarını belirleyin.
- Komut istemine trimvol -x (x-coordiante 1),( x-coordiante 2) -y (y-coordiante 1),( y-coordiante 2) -z (z-coordiante 1),( z-coordiante 2) inputfilename.mrc outputfilename.mrc yazarak haritayı daha önce elde edilen koordinatlarda kırpmak için IMOD komutunu Trimvol kullanın. Şimdi, harita çok daha küçük ve bu noktadan itibaren çalışmak daha kolay ve daha hızlı.
NOT: trimvol komutunun nasıl kullanılacağı hakkında daha fazla bilgi için, yardım menüsündeki seçeneği kullanın veya aşağıdaki URL'yi (https://bio3d.colorado.edu/imod/doc/man/trimvol.html) ziyaret edin. Bu adımlar, döndürülmüş ve kırpılmış bir .mrc dosyası oluşturur.
- Tomogram filtreleme
NOT: Bu adım, gürültü azaltma için IMOD tarafından sağlanan doğrusal olmayan bir anizotropik difüzyon (NAD) filtresi kullanır. Başlangıç değeri olarak, aşağıdaki URL'de (https://bio3d.colorado.edu/imod/doc/NADexample.html) listelendiği gibi, doğrusal olmayan anizotropik difüzyon filtrelemesi için IMOD yardım sayfasında önerilenleri kullandık ve başlangıç değerleri olarak kullanıldı.- IMOD'un etomo grafik kullanıcı arayüzünü başlatmak için komut istemine etomo yazın.
- etomo menüsünden Doğrusal Olmayan Anizotropik Difüzyon seçeneğini belirleyin ve harita dosyasını seçin (Bir birim seçin).
- Görsel inceleme ile değerlendirildiği gibi en iyi filtreleme sonuçlarını sağlayan bir K değeri ve yineleme sayısını bulmak için, belirtilen NAD filtresi test hacmini çıkarmak için NAD filtresini daha küçük bir test hacmine uygulayın. 3dmod arayüzünün üst kısmındaki Lastik Bant Aracına tıklayın, test sesini seçmek için sol tıklayın sürükleyin ve Merhaba ve Lo Z diliminin test hacminin sınırı olduğunu belirtmek için düğmelere tıklayın (3dmod'dan Test Hacmi Aralığını Alın). Test Hacmini Ayıkla'ya tıklayarak test hacmini ayıklayın.
- 0.1, 1,5, 10, 15, 25, 30, 50 ve 75 gibi çeşitli K değerlerini (Test Hacmi için Bir K Değeri Bul > K değerleri listesi) kullanarak test hacmini filtreleyin. Belirtilen K değerlerinin her biri ile NAD filtrelemesi yapın (Farklı K Değerleriyle Çalıştırın > Farklı K Değerleri Test Sonuçlarını Görüntüleyin). NAD filtresinin performansını değerlendirmek için dilimleyici penceresini kullanın ve tüm birimi filtrelemek için kullanılacak bir K değeri seçin.
- Adım 2.2.4'te tanımlanan K değerini kullanın ve 2, 5, 8, 11, 15 ve 21 gibi farklı sayıda yinelemeyi test edin (Test Hacmi için Yineleme Numarası Bul > Yineleme listesi). Farklı Yineleme Test Sonuçlarını Görüntüle'ye tıklayarak veya bir dilimleyici aracı kullanarak farklı yinelemeler için NAD filtresi performansını değerlendirin. Farklı sayıda yineleme için bir değer seçin.
- Tanımlanan K değerini ve yineleme sayısını kullanın (Tam Birimi Filtrele > K değeri ve Yinelemeler) ve tüm birimi filtreleyin (Tam Birimi Filtrele). Yeni filtrelenmiş bir birim, yüklenen tam birimin bulunduğu dizinde ve .mrc dosyasına iliştirilmiş bir .nad dosya uzantısıyla birlikte görünür.
NOT: Bu adımlar, bir NAD filtresiyle filtrelenmiş ve daha düşük bir gürültü ve dolayısıyla iyileştirilmiş bir sinyal-gürültü oranı görüntüleyen bir .mrc dosyası oluşturur.
3. Hacimsel model oluşturma
- 3D model yerleştirme hazırlığı
NOT: Bu adımın amacı, UCSF Chimera programını kullanarak 3B hacimsel modelleri döndürülmüş ve gürültüden arındırılmış yoğunluk haritasına yerleştirmek için haritayı hazırlamak ve analiz etmektir.- Dilimleyici penceresini kullanarak, aktin filament modelinin yerleştirileceği yoğunluk levhasının merkezinin Z-koordinatlarını belirlemek için bir kesit görünümü (30 dilim/28,4 nm'lik levha) elde edin. Filament yoğunluğunun merkezine tıklayın ve IMOD ana penceresindeki Z koordinatlarını not edin.
- UCSF Chimera'da gelecekteki model yerleşimi için belirlenen koordinatları not edin.
NOT: Bu adımda not edilen Z-koordinatları, önceden hazırlanmış aktin filament modellerini doğru Z-yüksekliklerine kolayca bulmak ve yerleştirmek için 4. adımda kullanılır, bu da model yerleşimini daha hızlı hale getirir.
- 3D model yerleştirme
NOT: Bu adımın amacı, UCSF Chimera kullanarak hazırlanan yoğunluk haritasına 3 boyutlu hacimsel bir model yerleştirmektir.- UCSF Chimera'da filtrelenmiş, döndürülmüş haritayı açın (Dosya > Aç)
- Özellik ve koordinatlar arayüzünü açmak için Volume Viewer'da Özellikler > Düzlemler ve Özellik > Koordinatları'nı seçerek harita parametrelerinin doğru ayarlanıp ayarlanmadığını kontrol edin. Komut istemindeki (IMOD'da) başlık komutu kullanılarak doğru voksel aralığının ayarlanıp ayarlanmadığını görmek için Voksel Boyutu'nu kontrol edin. Değilse, Voksel Boyutu arayüzünü doğru voksel aralığına düzeltin. Kamerayı görüntüleme penceresinin ortasına yerleştirmek için haritayı ortalayın (Origin Index > Center).
- Kamera Kontrolü penceresini açın (Araçlar > Görüntüleme Kontrolü > Kamera). Ardından, kamera görünümünü ortografik görünüme ayarlayın (projeksiyon > ortografik)
- Önceden hazırlanmış python betik modelini yükleyin (Dosya > Aç > ActinFilamentPlane.py) ve haritada aktin filament düzlemleri olduğu kadar çok aktin filament düzlemi betiği açın.
- Hareket Fare Modu'nu (Hareket Fare Modu > Araçlar >) açarak yüklenen modellerin yerini değiştirin. Hareket faresi seçeneğini etkinleştirin (Hareket Fare Modunu Ayarla > Seçimi Taşı). Oturumda bulunan modelleri çoklu seçmek için Ctrl + Sürükle tuşlarını kullanın ve modelleri istediğiniz yönde hareket ettirmek için sağ tıklayın (veya Fare Tercihi'nde belirtilen başka bir tuşa) > tıklayın.
NOT: Modeller, belirli kalınlıkta (ActinFilamentPlane.py yazının yarıçapı ) ve belirli aralıklarla (ActinFilamentPlane.py yazının Lattice_Spacing ) toplar ve çubuklarla oluşturulmuştur. - Yalnızca ilgilendiğiniz modeli görselleştirmek için Model Panelini açın (Araçlar > Genel Kontroller > Model Paneli). Belirli bir modelin görünürlüğünü etkinleştirmek veya devre dışı bırakmak için S (Göster) altındaki onay kutularını tıklayın.
- Komut satırı panelini etkinleştirin (Araçlar > Genel Kontroller > Komut Satırı).
- Her aktin filament düzlemi modelini uygun Z yüksekliğine yerleştirdikten sonra, modellerin fazla kısmını seçerek ve UCSF Chimera ana görünüm penceresinin altındaki komut satırına del sel (Seçileni Sil) yazarak her bir aktin filament düzlemi modelindeki fazla aktin filamentini silin.
- Model Paneli penceresine gidip diğer tüm aktin filament modellerini seçip gizleyerek (Ctrl + Tıkla > Gizle) bir seferde yalnızca bir aktin filament düzlemini görselleştirin.
- İlgilenilen tek aktin filament düzlemi modelinin hemen yakınında bulunan harita yoğunluğunun bölümlerini görselleştirmek için modeli seçin (Model Paneli > seçin) ve Birim Görüntüleyici'de (Birim Görüntüleyici > Özellikler > Bölge) bölge kontrol panelini açın. Bölge yarıçapını 100 şolarak ayarlayın, ardından haritayı seçili modelden 100 ş(= 10 nm) yarıçapa yerleştirmek için Bölge'ye tıklayın. Chimera'daki boyutun Angstrøm (1 ş= 0,1 nm) olarak ayarlandığını unutmayın.
- Gerekirse, Hareket Fare Modu'nu açarak modeli yoğunluk haritasına sığacak şekilde ayarlayın. Bölgeli yoğunluk haritasının içine düzgün bir şekilde yerleştirilecek modelleri taşımak için uygun konumda olmayan modelleri çoklu seçin.
- ActinFilamentPlane.py'daki bir programlama hatasının üstesinden gelmek için, "atomların" (top ve sopa modelindeki toplar) eksik MarkerID'sini düzelten FixingMarkerID.py komut dosyasını çalıştırın. Modelin tamamını seçin ve komut satırına runscript FixingMarkerID.py yazın (betik doğru dizin tarafından belirtilmelidir, örneğin C: \ directory \ Script.py).
- Yoğunluk haritası aktin filamentinin kavisli olduğunu gösteriyorsa, istenen modelleri seçerek ve runscript dividelinks.py numarasını yazarak eğriliğe uyum sağlamak için düz aktin filamentini birden çok parçaya böldüğünüzden emin olun. Ek işaretler (komuttaki sayı ile belirtilen) aktin modelinin içine birbirinden eşit uzaklıkta yerleştirilir ve modelin bükülebileceği "eklemler" oluşturur.
- Hareket Fare Modu'nu kullanarak ek işaretleyici "eklemleri" bölgeli harita boyunca hareket ettirerek eklemleri eğriliği yansıtacak şekilde ayarlayın. dividelinks.py betiğini kullanarak modele gerektiği gibi ek işaretçiler ekleyin.
- Tüm aktin filamentlerinin aktin yoğunluklarının içine düzgün bir şekilde yerleştirildiğini doğrulamak için, Kamera Kontrolü penceresini açmak için UCSF Chimera açılır menüsünde Araçlar > Görüntüleme Kontrolü > Kamera'ya tıklayarak kırpma çubuklarını kullanın, ardından kameranın iki kırpma çubuğunu (yakın ve uzak kırpma düzlemleri) görmek için Yan Görünüm'ü seçin. Sol tıklayarak iki sarı çubuğu dar bir yarığa sürükleyin ve modelleri her seferinde küçük bir parça incelemek için Orta Fare Düğmesini kullanarak kırpma kısmını sürükleyin.
NOT: Stereosilium aktin çekirdek modeli artık tamamlanmıştır.
- Membran segmentasyonu
NOT: Bu adımın amacı, stereocilia zarının bir yüzey modelini oluşturmaktır.- Yüzeylerin segmentasyonu, Volume Tracer aracını kullanır (Hacim Verileri > Araçlar > Volume Tracer).
- Volume Tracer (Hacim İzleyici) penceresinde, Fare'ye tıklayın ve yalnızca İşaretçileri Veri Düzlemlerine Yerleştir ve Yeni işaretçiyi seçili işaretçiye bağla seçeneklerini işaretleyin. Bu, işaretleyicilerin aynı Z düzlemine (tek bir Z düzlemi gösterildiğinde) hassas bir şekilde yerleştirilmesine izin verir. Ayrıca, Volume Tracer (Ses İzleyici) penceresindeki Place Markers Using the Middle Mouse Button (Orta Fare Düğmesini Kullanarak İşaretleyicileri Yerleştir) seçeneğini de işaretleyin.
- Yeni bir işaretçi kümesi oluşturun (Dosya > Yeni İşaretçi kümesi). İşaretleyici seti 1, Volume Tracer penceresinde oluşturulmalı ve seçilmelidir.
- Ana görüntüleme penceresinde tek Z düzlemlerini görüntülemek için Ses Görüntüleyici'yi kullanın (Eksenden Z'ye > Bir.)
- Orta Fare Düğmesini kullanarak, zarı görünür zarın bir ucundan diğerine kadar takip edin. Tüm işaretçiler bir uçtan diğerine bağlantılarla bağlanmalıdır.
- Tek bir seçilebilir öğenin bulunmadığı arka planda Ctrl + Click tuşlarına basarak son yerleştirilen işaretçinin seçimini kaldırın. Bu, yerleştirilecek bir sonraki işaretleyicinin, başka bir Z-düzleminin önceki bir zar iziyle değil, yeni düzlemdeki bağlantılarla bağlanmasını sağlar.
- Görünür zarın bir ucundan diğer tarafına aynı izleme yönünde her 10 nm'de bir membran izlemeyi tekrarlayın.
- Birçok paralel membran bandı segmentlere ayrıldıktan sonra, Volume Tracer penceresine gidin ve Özellikler > Yüzeyler'e tıklayın. Bu, Volume Tracer penceresindeki yüzey arabirimini etkinleştirir. Membran segmentasyon bantları arasında yüzeyler oluşturmak için Yüzeyler arayüzünün yanındaki Oluştur'a tıklayın. Bu, aralıklı membran izlerini dolduran yüzey modelinin ince bir kanadını oluşturur.
NOT: Stereosilium membran yüzey modeli oluşturma işlemi tamamlanmıştır.
- Çapraz bağlayıcı modelleme
NOT: Bu adımın amacı, aktin filamentleri arasındaki çapraz bağlayıcıları modellemektir.- Çapraz bağlayıcılar, Volume Tracer aracı kullanılarak da yerleştirilebilir.
- Volume Tracer (Hacim İzleyici) penceresinde, yalnızca Place Markers on Surfaces (İşaretçileri Yüzeylere Yerleştir) ve Link new marker to selected marker (Yeni işaretçiyi seçili işaretçiye bağla) seçeneklerini işaretlemek için sol tıklayın. Ardından, Volume Tracer (Ses Kaydedici) penceresindeki Place Markers Using the Middle Mouse Button (Orta Fare Düğmesini Kullanarak İşaretleyicileri Yerleştir) seçeneğini işaretleyin.
- Bölge fonksiyonunu kullanarak yalnızca ilgilenilen tek aktin filament düzlemi modelinin etrafındaki yoğunlukları görselleştirin. Haritayı seçilen modelden 100 Å'lik bir yarıçap içinde görselleştirmek için bölge yarıçapını 100 şolarak ayarlayın.
- Orta Fare Düğmesini tıklatarak işaretçileri çapraz bağlayıcıların görünür yoğunlukları boyunca yerleştirin. İki işaretçi yerleştirildikten sonra (ve böylece tek bir çapraz bağlayıcı modellendikten sonra), bağlantıların sürekli olarak bir sonraki çapraz bağlayıcı modeline bağlanmasını önlemek için ikinci işaretleyicinin seçimini kaldırdığınızdan emin olun.
- Her aktin filament düzlemi için yukarıdaki adımları tekrarlayın ve yukarıdaki işlemin tamamını, çapraz bağlayıcıların yerleştirilebileceği üç ana yönün tümüne tekrarlayın. Aktin filament düzlemlerini çapraz yönde imar ederken, aktin filament modellerini ayrı ayrı manuel olarak seçin.
NOT: Aktin çapraz bağlayıcı modellerinin her biri için, varsayılan çapraz bağlayıcıların bağlandığı aktin filamentleri üzerindeki konumu kaydedebiliriz. Bu bilgiler, prensip olarak, bu çalışmanın kapsamı dışında kalan alt tomogram ortalaması kullanılarak bir takip analizi için çıkarılabilir.
4. 3D modelin kantitatif analizi
- Boşluk analizi
NOT: Bu adımın amacı, aktin filamentlerindeki boşlukları tespit etmektir.- Seçilen öğeler analiz edilebilir. Seçimi İncele aracını (Eylem > İncele) kullanarak renkleri gibi parametrelerini de belirtin.
- Seçilen öğenin rengi, UCSF Chimera'da herhangi bir seçilebilir öğe seçilerek ve Seçimi İncele penceresinin Renk bölümünün yanındaki renk değiştirilerek belirlenebilir.
- UCSF Chimera'da herhangi bir işaretçi ve bağlantı seçerek ve birimlerin Angstrøm'da bulunduğu Seçimi İncele penceresinin yarıçap bölümünün yanındaki değerleri değiştirerek işaretçilerin ve bağlantıların boyutunu belirtin.
- Atom Konumlarındaki Değerler işlevini (Atom Konumlarındaki Değerler > Hacim Verileri > Araçlar) kullanarak yoğunluk boşluklarını otomatik olarak algılayarak boşluk analizi gerçekleştirin.
- Atom Konumlarındaki Değerler penceresinin Hacim Verileri bölümünün yanında istediğiniz haritayı seçerek değerlerin okunacağı haritayı seçin. Molekül bölümünün yanında istenen modeli seçerek tüm işaretçilere bir parametre atanacak modeli seçin.
- Atom Konumlarındaki Değerler penceresinin altındaki Histogram düğmesiyle, Özniteliğe Göre Oluştur/Seç adlı yeni bir pencerede bir histogram oluşturulur. Histograma Ctrl + Tıklama, farklı renklerin seçilebileceği kesme noktaları ekleyecek veya kaldıracaktır. Sahneye saydam bir renk uygulamak için Opak Tut seçeneğinin işaretini kaldırın. Boşlukları temsil eden belirli bir eşiğin altında bir yoğunluğa sahip işaretler sadece renklendirilemez, aynı zamanda seçilebilir ve böylece boşluk bölgesindeki sayı işaretleyicilerini saymamıza izin verir. Seçmek için > Özellik Seç'e tıklayın > value_mapname.
- Aktinler arası mesafelerin belirlenmesi
- Aktinler arası mesafeleri ölçmek için, Hacim İzleyici penceresini kullanarak, Fare'yi tıklatarak ve yalnızca Veri Düzlemlerine İşaretleyicileri Yerleştir'i işaretleyerek ve Yeni işaretçiyi seçili işaretçiye bağla seçeneğinin işaretini kaldırarak işaretçileri tek bir XZ düzlemine (UCSF Chimera'da Y-düzlemi olarak anılır) yerleştirin. Bu, işaretçilerin aynı Y düzlemine hassas bir şekilde yerleştirilmesine izin verir (tek bir Z düzlemi gösterildiğinde). Ayrıca, Volume Tracer penceresinde Orta Fare Düğmesini Kullanarak İşaretleyicileri Yerleştir'i işaretleyin ve işaretçileri, aktinler arası mesafe ölçümü için ilgilenilen bir hedef Y düzlemine yerleştirin.
- Tek Y düzleminin aktin konumları başarıyla işaretlendikten sonra, tüm işaretçileri seçin ve şu komutu yazın: findclash #model-spec test self overlapCutoff -200 hbondAllowance 0 log true Linewidth 10 pbColor, aktin filament düzleminin altındaki komut satırına siyah. Bu, 200 ş(overlapCutoff -200) mesafesindeki tüm işaretçiler arası çakışmaları bulur ve işaretçiler arasında 10 ş(Çizgi Genişliği 10) kalınlığında siyah çizgiler (pbColor black) oluşturur.
- Verilen 200 şaralığında çakışan işaretçiler olduğundan, yalnızca en yakın, bitişik işaretçiler arasında bağlantı olmayan gereksiz ölçümleri kaldırın. Bunu, UCSF Chimera web sitesinde sağlanan ve bitişik işaretçileriyle en kısa bağlantı olmayan tüm çakışan bağlantıları kaldıran RemoveCross.py komut dosyasını kullanarak yapın.
- Kalan bağların mesafesini ölçmek için tüm sahte bağları (findclash tarafından oluşturulan bağlantılar) seçin ve runscript pblengths.py yazarak python betiğini pblengths.py kullanın. Yanıt Günlüğü'nden (Araçlar > Yardımcı Programlar > Yanıt Günlüğü) ölçülen değerlere bakın. Ardından, elde edilen değerleri Yanıt Günlüğünden kopyalayın ve bir histogram oluşturmak için bir elektronik tabloya veya başka bir uygun araca aktarın.
Sonuçlar
Camsı buza gömülü boyanmamış, donmuş hidratlı bireysel stereosilyaların kriyo-elektron tomografisini kullanarak, çapraz bağlayıcı proteinler23 ile bağlanan altıgen olarak düzenlenmiş aktin filamentleri ile aktin demetinin yoğunluk haritalarını elde ettik. Tek bir vokselin boyutu 0.947 nm idi. Tüm tomogramın (400 dilim / 379 nm) bir hacim görüntüsünün IMOD dilimleyici programında görsel olarak incelenmesi, uzunlamasına görünümlerde görülebileceği gibi, stereosilinin uzunlamasına ekseni ile hizalanan filamentli yapıların varlığını gösterdi (XY düzlemi; Şekil 1A-C, üst paneller) ve kesit görünümlerinde (XZ-düzlemi; Şekil 1A-C, alt paneller). 400 dilim/379 nm filament ağından projeksiyon görünümünün, yeniden yapılandırılmış orijinal hacmi X ekseni etrafında -6°, Y ekseni etrafında -13,5° ve Z ekseni etrafında 5° döndürdüğümüzde en net hale geldiğini fark ettik. Bu açıda, tüm filamentler üst üste hizalanır ve bu nedenle, kesit görünümlerinden de anlaşılacağı gibi kontrast maksimumdur (Şekil 1B). Tek kesitli dilimler, aktin filamentlerini açık bir şekilde ayırt etmek için yeterli sinyale sahip olmadığından, enine kesit görünümünde altıgen bir deseni açıkça gösteren 30 dilim/28,4 nm'lik bir levhayı hacim olarak oluşturmayı seçtik. Şekil 1C'deki (üst paneller) mavi çizgiler, alt panellerdeki karşılık gelen 30 dilim/28.4 nm kesitli levhaların ortasının konumunu gösterir.
Bu optimal görüş açısından ±2° kadar küçük sapmalar, aktin filament ağının algılanan sırasını önemli ölçüde azaltmıştır (Şekil 1A,C), bu da tomogramın 3D hacminde kaybolmanın ne kadar kolay olduğunun bir göstergesidir.
Havza segmentasyonu gibi otomatik segmentasyon yaklaşımlarını kullanmanın zorluğunu göstermek için, UCSF Chimera yazılım paketinde (Segger > Segmenti > Hacim Verileri Araçları >) uygulandığı gibi, havza segmentasyonu için küçük bir alt hacim (altın olarak tasvir edilmiştir) seçtik. Alt hacmin tüm stereocilia haritasına göre konumu, Şekil 1B'deki küçük iç kısım ile gösterilir.
Şekil 1D-F, seçilen alt hacmi farklı yönlerde gösterir, Şekil 1D,E uzunlamasına görüntüleme yönünü ve Şekil 1F enine kesitsel görüntüleme yönünü gösterir. Şekil 1D-F'nin sol tarafındaki oklar, aktin filamentlerinin yönünü gösterir.
Şekil 1D-F (sağ paneller) havza segmentasyonunun sonuçlarını göstermektedir. Alt hacim, nesne kimliğine göre renk kodluyor ve renkler farklı nesnelere rastgele atanıyor. Farklı renkler farklı bir nesne kimliğini gösterir, bu nedenle Şekil 1D-F'den, filamentler için harita yoğunluklarının her ikisinin de filament ekseni boyunca parçalandığı, buna karşılık aynı renk ve dolayısıyla nesne kimliğinin, komşu filamentleri birbirine bağlayan harita yoğunluklarına verildiği anlaşılmaktadır. Başka bir deyişle, havza segmentasyon algoritması, aktin filamentlerinin yoğunluk haritasını uzun bir süre boyunca takip edemedi ve bunun yerine komşu filamentlerden gelen yoğunlukların bağlanmasına yol açtı. Seçimi manuel olarak düzenlemek mümkün olsa da (örneğin, nesneleri silerek veya birleştirerek), bu yaklaşım oldukça emek yoğun ve dolayısıyla zaman alıcıdır.
Hacimsel model oluşturma stratejimizin çalışması için kesinlikle gerekli olmasa da, aktin filament ağ ekseninin Y ekseni ile hizalanması ve aktin filament model düzlemlerinin tomogramın XY düzlemi ile hizalanması için 3B haritanın yeniden yönlendirilmesine (döndürülmesine) yardımcı oldu. Bu oryantasyonu stereocilia tomografik ekran için standart oryantasyon olarak adlandırıyoruz.
Bu nedenle, aktin filamentlerinin düzenli aralık ve tanımlanmış genel demet oryantasyonu ile genel bir düzenli organizasyon (altıgen paketleme) sergilediği gerçeğinden yararlanarak görüntü segmentasyonu için farklı bir strateji keşfetmeye karar verdik. Stratejimiz, bir filament dizisi olarak bir aktin demetinin modellerinin genel bir uyumunu bulmak ve ardından deneysel yoğunluk haritasına uyacak şekilde model konumunun bölgesel ve ardından yerel ayarlamalarını yapmaktı. Genel bir modeli ilk sıraya yerleştirerek, yerel harita belirsizliklerinin üstesinden gelebilir ve filament bükme gibi modelin orijinal organizasyonundan sapmalarının bölgesel eğilimlerini tespit edebiliriz.
Modeli yerleştirmek için, düzenli aralıklı, düz aktin filament modellerinden oluşan bir tabakanın yerleştirildiği aktin filamentlerinin tek bir tabakasının kalınlığına karşılık gelen standart oryantasyonda yoğunluk levhalarını (10 dilim / 9.47 nm) sergiledik. Bu elbette, her biri sarmal simetriye sahip doğrusal bir aktin monomerleri dizisinden oluşan aktin filamentlerinin aşırı basitleştirilmesidir. Şekil 2A-C, aktin filamentlerini temsil eden kırmızı renkli çubuklarla farklı Z-yüksekliklerinde üç temsili katmanı göstermektedir. ~ 30 dilim / 28.4 nm kalınlığında kesitleri gösteren üst paneller, 19 çubuktan oluşan ayrı bir aktin model katmanının hangi Z-yüksekliğine yerleştirildiğini gösterirken, alt paneller uzunlamasına bir yönelim gösterir (perspektif görünümde gösterilmiş olsa da). Şekil 2D, hem kesitsel (üst panel) hem de uzunlamasına perspektif görünümlerinde (alt panel) basitleştirilmiş modelin tamamını göstermektedir. Kesitsel oryantasyon, filamentleri iyi bir güvenle konumlandırmamızı sağladı. Burada, tüm hacmi tomogramların ana eksenleriyle çakışacak şekilde yeniden yönlendirmek için yaptığımız orijinal hamlenin yardımcı olduğu kanıtlandı, çünkü bu, modelimizin standart görüntüleme yönündeki oryantasyonunun da ana eksenlere paralel olduğu anlamına geliyordu. Bununla birlikte, kesin olarak konuşursak, yaklaşımımız tomogramın yeniden yönlendirilmesi olmadan da işe yarayacaktı, sadece modelin yoğunluğa yerleştirilmesi daha zor olacaktı.
Yoğunluk haritasının tek tek levhalarının dikkatli bir şekilde incelenmesi üzerine, mükemmel düz bir aktin modelinin, stereosilia'nın proksimal ucundan distal ucuna (yani ucuna doğru) hareket eden gözlemlenen yoğunluk haritasına uymadığını fark ettik (Şekil 3A-C). Stereosilianın ucuna yakın bir yerde, filamentler için harita yoğunluğu 13 nm'den (aktin-aktin aralığı) daha fazla yer değiştirdi, bu da stereosilia yoğunluk haritasının proksimal kısmından distal kısmına geçerken modeli ayarlayarak telafi edebildik, böylece aktin modelimize küçük ama ayırt edilebilir kademeli bir eğrilik getirdik. Şekil 3D, yoğunluk haritasına takılan hacimsel bir model ile aktin filamentinin harita yoğunluğunun tek bir levhasını göstermektedir. Düz (kırmızı) ve kavisli (sarı) model arasında bir karşılaştırma Şekil 3E'de gösterilmiştir. Bu eğrilik en iyi şekilde, yerleştirilmiş modelle birlikte yoğunluk haritasının bir levhasının X ekseni etrafında 80° eğilmesiyle takdir edilir, bu da aktin filamentlerinin yönü boyunca perspektif bir görünüme sahip olmayı sağlar (Şekil 3D,E).
İki modelin sapması, aktin modelinin uca yakın konumunun, aktin filamentlerinin aralığı ile yaklaşık olarak aynı mesafede kaydırılması, yaptığımız gibi ilerlememiş olsaydık, çok fazla kafa karışıklığına neden olabilirdi. Aktin filament modelinin bir katmanının bu "küresel" konumlandırılması ve ardından "bölgesel" ayarlama, uzunlamasına veya enine kesit görünümünde zar zor fark edilen bu eğriliği tespit etmemizi sağladı. Bununla birlikte, Şekil 3E'de gösterildiği gibi iki modelin üst üste bindirilmesi, ince farkı ortaya koymaktadır.
Bu yaklaşımın birden çok katman için tekrarlanması, enine kesit oryantasyonunda görüntülendiğinde, yalnızca stereosilianın en üstündeki ve altındaki veri belirsizliği ile sınırlı olan eksiksiz bir 3B modelin elde edilmesine izin verir (Şekil 3F). Bu yoğunluk eksikliği, (tek eksenli) tomografik veri toplamadaki eksik kama ve karşılık gelen veri çözünürlüğü anizotropisinden kaynaklanır ve etkisi, stereosilia membranları için iyi tanımlanmış bir harita yoğunluğunun olmaması ile gösterilir.
Bir 3D modele sahip olduğumuzda, hacimsel modelin her bir konumunu o konumdaki harita yoğunluğu değerine göre renk kodladık. Modelin altta yatan zayıf harita yoğunluğuna sahip bölgeleri kırmızı renkteyken, modelin güçlü harita yoğunluğu sinyaline sahip bölgeleri sarı renkte boyanmıştır (Şekil 4A). Onlarca nanometreye kadar uzayabilen bu tür kırmızı renkli bölgeleri, kapsamları nedeniyle, bir kriyo-EM haritasının yüksek gürültülü ortamında sıklıkla karşılaşılan yoğunluk değişimlerine atfedilemeyen aktin filament yapılarındaki boşluklar olarak yorumluyoruz. Gürültü, tek tek vokselleri veya küçük voksel gruplarını etkileme eğilimindedir, ancak filament yoğunluğunun eksik olduğu yüzlerce vokselden oluşan hacimlerin kaynağı olması olası değildir. Bunun yerine, bu tür boşlukların stereocilia aktin ağının gerçek bir özelliği olması muhtemeldir ve aktin devir bölgeleri oluşturabilir. Şekil 4A , açık mavi ve koyu mavi ile gösterilen iki farklı harita yoğunluğu değerine sahiptir. Açıkça belirtmek gerekir ki, hacimsel model oluşturma yaklaşımımız, modelimizin zayıf yoğunluklu bölgelerde otomatik renk kodlaması ile birleştiğinde, aksi takdirde çok zor olacak olan aktin filament modelindeki bu tür boşlukların dağılımını tespit etmek ve görselleştirmek için hızlı ve kullanışlı bir yoldur.
Şekil 4B'de gösterildiği gibi, hacimsel modelin nispeten zayıf yoğunluğa sahip konumlardaki kısımları, Şekil 4A'da elde edilen sonuçlara dayanarak kolayca gizlenebilir. Bu daha sonra stereocilia'daki aktin modelini daha gerçekçi bir şekilde tasvir edebilen daha parçalı bir modelle sonuçlanır. Küçük aktin filamentleri oluşturma alternatifi, Şekil 1'i tarif ederken tartışılan problemler nedeniyle çok emek yoğun olurdu ve tamamen başarısız olabilirdi.
Ek olarak, hacimsel model, çapraz bağlantının her iki tarafındaki aktin filament modelinin model noktası konumları arasına basitçe bir bağlantı (kırmızı ile gösterilmiştir) yerleştirerek çapraz konektörleri kolayca modellememize olanak tanır (Şekil 4C). Basitleştirilmiş yaklaşımımızda, daha yüksek çözünürlük ve / veya karmaşık etiketleme yaklaşımları gerektirecek olan her bir çapraz bağlanma proteininin tam kimliği hakkında herhangi bir varsayımda bulunmamıza gerek yoktur. Bunun yerine, belirlememiz gereken tek şey, bitişik aktin filamentleri arasında köprü kuran bir yoğunluğun var olup olmadığıdır; Varsa, bir filamentten bitişik muadili ile kısa bir bağlantı kurabiliriz. Şekil 4D'de, çapraz bağları ile birlikte beş aktin filamentinin bir modeli gösterilmektedir, bu da aktin filament ekseni boyunca çapraz konektörlerin dağılımı hakkında bir izlenim verir.
Aktin demetinin hacimsel bir modelini oluşturmanın bir başka avantajı, bitişik aktin filamentleri arasındaki boşluğun hızlı bir şekilde belirlenebilmesidir (Şekil 4E-H). Şekil 4E,F, sırasıyla, harita yoğunluğunun altıgen kafesine takılan bir model olmadan ve bu modelle yoğunluk haritasının enine kesit görünümünü göstermektedir. Şekil 4G, en yakın komşu toplar arasındaki bağlantılara sahip modeli göstermektedir. UCSF Chimera, en yakın komşu merkezlerin mesafesinin otomatik olarak hesaplanmasına izin verir, bunun sonucu daha sonra bir mesafe dağılımı olarak çizilebilir (Şekil 4H). İki ek veri kümesi için model oluşturma, Ek Şekil 1 ve Ek Şekil 2'de gösterilmiştir.
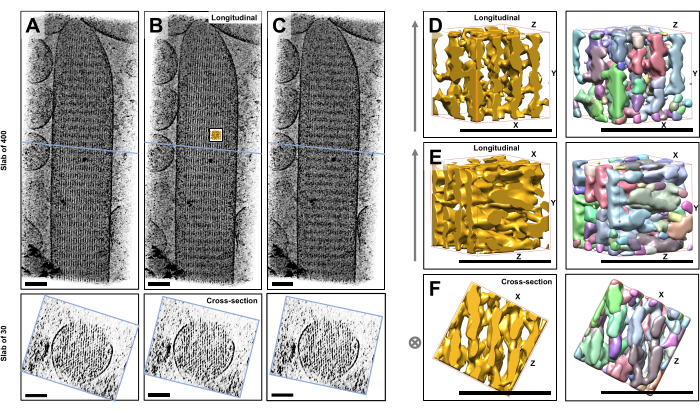
Şekil 1: Saç hücresi stereocilia tomogramlarının havza segmentasyonunda karşılaşılan zorluklar. (A-C) XY düzleminde (üst paneller) tomografik 3B harita aracılığıyla uzunlamasına projeksiyonlar (400 dilim/379 nm) ve XZ düzleminde (alt paneller) kesit görünümleri (30 dilim/28,4 nm). (A) Tomografik harita, Y ekseni boyunca optimum yönünden -2° döndürülmüştür. (B) X, Y ve Z eksenlerinin dönüş açılarının ayarlanmasıyla (X = -6°, Y = -13.5° ve Z= 5°) belirlenen ve yoğunluk haritasında yüksek derecede bir düzen ortaya çıkaran optimal oryantasyondaki tomografik harita, yüksek derecede düzenli bir aktin filament ağı olduğunu düşündürür. (C) Tomografik harita, Y ekseni boyunca optimum yönünden +2° döndürülmüş; Y ekseni etrafında optimum görüntüleme yönünden uzağa 2° kadar küçük bir dönüş, yoğunluk haritasının algılanan düzenliliğini ciddi şekilde tehlikeye atar. Alt paneller, enine kesit yönünde bakıldığında aktin filament dizisinin düzenliliğini ortaya koymaktadır. AC'deki mavi çizgi, kesit levhasının konumunu gösterir. (D-F) 50 nm x 50 nm x 50 nm'lik bir küp, havza segmentasyonundan önce (sol paneller) ve sonra (sağ paneller) üç farklı yönden görüntülendi. Havza segmentasyonunun sürekli aktin filament yoğunluğunu tespit edemediğini, bitişik aktin filamentlerinin ve bunların çapraz bağlantılarının aynı nesne kimliğini paylaştığını ve bu da havza segmentasyonunun tomogram segmentasyonu için uygun bir yaklaşım olmadığını düşündürdüğünü unutmayın. D-F panellerinde, Chimera'daki yoğunluk haritası "Yüzey" harita stili olarak gösterilir. (A-C) Ölçek çubukları = 100 nm. (D-F) Ölçek çubukları = 50 nm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
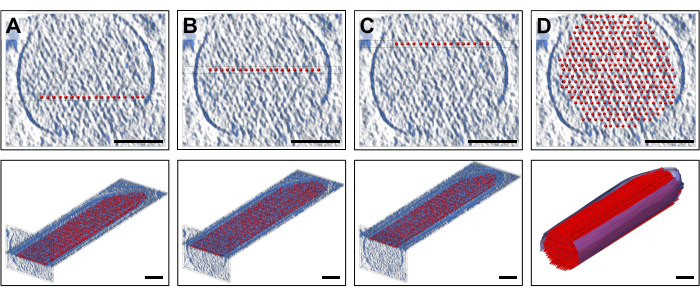
Şekil 2: Hacimsel bir top ve çubuk aktin filament modelinin oluşturulması. (A-C) Üst: farklı bir Z-yüksekliğine yerleştirilmiş tek katmanlı basitleştirilmiş aktin filament modeli ile yoğunluk haritasının kesitsel 30 dilim/28,4 nm görünümleri. Alt: tek katmanlı basitleştirilmiş aktin filament modeli ile yoğunluk haritasının tek bir 10 dilim/9.47 nm levhası. (D) 30 dilim/28,4 nm kesit görünümünde (üstte) ve perspektif görünümde (altta) eksiksiz düz aktin filament modeli; Ölçek çubukları = 100 nm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
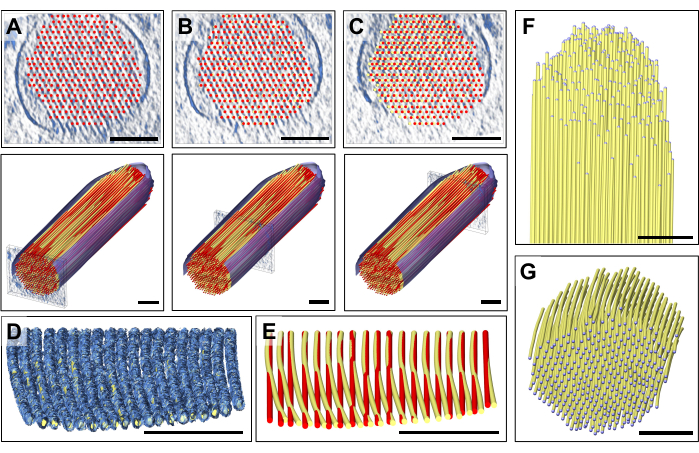
Şekil 3: Aktin filament yoğunluğunun tespit edilen bükülmesini düzeltmek için model ayarı. (A-C) Modelin enine kesit görünümünde (üst paneller) veya uzunlamasına görünümde (alt paneller) dikkatli bir şekilde görsel olarak incelenmesi, modelin stereocilia'nın proksimal ucundaki yoğunluk haritası ile iyi bir uyum sağladığını ortaya çıkardı. Bununla birlikte, stereosilinin distal ucuna doğru hareket ettikçe, uyum tüm aktin filament modelleri için giderek daha da kötüleşir. Bu, top ve çubuk modelinin toplarını doğru yoğunluk haritası konumuna hareket ettirerek düzeltilebilir, bu da hafif kavisli bir aktin filament modeli ile sonuçlanır. Düz model kırmızı renkle gösterilir ve düzeltilmiş bükülmüş model sarı renkle gösterilir. (D) Aktin yoğunluğunun stereosilinin ucuna doğru eğriliğini ortaya çıkaran, üzerine eğri model takılmış yoğunluk haritasının tek levhası. Aktin modeli, aktin filamentlerinin bu ince ama önemli bükülmesini daha iyi göstermek için X ekseni etrafında 80° döndürülmüştür. (E) Kırmızı ile gösterilen düz, düzeltilmemiş aktin modeli ile sarı ile gösterilen kavisli, düzeltilmiş aktin modeli arasındaki karşılaştırma. Netlik nedeniyle, aktin filament modelinin sadece bir katmanı gösterilmiştir. (F-G) Uzunlamasına (F) ve enine kesitsel (G) oryantasyonda gösterilen kavisli, düzeltilmiş aktin filamentlerine sahip aktin demeti modeli. Segmentli zar mavi renkle gösterilmiştir. Panel D'de, Chimera'daki yoğunluk haritası "Mesh" harita stili olarak gösterilmiştir. Ölçek çubukları = 100 nm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
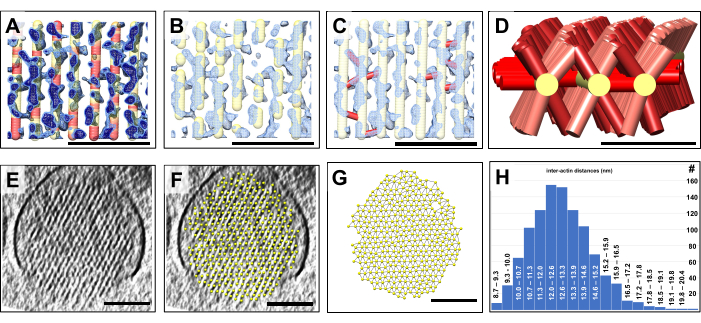
Şekil 4: Hacimsel model oluşturma kullanılarak aktin demetindeki boşlukların tespiti. Bilye-sopa modelleri kullanılarak hacimsel özelliklerin ölçülmesi. (A-C) Stereocilia yoğunluk haritasının ~ 10 dilim / 9.47 nm kalınlığında uzunlamasına bir levhasının küçük bir bölgesi, kavisli, düzeltilmiş aktin filament modeli ile birlikte mavi renkle gösterilmiştir. (A) Yoğunluk haritası, daha düşük bir yoğunluk eşiğinde (açık mavi) ve daha yüksek bir yoğunluk eşiğinde (koyu mavi) gösterilir. Aktin filament modelinin, karşılık gelen yoğunluğun olmadığı bölgeleri vardır. Bu tür konumlarda, model yoğunluk eksikliğini temsil etmek için kırmızı renk kodludur. Bu konumları aktin filamentlerindeki boşluklar olarak yorumluyoruz. (B) Bu tür boşluk konumlarında aktin filament yoğunluğunun eksikliğini yansıtan, parçalanmış görünen aktin filamentlerinin modeli. (C) Bitişik aktin filamentlerini köprülemek için güçlü bir yoğunluğun bulunduğu yerlere eklenen çapraz bağlama bağlantılarına (kırmızı ile gösterilmiştir) sahip aktin filamentlerinin modeli. (D) Seçilen üç model aktin filamenti, bitişik aktin filamentlerine çapraz konektörlerle gösterilmiştir (bunlar netlik için gösterilmemiştir). Hepsi olmasa da birçok olası interaktin filament pozisyonunun bağlayıcı proteinler tarafından işgal edildiğine dikkat edin; (E) Stereocilia yoğunluğunun enine kesitsel 30 dilim / 28.4 nm levhası. (F) Kesitli 30 dilim/28.4 nm stereocilia yoğunluk levhasına takılan aktin filamentlerinin modeli. (G) Altta yatan harita yoğunluğu olmayan aktin filamentlerinin modeli. Aktin filament model topları arasındaki ince bağlantılarla gösterildiği gibi, en yakın komşu aktin filament mesafelerinin otomatik olarak algılanması. (H) Aktin filament model mesafelerinin histogramı. A-C panellerinde, Chimera'daki yoğunluk haritası "Mesh" harita stili olarak gösterilir. (A-C) Ölçek çubukları = 50 nm. (D) Ölçek çubuğu = 25 nm. (E-G) Ölçek çubukları = 100 nm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Şekil 1: İki ek stereocilia veri setinden ilki için model oluşturma. (A-C) Stereocilia yoğunluk haritasının ~ 10 dilim / 9.47 nm kalınlığında uzunlamasına bir levhanın küçük bir bölgesi, ağ modu ekranı kullanılarak mavi renkle gösterilir. Başlangıçta yerleştirilen model kırmızı renkle gösterilir ve düzeltilmiş model sarı renkle gösterilir. (A) Yalnızca harita yoğunluğu. (B) Yoğunluk haritasına yerleştirilen ilk model. (C) Yoğunluk haritasına yerleştirilen düzeltilmiş model. (D-E) (D) ve (E) olmadan daha büyük stereocilia bölgesi, stereocilia yoğunluk haritasının ~ 10 dilim / 9.47 nm kalınlığında uzunlamasına bir levhasına takılan düzeltilmiş model. (F-G) Tüm stereocilia tomografi bölgesi gösterilir. (F) Yalnızca harita. (G) Düzeltilmiş model ile harita. (H) İlk ve düzeltilmiş modellerin üst üste binmesi. Ölçek çubukları = 100 nm Bu Dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 2: İki ek stereocilia veri setinden ikincisi için model oluşturma. (A-C) Stereocilia yoğunluk haritasının ~ 10 dilim / 9.47 nm kalınlığında uzunlamasına bir levhanın küçük bir bölgesi, ağ modu ekranı kullanılarak mavi renkle gösterilir. Başlangıçta yerleştirilen model kırmızı renkle gösterilir ve düzeltilmiş model sarı renkle gösterilir; (A) Yalnızca harita yoğunluğu. (B) Yoğunluk haritasına yerleştirilen ilk model. (C) Yoğunluk haritasına yerleştirilen düzeltilmiş model. (D-E) (D) ve (E) olmadan daha büyük stereocilia bölgesi, stereocilia yoğunluk haritasının ~ 10 dilim / 9.47 nm kalınlığında uzunlamasına bir levhasına takılan düzeltilmiş model. (F-G) Tüm stereocilia tomografi bölgesi gösterilir. (F) Yalnızca harita. (G) Düzeltilmiş model ile harita. (H) İlk ve düzeltilmiş modelin üst üste binmesi. Ölçek çubukları = 100 nm. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 1: Stereocilia modelleme için UCSF Chimera python betikleri. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Kodlama Dosyası 1: pblengths.py. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Kodlama Dosyası 2: RemoveCross.py. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Kodlama Dosyası 3: ActinFilamentPlane.py. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Kodlama Dosyası 4: dividelinks.py. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Kodlama Dosyası 5: FixingMarkerID.py. Bu dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Havza segmentasyonu gibi segmentasyon için otomatik yaklaşımların, saç hücresi stereocilia kriyo-elektron tomogramlarının yüksek gürültülü ve yüksek karmaşıklıklı ortamında başarısız olabileceğini gösterdik. Bu ipliksi ağın hangi kısmının aktin filamentlerini temsil ettiğini ve yerel çevre düzeyinde çapraz bağları neyin oluşturduğunu ayırt etmek, sadece küçük tomografik alt hacimleri incelerken en iyi ihtimalle zor görünmektedir. Bu çalışmada kullanılan model oluşturma yaklaşımı, aktin filamentlerinin oryantasyonu ve çapraz bağlayıcı yoğunlukları hakkında bir beklenti geliştirmeye yardımcı olan aktin demetinin büyük ölçekli düzenine ilişkin önceki bilgilerden yararlanır. Belki daha da önemlisi, bir insan beyninin yerel yoğunluk dağılımının ötesindeki daha geniş bağlamı göz önünde bulundurarak kalıpları kolayca bulabilmesidir, oysa bir bilgisayar algoritması yalnızca algoritma tarafından dikkate alınan nispeten küçük bir bölge için çalışır; bu nedenle, daha büyük ölçekli eğilimler kolayca dikkate alınamaz. Bir modeli küresel olarak bir yoğunluk katmanına sığdırarak, bir seferde tek aktin filamentlerinin küçük bölümleri için bir model oluşturmaya çalışırken ortaya çıkabilecek karışıklığı önledik. Tabii ki, bu tür bir küresel uyum, büyük mesafelere yayılan bir düzen varsayar. Bununla birlikte, aktin filamentlerinde beklenmedik küçük ama önemli bir kademeli bükülme olduğu için, küresel uyum yalnızca bir başlangıç yaklaşımıydı ve yoğunluk haritasına uyması için modelin yerel ayarlamalarını gerektirdi. İlk model iyi bir başlangıç noktası olduğundan, ayarlamalar yüksek güvenle yapılabilir. Yaklaşımımızın en büyük avantajlarından biri, yalnızca belirli bir yoğunluk bölgesini görüntülemeyi seçebilmemizdi, bu da manzaranın karmaşıklığını azaltmaya yardımcı oldu. Ayrıca, filament model ekseni boyunca harita yoğunluğu levhasının görüntülenmesi, daha küçük alt hacimleri görüntülerken büyük olasılıkla gözden kaçıracağımız beklenmedik eğriliğin belirlenmesine yardımcı oldu. İlk modelin yerleştirilmesi, ilgili aktin filament katmanının genel görünümü ile model ayarlamaları yapmak için ayrıntılı görünümler arasında geçiş yapmak için hızlı yakınlaştırma ve uzaklaştırmayı da kolaylaştırdı.
Protokol dahilindeki kritik adımlar, görsel incelemeden sonra haritanın döndürülmesini, modelin yoğunluk haritasına oluşturulmasını ve yerleştirilmesini ve ayrıca filament modelinin daha küçük segmentlere bölünmesini içeriyordu. Segmentlerin atom konumu daha sonra yoğunluk haritasına uyacak şekilde uzamsal olarak ayarlanabilir ve/veya boşlukları tespit etmek için renk kodlu hale getirilebilir.
Aktin modeli oluşturmanın bu yaklaşımı, 10-30 dilim/9.47-28.4 nm ortalama yoğunluk levhasının enine kesit görünümü kullanılarak filament yoğunluklarına bir dizi "atom" (yani top ve çubuk modelinin topları) yerleştirilerek de değiştirilebilir, bu daha sonra bağlarla bağlanabilir (yani, top ve çubuk modelinin çubukları). Burada ayrıntılı olarak açıklanan protokolün bir modifikasyonu olan bu yaklaşımı, saç hücresi stereocilia23'ün konik bölgesinde hacimsel model oluşturma için kullandık. Ayrıca, burada açıkladığımız gibi, hacimsel model oluşturma yaklaşımımız, membranların segmentasyonu ve model inşası için de çok uygundur.
Hacimsel model oluşturma, filamentli özellikler gösteren herhangi bir yoğunluk haritasına uygulanabilirken, burada tanımladığımız teknik, hacimsel bir modelin küresel bir uyumunun elde edilebildiği bir dizi düzenli aralıklı filamente sahip olduğumuzda en verimlidir. Aynı zamanda, yönlülüklerini kademeli bir şekilde değiştirmek için ipliksi özelliklere de bağlıdır. Filamentli yapılarda ani bükülmeler ve keskin dönüşler varsa, yaklaşımımız segmentasyon için özellikle yardımcı olmayabilir.
Bu arada, işbirlikçilerimiz, burada manuel segmentasyon için kullanılan benzer bir konsepti takip eden otomatik filament izleme için otomatik bir yaklaşım geliştirdiler30,31. İleriye dönük olarak, en iyi yaklaşım, başlangıçtaki seyrek bir modelin (hatta sadece birkaç topun) bir başlangıç noktası olarak yoğunluğa manuel olarak tanımlanması ve yerleştirilmesinin bir melezi olabilir ve ardından bir arama ve yerleştirme algoritmasının filamentlerin izlenmesini tamamlamasına izin verebilir.
Basitleştirilmiş hacimsel modeller, bir sistemin karmaşıklığını azaltır ve aktin filamentinin uca yakın bükülmesi gibi belirli modellerin daha iyi takdir edilmesini sağlar. Ayrıca, hacimsel model, seçilen ankraj bilyesi ve çubuk modeli etrafında bir yoğunluk bölgesini görüntülemek için bir "ankraj" olarak kullanılabilir, bu da bitişik aktin filamentleri arasındaki çapraz bağlayıcı yoğunluklarının algılanmasına ve görselleştirilmesine olanak tanır. Tek tek filamentleri seçme ve yoğunluğun tekrar görüntülendiği bir bölge olarak uygun yarıçapları ayarlama yeteneği, manzaranın ezici karmaşıklığının yönetilebilir bir düzeye indirilmesine olanak tanır.
Küresel uydurmanın bu hacimsel model oluşturma yaklaşımının ve ardından yerel ayarlamaların bir avantajı, aktin filamentlerinin kesintiye uğramış gibi göründüğü bölgeleri belirleyebilmemiz ve aktin filamentlerindeki önemli boşlukların harita yoğunluğunun olmaması ile gösterilmesiydi. Hacimsel bir top ve çubuk modeli yerleştirdiğimiz için, UCSF Chimera yazılım paketindeki her model top konumunu o konumdaki harita yoğunluğu değerine göre renk kodlayan bir rutinden yararlanabilirdik. Bu yaklaşım, kriyo-elektron tomogramımızda bulduğumuz biyolojik olarak önemli bir özellik olan ve geleneksel segmentasyon yaklaşımlarıyla tespit edilmesi ve görselleştirilmesi çok zor olan aktin demetindeki aktin filament boşluklarının hızlı bir şekilde tespit edilmesini ve görselleştirilmesini sağladı. Hacimsel modelimizin bir başka avantajı da, uzunluklar ve mesafeler dahil olmak üzere hacimsel özelliklerin kolayca elde edilebilmesidir, bu da gerçek sayılara ve dolayısıyla istatistiksel bir analizin yapılmasına olanak tanır.
Özetle, muhtemelen daha sonraki otomatik yerel uydurma ve filament izleme yetenekleriyle daha da artırılan etkileşimli manuel model noktası yerleştirme, elektron tomografik hücre altı hacimlerinin görselleştirilmesi ve kantitatif analizi için oldukça umut verici bir yaklaşımdır. Bunun nedeni, örüntü tanıma için insan beyninin gücünü ve model optimizasyonu için bilgisayar biliminin gücünü kullanmasıdır.
Açıklamalar
Yazarlar, rekabet eden finansal çıkarları veya diğer çıkar çatışmaları olmadığını beyan ederler.
Teşekkürler
Dr. Peter Barr-Gillespie ve ekibine numune hazırlamadaki rolleri için ve Auer laboratuvarı ve Dr. Dorit Hanein laboratuvarının eski üyelerine tomografik veri toplamadaki rolleri için teşekkür ederiz. Ayrıca, çeşitli UCSF Chimera komut dosyalarını sağladığı için UCSF Biyohesaplama, Görselleştirme ve Bilişim Kaynağı'ndan (RBVI) Tom Goddard'a teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/download.html | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=win64/chimera-1.16-win64.exe | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=mac64/chimera-1.16-mac64.dmg | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=linux_x86_64/chimera-1.16-linux_x86_64.bin | |
| Excel | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Falcon II | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/accessories-em/falcon-detector.html | |
| IMOD | University of Colorado | Version 4.11.1 https://bio3d.colorado.edu/imod/download.html | |
| PC Desktop | Intel | Windows 10, ver. 22H2 | |
| PC Laptop | Gigabyte | Windows 10, ver. 22H2 | |
| Powerpoint | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Titan Krios Electron Microscope | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/transmission-electron-microscopes/krios-g4-cryo-tem.html | |
| Word | Microsoft | Version 2211 https://www.office.com/?auth=1 |
Referanslar
- Downing, K. H., Sui, H., Auer, M. Electron tomography: A 3D view of the subcellular world. Analytical Chemistry. 79 (21), 7949-7957 (2007).
- Koning, R. I., Koster, A. J. Cellular nanoimaging by cryo electron tomography. Methods in Molecular Biology. 950, 227-251 (2013).
- Asano, S., Engel, B. D., Baumeister, W. In situ cryo-electron tomography: A post-reductionist approach to structural biology. Journal of Molecular Biology. 428 (2), 332-343 (2016).
- Serwas, D., Davies, K. M. Getting started with in situ cryo-electron tomography. Methods in Molecular Biology. 2215, 3-23 (2021).
- McDonald, K. L., Auer, M. High-pressure freezing, cellular tomography, and structural cell biology. Biotechniques. 41 (2), 137-143 (2006).
- Narasimha, R., et al. Evaluation of denoising algorithms for biological electron tomography. Journal of Structural Biology. 164 (1), 7-17 (2008).
- Frangakis, A. S. It's noisy out there! A review of denoising techniques in cryo-electron tomography. Journal of Structural Biology. 213 (4), 107804(2021).
- Frangakis, A. S., Hegerl, R. Noise reduction in electron tomographic reconstructions using nonlinear anisotropic diffusion. Journal of Structural Biology. 135 (3), 239-250 (2001).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. Journal of Structural Biology. 144 (1-2), 114-122 (2003).
- vander Heide, P., Xu, X. -P., Marsh, B. J., Hanein, D., Volkmann, N. Efficient automatic noise reduction of electron tomographic reconstructions based on iterative median filtering. Journal of Structural Biology. 158 (2), 196-204 (2007).
- Volkmann, N. Methods for segmentation and interpretation of electron tomographic reconstructions. Methods in Enzymology. 483, 31-46 (2010).
- Böhm, J., et al. Toward detecting and identifying macromolecules in a cellular context: template matching applied to electron tomograms. Proceedings of the National Academy of Sciences. 97 (26), 14245-14250 (2000).
- Frangakis, A. S., et al. Identification of macromolecular complexes in cryoelectron tomograms of phantom cells. Proceedings of the National Academy of Sciences. 99 (22), 14153-14158 (2002).
- Lebbink, M. N., et al. Template matching as a tool for annotation of tomograms of stained biological structures. Journal of Structural Biology. 158 (3), 327-335 (2007).
- Rigort, A., et al. Automated segmentation of electron tomograms for a quantitative description of actin filament networks. Journal of Structural Biology. 177 (1), 135-144 (2012).
- Volkmann, N. A novel three-dimensional variant of the watershed transform for segmentation of electron density maps. Journal of Structural Biology. 138 (1-2), 123-129 (2002).
- Bajaj, C., Yu, Z., Auer, M. Volumetric feature extraction and visualization of tomographic molecular imaging. Journal of Structural Biology. 144 (1-2), 132-143 (2003).
- Yu, Z., Frangakis, A. S. Classification of electron sub-tomograms with neural networks and its application to template-matching. Journal of Structural Biology. 174 (3), 494-504 (2011).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Tilney, L. G., Derosier, D. J., Mulroy, M. J. The organization of actin filaments in the stereocilia of cochlear hair cells. The Journal of Cell Biology. 86 (1), 244-259 (1980).
- Tilney, L. G., Tilney, M. S., DeRosier, D. J. Actin filaments, stereocilia, and hair cells: how cells count and measure. Annual Review of Cell Biology. 8, 257-274 (1992).
- Metlagel, Z., et al. Electron cryo-tomography of vestibular hair-cell stereocilia. Journal of Structural Biology. 206 (2), 149-155 (2019).
- Song, J., et al. A cryo-tomography-based volumetric model of the actin core of mouse vestibular hair cell stereocilia lacking plastin 1. Journal of Structural Biology. 210 (1), 107461(2020).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Agulleiro, J. I., Fernandez, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27 (4), 582-583 (2011).
- Fernandez, J. J. Computational methods for electron tomography. Micron. 43 (10), 1010-1030 (2012).
- Chen, H., Clyborne, W. K., Sedat, J. W., Agard, D. A. Priism: an integrated system for display and analysis of 3-D microscope images. Biomedical Image Processing and Three-Dimensional Microscopy. 1660, 784-790 (1992).
- Pettersen, E. F., et al. UCSF Chimera-a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25 (13), 1605-1612 (2004).
- Goddard, T. D., Huang, C. C., Ferrin, T. E. Visualizing density maps with UCSF Chimera. Journal of Structural Biology. 157 (1), 281-287 (2007).
- Sazzed, S., et al. Tracing actin filament bundles in three-dimensional electron tomography density maps of hair cell stereocilia. Molecules. 23 (4), 882(2018).
- Sazzed, S., Scheible, P., He, P., Wriggers, J. Spaghetti tracer: A framework for tracing semiregular filamentous densities in 3D tomograms. Biomolecules. 12 (8), 1022(2022).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır