Method Article
Les modèles volumétriques simplifiés comme stratégie efficace pour segmenter les réseaux d’actine dans les cryo-tomogrammes d’électrons
Dans cet article
Résumé
Ici, nous présentons un protocole permettant de placer des modèles volumétriques simplifiés dans des volumes 3D tomographiques complexes et bruyants. Cela permet la segmentation rapide des densités de filaments d’actine, la détection de la flexion systématique des filaments et des lacunes dans les filaments des faisceaux de cheveux, ainsi que la quantification pratique des propriétés du modèle volumétrique, telles que les distances.
Résumé
Des méthodes efficaces pour l’extraction de caractéristiques d’intérêt restent l’un des plus grands défis pour l’interprétation des cryo-tomogrammes électroniques. Diverses approches automatisées ont été proposées, dont beaucoup fonctionnent bien pour les ensembles de données à contraste élevé où les caractéristiques d’intérêt peuvent être facilement détectées et sont clairement séparées les unes des autres. Nos ensembles de données de cryo-tomographie d’électrons stéréocils de l’oreille interne sont caractérisés par un réseau dense de filaments d’actine hexagonaux qui sont fréquemment interconnectés. Ces caractéristiques rendent la segmentation automatisée très difficile, aggravée par l’environnement très bruyant des cryo-tomogrammes électroniques et la grande complexité des caractéristiques densément emballées. En utilisant des connaissances préalables sur l’organisation du faisceau d’actine, nous avons placé des couches d’un modèle d’actine hautement simplifié pour obtenir d’abord un ajustement global à la carte de densité, suivi d’ajustements régionaux et locaux du modèle. Nous montrons que la construction d’un modèle volumétrique nous permet non seulement de faire face à la grande complexité, mais fournit également des mesures précises et des statistiques sur le faisceau d’actine. Les modèles volumétriques servent également de points d’ancrage pour la segmentation locale, comme dans le cas des connecteurs croisés actine-actine. La construction de modèles volumétriques, en particulier lorsqu’elle est complétée par des approches d’ajustement automatisées basées sur ordinateur, peut être une alternative puissante lorsque les approches de segmentation automatisées conventionnelles ne sont pas couronnées de succès.
Introduction
La cryotomographie électronique permet de visualiser des organites entiers ou des parties de cellules et de tissus à des résolutions nanométriques dans leur état quasi natif 1,2,3 en utilisant soit la congélation en plongée4, soit la vitrification ultra-rapide par congélation à haute pression5. Étant donné que seule une dose limitée d’électrons peut être tolérée par l’échantillon cryoconservé, non coloré et hydraté par congélation, les données 3D tomographiques sont très bruitées. Ce bruit peut souvent être considérablement réduit par une variété d’algorithmes de filtrage du bruit 6,7, y compris la diffusion anisotrope non linéaire8, le filtrage bilatéral9 et le filtrage médian récursif10.
De plus, les limitations d’inclinaison de la platine du microscope, entraînant un manque d’information, et le fait que l’épaisseur de l’échantillon augmente à des angles d’inclinaison élevés, conduisent à des reconstructions 3D avec une résolution anisotrope. Cela signifie que la densité est étalée dans la troisième dimension en raison d’une résolution plus faible dans la direction Z. En conséquence, la forme des macromolécules semble déformée (c’est-à-dire moins bien définie et allongée dans la troisième dimension).
L’un des plus grands défis dans l’interprétation des données tomographiques est l’extraction automatisée des caractéristiques pertinentes, également connue sous le nom de segmentation11. Avec suffisamment de caractéristiques de forme uniques et un faible bruit, les machines macromoléculaires dans des volumes 3D complexes peuvent être identifiées par correspondance de modèle 12,13,14 ; Cependant, le succès de la correspondance des modèles dépend de la résolution du tomogramme, d’un modèle de recherche approprié, ainsi que des caractéristiques de taille et de forme des volumes de fonctionnalités. Si les caractéristiques d’intérêt sont suffisamment espacées et que les motifs répétitifs (tels que les grandes machines macromoléculaires) peuvent être facilement identifiés, les sous-volumes du tomogramme peuvent être combinés pour augmenter le rapport signal/bruit et pour calculer la moyenne des distorsions de forme des particules individuelles. La segmentation automatisée d’un réseau de filaments d’actine dans des tomogrammes électroniques du bord mince de cellules de Dictyostelium discoideum congelées-hydratées par correspondance de matrice a été rapportée15.
Cependant, si les caractéristiques d’intérêt sont rapprochées, l’anisotropie de résolution des données peut conduire à un étalement des densités de la carte dans la direction Z (le long de la direction du faisceau d’électrons), entraînant une fusion apparente de l’enveloppe de densité de machines macromoléculaires ou de complexes supramoléculaires rapprochés. Dans de tels cas, les approches automatisées de segmentation, telles que la segmentation des bassins versants16, la segmentation des limites17 ou diverses approches de classification basées sur l’apprentissage automatique18,19, peuvent ne pas être en mesure de reconnaître les caractéristiques d’intérêt ou d’établir une limite correcte autour d’un objet d’intérêt. Souvent, on se retrouve soit avec quelques très gros morceaux, soit avec un volume fortement sur-segmenté, où beaucoup d’efforts sont nécessaires pour fusionner de nombreux petits morceaux jusqu’à ce que le trait d’intérêt soit perçu comme complet. Une telle conservation manuelle des résultats de segmentation peut être très laborieuse et peut même échouer complètement lorsque la structure d’intérêt est un réseau de filaments étroitement espacés qui sont interconnectés via de courts linkers. Dans ce gigantesque réseau de structures filamenteuses, il peut être difficile de s’orienter. En effet, en raison de l’anisotropie de la résolution, les densités semblent se fondre les unes dans les autres, ce qui représente un formidable défi pour les approches de segmentation manuelle automatisées et interactives. En conséquence, on peut facilement « sauter » entre les filaments en n’inspectant que visuellement de petites régions.
Heureusement, dans le cas du faisceau d’actine dans les stéréocils des cellules ciliées de l’oreille interne, nous avons des connaissances sur l’organisation globale du faisceau d’actine et la directionnalité des filaments d’actine20,21. Le faisceau d’actine se compose de centaines de filaments d’actine hexagonalement denses de 6 à 8 nm de diamètre, qui sont espacés d’environ 12 à 13 nm les uns des autres22.
Cela nous a permis d’adopter une approche assez différente de la segmentation, basée sur des modèles simplifiés à bille et bâton pour représenter les filaments d’actine. La stratégie consistait à placer simultanément un ensemble régulier idéalisé de modèles de filaments dans des plaques de cartes de densité de cryo-tomographie électronique pour construire un modèle 3D du faisceau d’actine couche par couche. Nous nous sommes assurés que le modèle avait un ajustement général à la carte de densité avant d’effectuer des ajustements locaux sur des modèles de filaments individuels ou des groupes de modèles de filaments pour correspondre étroitement à la carte de densité. En codant automatiquement par couleur la valeur de densité de la carte à l’emplacement du modèle de filament, nous avons pu détecter facilement les lacunes apparentes dans le faisceau d’actine. Les modèles volumétriques permettent une analyse quantitative des propriétés volumétriques, telles que les distances entre les filaments d’actine, et conduisent également à un affichage simplifié de l’organisation globale du réseau filamenteux 3D.
En outre, les modèles peuvent également servir de structures d’ancrage pour la segmentation de caractéristiques supplémentaires, telles que les liaisons actine-actine, car des modèles de filaments individuels peuvent être sélectionnés, autour desquels des zones de densité de carte de rayon appropriées peuvent être générées pour l’inspection et une segmentation plus poussée.
Nous pensons que notre approche de segmentation basée sur un modèle volumétrique est particulièrement utile pour les grands réseaux de structures filamenteuses qui peuvent contenir des lacunes et des inter-connexions interfilamentaires. Les algorithmes de segmentation ont tendance à fonctionner localement, alors que le cerveau humain prend en compte de plus grandes zones et est donc supérieur aux ordinateurs lorsqu’il s’agit de reconnaître les structures des filaments, même dans un environnement complexe et très bruyant.
Protocole
Le protocole suit les directives du comité d’éthique de la recherche humaine de l’Université du Sud-Est.
1. Source de données de cryo-tomographie électronique pour la construction de modèles volumétriques
REMARQUE : Les reconstructions cryo-tomographiques d’électrons stéréocils utilisées pour la construction d’un modèle volumétrique ont déjà été publiées22,23 et ont été obtenues comme décrit par Metlagel et al.22.
Les scripts python UCSF Chimera pour la modélisation des stéréocils sont fournis dans le fichier supplémentaire 1, le fichier de codage supplémentaire 1, le fichier de codage supplémentaire 2, le fichier de codage supplémentaire 3, le fichier de codage supplémentaire 4 et le fichier de codage supplémentaire 5.
- Brièvement, épongez les stéréocils de la surface apicale de l’épithélium sensoriel de l’utricule de souris sur le film de support en dentelle de carbone d’une grille de microscope électronique (EM). Ensuite, effectuez une vitrification par congélation plongeante ultra-rapide et une collecte de données cryo-tomographiques électroniques mono-axe sur une microscopie électronique à transmission cryogénique (cryo-MET), opérée à 300 kV avec une défocalisation nominale de 3,5-4,5 μm, à l’aide d’une caméra de type CMOS en mode intégration, d’une taille de pixel de 0,47 à 0,59 nm (voir Tableau des matériaux).
- Une dose typique pour la collecte de données à axe unique est généralement de 80 à 100 électrons/Å2. Reconstruisez les volumes 3D du tomogramme à l’aide du progiciel IMOD24, soit par rétroprojection pondérée, soit par la méthode SIRT 25,26.
- Débruiter les tomogrammes à l’aide d’un filtrage récursif, médian ou bilatéral dans Priism27 ou de l’option de diffusion anisotrope non linéaire dans IMOD.
REMARQUE : Le principal progiciel utilisé pour la visualisation interactive, la construction de modèles volumétriques et l’analyse quantitative était le logiciel UCSF Chimera28,29. Tous les progiciels utilisés sont répertoriés dans la table des matériaux.
2. Préparation des données de cryo-tomographie électronique pour la construction de modèles volumétriques
- Rotation du tomogramme
REMARQUE : Le but de cette étape est de trouver les « angles optimaux » dans lesquels le noyau d’actine est aligné dans deux des trois axes (x et z), de sorte que le placement des modèles en 3D puisse se faire avec un seul axe à se soucier. Les étapes suivantes sont effectuées dans le progiciel IMOD.- Ouvrez 3dmod (IMOD) et ouvrez le fichier de pile d’images 3D au format de fichier .mrc. Assurez-vous que la pile 3D est en mode niveaux de gris 16 bits et qu’elle affiche les dimensions X, Y et Z correctes dans l’en-tête de son fichier image. Si nécessaire, apportez des corrections à l’aide de la commande alterheader : alterheader -d (x_pixelspacing),(y_pixelspacing),(z_pixelspacing) inputfilename.mrc dans l’invite de commande. Si la pile 3D est au format de fichier TIFF, utilisez la commande tif2mrc inputfilename.tif outputfilename.mrc dans l’invite de commande pour créer un fichier .mrc.
- Inspectez visuellement le tomogramme et ouvrez le « slicer » (en appuyant sur la barre oblique inverse « \ ») pour trouver les meilleurs angles de rotation dans les dimensions X, Y et Z pour l’alignement du plan du filament d’actine avec le plan Z. Manipulez la rotation X, la rotation Y, la barre de rotation Z pour trouver l’angle optimal auquel les filaments d’actine sont alignés sur les plans X et Z. Notez que la modification de l’épaisseur moyenne pour l’affichage du tomogramme (Thick : Img) peut aider à trouver une quantité idéale de moyenne de la densité dans le plan Z et donc à augmenter le contraste. Effectuez un zoom avant et arrière sur le tomogramme à l’aide des touches + et -, respectivement, du clavier.
- Une fois que les angles idéaux de rotation ont été identifiés, faites pivoter le tomogramme en tapant la commande IMOD rotatevol -a (Z angles de rotation),(Y angles de rotation),(X angles de rotation) -s (x-width),(y-height),(z-depth) inputfilename.mrc outputfilename.mrc dans l’invite de commande. Assurez-vous de donner à la carte suffisamment d’espace pour s’adapter à la rotation afin d’éviter de couper des parties de la carte lors de la rotation.
REMARQUE : Pour plus d’informations sur l’utilisation de la commande rotatevol, utilisez l’option du menu d’aide ou consultez l’URL suivante (https://bio3d.colorado.edu/imod/doc/man/rotatevol.html). Cette commande (rotatevol) ne modifie pas la carte actuellement affichée dans la fenêtre IMOD, mais crée plutôt un nouveau fichier de carte avec une carte de densité en cours de rotation. - À l’aide de la fenêtre de segmentation, identifiez les coordonnées X, Y et Z de deux coins opposés d’une carte pour identifier la zone de recadrage contenant le stéréocil à l’aide de la commande trimvol .
- Utilisez la commande IMOD Trimvol pour recadrer la carte aux coordonnées précédemment obtenues, en tapant trimvol -x (x-coordiante 1),( x-coordiante 2) -y (y-coordiante 1),( y-coordiante 2) -z (z-coordiante 1),( z-coordiante 2) inputfilename.mrc outputfilename.mrc dans l’invite de commande. Maintenant, la carte est beaucoup plus petite, et donc plus facile et plus rapide à utiliser à partir de maintenant.
REMARQUE : Pour plus d’informations sur l’utilisation de la commande trimvol, utilisez l’option du menu d’aide ou visitez l’URL suivante (https://bio3d.colorado.edu/imod/doc/man/trimvol.html). Ces étapes créent un fichier .mrc qui a été pivoté et tronqué.
- Filtrage par tomogramme
REMARQUE : Cette étape utilise un filtre de diffusion anisotrope non linéaire (NAD) fourni par IMOD pour la réduction du bruit. Nous avons utilisé comme valeur initiale celles recommandées dans la page d’aide d’IMOD pour le filtrage de diffusion anisotrope non linéaire, comme indiqué dans l’URL suivante (https://bio3d.colorado.edu/imod/doc/NADexample.html), et avons été utilisées comme valeurs initiales.- Tapez etomo dans l’invite de commande pour lancer l’interface utilisateur graphique etomo d’IMOD.
- Sélectionnez l’option Diffusion anisotrope non linéaire dans le menu etomo et sélectionnez le fichier de carte (Choisir un volume).
- Afin de trouver une valeur K et le nombre d’itérations qui fournissent les meilleurs résultats de filtrage, tels qu’ils sont jugés par l’inspection visuelle, appliquez le filtre NAD à un volume de test plus petit pour extraire le volume de test de filtre NAD spécifié. Cliquez sur l’outil Rubberband en haut de l’interface 3dmod, faites glisser le clic gauche pour sélectionner le volume de test, puis cliquez sur les boutons Hi et Lo pour indiquer que la tranche Z est la limite du volume de test (Obtenir la plage de volumes de test de 3dmod). Extrayez le volume de test en cliquant sur Extraire le volume de test.
- Filtrez le volume de test à l’aide de différentes valeurs K (Recherche d’une valeur K pour le volume de test > Liste des valeurs K), telles que 0,1, 1,5, 10, 15, 25, 30, 50 et 75. Effectuez un filtrage NAD avec chacune des valeurs K spécifiées (Exécuter avec différentes valeurs K > Afficher les résultats du test de différentes valeurs K). Utilisez la fenêtre de segment pour évaluer les performances du filtre NAD et sélectionnez une valeur K qui sera utilisée pour filtrer l’ensemble du volume.
- Utilisez la valeur K identifiée à l’étape 2.2.4 et testez différents nombres d’itérations, tels que 2, 5, 8, 11, 15 et 21 (Recherche d’un numéro d’itération pour le volume de test > Liste des itérations). Évaluez les performances du filtre NAD pour différentes itérations en cliquant sur Afficher les résultats des tests d’itération différente ou à l’aide d’un outil de segmentage. Choisissez une valeur pour un nombre différent d’itérations.
- Utilisez la valeur K identifiée et le nombre d’itérations (Filtrer le volume complet > la valeur K et les itérations) et filtrez l’ensemble du volume (Filtrer le volume complet). Un volume nouvellement filtré apparaîtra dans le même répertoire que celui où se trouve le volume complet chargé avec une extension de fichier .nad attachée au fichier .mrc.
REMARQUE : Ces étapes créent un fichier .mrc qui a été filtré avec un filtre NAD et qui affiche un bruit plus faible et donc un meilleur rapport signal/bruit.
3. Construction de modèles volumétriques
- Préparation du placement du modèle 3D
REMARQUE : Le but de cette étape est de préparer et d’analyser la carte pour placer des modèles volumétriques 3D dans la carte de densité pivotée et débruitée à l’aide du programme Chimera de l’UCSF.- À l’aide de la fenêtre du slicer, obtenez une vue en coupe transversale (plaque de 30 slices/28,4 nm) pour identifier les coordonnées Z du centre de la dalle de densité, dans laquelle le modèle de filament d’actine doit être placé. Cliquez sur le centre de la densité du filament et notez les coordonnées Z dans la fenêtre principale d’IMOD.
- Gardez note des coordonnées identifiées pour le placement futur du modèle dans UCSF Chimera.
REMARQUE : Les coordonnées Z notées à partir de cette étape sont utilisées à l’étape 4 pour trouver et placer facilement des modèles de filaments d’actine préfabriqués aux hauteurs Z correctes, ce qui accélère le placement du modèle.
- Placement du modèle 3D
REMARQUE : Le but de cette étape est de placer un modèle volumétrique 3D dans la carte de densité préparée à l’aide de UCSF Chimera.- Dans UCSF Chimera, ouvrez la carte filtrée et pivotée (Fichier > Ouvrir)
- Vérifiez si les paramètres de la carte sont correctement définis en sélectionnant Entités > plans et Entités > coordonnées dans le visualiseur de volume pour ouvrir l’interface des entités et des coordonnées. Vérifiez la taille du voxel pour voir si l’espacement correct des voxels est défini à l’aide de la commande d’en-tête dans l’invite de commande (dans IMOD). Si ce n’est pas le cas, corrigez l’interface de taille de voxel pour qu’elle s’adapte à l’espacement correct des voxels. Centrez la carte pour placer la caméra au centre de la fenêtre d’affichage (Origin Index > Center).
- Ouvrez la fenêtre Contrôle de l’appareil photo (Outils > Contrôle de l’affichage > appareil photo). Ensuite, réglez l’affichage de la caméra sur la vue orthographique (projection > orthographique)
- Chargez le modèle de script python prédéfini (Fichier > Ouvrir > ActinFilamentPlane.py) et ouvrez autant de scripts de plans de filaments d’actine qu’il y a de plans de filaments d’actine sur la carte.
- Déplacez les modèles chargés en activant le mode Mouvement de la souris (Outils > Mouvement > Mode Mouvement de la souris). Activez l’option de mouvement de la souris (Définir le mode de mouvement de la souris > Déplacer la sélection). Utilisez Ctrl + Glisser pour sélectionner plusieurs modèles présents dans la session et cliquez avec le bouton droit de la souris (ou toute autre touche spécifiée dans Préférence > souris) pour déplacer les modèles dans la direction souhaitée.
REMARQUE : Les modèles ont été créés avec des boules et des bâtons d’une certaine épaisseur (rayon de ActinFilamentPlane.py script) et avec certains espacements (Lattice_Spacing de ActinFilamentPlane.py script). - Pour visualiser uniquement le modèle qui vous intéresse, ouvrez le Panneau Modèle (Outils > Commandes générales > Panneau Modèle). Cochez les cases sous S (Afficher) pour activer ou désactiver la visibilité d’un modèle donné.
- Activez le panneau de ligne de commande (Outils > Commandes générales > Ligne de commande).
- Après avoir placé chaque modèle de plan de filament d’actine à sa hauteur Z appropriée, supprimez tous les filaments d’actine en excès dans chaque modèle de plan de filament d’actine en sélectionnant la partie excédentaire des modèles et en tapant del sel (Supprimer la sélection) dans la ligne de commande en bas de la fenêtre principale de l’UCSF Chimera.
- Ne visualisez qu’un seul plan de filament d’actine à la fois, en accédant à la fenêtre du panneau Modèle et en sélectionnant et en masquant tous les autres modèles de filaments d’actine (Ctrl + Clic > Masquer).
- Pour visualiser des parties de la densité cartographique qui se trouvent à proximité immédiate du modèle de plan de filament d’actine unique d’intérêt, sélectionnez le modèle (Panneau de modèle > sélectionner) et ouvrez le panneau de configuration de zone dans le Visualiseur de volume (Visualiseur de volume > Entités > Zone). Définissez le rayon de la zone sur 100 Å, puis cliquez sur Zone pour placer la carte dans un rayon de 100 Å (= 10 nm) par rapport au modèle sélectionné. Notez que la dimension dans Chimera est définie en Angstrøm (1 Å = 0,1 nm).
- Si nécessaire, ajustez le modèle pour l’adapter à la carte de densité en activant le mode Mouvement de la souris. Sélectionnez plusieurs modèles qui ne sont pas dans la bonne position pour déplacer les modèles à placer correctement à l’intérieur de la carte de densité de zone.
- Pour pallier un bug de programmation dans ActinFilamentPlane.py, exécutez le script FixingMarkerID.py , qui corrige le MarkerID manquant des « atomes » (balles dans le modèle à bille et bâton). Sélectionnez le modèle entier et tapez runscript FixingMarkerID.py dans la ligne de commande (le script doit être spécifié par le répertoire correct, tel que C : \ répertoire \ Script.py).
- Si la carte de densité indique que le filament d’actine est courbé, assurez-vous de diviser le filament d’actine droit en plusieurs parties pour tenir compte de la courbure en sélectionnant les modèles souhaités et en tapant runscript dividelinks.py numéro. Des marqueurs supplémentaires (spécifiés par le numéro dans la commande) sont placés à l’intérieur du modèle d’actine à égale distance les uns des autres, créant des « articulations » où le modèle peut être plié.
- Ajustez les articulations pour qu’elles reflètent la courbure en déplaçant des marqueurs supplémentaires le long de la carte zonée à l’aide du mode Mouvement de la souris. Ajoutez des marqueurs supplémentaires au modèle si nécessaire à l’aide du script dividelinks.py .
- Pour vérifier que tous les filaments d’actine sont correctement placés à l’intérieur des densités d’actine, utilisez les barres d’écrêtage en cliquant sur Outils > Contrôle d’affichage > caméra dans le menu déroulant UCSF Chimera pour ouvrir la fenêtre Contrôle de l’appareil photo , puis sélectionnez Vue latérale pour voir les deux barres d’écrêtage (plans d’écrêtage proche et éloigné) de l’appareil photo. Faites glisser deux barres jaunes dans une fente étroite en cliquant avec le bouton gauche de la souris et faites glisser la partie de découpage à l’aide du bouton central de la souris pour inspecter les modèles un petit bout à la fois.
REMARQUE : Le modèle de base d’actine stéréocilium est maintenant terminé.
- Segmentation membranaire
REMARQUE : Le but de cette étape est de créer un modèle de surface de la membrane des stéréocils.- La segmentation des surfaces utilise l’outil Traceur de volume (Outils > Données de volume > Traceur de volume).
- Dans la fenêtre Traceur de volume , cliquez sur Souris et cochez uniquement les options Placer les marqueurs sur les plans de données et Lier le nouveau marqueur au marqueur sélectionné. Cela permet de placer précisément les marqueurs sur le même plan Z (lorsqu’un seul plan Z est affiché). Cochez également la case Placer les marqueurs à l’aide du bouton central de la souris dans la fenêtre du traceur de volume .
- Créez un nouveau jeu de marqueurs (Fichier > Nouveau jeu de marqueurs). Le jeu de marqueurs 1 doit être créé et sélectionné dans la fenêtre Traceur de volume .
- Utilisez le visualiseur de volume pour afficher des plans Z uniques dans la fenêtre d’affichage principale (Axe à Z > Un.)
- À l’aide du bouton central de la souris, tracez la membrane d’une extrémité à l’autre de la membrane visible. Tous les marqueurs doivent être reliés par des liens d’un bout à l’autre.
- Désélectionnez le dernier marqueur placé en appuyant sur Ctrl + Clic sur l’arrière-plan où se trouve aucun élément sélectionnable. Cela garantit que le prochain marqueur à placer est connecté avec des liens dans le nouveau plan, et non avec une trace de membrane précédente d’un autre plan Z.
- Répétez le traçage de la membrane tous les 10 nm dans le même sens que le traçage d’une extrémité de la membrane visible à l’autre côté.
- Une fois que de nombreuses bandes parallèles de membranes sont segmentées, accédez à la fenêtre Traceur de volume et cliquez sur Entités > surfaces. Cela active l’interface de surface dans la fenêtre Traceur de volume . Cliquez sur Créer en regard de l’interface Surfaces pour créer des surfaces entre les bandes de segmentation de membrane. Cela crée un mince rabat du modèle de surface qui remplit les traces de membrane espacées.
REMARQUE : La création du modèle de surface de la membrane de stéréocilium est maintenant terminée.
- Modélisation de l’agent de réticulation
REMARQUE : Le but de cette étape est de modéliser les réticulants entre les filaments d’actine.- Les réticulants peuvent également être placés à l’aide de l’outil Volume Tracer .
- Dans la fenêtre Traceur de volume , cliquez avec le bouton gauche de la souris pour cocher uniquement Placer des marqueurs sur les surfaces et Lier un nouveau marqueur au marqueur sélectionné. Ensuite, cochez Placer les marqueurs à l’aide du bouton central de la souris dans la fenêtre du traceur de volume .
- Visualisez uniquement les densités autour du modèle de plan de filament d’actine unique d’intérêt à l’aide de la fonction zone. Réglez le rayon de la zone sur 100 Å pour visualiser la carte dans un rayon de 100 Å à partir du modèle sélectionné.
- Placez des marqueurs sur les densités visibles de maillonneurs en cliquant sur le bouton central de la souris. Une fois que deux marqueurs sont placés (et qu’un seul réticulant est modélisé), assurez-vous de désélectionner le deuxième marqueur pour éviter que les liens ne soient continuellement connectés au modèle de réticulant suivant.
- Répétez les étapes ci-dessus pour chaque plan de filament d’actine et répétez tout le processus ci-dessus dans les trois principales directions dans lesquelles les agents de réticulation peuvent être localisés. Lorsque vous zonez les plans des filaments d’actine dans la direction diagonale, sélectionnez manuellement les modèles de filaments d’actine individuellement.
REMARQUE : Pour chacun des modèles de réticulants d’actine, nous pouvons enregistrer la position sur les filaments d’actine auxquels les réticulants présumés se connectent. Ces informations pourraient, en principe, être extraites pour une analyse de suivi à l’aide de la moyenne des sous-tomogrammes, ce qui dépasse le cadre de la présente étude.
4. Analyse quantitative du modèle 3D
- Analyse des écarts
REMARQUE : Le but de cette étape est de détecter les lacunes dans les filaments d’actine.- Les éléments sélectionnés peuvent être analysés. Spécifiez leurs paramètres, comme leur couleur, à l’aide de l’outil Inspecter la sélection (Action > Inspecter).
- La couleur de l’élément sélectionné peut être spécifiée en sélectionnant n’importe quel élément sélectionnable dans UCSF Chimera et en modifiant la couleur en regard de la section Couleur de la fenêtre Inspecter la sélection .
- Spécifiez la taille des marqueurs et des liens en sélectionnant des marqueurs et des liens dans UCSF Chimera et en modifiant les valeurs en regard de la section rayon de la fenêtre Inspecter la sélection , où se trouvent les unités en Angstrøm.
- Effectuez une analyse des écarts par détection automatique des écarts de densité, à l’aide de la fonction Valeurs aux positions atomiques (Outils > les données de volume > Valeurs aux positions des atomes).
- Sélectionnez la carte dans laquelle les valeurs seront lues en sélectionnant la carte souhaitée en regard de la section Données de volume de la fenêtre Valeurs aux positions atomiques . Sélectionnez le modèle dans lequel tous les marqueurs seront affectés à un paramètre en sélectionnant le modèle souhaité à côté de la section Molécule .
- Avec le bouton Histogramme en bas de la fenêtre Valeurs aux positions atomiques , un histogramme est créé dans une nouvelle fenêtre appelée Rendu/Sélection par attribut. Ctrl + Clic sur l’histogramme ajoutera ou supprimera des points de coupure, pour lesquels différentes couleurs peuvent être sélectionnées. Décochez la case Garder opaque pour appliquer une couleur transparente à la scène. Les marqueurs ayant une densité inférieure à un seuil donné, qui représentent des lacunes, peuvent non seulement être colorés, mais aussi sélectionnés, et ainsi nous permettre de compter les marqueurs numériques dans la région de l’espace. Pour sélectionner, cliquez sur Sélectionner > Attribut > value_mapname.
- Détermination des distances d’inter-actine
- Pour mesurer les distances d’inter-actine, placez les marqueurs sur un seul plan XZ (appelé plan Y dans UCSF Chimera) à l’aide de la fenêtre Traceur de volume , en cliquant sur Souris, en cochant uniquement Placer les marqueurs sur les plans de données et en décochant Lier le nouveau marqueur au marqueur sélectionné. Cela permet de placer avec précision les marqueurs sur le même plan Y (lorsqu’un seul plan Z est affiché). Cochez également la case Placer les marqueurs à l’aide du bouton central de la souris dans la fenêtre Traceur de volume et placez les marqueurs sur un plan Y cible d’intérêt pour la mesure de la distance d’inter-actine.
- Une fois que les positions d’actine d’un seul plan Y sont marquées avec succès, sélectionnez tous les marqueurs et tapez la commande findclash #model-spec test self overlapCutoff -200 hbondAllowance 0 log true Linewidth 10 pbColor black dans la ligne de commande en bas du plan de filament d’actine. Cela permet de trouver tous les conflits entre marqueurs à une distance de 200 Å (overlapCutoff -200) et de créer des lignes noires (pbColor black) d’une épaisseur de 10 Å (largeur de ligne 10) entre les marqueurs.
- Comme il y a des marqueurs qui se chevauchent dans la plage donnée de 200 Å, supprimez les mesures redondantes qui ne sont pas des liens entre les marqueurs adjacents les plus proches. Pour ce faire, utilisez le script RemoveCross.py fourni sur le site Web UCSF Chimera, qui supprime tous les liens qui se chevauchent et qui ne sont pas le lien le plus court avec ses marqueurs adjacents.
- Pour mesurer la distance des liaisons restantes, sélectionnez toutes les pseudo-liaisons (liens créés par findclash) et utilisez le script python pblengths.py en tapant runscript pblengths.py. Consultez les valeurs mesurées à partir du journal des réponses (Outils > utilitaires > Journal des réponses). Ensuite, copiez les valeurs obtenues à partir du journal des réponses et transférez-les dans une feuille de calcul ou tout autre outil approprié pour créer un histogramme.
Résultats
En utilisant la cryotomographie électronique de stéréocils individuels non colorés, gelés-hydratés et intégrés dans de la glace vitrée, nous avons obtenu des cartes de densité du faisceau d’actine avec ses filaments d’actine disposés hexagonalement, reliés par des protéines de connexion croisée23. La dimension d’un voxel individuel était de 0,947 nm. L’inspection visuelle dans le programme de découpage IMOD d’un rendu volumique de l’ensemble du tomogramme (400 tranches/379 nm) a indiqué la présence de structures filamenteuses alignées avec l’axe longitudinal des stéréocils, comme on peut le voir dans les vues longitudinales (plan XY ; Figure 1A-C, panneaux supérieurs) ainsi que dans les vues en coupe (plan XZ ; Figure 1A-C, panneaux inférieurs). Nous avons remarqué que la vue de projection à travers le réseau de filaments 400 coupes/379 nm devenait plus claire lors de la rotation du volume reconstruit d’origine de -6° autour de l’axe X, de -13,5° autour de l’axe Y et de 5° autour de l’axe Z. À cet angle, tous les filaments sont alignés les uns sur les autres, et le contraste est donc maximal, comme on peut l’apprécier à partir des vues en coupe transversale (Figure 1B). Étant donné que les coupes transversales simples n’ont pas assez de signal pour distinguer sans ambiguïté les filaments d’actine, nous avons choisi de rendre en volume une plaque de 30 tranches/28,4 nm, qui montre clairement un motif hexagonal dans la vue en coupe transversale. Les lignes bleues de la figure 1C (panneaux supérieurs) indiquent la position du milieu des dalles de section transversale correspondantes de 30 tranches/28,4 nm dans les panneaux inférieurs.
De petits écarts par rapport à cet angle de vision optimal, d’aussi peu que ±2°, ont considérablement réduit l’ordre perçu du réseau de filaments d’actine (Figure 1A,C), ce qui indique à quel point il est facile de se perdre dans le volume 3D du tomogramme.
Pour illustrer le défi de l’utilisation d’approches de segmentation automatisées, telles que la segmentation des bassins versants, nous avons choisi un petit sous-volume (représenté en or) pour la segmentation des bassins versants, tel qu’il est mis en œuvre dans le progiciel UCSF Chimera (Tools > Volume Data > Segger > Segment). La position du sous-volume par rapport à l’ensemble de la carte des stéréocils est indiquée par le petit encart de la figure 1B.
La figure 1D-F montre le sous-volume choisi dans différentes orientations, tandis que les figures 1D, E montrent la direction de vision longitudinale et la figure 1F montre une direction de vue en coupe transversale. Les flèches sur le côté gauche de la figure 1D-F indiquent la direction des filaments d’actine.
La figure 1D-F (panneaux de droite) montre les résultats de la segmentation des bassins versants. Le sous-volume est codé par couleur en fonction de l’identité de l’objet, les couleurs étant attribuées de manière aléatoire aux différents objets. Des couleurs différentes indiquent une identité d’objet différente, d’où l’évidence de la figure 1D-F que les densités de carte pour les filaments étaient toutes deux fragmentées le long de l’axe du filament, tandis que la même couleur et donc l’identité de l’objet étaient données aux densités de carte qui reliaient les filaments voisins. En d’autres termes, l’algorithme de segmentation des bassins versants n’a pas été capable de suivre la carte de densité des filaments d’actine pendant une période prolongée, et a plutôt conduit à des densités de connexion de filaments voisins. Bien qu’il soit possible d’organiser manuellement la sélection (par exemple, en supprimant ou en fusionnant des objets), cette approche est plutôt laborieuse et prend donc beaucoup de temps.
Bien que cela ne soit pas absolument nécessaire pour que notre stratégie de construction de modèles volumétriques fonctionne, cela a permis de réorienter (tourner) la carte 3D de sorte que l’axe du réseau de filaments d’actine soit aligné avec l’axe Y et que les plans du modèle de filaments d’actine soient alignés avec le plan X-Y du tomogramme. Nous nous référons à cette orientation comme l’orientation standard pour l’affichage tomographique des stéréocils.
Nous avons donc décidé d’explorer une stratégie différente pour la segmentation des images, en exploitant le fait que les filaments d’actine présentaient une organisation globale régulière (empilement hexagonal), avec un espacement régulier et une orientation globale définie des faisceaux. Notre stratégie consistait à trouver un ajustement global des modèles d’un faisceau d’actine, sous la forme d’un ensemble de filaments, suivi d’ajustements régionaux puis locaux de la position du modèle pour s’adapter à la carte de densité expérimentale. En plaçant un modèle global en premier, nous pouvons surmonter les ambiguïtés de la carte locale et détecter les tendances régionales des écarts du modèle par rapport à son organisation d’origine, comme la flexion du filament.
Pour placer le modèle, nous avons affiché des dalles de densité (10 tranches/9,47 nm) dans l’orientation standard qui correspondaient à l’épaisseur d’une seule couche de filaments d’actine, à laquelle une couche de modèles de filaments d’actine droits régulièrement espacés a été ajustée. Il s’agit bien sûr d’une simplification excessive des filaments d’actine, chacun d’entre eux étant constitué d’un réseau linéaire de monomères d’actine à symétrie hélicoïdale. Les figures 2A-C montrent trois couches représentatives à différentes hauteurs Z, les bâtonnets de couleur rouge représentant les filaments d’actine. Les panneaux supérieurs, représentant des sections transversales de ~30 tranches/28,4 nm d’épaisseur, montrent à quelle hauteur Z une couche de modèle d’actine individuelle de 19 tiges a été placée, tandis que les panneaux inférieurs montrent une orientation longitudinale (bien que montrée en vue en perspective). La figure 2D montre le modèle simplifié complet, à la fois dans les vues en coupe transversale (panneau supérieur) et en perspective longitudinale (panneau inférieur). L’orientation transversale nous a permis de positionner les filaments avec une bonne confiance. Ici, notre décision initiale de réorienter l’ensemble du volume pour qu’il coïncide avec les grands axes des tomogrammes s’est avérée utile, car elle signifiait que l’orientation de notre modèle dans notre direction de visualisation standard était également parallèle aux axes principaux. Cependant, à proprement parler, notre approche aurait également fonctionné sans la réorientation du tomogramme, juste le placement du modèle par rapport à la densité aurait été plus difficile.
Après une inspection minutieuse des dalles individuelles de la carte de densité, nous avons remarqué qu’un modèle d’actine parfaitement droit ne correspondait pas à la carte de densité observée se déplaçant de l’extrémité proximale à l’extrémité distale (c’est-à-dire vers l’extrémité) des stéréocils (Figure 3A-C). Près de l’extrémité des stéréocils, la densité de la carte pour les filaments a été déplacée de plus de 13 nm (espacement actine-actine), ce que nous avons pu compenser en ajustant le modèle tout en passant de la partie proximale à la partie distale de la carte de densité des stéréocils, introduisant ainsi une courbure progressive petite mais distincte dans notre modèle d’actine. La figure 3D montre une seule dalle de la carte de densité du filament d’actine, avec un modèle volumétrique ajusté à la carte de densité. Une comparaison entre le modèle droit (rouge) et le modèle courbe (jaune) est illustrée à la figure 3E. Cette courbure est mieux appréciée en inclinant une plaque de la carte de densité avec le modèle placé de 80° autour de l’axe des X, ce qui permet d’avoir une vue en perspective le long de la direction des filaments d’actine (Figure 3D,E).
La déviation des deux modèles, avec la position du modèle d’actine près de la pointe étant décalée d’à peu près de la même distance que l’espacement des filaments d’actine, aurait pu causer beaucoup de confusion si nous n’avions pas procédé comme nous l’avons fait. Ce positionnement « global » d’une couche du modèle de filament d’actine, suivi d’un ajustement « régional », nous a permis de détecter cette courbure, à peine perceptible en vue longitudinale ou en coupe. Cependant, la superposition des deux modèles, comme le montre la figure 3E, révèle la différence subtile.
La répétition de cette approche pour plusieurs couches permet d’obtenir un modèle 3D complet (Figure 3F), uniquement limité par l’incertitude des données tout en haut et en bas des stéréocils, lorsqu’il est vu dans l’orientation de la section transversale (Figure 3G). Ce manque de densité est causé par l’absence de coin dans la collecte de données tomographiques (à axe unique) et l’anisotropie de résolution des données correspondante, et son effet est indiqué par l’absence d’une densité cartographique bien définie pour les membranes de stéréocils.
Une fois que nous avons eu un modèle 3D, nous avons codé en couleur chaque emplacement du modèle volumétrique en fonction de la valeur de densité de la carte à cette position. Les régions du modèle avec une faible densité de carte sous-jacente ont été colorées en rouge, tandis que les régions du modèle avec un signal de forte densité de carte ont été colorées en jaune (Figure 4A). Nous interprétons ces régions de couleur rouge, qui peuvent s’étendre jusqu’à des dizaines de nanomètres, comme des lacunes dans les structures des filaments d’actine qui, en raison de leur étendue, ne peuvent pas être attribuées aux variations de densité fréquemment rencontrées dans l’environnement à fort bruit d’une carte cryo-EM. Le bruit a tendance à affecter des voxels individuels ou de petits groupes de voxels, mais il est peu probable qu’il soit la source de volumes composés de centaines de voxels, pour lesquels la densité du filament est manquante. Au lieu de cela, de tels écarts sont susceptibles d’être une caractéristique réelle du réseau d’actine des stéréocils et peuvent constituer des sites de renouvellement de l’actine. La figure 4A présente deux valeurs de densité de carte différentes, en bleu clair et en bleu foncé. Il convient de noter explicitement que notre approche de construction de modèles volumétriques, combinée à un codage couleur automatisé de notre modèle dans les régions de faible densité, est un moyen rapide et pratique de détecter et de visualiser la distribution de tels écarts dans le modèle de filaments d’actine, ce qui aurait été très difficile autrement.
Comme le montre la figure 4B, des parties du modèle volumétrique à des endroits où la densité est comparativement faible peuvent être facilement cachées sur la base des résultats obtenus à la figure 4A. Il en résulte alors un modèle plus fragmenté qui peut représenter de manière plus réaliste le modèle d’actine dans les stéréocils. L’alternative consistant à construire de petits tronçons de filaments d’actine aurait été très laborieuse et aurait peut-être échoué complètement, en raison des problèmes discutés lors de la description de la figure 1.
De plus, le modèle volumétrique nous permet de modéliser facilement les connecteurs croisés en plaçant simplement une connexion (en rouge) entre les positions des points du modèle de filament d’actine de chaque côté de la connexion transversale (Figure 4C). Dans notre approche simplifiée, nous n’avons pas besoin de faire d’hypothèses sur l’identité exacte de chaque protéine de interconnexion, ce qui nécessiterait une résolution plus élevée et/ou des approches de marquage sophistiquées. Au lieu de cela, tout ce que nous avons besoin de déterminer est s’il existe une densité qui relie les filaments d’actine adjacents ; S’il y en a un, nous pouvons placer une courte connexion d’un filament à son homologue adjacent. La figure 4D montre un modèle de cinq filaments d’actine avec leurs connecteurs transversaux, ce qui donne une impression de la distribution des connecteurs transversaux le long de l’axe du filament d’actine.
Un autre avantage de la construction d’un modèle volumétrique du faisceau d’actine est que l’on peut rapidement déterminer l’espacement entre les filaments d’actine adjacents (Figure 4E-H). La figure 4E,F montre une vue en coupe transversale de la carte de densité sans et avec un modèle ajusté au réseau hexagonal de la densité de la carte, respectivement. La figure 4G montre le modèle avec les connexions entre les balles voisines les plus proches. UCSF Chimera permet le calcul automatique de la distance des centres des voisins les plus proches, dont le résultat peut ensuite être tracé sous forme de distribution de distance (Figure 4H). La construction de modèles pour deux ensembles de données supplémentaires est illustrée dans la figure supplémentaire 1 et la figure supplémentaire 2.
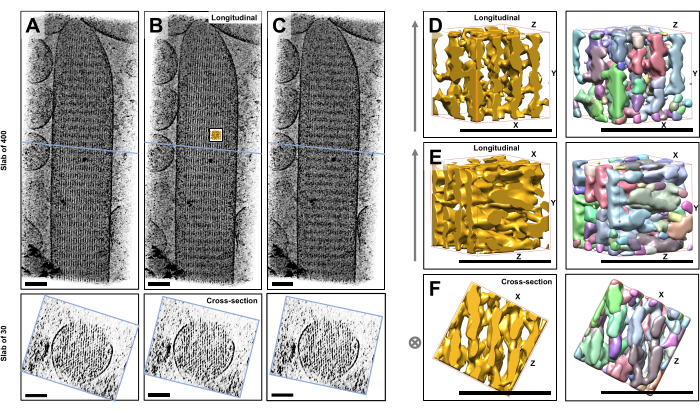
Figure 1 : Les défis rencontrés par la segmentation des bassins versants des tomogrammes de stéréocils de cellules ciliées (A-C) Projections longitudinales (400 coupes/379 nm) à travers la carte tomographique 3D dans le plan XY (panneaux supérieurs) et vues en coupe transversale (30 coupes/28,4 nm) dans le plan XZ (panneaux inférieurs). (A) Carte tomographique tournée de -2° le long de l’axe Y par rapport à son orientation optimale. (B) Carte tomographique dans l’orientation optimale, déterminée par l’ajustement des angles de rotation des axes X, Y et Z (X = -6°, Y = -13,5° et Z = 5°) et révélant un haut degré d’ordre dans la carte de densité, suggérant un réseau de filaments d’actine hautement ordonné. (C) Carte tomographique tournée de +2° le long de l’axe Y par rapport à son orientation optimale ; la rotation d’aussi peu que 2° autour de l’axe Y par rapport à la direction de visualisation optimale compromet gravement la régularité perçue de la carte de densité. Les panneaux inférieurs révèlent la régularité du réseau de filaments d’actine lorsqu’ils sont observés dans le sens de la section transversale. La ligne bleue en A-C indique la position de la dalle de section transversale. (D-F) Un cube de 50 nm x 50 nm x 50 nm vu de trois directions différentes avant (panneaux de gauche) et après (panneaux de droite) la segmentation du bassin versant. Notez que la segmentation des bassins versants ne permet pas de détecter la densité continue des filaments d’actine, tandis que les filaments d’actine adjacents et leur interconnexion partagent la même identité d’objet, ce qui suggère que la segmentation des bassins versants n’est pas une approche appropriée pour la segmentation par tomogramme. Dans les panneaux D-F, la carte de densité dans Chimera est représentée par le style de carte « Surface ». (A-C) Barres d’échelle = 100 nm. (D-F) Barres d’échelle = 50 nm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
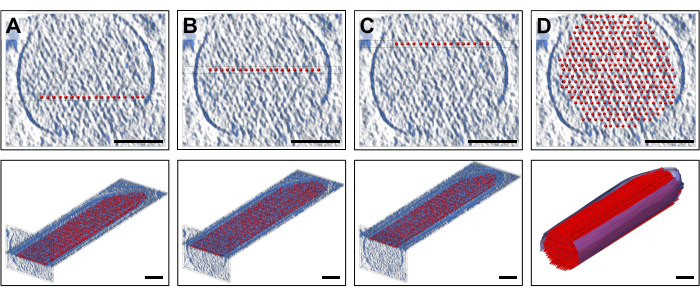
Figure 2 : Construction d’un modèle volumétrique de filament d’actine en forme de boule et de bâton. (A-C) En haut : vues en coupe transversale de 30 coupes/28,4 nm de la carte de densité avec un modèle simplifié de filament d’actine monocouche placé à une hauteur Z différente. En bas : une seule dalle de 10 coupes/9,47 nm de la carte de densité avec un modèle simplifié de filament d’actine monocouche. (D) Modèle complet de filament d’actine droit dans une vue en coupe transversale de 30 coupes/28,4 nm (en haut) et en perspective (en bas) ; Barres d’échelle = 100 nm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
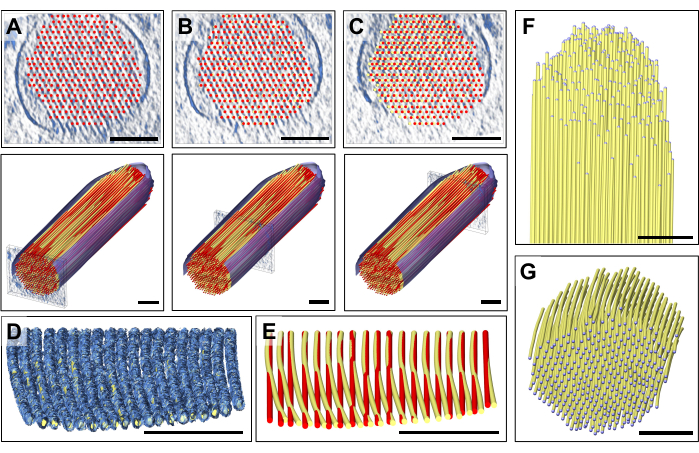
Figure 3 : Ajustement du modèle pour corriger la courbure détectée de la densité des filaments d’actine. (A-C) Une inspection visuelle minutieuse du modèle, soit en coupe transversale (panneaux supérieurs), soit en vue longitudinale (panneaux inférieurs), a révélé un bon ajustement du modèle avec la carte de densité à l’extrémité proximale des stéréocils. Cependant, à mesure que l’on se rapproche de l’extrémité distale des stéréocils, l’ajustement s’aggrave de plus en plus pour tous les modèles de filaments d’actine. Cela peut être corrigé en déplaçant les boules du modèle à bille et bâton dans la position correcte de la carte de densité, ce qui donne un modèle de filament d’actine légèrement incurvé. Le modèle droit est représenté en rouge et le modèle courbé corrigé est représenté en jaune. (D) Une seule plaque de la carte de densité avec le modèle incurvé ajusté à celle-ci, révélant la courbure de la densité d’actine vers l’extrémité des stéréocils. Le modèle d’actine a été tourné de 80° autour de l’axe X pour mieux montrer cette courbure subtile mais significative des filaments d’actine. (E) Comparaison entre le modèle d’actine droit et non corrigé illustré en rouge et le modèle d’actine incurvé et corrigé illustré en jaune. Pour des raisons de clarté, une seule couche du modèle de filament d’actine est représentée. (F-G) Modèle de faisceau d’actine avec des filaments d’actine incurvés et corrigés représentés en orientation longitudinale (F) et transversale (G). La membrane segmentée est représentée en bleu. Dans le panneau D, la carte de densité dans Chimera est représentée par le style de carte « Mesh ». Barres d’échelle = 100 nm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
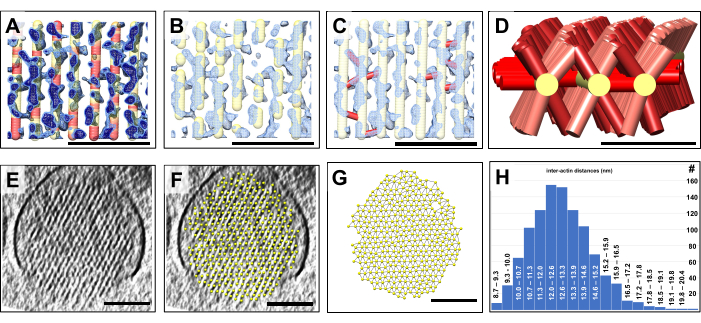
Figure 4 : Détection des lacunes dans le faisceau d’actine à l’aide de la construction d’un modèle volumétrique. Quantification des propriétés volumétriques à l’aide de modèles à bille et bâton. (A-C) Une petite région d’une dalle longitudinale de ~10 tranches/9,47 nm d’épaisseur de la carte de densité des stéréocils est représentée en bleu, ainsi que le modèle de filament d’actine incurvé et corrigé. (A) La carte de densité est représentée à un seuil de densité inférieur (bleu clair) et à un seuil de densité plus élevé (bleu foncé). Il existe des régions du modèle de filament d’actine pour lesquelles il n’y a pas de densité correspondante. À ces endroits, le modèle a été codé en rouge pour représenter un manque de densité. Nous interprétons ces emplacements comme des lacunes dans les filaments d’actine. (B) Modèle des filaments d’actine qui semble fragmenté, reflétant l’absence de densité de filaments d’actine à ces endroits troués. (C) Modèle des filaments d’actine avec des liens d’interconnexion (en rouge) ajoutés aux endroits où une forte densité a été trouvée pour relier les filaments d’actine adjacents. (D) Trois filaments d’actine modèles sélectionnés sont représentés avec des connecteurs croisés vers des filaments d’actine adjacents (qui ne sont pas représentés pour plus de clarté). Notez que de nombreuses positions possibles des filaments d’inter-actine, mais pas toutes, sont occupées par des protéines de connexion ; (E) Une section transversale de 30 coupes/28,4 nm de la densité des stéréocils. (F) Modèle de filaments d’actine montés sur la plaque de densité de stéréocils de 30 tranches/28,4 nm en coupe transversale. (G) Modèle de filaments d’actine sans la densité de carte sous-jacente. Détection automatique des distances des filaments d’actine du voisin le plus proche, comme indiqué par les connexions minces entre les boules du modèle de filament d’actine. (H) Histogramme des distances des modèles de filaments d’actine. Dans les panneaux A-C, la carte de densité dans Chimera est affichée sous la forme du style de carte « Mesh ». (A-C) Barres d’échelle = 50 nm. (D) Barre d’échelle = 25 nm. (E-G) Barres d’échelle = 100 nm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Construction d’un modèle pour le premier de deux ensembles de données supplémentaires sur les stéréocils. (A-C) Une petite région d’une dalle longitudinale de ~10 coupes/9,47 nm d’épaisseur de la carte de densité des stéréocils est représentée en bleu à l’aide de l’affichage en mode maillé. Le modèle initialement placé est représenté en rouge et le modèle corrigé en jaune. (A) Densité de la carte uniquement. (B) Modèle initial placé dans la carte de densité. (C) Modèle corrigé placé dans la carte de densité. (D-E) Région de stéréocils plus grande sans (D) et avec (E) le modèle corrigé ajusté à une plaque longitudinale de ~10 coupes/9,47 nm d’épaisseur de la carte de densité des stéréocils. (F-G) La région entière du tomogramme des stéréocils est représentée. (F) Carte uniquement. (G) Carte avec le modèle corrigé. (H) Superposition des modèles initiaux et corrigés. Barres d’échelle = 100 nm Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2 : Construction d’un modèle pour le deuxième des deux ensembles de données supplémentaires sur les stéréocils. (A-C) Une petite région d’une dalle longitudinale de ~10 coupes/9,47 nm d’épaisseur de la carte de densité des stéréocils est représentée en bleu à l’aide de l’affichage en mode maillé. Le modèle initialement placé est représenté en rouge et le modèle corrigé est représenté en jaune ; (A) Densité de la carte uniquement. (B) Modèle initial placé dans la carte de densité. (C) Modèle corrigé placé dans la carte de densité. (D-E) Région de stéréocils plus grande sans (D) et avec (E) le modèle corrigé ajusté à une plaque longitudinale de ~10 coupes/9,47 nm d’épaisseur de la carte de densité des stéréocils. (F-G) La région entière du tomogramme des stéréocils est représentée. (F) Carte uniquement. (G) Carte avec le modèle corrigé. (H) Superposition du modèle initial et du modèle corrigé. Barres d’échelle = 100 nm. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 1 : Scripts python UCSF Chimera pour la modélisation des stéréocils. Veuillez cliquer ici pour télécharger ce fichier.
Fichier de codage supplémentaire 1 : pblengths.py. Veuillez cliquer ici pour télécharger ce fichier.
Fichier de codage supplémentaire 2 : RemoveCross.py. Veuillez cliquer ici pour télécharger ce fichier.
Fichier de codage supplémentaire 3 : ActinFilamentPlane.py. Veuillez cliquer ici pour télécharger ce fichier.
Fichier de codage supplémentaire 4 : dividelinks.py. Veuillez cliquer ici pour télécharger ce fichier.
Fichier de codage supplémentaire 5 : FixingMarkerID.py. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Nous avons montré que les approches automatisées de segmentation, telles que la segmentation des bassins versants, peuvent échouer dans l’environnement à fort bruit et à haute complexité des stéréocils, des cryo-atomographies d’électrons de cellules ciliées. Distinguer quelle partie de ce réseau filamenteux représente des filaments d’actine et ce qui constitue des réticulations au niveau de l’environnement local semble au mieux difficile lorsqu’on se contente d’inspecter de petits sous-volumes tomographiques. L’approche de construction de modèles utilisée dans cette étude bénéficie d’une connaissance préalable de l’ordre à grande échelle du faisceau d’actine, ce qui aide à développer une attente sur l’orientation des filaments d’actine et les densités de réticulants. Ce qui est peut-être encore plus significatif, c’est qu’un cerveau humain peut facilement trouver des modèles en considérant le contexte plus large au-delà de la distribution de densité locale, alors qu’un algorithme informatique ne fonctionne que pour une région relativement petite qui est considérée par l’algorithme ; Par conséquent, les tendances à plus grande échelle ne peuvent pas être facilement prises en considération. En ajustant un modèle globalement à une couche de densité, nous avons évité la confusion qui peut survenir lorsque l’on tente de créer un modèle pour de petites portions de filaments d’actine uniques à la fois. Bien sûr, un tel ajustement global suppose un ordre qui s’étend sur de grandes distances. Cependant, comme nous avons eu une courbure progressive inattendue, petite mais significative, des filaments d’actine, l’ajustement global n’était qu’une approximation initiale et a nécessité des ajustements locaux du modèle pour s’adapter à la carte de densité. Comme le modèle initial était un bon point de départ, les ajustements ont pu être effectués en toute confiance. L’un des grands avantages de notre approche était que nous pouvions choisir de n’afficher qu’une zone de densité définie, ce qui a permis de réduire la complexité du paysage. De plus, l’observation de la dalle de densité de la carte le long de l’axe du modèle de filament a permis d’identifier la courbure inattendue, que nous aurions probablement manquée en affichant simplement des sous-volumes plus petits. Le placement du modèle initial a également facilité un zoom avant et arrière rapide, afin d’alterner entre une vue d’ensemble de la couche respective de filaments d’actine et des vues détaillées pour effectuer des ajustements au modèle.
Les étapes critiques du protocole comprenaient la rotation de la carte après inspection visuelle, la création et le placement du modèle dans la carte de densité, ainsi que la division du modèle de filament en segments plus petits. La position des atomes des segments pourrait ensuite être ajustée spatialement pour s’adapter à la carte de densité et/ou codée par couleur pour détecter les écarts.
Cette approche de construction du modèle d’actine peut également être modifiée en plaçant un ensemble d'« atomes » (c’est-à-dire les boules du modèle boule et bâton) dans des densités de filaments en utilisant une vue en coupe transversale d’une tranche de 10-30/9,47-28,4 nm de densité moyenne, qui peut ensuite être connectée par des liaisons (c’est-à-dire les bâtons du modèle boule et bâton). Nous avons utilisé cette approche, qui est une modification du protocole décrit ici en détail, pour la construction du modèle volumétrique dans la région conique des stéréocils des cellules ciliées23. De plus, comme nous l’avons décrit ici, notre approche de construction de modèles volumétriques est également bien adaptée à la segmentation et à la construction de modèles de membranes.
Bien que la construction de modèles volumétriques puisse être appliquée à n’importe quelle carte de densité qui montre des caractéristiques filamenteuses, la technique que nous avons décrite ici est plus efficace lorsque nous disposons d’un réseau de filaments régulièrement espacés, pour lesquels un ajustement global d’un modèle volumétrique peut être obtenu. Il dépend également des caractéristiques filamenteuses pour modifier leur directionnalité de manière progressive. S’il y a des plis soudains et des virages brusques dans les structures filamenteuses, notre approche peut ne pas être particulièrement utile pour la segmentation.
Entre-temps, nos collaborateurs ont développé une approche automatisée pour le traçage automatisé des filaments qui suit un concept similaire utilisé ici pour la segmentation manuelle30,31. À l’avenir, la meilleure approche pourrait bien être un hybride d’identification manuelle et de placement d’un modèle initial clairsemé (même seulement quelques boules) dans la densité comme point de départ, puis de laisser un algorithme de recherche et d’ajustement terminer le traçage des filaments.
Les modèles volumétriques simplifiés réduisent la complexité d’un système et permettent de mieux apprécier certains motifs, tels que la flexion du filament d’actine près de la pointe. De plus, le modèle volumétrique peut être utilisé comme « ancre » pour afficher une zone de densité autour du modèle d’ancrage choisi, ce qui permet de détecter et de visualiser les densités de réticulants entre les filaments d’actine adjacents. La possibilité de sélectionner des filaments individuels et de définir des rayons appropriés comme zone dans laquelle la densité est à nouveau affichée permet de réduire la complexité écrasante du paysage à un niveau gérable.
L’un des avantages de cette approche de construction de modèles volumétriques d’ajustement global, suivi d’ajustements locaux, est que nous avons pu identifier les régions où les filaments d’actine semblaient être interrompus, et des lacunes significatives dans les filaments d’actine ont été indiquées par l’absence de densité de carte. Comme nous avions placé un modèle volumétrique à bille et bâton, nous avons pu utiliser une routine dans le progiciel UCSF Chimera qui code par couleur la position de chaque balle de modèle en fonction de la valeur de densité de la carte à cet endroit. Cette approche a permis une détection et une visualisation rapides des lacunes des filaments d’actine dans le faisceau d’actine, ce qui est une caractéristique biologiquement significative que nous avons trouvée dans notre cryo-tomogramme électronique, et qui aurait été très difficile à détecter et à visualiser avec les approches de segmentation traditionnelles. Un autre avantage de notre modèle volumétrique est qu’il est facile d’obtenir des propriétés volumétriques, y compris les longueurs et les distances, ce qui permet d’obtenir des nombres réels et donc d’effectuer une analyse statistique.
En résumé, le placement manuel interactif des points du modèle, éventuellement complété par des capacités ultérieures d’ajustement local automatisé et de traçage des filaments, est une approche plutôt prometteuse pour la visualisation et l’analyse quantitative des volumes subcellulaires tomographiques électroniques. En effet, il utilise la puissance du cerveau humain pour la reconnaissance des formes et la puissance de l’informatique pour l’optimisation des modèles.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents ou d’autres conflits d’intérêts.
Remerciements
Nous tenons à remercier le Dr Peter Barr-Gillespie et son équipe pour leur rôle dans la préparation des échantillons, ainsi que les anciens membres du laboratoire Auer et du laboratoire de la Dre Dorit Hanein pour leur rôle dans la collecte de données tomographiques. Nous tenons également à remercier Tom Goddard de UCSF Resource for Biocomputing, Visualization, and Informatics (RBVI) pour avoir fourni divers scripts UCSF Chimera.
matériels
| Name | Company | Catalog Number | Comments |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/download.html | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=win64/chimera-1.16-win64.exe | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=mac64/chimera-1.16-mac64.dmg | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=linux_x86_64/chimera-1.16-linux_x86_64.bin | |
| Excel | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Falcon II | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/accessories-em/falcon-detector.html | |
| IMOD | University of Colorado | Version 4.11.1 https://bio3d.colorado.edu/imod/download.html | |
| PC Desktop | Intel | Windows 10, ver. 22H2 | |
| PC Laptop | Gigabyte | Windows 10, ver. 22H2 | |
| Powerpoint | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Titan Krios Electron Microscope | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/transmission-electron-microscopes/krios-g4-cryo-tem.html | |
| Word | Microsoft | Version 2211 https://www.office.com/?auth=1 |
Références
- Downing, K. H., Sui, H., Auer, M. Electron tomography: A 3D view of the subcellular world. Analytical Chemistry. 79 (21), 7949-7957 (2007).
- Koning, R. I., Koster, A. J. Cellular nanoimaging by cryo electron tomography. Methods in Molecular Biology. 950, 227-251 (2013).
- Asano, S., Engel, B. D., Baumeister, W. In situ cryo-electron tomography: A post-reductionist approach to structural biology. Journal of Molecular Biology. 428 (2), 332-343 (2016).
- Serwas, D., Davies, K. M. Getting started with in situ cryo-electron tomography. Methods in Molecular Biology. 2215, 3-23 (2021).
- McDonald, K. L., Auer, M. High-pressure freezing, cellular tomography, and structural cell biology. Biotechniques. 41 (2), 137-143 (2006).
- Narasimha, R., et al. Evaluation of denoising algorithms for biological electron tomography. Journal of Structural Biology. 164 (1), 7-17 (2008).
- Frangakis, A. S. It's noisy out there! A review of denoising techniques in cryo-electron tomography. Journal of Structural Biology. 213 (4), 107804 (2021).
- Frangakis, A. S., Hegerl, R. Noise reduction in electron tomographic reconstructions using nonlinear anisotropic diffusion. Journal of Structural Biology. 135 (3), 239-250 (2001).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. Journal of Structural Biology. 144 (1-2), 114-122 (2003).
- vander Heide, P., Xu, X. -. P., Marsh, B. J., Hanein, D., Volkmann, N. Efficient automatic noise reduction of electron tomographic reconstructions based on iterative median filtering. Journal of Structural Biology. 158 (2), 196-204 (2007).
- Volkmann, N. Methods for segmentation and interpretation of electron tomographic reconstructions. Methods in Enzymology. 483, 31-46 (2010).
- Böhm, J., et al. Toward detecting and identifying macromolecules in a cellular context: template matching applied to electron tomograms. Proceedings of the National Academy of Sciences. 97 (26), 14245-14250 (2000).
- Frangakis, A. S., et al. Identification of macromolecular complexes in cryoelectron tomograms of phantom cells. Proceedings of the National Academy of Sciences. 99 (22), 14153-14158 (2002).
- Lebbink, M. N., et al. Template matching as a tool for annotation of tomograms of stained biological structures. Journal of Structural Biology. 158 (3), 327-335 (2007).
- Rigort, A., et al. Automated segmentation of electron tomograms for a quantitative description of actin filament networks. Journal of Structural Biology. 177 (1), 135-144 (2012).
- Volkmann, N. A novel three-dimensional variant of the watershed transform for segmentation of electron density maps. Journal of Structural Biology. 138 (1-2), 123-129 (2002).
- Bajaj, C., Yu, Z., Auer, M. Volumetric feature extraction and visualization of tomographic molecular imaging. Journal of Structural Biology. 144 (1-2), 132-143 (2003).
- Yu, Z., Frangakis, A. S. Classification of electron sub-tomograms with neural networks and its application to template-matching. Journal of Structural Biology. 174 (3), 494-504 (2011).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Tilney, L. G., Derosier, D. J., Mulroy, M. J. The organization of actin filaments in the stereocilia of cochlear hair cells. The Journal of Cell Biology. 86 (1), 244-259 (1980).
- Tilney, L. G., Tilney, M. S., DeRosier, D. J. Actin filaments, stereocilia, and hair cells: how cells count and measure. Annual Review of Cell Biology. 8, 257-274 (1992).
- Metlagel, Z., et al. Electron cryo-tomography of vestibular hair-cell stereocilia. Journal of Structural Biology. 206 (2), 149-155 (2019).
- Song, J., et al. A cryo-tomography-based volumetric model of the actin core of mouse vestibular hair cell stereocilia lacking plastin 1. Journal of Structural Biology. 210 (1), 107461 (2020).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Agulleiro, J. I., Fernandez, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27 (4), 582-583 (2011).
- Fernandez, J. J. Computational methods for electron tomography. Micron. 43 (10), 1010-1030 (2012).
- Chen, H., Clyborne, W. K., Sedat, J. W., Agard, D. A. Priism: an integrated system for display and analysis of 3-D microscope images. Biomedical Image Processing and Three-Dimensional Microscopy. 1660, 784-790 (1992).
- Pettersen, E. F., et al. UCSF Chimera-a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25 (13), 1605-1612 (2004).
- Goddard, T. D., Huang, C. C., Ferrin, T. E. Visualizing density maps with UCSF Chimera. Journal of Structural Biology. 157 (1), 281-287 (2007).
- Sazzed, S., et al. Tracing actin filament bundles in three-dimensional electron tomography density maps of hair cell stereocilia. Molecules. 23 (4), 882 (2018).
- Sazzed, S., Scheible, P., He, P., Wriggers, J. Spaghetti tracer: A framework for tracing semiregular filamentous densities in 3D tomograms. Biomolecules. 12 (8), 1022 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon