Method Article
Vereinfachte volumetrische Modelle als effektive Strategie zur Segmentierung von Aktinnetzwerken in Kryo-Elektronen-Tomogrammen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll vor, um vereinfachte volumetrische Modelle in verrauschte, komplexe, tomographische 3D-Volumina zu platzieren. Dies ermöglicht die schnelle Segmentierung von Aktinfilamentdichten, die Detektion von systematischer Filamentbiegung und von Lücken in Haarbündelfilamenten sowie die komfortable Quantifizierung von volumetrischen Modelleigenschaften, wie z.B. Abständen.
Zusammenfassung
Effiziente Methoden zur Extraktion von Merkmalen von Interesse sind nach wie vor eine der größten Herausforderungen für die Interpretation von Kryo-Elektronen-Tomogrammen. Es wurden verschiedene automatisierte Ansätze vorgeschlagen, von denen viele gut für kontrastreiche Datensätze geeignet sind, bei denen die interessierenden Merkmale leicht zu erkennen sind und klar voneinander getrennt sind. Unsere Kryo-Elektronen-Tomographie-Datensätze für Stereozilien des Innenohrs zeichnen sich durch eine dichte Anordnung von hexagonal gepackten Aktinfilamenten aus, die häufig miteinander verbunden sind. Diese Merkmale machen die automatisierte Segmentierung zu einer großen Herausforderung, was durch die rauschreiche Umgebung von Kryo-Elektronen-Tomogrammen und die hohe Komplexität der dicht gepackten Merkmale noch verschärft wird. Unter Verwendung von Vorkenntnissen über die Organisation von Aktinbündeln haben wir Schichten eines stark vereinfachten Ball-and-Stick-Aktinmodells platziert, um zunächst eine globale Anpassung an die Dichtekarte zu erhalten, gefolgt von regionalen und lokalen Anpassungen des Modells. Wir zeigen, dass die volumetrische Modellbildung es uns nicht nur ermöglicht, mit der hohen Komplexität umzugehen, sondern auch präzise Messungen und Statistiken über das Aktinbündel liefert. Volumetrische Modelle dienen auch als Ankerpunkte für die lokale Segmentierung, wie z. B. im Fall der Aktin-Aktin-Kreuzverbinder. Die Erstellung volumetrischer Modelle, insbesondere wenn sie durch computergestützte automatisierte Anpassungsansätze ergänzt wird, kann eine leistungsstarke Alternative sein, wenn herkömmliche automatisierte Segmentierungsansätze nicht erfolgreich sind.
Einleitung
Die Kryo-Elektronentomographie ermöglicht es, ganze Organellen oder Teile von Zellen und Geweben mit Nanometerauflösungen in ihrem nahezu nativen Zustand 1,2,3 entweder durch Tauchgefrieren4 oder Hochdruckgefrieren mit ultraschneller Vitrifikation5 sichtbar zu machen. Da die kryokonservierte, ungefärbte, gefroren hydratisierte Probe nur eine begrenzte Elektronendosis toleriert, sind die tomographischen 3D-Daten sehr verrauscht. Dieses Rauschen kann häufig durch eine Vielzahl von Rauschfilteralgorithmen 6,7 erheblich reduziert werden, einschließlich der nichtlinearen anisotropen Diffusion8, der bilateralen Filterung9 und der rekursiven Medianfilterung10.
Darüber hinaus führen die Neigungsbeschränkungen des Mikroskoptisches, die zu einem fehlenden Informationskeil führen, und die Tatsache, dass die Probendicke bei hohen Neigungswinkeln zunimmt, zu 3D-Rekonstruktionen mit anisotroper Auflösung. Das bedeutet, dass die Dichte in der dritten Dimension aufgrund der geringeren Auflösung in Z-Richtung verschmiert wird. Infolgedessen erscheint die Form von Makromolekülen verzerrt (d. h. in der dritten Dimension weniger gut definiert und länglich).
Eine der größten Herausforderungen bei der Interpretation tomographischer Daten ist die automatisierte Extraktion der relevanten Merkmale, auch Segmentierung11 genannt. Mit ausreichenden einzigartigen Formmerkmalen und geringem Rauschen können makromolekulare Maschinen in komplexen 3D-Volumina durch den Vorlagenabgleich 12,13,14 identifiziert werden; Der Erfolg des Vorlagenabgleichs hängt jedoch von der Tomogrammauflösung, einem geeigneten Suchmodell sowie den Größen- und Formmerkmalen der Feature-Volumina ab. Wenn die interessierenden Merkmale ausreichend voneinander entfernt sind und sich wiederholende Motive (z. B. große makromolekulare Maschinen) leicht identifiziert werden können, können Tomogramm-Subvolumina kombiniert werden, um das Signal-Rausch-Verhältnis zu erhöhen und individuelle Formverzerrungen der einzelnen Partikel auszugleichen. Es wurde über eine automatisierte Segmentierung eines Aktinfilamentnetzwerks in Elektronentomogrammen des dünnen Randes von gefrorenen hydratisierten Dictyostelium discoideum-Zellen durch Template-Matching berichtet15.
Wenn jedoch die interessierenden Merkmale eng beieinander liegen, kann die Anisotropie der Datenauflösung zu einer Verschmierung der Kartendichten in Z-Richtung (entlang der Richtung des Elektronenstrahls) führen, was zu einer scheinbaren Verschmelzung der Dichtehülle von eng beieinander liegenden makromolekularen Maschinen oder supramolekularen Komplexen führt. In solchen Fällen sind automatisierte Ansätze für die Segmentierung, wie z. B. Wasserscheide16, Grenzsegmentierung17 oder eine Vielzahl von auf maschinellem Lernen basierenden Klassifizierungsansätzen18, 19, möglicherweise nicht in der Lage, die interessierenden Merkmale zu erkennen oder eine korrekte Grenze um ein Objekt von Interesse festzulegen. Oft hat man entweder einige sehr große Stücke oder ein stark übersegmentiertes Volumen, bei dem viel Aufwand erforderlich ist, um viele kleine Stücke zusammenzuführen, bis das interessante Merkmal als vollständig wahrgenommen wird. Eine solche manuelle Kuratierung von Segmentierungsergebnissen kann sehr arbeitsintensiv sein und sogar ganz scheitern, wenn die interessierende Struktur ein Array von eng beieinander liegenden Filamenten ist, die über kurze Linker miteinander verbunden sind. In diesem gigantischen Geflecht aus fadenförmigen Strukturen kann es schwierig sein, sich zu orientieren. Dies liegt daran, dass die Dichten aufgrund der Auflösungsanisotropie ineinander überzugehen scheinen, was sowohl für automatisierte als auch für interaktive manuelle Segmentierungsansätze eine große Herausforderung darstellt. Infolgedessen kann man leicht zwischen den Filamenten "springen", wenn man nur kleine Bereiche visuell inspiziert.
Glücklicherweise haben wir im Fall des Aktinbündels in den Stereozilien der Innenohr-Haarzellen Kenntnisse über die Gesamtorganisation des Aktinbündels und die Richtungsabhängigkeit der Aktinfilamente20,21. Das Aktinbündel besteht aus Hunderten von hexagonalen, dicht gepackten Aktinfilamenten mit einem Durchmesser von 6-8 nm, die etwa 12-13 nm voneinander entfernt sind22.
Dies ermöglichte es uns, einen ganz anderen Ansatz für die Segmentierung zu wählen, der auf vereinfachten Ball-and-Stick-Modellen basiert, um Aktinfilamente darzustellen. Die Strategie bestand darin, gleichzeitig ein idealisiertes regelmäßiges Array von Filamentmodellen in Platten der Kryo-Elektronentomographie-Dichtekarten zu platzieren, um Schicht für Schicht ein 3D-Modell des Aktinbündels aufzubauen. Wir stellten sicher, dass das Modell eine allgemeine Gesamtanpassung an die Dichtekarte aufwies, bevor wir lokale Anpassungen an einzelnen Filamentmodellen oder Gruppen von Filamentmodellen vornahmen, um der Dichtekarte nahe zu kommen. Durch die automatische Farbcodierung des Kartendichtewerts an der Position des Filamentmodells konnten wir offensichtliche Lücken im Aktinbündel leicht erkennen. Volumetrische Modelle ermöglichen eine quantitative Analyse volumetrischer Eigenschaften, wie z.B. Abstände zwischen Aktinfilamenten, und führen auch zu einer vereinfachten Darstellung der gesamten 3D-filamentösen Netzwerkorganisation.
Darüber hinaus können Modelle auch als Verankerungsstrukturen für die Segmentierung von zusätzlichen Merkmalen, wie z.B. Aktin-Aktin-Linkern, dienen, da (Teile von) einzelnen Filamentmodellen ausgewählt werden können, um die geeignete Radiuskarten-Dichtezonen für die Inspektion und weitere Segmentierung generiert werden können.
Wir glauben, dass unser volumetrischer modellbasierter Segmentierungsansatz besonders nützlich für große filamentöse Strukturnetzwerke ist, die Lücken und Querverbindungen zwischen den Filamenten enthalten können. Segmentierungsalgorithmen arbeiten in der Regel lokal, während das menschliche Gehirn größere Bereiche berücksichtigt und damit Computern überlegen ist, wenn es darum geht, Filamentstrukturen zu erkennen, selbst in einer komplexen, rauschreichen Umgebung.
Protokoll
Das Protokoll folgt den Richtlinien der Ethikkommission der Southeast University.
1. Kryo-Elektronentomographie-Datenquelle für den volumetrischen Modellbau
HINWEIS: Die Stereozilien-Kryo-Elektronen-Tomographie-Rekonstruktionen, die für einen volumetrischen Modellbau verwendet wurden, wurden zuvor veröffentlicht22,23 und wurden wie von Metlagel et al.22 beschrieben erhalten.
Die UCSF Chimera-Python-Skripte für die Stereozilienmodellierung werden in der Ergänzenden Datei 1, der Ergänzenden Codierungsdatei 1, der Ergänzenden Codierungsdatei 2, der Ergänzenden Codierungsdatei 3, der Ergänzenden Codierungsdatei 4 und der Ergänzenden Codierungsdatei 5 bereitgestellt.
- Kurz gesagt, tupfen Sie Stereozilien von der apikalen Oberfläche der sensorischen Epithelien des Utrikels der Maus auf den lacey Carbon-Stützfilm eines Elektronenmikroskop (EM)-Gitters. Führen Sie dann eine ultraschnelle Tauch-Gefrier-Vitrifikation und eine einachsige Kryo-Elektronentomographie-Datenerfassung auf einer kryogenen Transmissionselektronenmikroskopie (Kryo-TEM) durch, die bei 300 kV mit einer nominalen Defokussierung von 3,5 bis 4,5 μm betrieben wird, unter Verwendung einer CMOS-Kamera im Integrationsmodus mit einer Pixelgröße von 0,47 bis 0,59 nm (siehe Materialtabelle).
- Eine typische Dosis für die einachsige Datenerfassung beträgt typischerweise 80-100 Elektronen/Å 2. Rekonstruieren Sie die Tomogramm-3D-Volumina mit dem Softwarepaket IMOD24, entweder durch gewichtete Rückprojektion oder durch die SIRT-Methode25,26.
- Entrauschen Sie die Tomogramme mit rekursiver medianer oder bilateraler Filterung in Priism27 oder der Option für nichtlineare anisotrope Diffusion in IMOD.
HINWEIS: Das wichtigste Softwarepaket, das für die interaktive Visualisierung, die Erstellung volumetrischer Modelle und die quantitative Analyse verwendet wurde, war die UCSF Chimera-Software28,29. Alle verwendeten Softwarepakete sind in der Materialtabelle aufgeführt.
2. Datenaufbereitung der Kryo-Elektronentomographie für den volumetrischen Modellbau
- Tomogramm-Rotation
HINWEIS: Der Zweck dieses Schritts besteht darin, die "optimalen Winkel" zu finden, in denen der Aktinkern in zwei der drei Achsen (x und z) ausgerichtet ist, so dass das Platzieren der Modelle in 3D mit nur einer Achse erfolgen kann. Die folgenden Schritte werden innerhalb des IMOD-Softwarepakets ausgeführt.- Öffnen Sie 3dmod (IMOD) und öffnen Sie die 3D-Bildstapeldatei im .mrc-Dateiformat. Stellen Sie sicher, dass sich der 3D-Stapel im 16-Bit-Graustufenmodus befindet und die korrekten X-, Y- und Z-Abmessungen im Kopfbereich der Bilddatei anzeigt. Nehmen Sie ggf. Korrekturen mit dem Befehl alterheader vor: alterheader -d (x_pixelspacing),(y_pixelspacing),(z_pixelspacing) inputfilename.mrc in der Eingabeaufforderung. Wenn der 3D-Stapel im TIFF-Dateiformat vorliegt, verwenden Sie den Befehl tif2mrc inputfilename.tif outputfilename.mrc in der Eingabeaufforderung, um eine .mrc-Datei zu erstellen.
- Überprüfen Sie das Tomogramm visuell und öffnen Sie den "Slicer" (durch Drücken des Backslash "\"), um die besten Drehwinkel in den X-, Y- und Z-Dimensionen für die Ausrichtung der Aktinfilamentebene mit der Z-Ebene zu finden. Manipulieren Sie die X-Rotation, Y-Rotation, Z-Rotationsleiste, um den optimalen Winkel zu ermitteln, in dem die Aktinfilamente auf der X- und Z-Ebene ausgerichtet sind. Beachten Sie, dass das Ändern der durchschnittlichen Dicke für die Anzeige des Tomogramms (Dick : Bild) dazu beitragen kann, eine ideale Mittelung der Dichte in der Z-Ebene und damit einen erhöhten Kontrast zu finden. Vergrößern und verkleinern Sie das Tomogramm, indem Sie die Tasten + bzw. - auf der Tastatur verwenden.
- Nachdem Sie die idealen Winkel für die Drehung identifiziert haben, drehen Sie das Tomogramm, indem Sie den IMOD-Befehl rotatevol -a (Z-Drehwinkel),(Y-Drehwinkel),(X-Drehwinkel) -s (x-Breite),(y-Höhe),(z-Tiefe) inputDateiname.mrc Ausgabedateiname.mrc in die Eingabeaufforderung eingeben. Stellen Sie sicher, dass die Karte ausreichend Platz für die Drehung hat, um zu vermeiden, dass Teile der Karte bei der Drehung abgeschnitten werden.
HINWEIS: Weitere Informationen zur Verwendung des Befehls rotatevol finden Sie unter der Option im Hilfemenü oder unter der folgenden URL (https://bio3d.colorado.edu/imod/doc/man/rotatevol.html). Dieser Befehl (rotatevol) ändert nicht die aktuell im IMOD-Fenster angezeigte Map, sondern erstellt eine neue Map-Datei mit einer Dichte-Map, die gedreht wird. - Identifizieren Sie im Slicer-Fenster die X-, Y- und Z-Koordinaten von zwei gegenüberliegenden Ecken einer Map, um mit dem Befehl trimvol den Zuschneidebereich zu identifizieren, der das Stereocilium enthält.
- Verwenden Sie den IMOD-Befehl Trimvol , um die Karte an den zuvor erhaltenen Koordinaten zuzuschneiden, indem Sie trimvol -x (x-coordiante 1),( x-coordiante 2) -y (y-coordiante 1),( y-coordiante 2) -z (z-coordiante 1),( z-coordiante 2) inputfilename.mrc outputfilename.mrc in die Eingabeaufforderung eingeben. Jetzt ist die Karte viel kleiner und somit einfacher und schneller zu bearbeiten.
HINWEIS: Weitere Informationen zur Verwendung des Befehls trimvol finden Sie unter der Option im Hilfemenü oder unter der folgenden URL (https://bio3d.colorado.edu/imod/doc/man/trimvol.html). Mit diesen Schritten wird eine MRC-Datei erstellt, die gedreht und gekürzt wurde.
- Tomogramm-Filterung
HINWEIS: In diesem Schritt wird ein nichtlinearer anisotroper Diffusionsfilter (NAD) verwendet, der von IMOD zur Rauschunterdrückung bereitgestellt wird. Wir haben als Anfangswert die auf der IMOD-Hilfeseite empfohlenen Werte für die nichtlineare anisotrope Diffusionsfilterung verwendet, wie in der folgenden URL (https://bio3d.colorado.edu/imod/doc/NADexample.html) aufgeführt, und wurden als Anfangswerte verwendet.- Geben Sie etomo in die Eingabeaufforderung ein, um die grafische Benutzeroberfläche von etomo von IMOD zu starten.
- Wählen Sie die Option Nichtlineare anisotrope Diffusion aus dem etomo-Menü und wählen Sie die Map-Datei aus (Wählen Sie ein Volumen).
- Um einen K-Wert und die Anzahl der Iterationen zu finden, die die besten Filterergebnisse liefern, wenden Sie den NAD-Filter auf ein kleineres Testvolumen an, um das angegebene NAD-Filtertestvolumen zu extrahieren. Klicken Sie auf das Gummiband-Werkzeug oben in der 3dmod-Oberfläche, klicken Sie mit der linken Maustaste auf Ziehen, um das Testvolumen auszuwählen, und klicken Sie auf die Schaltflächen Hi und Lo , um das Z-Slice als Begrenzung des Testvolumens anzugeben (Testvolumenbereich von 3dmod abrufen). Extrahieren Sie das Testvolumen, indem Sie auf Testvolume extrahieren klicken.
- Filtern Sie das Testvolumen mit verschiedenen K-Werten (Suchen eines K-Werts für das Testvolumen > Liste der K-Werte), z. B. 0,1, 1,5, 10, 15, 25, 30, 50 und 75. Führen Sie die NAD-Filterung mit jedem der angegebenen K-Werte durch (Mit unterschiedlichen K-Werten ausführen > Testergebnisse mit unterschiedlichen K-Werten anzeigen). Verwenden Sie das Slicer-Fenster, um die Leistung des NAD-Filters zu bewerten, und wählen Sie einen K-Wert aus, der zum Filtern des gesamten Volumes verwendet wird.
- Verwenden Sie den in Schritt 2.2.4 identifizierten K-Wert, und testen Sie eine unterschiedliche Anzahl von Iterationen, z. B. 2, 5, 8, 11, 15 und 21 (Suchen Sie eine Iterationsnummer für das Testvolumen > Liste der Iterationen). Werten Sie die Leistung des NAD-Filters für verschiedene Iterationen aus, indem Sie entweder auf Unterschiedliche Iterationstestergebnisse anzeigen klicken oder ein Slicer-Tool verwenden. Wählen Sie einen Wert für eine andere Anzahl von Iterationen aus.
- Verwenden Sie den identifizierten K-Wert und die Anzahl der Iterationen (Volles Volumen filtern > K-Wert und Iterationen), und filtern Sie das gesamte Volumen (Volles Volumen filtern). Ein neu gefiltertes Volume wird im selben Verzeichnis angezeigt, in dem sich das geladene vollständige Volume befindet, mit der Dateierweiterung .nad, die an die .mrc-Datei angehängt ist.
HINWEIS: Mit diesen Schritten wird eine .mrc-Datei erstellt, die mit einem NAD-Filter gefiltert wurde und ein geringeres Rauschen und damit ein verbessertes Signal-Rausch-Verhältnis aufweist.
3. Volumetrischer Modellbau
- Vorbereitung der Platzierung von 3D-Modellen
HINWEIS: Der Zweck dieses Schritts besteht darin, das Map für das Platzieren von volumetrischen 3D-Modellen in das rotierte und entrauschte Dichte-Map mit dem UCSF Chimera-Programm vorzubereiten und zu analysieren.- Erhalten Sie mit Hilfe des Slicer-Fensters eine Querschnittsansicht (Platte mit 30 Schichten/28,4 nm), um die Z-Koordinaten des Mittelpunkts der Dichteplatte zu identifizieren, in der das Aktinfilamentmodell platziert werden soll. Klicken Sie auf die Mitte der Filamentdichte und notieren Sie sich die Z-Koordinaten im IMOD-Hauptfenster.
- Notieren Sie sich die Koordinaten, die für die zukünftige Modellplatzierung in UCSF Chimera identifiziert wurden.
HINWEIS: Die in diesem Schritt notierten Z-Koordinaten werden in Schritt 4 verwendet, um vorgefertigte Aktin-Filament-Modelle in den richtigen Z-Höhen leicht zu finden und zu platzieren, wodurch die Modellplatzierung beschleunigt wird.
- Platzierung von 3D-Modellen
HINWEIS: Der Zweck dieses Schritts besteht darin, ein volumetrisches 3D-Modell mit UCSF Chimera in die vorbereitete Dichtekarte einzufügen.- Öffnen Sie in UCSF Chimera die gefilterte, gedrehte Map (Datei > Öffnen)
- Überprüfen Sie, ob die Parameter der Karte korrekt eingestellt sind, indem Sie in der Volumenanzeige Features > Ebenen und Feature->-Koordinaten auswählen, um die Benutzeroberfläche für Features und Koordinaten zu öffnen. Überprüfen Sie Voxelgröße , um festzustellen, ob der richtige Voxelabstand mithilfe des Befehls header in der Eingabeaufforderung (im IMOD) festgelegt ist. Wenn nicht, korrigieren Sie die Benutzeroberfläche für die Voxelgröße auf den richtigen Voxelabstand. Zentrieren Sie die Map, um die Kamera in der Mitte des Ansichtsfensters zu platzieren (Ursprungsindex > Mitte).
- Öffnen Sie das Fenster Kamerasteuerung (Extras > Anzeigesteuerung > Kamera). Legen Sie dann die Kameraansicht auf die orthogonale Ansicht fest (Projektion > orthogonal)
- Laden Sie das vorgefertigte Python-Skriptmodell (Datei > Öffnen > ActinFilamentPlane.py), und öffnen Sie so viele Aktin-Filament-Ebenen-Skripte, wie Aktin-Filamentebenen in der Karte vorhanden sind.
- Verschieben Sie die geladenen Modelle, indem Sie den Bewegungsmausmodus aktivieren (Extras > Bewegung > Bewegungsmausmodus). Aktivieren Sie die Option "Bewegungsmaus" (Bewegungsmausmodus einstellen > Auswahl verschieben). Verwenden Sie Strg + Ziehen , um alle in der Sitzung vorhandenen Modelle auszuwählen, und klicken Sie mit der rechten Maustaste (oder einer anderen Taste, die in den Einstellungen > Maus angegeben ist), um die Modelle in die gewünschte Richtung zu verschieben.
HINWEIS: Die Modelle wurden mit Kugeln und Stöcken mit einer bestimmten Dicke (Radius ActinFilamentPlane.py Schrift) und mit bestimmten Abständen (Lattice_Spacing ActinFilamentPlane.py Schrift) erstellt. - Um nur das Modell zu visualisieren, an dem man interessiert ist, öffnen Sie die Gruppe Modell (Extras > Allgemeine Steuerelemente > Gruppe Modell). Klicken Sie auf die Kontrollkästchen unter S (Anzeigen), um die Sichtbarkeit eines bestimmten Modells zu aktivieren oder zu deaktivieren.
- Aktivieren Sie das Befehlszeilenfenster (Extras > Allgemeine Steuerelemente > Befehlszeile).
- Nachdem Sie jedes Aktin-Filament-Ebenenmodell auf die richtige Z-Höhe gebracht haben, löschen Sie alle überschüssigen Aktin-Filamente in jedem Aktin-Filament-Ebenenmodell, indem Sie den überschüssigen Teil der Modelle auswählen und in der Befehlszeile am unteren Rand des UCSF-Chimera-Hauptansichtsfensters del sel (Delete Selected) eingeben.
- Visualisieren Sie jeweils nur eine Aktin-Filamentebene, indem Sie zum Modell-Panel-Fenster gehen und alle anderen Aktin-Filamentmodelle auswählen und ausblenden (Strg + Klick > Ausblenden).
- Um Teile der Map-Dichte zu visualisieren, die sich in unmittelbarer Nähe des gewünschten Modells mit einer einzelnen Aktinfilamentebene befinden, wählen Sie das Modell aus (Gruppe Modell > auswählen), und öffnen Sie das Zonensteuerungsfeld in der Volumenanzeige (Volumenanzeige > Funktionen > Zone). Legen Sie den Zonenradius auf 100 Å fest, und klicken Sie dann auf Zone , um die Karte in einem Radius von 100 Å (= 10 nm) vom ausgewählten Modell zu zonieren. Beachten Sie, dass die Dimension in Chimera in Angstrøm festgelegt ist (1 Å = 0,1 nm).
- Passen Sie das Modell bei Bedarf an die Dichtekarte an, indem Sie den Bewegungsmausmodus aktivieren. Wählen Sie alle Modelle mehrfach aus, die sich nicht an der richtigen Position befinden, um die Modelle so zu verschieben, dass sie ordnungsgemäß in der zonierten Dichtekarte platziert werden.
- Um einen Programmierfehler in ActinFilamentPlane.py zu beheben, führen Sie das Skript FixingMarkerID.py aus, das die fehlende MarkerID der "Atome" (Kugeln im Ball-and-Stick-Modell) behebt. Wählen Sie das gesamte Modell aus, und geben Sie runscript FixingMarkerID.py in die Befehlszeile ein (das Skript sollte im richtigen Verzeichnis angegeben werden, z. B. C: \ Verzeichnis \ Script.py).
- Wenn die Dichtekarte anzeigt, dass das Aktinfilament gekrümmt ist, stellen Sie sicher, dass Sie das gerade Aktinfilament in mehrere Teile aufteilen, um die Krümmung auszugleichen, indem Sie die gewünschten Modelle auswählen und runscript dividelinks.py Nummer eingeben. Zusätzliche Marker (angegeben durch die Zahl im Befehl) werden innerhalb des Aktinmodells in gleichem Abstand zueinander platziert, wodurch "Gelenke" entstehen, an denen das Modell gebogen werden kann.
- Passen Sie die Gelenke an, um die Krümmung widerzuspiegeln, indem Sie mithilfe des Bewegungsmausmodus zusätzliche Markierungs-"Gelenke" entlang der Zonenkarte verschieben. Fügen Sie dem Modell bei Bedarf zusätzliche Marker hinzu, indem Sie das Skript dividelinks.py verwenden.
- Um zu überprüfen, ob alle Aktinfilamente korrekt innerhalb der Aktindichten platziert sind, verwenden Sie Schnittleisten, indem Sie im Dropdown-Menü UCSF Chimera auf Werkzeuge > Anzeigesteuerung > Kamera klicken, um das Fenster Kamerasteuerung zu öffnen, und dann Seitenansicht auswählen, um die beiden Schnittbalken (Nah- und Fernschnittebene) der Kamera anzuzeigen. Ziehen Sie zwei gelbe Balken in einen schmalen Schlitz, indem Sie mit der linken Maustaste klicken, und ziehen Sie den Schnittteil mit der mittleren Maustaste , um die Modelle ein kleines Stück nach dem anderen zu untersuchen.
HINWEIS: Das Stereozilium-Aktin-Kernmodell ist nun vollständig.
- Segmentierung der Membran
HINWEIS: Der Zweck dieses Schritts besteht darin, ein Oberflächenmodell der Stereozilienmembran zu erstellen.- Für die Segmentierung von Oberflächen wird das Werkzeug Volumen-Tracer verwendet (Extras > Volumendaten > Volumen-Tracer).
- Klicken Sie im Fenster Volume Tracer auf Maus, und aktivieren Sie nur die Optionen Marker auf Datenebenen platzieren und Neue Marker mit ausgewählter Marker verknüpfen. Dies ermöglicht es, die Marker präzise auf der gleichen Z-Ebene zu platzieren (wenn eine einzelne Z-Ebene angezeigt wird). Aktivieren Sie auch die Option Marker mit der mittleren Maustaste im Fenster Volume Tracer platzieren.
- Erstellen Sie einen neuen Markierungssatz (Datei > Neuer Markierungssatz). Marker-Set 1 sollte erstellt und im Fenster Volume Tracer ausgewählt werden.
- Verwenden Sie die Volumenanzeige, um einzelne Z-Ebenen im Hauptanzeigefenster anzuzeigen (Achse zu Z > Eins).
- Zeichnen Sie mit der mittleren Maustaste die Membran von einem Ende der sichtbaren Membran zum anderen nach. Alle Marker sollten durch Glieder von einem Ende zum anderen verbunden sein.
- Heben Sie die Auswahl der zuletzt platzierten Markierung auf, indem Sie Strg + Klick auf den Hintergrund drücken, an dem sich kein einzelnes auswählbares Element befindet. Dadurch wird sichergestellt, dass der nächste Marker, der platziert werden soll, mit Verknüpfungen in der neuen Ebene verbunden ist und nicht mit einer vorherigen Membranbahn einer anderen Z-Ebene.
- Wiederholen Sie die Membranverfolgung alle 10 nm in der gleichen Richtung wie die Verfolgung von einem Ende der sichtbaren Membran zu einer anderen Seite.
- Nachdem viele parallele Membranbänder segmentiert sind, wechseln Sie zum Fenster Volumenverfolgung, und klicken Sie auf Features > Oberflächen. Dadurch wird die Oberflächenschnittstelle im Fenster Volume Tracer aktiviert. Klicken Sie neben der Benutzeroberfläche Oberflächen auf Erstellen, um Flächen zwischen den Bändern der Membransegmentierung zu erstellen. Dadurch entsteht eine dünne Klappe des Oberflächenmodells, die die beabstandeten Membranspuren ausfüllt.
HINWEIS: Die Erstellung des Oberflächenmodells der Stereoziliummembran ist nun abgeschlossen.
- Crosslinker-Modellierung
HINWEIS: Der Zweck dieses Schritts besteht darin, die Vernetzer zwischen Aktinfilamenten zu modellieren.- Vernetzer können auch mit dem Volume Tracer-Werkzeug platziert werden.
- Klicken Sie im Fenster Volumenverfolgung mit der linken Maustaste, um nur die Optionen Marker auf Flächen platzieren und Neuen Marker mit ausgewähltem Marker verknüpfen zu aktivieren. Aktivieren Sie dann die Option Marker mit der mittleren Maustaste platzieren im Fenster Volume Tracer .
- Visualisieren Sie die Dichten um das interessierende Modell der einzelnen Aktinfilamentebene nur mithilfe der Zonenfunktion. Legen Sie den Zonenradius auf 100 Å fest, um die Karte in einem Radius von 100 Å vom ausgewählten Modell zu visualisieren.
- Platzieren Sie Markierungen in den sichtbaren Dichten von Vernetzern, indem Sie mit der mittleren Maustaste klicken. Sobald zwei Marker platziert sind (und somit ein einzelner Vernetzer modelliert wird), stellen Sie sicher, dass Sie die Auswahl des zweiten Markers aufheben, um zu verhindern, dass Links kontinuierlich mit dem nächsten Vernetzermodell verbunden werden.
- Wiederholen Sie die obigen Schritte für jede Aktin-Filamentebene und wiederholen Sie den gesamten obigen Vorgang in alle drei Hauptrichtungen, in denen sich Vernetzer befinden können. Wenn Sie Aktinfilamentebenen in diagonaler Richtung zonieren, wählen Sie die Aktinfilamentmodelle manuell einzeln aus.
HINWEIS: Für jedes der Aktinvernetzermodelle können wir die Position auf den Aktinfilamenten aufzeichnen, mit der sich die mutmaßlichen Vernetzer verbinden. Diese Informationen könnten prinzipiell für eine Folgeanalyse mittels Sub-Tomogramm-Mittelung extrahiert werden, was den Rahmen dieser Studie sprengen würde.
4. Quantitative Analyse des 3D-Modells
- Gap-Analyse
HINWEIS: Der Zweck dieses Schritts besteht darin, Lücken in den Aktinfilamenten zu erkennen.- Ausgewählte Elemente können analysiert werden. Geben Sie ihre Parameter, wie z. B. ihre Farbe, mit dem Werkzeug "Auswahl prüfen " (Aktion > "Prüfen") an.
- Die Farbe des ausgewählten Elements kann angegeben werden, indem Sie ein beliebiges auswählbares Element in UCSF Chimera auswählen und die Farbe neben dem Abschnitt "Farbe " des Fensters "Auswahl prüfen " ändern.
- Geben Sie die Größe der Marker und Verknüpfungen an, indem Sie alle Markierungen und Verknüpfungen in UCSF Chimera auswählen und die Werte neben dem Abschnitt "Radius " des Fensters "Auswahl prüfen " ändern, in dem sich die Einheiten in Angstrøm befinden.
- Führen Sie eine Lückenanalyse durch, indem Sie Dichtelücken automatisch erkennen, indem Sie die Funktion "Werte an Atompositionen " verwenden (Extras > Volumendaten > Werte an Atompositionen).
- Wählen Sie die Map aus, in der die Werte gelesen werden sollen, indem Sie die gewünschte Map neben dem Abschnitt "Volumendaten " des Fensters "Werte an Atompositionen " auswählen. Wählen Sie das Modell aus, in dem allen Markern ein Parameter zugewiesen werden soll, indem Sie das gewünschte Modell neben dem Abschnitt Molekül auswählen.
- Mit der Schaltfläche "Histogramm" am unteren Rand des Fensters "Werte an Atompositionen" wird ein Histogramm in einem neuen Fenster mit dem Namen "Rendern/Nach Attribut auswählen" erstellt. Strg + Klick auf das Histogramm fügt Cutoff-Punkte hinzu oder entfernt sie, für die verschiedene Farben ausgewählt werden können. Deaktivieren Sie "Undurchsichtig halten", um eine transparente Farbe auf die Szene anzuwenden. Marker mit einer Dichte unterhalb eines bestimmten Schwellenwerts, der Lücken darstellt, können nicht nur eingefärbt, sondern auch ausgewählt werden und ermöglichen es uns so, die Anzahl der Marker im Lückenbereich zu zählen. Um auszuwählen, klicken Sie auf > Attribut auswählen > value_mapname.
- Bestimmung von Inter-Aktin-Abständen
- Um Abstände zwischen den Aktinen zu messen, platzieren Sie Marker auf einer einzelnen XZ-Ebene (in UCSF Chimera als Y-Ebene bezeichnet), indem Sie das Fenster Volume Tracer verwenden, mit der Maus klicken und nur Marker auf Datenebenen platzieren aktivieren und die Option Neue Markierung mit ausgewähltem Marker verknüpfen deaktivieren. Dies ermöglicht es, die Marker präzise auf der gleichen Y-Ebene zu platzieren (wenn eine einzelne Z-Ebene angezeigt wird). Aktivieren Sie auch die Option Marker mit mittlerer Maustaste platzieren im Fenster Volume Tracer und platzieren Sie die Marker auf einer Ziel-Y-Ebene, die für die Messung des Inter-Aktin-Abstands von Interesse ist.
- Sobald die Aktinpositionen einer einzelnen Y-Ebene erfolgreich markiert wurden, wählen Sie alle Markierungen aus und geben Sie den Befehl findclash #model-spec test self overlapCutoff -200 hbondAllowance 0 log true Linewidth 10 pbColor black in die Befehlszeile am unteren Rand der Aktinfilamentebene ein. Dadurch werden alle Kollisionen zwischen den Markern in einem Abstand von 200 Å (overlapCutoff -200) gefunden und es werden schwarze Linien (pbColor black) mit einer Dicke von 10 Å (Linienbreite 10) zwischen den Markern erstellt.
- Da es überlappende Marker innerhalb des angegebenen Bereichs von 200 Å gibt, entfernen Sie redundante Messungen, die nicht nur Verbindungen zwischen den nächstgelegenen, benachbarten Markern sind. Verwenden Sie dazu das RemoveCross.py Skript, das auf der UCSF Chimera-Website bereitgestellt wird und alle überlappenden Links entfernt, die nicht die kürzeste Verbindung mit den angrenzenden Markierungen sind.
- Um den Abstand der verbleibenden Bindungen zu messen, wählen Sie alle Pseudobindungen (Links, die von findclash erstellt werden) aus und verwenden Sie das Python-Skript pblengths.py , indem Sie runscript pblengths.py eingeben. Sehen Sie sich die gemessenen Werte aus dem Antwortprotokoll an (Extras > Dienstprogramme > Antwortprotokoll). Kopieren Sie dann die erhaltenen Werte aus dem Antwortprotokoll und übertragen Sie sie in eine Tabelle oder ein anderes geeignetes Tool, um ein Histogramm zu erstellen.
Ergebnisse
Mit Hilfe der Kryo-Elektronentomographie von ungefärbten, gefroren hydratisierten einzelnen Stereozilien, die in Glaseis eingebettet sind, erhielten wir Dichtekarten des Aktinbündels mit seinen hexagonal angeordneten Aktinfilamenten, die durch Kreuzkonnektorproteine verbunden sind23. Die Abmessung eines einzelnen Voxels betrug 0,947 nm. Die visuelle Inspektion eines Volumen-Renderings des gesamten Tomogramms (400 Schichten/379 nm) im IMOD-Slicer-Programm zeigte das Vorhandensein von filamentösen Strukturen, die mit der Längsachse der Stereozilien ausgerichtet sind, wie in Längsansichten (XY-Ebene; Abbildung 1A-C, obere Felder) sowie in Schnittansichten (XZ-Ebene; Abbildung 1A-C, Bodenplatten). Wir stellten fest, dass die Projektionsansicht durch das 400 Scheiben/379 nm Filamentnetzwerk am klarsten wurde, wenn das original rekonstruierte Volumen um -6° um die X-Achse, -13,5° um die Y-Achse und 5° um die Z-Achse gedreht wurde. In diesem Winkel sind alle Filamente übereinander ausgerichtet, und daher ist der Kontrast maximal, wie man an den Schnittansichten erkennen kann (Abbildung 1B). Da einzelne Querschnittsschnitte nicht über genügend Signal verfügen, um Aktinfilamente eindeutig zu unterscheiden, haben wir uns für das Volumen-Rendering einer Platte von 30 Schichten/28,4 nm entschieden, die in der Querschnittsansicht deutlich ein hexagonales Muster zeigt. Die blauen Linien in Abbildung 1C (obere Felder) zeigen die Position der Mitte der entsprechenden Platten mit 30 Schichten/28,4 nm Querschnitt in den unteren Platten.
Kleine Abweichungen von diesem optimalen Betrachtungswinkel von nur ±2° reduzierten die wahrgenommene Ordnung des Aktinfilament-Netzwerks signifikant (Abbildung 1A,C), was ein Hinweis darauf ist, wie leicht es ist, sich im 3D-Volumen des Tomogramms zu verlieren.
Um die Herausforderung bei der Verwendung automatisierter Segmentierungsansätze, wie z. B. der Segmentierung von Wassereinzugsgebieten, zu veranschaulichen, haben wir ein kleines Teilvolumen (als Gold dargestellt) für die Segmentierung von Wassereinzugsgebieten ausgewählt, wie es im UCSF Chimera-Softwarepaket implementiert ist (Tools > Volume Data > Segger > Segment). Die Position des Subvolumens relativ zur gesamten Stereozilienkarte ist durch den kleinen Einschub in Abbildung 1B dargestellt.
Abbildung 1D-F zeigt das gewählte Subvolumen in verschiedenen Ausrichtungen, wobei Abbildung 1D,E die Betrachtungsrichtung in Längsrichtung und Abbildung 1F eine Querschnittsbetrachtungsrichtung zeigt. Die Pfeile auf der linken Seite von Abbildung 1D-F zeigen die Richtung der Aktinfilamente.
Abbildung 1D-F (rechte Felder) zeigt die Ergebnisse der Segmentierung von Wassereinzugsgebieten. Das Subvolume wird nach Objektidentität farbcodiert, wobei die Farben den verschiedenen Objekten nach dem Zufallsprinzip zugewiesen werden. Unterschiedliche Farben deuten auf eine unterschiedliche Objektidentität hin, daher wird aus Abbildung 1D-F deutlich, dass die Kartendichten für Filamente beide entlang der Filamentachse fragmentiert waren, während die gleiche Farbe und damit Objektidentität für Kartendichten gegeben wurde, die benachbarte Filamente miteinander verbanden. Mit anderen Worten, der Segmentierungsalgorithmus des Wassereinzugsgebiets war nicht in der Lage, der Dichtekarte von Aktinfilamenten über einen längeren Zeitraum zu folgen, und führte stattdessen dazu, dass die Dichten von benachbarten Filamenten verbunden wurden. Es ist zwar möglich, die Auswahl manuell zu kuratieren (z.B. durch Löschen oder Zusammenführen von Objekten), aber dieser Ansatz ist ziemlich arbeitsintensiv und damit zeitaufwändig.
Obwohl es für unsere volumetrische Modellerstellungsstrategie nicht unbedingt notwendig ist, hat es geholfen, die 3D-Karte neu auszurichten (zu drehen), so dass die Netzwerkachse des Aktinfilaments mit der Y-Achse und die Modellebenen des Aktinfilaments mit der X-Y-Ebene des Tomogramms ausgerichtet sind. Wir bezeichnen diese Orientierung als Standardausrichtung für die stereozilientomographische Darstellung.
Wir beschlossen daher, eine andere Strategie für die Bildsegmentierung zu erforschen und die Tatsache auszunutzen, dass die Aktinfilamente eine insgesamt regelmäßige Organisation (hexagonale Packung) mit regelmäßigen Abständen und definierter Gesamtbündelausrichtung aufwiesen. Unsere Strategie bestand darin, eine Gesamtanpassung der Modelle eines Aktinbündels als Array von Filamenten zu finden, gefolgt von regionalen und dann lokalen Anpassungen der Modellposition, um sie an die experimentelle Dichtekarte anzupassen. Indem wir ein Gesamtmodell an die erste Stelle setzen, können wir lokale Kartenmehrdeutigkeiten überwinden und regionale Trends bei Abweichungen des Modells von seiner ursprünglichen Organisation, wie z. B. Filamentbiegung, erkennen.
Um das Modell zu platzieren, zeigten wir Platten mit der Dichte (10 Scheiben/9,47 nm) in der Standardausrichtung, die einer Dicke einer einzelnen Schicht der Aktinfilamente entsprach, an die eine Schicht aus regelmäßig verteilten, geraden Aktinfilamentmodellen angepasst wurde. Dies ist natürlich eine zu starke Vereinfachung von Aktinfilamenten, von denen jedes aus einer linearen Anordnung von Aktinmonomeren mit helikaler Symmetrie besteht. Abbildung 2A-C zeigt drei repräsentative Schichten in unterschiedlichen Z-Höhen, wobei die rot gefärbten Stäbchen die Aktinfilamente darstellen. Die oberen Felder, die ~30 Scheiben/28,4 nm dicke Querschnitte darstellen, zeigen, auf welcher Z-Höhe eine einzelne Aktinmodellschicht von 19 Stäben platziert wurde, während die unteren Tafeln eine Längsausrichtung zeigen (wenn auch in perspektivischer Ansicht dargestellt). Abbildung 2D zeigt das vollständige vereinfachte Modell, sowohl in der Querschnittsansicht (oberes Bild) als auch in der längsperspektivischen Ansicht (unteres Bild). Die Querschnittsausrichtung ermöglichte es uns, die Filamente mit guter Sicherheit zu positionieren. Hier erwies sich unser ursprünglicher Schritt, das gesamte Volumen so auszurichten, dass es mit den Hauptachsen des Tomogramms übereinstimmte, als hilfreich, da dies bedeutete, dass die Ausrichtung unseres Modells in unserer Standardbetrachtungsrichtung auch parallel zu den Hauptachsen verlief. Streng genommen hätte unser Ansatz aber auch ohne die Neuausrichtung des Tomogramms funktioniert, allein die Platzierung des Modells zur Dichte wäre herausfordernder gewesen.
Bei sorgfältiger Untersuchung einzelner Platten der Dichtekarte stellten wir fest, dass ein perfekt gerades Aktinmodell nicht zu der beobachteten Dichtekarte passte, die sich vom proximalen Ende zum distalen Ende (d.h. zur Spitze) der Stereozilien bewegte (Abbildung 3A-C). In der Nähe der Spitze der Stereozilien war die Kartendichte für Filamente um mehr als 13 nm verschoben (Aktin-Aktin-Abstand), was wir durch Anpassung des Modells kompensieren konnten, während wir vom proximalen zum distalen Teil der Stereozilien-Dichtekarte übergingen, wodurch eine kleine, aber unterscheidbare allmähliche Krümmung in unser Aktinmodell eingeführt wurde. Abbildung 3D zeigt eine einzelne Platte der Dichtekarte des Aktinfilaments, mit einem volumetrischen Modell, das an die Dichtekarte angepasst ist. Ein Vergleich zwischen dem geraden (rot) und dem gekrümmten (gelb) Modell ist in Abbildung 3E dargestellt. Diese Krümmung wird am besten erkannt, indem man eine Platte der Dichtekarte mit dem platzierten Modell um 80° um die X-Achse neigt, was einen perspektivischen Blick entlang der Richtung der Aktinfilamente ermöglicht (Abbildung 3D,E).
Die Abweichung der beiden Modelle, bei der die Position des Aktinmodells in der Nähe der Spitze um etwa den gleichen Abstand wie der Abstand der Aktinfilamente verschoben wurde, hätte viel Verwirrung stiften können, wenn wir nicht so vorgegangen wären, wie wir es getan haben. Diese "globale" Positionierung einer Schicht des Aktinfilamentmodells, gefolgt von einer "regionalen" Anpassung, ermöglichte es uns, diese Krümmung zu erkennen, die in der Längs- oder Querschnittsansicht kaum wahrnehmbar ist. Überlagert man jedoch die beiden Modelle, wie in Abbildung 3E gezeigt, erkennt man den feinen Unterschied.
Die Wiederholung dieses Ansatzes für mehrere Schichten ermöglicht es, ein vollständiges 3D-Modell zu erhalten (Abbildung 3F), das nur durch die Datenunsicherheit am oberen und unteren Rand der Stereozilien begrenzt ist, wenn es in der Querschnittsausrichtung betrachtet wird (Abbildung 3G). Dieser Mangel an Dichte wird durch den fehlenden Keil in der (einachsigen) tomographischen Datenerfassung und die entsprechende Anisotropie der Datenauflösung verursacht, und sein Effekt wird durch das Fehlen einer gut definierten Kartendichte für Stereozilienmembranen angezeigt.
Sobald wir ein 3D-Modell hatten, haben wir jede Position des volumetrischen Modells entsprechend dem Wert der Kartendichte an dieser Position farbcodiert. Regionen des Modells mit zugrundeliegender schwacher Kartendichte wurden rot eingefärbt, während Regionen des Modells mit starkem Kartendichtesignal gelb eingefärbt wurden (Abbildung 4A). Wir interpretieren solche rot gefärbten Bereiche, die sich bis zu mehreren Dutzend Nanometern erstrecken können, als Lücken in den Aktinfilamentstrukturen, die aufgrund ihrer Ausdehnung nicht auf Dichteschwankungen zurückgeführt werden können, die in der rauschreichen Umgebung einer Kryo-EM-Karte häufig auftreten. Rauschen neigt dazu, einzelne Voxel oder kleine Gruppen von Voxeln zu beeinflussen, aber es ist unwahrscheinlich, dass es die Quelle für Volumina ist, die aus Hunderten von Voxeln bestehen und für die die Filamentdichte fehlt. Stattdessen sind solche Lücken wahrscheinlich ein reales Merkmal des Stereozilien-Aktin-Geflechts und können Orte des Aktinumsatzes darstellen. Abbildung 4A zeigt zwei unterschiedliche Kartendichtewerte, die in Hellblau und Dunkelblau dargestellt sind. Es sollte ausdrücklich darauf hingewiesen werden, dass unser Ansatz zur Erstellung volumetrischer Modelle, kombiniert mit einer automatisierten Farbcodierung unseres Modells in Regionen mit schwacher Dichte, eine schnelle und bequeme Möglichkeit ist, die Verteilung solcher Lücken im Aktinfilament-Modell zu erkennen und zu visualisieren, was sonst sehr schwierig gewesen wäre.
Wie in Abbildung 4B gezeigt, können Teile des volumetrischen Modells an Stellen mit einer vergleichbar schwachen Dichte auf der Grundlage der in Abbildung 4A erzielten Ergebnisse leicht verborgen werden. Dies führt dann zu einem fragmentierteren Modell, das das Aktinmodell bei Stereozilien realistischer abbilden kann. Die Alternative, kleine Abschnitte von Aktinfilamenten zu bauen, wäre sehr arbeitsintensiv gewesen und hätte aufgrund der Probleme, die in Abbildung 1 diskutiert wurden, möglicherweise ganz gescheitert.
Darüber hinaus ermöglicht uns das volumetrische Modell eine einfache Modellierung der Querverbinder, indem wir einfach eine Verbindung (rot dargestellt) zwischen den Modellpunktpositionen des Aktinfilamentmodells auf beiden Seiten der Querverbindung platzieren (Abbildung 4C). In unserem vereinfachten Ansatz müssen wir keine Annahmen über die genaue Identität jedes anderen Proteins treffen, was eine höhere Auflösung und/oder ausgefeilte Markierungsansätze erfordern würde. Stattdessen müssen wir nur feststellen, ob eine Dichte vorhanden ist, die benachbarte Aktinfilamente überbrückt. Wenn ja, können wir eine kurze Verbindung von einem Filament zu seinem benachbarten Gegenstück herstellen. In Abbildung 4D ist ein Modell von fünf Aktinfilamenten mit ihren Querverbindern dargestellt, das einen Eindruck von der Verteilung der Querverbinder entlang der Aktinfilamentachse vermittelt.
Ein weiterer Vorteil bei der Erstellung eines volumetrischen Modells des Aktinbündels besteht darin, dass man schnell den Abstand zwischen benachbarten Aktinfilamenten bestimmen kann (Abbildung 4E-H). Abbildung 4E,F zeigt eine Querschnittsansicht der Dichtekarte ohne bzw. mit einem Modell, das an das hexagonale Gitter der Kartendichte angepasst ist. Abbildung 4G zeigt das Modell mit Verbindungen zwischen den nächstgelegenen benachbarten Kugeln. UCSF Chimera ermöglicht die automatische Berechnung der Entfernung von Nächste-Nachbar-Zentren, deren Ergebnis dann als Entfernungsverteilung dargestellt werden kann (Abbildung 4H). Die Modellerstellung für zwei zusätzliche Datensätze ist in der ergänzenden Abbildung 1 und der ergänzenden Abbildung 2 dargestellt.
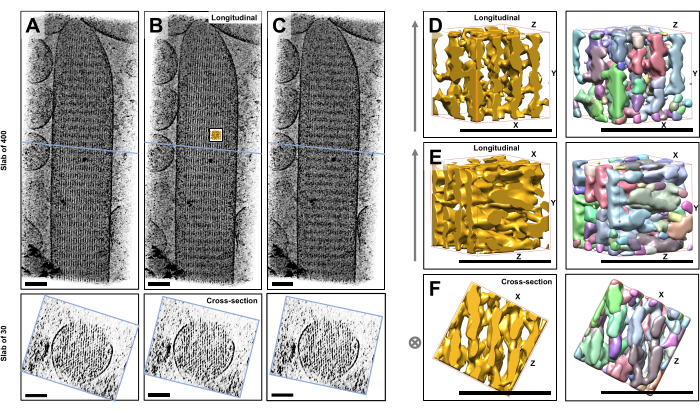
Abbildung 1: Die Herausforderungen bei der Segmentierung von Stereozilien von Haarzellen-Tomogrammen. (A-C) Längsprojektionen (400 Schichten/379 nm) durch die tomographische 3D-Karte in der XY-Ebene (obere Felder) und Querschnittsansichten (30 Schichten/28,4 nm) in der XZ-Ebene (untere Felder). (A) Tomographische Karte, die um -2° entlang der Y-Achse von ihrer optimalen Ausrichtung gedreht wurde. (B) Tomographische Karte in optimaler Orientierung, bestimmt durch Anpassung der Rotationswinkel der X-, Y- und Z-Achse (X = -6°, Y = -13,5° und Z= 5°) und zeigt einen hohen Grad an Ordnung in der Dichtekarte, was auf ein hochgeordnetes Aktinfilament-Netzwerk hindeutet. (C) Tomographische Karte, die um +2° entlang der Y-Achse von ihrer optimalen Ausrichtung gedreht wurde; Die Drehung von nur 2° um die Y-Achse weg von der optimalen Blickrichtung beeinträchtigt die wahrgenommene Regelmäßigkeit der Dichtekarte erheblich. Die unteren Felder zeigen die Regelmäßigkeit des Aktinfilament-Arrays, wenn man es in Querschnittsrichtung betrachtet. Die blaue Linie in A-C zeigt die Position der Querschnittsplatte an. (D-F) Ein Würfel von 50 nm x 50 nm x 50 nm, betrachtet aus drei verschiedenen Richtungen vor (linke Felder) und nach (rechte Felder) Segmentierung. Beachten Sie, dass die Segmentierung von Wassereinzugsgebieten die Dichte der kontinuierlichen Aktinfilamente nicht erkennt, während benachbarte Aktinfilamente und ihre Querverbindung die gleiche Objektidentität aufweisen, was darauf hindeutet, dass die Segmentierung von Wassereinzugsgebieten kein geeigneter Ansatz für die Tomogrammsegmentierung ist. In den Feldern D-F wird das Dichte-Map in Chimera als Map-Stil "Oberfläche" angezeigt. (A-C) Maßstabsbalken = 100 nm. (D-F) Maßstabsbalken = 50 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
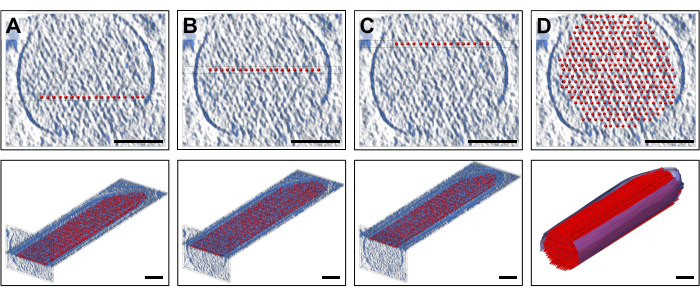
Abbildung 2: Aufbau eines volumetrischen Kugel-und-Stick-Aktin-Filament-Modells. (A-C) Oben: Querschnittsansichten der Dichtekarte mit 30 Schichten/28,4 nm mit einem einlagigen, vereinfachten Aktin-Filament-Modell, das auf einer anderen Z-Höhe platziert ist. Unten: eine einzelne 10-Schicht-/9,47-nm-Platte der Dichtekarte mit einem einlagigen, vereinfachten Aktin-Filament-Modell. (D) Vollständiges Modell eines geraden Aktinfilaments in einer Querschnittsansicht von 30 Schichten/28,4 nm (oben) und perspektivischer Ansicht (unten); Maßstabsbalken = 100 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
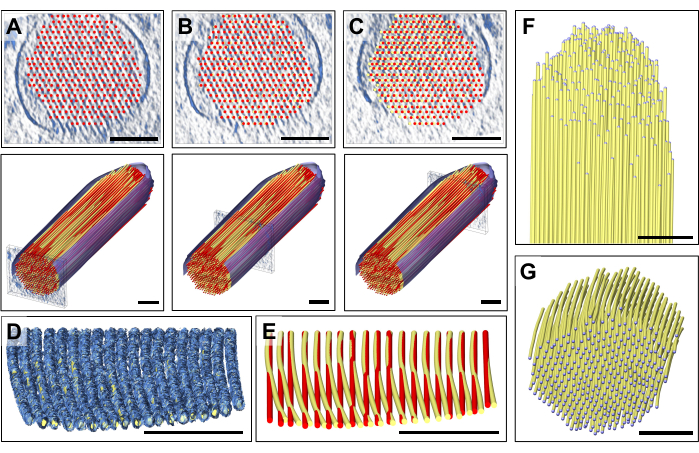
Abbildung 3: Modellanpassung zur Korrektur der erkannten Biegung der Aktinfilamentdichte. (A-C) Eine sorgfältige visuelle Inspektion des Modells, entweder in der Querschnittsansicht (obere Felder) oder in der Längsansicht (untere Felder), ergab eine gute Übereinstimmung des Modells mit der Dichtekarte am proximalen Ende der Stereozilien. Je näher man sich jedoch dem distalen Ende der Stereozilien nähert, desto schlechter wird die Passform bei allen Aktinfilamentmodellen. Dies kann korrigiert werden, indem die Kugeln des Ball-and-Stick-Modells in die richtige Position der Dichtekarte verschoben werden, was zu einem leicht gekrümmten Aktinfilamentmodell führt. Das gerade Modell wird in Rot und das korrigierte gebogene Modell in Gelb dargestellt. (D) Einzelne Platte der Dichtekarte mit dem daran angepassten gekrümmten Modell, das die Krümmung der Aktindichte zur Spitze der Stereozilien hin zeigt. Das Aktin-Modell wurde um 80° um die X-Achse gedreht, um diese subtile, aber signifikante Biegung der Aktinfilamente besser zu zeigen. (E) Vergleich zwischen dem geraden, unkorrigierten Aktinmodell, das in Rot dargestellt ist, und dem gekrümmten, korrigierten Aktinmodell, das in Gelb dargestellt ist. Aus Gründen der Übersichtlichkeit ist nur eine Schicht des Aktinfilamentmodells dargestellt. (F-G) Aktinbündelmodell mit gekrümmten, korrigierten Aktinfilamenten, dargestellt in Längs- (F) und Querschnittsausrichtung (G). Die segmentierte Membran ist blau dargestellt. In Panel D wird die Dichtekarte in Chimera als Kartenstil "Mesh" angezeigt. Maßstabsbalken = 100 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
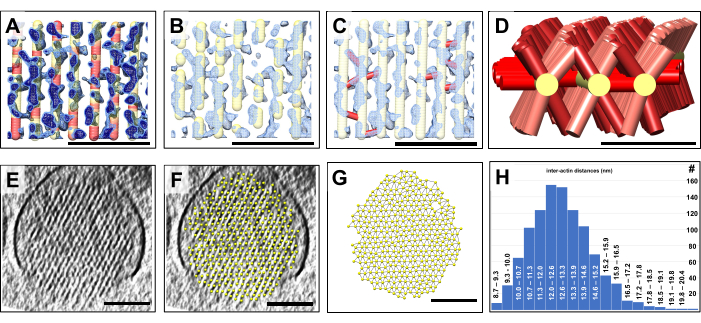
Abbildung 4: Detektion von Lücken im Aktinbündel mittels volumetrischer Modellbildung. Quantifizierung volumetrischer Eigenschaften mit Hilfe von Ball-and-Stick-Modellen. (A-C) Ein kleiner Bereich einer ~10 Schichten/9,47 nm dicken Längsplatte der Stereozilien-Dichtekarte ist in Blau dargestellt, zusammen mit dem gekrümmten, korrigierten Aktinfilamentmodell. (A) Die Dichtekarte wird bei einer niedrigeren Dichteschwelle (hellblau) und einer höheren Dichteschwelle (dunkelblau) dargestellt. Es gibt Regionen des Aktinfilament-Modells, für die es keine entsprechende Dichte gibt. An solchen Stellen wurde das Modell rot farbcodiert, um einen Mangel an Dichte darzustellen. Wir interpretieren diese Stellen als Lücken in den Aktinfilamenten. (B) Modell der Aktinfilamente, das fragmentiert erscheint, was den Mangel an Aktinfilamentdichte an solchen Lückenstellen widerspiegelt. (C) Modell der Aktinfilamente mit Querverbindungen (rot dargestellt), die an Stellen hinzugefügt wurden, an denen eine starke Dichte zur Überbrückung benachbarter Aktinfilamente festgestellt wurde. (D) Drei ausgewählte Modell-Aktinfilamente werden mit Querverbindern zu benachbarten Aktinfilamenten dargestellt (die aus Gründen der Übersichtlichkeit nicht dargestellt sind). Es ist zu beachten, dass viele, aber nicht alle möglichen Positionen der Inter-Aktin-Filamente von Konnektorproteinen besetzt werden. (E) Eine Querschnittsplatte mit 30 Schichten/28,4 nm der Stereoziliendichte. (F) Modell von Aktinfilamenten, die an die Querschnittsplatte mit 30 Schichten/28,4 nm Stereoziliendichte angepasst wurden. (G) Modell von Aktinfilamenten ohne die zugrundeliegende Kartendichte. Automatische Erkennung der nächsten benachbarten Aktinfilamentabstände, was durch dünne Verbindungen zwischen den Aktinfilament-Modellkugeln angezeigt wird. (H) Histogramm der Abstände des Aktinfilamentmodells. In den Feldern A-C wird das Dichte-Map in Chimera als Map-Stil "Mesh" angezeigt. (A-C) Maßstabsbalken = 50 nm. (D) Maßstabsleiste = 25 nm. (E-G) Maßstabsbalken = 100 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Modellbildung für den ersten von zwei zusätzlichen Stereozilien-Datensätzen. (A-C) Ein kleiner Bereich einer ~10 Schichten/9,47 nm dicken Längsplatte der Stereozilien-Dichtekarte wird mit Hilfe der Mesh-Modus-Anzeige in Blau dargestellt. Das ursprünglich platzierte Modell wird in Rot und das korrigierte Modell in Gelb dargestellt. (A) Nur Kartendichte. (B) Erstes Modell, das in die Dichtekarte eingefügt wurde. (C) Korrigiertes Modell, das in die Dichtekarte eingefügt wurde. (D-E) Größere Stereozilienregion ohne (D) und mit (E) das korrigierte Modell, angepasst an eine ~10 Schichten/9,47 nm dicke Längsplatte der Stereozilien-Dichtekarte. (F-G) Dargestellt ist der gesamte Bereich des Stereozilien-Tomogramms. (F) Nur Karte. (G) Karte mit dem korrigierten Modell. (H) Überlagerung des ursprünglichen und des korrigierten Modells. Maßstabsbalken = 100 nm Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 2: Modellbildung für den zweiten von zwei zusätzlichen Stereozilien-Datensätzen. (A-C) Ein kleiner Bereich einer ~10 Schichten/9,47 nm dicken Längsplatte der Stereozilien-Dichtekarte wird mit Hilfe der Mesh-Modus-Anzeige in Blau dargestellt. Das ursprünglich platzierte Modell wird in Rot und das korrigierte Modell in Gelb dargestellt. (A) Nur Kartendichte. (B) Erstes Modell, das in die Dichtekarte eingefügt wurde. (C) Korrigiertes Modell, das in die Dichtekarte eingefügt wurde. (D-E) Größere Stereozilienregion ohne (D) und mit (E) das korrigierte Modell, angepasst an eine ~10 Schichten/9,47 nm dicke Längsplatte der Stereozilien-Dichtekarte. (F-G) Dargestellt ist der gesamte Bereich des Stereozilien-Tomogramms. (F) Nur Karte. (G) Karte mit dem korrigierten Modell. (H) Überlagerung des ursprünglichen und des korrigierten Modells. Maßstabsbalken = 100 nm. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 1: UCSF Chimera Python-Skripte für die Stereozilien-Modellierung. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Codierungsdatei 1: pblengths.py. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Codierungsdatei 2: RemoveCross.py. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Codierungsdatei 3: ActinFilamentPlane.py. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Codierungsdatei 4: dividelinks.py. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Codierungsdatei 5: FixingMarkerID.py. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Wir haben gezeigt, dass automatisierte Ansätze zur Segmentierung, wie z. B. die Segmentierung von Wasserscheiden, in der rauschreichen und hochkomplexen Umgebung von Kryo-Elektronen-Tomogrammen von Haarzell-Stereozilien versagen können. Zu unterscheiden, welcher Teil dieses filamentösen Netzwerks Aktinfilamente repräsentiert und was Vernetzungen auf lokaler Umweltebene darstellt, scheint bestenfalls bei der Untersuchung kleiner tomographischer Subvolumina eine Herausforderung zu sein. Der in dieser Studie verwendete Modellbildungsansatz profitiert von Vorkenntnissen über die großräumige Ordnung des Aktinbündels, was dazu beiträgt, eine Erwartung über die Orientierung von Aktinfilamenten und die Vernetzerdichten zu entwickeln. Vielleicht noch wichtiger ist, dass ein menschliches Gehirn leicht Muster finden kann, indem es den größeren Kontext über die lokale Dichteverteilung hinaus berücksichtigt, während ein Computeralgorithmus nur für einen relativ kleinen Bereich funktioniert, der vom Algorithmus berücksichtigt wird; Daher können größere Trends nicht ohne weiteres berücksichtigt werden. Durch die globale Anpassung eines Modells an eine Dichteschicht haben wir die Verwirrung vermieden, die auftreten kann, wenn versucht wird, ein Modell für jeweils kleine Teile einzelner Aktinfilamente zu erstellen. Natürlich setzt eine solche globale Anpassung eine Ordnung voraus, die sich über große Entfernungen erstreckt. Da wir jedoch eine unerwartete kleine, aber signifikante allmähliche Biegung der Aktinfilamente hatten, war die globale Anpassung nur eine anfängliche Annäherung und erforderte lokale Anpassungen des Modells, um sie an die Dichtekarte anzupassen. Da das erste Modell ein guter Ausgangspunkt war, konnten die Anpassungen mit hoher Sicherheit vorgenommen werden. Ein großer Vorteil unseres Ansatzes war, dass wir uns dafür entscheiden konnten, nur eine definierte Dichtezone anzuzeigen, was dazu beitrug, die Komplexität der Szenerie zu reduzieren. Darüber hinaus half die Betrachtung der Kartendichteplatte entlang der Filamentmodellachse, die unerwartete Krümmung zu identifizieren, die wir bei der Darstellung kleinerer Teilvolumina höchstwahrscheinlich übersehen hätten. Die Platzierung des Ausgangsmodells ermöglichte auch ein schnelles Vergrößern und Verkleinern, um zwischen einer Gesamtansicht der jeweiligen Schicht von Aktinfilamenten und den Detailansichten zu wechseln, um Modellanpassungen vorzunehmen.
Zu den kritischen Schritten innerhalb des Protokolls gehörten die Drehung der Karte nach der visuellen Inspektion, die Erstellung und Platzierung des Modells in der Dichtekarte sowie die Aufteilung des Filamentmodells in kleinere Segmente. Die Atomposition der Segmente könnte dann räumlich angepasst werden, um sie an die Dichtekarte anzupassen, und/oder farbcodiert werden, um Lücken zu erkennen.
Dieser Ansatz der Aktin-Modellbildung kann auch modifiziert werden, indem ein Satz von "Atomen" (d.h. die Kugeln des Kugel-und-Stick-Modells) in Filamentdichten platziert wird, indem eine Querschnittsansicht einer 10-30 Schichten/9,47-28,4 nm gemittelten Dichteplatte verwendet wird, die dann durch Bindungen (d.h. die Stäbe des Kugel-und-Stab-Modells) verbunden werden können. Wir haben diesen Ansatz, der eine Modifikation des hier ausführlich beschriebenen Protokolls darstellt, für die volumetrische Modellbildung im konischen Bereich der Haarzellstereozilien23 verwendet. Darüber hinaus eignet sich unser Ansatz der volumetrischen Modellbildung, wie wir hier beschrieben haben, auch gut für die Segmentierung und Modellbildung von Membranen.
Während die Erstellung volumetrischer Modelle auf jede Dichtekarte angewendet werden kann, die filamentöse Merkmale zeigt, ist die hier beschriebene Technik am effizientesten, wenn wir ein Array von Filamenten mit regelmäßigen Abständen haben, für die eine globale Anpassung eines volumetrischen Modells erhalten werden kann. Es hängt auch davon ab, dass die filamentösen Merkmale ihre Richtungsabhängigkeit allmählich ändern. Wenn es zu plötzlichen Knicken und scharfen Kurven in den filamentösen Strukturen kommt, ist unser Ansatz möglicherweise nicht besonders hilfreich für die Segmentierung.
In der Zwischenzeit haben unsere Mitarbeiter einen automatisierten Ansatz für das automatisierte Filament-Tracing entwickelt, der einem ähnlichen Konzept folgt, das hier für die manuelle Segmentierung verwendet wird30,31. Für die Zukunft könnte der beste Ansatz eine Mischung aus manueller Identifizierung und Platzierung eines anfänglichen spärlichen Modells (auch nur ein paar Kugeln) in der Dichte als Ausgangspunkt sein und dann einen Such- und Anpassungsalgorithmus die Verfolgung der Filamente abschließen lassen.
Vereinfachte volumetrische Modelle reduzieren die Komplexität eines Systems und ermöglichen es, bestimmte Muster, wie z. B. die Biegung des Aktinfilaments in der Nähe der Spitze, besser zu erkennen. Das volumetrische Modell kann auch als "Anker" verwendet werden, um eine Dichtezone um das gewählte Verankerungskugel-und-Stab-Modell darzustellen, was die Detektion und Visualisierung von Vernetzerdichten zwischen benachbarten Aktinfilamenten ermöglicht. Durch die Möglichkeit, einzelne Filamente auszuwählen und entsprechende Radien als Zone einzustellen, in der die Dichte wieder angezeigt wird, kann die überwältigende Komplexität der Szenerie auf ein überschaubares Maß reduziert werden.
Ein Vorteil dieses volumetrischen Modellbildungsansatzes mit globaler Anpassung, gefolgt von lokalen Anpassungen, bestand darin, dass wir Regionen identifizieren konnten, in denen Aktinfilamente unterbrochen zu sein schienen, und signifikante Lücken in den Aktinfilamenten durch das Fehlen einer Kartendichte angezeigt wurden. Da wir ein volumetrisches Kugel-und-Stick-Modell platziert hatten, konnten wir eine Routine im UCSF Chimera-Softwarepaket verwenden, die jede Modellballposition entsprechend dem Dichtewert der Karte an dieser Position farbcodiert. Dieser Ansatz ermöglichte eine schnelle Detektion und Visualisierung von Aktinfilamentlücken im Aktinbündel, was ein biologisch signifikantes Merkmal ist, das wir in unserem Kryo-Elektronen-Tomogramm gefunden haben und das mit herkömmlichen Segmentierungsansätzen sehr schwer zu erkennen und zu visualisieren gewesen wäre. Ein weiterer Vorteil unseres volumetrischen Modells besteht darin, dass volumetrische Eigenschaften wie Längen und Entfernungen leicht ermittelt werden können, was es ermöglicht, tatsächliche Zahlen und damit eine statistische Analyse durchzuführen.
Zusammenfassend lässt sich sagen, dass die interaktive manuelle Platzierung von Modellpunkten, möglicherweise ergänzt durch nachträgliche automatisierte lokale Anpassung und Filamentverfolgung, ein vielversprechender Ansatz für die Visualisierung und quantitative Analyse von elektronentomographischen subzellulären Volumina ist. Dies liegt daran, dass es die Leistungsfähigkeit des menschlichen Gehirns für die Mustererkennung und die Leistungsfähigkeit der Informatik für die Modelloptimierung nutzt.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen oder sonstige Interessenkonflikte haben.
Danksagungen
Wir danken Dr. Peter Barr-Gillespie und seinem Team für ihre Rolle bei der Probenvorbereitung und den ehemaligen Mitgliedern des Auer-Labors und des Dr. Dorit Hanein-Labors für ihre Rolle bei der tomographischen Datenerfassung. Wir möchten uns auch bei Tom Goddard von UCSF Resource for Biocomputing, Visualization, and Informatics (RBVI) für die Bereitstellung verschiedener UCSF Chimera-Skripte bedanken.
Materialien
| Name | Company | Catalog Number | Comments |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/download.html | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=win64/chimera-1.16-win64.exe | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=mac64/chimera-1.16-mac64.dmg | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=linux_x86_64/chimera-1.16-linux_x86_64.bin | |
| Excel | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Falcon II | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/accessories-em/falcon-detector.html | |
| IMOD | University of Colorado | Version 4.11.1 https://bio3d.colorado.edu/imod/download.html | |
| PC Desktop | Intel | Windows 10, ver. 22H2 | |
| PC Laptop | Gigabyte | Windows 10, ver. 22H2 | |
| Powerpoint | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Titan Krios Electron Microscope | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/transmission-electron-microscopes/krios-g4-cryo-tem.html | |
| Word | Microsoft | Version 2211 https://www.office.com/?auth=1 |
Referenzen
- Downing, K. H., Sui, H., Auer, M. Electron tomography: A 3D view of the subcellular world. Analytical Chemistry. 79 (21), 7949-7957 (2007).
- Koning, R. I., Koster, A. J. Cellular nanoimaging by cryo electron tomography. Methods in Molecular Biology. 950, 227-251 (2013).
- Asano, S., Engel, B. D., Baumeister, W. In situ cryo-electron tomography: A post-reductionist approach to structural biology. Journal of Molecular Biology. 428 (2), 332-343 (2016).
- Serwas, D., Davies, K. M. Getting started with in situ cryo-electron tomography. Methods in Molecular Biology. 2215, 3-23 (2021).
- McDonald, K. L., Auer, M. High-pressure freezing, cellular tomography, and structural cell biology. Biotechniques. 41 (2), 137-143 (2006).
- Narasimha, R., et al. Evaluation of denoising algorithms for biological electron tomography. Journal of Structural Biology. 164 (1), 7-17 (2008).
- Frangakis, A. S. It's noisy out there! A review of denoising techniques in cryo-electron tomography. Journal of Structural Biology. 213 (4), 107804 (2021).
- Frangakis, A. S., Hegerl, R. Noise reduction in electron tomographic reconstructions using nonlinear anisotropic diffusion. Journal of Structural Biology. 135 (3), 239-250 (2001).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. Journal of Structural Biology. 144 (1-2), 114-122 (2003).
- vander Heide, P., Xu, X. -. P., Marsh, B. J., Hanein, D., Volkmann, N. Efficient automatic noise reduction of electron tomographic reconstructions based on iterative median filtering. Journal of Structural Biology. 158 (2), 196-204 (2007).
- Volkmann, N. Methods for segmentation and interpretation of electron tomographic reconstructions. Methods in Enzymology. 483, 31-46 (2010).
- Böhm, J., et al. Toward detecting and identifying macromolecules in a cellular context: template matching applied to electron tomograms. Proceedings of the National Academy of Sciences. 97 (26), 14245-14250 (2000).
- Frangakis, A. S., et al. Identification of macromolecular complexes in cryoelectron tomograms of phantom cells. Proceedings of the National Academy of Sciences. 99 (22), 14153-14158 (2002).
- Lebbink, M. N., et al. Template matching as a tool for annotation of tomograms of stained biological structures. Journal of Structural Biology. 158 (3), 327-335 (2007).
- Rigort, A., et al. Automated segmentation of electron tomograms for a quantitative description of actin filament networks. Journal of Structural Biology. 177 (1), 135-144 (2012).
- Volkmann, N. A novel three-dimensional variant of the watershed transform for segmentation of electron density maps. Journal of Structural Biology. 138 (1-2), 123-129 (2002).
- Bajaj, C., Yu, Z., Auer, M. Volumetric feature extraction and visualization of tomographic molecular imaging. Journal of Structural Biology. 144 (1-2), 132-143 (2003).
- Yu, Z., Frangakis, A. S. Classification of electron sub-tomograms with neural networks and its application to template-matching. Journal of Structural Biology. 174 (3), 494-504 (2011).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Tilney, L. G., Derosier, D. J., Mulroy, M. J. The organization of actin filaments in the stereocilia of cochlear hair cells. The Journal of Cell Biology. 86 (1), 244-259 (1980).
- Tilney, L. G., Tilney, M. S., DeRosier, D. J. Actin filaments, stereocilia, and hair cells: how cells count and measure. Annual Review of Cell Biology. 8, 257-274 (1992).
- Metlagel, Z., et al. Electron cryo-tomography of vestibular hair-cell stereocilia. Journal of Structural Biology. 206 (2), 149-155 (2019).
- Song, J., et al. A cryo-tomography-based volumetric model of the actin core of mouse vestibular hair cell stereocilia lacking plastin 1. Journal of Structural Biology. 210 (1), 107461 (2020).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Agulleiro, J. I., Fernandez, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27 (4), 582-583 (2011).
- Fernandez, J. J. Computational methods for electron tomography. Micron. 43 (10), 1010-1030 (2012).
- Chen, H., Clyborne, W. K., Sedat, J. W., Agard, D. A. Priism: an integrated system for display and analysis of 3-D microscope images. Biomedical Image Processing and Three-Dimensional Microscopy. 1660, 784-790 (1992).
- Pettersen, E. F., et al. UCSF Chimera-a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25 (13), 1605-1612 (2004).
- Goddard, T. D., Huang, C. C., Ferrin, T. E. Visualizing density maps with UCSF Chimera. Journal of Structural Biology. 157 (1), 281-287 (2007).
- Sazzed, S., et al. Tracing actin filament bundles in three-dimensional electron tomography density maps of hair cell stereocilia. Molecules. 23 (4), 882 (2018).
- Sazzed, S., Scheible, P., He, P., Wriggers, J. Spaghetti tracer: A framework for tracing semiregular filamentous densities in 3D tomograms. Biomolecules. 12 (8), 1022 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten