Method Article
Modelli volumetrici semplificati come strategia efficace per la segmentazione delle reti di actina nei tomogrammi crioelettronici
In questo articolo
Riepilogo
Qui presentiamo un protocollo per posizionare modelli volumetrici semplificati in volumi 3D rumorosi e complessi e tomografici. Ciò consente la segmentazione rapida delle densità dei filamenti di actina, il rilevamento della flessione sistematica dei filamenti e delle lacune nei filamenti dei fasci di capelli, nonché una comoda quantificazione delle proprietà volumetriche del modello, come le distanze.
Abstract
Metodi efficienti per l'estrazione di caratteristiche di interesse rimangono una delle maggiori sfide per l'interpretazione dei tomogrammi crioelettronici. Sono stati proposti vari approcci automatizzati, molti dei quali funzionano bene per set di dati ad alto contrasto in cui le caratteristiche di interesse possono essere facilmente rilevate e sono chiaramente separate l'una dall'altra. I nostri set di dati tomografici crioelettronici per stereociglia dell'orecchio interno sono caratterizzati da una fitta serie di filamenti di actina impacchettati esagonalmente che sono spesso collegati in modo incrociato. Queste caratteristiche rendono la segmentazione automatizzata molto impegnativa, ulteriormente aggravata dall'ambiente ad alto rumore dei tomogrammi crioelettronici e dall'elevata complessità delle caratteristiche densamente impacchettate. Utilizzando le conoscenze preliminari sull'organizzazione del fascio di actina, abbiamo posizionato strati di un modello di actina a sfera e bastone altamente semplificato per ottenere prima un adattamento globale alla mappa di densità, seguito da aggiustamenti regionali e locali del modello. Dimostriamo che la costruzione di modelli volumetrici non solo ci permette di affrontare l'elevata complessità, ma fornisce anche misurazioni e statistiche precise sul fascio di actina. I modelli volumetrici fungono anche da punti di ancoraggio per la segmentazione locale, come nel caso dei connettori incrociati actina-actina. La costruzione di modelli volumetrici, in particolare se ulteriormente potenziata da approcci di fitting automatizzato basati su computer, può essere una valida alternativa quando gli approcci convenzionali di segmentazione automatizzata non hanno successo.
Introduzione
La tomografia crioelettronica consente di visualizzare interi organelli o parti di cellule e tessuti a risoluzioni nanometriche nel loro stato quasi nativo 1,2,3 utilizzando il congelamento a tuffo4 o il congelamento ad alta pressione di vetrificazione ultrarapida5. Poiché solo una dose limitata di elettroni può essere tollerata dal campione crioconservato, non colorato e congelato-idratato, i dati tomografici 3D sono molto rumorosi. Questo rumore spesso può essere significativamente ridotto da una varietà di algoritmi di filtraggio del rumore 6,7, tra cui la diffusione anisotropa non lineare8, il filtraggio bilaterale9 e il filtraggio mediano ricorsivo10.
Inoltre, le limitazioni di inclinazione del tavolino del microscopio, che risultano in un cuneo mancante di informazioni, e il fatto che lo spessore del campione aumenta ad alti angoli di inclinazione, portano a ricostruzioni 3D con risoluzione anisotropa. Ciò significa che la densità è spalmata nella terza dimensione a causa della minore risoluzione nella direzione Z. Di conseguenza, la forma delle macromolecole appare distorta (cioè meno ben definita e allungata nella terza dimensione).
Tra le maggiori sfide nell'interpretazione dei dati tomografici c'è l'estrazione automatizzata delle caratteristiche rilevanti, nota anche come segmentazione11. Con sufficienti caratteristiche di forma uniche e bassa rumorosità, le macchine macromolecolari in volumi 3D complessi possono essere identificate mediante la corrispondenza di modelli 12,13,14; Tuttavia, il successo della corrispondenza dei modelli dipende dalla risoluzione del tomogramma, da un modello di ricerca adatto e dalle caratteristiche di dimensioni e forma dei volumi delle funzioni. Se le caratteristiche di interesse sono sufficientemente distanziate tra loro e i motivi ripetuti (come grandi macchine macromolecolari) possono essere facilmente identificati, i sottovolumi del tomogramma possono essere combinati per aumentare il rapporto segnale/rumore e per mediare le distorsioni della forma delle singole particelle. È stata riportata la segmentazione automatizzata di una rete di filamenti di actina in tomogrammi elettronici del bordo sottile di cellule di Dictyostelium discoideum congelate-idratate mediante corrispondenza del modello15.
Tuttavia, se le caratteristiche di interesse sono ravvicinate, l'anisotropia della risoluzione dei dati può portare a una sbavatura delle densità della mappa nella direzione Z (lungo la direzione del fascio di elettroni), con conseguente apparente fusione dell'inviluppo di densità di macchine macromolecolari ravvicinate o complessi supramolecolari. In questi casi, gli approcci automatizzati per la segmentazione, come il bacino idrografico16, la segmentazione dei confini17 o una varietà di approcci di classificazione basati sull'apprendimento automatico18,19, potrebbero non essere in grado di riconoscere le caratteristiche di interesse o stabilire un confine corretto attorno a un oggetto di interesse. Spesso, ci si ritrova con pochi pezzi molto grandi o con un volume fortemente sovrasegmentato, dove è necessario molto sforzo per unire molti pezzi piccoli fino a quando la caratteristica di interesse non viene percepita come completa. Tale cura manuale dei risultati della segmentazione può essere molto laboriosa e può anche fallire del tutto quando la struttura di interesse è una matrice di filamenti ravvicinati che sono interconnessi tramite brevi linker. In questa gigantesca rete di strutture filamentose, può essere difficile orientarsi. Questo perché, a causa dell'anisotropia della risoluzione, le densità sembrano fondersi l'una con l'altra, presentando una sfida formidabile sia per gli approcci di segmentazione manuale automatizzati che per quelli interattivi. Di conseguenza, si può facilmente "saltare" tra i filamenti quando si ispezionano visivamente solo piccole regioni.
Fortunatamente, nel caso del fascio di actina nelle stereociglia delle cellule ciliate dell'orecchio interno, abbiamo conoscenze sull'organizzazione complessiva del fascio di actina e sulla direzionalità dei filamenti di actina20,21. Il fascio di actina è costituito da centinaia di filamenti di actina esagonali e densamente impacchettati di 6-8 nm di diametro, che sono distanziati di circa 12-13 nm l'uno dall'altro22.
Questo ci ha permesso di adottare un approccio piuttosto diverso alla segmentazione, basato su modelli semplificati a sfera e bastone per rappresentare i filamenti di actina. La strategia prevedeva l'inserimento simultaneo di una serie regolare idealizzata di modelli di filamenti in lastre delle mappe di densità della tomografia crioelettronica per costruire un modello 3D del fascio di actina strato per strato. Ci siamo assicurati che il modello si adattasse complessivamente alla mappa di densità prima di apportare modifiche locali a singoli modelli di filamenti o gruppi di modelli di filamenti in modo che corrispondessero il più possibile alla mappa di densità. Mediante la codifica automatica a colori del valore di densità della mappa nella posizione del modello di filamento, siamo stati in grado di rilevare facilmente le lacune apparenti nel fascio di actina. I modelli volumetrici consentono un'analisi quantitativa delle proprietà volumetriche, come le distanze tra i filamenti di actina, e portano anche a una visualizzazione semplificata dell'organizzazione complessiva della rete filamentosa 3D.
Inoltre, i modelli possono anche fungere da strutture di ancoraggio per la segmentazione di caratteristiche aggiuntive, come i linker actina-actina, in quanto è possibile selezionare (porzioni di) singoli modelli di filamenti, attorno ai quali è possibile generare zone di densità della mappa del raggio appropriate per l'ispezione e un'ulteriore segmentazione.
Riteniamo che il nostro approccio di segmentazione basato su modelli volumetrici sia particolarmente utile per reti di grandi strutture filamentose che possono contenere lacune e connessioni incrociate tra filamenti. Gli algoritmi di segmentazione tendono a operare localmente, mentre il cervello umano prende in considerazione aree più grandi e quindi è superiore ai computer quando si tratta di riconoscere le strutture dei filamenti, anche in un ambiente complesso e ad alto rumore.
Protocollo
Il protocollo segue le linee guida del comitato etico per la ricerca umana della Southeast University.
1. Fonte di dati di tomografia crioelettronica per la costruzione di modelli volumetrici
NOTA: Le ricostruzioni tomografiche crioelettroniche delle stereociglia utilizzate per la costruzione di un modello volumetrico sono state precedentemente pubblicate22,23, e sono state ottenute come descritto da Metlagel et al.22.
Gli script Python UCSF Chimera per la modellazione delle stereociglia sono forniti in File supplementare 1, File di codifica supplementare 1, File di codifica supplementare 2, File di codifica supplementare 3, File di codifica supplementare 4 e File di codifica supplementare 5.
- In breve, tamponare le stereociglia dalla superficie apicale degli epiteli sensoriali dell'utricolo di topo sulla pellicola di supporto in carbonio lacey di una griglia per microscopio elettronico (EM). Quindi, eseguire la vetrificazione ultrarapida con congelamento a tuffo e la raccolta di dati tomografici crioelettronici su un singolo asse su una microscopia elettronica a trasmissione criogenica (cryo-TEM), operata a 300 kV con una sfocatura nominale di 3,5-4,5 μm, utilizzando una fotocamera di tipo CMOS in modalità di integrazione, con una dimensione dei pixel da 0,47 a 0,59 nm (vedi Tabella dei materiali).
- Una dose tipica per la raccolta di dati su un singolo asse è tipicamente di 80-100 elettroni/Å2. Ricostruire i volumi 3D del tomogramma utilizzando il pacchetto software IMOD24, sia mediante retroproiezione pesata che con il metodo SIRT 25,26.
- Ridurre il rumore dei tomogrammi utilizzando il filtraggio mediano o bilaterale ricorsivo in Priism27 o l'opzione di diffusione anisotropa non lineare in IMOD.
NOTA: Il pacchetto software chiave utilizzato per la visualizzazione interattiva, la costruzione di modelli volumetrici e l'analisi quantitativa è stato il software UCSF Chimera28,29. Tutti i pacchetti software utilizzati sono elencati nella Tabella dei materiali.
2. Preparazione dei dati di tomografia crioelettronica per la costruzione di modelli volumetrici
- Rotazione del tomogramma
NOTA: Lo scopo di questo passaggio è quello di trovare gli "angoli ottimali" in cui il nucleo di actina è allineato in due dei tre assi (x e z), in modo che il posizionamento dei modelli in 3D possa essere fatto con un solo asse di cui preoccuparsi. I seguenti passaggi vengono eseguiti all'interno del pacchetto software IMOD.- Apri 3dmod (IMOD) e apri il file della pila di immagini 3D in formato .mrc. Assicurati che lo stack 3D sia in modalità scala di grigi a 16 bit e che visualizzi le dimensioni X, Y e Z corrette nell'intestazione del file immagine. Se necessario, apportare correzioni utilizzando il comando alterheader : alterheader -d (x_pixelspacing),(y_pixelspacing),(z_pixelspacing) inputfilename.mrc nel prompt dei comandi. Se la pila 3D è in formato file TIFF, utilizzare il comando tif2mrc inputfilename.tif outputfilename.mrc nel prompt dei comandi per creare un file .mrc.
- Ispezionare visivamente il tomogramma e aprire il "filtro dei dati" (premendo la barra rovesciata "\") per trovare i migliori angoli di rotazione nelle dimensioni X, Y e Z per l'allineamento del piano del filamento di actina con il piano Z. Manipola la rotazione X, la rotazione Y, la barra di rotazione Z per scoprire l'angolo ottimale con cui i filamenti di actina sono allineati sui piani X e Z. Si noti che la modifica dello spessore medio per la visualizzazione del tomogramma (Spessore: Img) può aiutare a trovare una quantità ideale di media della densità nel piano Z e quindi un aumento del contrasto. Ingrandisci e rimpicciolisci il tomogramma utilizzando rispettivamente i tasti + e - sulla tastiera.
- Una volta identificati gli angoli ideali per la rotazione, ruotare il tomogramma digitando il comando IMOD rotatevol -a (angoli di rotazione Z),(angoli di rotazione Y),(angoli di rotazione X) -s (larghezza x),(altezza y),(profondità z) inputfilename.mrc outputfilename.mrc nel prompt dei comandi. Assicurati di lasciare alla mappa molto spazio per ospitare la rotazione per evitare di tagliare parti della mappa durante la rotazione.
NOTA: Per ulteriori informazioni su come utilizzare il comando rotatevol, utilizzare l'opzione dal menu di aiuto o visitare il seguente URL (https://bio3d.colorado.edu/imod/doc/man/rotatevol.html). Questo comando (rotatevol) non modifica la mappa attualmente visualizzata nella finestra IMOD, ma crea un nuovo file mappa con una mappa di densità che viene ruotata. - Utilizzando la finestra del filtro dei dati, identificare le coordinate X, Y e Z di due angoli opposti di una mappa per identificare la regione di ritaglio contenente lo stereocilio utilizzando il comando trimvol .
- Utilizzare il comando IMOD Trimvol per ritagliare la mappa alle coordinate ottenute in precedenza, digitando trimvol -x (x-coordiante 1),( x-coordiante 2) -y (y-coordiante 1),( y-coordiante 2) -z (z-coordiante 1),( z-coordiante 2) inputfilename.mrc outputfilename.mrc nel prompt dei comandi. Ora, la mappa è molto più piccola, e quindi più facile e veloce da lavorare da questo punto in poi.
NOTA: Per ulteriori informazioni su come utilizzare il comando trimvol, utilizzare l'opzione dal menu di aiuto o visitare il seguente URL (https://bio3d.colorado.edu/imod/doc/man/trimvol.html). Questi passaggi creano un file .mrc che è stato ruotato e tagliato.
- Filtraggio dei tomogrammi
NOTA: Questo passaggio utilizza un filtro di diffusione anisotropa non lineare (NAD) fornito da IMOD per la riduzione del rumore. Abbiamo usato come valore iniziale quelli raccomandati nella pagina di aiuto IMOD per il filtraggio della diffusione anisotropa non lineare, come elencato nel seguente URL (https://bio3d.colorado.edu/imod/doc/NADexample.html), e sono stati usati come valori iniziali.- Digita etomo nel prompt dei comandi per avviare l'interfaccia utente grafica etomo di IMOD.
- Selezionare l'opzione Diffusione anisotropa non lineare dal menu etomo e selezionare il file mappa (Scegli un volume).
- Per trovare un valore K e il numero di iterazioni che forniscono i migliori risultati di filtraggio, come giudicato dall'ispezione visiva, applicare il filtro NAD a un volume di test più piccolo per estrarre il volume di test del filtro NAD specificato. Fare clic sullo strumento Elastico nella parte superiore dell'interfaccia 3dmod, fare clic con il pulsante sinistro del mouse per selezionare il volume di test e fare clic sui pulsanti Hi e Lo per indicare che la sezione Z deve essere il limite del volume di test (Ottieni intervallo volume di test da 3dmod). Estrarre il volume di test facendo clic su Estrai volume di test.
- Filtrare il volume di test utilizzando vari valori K (Trova un valore K per il volume di test > Elenco di valori K), ad esempio 0,1, 1,5, 10, 15, 25, 30, 50 e 75. Eseguire il filtraggio NAD con ciascuno dei valori K specificati (Esecuzione con valori K diversi > visualizzare i risultati del test con valori K diversi). Utilizzare la finestra dello slicer per valutare le prestazioni del filtro NAD e selezionare un valore K che verrà utilizzato per filtrare l'intero volume.
- Utilizzare il valore K identificato nel passaggio 2.2.4 e testare un numero diverso di iterazioni, ad esempio 2, 5, 8, 11, 15 e 21 (Trovare un numero di iterazione per il volume di test > Elenco delle iterazioni). Valutare le prestazioni del filtro NAD per le diverse iterazioni facendo clic su Visualizza risultati diversi del test di iterazione o utilizzando uno strumento di sezionamento. Scegliere un valore per un numero diverso di iterazioni.
- Utilizzare il valore K identificato e il numero di iterazioni (Filtra volume completo > valore K e Iterazioni) e filtra l'intero volume (Filtra volume completo). Un volume appena filtrato apparirà nella stessa directory in cui si trova il volume completo caricato con un'estensione di file .nad collegata al file .mrc.
NOTA: In questi passaggi viene creato un file .mrc che è stato filtrato con un filtro NAD e visualizza un rumore inferiore e quindi un rapporto segnale/rumore migliorato.
3. Modellismo volumetrico
- Preparazione al posizionamento del modello 3D
NOTA: Lo scopo di questo passaggio è preparare e analizzare la mappa per il posizionamento di modelli volumetrici 3D nella mappa di densità ruotata e denoised utilizzando il programma UCSF Chimera.- Utilizzando la finestra dello slicer, ottenere una vista in sezione trasversale (lastra di 30 fette/28,4 nm) per identificare le coordinate Z del centro della lastra di densità, in cui deve essere posizionato il modello del filamento di actina. Fare clic sul centro della densità del filamento e annotare le coordinate Z nella finestra principale di IMOD.
- Prendere nota delle coordinate identificate per il futuro posizionamento del modello in UCSF Chimera.
NOTA: Le coordinate Z annotate da questo passaggio vengono utilizzate durante il passaggio 4 per trovare e posizionare facilmente i modelli di filamenti di actina prefabbricati alle altezze Z corrette, rendendo il posizionamento del modello più veloce.
- Posizionamento del modello 3D
NOTA: Lo scopo di questo passaggio è quello di posizionare un modello volumetrico 3D nella mappa di densità preparata utilizzando UCSF Chimera.- In UCSF Chimera, aprire la mappa filtrata e ruotata (File > Apri)
- Verificare se i parametri della mappa sono impostati correttamente selezionando Funzioni > Piani e Caratteristiche > Coordinate nel Visualizzatore volumi per aprire l'interfaccia delle funzioni e delle coordinate. Controllare le dimensioni del voxel per verificare se è impostata la spaziatura corretta dei voxel utilizzando il comando header nel prompt dei comandi (in IMOD). In caso contrario, correggere l'interfaccia Voxel Size con la spaziatura voxel corretta. Centrare la mappa per impostare la telecamera al centro della finestra di visualizzazione (Indice di origine > Centro).
- Aprire la finestra Controllo telecamera (Strumenti > Controllo visualizzazione > Telecamera). Quindi, impostare la visualizzazione della telecamera sulla vista ortogonale (proiezione > ortogonale)
- Carica il modello di script python predefinito (File > Open > ActinFilamentPlane.py) e apri tanti script del piano del filamento di actina quanti sono i piani del filamento di actina nella mappa.
- Riposiziona i modelli caricati attivando la modalità Mouse di movimento (Strumenti > Movimento > Modalità mouse di movimento). Abilita l'opzione di movimento del mouse (Imposta la modalità di movimento del mouse > Sposta selezione). Utilizzare Ctrl + Trascinamento per selezionare più modelli presenti nella sessione e fare clic con il pulsante destro del mouse (o qualsiasi altro tasto specificato in Preferenze > mouse) per spostare i modelli nella direzione desiderata.
NOTA: I modelli sono stati creati con palle e bastoncini con un certo spessore (raggio di ActinFilamentPlane.py script) e con determinate distanze (Lattice_Spacing di ActinFilamentPlane.py script). - Per visualizzare solo il modello a cui si è interessati, aprire il Pannello Modello (Strumenti > Controlli generali > Pannello Modello). Fare clic sulle caselle di controllo sotto S (Mostra) per abilitare o disabilitare la visibilità di un determinato modello.
- Attivare il pannello della riga di comando (Strumenti > Controlli generali > Riga di comando).
- Dopo aver posizionato ciascun modello di piano di filamento di actina alla corretta altezza Z, eliminare eventuali filamenti di actina in eccesso in ciascun modello di piano di filamento di actina selezionando la parte in eccesso dei modelli e digitando del sel (Elimina selezionati) nella riga di comando nella parte inferiore della finestra di visualizzazione principale di UCSF Chimera.
- Visualizza solo un piano di filamento di actina alla volta, andando alla finestra del pannello del modello e selezionando e nascondendo tutti gli altri modelli di filamenti di actina (Ctrl + clic > Nascondi).
- Per visualizzare le porzioni della densità della mappa che si trovano nelle immediate vicinanze del modello del singolo filamento di actina di interesse, selezionare il modello (Pannello Modello > selezionare) e aprire il pannello di controllo della zona in Visualizzatore volumi (Visualizzatore volume > Funzioni > Zona). Impostare il raggio della zona su 100 Å, quindi fare clic su Zona per suddividere la mappa in zone in un raggio di 100 Å (= 10 nm) dal modello selezionato. Si noti che la dimensione in Chimera è impostata in Angstrøm (1 Å = 0,1 nm).
- Se necessario, regolare il modello per adattarlo alla mappa di densità attivando la modalità Mouse di movimento. Selezionare più modelli che non si trovano nella posizione corretta per spostare i modelli da posizionare correttamente all'interno della mappa di densità suddivisa in zone.
- Per superare un bug di programmazione in ActinFilamentPlane.py, eseguire lo script FixingMarkerID.py , che corregge il MarkerID mancante degli "atomi" (palline nel modello a sfera e bastone). Selezionare l'intero modello e digitare runscript FixingMarkerID.py nella riga di comando (lo script deve essere specificato dalla directory corretta, ad esempio C: \ directory \ Script.py).
- Se la mappa di densità indica che il filamento di actina è curvo, assicurati di dividere il filamento di actina dritto in più parti per adattarsi alla curvatura selezionando i modelli desiderati e digitando il numero dividelinks.py runscript. Ulteriori marcatori (specificati dal numero nel comando) vengono posizionati all'interno del modello di actina equidistanti l'uno dall'altro, creando "giunti" in cui il modello può essere piegato.
- Regola le giunzioni in modo che riflettano la curvatura spostando ulteriori "giunti" di marcatore lungo la mappa suddivisa in zone utilizzando la modalità Movimento del mouse. Se necessario, aggiungere altri marcatori al modello utilizzando lo script dividelinks.py .
- Per verificare che tutti i filamenti di actina siano posizionati correttamente all'interno delle densità di actina, utilizzare le barre di ritaglio facendo clic su Strumenti > Controllo visualizzazione > telecamera nel menu a discesa UCSF Chimera per aprire la finestra Controllo telecamera , quindi selezionare Vista laterale per visualizzare le due barre di ritaglio (piani di ritaglio vicino e lontano) della telecamera. Trascinate due barre gialle in una fessura stretta facendo clic con il pulsante sinistro del mouse e trascinate la parte di ritaglio utilizzando il pulsante centrale del mouse per esaminare i modelli un po' alla volta.
NOTA: Il modello del nucleo di actina stereocilio è ora completo.
- Segmentazione a membrana
NOTA: Lo scopo di questo passaggio è quello di creare un modello di superficie della membrana delle stereociglia.- La segmentazione delle superfici utilizza lo strumento Tracciatore volume (Strumenti > Dati volume > Tracciatore volume).
- Nella finestra Traccia volume , fare clic su Mouse e selezionare solo le opzioni Posiziona marcatori sui piani dati e Collega nuovo marcatore al marcatore selezionato. Ciò consente di posizionare con precisione i marcatori sullo stesso piano Z (quando viene visualizzato un singolo piano Z). Selezionare anche i marcatori posizionali utilizzando il pulsante centrale del mouse nella finestra Tracciatore volume .
- Creare un nuovo set di marker (File > Nuovo set di marker). Il set di marker 1 deve essere creato e selezionato nella finestra Tracciatore volume .
- Utilizzare il Visualizzatore volume per visualizzare i singoli piani Z nella finestra di visualizzazione principale (Asse alla Z > Uno).
- Utilizzando il pulsante centrale del mouse, tracciare la membrana da un'estremità all'altra della membrana visibile. Tutti i marcatori devono essere collegati da collegamenti da un'estremità all'altra.
- Deseleziona l'ultimo marcatore posizionato premendo Ctrl + clic sullo sfondo in cui non si trova un singolo elemento selezionabile. Ciò garantisce che il prossimo marcatore da posizionare sia collegato con collegamenti nel nuovo piano e non con una precedente traccia di membrana di un altro piano Z.
- Ripetere il tracciamento della membrana per ogni 10 nm nella stessa direzione del tracciamento da un'estremità all'altra della membrana visibile.
- Una volta segmentate molte bande parallele di membrane, accedere alla finestra Tracciatore volume (Volume Tracer ) e fare clic su Feature > Superfici (Surfaces). In questo modo viene abilitata l'interfaccia di superficie nella finestra Traccia volume . Fare clic su Crea accanto all'interfaccia Superfici per creare superfici tra le bande di segmentazione della membrana. Questo crea un sottile lembo del modello di superficie che riempie le tracce distanziate della membrana.
NOTA: La creazione del modello di superficie della membrana stereocilio è ora completa.
- Modellazione reticolante
NOTA: Lo scopo di questo passaggio è quello di modellare i reticolanti tra i filamenti di actina.- I reticolanti possono essere posizionati anche utilizzando lo strumento Volume Tracer .
- Nella finestra Traccia volume , fare clic con il pulsante sinistro del mouse per selezionare solo Posiziona marker su superfici e Collega nuovo marker al marker selezionato. Quindi, selezionare Posiziona marcatori utilizzando il pulsante centrale del mouse nella finestra Tracciatore volume .
- Visualizza le densità attorno al modello del singolo filamento di actina di interesse solo utilizzando la funzione di zona. Impostare il raggio della zona su 100 Å per visualizzare la mappa entro un raggio di 100 Å dal modello selezionato.
- Posiziona i marcatori sulle densità visibili dei reticolanti facendo clic con il pulsante centrale del mouse. Una volta posizionati due marcatori (e quindi viene modellato un singolo reticolante), assicurati di deselezionare il secondo marcatore per evitare che i collegamenti vengano continuamente collegati al modello di reticolante successivo.
- Ripetere i passaggi precedenti per ogni piano di filamento di actina e ripetere l'intero processo sopra in tutte e tre le direzioni principali in cui possono essere posizionati i reticolanti. Quando si suddividono in zone i piani dei filamenti di actina in direzione diagonale, selezionare manualmente i modelli di filamenti di actina individualmente.
NOTA: Per ciascuno dei modelli di reticolante di actina, possiamo registrare la posizione sui filamenti di actina a cui si collegano i presunti reticolanti. Queste informazioni potrebbero, in linea di principio, essere estratte per un'analisi di follow-up utilizzando la media del sottotomogramma, che esula dallo scopo di questo studio.
4. Analisi quantitativa del modello 3D
- Analisi delle lacune
NOTA: Lo scopo di questo passaggio è rilevare le lacune nei filamenti di actina.- Gli elementi selezionati possono essere analizzati. Specificate i loro parametri, come il loro colore, utilizzando lo strumento Ispeziona selezione (Azione > Ispeziona).
- Il colore dell'elemento selezionato può essere specificato selezionando qualsiasi elemento selezionabile in UCSF Chimera e modificando il colore accanto alla sezione Colore della finestra Ispeziona selezione .
- Specificare la dimensione dei marker e dei collegamenti selezionando i marker e i collegamenti in UCSF Chimera e modificando i valori accanto alla sezione del raggio della finestra Ispeziona selezione , dove le unità si trovano in Angstrøm.
- Esegui l'analisi delle lacune mediante il rilevamento automatico delle lacune di densità, utilizzando la funzione Valori alle posizioni degli atomi (Strumenti > Dati di volume > Valori alle posizioni degli atomi).
- Selezionare la mappa in cui verranno letti i valori selezionando la mappa desiderata accanto alla sezione Dati volume della finestra Valori alle posizioni degli atomi . Seleziona il modello in cui a tutti i marcatori verrà assegnato un parametro selezionando il modello desiderato accanto alla sezione Molecola .
- Con il pulsante Istogramma nella parte inferiore della finestra Valori alle Posizioni Atom , viene creato un istogramma in una nuova finestra chiamata Rendering/Seleziona per Attributo. Ctrl + clic sull'istogramma aggiungerà o rimuoverà i punti di taglio, per i quali è possibile selezionare colori diversi. Deselezionate Mantieni opaco per applicare un colore trasparente alla scena. I marcatori con una densità inferiore a una data soglia, che rappresenta gli spazi vuoti, non solo possono essere colorati ma possono anche essere selezionati, e quindi ci permettono di contare i marcatori numerici nella regione del vuoto. Per selezionare, fare clic su Seleziona >attributo > value_mapname.
- Determinazione delle distanze tra le actrine
- Per misurare le distanze tra le attine, posizionare i marcatori su un singolo piano XZ (indicato in UCSF Chimera come piano Y) utilizzando la finestra Tracciatore volume , facendo clic con il mouse e selezionando solo Posiziona marcatori sui piani dati e deselezionando Collega nuovo marcatore al marcatore selezionato. Ciò consente di posizionare con precisione i marcatori sullo stesso piano Y (quando viene visualizzato un singolo piano Z). Selezionare anche Posiziona marcatori utilizzando il pulsante centrale del mouse nella finestra Tracciatore volume e posizionare i marcatori su un piano Y di interesse per la misurazione della distanza interactina.
- Una volta che le posizioni di actina del singolo piano Y sono state contrassegnate con successo, selezionare tutti i marcatori e digitare il comando findclash #model-spec test self overlapCutoff -200 hbondAllowance 0 log true Linewidth 10 pbColor black nella riga di comando nella parte inferiore del piano del filamento di actina. In questo modo vengono rilevate tutte le interferenze tra i marcatori entro una distanza di 200 Å (overlapCutoff -200) e vengono create linee nere (pbColor black) con uno spessore di 10 Å (Larghezza linea 10) tra i marcatori.
- Poiché ci sono marcatori sovrapposti all'interno dell'intervallo dato di 200 Å, rimuovere le misurazioni ridondanti che non sono collegamenti solo tra i marcatori adiacenti più vicini. A tale scopo, utilizzare lo script RemoveCross.py fornito sul sito Web UCSF Chimera, che rimuove tutti i collegamenti sovrapposti che non sono il collegamento più breve con i suoi marcatori adiacenti.
- Per misurare la distanza dei legami rimasti, selezionare tutti gli pseudolegami (collegamenti creati da findclash) e utilizzare lo script python pblengths.py digitando runscript pblengths.py. Vedere i valori misurati dal Registro delle risposte (Strumenti > Utilità > Registro delle risposte). Quindi, copia i valori ottenuti dal registro delle risposte e trasferiscili su un foglio di calcolo o qualsiasi altro strumento adatto per creare un istogramma.
Risultati
Utilizzando la tomografia crioelettronica di singole stereociglia non colorate, congelate-idratate incorporate nel ghiaccio vitreo, abbiamo ottenuto mappe di densità del fascio di actina con i suoi filamenti di actina disposti esagonalmente, legati da proteine cross-connector23. La dimensione di un singolo voxel era di 0,947 nm. L'ispezione visiva nel programma di affettatura IMOD di un rendering volumetrico dell'intero tomogramma (400 sezioni/379 nm) ha indicato la presenza di strutture filamentose allineate con l'asse longitudinale delle stereociglia, come si può vedere nelle viste longitudinali (piano XY; Figura 1A-C, pannelli superiori) e nelle viste in sezione trasversale (piano XZ; Figura 1A-C, pannelli inferiori). Abbiamo notato che la vista di proiezione attraverso la rete di filamenti a 400 fette/379 nm diventava più chiara ruotando il volume ricostruito originale di -6° attorno all'asse X, -13,5° attorno all'asse Y e 5° attorno all'asse Z. A questo angolo, tutti i filamenti sono allineati uno sopra l'altro, e quindi il contrasto è massimo, come si può apprezzare dalle viste in sezione trasversale (Figura 1B). Poiché le singole sezioni trasversali non hanno un segnale sufficiente per distinguere in modo univoco i filamenti di actina, abbiamo scelto di eseguire il rendering volumetrico di una lastra di 30 fette/28,4 nm, che mostra chiaramente un modello esagonale nella vista della sezione trasversale. Le linee blu nella Figura 1C (pannelli superiori) indicano la posizione del centro delle corrispondenti lastre in sezione trasversale da 30 fette/28,4 nm nei pannelli inferiori.
Piccole deviazioni da questo angolo di visione ottimale, di appena ±2°, hanno ridotto significativamente l'ordine percepito della rete di filamenti di actina (Figura 1A, C), il che è un'indicazione di quanto sia facile perdersi nel volume 3D del tomogramma.
Per illustrare la sfida dell'utilizzo di approcci di segmentazione automatizzata, come la segmentazione dei bacini idrografici, abbiamo scelto un piccolo sottovolume (raffigurato come oro) per la segmentazione dei bacini idrografici, come implementato nel pacchetto software UCSF Chimera (Tools > Volume Data > Segger > Segment). La posizione del sottovolume rispetto all'intera mappa delle stereociglia è indicata dal piccolo riquadro nella Figura 1B.
La Figura 1D-F mostra il sottovolume scelto in diversi orientamenti, con la Figura 1D, E che mostra la direzione di visione longitudinale e la Figura 1F che mostra una direzione di visione in sezione trasversale. Le frecce sul lato sinistro della Figura 1D-F indicano la direzione dei filamenti di actina.
Le figure 1D-F (pannelli a destra) mostrano i risultati della segmentazione dei bacini idrografici. Il sottovolume viene codificato a colori in base all'identità dell'oggetto, con i colori assegnati in modo casuale ai diversi oggetti. Colori diversi indicano una diversa identità dell'oggetto, quindi diventa evidente dalla Figura 1D-F che le densità della mappa per i filamenti erano entrambe frammentate lungo l'asse del filamento, mentre lo stesso colore e quindi l'identità dell'oggetto erano dati alle densità della mappa che collegavano i filamenti vicini. In altre parole, l'algoritmo di segmentazione del bacino idrografico non era in grado di seguire la mappa di densità dei filamenti di actina per un periodo prolungato, e portava invece a collegare densità dai filamenti vicini. Sebbene sia possibile curare manualmente la selezione (ad esempio, eliminando o unendo gli oggetti), questo approccio è piuttosto laborioso e quindi dispendioso in termini di tempo.
Sebbene non sia assolutamente necessario per il funzionamento della nostra strategia di costruzione del modello volumetrico, è stato utile riorientare (ruotare) la mappa 3D in modo che l'asse della rete del filamento di actina fosse allineato con l'asse Y e i piani del modello del filamento di actina allineati con il piano X-Y del tomogramma. Ci riferiamo a questo orientamento come orientamento standard per la visualizzazione tomografica delle stereociglia.
Abbiamo quindi deciso di esplorare una strategia diversa per la segmentazione dell'immagine, sfruttando il fatto che i filamenti di actina mostravano un'organizzazione complessivamente regolare (impacchettamento esagonale), con spaziatura regolare e orientamento complessivo del fascio definito. La nostra strategia è stata quella di trovare un adattamento complessivo dei modelli di un fascio di actina, come una matrice di filamenti, seguito da aggiustamenti regionali e poi locali della posizione del modello per adattarsi alla mappa di densità sperimentale. Mettendo al primo posto un modello complessivo, possiamo superare le ambiguità della mappa locale e rilevare le tendenze regionali delle deviazioni del modello dalla sua organizzazione originale, come la flessione del filamento.
Per posizionare il modello, abbiamo visualizzato lastre della densità (10 fette/9,47 nm) nell'orientamento standard che corrispondeva a uno spessore di un singolo strato di filamenti di actina, a cui è stato montato uno strato di modelli di filamenti di actina diritti e regolarmente distanziati. Questa è ovviamente una semplificazione eccessiva dei filamenti di actina, ognuno dei quali è costituito da una matrice lineare di monomeri di actina con simmetria elicoidale. Le figure 2A-C mostrano tre strati rappresentativi a diverse altezze Z, con i bastoncelli di colore rosso che rappresentano i filamenti di actina. I pannelli superiori, raffiguranti ~30 fette/sezioni trasversali spesse 28,4 nm, mostrano a quale altezza Z è stato posizionato un singolo strato di modello di actina di 19 aste, mentre i pannelli inferiori mostrano un orientamento longitudinale (anche se mostrato in vista prospettica). La Figura 2D mostra il modello semplificato completo, sia nella vista prospettica in sezione trasversale (pannello superiore) che in quella longitudinale (pannello inferiore). L'orientamento della sezione trasversale ci ha permesso di posizionare i filamenti con buona sicurezza. In questo caso, la nostra mossa originale di riorientare l'intero volume in modo che coincida con gli assi maggiori dei tomogrammi si è rivelata utile, in quanto significava che anche l'orientamento del nostro modello nella nostra direzione di visualizzazione standard era parallelo agli assi principali. Tuttavia, a rigor di termini, il nostro approccio avrebbe funzionato anche senza il riorientamento del tomogramma, solo il posizionamento del modello alla densità sarebbe stato più impegnativo.
Dopo un'attenta ispezione delle singole lastre della mappa di densità, abbiamo notato che un modello di actina perfettamente dritto non si adattava alla mappa di densità osservata spostandosi dall'estremità prossimale all'estremità distale (cioè verso la punta) delle stereociglia (Figura 3A-C). Vicino alla punta delle stereociglia, la densità della mappa per i filamenti è stata spostata di oltre 13 nm (spaziatura actina-actina), che abbiamo potuto compensare regolando il modello mentre ci spostavamo dalla porzione prossimale a quella distale della mappa di densità delle stereociglia, introducendo così una curvatura graduale piccola ma distinguibile nel nostro modello di actina. La Figura 3D mostra una singola lastra della densità della mappa del filamento di actina, con un modello volumetrico adattato alla mappa di densità. Un confronto tra il modello dritto (rosso) e quello curvo (giallo) è mostrato nella Figura 3E. Questa curvatura si apprezza al meglio inclinando una lastra della mappa di densità con il modello posizionato di 80° attorno all'asse X, il che consente di avere una vista prospettica lungo la direzione dei filamenti di actina (Figura 3D, E).
La deviazione dei due modelli, con la posizione del modello di actina vicino alla punta spostata di circa la stessa distanza della spaziatura dei filamenti di actina, avrebbe potuto causare molta confusione se non avessimo proceduto nel modo in cui abbiamo proceduto. Questo posizionamento "globale" di uno strato del modello del filamento di actina, seguito da una regolazione "regionale", ci ha permesso di rilevare questa curvatura, che è appena percettibile nella vista longitudinale o in sezione trasversale. Tuttavia, la sovrapposizione dei due modelli, come mostrato nella Figura 3E, rivela la sottile differenza.
La ripetizione di questo approccio per più strati consente di ottenere un modello 3D completo (Figura 3F), limitato solo dall'incertezza dei dati nella parte superiore e inferiore delle stereociglia, quando viene visualizzato nell'orientamento della sezione trasversale (Figura 3G). Questa mancanza di densità è causata dalla mancanza di un cuneo nella raccolta dei dati tomografici (ad asse singolo) e dalla corrispondente anisotropia della risoluzione dei dati, e il suo effetto è indicato dall'assenza di una densità di mappa ben definita per le membrane delle stereociglia.
Una volta ottenuto un modello 3D, abbiamo codificato a colori ogni posizione del modello volumetrico in base al valore di densità della mappa in quella posizione. Le regioni del modello con una debole densità della mappa sottostante erano colorate di rosso, mentre le regioni del modello con un forte segnale di densità della mappa erano colorate di giallo (Figura 4A). Interpretiamo tali regioni di colore rosso, che possono estendersi fino a decine di nanometri, come lacune nelle strutture dei filamenti di actina che, a causa della loro estensione, non possono essere attribuite a variazioni di densità frequentemente incontrate nell'ambiente ad alto rumore di una mappa crio-EM. Il rumore tende a influenzare singoli voxel o piccoli gruppi di voxel, ma è improbabile che sia la fonte di volumi costituiti da centinaia di voxel, per i quali manca la densità del filamento. Invece, è probabile che tali lacune siano una caratteristica reale della rete di actina delle stereociglia e possano costituire siti di ricambio dell'actina. La Figura 4A ha due diversi valori di densità della mappa, mostrati in blu chiaro e blu scuro. Va notato esplicitamente che il nostro approccio alla costruzione di modelli volumetrici, combinato con una codifica a colori automatizzata del nostro modello in regioni di debole densità, è un modo rapido e conveniente per rilevare e visualizzare la distribuzione di tali lacune nel modello di filamento di actina, che altrimenti sarebbe stato molto difficile.
Come mostrato nella Figura 4B, porzioni del modello volumetrico in luoghi con una densità relativamente debole possono essere facilmente nascoste in base ai risultati ottenuti nella Figura 4A. Ciò si traduce in un modello più frammentato che può rappresentare in modo più realistico il modello di actina nelle stereociglia. L'alternativa di costruire piccoli tratti di filamenti di actina sarebbe stata molto laboriosa e potrebbe essere fallita del tutto, a causa dei problemi discussi durante la descrizione della Figura 1.
Inoltre, il modello volumetrico ci consente di modellare facilmente i connettori trasversali semplicemente posizionando una connessione (mostrata in rosso) tra le posizioni dei punti del modello del modello di filamento di actina su entrambi i lati della connessione trasversale (Figura 4C). Nel nostro approccio semplificato, non abbiamo bisogno di fare alcuna ipotesi sull'esatta identità di ciascuna proteina di cross-linking, il che richiederebbe una risoluzione più elevata e/o approcci di marcatura sofisticati. Invece, tutto ciò di cui abbiamo bisogno per determinare è se esiste una densità che collega i filamenti di actina adiacenti; Se c'è, possiamo posizionare una breve connessione da un filamento alla sua controparte adiacente. Nella Figura 4D, è mostrato un modello di cinque filamenti di actina con i loro connettori incrociati, che dà un'impressione della distribuzione dei connettori incrociati lungo l'asse del filamento di actina.
Un altro vantaggio della costruzione di un modello volumetrico del fascio di actina è che si può determinare rapidamente la spaziatura tra i filamenti di actina adiacenti (Figura 4E-H). La Figura 4E,F mostra una vista in sezione trasversale della mappa di densità senza e con un modello adattato al reticolo esagonale della densità della mappa, rispettivamente. La Figura 4G mostra il modello con le connessioni tra le sfere vicine più vicine. UCSF Chimera consente il calcolo automatico della distanza dei centri vicini più vicini, il cui risultato può quindi essere tracciato come una distribuzione della distanza (Figura 4H). La creazione di modelli per due set di dati aggiuntivi è illustrata nella Figura 1 e nella Figura 2 supplementare.
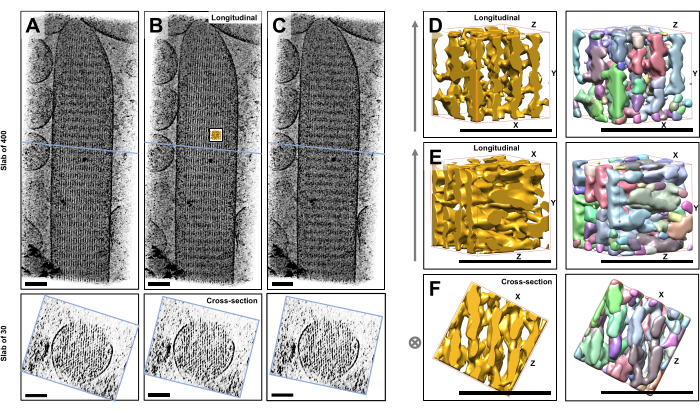
Figura 1: Le sfide incontrate dalla segmentazione dei bacini idrografici dei tomogrammi delle stereociglia delle cellule ciliate. (A-C) Proiezioni longitudinali (400 sezioni/379 nm) attraverso la mappa 3D tomografica nel piano XY (pannelli superiori) e viste in sezione trasversale (30 sezioni/28,4 nm) nel piano XZ (pannelli inferiori). (A) Mappa tomografica ruotata di -2° lungo l'asse Y rispetto al suo orientamento ottimale. (B) Mappa tomografica nell'orientamento ottimale, determinata dalla regolazione degli angoli di rotazione degli assi X, Y e Z (X = -6°, Y = -13,5° e Z= 5°) e rivelando un alto grado di ordine nella mappa di densità, suggerendo una rete di filamenti di actina altamente ordinata. (C) Mappa tomografica ruotata di +2° lungo l'asse Y rispetto al suo orientamento ottimale; la rotazione di soli 2° attorno all'asse Y rispetto alla direzione di visione ottimale compromette gravemente la regolarità percepita della mappa di densità. I pannelli inferiori rivelano la regolarità dell'array di filamenti di actina se visti nella direzione della sezione trasversale. La linea blu in A-C indica la posizione della soletta della sezione trasversale. (D-F) Un cubo di 50 nm x 50 nm x 50 nm visto da tre diverse direzioni prima (pannelli a sinistra) e dopo (pannelli a destra) la segmentazione del bacino idrografico. Si noti che la segmentazione dei bacini idrografici non riesce a rilevare la densità continua dei filamenti di actina, mentre i filamenti di actina adiacenti e la loro connessione incrociata condividono la stessa identità dell'oggetto, suggerendo che la segmentazione dei bacini idrografici non è un approccio adatto per la segmentazione del tomogramma. Nei pannelli D-F, la mappa di densità in Chimera è mostrata come stile di mappa "Superficie". (A-C) Barre di scala = 100 nm. (D-F) Barre di scala = 50 nm. Clicca qui per visualizzare una versione più grande di questa figura.
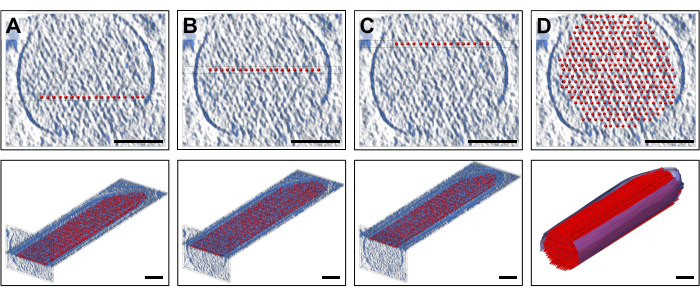
Figura 2: Costruzione di un modello volumetrico di filamento di actina ball-and-stick. (A-C) In alto: viste in sezione trasversale a 30 fette/28,4 nm della mappa di densità con un modello di filamento di actina semplificato a strato singolo posizionato a un'altezza Z diversa. In basso: una singola lastra da 10 slice/9,47 nm della mappa di densità con un modello di filamento di actina semplificato a strato singolo. (D) Modello completo di filamento di actina diritto in una vista in sezione trasversale a 30 fette/28,4 nm (in alto) e in prospettiva (in basso); Barre di scala = 100 nm. Clicca qui per visualizzare una versione più grande di questa figura.
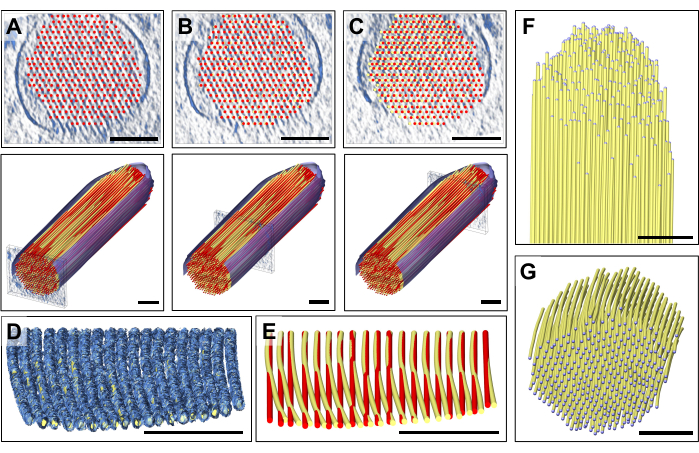
Figura 3: Regolazione del modello per correggere la flessione rilevata della densità del filamento di actina. (A-C) Un'attenta ispezione visiva del modello, sia in vista in sezione trasversale (pannelli superiori) che in vista longitudinale (pannelli in basso), ha rivelato un buon adattamento del modello con la mappa di densità all'estremità prossimale delle stereociglia. Tuttavia, man mano che ci si sposta verso l'estremità distale delle stereociglia, l'adattamento diventa sempre peggiore per tutti i modelli di filamenti di actina. Questo può essere corretto spostando le sfere del modello a sfera e bastone nella corretta posizione della mappa di densità, il che si traduce in un modello di filamento di actina leggermente curvo. Il modello diritto viene visualizzato in rosso, mentre il modello piegato corretto viene visualizzato in giallo. (D) Singola lastra della mappa di densità con il modello curvo montato su di essa, che rivela la curvatura della densità di actina verso la punta delle stereociglia. Il modello di actina è stato ruotato di 80° attorno all'asse X per mostrare meglio questa sottile ma significativa flessione dei filamenti di actina. (E) Confronto tra il modello di actina dritto e non corretto mostrato in rosso e il modello di actina curvo e corretto mostrato in giallo. Per motivi di chiarezza, viene mostrato solo uno strato del modello del filamento di actina. (F-G) Modello di fascio di actina con filamenti di actina curvi e corretti mostrati con orientamento longitudinale (F) e in sezione trasversale (G). La membrana segmentata è mostrata in blu. Nel pannello D, la mappa di densità in Chimera viene mostrata come stile di mappa "Mesh". Barre di scala = 100 nm. Clicca qui per visualizzare una versione più grande di questa figura.
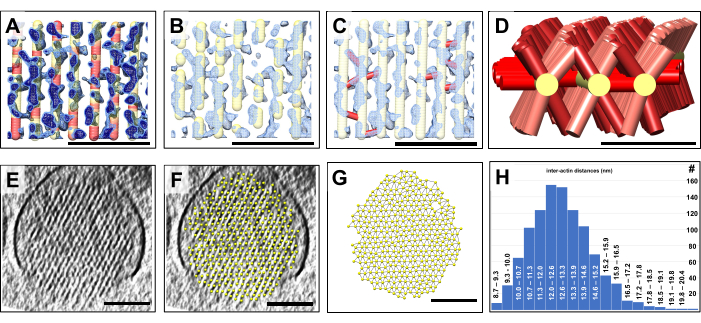
Figura 4: Rilevamento di lacune nel fascio di actina utilizzando la costruzione di modelli volumetrici. Quantificazione delle proprietà volumetriche mediante modelli a sfera e bastone. (A-C) Una piccola regione di una lastra longitudinale spessa ~10 fette/9,47 nm della mappa di densità delle stereociglia è mostrata in blu, insieme al modello curvo e corretto del filamento di actina. (A) La mappa di densità viene visualizzata a una soglia di densità inferiore (azzurro) e a una soglia di densità superiore (blu scuro). Ci sono regioni del modello di filamento di actina per le quali non esiste una densità corrispondente. In tali posizioni, il modello è stato codificato a colori in rosso per rappresentare una mancanza di densità. Interpretiamo queste posizioni come lacune nei filamenti di actina. (B) Modello dei filamenti di actina che appaiono frammentati, riflettendo la mancanza di densità dei filamenti di actina in tali punti di interruzione. (C) Modello dei filamenti di actina con legami di interconnessione incrociata (mostrati in rosso) aggiunti in punti in cui è stata trovata una forte densità per collegare i filamenti di actina adiacenti. (D) Tre filamenti di actina selezionati sono mostrati con connettori incrociati ai filamenti di actina adiacenti (che non sono mostrati per chiarezza). Si noti che molte, ma non tutte, le possibili posizioni dei filamenti inter-actina sono occupate da proteine connettore; (E) Una sezione trasversale di 30 slice/28,4 nm della densità delle stereociglia. (F) Modello di filamenti di actina montati sulla lastra di densità delle stereociglia a sezione trasversale da 30 slice/28,4 nm. (G) Modello di filamenti di actina senza la densità della mappa sottostante. Rilevamento automatico delle distanze dei filamenti di actina più vicini, come indicato da sottili connessioni tra le sfere del modello di filamento di actina. (H) Istogramma delle distanze del modello di filamento di actina. Nei pannelli A-C, la mappa di densità in Chimera viene mostrata come stile mappa "Mesh". (A-C) Barre di scala = 50 nm. (D) Barra della scala = 25 nm. (E-G) Barre di scala = 100 nm. Clicca qui per visualizzare una versione più grande di questa figura.
Figura 1 supplementare: Costruzione di modelli per il primo di due set di dati aggiuntivi di stereociglia. (A-C) Una piccola regione di una lastra longitudinale spessa ~10 fette/9,47 nm della mappa di densità delle stereociglia viene mostrata in blu utilizzando la visualizzazione in modalità mesh. Il modello posizionato inizialmente viene visualizzato in rosso e il modello corretto viene visualizzato in giallo. (A) Solo densità della mappa. (B) Modello iniziale inserito nella mappa di densità. (C) Modello corretto inserito nella mappa di densità. (D-E) Regione delle stereociglia più grande senza (D) e con (E) il modello corretto si adatta a una lastra longitudinale spessa ~10 fette/9,47 nm della mappa di densità delle stereociglia. (F-G) Viene mostrata l'intera regione del tomogramma delle stereociglia. (F) Solo mappa. (G) Mappa con il modello corretto. (H) Sovrapposizione dei modelli iniziali e corretti. Barre di scala = 100 nm Clicca qui per scaricare questo file.
Figura 2 supplementare: Costruzione di modelli per il secondo di due set di dati aggiuntivi di stereociglia. (A-C) Una piccola regione di una lastra longitudinale spessa ~10 fette/9,47 nm della mappa di densità delle stereociglia viene mostrata in blu utilizzando la visualizzazione in modalità mesh. Il modello inizialmente posizionato viene visualizzato in rosso e il modello corretto viene visualizzato in giallo; (A) Solo densità della mappa. (B) Modello iniziale inserito nella mappa di densità. (C) Modello corretto inserito nella mappa di densità. (D-E) Regione delle stereociglia più grande senza (D) e con (E) il modello corretto si adatta a una lastra longitudinale spessa ~10 fette/9,47 nm della mappa di densità delle stereociglia. (F-G) Viene mostrata l'intera regione del tomogramma delle stereociglia. (F) Solo mappa. (G) Mappa con il modello corretto. (H) Sovrapposizione del modello iniziale e corretto. Barre di scala = 100 nm. Clicca qui per scaricare questo file.
File supplementare 1: Script Python UCSF Chimera per la modellazione delle stereociglia. Clicca qui per scaricare questo file.
File di codifica supplementare 1: pblengths.py. Clicca qui per scaricare questo file.
File di codifica supplementare 2: RemoveCross.py. Clicca qui per scaricare questo file.
File di codifica supplementare 3: ActinFilamentPlane.py. Clicca qui per scaricare questo file.
File di codifica supplementare 4: dividelinks.py. Clicca qui per scaricare questo file.
File di codifica supplementare 5: FixingMarkerID.py. Clicca qui per scaricare questo file.
Discussione
Abbiamo dimostrato che gli approcci automatizzati per la segmentazione, come la segmentazione dei bacini idrografici, possono fallire nell'ambiente ad alto rumore e ad alta complessità dei tomogrammi crioelettronici delle stereociglia delle cellule ciliate. Distinguere quale parte di questa rete filamentosa rappresenti i filamenti di actina e cosa costituisca i legami incrociati a livello di ambiente locale sembra difficile nella migliore delle ipotesi quando si ispezionano piccoli sottovolumi tomografici. L'approccio di costruzione del modello utilizzato in questo studio beneficia della conoscenza preliminare dell'ordine su larga scala del fascio di actina, che aiuta a sviluppare un'aspettativa sull'orientamento dei filamenti di actina e sulle densità dei reticolanti. Forse ancora più significativo è che un cervello umano può facilmente trovare modelli considerando il contesto più ampio oltre la distribuzione locale della densità, mentre un algoritmo informatico funziona solo per una regione relativamente piccola che viene considerata dall'algoritmo; Pertanto, le tendenze su larga scala non possono essere facilmente prese in considerazione. Adattando un modello globalmente a uno strato di densità, abbiamo evitato la confusione che può verificarsi quando si tenta di creare un modello per piccole porzioni di singoli filamenti di actina alla volta. Naturalmente, tale adattamento globale presuppone un ordine che si estende su grandi distanze. Tuttavia, poiché abbiamo avuto una piccola ma significativa curvatura graduale inaspettata dei filamenti di actina, l'adattamento globale è stato solo un'approssimazione iniziale e ha richiesto aggiustamenti locali del modello per adattarsi alla mappa di densità. Poiché il modello iniziale era un buon punto di partenza, le regolazioni potevano essere effettuate con grande sicurezza. Un grande vantaggio del nostro approccio è stato che abbiamo potuto scegliere di visualizzare solo una zona di densità definita, il che ha contribuito a ridurre la complessità dello scenario. Inoltre, la visualizzazione della lastra di densità della mappa lungo l'asse del modello del filamento ha aiutato a identificare la curvatura inaspettata, che molto probabilmente ci sarebbe sfuggita visualizzando semplicemente sottovolumi più piccoli. Il posizionamento del modello iniziale ha anche facilitato l'ingrandimento e la riduzione rapidi, per alternare tra una visione d'insieme del rispettivo strato di filamenti di actina e le viste dettagliate per apportare modifiche al modello.
Le fasi critiche all'interno del protocollo includevano la rotazione della mappa dopo l'ispezione visiva, la creazione e il posizionamento del modello nella mappa di densità, nonché la divisione del modello a filamento in segmenti più piccoli. La posizione dell'atomo dei segmenti potrebbe quindi essere regolata spazialmente per adattarsi alla mappa di densità e/o codificata a colori per rilevare le lacune.
Questo approccio alla costruzione di modelli di actina può anche essere modificato posizionando un insieme di "atomi" (cioè le sfere del modello a palla e bastone) nelle densità dei filamenti utilizzando una vista in sezione trasversale di una fetta di 10-30 / 9,47-28,4 nm media di densità, che può poi essere collegata da legami (cioè i bastoncini del modello a palla e bastone). Abbiamo utilizzato questo approccio, che è una modifica rispetto al protocollo qui descritto in dettaglio, per la costruzione di modelli volumetrici nella regione conica delle stereociglia23 delle cellule ciliate. Inoltre, come abbiamo descritto qui, il nostro approccio alla costruzione di modelli volumetrici è adatto anche per la segmentazione e la costruzione di modelli di membrane.
Mentre la costruzione di modelli volumetrici può essere applicata a qualsiasi mappa di densità che mostri caratteristiche filamentose, la tecnica che abbiamo descritto qui è più efficiente quando abbiamo una serie di filamenti regolarmente distanziati, per i quali è possibile ottenere un adattamento globale di un modello volumetrico. Dipende anche dalle caratteristiche filamentose alterare la loro direzionalità in modo graduale. Se ci sono pieghe improvvise e curve brusche nelle strutture filamentose, il nostro approccio potrebbe non essere particolarmente utile per la segmentazione.
Nel frattempo, i nostri collaboratori hanno sviluppato un approccio automatizzato per il tracciamento automatizzato dei filamenti che segue un concetto simile utilizzato qui per la segmentazione manuale30,31. Andando avanti, l'approccio migliore potrebbe essere un ibrido di identificazione manuale e posizionamento di un modello sparso iniziale (anche solo poche sfere) nella densità come punto di partenza, e quindi lasciare che un algoritmo di ricerca e adattamento finisca il tracciamento dei filamenti.
I modelli volumetrici semplificati riducono la complessità di un sistema e consentono di apprezzare meglio alcuni modelli, come la flessione del filamento di actina vicino alla punta. Inoltre, il modello volumetrico può essere utilizzato come "ancoraggio" per visualizzare una zona di densità attorno al modello di ancoraggio a sfera e bastone scelto, che consente il rilevamento e la visualizzazione delle densità di reticolanti tra filamenti di actina adiacenti. La capacità di selezionare i singoli filamenti e di impostare i raggi appropriati come zona in cui la densità viene nuovamente visualizzata consente di ridurre la travolgente complessità dello scenario a un livello gestibile.
Un vantaggio di questo approccio di costruzione del modello volumetrico di adattamento globale, seguito da aggiustamenti locali, è stato che siamo stati in grado di identificare le regioni in cui i filamenti di actina sembravano essere interrotti e lacune significative nei filamenti di actina erano indicate dall'assenza di densità della mappa. Poiché avevamo posizionato un modello volumetrico a sfera e bastone, abbiamo potuto utilizzare una routine nel pacchetto software UCSF Chimera che codifica a colori la posizione della sfera di ogni modello in base al valore di densità della mappa in quella posizione. Questo approccio ha permesso una rapida rilevazione e visualizzazione delle lacune del filamento di actina nel fascio di actina, che è una caratteristica biologicamente significativa che abbiamo trovato nel nostro tomogramma crioelettronico e che sarebbe stata molto difficile da rilevare e visualizzare con gli approcci di segmentazione tradizionali. Un altro vantaggio del nostro modello volumetrico è che le proprietà volumetriche, comprese le lunghezze e le distanze, possono essere facilmente ottenute, il che consente di ottenere numeri effettivi e quindi di eseguire un'analisi statistica.
In sintesi, il posizionamento manuale interattivo dei punti del modello, possibilmente ulteriormente potenziato da successive capacità automatizzate di adattamento locale e tracciamento dei filamenti, è un approccio piuttosto promettente per la visualizzazione e l'analisi quantitativa dei volumi subcellulari tomografici degli elettroni. Questo perché utilizza la potenza del cervello umano per il riconoscimento dei modelli e la potenza dell'informatica per l'ottimizzazione dei modelli.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti o altri conflitti di interesse.
Riconoscimenti
Ringraziamo il Dr. Peter Barr-Gillespie e il suo team per il loro ruolo nella preparazione dei campioni e gli ex membri del laboratorio Auer e del laboratorio Dr. Dorit Hanein per il loro ruolo nella raccolta dei dati tomografici. Vorremmo anche ringraziare Tom Goddard di UCSF Resource for Biocomputing, Visualization, and Informatics (RBVI) per aver fornito vari script UCSF Chimera.
Materiali
| Name | Company | Catalog Number | Comments |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/download.html | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=win64/chimera-1.16-win64.exe | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=mac64/chimera-1.16-mac64.dmg | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=linux_x86_64/chimera-1.16-linux_x86_64.bin | |
| Excel | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Falcon II | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/accessories-em/falcon-detector.html | |
| IMOD | University of Colorado | Version 4.11.1 https://bio3d.colorado.edu/imod/download.html | |
| PC Desktop | Intel | Windows 10, ver. 22H2 | |
| PC Laptop | Gigabyte | Windows 10, ver. 22H2 | |
| Powerpoint | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Titan Krios Electron Microscope | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/transmission-electron-microscopes/krios-g4-cryo-tem.html | |
| Word | Microsoft | Version 2211 https://www.office.com/?auth=1 |
Riferimenti
- Downing, K. H., Sui, H., Auer, M. Electron tomography: A 3D view of the subcellular world. Analytical Chemistry. 79 (21), 7949-7957 (2007).
- Koning, R. I., Koster, A. J. Cellular nanoimaging by cryo electron tomography. Methods in Molecular Biology. 950, 227-251 (2013).
- Asano, S., Engel, B. D., Baumeister, W. In situ cryo-electron tomography: A post-reductionist approach to structural biology. Journal of Molecular Biology. 428 (2), 332-343 (2016).
- Serwas, D., Davies, K. M. Getting started with in situ cryo-electron tomography. Methods in Molecular Biology. 2215, 3-23 (2021).
- McDonald, K. L., Auer, M. High-pressure freezing, cellular tomography, and structural cell biology. Biotechniques. 41 (2), 137-143 (2006).
- Narasimha, R., et al. Evaluation of denoising algorithms for biological electron tomography. Journal of Structural Biology. 164 (1), 7-17 (2008).
- Frangakis, A. S. It's noisy out there! A review of denoising techniques in cryo-electron tomography. Journal of Structural Biology. 213 (4), 107804 (2021).
- Frangakis, A. S., Hegerl, R. Noise reduction in electron tomographic reconstructions using nonlinear anisotropic diffusion. Journal of Structural Biology. 135 (3), 239-250 (2001).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. Journal of Structural Biology. 144 (1-2), 114-122 (2003).
- vander Heide, P., Xu, X. -. P., Marsh, B. J., Hanein, D., Volkmann, N. Efficient automatic noise reduction of electron tomographic reconstructions based on iterative median filtering. Journal of Structural Biology. 158 (2), 196-204 (2007).
- Volkmann, N. Methods for segmentation and interpretation of electron tomographic reconstructions. Methods in Enzymology. 483, 31-46 (2010).
- Böhm, J., et al. Toward detecting and identifying macromolecules in a cellular context: template matching applied to electron tomograms. Proceedings of the National Academy of Sciences. 97 (26), 14245-14250 (2000).
- Frangakis, A. S., et al. Identification of macromolecular complexes in cryoelectron tomograms of phantom cells. Proceedings of the National Academy of Sciences. 99 (22), 14153-14158 (2002).
- Lebbink, M. N., et al. Template matching as a tool for annotation of tomograms of stained biological structures. Journal of Structural Biology. 158 (3), 327-335 (2007).
- Rigort, A., et al. Automated segmentation of electron tomograms for a quantitative description of actin filament networks. Journal of Structural Biology. 177 (1), 135-144 (2012).
- Volkmann, N. A novel three-dimensional variant of the watershed transform for segmentation of electron density maps. Journal of Structural Biology. 138 (1-2), 123-129 (2002).
- Bajaj, C., Yu, Z., Auer, M. Volumetric feature extraction and visualization of tomographic molecular imaging. Journal of Structural Biology. 144 (1-2), 132-143 (2003).
- Yu, Z., Frangakis, A. S. Classification of electron sub-tomograms with neural networks and its application to template-matching. Journal of Structural Biology. 174 (3), 494-504 (2011).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Tilney, L. G., Derosier, D. J., Mulroy, M. J. The organization of actin filaments in the stereocilia of cochlear hair cells. The Journal of Cell Biology. 86 (1), 244-259 (1980).
- Tilney, L. G., Tilney, M. S., DeRosier, D. J. Actin filaments, stereocilia, and hair cells: how cells count and measure. Annual Review of Cell Biology. 8, 257-274 (1992).
- Metlagel, Z., et al. Electron cryo-tomography of vestibular hair-cell stereocilia. Journal of Structural Biology. 206 (2), 149-155 (2019).
- Song, J., et al. A cryo-tomography-based volumetric model of the actin core of mouse vestibular hair cell stereocilia lacking plastin 1. Journal of Structural Biology. 210 (1), 107461 (2020).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Agulleiro, J. I., Fernandez, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27 (4), 582-583 (2011).
- Fernandez, J. J. Computational methods for electron tomography. Micron. 43 (10), 1010-1030 (2012).
- Chen, H., Clyborne, W. K., Sedat, J. W., Agard, D. A. Priism: an integrated system for display and analysis of 3-D microscope images. Biomedical Image Processing and Three-Dimensional Microscopy. 1660, 784-790 (1992).
- Pettersen, E. F., et al. UCSF Chimera-a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25 (13), 1605-1612 (2004).
- Goddard, T. D., Huang, C. C., Ferrin, T. E. Visualizing density maps with UCSF Chimera. Journal of Structural Biology. 157 (1), 281-287 (2007).
- Sazzed, S., et al. Tracing actin filament bundles in three-dimensional electron tomography density maps of hair cell stereocilia. Molecules. 23 (4), 882 (2018).
- Sazzed, S., Scheible, P., He, P., Wriggers, J. Spaghetti tracer: A framework for tracing semiregular filamentous densities in 3D tomograms. Biomolecules. 12 (8), 1022 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon