Method Article
Simplified volumetric models as an effective strategy as an effective strategy for segmenting actin Networks in cryo-electron tomograms(초저온 전자 단층촬영에서 액틴 네트워크 분할을 위한 효과적인 전략으로서의 단순화된 체적 모델)
요약
여기에서는 단순화된 체적 모델을 잡음이 많고 복잡한 단층 촬영 3D 볼륨에 배치하는 프로토콜을 제시합니다. 이를 통해 액틴 필라멘트 밀도를 빠르게 세분화하고, 체계적인 필라멘트 굽힘 및 헤어 번들 필라멘트의 갭을 감지하고, 거리와 같은 체적 모델 속성을 편리하게 정량화할 수 있습니다.
초록
관심 있는 특징을 추출하기 위한 효율적인 방법은 초저온 전자 단층촬영(cryo-electron tomograms) 해석의 가장 큰 과제 중 하나로 남아 있습니다. 다양한 자동화 접근 방식이 제안되었으며, 그 중 다수는 관심 기능을 쉽게 감지할 수 있고 서로 명확하게 구분될 수 있는 고대비 데이터 세트에 적합합니다. 우리의 내이 입체섬모 초저온 전자 단층 촬영 데이터 세트는 자주 교차 연결되는 육각형으로 채워진 액틴 필라멘트의 조밀한 배열이 특징입니다. 이러한 기능은 자동 분할을 매우 어렵게 만들며, 초저온 전자 단층촬영의 고잡음 환경과 조밀하게 밀집된 기능의 높은 복잡성으로 인해 더욱 악화됩니다. 액틴 번들 조직에 대한 사전 지식을 사용하여 매우 단순화된 볼-스틱(ball-and-stick) 액틴 모델의 레이어를 배치하여 먼저 밀도 맵에 대한 글로벌 피팅을 얻은 다음 모델의 지역 및 로컬 조정을 수행했습니다. 우리는 체적 모델 구축을 통해 높은 복잡성을 처리할 수 있을 뿐만 아니라 actin 번들에 대한 정확한 측정 및 통계를 제공한다는 것을 보여줍니다. 체적 모델은 또한 actin-actin 교차 커넥터의 경우와 같이 로컬 분할을 위한 앵커링 포인트 역할을 합니다. 체적 모델 구축은 특히 컴퓨터 기반 자동 피팅 접근 방식으로 더욱 강화될 때 기존의 자동 세분화 접근 방식이 성공적이지 않을 때 강력한 대안이 될 수 있습니다.
서문
초저온 전자 단층촬영(cryo-electron tomography)을 사용하면 플런지 동결(plunge-freesing)4 또는 고압 동결(high-pressure freesing), 초고속 유리화(vitrification)5를 사용하여 전체 소기관 또는 세포 및 조직의 일부를 나노미터 해상도로 거의 네이티브 상태 1,2,3로 시각화할 수 있습니다. 극저온 보존, 염색되지 않은, 동결된 수화 샘플은 제한된 전자 선량만 허용할 수 있기 때문에 단층 촬영 3D 데이터는 매우 시끄럽습니다. 이러한 잡음은 종종 비선형 비등방성 확산(nonlinear anisotropic diffusion)8, 양측 필터링(bilateral filtering)9, 재귀 중앙값 필터링(recursive median filtering)10을 포함한 다양한 잡음 필터링 알고리즘(noise)6,7에 의해 현저히 감소될 수 있다.
또한 현미경 스테이지의 기울기 제한으로 인해 정보 쐐기가 누락되고 높은 기울기 각도에서 표본 두께가 증가한다는 사실로 인해 이방성 해상도로 3D 재구성이 가능합니다. 이것은 밀도가 Z 방향의 낮은 해상도로 인해 3차원에서 번진다는 것을 의미합니다. 그 결과, 거대분자의 모양이 왜곡된 것처럼 보입니다(즉, 3차원에서 덜 명확하고 길쭉한 것처럼 보입니다).
단층 촬영 데이터 해석에서 가장 큰 과제 중 하나는 세분화11이라고도 하는 관련 특징을 자동으로 추출하는 것입니다. 충분한 독특한 형상 특징과 낮은 소음으로 복잡한 3D 볼륨의 고분자 기계는 12,13,14 와 일치하는 템플릿으로 식별할 수 있습니다. 그러나 템플릿 일치의 성공 여부는 단층촬영 해상도, 적절한 검색 모델, 피처 볼륨의 크기 및 모양 특성에 따라 달라집니다. 관심 있는 특징이 충분히 떨어져 있고 반복되는 모티프(예: 대형 고분자 기계)를 쉽게 식별할 수 있는 경우, 단층 촬영 하위 볼륨을 결합하여 신호 대 잡음 비율을 높이고 개별 입자 형상 왜곡을 평균화할 수 있습니다. 주형 매칭에 의한 동결 수화된 딕티오스텔리움 디스코이데움 세포의 얇은 가장자리에 대한 전자 단층촬영에서 액틴 필라멘트 네트워크의 자동 분할이 보고되었습니다15.
그러나 관심 피쳐가 밀접하게 배치되어 있는 경우 데이터 해상도 이방성으로 인해 Z 방향(전자빔 방향을 따라)의 맵 밀도가 번져 밀접하게 배치된 고분자 기계 또는 초분자 복합체의 밀도 포락선이 명백하게 병합될 수 있습니다. 이러한 경우, 유역(watershed)16, 경계 분할(boundary segmentation)17, 또는 다양한 머신러닝-기반 분류 접근법(18,19)과 같은 세분화를 위한 자동화된 접근법들은 관심있는 특징들을 인식하거나 관심있는 객체 주위에 정확한 경계를 설정할 수 없을 수 있다. 종종, 몇 개의 매우 큰 조각이나 심하게 과도하게 세그먼트 화 된 볼륨으로 끝나며, 관심있는 기능이 완성 된 것으로 인식 될 때까지 많은 작은 조각을 병합하는 데 많은 노력이 필요합니다. 세분화 결과의 이러한 수동 큐레이션은 매우 노동 집약적일 수 있으며 관심 구조가 짧은 링커를 통해 상호 연결된 밀접한 간격의 필라멘트 배열인 경우 완전히 실패할 수도 있습니다. 이 거대한 필라멘트 구조의 네트워크에서는 방향을 잡기가 어려울 수 있습니다. 이는 해상도 이방성으로 인해 밀도가 서로 혼합되는 것처럼 보이기 때문이며, 이는 자동화 및 대화형 수동 세분화 접근 방식 모두에 엄청난 도전을 제시합니다. 결과적으로, 작은 영역만 육안으로 검사할 때 필라멘트 사이를 쉽게 "점프"할 수 있습니다.
다행히도, 내이 유모 세포 입체섬모에 있는 액틴 다발의 경우, 우리는 전반적인 액틴 다발 조직과 액틴 필라멘트20,21의 방향성에 대한 지식을 가지고 있습니다. 액틴 다발은 직경 6-8nm의 수백 개의 육각형으로 조밀하게 채워진 액틴 필라멘트로 구성되며, 이들은 서로 약 12-13nm 떨어져 있습니다22.
이를 통해 액틴 필라멘트를 표현하기 위해 단순화된 볼-앤-스틱 모델을 기반으로 하는 세분화에 대해 다소 다른 접근 방식을 취할 수 있었습니다. 이 전략은 극저온 전자 단층 촬영 밀도 맵의 슬래브에 이상화된 규칙적인 필라멘트 모델 배열을 동시에 배치하여 액틴 번들의 3D 모델을 층별로 구축하는 것이었습니다. 우리는 밀도 맵과 밀접하게 일치하도록 개별 필라멘트 모델 또는 필라멘트 모델 그룹을 지역적으로 조정하기 전에 모델이 밀도 맵에 전반적으로 전반적으로 맞는지 확인했습니다. 필라멘트 모델 위치에서 맵 밀도 값의 자동 색상 코딩을 통해 액틴 번들에서 명백한 갭을 쉽게 감지할 수 있었습니다. 체적 모델을 사용하면 액틴 필라멘트 사이의 거리와 같은 체적 특성을 정량적으로 분석할 수 있으며 전체 3D 필라멘트 네트워크 조직을 단순화하여 표시할 수 있습니다.
또한, 모델은 개별 필라멘트 모델(의 일부)을 선택할 수 있으므로 액틴-액틴 링커와 같은 추가 기능의 분할을 위한 앵커링 구조 역할을 할 수도 있으며, 그 주위에 검사 및 추가 분할을 위해 적절한 반경 맵 밀도 영역을 생성할 수 있습니다.
우리는 체적 모델 기반 세분화 접근 방식이 갭과 필라멘트 간 교차 연결을 포함할 수 있는 대규모 필라멘트 구조 네트워크에 특히 유용하다고 생각합니다. 세분화 알고리즘은 로컬에서 작동하는 경향이 있는 반면, 인간의 두뇌는 더 넓은 영역을 고려하기 때문에 복잡하고 노이즈가 많은 환경에서도 필라멘트 구조를 인식하는 데 있어 컴퓨터보다 우수합니다.
프로토콜
이 프로토콜은 동남대학교의 인간 연구 윤리 위원회의 지침을 따릅니다.
1. 체적 모델 구축을 위한 초저온 전자 단층 촬영 데이터 소스
참고: 체적 모델 건물에 사용된 입체섬모 초저온 전자 단층 촬영 재구성은 이전에 발표되었으며(22,23), Metlagel et al.22에 의해 설명된 대로 얻어졌습니다.
입체섬모 모델링을 위한 UCSF 키메라 파이썬 스크립트는 보충 파일 1, 보충 코딩 파일 1, 보충 코딩 파일 2, 보충 코딩 파일 3, 보충 코딩 파일 4 및 보충 코딩 파일 5에 제공됩니다.
- 간단히 말해서, 마우스 난소 감각 상피의 정점 표면에서 전자 현미경(EM) 그리드의 레이시 탄소 지지 필름으로 입체섬모를 닦아냅니다. 그런 다음 0.47 - 0.59nm 픽셀 크기의 통합 모드에서 CMOS 유형 카메라를 사용하여 3.5-4.5μm의 공칭 디포커스로 300kV에서 작동하는 극저온 투과 전자 현미경(cryo-TEM)에서 초고속 급락 동결 유리화 및 단일 축 초저온 전자 단층 촬영 데이터 수집을 수행합니다( 재료 표 참조).
- 단축 데이터 수집을 위한 일반적인 선량은 일반적으로 80-100전자/Å2입니다. 소프트웨어 패키지 IMOD24를 사용하여 가중 역투영 또는 SIRT 방법25,26을 사용하여 단층촬영 3D 볼륨을 재구성합니다.
- Priism27 의 재귀적 중앙값 또는 양측 필터링 또는 IMOD의 비선형 비등방성 확산 옵션을 사용하여 단층촬영의 노이즈를 제거합니다.
참고: 대화형 시각화, 체적 모델 구축 및 정량 분석에 사용된 주요 소프트웨어 패키지는 UCSF Chimera 소프트웨어28,29였습니다. 사용된 모든 소프트웨어 패키지는 Table of Materials에 나열되어 있습니다.
2. 체적 모델 구축을 위한 초저온 전자 단층 촬영 데이터 준비
- 단층 촬영 회전
참고: 이 단계의 목적은 액틴 코어가 3개의 축(x 및 z) 중 2개(x 및 z)에 정렬되는 "최적의 각도"를 찾아 하나의 축만 사용하여 모델을 3D로 배치할 수 있도록 하는 것입니다. 다음 단계는 IMOD 소프트웨어 패키지 내에서 수행됩니다.- 3dmod(IMOD)를 열고 3D 이미지 스택 파일을 .mrc 파일 형식으로 엽니다. 3D 스택이 16비트 회색 음영 모드이고 이미지 파일 헤더에 올바른 X, Y, Z 치수를 표시하는지 확인합니다. 필요한 경우 명령 프롬프트에서 alterheader 명령 alterheader -d (x_pixelspacing),(y_pixelspacing),(z_pixelspacing) inputfilename.mrc를 사용하여 수정합니다. 3D 스택이 TIFF 파일 형식인 경우 명령 프롬프트에서 tif2mrc inputfilename.tif outputfilename.mrc 명령을 사용하여 .mrc 파일을 만듭니다.
- 단층 촬영을 육안으로 검사하고 "슬라이서"를 열고(백슬래시 "\"를 눌러) X, Y, Z 차원에서 액틴 필라멘트 평면을 Z 평면과 정렬하기 위한 최상의 회전 각도를 찾습니다. X 회전, Y 회전, Z 회전 막대를 조작하여 액틴 필라멘트가 X 및 Z 평면에서 정렬되는 최적의 각도를 찾습니다. 단층 촬영(Thick : Img) 표시의 평균 두께를 변경하면 Z-평면의 밀도 평균을 이상적으로 구하는 데 도움이 될 수 있으므로 대비가 증가할 수 있습니다. 단층촬영을 확대 및 축소하려면 키보드의 + 및 - 키를 각각 사용합니다.
- 이상적인 회전 각도가 확인되면, 명령 프롬프트에 IMOD 명령 rotatevol -a (Z rotation angles),(Y rotation angles),(X rotation angles) -s (x-width),(y-height),(z-depth) inputfilename.mrc outputfilename.mrc 를 입력하여 단층 촬영을 회전합니다. 회전 시 지도의 일부가 잘리지 않도록 회전을 수용할 수 있는 충분한 공간을 지도에 제공해야 합니다.
참고: rotatevol 명령을 사용하는 방법에 대한 자세한 내용은 도움말 메뉴의 옵션을 사용하거나 다음 URL(https://bio3d.colorado.edu/imod/doc/man/rotatevol.html)을 참조하십시오. 이 명령 ( rotatevol )은 IMOD 창에 현재 표시된 맵을 변경하지 않고 회전중인 밀도 맵이있는 새 맵 파일을 만듭니다. - 슬라이서 창을 사용하여 지도의 서로 반대쪽 두 모서리의 X, Y, Z 좌표를 식별하고, trimvol 명령을 사용하여 입체실모를 포함하는 자르기 영역을 식별합니다.
- IMOD 명령 Trimvol 을 사용하여 명령 프롬프트에 trimvol -x (x-coordiante 1),( x-coordiante 2) -y (y-coordiante 1),( y-coordiante 2) -z (z-coordiante 1),( z-coordiante 2) inputfilename.mrc outputfilename.mrc 를 입력하여 이전에 얻은 좌표에서 지도를 자릅니다. 이제 지도가 훨씬 작아졌으므로 이 시점부터 작업하기가 더 쉽고 빠릅니다.
참고: trimvol 명령을 사용하는 방법에 대한 자세한 내용은 도움말 메뉴의 옵션을 사용하거나 다음 URL(https://bio3d.colorado.edu/imod/doc/man/trimvol.html)을 참조하십시오. 이러한 단계를 수행하면 회전 및 트리밍된 .mrc 파일이 만들어집니다.
- 단층 촬영 필터링
참고: 이 단계에서는 노이즈 감소를 위해 IMOD에서 제공하는 비선형 비등방성 확산(NAD) 필터를 사용합니다. 다음 URL(https://bio3d.colorado.edu/imod/doc/NADexample.html)에 나열된 대로 비선형 비등방성 확산 필터링을 위해 IMOD 도움말 페이지에서 권장하는 것을 초기 값으로 사용했으며 초기 값으로 사용되었습니다.- 명령 프롬프트에 etomo 를 입력하여 IMOD의 etomo 그래픽 사용자 인터페이스를 시작합니다.
- etomo 메뉴에서 Nonlinear Anisotropic Diffusion 옵션을 선택하고 맵 파일(볼륨 선택)을 선택합니다.
- 육안 검사로 판단할 때 최상의 필터링 결과를 제공하는 K 값과 반복 횟수를 찾으려면 NAD 필터를 더 작은 테스트 볼륨에 적용하여 지정된 NAD 필터 테스트 볼륨을 추출합니다. 3dmod 인터페이스 상단의 Rubberband Tool을 클릭하고, 마우스 왼쪽 버튼을 클릭하여 드래그하여 테스트 볼륨을 선택하고, Hi 및 Lo 버튼을 클릭하여 Z-slice를 테스트 볼륨의 경계로 표시합니다(3dmod에서 테스트 볼륨 범위 가져오기). Extract Test Volume(테스트 볼륨 추출)을 클릭하여 테스트 볼륨을 추출합니다.
- 0.1, 1,5, 10, 15, 25, 30, 50, 75와 같은 다양한 K 값(테스트 볼륨 > K 값 목록에 대한 K 값 찾기)을 사용하여 테스트 볼륨을 필터링합니다. 지정된 각 K 값으로 NAD 필터링을 수행합니다(다른 K 값으로 실행 > 다른 K 값 테스트 결과 보기). 슬라이서 창을 사용하여 NAD 필터의 성능을 평가하고 전체 볼륨을 필터링하는 데 사용할 K 값을 선택합니다.
- 2.2.4단계에서 식별된 K 값을 사용하여 2, 5, 8, 11, 15 및 21과 같은 다양한 반복 횟수를 테스트합니다(테스트 볼륨 > 반복 목록에 대한 반복 횟수 찾기). 다른 반복 테스트 결과 보기를 클릭하거나 슬라이서 도구를 사용하여 다른 반복에 대한 NAD 필터 성능을 평가합니다. 다른 반복 횟수에 대한 값을 선택합니다.
- 식별된 K 값과 반복 횟수(Filter Full Volume > K 값 및 Iterations)를 사용하고 전체 볼륨을 필터링합니다(Filter Full Volume). 새로 필터링된 볼륨은 로드된 전체 볼륨이 있는 동일한 디렉터리에 .mrc 파일에 .nad 파일 확장자가 첨부되어 나타납니다.
참고: 이러한 단계를 수행하면 NAD 필터로 필터링된 .mrc 파일이 만들어지고 노이즈가 낮아져 신호 대 노이즈 비율이 향상됩니다.
3. 체적 모델 구축
- 3D 모델 배치 준비
참고: 이 단계의 목적은 UCSF Chimera 프로그램을 사용하여 회전 및 노이즈 제거된 밀도 맵에 3D 체적 모델을 배치하기 위한 맵을 준비하고 분석하는 것입니다.- 슬라이서 창을 사용하여 단면도(30개 슬라이스/28.4nm의 슬래브)를 얻어 액틴 필라멘트 모델이 배치될 밀도 슬래브 중심의 Z 좌표를 식별합니다. 필라멘트 밀도의 중심을 클릭하고 IMOD 메인 창에서 Z 좌표를 확인합니다.
- UCSF Chimera에서 향후 모델 배치를 위해 식별된 좌표를 기록해 둡니다.
참고: 이 단계에서 언급된 Z 좌표는 4단계에서 사전 제작된 액틴 필라멘트 모델을 올바른 Z 높이로 쉽게 찾고 배치하는 데 사용되므로 모델을 더 빠르게 배치할 수 있습니다.
- 3D 모델 배치
참고: 이 단계의 목적은 UCSF Chimera를 사용하여 준비된 밀도 맵에 3D 체적 모델을 배치하는 것입니다.- UCSF Chimera에서 필터링되고 회전된 맵(File > Open)을 엽니다.
- Volume Viewer에서 Features > Planes 및 Feature > Coordinates 를 선택하여 피처 및 좌표 인터페이스를 열어 맵의 매개변수가 올바르게 설정되었는지 확인합니다. 복셀 크기를 확인하여 명령 프롬프트(IMOD)에서 헤더 명령을 사용하여 올바른 복셀 간격이 설정되어 있는지 확인합니다. 그렇지 않은 경우 복셀 크기 인터페이스를 올바른 복셀 간격으로 수정하십시오. 지도를 중앙에 배치하여 카메라를 보기 창의 중앙(Origin Index > Center)에 설정합니다.
- 카메라 제어 창(Tools > Viewing Control > Camera)을 엽니다. 그런 다음 카메라 보기를 직교 보기(투영 > 직교)로 설정합니다
- 미리 만들어진 Python 스크립트 모델(File > Open > ActinFilamentPlane.py) 을 로드하고 맵에 있는 actin 필라멘트 평면만큼 actin 필라멘트 평면 스크립트를 엽니다.
- 이동 마우스 모드(Tools > Movement > Movement Mouse Mode)를 켜서 로드된 모델을 재배치합니다. 이동 마우스 옵션(이동 마우스 모드 설정 > 선택 항목 이동)을 활성화합니다. Ctrl + Drag를 사용하여 세션에 있는 모델을 여러 개 선택하고 마우스 오른쪽 버튼을 클릭(또는 마우스 기본 설정에 지정된 다른 키)> 클릭하여 모델을 원하는 방향으로 이동합니다.
참고: 모델은 특정 두께(ActinFilamentPlane.py 스크립트의 반경 )와 특정 간격(ActinFilamentPlane.py 스크립트의 Lattice_Spacing )을 가진 공과 막대기로 만들어졌습니다. - 관심 있는 모델만 시각화하려면 모델 패널(도구 > 일반 컨트롤 > 모델 패널)을 엽니다. S (Show) 아래의 확인란을 클릭하여 지정된 모델의 가시성을 활성화하거나 비활성화합니다.
- 명령행 패널(Tools > General Controls > Command Line)을 활성화합니다.
- 각 액틴 필라멘트 평면 모델을 적절한 Z 높이로 배치한 후 모델의 초과 부분을 선택하고 UCSF Chimera 주 뷰 창 하단의 명령행에 del sel (선택 항목 삭제)을 입력하여 각 액틴 필라멘트 평면 모델에서 초과된 액틴 필라멘트를 삭제합니다.
- 모델 패널 창으로 이동하여 다른 모든 액틴 필라멘트 모델을 선택하고 숨기거나(Ctrl + 클릭 > 숨기기) 한 번에 하나의 액틴 필라멘트 평면만 시각화합니다.
- 관심 있는 단일 액틴 필라멘트 평면 모형에 바로 근접한 맵 밀도 부분을 시각화하려면 모형을 선택하고(모델 패널 > 선택) Volume Viewer(Volume Viewer > Features > Zone)에서 구역 제어 패널을 엽니다. 구역 반경 을 100 Å로 설정한 다음 구역 을 클릭하여 선택한 모델에서 반경 100 Å(= 10 nm) 내에 맵을 구역화합니다. Chimera의 치수는 Angstrøm(1 Å = 0.1 nm)으로 설정됩니다.
- 필요한 경우 이동 마우스 모드를 켜서 밀도 맵에 맞게 모델을 조정합니다. 적절한 위치에 있지 않은 모델을 다중 선택하여 모델을 이동하여 zoned density map 내부에 적절하게 배치합니다.
- ActinFilamentPlane.py 의 프로그래밍 버그를 극복하려면 "atoms"(공과 막대기 모델의 공)의 누락된 MarkerID를 수정하는 FixingMarkerID.py 스크립트를 실행합니다. 전체 모델을 선택하고 명령줄에서 runscript FixingMarkerID.py 입력합니다(스크립트는 C: \ directory \ Script.py 와 같은 올바른 디렉터리로 지정해야 함).
- 밀도 맵에 액틴 필라멘트가 곡선으로 표시되어 있으면 원하는 모델을 선택하고 runscript dividelinks.py 번호를 입력하여 곡률을 수용할 수 있도록 직선 액틴 필라멘트를 여러 부분으로 나눕니다. 추가 마커(명령의 숫자로 지정됨)는 서로 등거리에 있는 actin 모델 내부에 배치되어 모델을 구부릴 수 있는 "조인트"를 만듭니다.
- Movement Mouse Mode를 사용하여 구역 맵을 따라 추가 마커 "joints"를 이동하여 곡률을 반영하도록 관절을 조정합니다. 필요에 따라 dividelinks.py 스크립트를 사용하여 모델에 표식기를 추가합니다.
- 모든 액틴 필라멘트가 액틴 밀도 내에 제대로 배치되었는지 확인하려면 UCSF Chimera 드롭다운 메뉴에서 Tools > Viewing Control > Camera 를 클릭하여 클리핑 바를 사용하여 Camera Control 창을 연 다음 Side View 를 선택하여 카메라의 두 클리핑 바(근거리 및 원거리 클리핑 평면)를 확인합니다. 마우스 왼쪽 버튼을 클릭하여 두 개의 노란색 막대를 좁은 슬릿으로 드래그하고 마우스 가운데 버튼을 사용하여 클리핑 부분을 드래그하여 모델을 한 번에 하나씩 조금씩 검사합니다.
참고: 스테레오실리움 액틴 코어 모델이 완성되었습니다.
- 멤브레인 세분화
참고: 이 단계의 목적은 입체섬모막의 표면 모델을 만드는 것입니다.- 표면 세분화는 볼륨 추적기 도구(도구 > 볼륨 데이터 > 볼륨 추적기)를 사용합니다.
- Volume Tracer 창에서 Mouse를 클릭하고 Place Markers on Data Planes 및 Link new marker to selected marker 옵션만 선택합니다. 이렇게 하면 동일한 Z 평면에 마커를 정확하게 배치할 수 있습니다(단일 Z 평면이 표시되는 경우). 또한 볼륨 트레이서 창에서 마우스 가운데 버튼을 사용하여 마커 배치를 확인합니다.
- 새 마커 세트를 만듭니다(파일 > 새 마커 세트). 마커 세트 1은 볼륨 트레이서 창에서 생성하고 선택해야 합니다.
- Volume Viewer를 사용하여 기본 보기 창에 단일 Z-평면을 표시합니다(Axis to Z > One).
- 가운데 마우스 버튼을 사용하여 보이는 멤브레인의 한쪽 끝에서 다른 쪽 끝으로 멤브레인을 추적합니다. 모든 마커는 한쪽 끝에서 다른 쪽 끝으로 링크로 연결되어야 합니다.
- 선택 가능한 항목이 하나도 없는 배경에서 Ctrl + 클릭 을 눌러 마지막으로 배치된 마커를 선택 취소합니다. 이렇게 하면 배치할 다음 마커가 다른 Z 평면의 이전 멤브레인 트레이스가 아닌 새 평면의 링크와 연결됩니다.
- 보이는 막의 한쪽 끝에서 다른 쪽으로 추적하는 동일한 방향으로 10nm마다 멤브레인 추적을 반복합니다.
- 많은 병렬 밴드 멤브레인이 분할되면 Volume Tracer 창으로 이동하여 Features > Surfaces를 클릭합니다. 이렇게 하면 볼륨 트레이서 창에서 표면 인터페이스를 사용할 수 있습니다. Surfaces 인터페이스 옆에 있는 Create를 클릭하여 멤브레인 세분화 밴드 사이에 표면을 만듭니다. 이렇게 하면 이격된 멤브레인 트레이스를 채우는 표면 모델의 얇은 플랩이 만들어집니다.
참고: 이제 입체섬모 멤브레인 표면 모델 생성이 완료되었습니다.
- Crosslinker 모델링
참고: 이 단계의 목적은 액틴 필라멘트 사이의 가교결합자를 모델링하는 것입니다.- 가교제는 볼륨 트레이서 도구를 사용하여 배치할 수도 있습니다.
- 볼륨 트레이서 창에서 마우스 왼쪽 버튼을 클릭하여 표면에 마커 배치(Place Markers on Surfaces) 및 선택한 마커에 새 마커 연결(Link new marker to selected marker) 만 선택합니다. 그런 다음 볼륨 트레이서 창에서 마우스 가운데 버튼을 사용하여 마커 배치를 선택합니다.
- 관심 있는 단일 액틴 필라멘트 평면 모델 주위의 밀도는 zone 함수를 사용해서만 시각화합니다. 영역 반경 을 100 Å로 설정하여 선택한 모델에서 반경 100 Å 내에 있는 맵을 시각화합니다.
- 교차결합제의 보이는 밀도를 가로질러 마커를 배치하려면 Middle Mouse Button을 클릭합니다. 두 개의 마커가 배치되면(따라서 단일 가교제가 모델링됨) 두 번째 마커를 선택 취소하여 링크가 다음 가교 모델에 계속 연결되지 않도록 해야 합니다.
- 모든 액틴 필라멘트 평면에 대해 위의 단계를 반복하고 교차결합제가 위치할 수 있는 세 가지 주요 방향 모두에 대해 위의 전체 프로세스를 반복합니다. actin 필라멘트 평면을 대각선 방향으로 구역화할 때 actin 필라멘트 모델을 개별적으로 수동으로 선택합니다.
참고: 각 actin crosslinker 모델에 대해 추정 crosslinker가 연결되는 actin 필라멘트의 위치를 기록할 수 있습니다. 이 정보는 원칙적으로 sub-tomogram 평균을 사용하여 후속 분석을 위해 추출할 수 있으며, 이는 이 연구의 범위를 벗어납니다.
4. 3D 모델의 정량 분석
- 갭 분석
참고: 이 단계의 목적은 액틴 필라멘트의 갭을 감지하는 것입니다.- 선택한 항목을 분석할 수 있습니다. Inspect Selection 도구(Action > Inspect)를 사용하여 색상과 같은 매개변수를 지정합니다.
- 선택한 항목의 색상은 UCSF Chimera에서 선택 가능한 항목을 선택하고 Inspect Selection 창의 Color 섹션 옆에 있는 색상을 변경하여 지정할 수 있습니다.
- UCSF Chimera에서 마커 및 링크를 선택하고 Angstrøm에 단위가 있는 Inspect Selection 창의 반지름 섹션 옆에 있는 값을 변경하여 마커 및 링크의 크기를 지정합니다.
- Values at Atom Positions 기능(Volume Data > Values at Atom Positions을 > 도구)을 활용하여 밀도 갭을 자동으로 감지하여 갭 분석을 수행합니다.
- Values at Atom Positions 창의 Volume Data(볼륨 데이터) 섹션 옆에 있는 원하는 맵을 선택하여 값을 읽을 맵을 선택합니다. Molecule 섹션 옆에 있는 원하는 모델을 선택하여 모든 마커에 매개변수가 할당될 모델을 선택합니다.
- Values at Atom Positions 창의 맨 아래에 있는 Histogram 버튼을 사용하면 Render/Select by Attribute라는 새 창에 히스토그램이 만들어집니다. 히스토그램을 Ctrl + 클릭하면 다른 색상을 선택할 수 있는 컷오프 포인트가 추가되거나 제거됩니다. Keep Opaque를 선택 취소하여 장면에 투명한 색상을 적용합니다. 갭을 나타내는 주어진 임계값 미만의 밀도를 가진 마커는 색상을 지정할 수 있을 뿐만 아니라 선택할 수도 있으므로 갭 영역의 숫자 마커를 계산할 수 있습니다. 선택하려면 > 속성 선택 > value_mapname 클릭합니다.
- inter-actin 거리 결정
- 액틴 간 거리를 측정하려면 볼륨 추적기 창을 사용하여 마우스를 클릭하고 데이터 평면에 표식기 배치만 선택하고 선택한 표식기에 새 표식기 연결을 선택 취소하여 단일 XZ 평면(UCSF Chimera에서는 Y 평면이라고 함)에 표식기를 배치합니다. 이렇게 하면 동일한 Y-평면에 마커를 정확하게 배치할 수 있습니다(단일 Z-평면이 표시되는 경우). 또한 볼륨 트레이서 창에서 마우스 가운데 버튼을 사용하여 마커 배치를 선택하고 인터액틴 거리 측정을 위해 관심 있는 대상 Y 평면에 마커를 배치합니다.
- 단일 Y-plane의 액틴 위치가 성공적으로 표시되면 모든 마커를 선택하고 actin 필라멘트 평면 하단의 명령줄에 findclash #model-spec test self overlapCutoff -200 hbondAllowance 0 log true Linewidth 10 pbColor black 명령을 입력합니다. 이렇게 하면 200Å(overlapCutoff -200) 거리 내에 있는 모든 마커 간 간섭이 검색되고 마커 사이에 두께가 10Å(Linewidth 10)인 검은색 선(pbColor black)이 생성됩니다.
- 주어진 200 Å 범위 내에 겹치는 마커가 있으므로 가장 가깝고 인접한 마커 사이의 링크가 아닌 중복 측정을 제거합니다. UCSF Chimera 웹 사이트에 제공된 RemoveCross.py 스크립트를 사용하여 이 작업을 수행합니다. 이 스크립트는 인접 마커와 함께 가장 짧은 링크가 아닌 모든 겹치는 링크를 제거합니다.
- 남아 있는 본드의 거리를 측정하려면 모든 유사 본드(findclash에 의해 생성된 링크)를 선택하고 runscript pblengths.py 를 입력하여 pblengths.py python 스크립트를 사용합니다. Reply Log(응답 로그)에서 측정된 값을 확인합니다(Tools > Utilities > Reply Log). 그런 다음 회신 로그에서 얻은 값을 복사하고 스프레드시트 또는 기타 적절한 도구로 전송하여 히스토그램을 만듭니다.
결과
유리체 얼음에 박힌 염색되지 않고 얼어붙은 수화된 개별 입체섬모의 초저온 전자 단층촬영을 사용하여 교차 커넥터 단백질23으로 연결된 육각형으로 배열된 액틴 필라멘트가 있는 액틴 다발의 밀도 지도를 얻었습니다. 개별 복셀의 치수는 0.947nm였습니다. 전체 단층촬영(400 슬라이스/379 nm)의 볼륨 렌더링에 대한 IMOD 슬라이서 프로그램의 육안 검사는 세로 보기(XY 평면; 그림 1A-C, 상단 패널) 및 단면도(XZ-평면; 그림 1A-C, 하단 패널). 400 슬라이스/379nm 필라멘트 네트워크를 통한 프로젝션 뷰는 원래 재구성된 볼륨을 X축을 중심으로 -6°, Y축을 중심으로 -13.5°, Z축을 중심으로 5°까지 회전할 때 가장 선명해졌습니다. 이 각도에서 모든 필라멘트는 서로 위에 정렬되므로 단면도에서 알 수 있듯이 대비가 최대입니다(그림 1B). 단일 단면 슬라이스는 액틴 필라멘트를 명확하게 구별할 수 있는 충분한 신호를 가지고 있지 않기 때문에 단면도에서 육각형 패턴을 명확하게 보여주는 30 슬라이스/28.4nm의 슬래브를 볼륨 렌더링하기로 결정했습니다. 그림 1C(상단 패널)의 파란색 선은 하단 패널에서 해당 30개 슬라이스/28.4nm 단면 슬래브의 중간 위치를 나타냅니다.
이 최적 시야각에서 ±2°만으로도 액틴 필라멘트 네트워크의 인지된 순서가 크게 감소했으며(그림 1A, C), 이는 단층 촬영의 3D 볼륨에서 얼마나 쉽게 길을 잃을 수 있는지를 나타냅니다.
유역 세분화와 같은 자동화된 세분화 접근 방식을 사용하는 데 따르는 어려움을 설명하기 위해 UCSF Chimera 소프트웨어 패키지(Tools > Volume Data > Segger > Segment)에서 구현된 대로 유역 세분화를 위해 작은 하위 볼륨(금으로 표시)을 선택했습니다. 전체 입체섬모 맵에 상대적인 하위 볼륨의 위치는 그림 1B의 작은 삽입으로 표시됩니다.
그림 1D-F는 서로 다른 방향에서 선택된 하위 부피를 보여주며, 그림 1D, E는 세로 시야 방향을 나타내고 그림 1F는 단면 시야 방향을 나타냅니다. 그림 1D-F의 왼쪽에 있는 화살표는 액틴 필라멘트의 방향을 나타냅니다.
그림 1D-F(오른쪽 패널)는 유역 세분화의 결과를 보여줍니다. 하위 볼륨은 개체 ID에 따라 색상으로 구분되며, 색상은 다른 개체에 무작위로 할당됩니다. 색상이 다르면 물체 정체성이 다르다는 것을 나타내므로 그림 1D-F에서 필라멘트의 맵 밀도는 모두 필라멘트 축을 따라 조각화되는 반면, 동일한 색상과 객체 정체성은 인접한 필라멘트를 연결하는 밀도를 매핑하기 위해 주어졌음을 알 수 있습니다. 즉, 유역 분할 알고리즘은 장기간 동안 액틴 필라멘트의 밀도 맵을 따라갈 수 없었고, 대신 이웃 필라멘트의 밀도를 연결하도록 유도했습니다. 선택 영역을 수동으로 선별하는 것(예: 개체 삭제 또는 병합)이 가능하지만 이 접근 방식은 다소 노동 집약적이므로 시간이 많이 걸립니다.
체적 모델 구축 전략이 반드시 필요한 것은 아니지만, 액틴 필라멘트 네트워크 축이 Y축에 정렬되고 액틴 필라멘트 모델 평면이 단층 촬영의 X-Y 평면에 정렬되도록 3D 맵의 방향을 변경(회전)하는 데 도움이 되었습니다. 우리는 이 방향을 입체 섬모 단층 촬영 디스플레이의 표준 방향이라고 합니다.
따라서 우리는 액틴 필라멘트가 규칙적인 간격과 정의된 전체 번들 방향을 가진 전체적으로 규칙적인 조직(육각형 패킹)을 나타낸다는 사실을 활용하여 이미지 분할을 위한 다른 전략을 모색하기로 결정했습니다. 우리의 전략은 필라멘트 배열로 액틴 다발의 모델의 전체 적합도를 찾은 다음, 실험 밀도 맵에 맞게 모델 위치를 지역적으로 조정한 다음 국부적으로 조정하는 것이었습니다. 전체 모델을 먼저 배치하면 로컬 맵의 모호성을 극복하고 필라멘트 굽힘과 같은 원래 조직에서 모델의 편차에 대한 지역적 추세를 감지할 수 있습니다.
모델을 배치하기 위해 표준 방향에서 액틴 필라멘트의 단일 층 두께에 해당하는 밀도(10 슬라이스/9.47nm)의 슬래브를 표시했으며, 여기에 규칙적인 간격의 직선 액틴 필라멘트 모델 층이 장착되었습니다. 이것은 물론 액틴 필라멘트를 지나치게 단순화한 것이며, 각 필라멘트는 나선형 대칭을 가진 액틴 단량체의 선형 배열로 구성됩니다. 그림 2A-C는 서로 다른 Z-높이에 있는 세 개의 대표 층을 보여주며, 빨간색 막대는 액틴 필라멘트를 나타냅니다. ~30 슬라이스/28.4 nm 두께의 단면을 묘사하는 상단 패널은 19개의 막대로 구성된 개별 액틴 모델 레이어가 배치된 Z 높이를 보여주는 반면, 하단 패널은 세로 방향을 보여줍니다(투시도로 표시되었지만). 그림 2D는 횡단면(상단 패널)과 세로 투시도(하단 패널) 모두에서 완전히 단순화된 모델을 보여줍니다. 단면 방향을 통해 자신 있게 필라멘트를 배치할 수 있었습니다. 여기서, 전체 볼륨의 방향을 단층도(tomograms) 장축과 일치시키도록 재배치한 원래의 움직임은 표준 시청 방향에서 모델의 방향이 주축과 평행하다는 것을 의미했기 때문에 도움이 되는 것으로 판명되었습니다. 그러나 엄밀히 말하면 우리의 접근 방식은 단층 촬영의 방향을 바꾸지 않고도 효과가 있었을 것이며, 모델을 밀도에 배치하는 것만으로도 더 어려웠을 것입니다.
밀도 맵의 개별 슬래브를 주의 깊게 조사한 결과, 완벽하게 직선인 액틴 모델이 입체섬모의 근위부 끝에서 원위 끝(즉, 끝 쪽으로)으로 이동하는 관찰된 밀도 맵에 맞지 않는다는 것을 발견했습니다(그림 3A-C). 입체섬모의 끝 부근에서 필라멘트의 맵 밀도는 13nm 이상(액틴-액틴 간격) 변위되었는데, 이는 입체섬모 밀도 맵의 근위부에서 원위 부분으로 이동하면서 모델을 조정하여 보상할 수 있었으며, 따라서 작지만 구별 가능한 점진적인 곡률을 액틴 모델에 도입했습니다. 그림 3D는 밀도 맵에 맞는 체적 모델과 함께 액틴 필라멘트의 맵 밀도의 단일 슬래브를 보여줍니다. 직선(빨간색)과 곡선(노란색) 모델의 비교가 그림 3E에 나와 있습니다. 이 곡률은 배치된 모델이 있는 밀도 맵의 슬래브를 X축을 중심으로 80° 기울일 때 가장 잘 알 수 있으며, 이를 통해 액틴 필라멘트의 방향을 따라 투시도를 볼 수 있습니다(그림 3D, E).
팁 근처의 액틴 모델의 위치가 액틴 필라멘트의 간격과 거의 같은 거리만큼 이동되는 두 모델의 편차는 우리가 한 방식으로 진행하지 않았다면 많은 혼란을 야기했을 것입니다. 액틴 필라멘트 모델 레이어의 이러한 "전역적" 포지셔닝과 "지역적" 조정을 통해 세로 또는 단면 보기에서 거의 눈에 띄지 않는 이 곡률을 감지할 수 있었습니다. 그러나 그림 3E와 같이 두 모델을 오버레이하면 미묘한 차이가 드러납니다.
여러 레이어에 대해 이 접근 방식을 반복하면 완전한 3D 모델을 얻을 수 있으며(그림 3F), 단면 방향에서 볼 때 입체섬모의 맨 위와 맨 아래에 있는 데이터 불확실성에 의해서만 제한됩니다(그림 3G). 이러한 밀도 부족은 (단일 축) 단층 촬영 데이터 수집에서 누락된 쐐기 및 해당 데이터 해상도 이방성으로 인해 발생하며, 그 영향은 입체섬모막에 대한 잘 정의된 맵 밀도가 없는 것으로 표시됩니다.
3D 모델이 준비되면 해당 위치의 지도 밀도 값에 따라 체적 모델의 각 위치를 색상으로 구분했습니다. 기본이 약한 지도 밀도가 있는 모델 영역은 빨간색으로 표시한 반면, 강한 지도 밀도 신호가 있는 모델 영역은 노란색으로 표시했습니다(그림 4A). 우리는 수십 나노미터까지 확장될 수 있는 이러한 붉은색 영역을 액틴 필라멘트 구조의 갭으로 해석하며, 그 범위로 인해 Cryo-EM 맵의 고노이즈 환경에서 자주 발생하는 밀도 변화에 기인할 수 없습니다. 소음은 개별 복셀 또는 작은 복셀 그룹에 영향을 미치는 경향이 있지만, 필라멘트 밀도가 누락된 수백 개의 복셀로 구성된 볼륨의 원인이 될 가능성은 거의 없습니다. 대신, 이러한 갭은 입체섬모 액틴 그물망의 실제 특징일 가능성이 높으며 액틴 회전율의 위치를 구성할 수 있습니다. 그림 4A 에는 연한 파란색과 진한 파란색으로 표시된 두 가지 다른 맵 밀도 값이 있습니다. 밀도가 약한 영역에서 모델의 자동화된 색상 코딩과 결합된 체적 모델 구축 접근 방식은 그렇지 않으면 매우 어려웠을 액틴 필라멘트 모델에서 이러한 간격의 분포를 감지하고 시각화하는 빠르고 편리한 방법이라는 점에 명시적으로 유의해야 합니다.
그림 4B에서 볼 수 있듯이, 밀도가 비교적 약한 위치에 있는 체적 모델의 일부는 그림 4A에서 얻은 결과를 기반으로 쉽게 숨길 수 있습니다. 그러면 입체섬모에서 액틴 모델을 보다 사실적으로 묘사할 수 있는 더 파편화된 모델이 생성됩니다. 액틴 필라멘트를 작게 만드는 대안은 매우 노동 집약적이었을 것이며 그림 1을 설명할 때 논의된 문제로 인해 완전히 실패했을 수 있습니다.
또한 체적 모델을 사용하면 교차 연결의 양쪽에 있는 액틴 필라멘트 모델의 모델 포인트 위치 사이에 연결(빨간색으로 표시)을 배치하기만 하면 교차 커넥터를 쉽게 모델링할 수 있습니다(그림 4C). 당사의 단순화된 접근 방식에서는 각 교차 결합 단백질의 정확한 정체에 대해 어떠한 가정도 할 필요가 없으며, 이를 위해서는 더 높은 분해능 및/또는 정교한 라벨링 접근 방식이 필요합니다. 대신, 우리가 결정해야 할 것은 인접한 액틴 필라멘트를 연결하는 밀도가 존재하는지 여부입니다. 있는 경우 한 필라멘트에서 인접한 필라멘트로 짧은 연결을 배치할 수 있습니다. 그림 4D에는 교차 커넥터가 있는 5개의 액틴 필라멘트 모델이 표시되어 있으며, 이는 액틴 필라멘트 축을 따라 교차 커넥터의 분포를 보여줍니다.
액틴 다발의 체적 모델을 구축하는 또 다른 이점은 인접한 액틴 필라멘트 사이의 간격을 빠르게 결정할 수 있다는 것입니다(그림 4E-H). 그림 4E,F는 맵 밀도의 육각형 격자에 각각 모델이 맞지 않은 경우와 있는 밀도 맵의 단면도를 보여줍니다. 그림 4G는 가장 가까운 이웃 공 사이의 연결이 있는 모델을 보여줍니다. UCSF Chimera를 사용하면 가장 가까운 이웃 중심의 거리를 자동으로 계산할 수 있으며, 그 결과를 거리 분포로 표시할 수 있습니다(그림 4H). 두 개의 추가 데이터 세트에 대한 모델 구축은 보충 그림 1 및 보충 그림 2에 나와 있습니다.
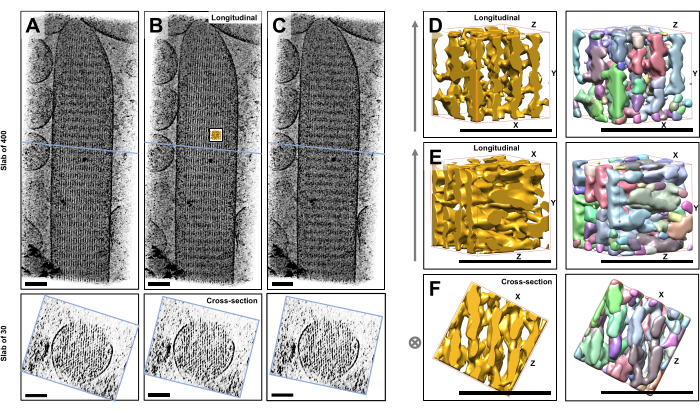
그림 1: 유모 세포 입체 토모그램의 유역 분할이 직면한 과제. (A-C) XY 평면(상단 패널)의 단층 촬영 3D 맵을 통한 세로 투영(400 슬라이스/379 nm) 및 XZ 평면(하단 패널)의 단면도(30 슬라이스/28.4 nm). (A) 최적 방향에서 Y축을 따라 -2° 회전한 단층 촬영 맵. (B) X축, Y축, Z축(X = -6°, Y = -13.5°, Z= 5°)의 회전 각도 조정에 의해 결정되고 밀도 맵에서 높은 차수를 나타내어 고도로 정렬된 액틴 필라멘트 네트워크를 제안하는 최적 방향의 단층 촬영 맵. (C) 최적 방향에서 Y축을 따라 +2° 회전한 단층 촬영 맵; 최적의 보기 방향에서 Y축을 중심으로 2°만 회전하면 밀도 맵의 인식된 규칙성이 심각하게 손상됩니다. 하단 패널은 단면 방향으로 볼 때 actin 필라멘트 어레이의 규칙성을 보여줍니다. A-C의 파란색 선은 단면 슬래브의 위치를 나타냅니다. (D-F) 유역 분할 전(왼쪽 패널)과 후(오른쪽 패널)의 세 가지 다른 방향에서 본 50nm x 50nm x 50nm의 큐브. 유역 분할은 연속적인 액틴 필라멘트 밀도를 감지하지 못하는 반면, 인접한 액틴 필라멘트와 교차 연결은 동일한 객체 정체성을 공유하므로 유역 분할이 단층촬영 분할에 적합한 접근 방식이 아님을 시사합니다. 패널 D-F에서 Chimera의 밀도 맵은 맵 스타일 "Surface"로 표시됩니다. (에이씨) 스케일 바 = 100nm. (D-F) 스케일 바 = 50nm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
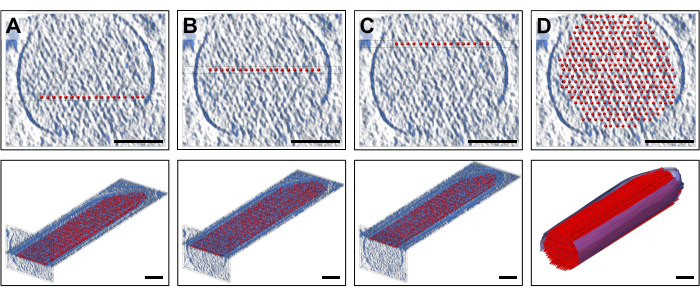
그림 2: 체적 ball-and-stick 액틴 필라멘트 모델 구축.(A-C) 상단: 다른 Z 높이에 배치된 단층 단순화된 액틴 필라멘트 모델이 있는 밀도 맵의 단면 30 슬라이스/28.4nm 보기. 아래: 단일 레이어 단순화된 액틴 필라멘트 모델이 있는 밀도 맵의 단일 10 슬라이스/9.47nm 슬래브. (D) 30 슬라이스/28.4nm 단면도(위) 및 투시도(아래)에서 완전한 직선 액틴 필라멘트 모델; 스케일 바 = 100nm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
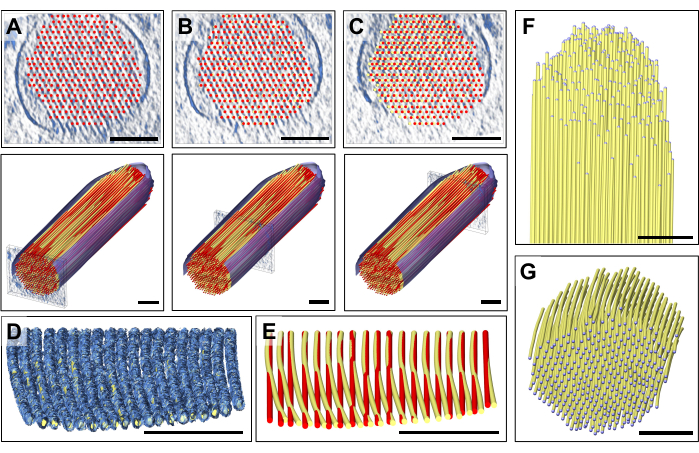
그림 3: 감지된 액틴 필라멘트 밀도의 굽힘을 보정하기 위한 모델 조정.(A-C) 단면도(상단 패널) 또는 종단면도(하단 패널)에서 모델을 주의 깊게 육안으로 검사한 결과, 입체섬모의 근위 끝에 있는 밀도 맵과 모델이 잘 맞았음이 드러났습니다. 그러나 입체섬모의 말단으로 갈수록 모든 액틴 필라멘트 모델에서 적합도가 점점 더 나빠집니다. 이는 ball-and-stick 모델의 볼을 올바른 밀도 맵 위치로 이동하여 수정할 수 있으며, 그 결과 약간 구부러진 액틴 필라멘트 모델이 생성됩니다. 직선 모델은 빨간색으로 표시되고 수정된 절곡부 모델은 노란색으로 표시됩니다. (D) 곡선 모델이 장착된 밀도 맵의 단일 슬래브, 입체섬모의 끝을 향한 액틴 밀도의 곡률을 나타냅니다. 액틴 모델은 액틴 필라멘트의 미묘하면서도 중요한 굽힘을 더 잘 보여주기 위해 X축을 중심으로 80° 회전되었습니다. (E) 빨간색으로 표시된 직선의 보정되지 않은 액틴 모델과 노란색으로 표시된 곡선의 보정된 액틴 모델 간의 비교. 명확성을 위해 액틴 필라멘트 모델의 한 층만 표시됩니다. (F-G) 세로(F) 및 단면(G) 방향으로 표시된 곡선의 수정된 액틴 필라멘트가 있는 액틴 번들 모델. 분절된 멤브레인은 파란색으로 표시됩니다. 패널 D에서 Chimera의 밀도 맵은 맵 스타일 "Mesh"로 표시됩니다. 스케일 바 = 100nm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
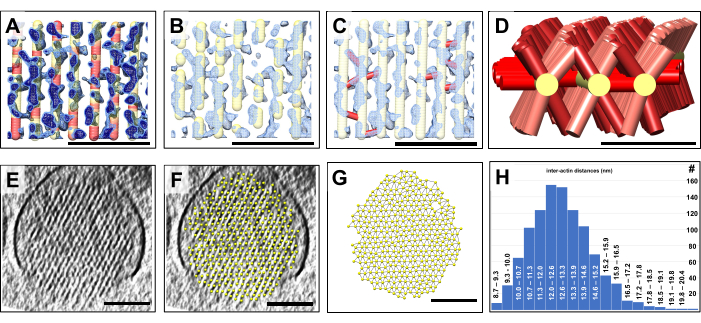
그림 4: 체적 모델 구축을 사용한 액틴 번들의 갭 감지. Ball-and-stick 모델을 사용한 체적 특성의 정량화. (에이씨) 입체섬모 밀도 맵의 ~10 슬라이스/9.47nm 두께의 세로 슬래브의 작은 영역은 곡선의 보정된 액틴 필라멘트 모델과 함께 파란색으로 표시됩니다. (A) 밀도 맵은 더 낮은 밀도 임계값(연한 파란색)과 더 높은 밀도 임계값(진한 파란색)으로 표시됩니다. 해당 밀도가 없는 액틴 필라멘트 모델의 영역이 있습니다. 이러한 위치에서 모델은 밀도 부족을 나타내기 위해 빨간색으로 색상으로 구분되었습니다. 우리는 이러한 위치를 액틴 필라멘트의 틈으로 해석합니다. (B) 단편적으로 보이는 액틴 필라멘트의 모델로, 이러한 갭 위치에서 액틴 필라멘트 밀도가 부족함을 반영합니다. (C) 인접한 액틴 필라멘트를 연결하기 위해 강한 밀도가 발견된 위치에 추가된 교차 연결 링크(빨간색으로 표시)가 있는 액틴 필라멘트 모델. (D) 3개의 선택된 모델 액틴 필라멘트가 인접한 액틴 필라멘트에 대한 교차 커넥터와 함께 표시됩니다(명확성을 위해 표시되지 않음). 전부는 아니지만 많은 가능한 인터액틴 필라멘트 위치가 커넥터 단백질에 의해 점유되고 있다는 점에 유의하십시오. (E) 입체섬모 밀도의 단면 30 슬라이스/28.4nm 슬래브. (F) 단면 30 슬라이스/28.4nm 입체섬모 밀도 슬래브에 장착된 액틴 필라멘트 모델. (G) 기본 맵 밀도가 없는 액틴 필라멘트 모델. 가장 가까운 이웃 액틴 필라멘트 거리의 자동 감지, 액틴 필라멘트 모델 볼 사이의 얇은 연결로 표시됩니다. (H) 액틴 필라멘트 모델 거리의 히스토그램. 패널 A-C에서 Chimera의 밀도 맵은 맵 스타일 "Mesh"로 표시됩니다. (에이씨) 스케일 바 = 50nm. (D) 스케일 바 = 25nm. (예) 스케일 바 = 100nm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 그림 1: 두 개의 추가 입체섬모 데이터 세트 중 첫 번째 데이터 세트에 대한 모델 구축. (에이씨) 입체섬모 밀도 맵의 ~10 슬라이스/9.47nm 두께의 세로 슬래브의 작은 영역은 메쉬 모드 디스플레이를 사용하여 파란색으로 표시됩니다. 초기에 배치된 모델은 빨간색으로 표시되고 수정된 모델은 노란색으로 표시됩니다. (A) 지도 밀도만 해당됩니다. (B) 밀도 맵에 배치된 초기 모델. (C) 밀도 맵에 배치된 수정된 모델. (D-E) (D)가 없고 (E)가 있는 더 큰 입체섬모 영역, 수정된 모델은 입체섬모 밀도 맵의 ~10 슬라이스/9.47nm 두께의 세로 슬래브에 맞았습니다. (F-G) 전체 입체섬모 단층촬영 영역이 도시되어 있습니다. (F) 지도만 해당됩니다. (G) 수정된 모델로 매핑합니다. (H) 초기 모델과 보정된 모델의 중첩. 스케일 바 = 100nm 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 2: 두 개의 추가 입체섬모 데이터 세트 중 두 번째 데이터 세트에 대한 모델 구축. (에이씨) 입체섬모 밀도 맵의 ~10 슬라이스/9.47nm 두께의 세로 슬래브의 작은 영역은 메쉬 모드 디스플레이를 사용하여 파란색으로 표시됩니다. 초기에 배치된 모델은 빨간색으로 표시되고 수정된 모델은 노란색으로 표시됩니다. (A) 지도 밀도만 해당됩니다. (B) 밀도 맵에 배치된 초기 모델. (C) 밀도 맵에 배치된 수정된 모델. (D-E) (D)가 없고 (E)가 있는 더 큰 입체섬모 영역, 수정된 모델은 입체섬모 밀도 맵의 ~10 슬라이스/9.47nm 두께의 세로 슬래브에 맞았습니다. (F-G) 전체 입체섬모 단층촬영 영역이 도시되어 있습니다. (F) 지도만 해당됩니다. (G) 수정된 모델로 매핑합니다. (H) 초기 및 보정된 모델의 중첩. 스케일 바 = 100nm. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: 입체섬모 모델링을 위한 UCSF 키메라 파이썬 스크립트. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 코딩 파일 1: pblengths.py. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 코딩 파일 2: RemoveCross.py. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 코딩 파일 3: ActinFilamentPlane.py. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 코딩 파일 4: dividelinks.py. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 코딩 파일 5: FixingMarkerID.py. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
우리는 유역 분할(watershed segmentation)과 같은 세분화를 위한 자동화된 접근 방식이 유모 세포 입체섬모 초저온 전자 단층촬영(haircell stereocilia cryo-electron tomograms)의 고잡음 및 고복잡성 환경에서 실패할 수 있음을 보여주었습니다. 이 필라멘트 네트워크의 어느 부분이 액틴 필라멘트를 나타내고 무엇이 국소 환경 수준에서 가교를 구성하는지 구별하는 것은 작은 단층 촬영 하위 부피를 검사할 때 기껏해야 어려운 것처럼 보입니다. 이 연구에서 사용된 모델 구축 접근 방식은 액틴 다발의 대규모 차수에 대한 사전 지식을 활용하여 액틴 필라멘트의 방향과 교차결합 밀도에 대한 기대치를 개발하는 데 도움이 됩니다. 어쩌면 더 중요한 것은 인간의 뇌는 국소 밀도 분포를 넘어서는 더 큰 맥락을 고려하여 패턴을 쉽게 찾을 수 있는 반면, 컴퓨터 알고리즘은 알고리즘이 고려하는 상대적으로 작은 영역에서만 작동한다는 것입니다. 따라서 더 큰 규모의 추세는 쉽게 고려될 수 없습니다. 모델을 밀도 층에 전역적으로 맞춤으로써, 한 번에 단일 액틴 필라멘트의 작은 부분에 대한 모델을 생성하려고 할 때 발생할 수 있는 혼란을 피할 수 있었습니다. 물론, 이러한 전역 피팅은 먼 거리에 걸쳐 확장되는 순서를 가정합니다. 그러나 액틴 필라멘트가 예상치 못한 작지만 상당한 점진적 굴곡이 있었기 때문에 전체 적합은 초기 근사치일 뿐이며 밀도 맵에 맞추기 위해 모델의 로컬 조정이 필요했습니다. 초기 모델이 좋은 출발점이었기 때문에 높은 신뢰도를 가지고 조정할 수 있었습니다. 이 접근 방식의 큰 장점 중 하나는 정의된 밀도 영역만 표시하도록 선택할 수 있어 풍경의 복잡성을 줄이는 데 도움이 된다는 것입니다. 또한 필라멘트 모델 축을 따라 맵 밀도 슬래브를 보는 것은 단순히 더 작은 하위 볼륨을 표시할 때 놓쳤을 가능성이 가장 높은 예기치 않은 곡률을 식별하는 데 도움이 되었습니다. 또한 초기 모델을 배치하면 빠르게 확대 및 축소할 수 있어 각 액틴 필라멘트 레이어의 전체 보기와 세부 보기를 번갈아 가며 모델을 조정할 수 있습니다.
프로토콜 내의 중요한 단계에는 육안 검사 후 맵 회전, 밀도 맵에 모델 생성 및 배치, 필라멘트 모델을 더 작은 세그먼트로 나누는 것이 포함되었습니다. 그런 다음 세그먼트의 원자 위치를 밀도 맵에 맞게 공간적으로 조정하거나 갭을 감지하기 위해 색상으로 구분할 수 있습니다.
액틴 모델 구축의 이러한 접근 방식은 10-30 슬라이스/9.47-28.4nm 평균 밀도 슬래브의 단면도를 사용하여 일련의 "원자"(즉, 볼과 스틱 모델의 볼)를 필라멘트 밀도에 배치하여 수정할 수도 있으며, 이는 본드(즉, 볼-앤-스틱 모델의 스틱)로 연결될 수 있습니다. 우리는 유모 세포 입체섬모23의 테이퍼 영역에서 체적 모델 구축을 위해 여기에 자세히 설명된 프로토콜의 변형인 이 접근 방식을 사용했습니다. 또한, 여기에서 설명한 바와 같이, 당사의 체적 모델 구축 접근 방식은 멤브레인의 세분화 및 모델 구축에도 매우 적합합니다.
체적 모델 구축은 필라멘트 기능을 보여주는 모든 밀도 맵에 적용할 수 있지만, 여기에서 설명한 기술은 규칙적인 간격의 필라멘트 배열이 있을 때 가장 효율적이며, 이를 위해 체적 모델의 전역 적합도를 얻을 수 있습니다. 또한 점진적인 방식으로 방향성을 변경하기 위해 필라멘트 기능에 따라 달라집니다. 필라멘트 구조에 갑작스러운 꼬임과 급격한 회전이 있는 경우 당사의 접근 방식은 분할에 특별히 도움이 되지 않을 수 있습니다.
그동안 공동 연구자들은 여기에서 수동 분할에 사용된 것과 유사한 개념을 따르는 자동화된 필라멘트 추적을 위한 자동화된 접근 방식을 개발했습니다30,31. 앞으로 가장 좋은 접근 방식은 초기 희소 모델(심지어 몇 개의 공)을 수동으로 식별하고 밀도에 시작점으로 배치한 다음 검색 및 피팅 알고리즘이 필라멘트 추적을 완료하도록 하는 하이브리드일 수 있습니다.
단순화된 체적 모델은 시스템의 복잡성을 줄이고 팁 근처의 액틴 필라멘트 굽힘과 같은 특정 패턴을 더 잘 이해할 수 있도록 합니다. 또한 체적 모델을 "앵커"로 사용하여 선택한 앵커링 볼-앤-스틱 모델 주변의 밀도 영역을 표시할 수 있으며, 이를 통해 인접한 액틴 필라멘트 사이의 가교 밀도를 감지하고 시각화할 수 있습니다. 개별 필라멘트를 선택하고 밀도가 다시 표시되는 영역으로 적절한 반경을 설정할 수 있는 기능을 통해 풍경의 압도적인 복잡성을 관리 가능한 수준으로 줄일 수 있습니다.
글로벌 피팅에 대한 이 체적 모델 구축 접근 방식의 한 가지 이점은 로컬 조정이 뒤따랐는데, 액틴 필라멘트가 중단된 것으로 보이는 영역을 식별할 수 있었고 맵 밀도가 없어 액틴 필라멘트의 상당한 갭이 표시되었다는 것입니다. 볼류메트릭 볼-앤-스틱 모델을 배치했기 때문에 UCSF Chimera 소프트웨어 패키지의 루틴을 사용하여 해당 위치의 맵 밀도 값에 따라 각 모델 볼 위치를 색상으로 구분할 수 있었습니다. 이 접근 방식을 통해 액틴 다발에서 액틴 필라멘트 갭을 빠르게 검출하고 시각화할 수 있었는데, 이는 초저온 전자 단층촬영에서 발견한 생물학적으로 중요한 기능이며 기존의 분할 접근 방식으로는 검출 및 시각화하기가 매우 어려웠을 것입니다. 체적 모델의 또 다른 장점은 길이와 거리를 포함한 체적 특성을 쉽게 얻을 수 있어 실제 수치를 얻을 수 있으므로 통계 분석을 수행할 수 있다는 것입니다.
요약하면, 후속 자동화된 로컬 피팅 및 필라멘트 추적 기능에 의해 더욱 강화될 수 있는 대화형 수동 모델 포인트 배치는 전자 단층 촬영 세포 이하 부피의 시각화 및 정량 분석을 위한 다소 유망한 접근 방식입니다. 패턴 인식을 위해서는 인간 두뇌의 힘을 사용하고 모델 최적화를 위해서는 컴퓨터 과학의 힘을 사용하기 때문입니다.
공개
저자는 경쟁하는 재정적 이해관계나 다른 이해상충이 없음을 선언합니다.
감사의 말
시료 준비에 기여한 Peter Barr-Gillespie 박사와 그의 팀, 단층 촬영 데이터 수집에 기여한 Auer 실험실과 Dorit Hanein 실험실의 전 구성원에게 감사드립니다. 또한 다양한 UCSF 키메라 스크립트를 제공해 주신 UCSF Resource for Biocomputing, Visualization, and Informatics(RBVI)의 Tom Goddard에게도 감사의 말씀을 전합니다.
자료
| Name | Company | Catalog Number | Comments |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/download.html | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=win64/chimera-1.16-win64.exe | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=mac64/chimera-1.16-mac64.dmg | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=linux_x86_64/chimera-1.16-linux_x86_64.bin | |
| Excel | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Falcon II | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/accessories-em/falcon-detector.html | |
| IMOD | University of Colorado | Version 4.11.1 https://bio3d.colorado.edu/imod/download.html | |
| PC Desktop | Intel | Windows 10, ver. 22H2 | |
| PC Laptop | Gigabyte | Windows 10, ver. 22H2 | |
| Powerpoint | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Titan Krios Electron Microscope | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/transmission-electron-microscopes/krios-g4-cryo-tem.html | |
| Word | Microsoft | Version 2211 https://www.office.com/?auth=1 |
참고문헌
- Downing, K. H., Sui, H., Auer, M. Electron tomography: A 3D view of the subcellular world. Analytical Chemistry. 79 (21), 7949-7957 (2007).
- Koning, R. I., Koster, A. J. Cellular nanoimaging by cryo electron tomography. Methods in Molecular Biology. 950, 227-251 (2013).
- Asano, S., Engel, B. D., Baumeister, W. In situ cryo-electron tomography: A post-reductionist approach to structural biology. Journal of Molecular Biology. 428 (2), 332-343 (2016).
- Serwas, D., Davies, K. M. Getting started with in situ cryo-electron tomography. Methods in Molecular Biology. 2215, 3-23 (2021).
- McDonald, K. L., Auer, M. High-pressure freezing, cellular tomography, and structural cell biology. Biotechniques. 41 (2), 137-143 (2006).
- Narasimha, R., et al. Evaluation of denoising algorithms for biological electron tomography. Journal of Structural Biology. 164 (1), 7-17 (2008).
- Frangakis, A. S. It's noisy out there! A review of denoising techniques in cryo-electron tomography. Journal of Structural Biology. 213 (4), 107804 (2021).
- Frangakis, A. S., Hegerl, R. Noise reduction in electron tomographic reconstructions using nonlinear anisotropic diffusion. Journal of Structural Biology. 135 (3), 239-250 (2001).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. Journal of Structural Biology. 144 (1-2), 114-122 (2003).
- vander Heide, P., Xu, X. -. P., Marsh, B. J., Hanein, D., Volkmann, N. Efficient automatic noise reduction of electron tomographic reconstructions based on iterative median filtering. Journal of Structural Biology. 158 (2), 196-204 (2007).
- Volkmann, N. Methods for segmentation and interpretation of electron tomographic reconstructions. Methods in Enzymology. 483, 31-46 (2010).
- Böhm, J., et al. Toward detecting and identifying macromolecules in a cellular context: template matching applied to electron tomograms. Proceedings of the National Academy of Sciences. 97 (26), 14245-14250 (2000).
- Frangakis, A. S., et al. Identification of macromolecular complexes in cryoelectron tomograms of phantom cells. Proceedings of the National Academy of Sciences. 99 (22), 14153-14158 (2002).
- Lebbink, M. N., et al. Template matching as a tool for annotation of tomograms of stained biological structures. Journal of Structural Biology. 158 (3), 327-335 (2007).
- Rigort, A., et al. Automated segmentation of electron tomograms for a quantitative description of actin filament networks. Journal of Structural Biology. 177 (1), 135-144 (2012).
- Volkmann, N. A novel three-dimensional variant of the watershed transform for segmentation of electron density maps. Journal of Structural Biology. 138 (1-2), 123-129 (2002).
- Bajaj, C., Yu, Z., Auer, M. Volumetric feature extraction and visualization of tomographic molecular imaging. Journal of Structural Biology. 144 (1-2), 132-143 (2003).
- Yu, Z., Frangakis, A. S. Classification of electron sub-tomograms with neural networks and its application to template-matching. Journal of Structural Biology. 174 (3), 494-504 (2011).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Tilney, L. G., Derosier, D. J., Mulroy, M. J. The organization of actin filaments in the stereocilia of cochlear hair cells. The Journal of Cell Biology. 86 (1), 244-259 (1980).
- Tilney, L. G., Tilney, M. S., DeRosier, D. J. Actin filaments, stereocilia, and hair cells: how cells count and measure. Annual Review of Cell Biology. 8, 257-274 (1992).
- Metlagel, Z., et al. Electron cryo-tomography of vestibular hair-cell stereocilia. Journal of Structural Biology. 206 (2), 149-155 (2019).
- Song, J., et al. A cryo-tomography-based volumetric model of the actin core of mouse vestibular hair cell stereocilia lacking plastin 1. Journal of Structural Biology. 210 (1), 107461 (2020).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Agulleiro, J. I., Fernandez, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27 (4), 582-583 (2011).
- Fernandez, J. J. Computational methods for electron tomography. Micron. 43 (10), 1010-1030 (2012).
- Chen, H., Clyborne, W. K., Sedat, J. W., Agard, D. A. Priism: an integrated system for display and analysis of 3-D microscope images. Biomedical Image Processing and Three-Dimensional Microscopy. 1660, 784-790 (1992).
- Pettersen, E. F., et al. UCSF Chimera-a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25 (13), 1605-1612 (2004).
- Goddard, T. D., Huang, C. C., Ferrin, T. E. Visualizing density maps with UCSF Chimera. Journal of Structural Biology. 157 (1), 281-287 (2007).
- Sazzed, S., et al. Tracing actin filament bundles in three-dimensional electron tomography density maps of hair cell stereocilia. Molecules. 23 (4), 882 (2018).
- Sazzed, S., Scheible, P., He, P., Wriggers, J. Spaghetti tracer: A framework for tracing semiregular filamentous densities in 3D tomograms. Biomolecules. 12 (8), 1022 (2022).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유