Method Article
Упрощенные волюметрические модели как эффективная стратегия сегментации актиновых сетей в криоэлектронных томограммах
В этой статье
Резюме
Здесь мы представляем протокол размещения упрощенных объемных моделей в шумные, сложные, томографические 3D-объемы. Это позволяет быстро сегментировать плотности актиновых филаментов, обнаруживать систематические изгибы филаментов и разрывы в филаментах пучка волос, а также удобную количественную оценку объемных свойств модели, таких как расстояния.
Аннотация
Эффективные методы извлечения интересующих признаков остаются одной из самых больших проблем при интерпретации криоэлектронных томограмм. Были предложены различные автоматизированные подходы, многие из которых хорошо работают для высококонтрастных наборов данных, где интересующие особенности могут быть легко обнаружены и четко отделены друг от друга. Наши наборы данных криоэлектронной томографии стереоцилии внутреннего уха характеризуются плотным массивом гексагонально упакованных актиновых филаментов, которые часто связаны друг с другом. Эти особенности делают автоматическую сегментацию очень сложной, что еще больше усугубляется высоким уровнем шума криоэлектронных томограмм и высокой сложностью плотно упакованных элементов. Используя предварительные знания об организации актиновых пучков, мы разместили слои сильно упрощенной модели актина «шарик и палочка», чтобы сначала получить глобальное соответствие карте плотности, а затем провести региональные и локальные корректировки модели. Показано, что построение объемной модели не только позволяет справиться с высокой сложностью, но и обеспечивает точные измерения и статистику по актиновому расслоению. Объемные модели также служат точками привязки для локальной сегментации, например, в случае перекрестных соединителей актин-актин. Построение объемных моделей, особенно когда оно дополняется компьютерными автоматизированными подходами к подгонке, может быть мощной альтернативой, когда традиционные автоматизированные подходы к сегментации не увенчались успехом.
Введение
Криоэлектронная томография позволяет визуализировать целые органеллы или части клеток и тканей с нанометровым разрешением в их состоянии, близком к нативному1,2,3 с использованием либо врезной заморозки4, либо сверхбыстрой витрификации с замораживанием под высокимдавлением5. Поскольку криоконсервированный, неокрашенный, замороженно-гидратированный образец может переносить только ограниченную дозу электронов, томографические 3D-данные очень зашумлены. Этот шум часто может быть значительно уменьшен с помощью различных алгоритмов фильтрации шума 6,7, включая нелинейную анизотропную диффузию8, билатеральную фильтрацию9 и рекурсивную медианную фильтрацию10.
Кроме того, ограничения по наклону предметного столика микроскопа, приводящие к отсутствующему клину информации, и тот факт, что толщина образца увеличивается при больших углах наклона, приводят к 3D-реконструкции с анизотропным разрешением. Это означает, что плотность размазывается в третьем измерении из-за более низкого разрешения в направлении Z. В результате форма макромолекул выглядит искаженной (т.е. менее четко очерченной и вытянутой в третьем измерении).
Одной из самых больших проблем при интерпретации томографических данных является автоматизированное извлечение соответствующих признаков, также известное как сегментация11. Обладая достаточными уникальными характеристиками формы и низким уровнем шума, высокомолекулярные машины в сложных 3D-объемах могут быть идентифицированы путем сопоставления шаблонов 12,13,14; Однако успех сопоставления шаблона зависит от разрешения томограммы, подходящей модели поиска, а также размерных и форменных характеристик объемов признаков. Если интересующие нас особенности расположены на достаточном расстоянии друг от друга и можно легко идентифицировать повторяющиеся мотивы (например, большие макромолекулярные машины), субобъемы томограмм могут быть объединены для увеличения отношения сигнал/шум и усреднения искажений формы отдельных частиц. Сообщалось об автоматизированной сегментации сети актиновых филаментов в электронных томограммах тонкого края замороженно-гидратированных клеток Dictyostelium discoideum путем сопоставления матриц15.
Однако, если интересующие нас объекты расположены близко друг к другу, анизотропия разрешения данных может привести к размазыванию плотностей карты в направлении Z (вдоль направления электронного пучка), что приводит к очевидному слиянию плотностной оболочки близко расположенных высокомолекулярных машин или супрамолекулярных комплексов. В таких случаях автоматизированные подходы к сегментации, такие как водораздел16, сегментация границ17 или различные подходы к классификации18,19 на основе машинного обучения, могут оказаться не в состоянии распознать интересующие их особенности или установить правильную границу вокруг интересующего объекта. Часто получается либо несколько очень больших частей, либо сильно раздробленный объем, где требуется много усилий, чтобы объединить множество мелких частей до тех пор, пока интересующая вас функция не будет восприниматься как завершенная. Такое ручное курирование результатов сегментации может быть очень трудоемким и даже может привести к полному краху, если интересующая нас структура представляет собой массив близко расположенных филаментов, которые соединены между собой с помощью коротких линкеров. В этой гигантской сети нитевидных структур может быть трудно сориентироваться. Это связано с тем, что из-за анизотропии разрешения плотности смешиваются друг с другом, что представляет собой сложную проблему как для автоматизированной, так и для интерактивной ручной сегментации. Как следствие, можно легко «прыгать» между нитями, только визуально рассматривая небольшие участки.
К счастью, в случае пучка актина в стереоцилии волосковых клеток внутреннего уха мы располагаем знаниями об общей организации актинового пучка и направленности актиновых филаментов 20,21. Актиновый пучок состоит из сотен гексагонально, плотно упакованных актиновых филаментов диаметром 6-8 нм, которые расположены на расстоянии около 12-13 нм друг от другана 22 нм.
Это позволило нам использовать совершенно иной подход к сегментации, основанный на упрощенных моделях шариков и палочек для представления актиновых филаментов. Стратегия заключалась в одновременном размещении идеализированного регулярного массива моделей филаментов в пластинах карт плотности криоэлектронной томографии для создания 3D-модели актинового пучка слой за слоем. Мы убедились, что модель в целом соответствует карте плотности, прежде чем вносить локальные корректировки в отдельные модели нитей накала или группы моделей нитей накала, чтобы они точно соответствовали карте плотности. С помощью автоматического цветового кодирования значения плотности карты в месте расположения модели филамента мы смогли легко обнаружить видимые пробелы в актиновом пучке. Объемные модели позволяют проводить количественный анализ объемных свойств, таких как расстояния между актиновыми филаментами, а также приводят к упрощенному отображению общей организации 3D-филаментной сети.
Кроме того, модели могут также служить в качестве якорных структур для сегментации дополнительных признаков, таких как актин-актиновые линкеры, поскольку могут быть выбраны (части) отдельных моделей филамента, вокруг которых могут быть сгенерированы соответствующие зоны плотности карты радиуса для проверки и дальнейшей сегментации.
Мы считаем, что наш подход к сегментации, основанный на объемных моделях, особенно полезен для сетей с большими нитчатыми структурами, которые могут содержать разрывы и межфиламентные перекрестные соединения. Алгоритмы сегментации, как правило, работают локально, в то время как человеческий мозг учитывает большие площади и, таким образом, превосходит компьютеры, когда дело доходит до распознавания филаментных структур даже в сложной среде с высоким уровнем шума.
протокол
Протокол соответствует руководящим принципам комитета по этике исследований человека Юго-Восточного университета.
1. Источник данных криоэлектронной томографии для построения объемных моделей
ПРИМЕЧАНИЕ: Криоэлектронные томографические реконструкции стереоцилии, использованные для построения объемной модели, были ранее опубликованы22,23 и были получены в том виде, в котором они описаны Metlagel et al.22.
Скрипты Python UCSF Chimera для моделирования стереоцилии представлены в Дополнительном файле 1, Дополнительном файле кодирования 1, Дополнительном файле кодирования 2, Дополнительном файле кодирования 3, Дополнительном файле кодирования 4 и Дополнительном файле кодирования 5.
- Вкратце, нанесите стереоцилию с апикальной поверхности сенсорного эпителия утрикула мыши на кружевную углеродную пленку-подложку сетки электронного микроскопа (ЭМ). Затем проводят сверхбыструю вскрытие методом плунжерной заморозки и сбор данных с помощью одноосевой криоэлектронной томографии на криогенной просвечивающей электронной микроскопии (крио-ПЭМ), работающей при напряжении 300 кВ с номинальной расфокусировкой 3,5-4,5 мкм, с помощью камеры типа CMOS в режиме интегрирования, с размером пикселя от 0,47 до 0,59 нм (см. таблицу материалов).
- Типичная доза для одноосевого сбора данных обычно составляет 80-100 электронов/Å2. Реконструируйте 3D объемы томограммы с помощью программного комплекса IMOD24, либо методом взвешенной обратной проекции, либо методом SIRT 25,26.
- Шумоподавление томограмм с помощью рекурсивной медианы или билатеральной фильтрации в Priism27 или опции нелинейной анизотропной диффузии в IMOD.
ПРИМЕЧАНИЕ: Ключевым программным пакетом, используемым для интерактивной визуализации, построения объемных моделей и количественного анализа, было программное обеспечение UCSF Chimera28,29. Все используемые программные пакеты перечислены в Таблице материалов.
2. Подготовка данных криоэлектронной томографии для построения объемной модели
- Вращение томограммы
ПРИМЕЧАНИЕ: Цель этого шага состоит в том, чтобы найти «оптимальные углы», под которыми ядро актина выровнено по двум из трех осей (x и z), чтобы размещение моделей в 3D могло быть выполнено только с одной осью, о которой нужно беспокоиться. Следующие шаги выполняются в программном пакете IMOD.- Откройте 3dmod (IMOD) и откройте файл стека 3D-изображений в формате .mrc. Убедитесь, что 3D-стек находится в 16-битном режиме оттенков серого и отображает правильные размеры X, Y, Z в заголовке файла изображения. При необходимости внесите исправления с помощью команды alterheader : alterheader -d (x_pixelspacing),(y_pixelspacing),(z_pixelspacing) inputfilename.mrc в командной строке. Если 3D-стек имеет формат файла TIFF, используйте команду tif2mrc inputfilename.tif outputfilename.mrc в командной строке для создания файла .mrc.
- Визуально осмотрите томограмму и откройте «срез» (нажав обратную косую черту "\"), чтобы найти наилучшие углы поворота в размерах X, Y и Z для выравнивания плоскости актинового филамента с плоскостью Z. Манипулируйте стержнем вращения X, Y и Z , чтобы найти оптимальный угол, под которым актиновые филаменты выравниваются в X- и Z-плоскостях. Обратите внимание, что изменение средней толщины для отображения томограммы (Thick : Img) может помочь в поиске идеальной величины усреднения плотности в Z-плоскости и, таким образом, увеличения контраста. Увеличивайте и уменьшайте масштаб томограммы, используя клавиши + и - на клавиатуре соответственно.
- После того, как идеальные углы поворота определены, поверните томограмму, введя в командную строку команду IMOD rotatevol -a (Z углов поворота),(Y углов поворота),(X углов поворота) -s (x-width),(y-height),(z-depth) inputfilename.mrc outputfilename.mrc в командной строке. Убедитесь, что на карте достаточно места для поворота, чтобы избежать отрезания частей карты при повороте.
ПРИМЕЧАНИЕ: Для получения дополнительной информации о том, как использовать команду rotatevol, воспользуйтесь опцией в меню справки или перейдите по следующему URL-адресу (https://bio3d.colorado.edu/imod/doc/man/rotatevol.html). Эта команда (rotatevol) не изменяет отображаемую в данный момент карту в окне IMOD, а скорее создает новый файл карты с поворачиваемой картой плотности. - С помощью окна среза определите координаты X, Y и Z двух противоположных углов карты, чтобы определить область обрезки, содержащую стереоцилию, с помощью команды trimvol .
- Используйте команду IMOD Trimvol для обрезки карты по ранее полученным координатам, набрав в командной строке trimvol -x (x-coordiante 1),( x-coordiante 2) -y (y-coordiante 1),( y-coordiante 2) -z (z-coordiante 1),( z-coordiante 2) inputfilename.mrc outputfilename.mrc в командной строке. Теперь карта стала намного меньше, и поэтому с этого момента работать с ней проще и быстрее.
ПРИМЕЧАНИЕ: Для получения дополнительной информации о том, как использовать команду trimvol, воспользуйтесь опцией в меню справки или посетите следующий URL-адрес (https://bio3d.colorado.edu/imod/doc/man/trimvol.html). В результате этих действий создается файл .mrc, который был повернут и обрезан.
- Фильтрация томограмм
ПРИМЕЧАНИЕ: На этом этапе используется фильтр нелинейной анизотропной диффузии (NAD), предоставленный IMOD для снижения шума. Мы использовали в качестве начальных значений те, которые рекомендованы на странице справки IMOD для нелинейной анизотропной диффузионной фильтрации, как указано в следующем URL (https://bio3d.colorado.edu/imod/doc/NADexample.html), и были использованы в качестве начальных значений.- Введите etomo в командной строке, чтобы запустить графический пользовательский интерфейс etomo IMOD.
- Выберите опцию Нелинейная анизотропная диффузия в меню этомо и выберите файл карты (Выберите объем).
- Чтобы найти значение K и количество итераций, которые обеспечивают наилучшие результаты фильтрации с точки зрения визуального осмотра, примените фильтр NAD к меньшему тестовому объему, чтобы извлечь указанный тестовый объем NAD-фильтра. Нажмите на инструмент Rubberband в верхней части интерфейса 3dmod, щелкните левой кнопкой мыши по перетаскиванию, чтобы выбрать тестовый объем, и нажмите кнопки Hi и Lo , чтобы указать, что Z-срез является границей тестового объема (Get Test Volume Range from 3dmod). Извлеките тестовый объем, нажав кнопку «Извлечь тестовый объем».
- Отфильтруйте тестовый объем с помощью различных значений K (Найти значение K для тестового объема > Список значений K), таких как 0,1, 1,5, 10, 15, 25, 30, 50 и 75. Выполните фильтрацию NAD с каждым из указанных значений K (Запуск с разными значениями K > Просмотр результатов теста с разными значениями K). Используйте окно среза для оценки производительности фильтра NAD и выберите значение K, которое будет использоваться для фильтрации всего объема.
- Используйте значение K, определенное на шаге 2.2.4, и протестируйте разное количество итераций, например 2, 5, 8, 11, 15 и 21 (Найдите номер итерации для параметра Test Volume > List of Iterations). Оцените производительность фильтра NAD для различных итераций, щелкнув «Просмотреть результаты теста на разных итерациях » или с помощью среза. Выберите значение для другого количества итераций.
- Используйте идентифицированное значение K и количество итераций (Filter Full Volume > K value and Iterations) и отфильтруйте весь объем (Filter Full Volume). Вновь отфильтрованный том появится в том же каталоге, где находится загруженный полный том, с расширением файла .nad, прикрепленным к файлу .mrc.
ПРИМЕЧАНИЕ: Эти шаги создают файл .mrc, который был отфильтрован с помощью фильтра NAD и отображает более низкий уровень шума и, следовательно, улучшенное соотношение сигнал/шум.
3. Построение объемной модели
- Подготовка к размещению 3D модели
ПРИМЕЧАНИЕ: Целью данного шага является подготовка и анализ карты для размещения 3D объемных моделей в карте плотности с вращением и шумоподавлением с помощью программы UCSF Chimera.- С помощью окна среза получить вид поперечного сечения (плита 30 срезов/28,4 нм) для определения Z-координат центра плотностной плиты, в которую будет помещена модель актинового филамента. Нажмите на центр плотности нити накала и обратите внимание на Z-координаты в главном окне IMOD.
- Запишите координаты, определенные для будущего размещения модели в UCSF Chimera.
ПРИМЕЧАНИЕ: Z-координаты, отмеченные на этом этапе, используются на шаге 4 для легкого поиска и размещения готовых моделей актиновых филаментов на правильных Z-высотах, что ускоряет размещение моделей.
- Размещение 3D моделей
ПРИМЕЧАНИЕ: Целью этого шага является размещение 3D объемной модели в подготовленной карте плотности с помощью UCSF Chimera.- В UCSF Chimera откройте отфильтрованную, повернутую карту (Файл > Открыть)
- Проверьте, правильно ли заданы параметры карты, выбрав Объекты > плоскости и Координаты объекта > в Просмотрщике объемов, чтобы открыть интерфейс функций и координат. Проверьте размер воксела , чтобы узнать, правильно ли задан интервал вокселов с помощью команды header в командной строке (в IMOD). Если нет, настройте интерфейс Voxel Size на правильный интервал вокселов. Карта центрируется по центру, чтобы установить камеру в центре окна просмотра (Origin Index > Center).
- Откройте окно «Управление камерой» (Инструменты > Управление просмотром > камеры). Затем установите ортогональный вид камеры (проекционный > ортогональный)
- Загрузите готовую модель скрипта Python (Файл > Открыть > ActinFilamentPlane.py) и откройте столько скриптов плоскости актинового филамента, сколько есть плоскостей актинового филамента на карте.
- Переместите загруженные модели, включив режим мыши для движения (Инструменты > Движение > Режим мыши для движения). Включите опцию перемещения мыши (Set Moving Mouse Mode > Move Selection). Используйте Ctrl + Drag для множественного выбора любых моделей, присутствующих в сеансе, и щелкните правой кнопкой мыши (или любой другой клавишей, указанной в разделе Настройки > мыши), чтобы переместить модели в нужном направлении.
ПРИМЕЧАНИЕ: Модели были созданы с шариками и палочками определенной толщины (радиус ActinFilamentPlane.py шрифта) и с определенными интервалами (Lattice_Spacing ActinFilamentPlane.py шрифта). - Чтобы визуализировать только ту модель, которая вас интересует, откройте Панель модели (Инструменты > Общие элементы управления > Панель модели). Установите флажки в разделе S (Показать), чтобы включить или отключить видимость данной модели.
- Активируйте панель командной строки (Сервис > Общие элементы управления > Командная строка).
- После размещения каждой модели плоскости актинового филамента на ее надлежащую Z-высоту, удалите все лишние актиновые филаменты в каждой модели плоскости актинового филамента, выбрав лишнюю часть моделей и набрав del sel (Удалить выбранное) в командной строке в нижней части главного окна просмотра UCSF Chimera.
- Визуализируйте только одну плоскость актинового филамента за раз, перейдя в окно «Панель моделей » и выбрав и скрыв все остальные модели актиновых филаментов (Ctrl + Click > Hide).
- Чтобы визуализировать участки плотности карты, которые находятся в непосредственной близости от интересующей модели плоскости актинового филамента, выберите модель (Model Panel > select) и откройте панель управления зонами в Volume Viewer (Volume Viewer > Features > Zone). Установите радиус зоны равным 100 Å, затем нажмите Zone , чтобы зонировать карту в радиусе 100 Å (= 10 nm) от выбранной модели. Обратите внимание, что размерность в Химере задается в Ангстреме (1 Å = 0,1 нм).
- При необходимости настройте модель в соответствии с картой плотности, включив режим движения мыши. Множественный выбор моделей, которые не находятся в правильном положении для перемещения моделей, которые должны быть правильно размещены внутри карты плотности зон.
- Чтобы устранить ошибку программирования в ActinFilamentPlane.py, запустите скрипт FixingMarkerID.py , который исправляет отсутствующий MarkerID «атомов» (мячей в модели «шар и палка»). Выберите всю модель и введите runscript FixingMarkerID.py в командной строке (скрипт должен быть указан по правильной директории, например, C:\directory \ Script.py).
- Если карта плотности показывает, что актиновая нить изогнута, обязательно разделите прямую актиновую нить на несколько частей, чтобы учесть кривизну, выбрав нужные модели и введя dividelinks.py номер runscript. Дополнительные маркеры (заданные числом в команде) размещаются внутри актиновой модели на равном расстоянии друг от друга, образуя «стыки», в которых модель может быть согнута.
- Отрегулируйте стыки так, чтобы они отражали кривизну, перемещая дополнительные маркеры «стыков» вдоль зонированной карты с помощью режима «Перемещение мыши». При необходимости добавьте в модель дополнительные маркеры с помощью скрипта dividelinks.py .
- Чтобы убедиться, что все актиновые филаменты правильно размещены внутри актиновых плотностей, используйте полосы обрезки, щелкнув «Инструменты» > «Управление просмотром > камерой » в раскрывающемся меню UCSF Chimera, чтобы открыть окно «Управление камерой », затем выберите «Вид сбоку », чтобы увидеть две полосы обрезки (ближнюю и дальнюю плоскости отсечения) камеры. Перетащите две желтые полосы в узкую щель, щелкнув левой кнопкой мыши, и перетащите обрезную часть, используя среднюю кнопку мыши , чтобы рассмотреть модели по одному маленькому кусочку за раз.
ПРИМЕЧАНИЕ: Модель актинового ядра stereocilium теперь завершена.
- Мембранная сегментация
ПРИМЕЧАНИЕ: Целью этого шага является создание модели поверхности стереоцилийной мембраны.- Для сегментации поверхностей используется инструмент Объемная трассировка (Tools > Volume Data > Volume Tracer).
- В окне Volume Tracer нажмите кнопку Мышь и отметьте только опции Разместить маркеры на плоскостях данных и Привязать новый маркер к выбранному маркеру. Это позволяет точно разместить маркеры на одной и той же Z-плоскости (когда отображается одна Z-плоскость). Также установите флажок Разместить маркеры с помощью средней кнопки мыши в окне Volume Tracer .
- Создайте новый набор маркеров (Файл > Новый набор маркеров). Набор маркеров 1 должен быть создан и выбран в окне Volume Tracer .
- Используйте средство просмотра объема для отображения отдельных Z-плоскостей в главном окне просмотра (от оси до Z > единицы).
- С помощью средней кнопки мыши обведите мембрану от одного конца видимой мембраны к другому. Все маркеры должны быть соединены звеньями с одного конца на другой.
- Отмените выделение последнего размещенного маркера, нажав Ctrl + ЛКМ на фоне, где нет ни одного элемента, доступного для выбора. Это гарантирует, что следующий маркер, который будет размещен, будет соединен со звеньями в новой плоскости, а не с предыдущей мембранной трассой другой Z-плоскости.
- Повторяйте трассировку мембраны для каждых 10 нм в том же направлении трассировки от одного конца видимой мембраны к другой стороне.
- После того как многие параллельные полосы мембран будут сегментированы, перейдите в окно Volume Tracer и щелкните Features > Surfaces. Это позволяет использовать интерфейс поверхности в окне Volume Tracer . Нажмите кнопку Создать рядом с интерфейсом Поверхности, чтобы создать поверхности между полосами сегментации мембраны. При этом создается тонкий лоскут модели поверхности, который заполняет разнесенные дорожки мембраны.
ПРИМЕЧАНИЕ: Создание модели поверхности стереоцилиевой мембраны завершено.
- Моделирование сшивающих агентов
Примечание: Целью данного этапа является моделирование сшивающих агентов между актиновыми филаментами.- Сшивающие агенты также могут быть размещены с помощью инструмента Volume Tracer .
- В окне Volume Tracer щелкните левой кнопкой мыши, чтобы отметить только Разместить маркеры на поверхностях и Связать новый маркер с выбранным маркером. Затем установите флажок «Разместить маркеры с помощью средней кнопки мыши » в окне «Трассировщик объема ».
- Визуализируйте плотности только вокруг интересующей модели плоскости одного актинового филамента с помощью функции зоны. Установите радиус зоны равным 100 Å, чтобы визуализировать карту в радиусе 100 Å от выбранной модели.
- Размещайте маркеры на видимых плотностях сшивающих агентов, нажимая среднюю кнопку мыши. После того, как два маркера размещены (и, таким образом, смоделирован один сшивающий агент), обязательно отмените выбор второго маркера, чтобы предотвратить постоянное соединение связей со следующей моделью сшивателя.
- Повторите описанные выше шаги для каждой плоскости актинового филамента и повторите весь описанный выше процесс для всех трех основных направлений, в которых могут быть расположены сшивающие агенты. При зонировании плоскостей актиновых филаментов в диагональном направлении вручную подбирайте модели актиновых филаментов по отдельности.
Примечание: Для каждой из моделей актиновых сшивающих агентов мы можем записать положение на актиновых филаментах, к которым подключаются предполагаемые сшивающие агенты. Эта информация, в принципе, может быть извлечена для последующего анализа с использованием усреднения субтомограммы, что выходит за рамки данного исследования.
4. Количественный анализ 3D модели
- Анализ пробелов
ПРИМЕЧАНИЕ: Целью этого шага является обнаружение разрывов в актиновых филаментах.- Выбранные элементы могут быть проанализированы. Укажите их параметры, например, цвет, с помощью инструмента «Осмотр выделения » (Action > Inspect).
- Цвет выбранного элемента можно указать, выбрав любой доступный для выбора элемент в UCSF Chimera, а также изменив цвет рядом с разделом «Цвет» в окне «Проверить выбор».
- Укажите размер маркеров и связей, выбрав любые маркеры и связи в UCSF Chimera, а также изменив значения рядом с разделом радиуса в окне Проверка выбора , где единицы измерения находятся в Ангстреме.
- Выполняйте анализ разрывов путем автоматического обнаружения разрывов плотности с помощью функции «Значения в позициях атомов » (Tools > Volume Data > Values at Atom Positions).
- Выберите карту, на которой будут считываться значения, выбрав нужную карту рядом с разделом «Данные об объеме » окна «Значения в позициях атомов ». Выберите модель, в которой всем маркерам будет присвоен параметр, выбрав нужную модель рядом с разделом Молекула .
- С помощью кнопки «Гистограмма » в нижней части окна «Значения в положениях атомов » гистограмма создается в новом окне под названием «Рендеринг/Выбор по атрибуту». Ctrl + Клик по гистограмме добавит или уберет точки отсечения, для которых можно выбрать разные цвета. Снимите галочку с опции Сохранить непрозрачность (Keep Opaque), чтобы применить прозрачный цвет к сцене. Маркеры с плотностью ниже заданного порога, представляющего пробелы, могут быть не только раскрашены, но и выбраны, и таким образом позволяют подсчитать количество маркеров в области разрыва. Чтобы выбрать, нажмите « Выбрать» > > value_mapname «Атрибут».
- Определение межактиновых расстояний
- Для измерения межактиновых расстояний разместите маркеры в одной XZ-плоскости (называемой в UCSF Chimera Y-плоскостью) с помощью окна Volume Tracer , щелкнув мышью и установив флажок Разместить маркеры только на плоскостях данных и сняв флажок Привязать новый маркер к выбранному маркеру. Это позволяет точно разместить маркеры в одной и той же плоскости Y (когда отображается одна плоскость Z). Также отметьте флажок «Разместить маркеры с помощью средней кнопки мыши » в окне «Трассировщик объема » и поместите маркеры в целевой плоскости Y, представляющей интерес для измерения расстояния между актами.
- После того, как положения актина в одной плоскости Y будут успешно отмечены, выберите все маркеры и введите команду findclash #model-spec test selfoverlapCutoff -200 hbondAllowance 0 log true Linewidth 10 pbColor black в командной строке в нижней части плоскости актиновой нити. При этом все межмаркерные коллизии находятся на расстоянии 200 Å (overlapCutoff -200) и между маркерами создаются черные линии (pbColor black) толщиной 10 Å (Linewidth 10).
- Поскольку в заданном диапазоне 200 Å есть перекрывающиеся маркеры, удалите избыточные измерения, которые не являются связующими звеньями только между ближайшими, соседними маркерами. Для этого используется RemoveCross.py скрипт, представленный на веб-сайте UCSF Chimera, который удаляет все перекрывающиеся ссылки, которые не являются самой короткой ссылкой с соседними маркерами.
- Чтобы измерить расстояние между оставшимися связями, выберите все псевдосвязи (ссылки, созданные с помощью findccollision) и используйте скрипт Python pblengths.py , набрав pblengths.py runscript. Смотрите измеренные значения из Журнала ответов (Инструменты > Утилиты > Журнал ответов). Затем скопируйте полученные значения из Журнала ответов и перенесите в таблицу или любой другой подходящий инструмент для создания гистограммы.
Результаты
Используя криоэлектронную томографию неокрашенных, замороженно-гидратированных отдельных стереоцилий, встроенных в стекловидный лед, мы получили карты плотности актинового пучка с его гексагонально расположенными актиновыми филаментами, связанными перекрестными белками23. Размерность отдельного вокселя составляла 0,947 нм. Визуальный осмотр в программе слайсера IMOD объемной визуализации всей томограммы (400 срезов/379 нм) показал наличие нитевидных структур, выравнивающихся по продольной оси стереоцилий, что видно на продольных видах (плоскость XY; Рисунок 1A-C, верхние панели), а также на видах поперечного сечения (плоскость XZ; Рисунок 1A-C, нижние панели). Мы заметили, что проекционный вид через сеть филаментов 400 срезов/379 нм становится наиболее четким при повороте исходного восстановленного объема на -6° вокруг оси X, -13,5° вокруг оси Y и на 5° вокруг оси Z. Под этим углом все нити выравниваются друг над другом, и, следовательно, контраст максимален, что можно оценить по поперечному сечению (рисунок 1B). Поскольку одиночные поперечные срезы не имеют достаточного сигнала для однозначного различения актиновых филаментов, мы выбрали объемный рендеринг плиты размером 30 срезов/28,4 нм, на которой четко виден гексагональный узор на виде поперечного сечения. Синие линии на рисунке 1C (верхние панели) указывают на положение середины соответствующих плит поперечного сечения 30 срезов/28,4 нм в нижних панелях.
Небольшие отклонения от этого оптимального угла обзора, всего на ±2°, значительно снижали воспринимаемый порядок сети актиновых филаментов (рис. 1A, C), что является показателем того, насколько легко можно заблудиться в трехмерном объеме томограммы.
Чтобы проиллюстрировать сложность использования автоматизированных подходов к сегментации, таких как сегментация водосборов, мы выбрали небольшой подобъем (изображенный золотом) для сегментации водосбора, как это реализовано в программном пакете UCSF Chimera (Tools > Volume Data > Segger > Segment). Положение субобъема относительно всей стереоцилиевой карты обозначено небольшой вставкой на рисунке 1B.
На рисунке 1D-F показан выбранный подобъем в различных ориентациях, на рисунке 1D,E показано продольное направление обзора, а на рисунке 1F показано направление обзора в поперечном сечении. Стрелки в левой части рисунка 1D-F указывают направление актиновых нитей.
На рисунке 1D-F (правые панели) показаны результаты сегментации водосбора. Подобъем имеет цветовую кодировку в соответствии с идентификатором объекта, при этом цвета случайным образом присваиваются различным объектам. Разные цвета указывают на различную идентичность объекта, следовательно, из рисунка 1D-F становится очевидным, что плотности карты для нитей были фрагментированы вдоль оси нити, в то время как один и тот же цвет и, следовательно, идентичность объекта были даны плотностям карты, которые соединяли соседние нити. Другими словами, алгоритм сегментации водосборного бассейна не был способен следовать карте плотности актиновых филаментов в течение длительного периода времени, а вместо этого приводил к получению плотностей соединения из соседних филаментов. Несмотря на то, что можно вручную курировать выделение (например, путем удаления или объединения объектов), этот подход довольно трудоемкий и, следовательно, занимает много времени.
Несмотря на то, что это не является абсолютно необходимым для того, чтобы наша стратегия построения объемной модели работала, она помогла переориентировать (повернуть) 3D-карту таким образом, чтобы ось сети актиновых филаментов была выровнена по оси Y, а плоскости модели актинового филамента были выровнены по плоскости X-Y томограммы. Мы называем эту ориентацию стандартной ориентацией для томографического отображения стереоцилий.
Поэтому мы решили изучить другую стратегию сегментации изображений, используя тот факт, что актиновые филаменты демонстрируют общую регулярную организацию (гексагональную упаковку) с регулярным расстоянием и определенной общей ориентацией пучка. Наша стратегия заключалась в том, чтобы найти общую аппроксимацию моделей актинового расслоения в виде массива филаментов с последующей региональной, а затем локальной корректировкой положения модели в соответствии с экспериментальной картой плотности. Поместив сначала общую модель, мы можем преодолеть неоднозначность локальной карты и обнаружить региональные тенденции отклонений модели от ее первоначальной организации, такие как изгиб нити.
Для размещения модели мы отобразили плиты плотности (10 срезов/9,47 нм) в стандартной ориентации, которая соответствовала толщине одного слоя актиновых филаментов, к которому был прикреплен слой равномерно расположенных прямых моделей актиновых филаментов. Это, конечно, чрезмерное упрощение актиновых филаментов, каждая из которых состоит из линейного массива актиновых мономеров со спиральной симметрией. На рисунке 2A-C показаны три репрезентативных слоя на разных Z-высотах, при этом красные стержни представляют актиновые филаменты. Верхние панели, изображающие поперечные сечения толщиной ~30 срезов/28,4 нм, показывают, на какой Z-высоте был размещен отдельный слой модели актина из 19 стержней, в то время как нижние панели показывают продольную ориентацию (хотя и показаны в перспективе). На рисунке 2D показана полная упрощенная модель, как в поперечном сечении (верхняя панель), так и в продольной перспективе (нижняя панель). Ориентация поперечного сечения позволила нам с хорошей уверенностью расположить нити. В данном случае наш первоначальный ход по переориентации всего объема так, чтобы он совпадал с основными осями томограммы, оказался полезным, так как это означало, что ориентация нашей модели в нашем стандартном направлении обзора также была параллельна основным осям. Однако, строго говоря, наш подход также работал бы без переориентации томограммы, просто размещение модели по плотности было бы более сложным.
При тщательном изучении отдельных пластин карты плотности мы заметили, что идеально прямая модель актина не соответствует наблюдаемой карте плотности, движущейся от проксимального конца к дистальному концу (т.е. к кончику) стереоцилий (рис. 3A-C). Вблизи кончика стереоцилий плотность карты для филаментов была смещена более чем на 13 нм (расстояние между актин-актином), что мы смогли компенсировать, корректируя модель при переходе от проксимальнее к дистальной части карты плотности стереоцилий, тем самым внося небольшую, но различимую постепенную кривизну в нашу актиновую модель. На рисунке 3D показана одна пластина плотности карты актинового волокна с объемной моделью, подогнанной к карте плотности. Сравнение прямой (красной) и изогнутой (желтой) моделей показано на рисунке 3E. Эту кривизну лучше всего оценить, наклонив плиту карты плотности с размещенной моделью на 80° вокруг оси X, что позволяет иметь перспективный вид вдоль направления актиновых филаментов (рис. 3D, E).
Отклонение двух моделей, при котором положение актиновой модели вблизи кончика смещается примерно на то же расстояние, что и расстояние между актиновыми филаментами, могло бы вызвать большую путаницу, если бы мы не поступили так, как поступили. Такое «глобальное» позиционирование слоя модели актинового филамента с последующей «региональной» корректировкой позволило обнаружить эту кривизну, которая едва заметна в продольном или поперечном сечении. Тем не менее, наложение двух моделей, как показано на рисунке 3E, показывает тонкую разницу.
Повторение этого подхода для нескольких слоев позволяет получить полную 3D-модель (рисунок 3F), ограниченную только неопределенностью данных в самом верху и внизу стереоцилий при просмотре в поперечной ориентации (рисунок 3G). Этот недостаток плотности вызван отсутствующим клином в (одноосевом) сборе томографических данных и соответствующей анизотропией разрешения данных, а его влияние проявляется в отсутствии четко определенной картографической плотности для стереоцилийных мембран.
Как только у нас была 3D-модель, мы раскрасили каждое местоположение объемной модели в соответствии со значением плотности карты в этом месте. Области модели со слабой плотностью карты были окрашены в красный цвет, в то время как области модели с сильным сигналом плотности карты были окрашены в желтый цвет (рис. 4A). Мы интерпретируем такие области красного цвета, которые могут простираться до десятков нанометров, как разрывы в структурах актиновых филаментов, которые из-за их протяженности не могут быть отнесены к изменениям плотности, часто встречающимся в среде с высоким уровнем шума крио-ЭМ карты. Шум имеет тенденцию воздействовать на отдельные воксели или небольшие группы вокселей, но вряд ли может быть источником объемов, состоящих из сотен вокселей, для которых отсутствует плотность филамента. Вместо этого такие промежутки, вероятно, являются реальной особенностью сети актиновых узоров stereocilia и могут представлять собой места оборота актина. На рисунке 4A представлены два разных значения плотности карты, показанные светло-голубым и темно-синим цветом. Следует особо отметить, что наш подход к построению объемной модели в сочетании с автоматизированным цветовым кодированием нашей модели в областях слабой плотности является быстрым и удобным способом обнаружения и визуализации распределения таких разрывов в модели актинового филамента, что в противном случае было бы очень сложно.
Как показано на рисунке 4В, участки объемной модели в местах со сравнительно слабой плотностью могут быть легко скрыты на основе результатов, полученных на рисунке 4А. Это приводит к более фрагментированной модели, которая может более реалистично отображать актиновую модель в стереоцилии. Альтернатива создания небольших участков актиновых филаментов была бы очень трудоемкой и, возможно, вообще потерпела бы неудачу из-за проблем, рассмотренных при описании рисунка 1.
Кроме того, объемная модель позволяет нам легко моделировать поперечные соединители, просто размещая соединение (показано красным) между позициями точек модели актинового волокна по обе стороны от поперечного соединения (рисунок 4C). В нашем упрощенном подходе нам не нужно делать никаких предположений о точной идентичности каждого перекрестного белка, что потребовало бы более высокого разрешения и/или сложных подходов к мечению. Вместо этого все, что нам нужно определить, это существует ли плотность, которая соединяет соседние актиновые филаменты; Если есть, то мы можем разместить короткое соединение от одной нити накаливания к ее соседнему аналогу. На рисунке 4D показана модель пяти актиновых филаментов с их поперечными коннекторами, что дает представление о распределении кросс-коннекторов вдоль оси актинового филамента.
Еще одним преимуществом построения объемной модели актинового расслоения является то, что можно быстро определить расстояние между соседними актиновыми филаментами (рис. 4E-H). На рисунках 4E,F показан вид в поперечном сечении карты плотности без и с моделью, подогнанной под гексагональную решетку плотности карты, соответственно. На рисунке 4G показана модель с соединениями между ближайшими соседними шарами. UCSF Chimera позволяет автоматически вычислять расстояния до ближайших соседних центров, результат чего затем может быть построен в виде распределения расстояний (рисунок 4H). Построение модели для двух дополнительных наборов данных показано на дополнительных рисунках 1 и 2.
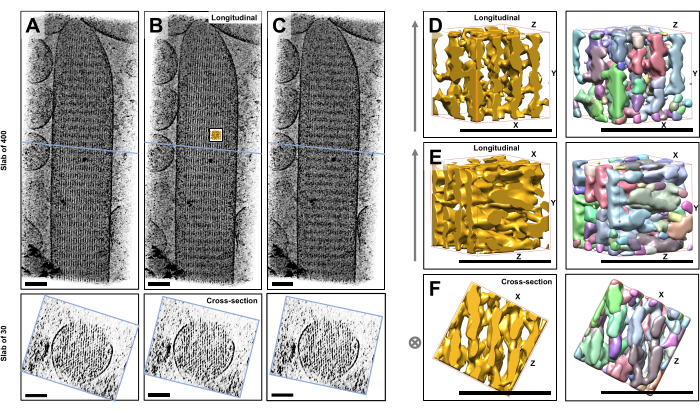
Рисунок 1: Проблемы, возникающие при водораздельной сегментации томограмм стереоцилии волосковых клеток. (A-C) Продольные проекции (400 срезов/379 нм) через томографическую 3D-карту в плоскости XY (верхние панели) и виды поперечного сечения (30 срезов/28,4 нм) в плоскости XZ (нижние панели). (A) Томографическая карта повернута на -2° вдоль оси Y от ее оптимальной ориентации. (B) Томографическая карта в оптимальной ориентации, определенная путем регулировки углов поворота осей X, Y и Z (X = -6°, Y = -13,5° и Z= 5°) и показывающая высокую степень упорядочения на карте плотности, предполагающую высокоупорядоченную сеть актиновых филаментов. (C) томографическая карта повернута на +2° вдоль оси Y от ее оптимальной ориентации; поворот всего на 2° вокруг оси Y в сторону от оптимального направления обзора серьезно нарушает воспринимаемую регулярность карты плотности. Нижние панели показывают регулярность массива актиновых филаментов при рассмотрении в направлении поперечного сечения. Синяя линия в точках A-C указывает положение плиты поперечного сечения. (Д-Ж) Куб размером 50 нм x 50 нм x 50 нм, вид с трех разных сторон до (левые панели) и после (правые панели) сегментации водораздела. Следует отметить, что сегментация водосборного бассейна не позволяет обнаружить непрерывную плотность актиновых филаментов, в то время как соседние актиновые филаменты и их перекрестные соединения имеют одну и ту же объектную идентичность, что позволяет предположить, что сегментация водосборного бассейна не является подходящим подходом для сегментации томограмм. На панелях D-F карта плотности в Химере отображается в виде стиля карты "Поверхность". (А-С) Масштабные линейки = 100 нм. (Д-Ж) Масштабные линейки = 50 нм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
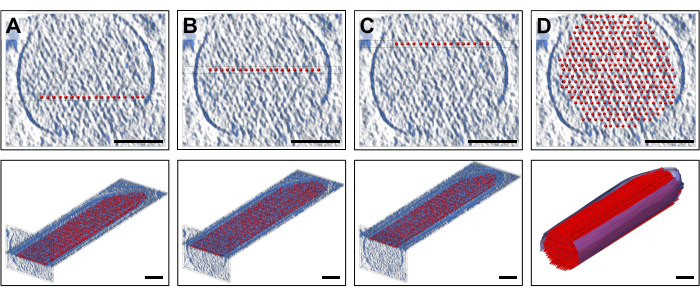
Рисунок 2: Построение объемной модели актинового филамента с шариком и палочкой. (A-C) Вверху: поперечное сечение 30 срезов/28,4 нм карты плотности с однослойной упрощенной моделью актинового филамента, размещенной на другой Z-высоте. Дно: одиночная плита 10 срезов/9,47 нм карты плотности с однослойной упрощенной моделью актинового филамента. (D) Полная модель прямого актинового волокна в поперечном сечении 30 срезов/28,4 нм (вверху) и перспективном виде (внизу); Масштабные линейки = 100 нм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
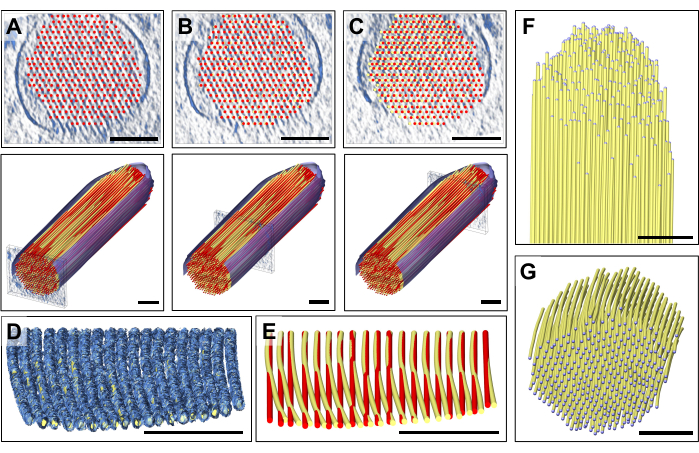
Рисунок 3: Корректировка модели для коррекции обнаруженного изгиба плотности актиновых нитей. (A-C) Тщательный визуальный осмотр модели, как в поперечном сечении (верхние панели), так и в продольном (нижние панели), показал хорошее соответствие модели карте плотности на проксимальном конце стереоцилии. Однако, по мере продвижения к дистальному концу стереоцилии, прилегание становится все хуже для всех моделей актиновых филаментов. Это можно исправить, переместив шарики модели «шарик и палочка» в правильное положение карты плотности, что приводит к слегка изогнутой модели актинового филамента. Прямая модель отображается красным цветом, а скорректированная изогнутая модель — желтым. (D) Одна плита карты плотности с прикрепленной к ней изогнутой моделью, показывающая кривизну плотности актина по направлению к кончику стереоцилии. Актиновая модель была повернута на 80° вокруг оси X, чтобы лучше показать этот тонкий, но значительный изгиб актиновых филаментов. (E) Сравнение между прямой, нескорректированной моделью актина, показанной красным цветом, и изогнутой, скорректированной моделью актина, показанной желтым цветом. Для ясности показан только один слой модели актинового филамента. (Ф-Г) Модель актинового расслоения с изогнутыми, скорректированными актиновыми филаментами, показанная в продольной (F) и поперечной (G) ориентации. Сегментированная мембрана показана синим цветом. На панели D карта плотности в Химере отображается в виде стиля карты "Mesh". Масштабные линейки = 100 нм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
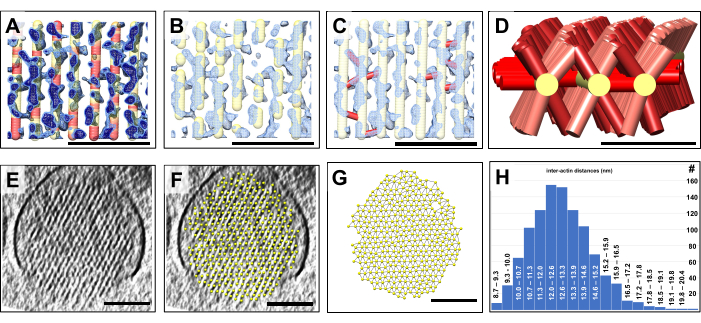
Рисунок 4: Обнаружение разрывов в актиновом пучке с помощью построения объемной модели. Количественная оценка объемных свойств с помощью моделей «шарик и палка». (А-С) Небольшая область продольной плиты толщиной ~10 срезов/9,47 нм карты плотности стереоцилий показана синим цветом вместе с изогнутой, скорректированной моделью актинового филамента. (A) Карта плотности отображается с более низким порогом плотности (светло-синий) и более высоким порогом плотности (темно-синий). Существуют области модели актинового филамента, для которых соответствующая плотность отсутствует. В таких местах модель имеет цветовую кодировку красного цвета, чтобы обозначить недостаточную плотность. Мы интерпретируем эти места как разрывы в актиновых филаментах. (B) Модель актиновых филаментов, которая выглядит фрагментированной, отражая недостаточную плотность актиновых филаментов в таких местах зазора. (C) Модель актиновых филаментов с поперечными соединительными звеньями (показаны красным цветом), добавленных в местах, где была обнаружена сильная плотность для моста соседних актиновых филаментов. (D) Три выбранные модели актиновых филаментов показаны с поперечными соединителями с соседними актиновыми филаментами (которые не показаны для ясности). Обратите внимание, что многие, но не все, возможные позиции интерактиновых филаментов заняты соединительными белками; (E) Поперечное сечение 30 срезов/28,4 нм плотности стереоцилий. (F) Модель актиновых филаментов, установленных на пластине плотности стереоцилий поперечного сечения 30 срезов/28,4 нм. (G) Модель актиновых филаментов без лежащей в их основе плотности карты. Автоматическое определение расстояний между ближайшими соседними актиновыми филаментами, на что указывают тонкие соединения между шариками модели актиновой филамента. (H) Гистограмма расстояний модели актинового филамента. На панелях A-C карта плотности в Chimera отображается в виде стиля карты "Mesh". (А-С) Масштабные линейки = 50 нм. (D) Масштабная линейка = 25 нм. (Э-Г) Масштабные линейки = 100 нм. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Дополнительный рисунок 1: Построение модели для первого из двух дополнительных наборов данных стереоцилий. (А-С) Небольшая область продольной плиты толщиной ~10 срезов/9,47 нм карты плотности стереоцилий показана синим цветом в режиме отображения в режиме сетки. Первоначально размещенная модель отображается красным цветом, а скорректированная модель — желтым. (A) Только плотность карты. (B) Исходная модель помещается в карту плотности. (C) Скорректированная модель помещена в карту плотности. (Д-Э) Большая область стереоцилий без (D) и с (E) скорректированная модель, подогнанная к продольной плите толщиной ~10 срезов/9,47 нм карты плотности стереоцилий. (Ф-Г) Отображается вся область томограммы стереоцилии. (F) Только карта. (G) Карта с скорректированной моделью. (H) Наложение исходной и скорректированной моделей. Масштабные линейки = 100 нм Пожалуйста, нажмите здесь, чтобы скачать этот файл.
Дополнительный рисунок 2: Построение модели для второго из двух дополнительных наборов данных стереоцилий. (А-С) Небольшая область продольной плиты толщиной ~10 срезов/9,47 нм карты плотности стереоцилий показана синим цветом в режиме отображения в режиме сетки. Первоначально размещенная модель отображается красным цветом, а скорректированная модель — желтым; (A) Только плотность карты. (B) Исходная модель помещается в карту плотности. (C) Скорректированная модель помещена в карту плотности. (Д-Э) Большая область стереоцилий без (D) и с (E) скорректированная модель, подогнанная к продольной плите толщиной ~10 срезов/9,47 нм карты плотности стереоцилий. (Ф-Г) Отображается вся область томограммы стереоцилии. (F) Только карта. (G) Карта с скорректированной моделью. (H) Наложение исходной и скорректированной модели. Масштабные линейки = 100 нм. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Дополнительный файл 1: Скрипты UCSF Chimera на Python для моделирования стереоцилий. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Файл дополнительного кодирования 1: pblengths.py. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Файл дополнительного кодирования 2: RemoveCross.py. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Файл дополнительного кодирования 3: ActinFilamentPlane.py. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Файл дополнительного кодирования 4: dividelinks.py. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Файл дополнительного кодирования 5: FixingMarkerID.py. Пожалуйста, нажмите здесь, чтобы загрузить этот файл.
Обсуждение
Мы показали, что автоматизированные подходы к сегментации, такие как сегментация водосбора, могут потерпеть неудачу в условиях высокого уровня шума и высокой сложности криоэлектронных томограмм стереоцилии волосковых клеток. Различение, какая часть этой нитевидной сети представляет собой актиновые филаменты, а какая представляет собой поперечные связи на уровне локальной среды, кажется в лучшем случае сложной задачей, когда речь идет только об небольших томографических подобъемах. Подход к построению моделей, использованный в этом исследовании, основан на предварительных знаниях о крупномасштабном порядке актинового пучка, что помогает в разработке математического ожидания относительно ориентации актиновых филаментов и плотностей сшивающих агентов. Возможно, еще более важным является то, что человеческий мозг может легко находить закономерности, рассматривая более широкий контекст за пределами локального распределения плотности, в то время как компьютерный алгоритм работает только для относительно небольшой области, которая рассматривается алгоритмом; Следовательно, более масштабные тенденции не могут быть легко приняты во внимание. Подогнав модель глобально к слою плотности, мы избежали путаницы, которая может возникнуть при попытке создать модель для небольших участков отдельных актиновых филаментов за один раз. Конечно, такая глобальная подгонка предполагает порядок, который распространяется на большие расстояния. Однако, поскольку у нас был небольшой, но значительный постепенный изгиб актиновых филаментов, глобальная аппроксимация была только начальным приближением и потребовала локальной корректировки модели для подгонки под карту плотности. Поскольку первоначальная модель была хорошей отправной точкой, корректировки можно было вносить с высокой степенью уверенности. Одним из больших преимуществ нашего подхода было то, что мы могли выбрать отображение только определенной зоны плотности, что помогло снизить сложность декораций. Кроме того, просмотр плиты плотности карты вдоль оси модели нити помог выявить неожиданную кривизну, которую мы, скорее всего, пропустили бы при простом отображении меньших подобъемов. Размещение исходной модели также способствовало быстрому увеличению и уменьшению масштаба, чтобы чередовать общий вид соответствующего слоя актиновых филаментов с подробными видами для внесения корректировок модели.
Важнейшие шаги в рамках протокола включали вращение карты после визуального осмотра, создание и размещение модели на карте плотности, а также разделение модели нити накала на более мелкие сегменты. Положение атомов сегментов может быть затем пространственно скорректировано в соответствии с картой плотности и/или цветовой кодировкой для обнаружения разрывов.
Этот подход к построению актиновой модели также может быть модифицирован путем помещения набора «атомов» (т.е. шариков модели «шарик и палочка») в плотности нитей с использованием вида поперечного сечения усредненной плиты плотности 10-30 срезов/9,47-28,4 нм, которая затем может быть соединена связями (т.е. палочками модели «шарик и палочка»). Мы использовали этот подход, который является модификацией подробно описанного здесь протокола, для построения объемной модели в области сужения стереоцилии волосковых клеток23. Кроме того, как мы уже описывали здесь, наш подход к построению объемных моделей также хорошо подходит для сегментации и построения моделей мембран.
В то время как построение объемной модели может быть применено к любой карте плотности, которая показывает филаментные особенности, метод, который мы здесь описали, наиболее эффективен, когда у нас есть массив регулярно расположенных филаментов, для которых можно получить глобальную аппроксимацию объемной модели. Это также зависит от нитевидных особенностей, чтобы постепенно изменять их направленность. Если в нитевидных структурах возникают внезапные изломы и резкие изгибы, наш подход может быть не особенно полезен для сегментации.
В то же время, наши сотрудники разработали автоматизированный подход к автоматизированному отслеживанию нитей накала, который следует аналогичной концепции, использованной здесь для ручной сегментации30,31. В перспективе лучшим подходом может быть гибрид ручной идентификации и помещения исходной разреженной модели (даже всего нескольких шаров) в плотность в качестве отправной точки, а затем позволить алгоритму поиска и подгонки завершить трассировку нитей.
Упрощенные объемные модели упрощают систему и позволяют лучше оценить определенные узоры, такие как изгиб актиновой нити возле кончика. Кроме того, объемная модель может быть использована в качестве «якоря» для отображения зоны плотности вокруг выбранной модели анкерного шара и палочки, что позволяет обнаруживать и визуализировать плотности сшивающих агентов между соседними актиновыми филаментами. Возможность выбора отдельных нитей накаливания и установки соответствующих радиусов в качестве зоны, в которой снова отображается плотность, позволяет снизить ошеломляющую сложность пейзажа до управляемого уровня.
Одним из преимуществ этого подхода к построению объемной модели с глобальной аппроксимацией с последующей локальной корректировкой было то, что мы смогли идентифицировать области, где актиновые филаменты, по-видимому, прерываются, а значительные разрывы в актиновых филаментах были обозначены отсутствием плотности карты. Поскольку мы разместили объемную модель шарика и палочки, мы могли использовать процедуру в программном пакете UCSF Chimera, которая кодирует цветом положение мяча каждой модели в соответствии со значением плотности карты в этом месте. Этот подход позволил быстро обнаружить и визуализировать разрывы актиновых филаментов в актиновом пучке, что является биологически значимой особенностью, которую мы обнаружили в нашей криоэлектронной томограмме, и которую было бы очень трудно обнаружить и визуализировать с помощью традиционных подходов к сегментации. Еще одно преимущество нашей объемной модели заключается в том, что можно легко получить объемные свойства, включая длины и расстояния, что позволяет получить фактические числа и, таким образом, выполнить статистический анализ.
Таким образом, интерактивное ручное размещение точек модели, возможно, в дальнейшем дополненное последующими возможностями автоматизированной локальной подгонки и отслеживания филаментов, является довольно перспективным подходом к визуализации и количественному анализу электронных томографических субклеточных объемов. Это связано с тем, что он использует возможности человеческого мозга для распознавания образов и возможности информатики для оптимизации моделей.
Раскрытие информации
Авторы заявляют, что у них нет конкурирующих финансовых интересов или других конфликтов интересов.
Благодарности
Мы хотели бы поблагодарить доктора Питера Барр-Гиллеспи и его команду за их роль в подготовке образцов, а также бывших сотрудников лаборатории Ауэра и лаборатории доктора Дорит Ханейн за их роль в сборе томографических данных. Мы также хотели бы поблагодарить Тома Годдарда (Tom Goddard) из UCSF Resource for Biocomputing, Visualization, and Informatics (RBVI) за предоставление различных сценариев UCSF Chimera.
Материалы
| Name | Company | Catalog Number | Comments |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/download.html | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=win64/chimera-1.16-win64.exe | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=mac64/chimera-1.16-mac64.dmg | |
| Chimera | RBVI | Version 1.16 https://www.cgl.ucsf.edu/chimera/cgi-bin/secure/chimera-get.py?file=linux_x86_64/chimera-1.16-linux_x86_64.bin | |
| Excel | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Falcon II | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/accessories-em/falcon-detector.html | |
| IMOD | University of Colorado | Version 4.11.1 https://bio3d.colorado.edu/imod/download.html | |
| PC Desktop | Intel | Windows 10, ver. 22H2 | |
| PC Laptop | Gigabyte | Windows 10, ver. 22H2 | |
| Powerpoint | Microsoft | Version 2211 https://www.office.com/?auth=1 | |
| Titan Krios Electron Microscope | Thermofisher | https://www.thermofisher.com/de/de/home/electron-microscopy/products/transmission-electron-microscopes/krios-g4-cryo-tem.html | |
| Word | Microsoft | Version 2211 https://www.office.com/?auth=1 |
Ссылки
- Downing, K. H., Sui, H., Auer, M. Electron tomography: A 3D view of the subcellular world. Analytical Chemistry. 79 (21), 7949-7957 (2007).
- Koning, R. I., Koster, A. J. Cellular nanoimaging by cryo electron tomography. Methods in Molecular Biology. 950, 227-251 (2013).
- Asano, S., Engel, B. D., Baumeister, W. In situ cryo-electron tomography: A post-reductionist approach to structural biology. Journal of Molecular Biology. 428 (2), 332-343 (2016).
- Serwas, D., Davies, K. M. Getting started with in situ cryo-electron tomography. Methods in Molecular Biology. 2215, 3-23 (2021).
- McDonald, K. L., Auer, M. High-pressure freezing, cellular tomography, and structural cell biology. Biotechniques. 41 (2), 137-143 (2006).
- Narasimha, R., et al. Evaluation of denoising algorithms for biological electron tomography. Journal of Structural Biology. 164 (1), 7-17 (2008).
- Frangakis, A. S. It's noisy out there! A review of denoising techniques in cryo-electron tomography. Journal of Structural Biology. 213 (4), 107804 (2021).
- Frangakis, A. S., Hegerl, R. Noise reduction in electron tomographic reconstructions using nonlinear anisotropic diffusion. Journal of Structural Biology. 135 (3), 239-250 (2001).
- Jiang, W., Baker, M. L., Wu, Q., Bajaj, C., Chiu, W. Applications of a bilateral denoising filter in biological electron microscopy. Journal of Structural Biology. 144 (1-2), 114-122 (2003).
- vander Heide, P., Xu, X. -. P., Marsh, B. J., Hanein, D., Volkmann, N. Efficient automatic noise reduction of electron tomographic reconstructions based on iterative median filtering. Journal of Structural Biology. 158 (2), 196-204 (2007).
- Volkmann, N. Methods for segmentation and interpretation of electron tomographic reconstructions. Methods in Enzymology. 483, 31-46 (2010).
- Böhm, J., et al. Toward detecting and identifying macromolecules in a cellular context: template matching applied to electron tomograms. Proceedings of the National Academy of Sciences. 97 (26), 14245-14250 (2000).
- Frangakis, A. S., et al. Identification of macromolecular complexes in cryoelectron tomograms of phantom cells. Proceedings of the National Academy of Sciences. 99 (22), 14153-14158 (2002).
- Lebbink, M. N., et al. Template matching as a tool for annotation of tomograms of stained biological structures. Journal of Structural Biology. 158 (3), 327-335 (2007).
- Rigort, A., et al. Automated segmentation of electron tomograms for a quantitative description of actin filament networks. Journal of Structural Biology. 177 (1), 135-144 (2012).
- Volkmann, N. A novel three-dimensional variant of the watershed transform for segmentation of electron density maps. Journal of Structural Biology. 138 (1-2), 123-129 (2002).
- Bajaj, C., Yu, Z., Auer, M. Volumetric feature extraction and visualization of tomographic molecular imaging. Journal of Structural Biology. 144 (1-2), 132-143 (2003).
- Yu, Z., Frangakis, A. S. Classification of electron sub-tomograms with neural networks and its application to template-matching. Journal of Structural Biology. 174 (3), 494-504 (2011).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Tilney, L. G., Derosier, D. J., Mulroy, M. J. The organization of actin filaments in the stereocilia of cochlear hair cells. The Journal of Cell Biology. 86 (1), 244-259 (1980).
- Tilney, L. G., Tilney, M. S., DeRosier, D. J. Actin filaments, stereocilia, and hair cells: how cells count and measure. Annual Review of Cell Biology. 8, 257-274 (1992).
- Metlagel, Z., et al. Electron cryo-tomography of vestibular hair-cell stereocilia. Journal of Structural Biology. 206 (2), 149-155 (2019).
- Song, J., et al. A cryo-tomography-based volumetric model of the actin core of mouse vestibular hair cell stereocilia lacking plastin 1. Journal of Structural Biology. 210 (1), 107461 (2020).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Agulleiro, J. I., Fernandez, J. J. Fast tomographic reconstruction on multicore computers. Bioinformatics. 27 (4), 582-583 (2011).
- Fernandez, J. J. Computational methods for electron tomography. Micron. 43 (10), 1010-1030 (2012).
- Chen, H., Clyborne, W. K., Sedat, J. W., Agard, D. A. Priism: an integrated system for display and analysis of 3-D microscope images. Biomedical Image Processing and Three-Dimensional Microscopy. 1660, 784-790 (1992).
- Pettersen, E. F., et al. UCSF Chimera-a visualization system for exploratory research and analysis. Journal of Computational Chemistry. 25 (13), 1605-1612 (2004).
- Goddard, T. D., Huang, C. C., Ferrin, T. E. Visualizing density maps with UCSF Chimera. Journal of Structural Biology. 157 (1), 281-287 (2007).
- Sazzed, S., et al. Tracing actin filament bundles in three-dimensional electron tomography density maps of hair cell stereocilia. Molecules. 23 (4), 882 (2018).
- Sazzed, S., Scheible, P., He, P., Wriggers, J. Spaghetti tracer: A framework for tracing semiregular filamentous densities in 3D tomograms. Biomolecules. 12 (8), 1022 (2022).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены