Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
DeepOmicsAE: Alzheimer Hastalığında Sinyal Modüllerinin Proteomik, Metabolomik ve Klinik Verilerin Derin Öğrenme Analizi ile Temsil Edilmesi
Bu Makalede
Özet
DeepOmicsAE, çoklu omik verilerin boyutluluğunu azaltmak için bir derin öğrenme yönteminin (yani bir otomatik kodlayıcı) uygulanmasına odaklanan bir iş akışıdır ve birden çok omik veri katmanını temsil eden tahmine dayalı modeller ve sinyal modülleri için bir temel sağlar.
Özet
Büyük omik veri kümeleri, insan sağlığına yönelik araştırmalar için giderek daha fazla kullanılabilir hale geliyor. Bu makale, proteomik, metabolomik ve klinik veriler dahil olmak üzere çoklu omik veri kümelerinin analizi için optimize edilmiş bir iş akışı olan DeepOmicsAE'yi sunmaktadır. Bu iş akışı, yüksek boyutlu çoklu omik girdi verilerinden kısa bir dizi özellik çıkarmak için otomatik kodlayıcı adı verilen bir tür sinir ağı kullanır. Ayrıca iş akışı, otomatik kodlayıcıyı uygulamak için gereken temel parametreleri optimize etmek için bir yöntem sağlar. Bu iş akışını sergilemek için, sağlıklı veya Alzheimer hastalığı teşhisi konmuş 142 kişiden oluşan bir kohorttan klinik veriler, ölüm sonrası beyin örneklerinin proteomu ve metabolomu ile birlikte analiz edildi. Otomatik kodlayıcının gizli katmanından çıkarılan özellikler, sağlıklı ve hastalıklı hastaları ayıran biyolojik bilgiyi korur. Ek olarak, bireysel ekstrakte edilen özellikler, her biri bireylerin klinik özellikleriyle benzersiz bir şekilde etkileşime giren ve proteomik, metabolomik ve klinik verileri entegre etmek için bir araç sağlayan farklı moleküler sinyal modüllerini temsil eder.
Giriş
Nüfusun giderek daha büyük bir kısmı yaşlanıyor ve nörodejenerasyon gibi yaşa bağlı hastalıkların yükünün önümüzdeki yıllarda keskin bir şekilde artması bekleniyor1. Alzheimer hastalığı en sık görülen nörodejeneratif hastalık türüdür2. Hastalığın başlangıcını ve ilerlemesini yönlendiren temel moleküler mekanizmaları yeterince anlamadığımız göz önüne alındığında, bir tedavi bulmadaki ilerleme yavaş olmuştur. Alzheimer hastalığı ile ilgili bilgilerin çoğu, nedenleri ve sonuçları ayırt etmeyi zor bir görev haline getiren beyin dokusunun incelenmesinden ölüm sonrası elde edilir3. Dini Tarikatlar Çalışması/Hafıza ve Yaşlanma Projesi (ROSMAP), her yıl tıbbi ve psikolojik muayenelerden geçmeyi ve ölümlerinden sonra beyinlerini araştırma için katkıda bulunmayı taahhüt eden binlerce kişinin çalışmasını içeren nörodejenerasyon hakkında daha geniş bir anlayış kazanmak için iddialı bir çabadır4. Çalışma, beynin normal işleyişinden Alzheimer hastalığınageçişe odaklanmaktadır 2. Proje kapsamında, postmortem beyin örnekleri, genomik, epigenomik, transkriptomik, proteomik5 ve metabolomik dahil olmak üzere çok sayıda omik yaklaşımla analiz edildi.
Hücresel durumların (yani proteomik ve metabolomiklerin) fonksiyonel okumalarını sunan omik teknolojileri6,7, protein ve metabolit bolluğu ile hücresel aktiviteler arasındaki doğrudan ilişki nedeniylehastalığı 8,9,10,11,12 yorumlamanın anahtarıdır. Proteinler, hücresel süreçlerin birincil yürütücüleridir, metabolitler ise biyokimyasal reaksiyonlar için substratlar ve ürünlerdir. Multi-omik veri analizi, proteomik ve metabolomik veriler arasındaki karmaşık ilişkileri, bunları tek başına değerlendirmek yerine anlama imkanı sunar. Multi-omik, moleküler veriler (genom dizisi ve mutasyonlar, transkriptom, proteom, metabolom), klinik görüntüleme verileri ve klinik özellikler dahil olmak üzere çok katmanlı yüksek boyutlu biyolojik verileri inceleyen bir disiplindir. Özellikle, multi-omik veri analizi, bu tür biyolojik veri katmanlarını entegre etmeyi, bunların karşılıklı düzenleme ve etkileşim dinamiklerini anlamayı ve hastalığın başlangıcı ve ilerlemesi hakkında bütünsel bir anlayış sunmayı amaçlar. Bununla birlikte, çoklu omik verileri entegre etme yöntemleri, geliştirmenin erken aşamalarında kalmaktadır13.
Bir tür denetimsiz sinir ağı14 olan otomatik kodlayıcılar, çoklu omik veri entegrasyonu için güçlü bir araçtır. Denetimli sinir ağlarının aksine, otomatik kodlayıcılar örnekleri belirli hedef değerlerle (sağlıklı veya hastalıklı gibi) eşlemez ve sonuçları tahmin etmek için kullanılmazlar. Birincil uygulamalarından biri boyutsallığın azaltılmasında yatmaktadır. Bununla birlikte, otomatik kodlayıcılar, temel bileşen analizi (PCA), t-dağıtılmış stokastik komşu gömme (tSNE) veya tekdüze manifold yaklaşımı ve projeksiyonu (UMAP) gibi daha basit boyutsallık azaltma yöntemlerine göre çeşitli avantajlar sunar. PCA'dan farklı olarak, otomatik kodlayıcılar veriler içindeki doğrusal olmayan ilişkileri yakalayabilir. tSNE ve UMAP'den farklı olarak, her biri doğrusal olmayan aktivasyon fonksiyonları içeren birden çok hesaplama birimi katmanına dayandıkları için veriler içindeki hiyerarşik ve çok modlu ilişkileri tespit edebilirler. Bu nedenle, multi-omik verilerin karmaşıklığını yakalamak için çekici modelleri temsil ederler. Son olarak, PCA, tSNE ve UMAP'nin birincil uygulaması verileri kümelemek olsa da, otomatik kodlayıcılar giriş verilerini aşağı akış tahmine dayalı görevler için çok uygun olan çıkarılmış özelliklere sıkıştırır15,16.
Kısaca, sinir ağları, her biri birden fazla hesaplama birimi veya "nöron" içeren birkaç katmandan oluşur. İlk ve son katmanlar sırasıyla giriş ve çıkış katmanları olarak adlandırılır. Otomatik kodlayıcılar, bir giriş katmanı, ardından bir ila üç gizli katman ve tipik olarak iki ila altı nöron içeren küçük bir "gizli" katmandan oluşan kum saati yapısına sahip sinir ağlarıdır. Bu yapının ilk yarısı kodlayıcı olarak bilinir ve kodlayıcıyı yansıtan bir kod çözücü ile birleştirilir. Kod çözücü, giriş katmanıyla aynı sayıda nöron içeren bir çıkış katmanıyla sona erer. Otomatik kodlayıcılar, girdiyi darboğazdan geçirir ve orijinal bilgileri mümkün olduğunca yakından yansıtan bir çıktı oluşturmak amacıyla çıktı katmanında yeniden oluşturur. Bu, "yeniden yapılanma kaybı" olarak adlandırılan bir parametrenin matematiksel olarak en aza indirilmesiyle elde edilir. Girdi, burada sergilenen uygulamada protein ve metabolit bollukları ve klinik özellikler (yani cinsiyet, eğitim ve ölüm yaşı) olacak bir dizi özellikten oluşur. Gizli katman, tahmine dayalı modeller17,18 gibi sonraki uygulamalar için kullanılabilecek girdinin sıkıştırılmış ve bilgi açısından zengin bir temsilini içerir.
Bu protokol, aşağıdakileri içeren bir iş akışı olan DeepOmicsAE'yi sunar: 1) makine öğrenimi analizi için tutarlı bir ölçekle veri elde etmek için proteomik, metabolomik ve klinik verilerin (yani normalleştirme, ölçekleme, aykırı değer kaldırma) ön işlenmesi; 2) özellik aşırı yüklenmesi ilgili hastalık modellerini gizleyebileceğinden, uygun otomatik kodlayıcı giriş özelliklerinin seçilmesi; 3) seçilecek en uygun protein ve metabolit sayısının ve gizli tabaka için nöronların belirlenmesi de dahil olmak üzere otomatik kodlayıcıyı optimize etmek ve eğitmek; 4) gizli tabakadan özelliklerin çıkarılması; ve 5) moleküler sinyal modüllerini ve bunların klinik özelliklerle ilişkilerini tanımlayarak biyolojik yorumlama için çıkarılan özelliklerin kullanılması.
Bu protokol, Python ile programlama konusunda temel bir anlayışa sahip, sınırlı hesaplama deneyimine sahip biyologlar tarafından basit ve uygulanabilir olmayı amaçlamaktadır. Protokol, proteomikler, metabolomikler ve klinik özellikler dahil olmak üzere çoklu omik verileri analiz etmeye odaklanır, ancak kullanımı, transkriptomik dahil olmak üzere diğer moleküler ekspresyon verileri türlerine genişletilebilir. Bu protokol tarafından sunulan önemli bir yeni uygulama, orijinal özelliklerin önem puanlarını gizli katmandaki bireysel nöronlara haritalamaktır. Sonuç olarak, gizli katmandaki her nöron, spesifik moleküler değişiklikler ile hastaların klinik özellikleri arasındaki etkileşimleri detaylandıran bir sinyal modülünü temsil eder. Moleküler sinyal modüllerinin biyolojik yorumu, zenginleştirilmiş metabolik ve hücre sinyal yollarını türetmek için gen/protein ve metabolit verilerini entegre eden halka açık bir araç olan MetaboAnalyst kullanılarak elde edilir17.
Protokol
NOT: Burada kullanılan veriler, AD Bilgi portalından indirilen ROSMAP verileridir. Verileri indirmek ve yeniden kullanmak için bilgilendirilmiş onay gerekli değildir. Burada sunulan protokol, çoklu omik verileri analiz etmek ve örneğin teşhislerine dayalı olarak belirli hasta veya numune gruplarını ayırt eden sinyal modüllerini tanımlamak için derin öğrenmeyi kullanır. Protokol ayrıca, orijinal büyük ölçekli verileri özetleyen ve makine öğrenimi algoritmalarını kullanarak tahmine dayalı bir modeli eğitmek gibi daha fazla analiz için kullanılabilen küçük bir dizi çıkarılmış özellik sunar (Şekil 1). Protokolü gerçekleştirmeden önce koda erişim ve hesaplama ortamının ayarlanması hakkında bilgi için Ek Dosya 1'e ve Malzeme Tablosuna bakın. Yöntemler aşağıda belirtilen sıraya göre gerçekleştirilmelidir.
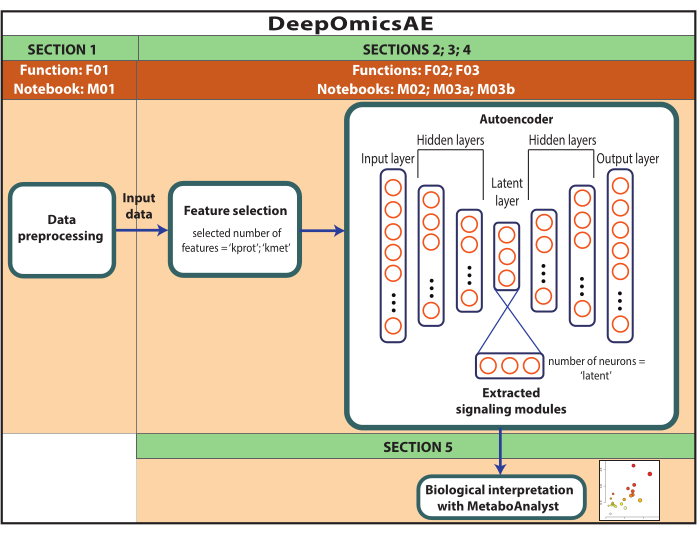
Şekil 1: DeepOmicsAE iş akışının şeması. İş akışını kullanarak çoklu omik verileri analiz etmek için iş akışının şematik gösterimi. Otomatik kodlayıcı tasvirinde, dikdörtgenler sinir ağının katmanlarını temsil eder ve daireler katmanlar içindeki nöronları temsil eder. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
1. Veri ön işleme
NOT: Bu bölümün amacı, eksik verilerin işlenmesi de dahil olmak üzere verileri önceden işlemektir; proteomik, metabolomik ekspresyon ve klinik verilerin normalleştirilmesi ve ölçeklendirilmesi; ve aykırı değerleri ortadan kaldırmak. Protokol, log2 (oran) olarak ifade edilen proteomik verileri içeren bir veri kümesi için tasarlanmıştır; kıvrım değişimi olarak ifade edilen metabolomik veriler; ve sürekli ve kategorik özellikler dahil olmak üzere klinik özellikler. Hastalar veya örnekler tanı veya diğer benzer parametrelere göre gruplandırılmalıdır. Numuneler veya hastalar satırlar boyunca ve sütunlar boyunca özellikler boyunca olmalıdır.
- Tarayıcıda yeni bir Jupyter Notebook örneği başlatmak için yeni bir terminal penceresi açın, aşağıdakini yazın ve Enter tuşuna basın.
Jupyter Not Defteri - Tarayıcıdaki Jupyter giriş sayfasında, yeni bir sekmede açmak için M01 - expression data pre-processing.ipynb not defterine tıklayın (Ek Dosya 2, Adım 1.1).
- Not defterinin ikinci hücresine , your_dataset_name.csv yerine veri kümesi dosyasının adını yazın.
- Not defterinin son hücresine , M01_output_data.csv yerine çıktı veri dosyasının istenen adını yazın.
- Not defterinin beşinci hücresinde , her veri türü için sütunların konumunu aşağıdaki gibi belirtin: proteomik veriler (cols_prot), metabolomik veriler (cols_met), sürekli klinik veriler (örn. yaş) (cols_clin_con), ikili klinik veriler (örn. cinsiyet) (cols_clin_bin). Her veri türü için col_start yerine ilk sütun dizinini ve col_end yerine son sütun dizinini girin; Örneğin: cols_prot = dilim(0, 8817). Dilim nesnelerinde belirtilen değerlerin, her veri türüne karşılık gelen ilk ve son sütun dizinlerine karşılık geldiğinden emin olun. Her veri türünün başlangıç ve bitiş konumunu belirlemek için aynı not defterinin dördüncü hücresindeki komutu (df.iloc[:, :]) kullanın (Ek Dosya 2, Adım 1.2).
- Hücre Seç | Belirtilen klasörde çıkış veri dosyasını oluşturmak için Jupyter'daki menü çubuğundan tümünü çalıştırın (Ek Dosya 2, Adım 1.3).
NOT: Bu veriler, bölüm 2, 3 veya 4'te açıklanan protokoller için girdi olarak kullanılacaktır.
2. İş akışının özel optimizasyonu (isteğe bağlı)
NOT: Bölüm 2, yoğun bilgisayar kullanımı gerektirdiğinden isteğe bağlıdır. Kullanıcılar, bölüm 2'yi gerçekleştirmemeye karar verirlerse doğrudan bölüm 4'e atlamalıdır. Bu protokol, iş akışını otomatik bir şekilde optimize etme konusunda kullanıcıya rehberlik edecektir. Özellikle, yöntem, örnek gruplarını iyi ayıran ayıklanmış özellikler oluşturma açısından otomatik kodlayıcının en iyi performansını sağlayan parametreleri tanımlar. Çıktı olarak oluşturulan optimize edilmiş parametreler, özellik seçimi için kullanılacak özelliklerin sayısını (k_prot ve k_met) ve otomatik kodlayıcı gizli katmanındaki nöronların sayısını (gizli) içerir. Bu parametreler daha sonra modeli oluşturmak için bölüm 3'te açıklanan protokolde kullanılabilir.
- Tarayıcıdaki Jupyter giriş sayfasında, yeni bir sekmede açmak için M02 - DeepOmicsAE model optimization.ipynb not defterine tıklayın (Ek Dosya 2, Adım 2.1).
- Not defterinin ikinci hücresine, M01_output_data.csv yerine giriş dosyasının adını yazın. Bu fonksiyonun girişi, bölüm 1'deki çıktı verileridir.
- Not defterinin beşinci hücresinde, her veri türü için sütunların konumunu aşağıdaki gibi belirtin: proteomik veriler (cols_X_prot), metabolomik veriler (cols_X_met), klinik veriler (cols_clin; tüm klinik verileri içerir), proteomik ve metabolomik veriler (cols_X_expr) dahil olmak üzere tüm moleküler ekspresyon verileri. Her veri türü için col_start yerine ilk sütun dizinini ve col_end yerine son sütun dizinini girin; Örneğin, cols_prot = dilim(0, 8817). Dilim nesnelerinde belirtilen değerlerin her veri türüne karşılık gelen ilk ve son sütun dizinine karşılık geldiğinden emin olun ve verileri araştırmak ve her veri türü için başlangıç ve bitiş konumlarını belirlemek için not defterinin üçüncü ve dördüncü hücrelerindeki komutları kullanın. y_column_name yerine hedef değişkeni içeren sütunun adını y_label olarak belirtin (Ek Dosya 2, Adım 2.2).
NOT: cols_X_prot, cols_X_met, cols_clin ve cols_X_expr'de belirtilen dizinlerin değerleri, veri ön işleme sırasında meydana gelen veri çerçevesinin yeniden şekillendirilmesi nedeniyle bölüm 1'de kullanılanlardan farklı olacaktır. - Not defterinin altıncı hücresinde, n_comb bir değer atayarak kaç iyileştirme turu gerçekleştirileceğini belirtin. İşleme süreleri 10 tur için yaklaşık 4-5 dakikadır; 50 tur için 20 dakika ve 100 tur için 40 dakika (Ek Dosya 2, Adım 2.3).
- Hücre Seç | Tümünü Jupyter'daki menü çubuğundan çalıştırın.
NOT: kprot, kmet ve latent çıkış değişkenleri saklanır ve analitik iş akışına devam etmek için kullanılacak diğer not defterlerinden erişilebilir. Çizim AE_optimization_plot.pdf oluşturulacak ve yerel klasöre kaydedilecektir (Şekil 2).
3. Özel olarak optimize edilmiş parametrelerle iş akışı uygulaması
NOT: Bu protokolü yalnızca yöntem optimizasyonunu izleyerek gerçekleştirin (bölüm 2). Kullanıcılar yöntem iyileştirmesi yapmamayı seçerse, doğrudan bölüm 4'e atlayın. Bu protokol, bölüm 2'den türetilen özel olarak optimize edilmiş parametreleri kullanarak bir model oluşturma konusunda kullanıcıya rehberlik edecektir. Otomatik kodlayıcı 1) orijinal verileri özetleyen bir dizi çıkarılmış özellik üretecek ve 2) gizli katmandaki her bir nöronu yönlendiren ve benzersiz sinyal modüllerini etkin bir şekilde temsil eden önemli özellikleri tanımlayacaktır. Sinyal modülleri, bölüm 5'te verilen protokol kullanılarak yorumlanacaktır.
- Tarayıcıdaki Jupyter giriş sayfasında, yeni bir sekmede açmak için özel olarak optimize edilmiş parameters.ipynb ile not defteri M03a - DeepOmicsAE uygulamasına tıklayın (Ek Dosya 2, Adım 3.1).
- Not defterinin ikinci hücresine, M01_output_data.csv yerine giriş dosyasının adını yazın. Bu fonksiyonun girişi, bölüm 1'deki çıktı verileridir.
- Not defterinin beşinci hücresinde, her veri türü için sütunların konumunu aşağıdaki gibi belirtin: proteomik veriler (cols_prot), metabolomik veriler (cols_met), klinik veriler (cols_clin; tüm klinik verileri içerir). Her veri türü için col_start yerine ilk sütun dizinini ve col_end yerine son sütun dizinini girin; Örneğin: cols_prot = dilim(0, 8817). Dilim nesnelerinde belirtilen değerlerin her veri türüne karşılık gelen ilk ve son sütun dizinlerine karşılık geldiğinden emin olun ve verileri araştırmak ve her veri türü için başlangıç ve bitiş konumlarını belirlemek için not defterinin üçüncü ve dördüncü hücrelerindeki komutları kullanın. Hedef değişkeni içeren sütunun adını (ör. sağlıklı veya hastalıklıya karşılık gelen 0 veya 1) y_column_name yerine y_label olarak belirtin.
NOT: cols_X_prot, cols_X_met, cols_clin ve cols_X_expr'de belirtilen dizinlerin değeri, veri ön işleme sırasında meydana gelen veri çerçevesinin yeniden şekillendirilmesi nedeniyle bölüm 1'de kullanılanlardan farklı olacaktır. - Hücre Seç | Yerel klasörde PCA_initial_data.pdf, PCA_extracted_features.pdf ve distribution_important_feature_scores.pdf grafikleri oluşturmak ve kaydetmek için Jupyter'daki menü çubuğundan tümünü çalıştırın (Şekil 3 ve Ek Şekil S1). Ek olarak, tanımlanan her bir sinyal modülü için önemli özelliklerin listeleri, module_n.txt adlı yerel klasördeki metin dosyalarında saklanacak ve burada n, modül numarası ile değiştirilecektir.
4. Önceden ayarlanmış parametrelerle iş akışı uygulaması
- Bu yöntemin nasıl çalıştırılacağına ilişkin ayrıntılı talimatlar için bölüm 3'e bakın (Ek Dosya 2, Adım 4.1). Bu iki protokol arasındaki tek fark, kprot, kmet ve latent parametrelerinin (not defterinin yedinci hücresinde) Şekil 2'de gösterildiği gibi gerçekleştirilen optimizasyonun sonuçlarına göre matematiksel olarak türetilmesidir.
NOT: Bölüm 4, örnek grupların yetersiz model performansını gösteren zayıf bir ayrımı sağlıyorsa, model optimizasyonunun (bölüm 2) en az 15 yineleme ve mümkünse en fazla 50 yineleme kullanılarak yürütülmesi önerilir.
5. MetaboAnalyst kullanarak biyolojik yorumlama
- MetaboAnalyst web sitesindeki Ortak Yol Analizi işlevine erişmek için tarayıcıyı açın ve aşağıdaki bağlantıya gidin: https://www.metaboanalyst.ca/MetaboAnalyst/upload/JointUploadView.xhtml.
- Yöntem 3 veya Yöntem 4'teki çıktı dosyalarının kaydedildiği klasöre erişin ve Yöntem 3 veya Yöntem 4 tarafından oluşturulan her sinyal modülü n için module_n.txt metin dosyalarını açın.
- Metin dosyalarındaki proteinleri bulun ve kopyalayın.
- Proteinlerin listesini MetaboAnalyst web sayfasındaki isteğe bağlı kat değişiklikleri olan genler/proteinler penceresine yapıştırın.
- Metabolitler için yukarıdaki adımı tekrarlayın ve bunları aynı web sayfasındaki isteğe bağlı katlama değişiklikleriyle Bileşik listeye yapıştırın.
- Uygun organizmayı ve kimlik türünü seçin, ardından sayfanın altındaki Gönder'e tıklayın (Ek Dosya 2, Adım 5.1).
NOT: Tanımlayıcıların MetaboAnalyst tarafından tanındığından emin olun. Tanınan tanımlayıcılar arasında Entrez ID, resmi gen sembolleri ve proteinler için Uniprot ID; metabolitler için bileşik ad, HMDB kimliği ve KEGG kimliği. Tanımlayıcılar bu türlerin dışındaysa, analizden önce uygun dönüştürme gereklidir. - Sonraki sayfada, tanımlayıcıların tanındığını doğrulamak için Devam Et'e tıklamadan önce kimlik eşlemesini kontrol edin.
- Parametre Ayarı sayfasında, girişin yalnızca metabolik yollara veya tüm sinyal yollarına katkısını sırasıyla görselleştirmek için Metabolik yollar (entegre) veya Tüm yollar (entegre) öğesini seçin (Ek Dosya 2, Adım 5.2). Algoritma seçimi panelinde Zenginleştirme analizi: Hipergeometrik test, Topoloji ölçüsü: Derece merkeziliği ve Entegrasyon yöntemi: p değerlerini birleştir (yol düzeyi) seçeneğini belirleyin. Sayfanın altındaki Gönder'e tıklayın.
- Son sayfa, zenginleştirme analizinin sonuçlarını sunan Sonuç Görünümü'dür. Zenginleştirilmiş yollar, etkilerine ve önemlerine göre çizilir ve yolların listesi de tablo biçiminde sağlanır.
Sonuçlar
Protokolü sergilemek için, sağlıklı veya Alzheimer hastalığı teşhisi konmuş 142 bireyin postmortem beyinlerinden elde edilen proteom, metabolom ve klinik bilgileri içeren bir veri setini analiz ettik.
Verileri ön işlemek için protokol bölüm 1'i gerçekleştirdikten sonra, veri seti 6.497 protein, 443 metabolit ve üç klinik özellik (cinsiyet, ölüm yaşı ve eğitim) içeriyordu. Hedef özellik, kogdx olarak kodlanmış, kogdx olarak kodlanm...
Tartışmalar
Veri kümesinin yapısı, protokolün başarısı için kritik öneme sahiptir ve dikkatlice kontrol edilmelidir. Veriler, protokol bölüm 1'de belirtildiği gibi biçimlendirilmelidir. Sütun konumlarının doğru atanması da yöntemin başarısı için kritik öneme sahiptir. Proteomik ve metabolomik veriler farklı şekilde önceden işlenir ve verilerin farklı doğası nedeniyle özellik seçimi ayrı ayrı yapılır. Bu nedenle, protokol adımları 1.5, 2.3 ve 3.3'te sütun konumlarını doğru bir şekilde atama...
Açıklamalar
Yazar, herhangi bir çıkar çatışması olmadığını beyan eder.
Teşekkürler
Bu çalışma, NIH hibe CA201402 ve Cornell Omurgalı Genomik Merkezi (CVG) Seçkin Bilim Adamı Ödülü tarafından desteklenmiştir. Burada yayınlanan sonuçlar tamamen veya kısmen AD Bilgi Portalı'ndan (https://adknowledgeportal.org) elde edilen verilere dayanmaktadır. Çalışma verileri, Rush Alzheimer Hastalığı Merkezi, Rush Üniversitesi Tıp Merkezi, Chicago tarafından sağlanan örneklere dayanarak AD için Hızlandırıcı Tıp Ortaklığı (U01AG046161 ve U01AG061357) aracılığıyla sağlandı. Veri toplama, NIA hibeleri P30AG10161, R01AG15819, R01AG17917, R01AG30146, R01AG36836, U01AG32984, U01AG46152, Illinois Halk Sağlığı Departmanı ve Translasyonel Genomik Araştırma Enstitüsü tarafından finanse edilerek desteklendi. Metabolomik veri seti Metabolon'da oluşturuldu ve ADMC tarafından önceden işlendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Computer | Apple | Mac Studio | Apple M1 Ultra with 20-core CPU, 48-core GPU, 32-core Neural Engine; 64 GB unified memory |
| Conda v23.3.1 | Anaconda, Inc. | N/A | package management system and environment manager |
| conda environment DeepOmicsAE | N/A | DeepOmicsAE_env.yml | contains packages necessary to run the worflow |
| github repository DeepOmicsAE | Microsoft | https://github.com/elepan84/DeepOmicsAE/ | provides scripts, Jupyter notebooks, and the conda environment file |
| Jupyter notebook v6.5.4 | Project Jupyter | N/A | a platform for interactive data science and scientific computing |
| DT01-metabolomics data | N/A | ROSMAP_Metabolon_HD4_Brain 514_assay_data.csv | This data was used to generate the Results reported in the article. Specifically, DT01-DT04 were merged by matching them based on the individualID. The column final consensus diagnosis (cogdx) was filtered to keep only patients classified as healthy or AD. Climnical features were filtered to keep the following: age at death, sex and education. Finally, age reported as 90+ was set to 91, then the age column was transformed to float64. The data is available at https://adknowledgeportal.synapse.org |
| DT02-TMT proteomics data | N/A | C2.median_polish_corrected_log2 (abundanceRatioCenteredOn MedianOfBatchMediansPer Protein)-8817x400.csv | |
| DT03-clinical data | N/A | ROSMAP_clinical.csv | |
| DT04-biospecimen metadata | N/A | ROSMAP_biospecimen_metadata .csv | |
| Python 3.11.3 | Python Software Foundation | N/A | programming language |
Referanslar
- Hou, Y., et al. Ageing as a risk factor for neurodegenerative disease. Nature Reviews Neurology. 15 (10), 565-581 (2019).
- Scheltens, P., et al. Alzheimer’s disease. The Lancet. 397 (10284), 1577-1590 (2021).
- Breijyeh, Z., Karaman, R. Comprehensive review on Alzheimer’s disease: causes and treatment. Molecules. 25 (24), 5789 (2020).
- Bennett, D. A., et al. Religious Orders Study and Rush Memory and Aging Project. Journal of Alzheimer’s Disease. 64 (s1), S161-S189 (2018).
- Higginbotham, L., et al. Integrated proteomics reveals brain-based cerebrospinal fluid biomarkers in asymptomatic and symptomatic Alzheimer’s disease. Science Advances. 6 (43), eaaz9360 (2020).
- Aebersold, R., et al. How many human proteoforms are there. Nature Chemical Biology. 14 (3), 206-214 (2018).
- Nusinow, D. P., et al. Quantitative proteomics of the cancer cell line encyclopedia. Cell. 180 (2), 387-402.e16 (2020).
- Johnson, E. C. B., et al. Large-scale proteomic analysis of Alzheimer’s disease brain and cerebrospinal fluid reveals early changes in energy metabolism associated with microglia and astrocyte activation. Nature Medicine. 26 (5), 769-780 (2020).
- Geyer, P. E., et al. Plasma proteome profiling to assess human health and disease. Cell Systems. 2 (3), 185-195 (2016).
- Akbani, R., et al. A pan-cancer proteomic perspective on the cancer genome atlas. Nature Communications. 5, 3887 (2014).
- Panizza, E., et al. Proteomic analysis reveals microvesicles containing NAMPT as mediators of radioresistance in glioma. Life Science Alliance. 6 (6), e202201680 (2023).
- Li, Z., Vacanti, N. M. A tale of three proteomes: visualizing protein and transcript abundance relationships in the Breast Cancer Proteome Portal. Journal of Proteome Research. 22 (8), 2727-2733 (2023).
- Subramanian, I., Verma, S., Kumar, S., Jere, A., Anamika, K. Multi-omics Data Integration, Interpretation, and Its Application. Bioinformatics and Biology Insights. 14, 1177932219899051 (2020).
- Wang, Y., Yao, H., Zhao, S. Auto-encoder based dimensionality reduction. Neurocomputing. 184, 232-242 (2016).
- Mulla, F. R., Gupta, A. K. A review paper on dimensionality reduction techniques. Journal of Pharmaceutical Negative Results. 13, 1263-1272 (2022).
- Shrestha, A., Mahmood, A. Review of deep learning algorithms and architectures. IEEE Access. 7, 53040-53065 (2019).
- Pang, Z., et al. MetaboAnalyst 5.0: Narrowing the gap between raw spectra and functional insights. Nucleic Acids Research. 49 (W1), W388-W396 (2021).
- Hinton, G. E., Salakhutdinov, R. R. Reducing the dimensionality of data with neural networks. Science. 313 (5786), 504-507 (2006).
- Altmann, A., Toloşi, L., Sander, O., Lengauer, T. Permutation importance: a corrected feature importance measure. Bioinformatics. 26 (10), 1340-1347 (2010).
- Lundberg, S. M., Allen, P. G., Lee, S. -. I. A unified approach to interpreting model predictions. , (2017).
- Wang, Q., et al. Deep learning-based brain transcriptomic signatures associated with the neuropathological and clinical severity of Alzheimer’s disease. Brain Communications. 4 (1), (2021).
- Beebe-Wang, N., et al. Unified AI framework to uncover deep interrelationships between gene expression and Alzheimer’s disease neuropathologies. Nature Communications. 12 (1), 5369 (2021).
- Camandola, S., Mattson, M. P. Brain metabolism in health, aging, and neurodegeneration. The EMBO Journal. 36 (11), 1474-1492 (2017).
- Verdin, E. NAD+ in aging, metabolism, and neurodegeneration. Science. 350 (6265), 1208-1213 (2015).
- Platten, M., Nollen, E. A. A., Röhrig, U. F., Fallarino, F., Opitz, C. A. Tryptophan metabolism as a common therapeutic target in cancer, neurodegeneration and beyond. Nature Reviews Drug Discovery. 18 (5), 379-401 (2019).
- Wang, R., Reddy, P. H. Role of glutamate and NMDA receptors in Alzheimer’s disease. Journal of Alzheimer’s Disease. 57 (4), 1041-1048 (2017).
- Skaper, S. D., Facci, L., Zusso, M., Giusti, P. Synaptic plasticity, dementia and Alzheimer disease. CNS & Neurological Disorders - Drug Targets. 16 (3), 220-233 (2017).
- Reisberg, B., et al. Memantine in moderate-to-severe Alzheimer’s disease. New England Journal of Medicine. 348 (14), 1333-1341 (2003).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır