A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
القوة الذرية المجهري التحقيقات من الحمض النووي الآفة الاعتراف في إصلاح ختان النوكليوتيدات
* These authors contributed equally
In This Article
Summary
Here, the study of different DNA lesion recognition approaches via single molecule AFM imaging is demonstrated with the nucleotide excision repair system as an example. The procedures of DNA and protein sample preparations and experimental as well as analytical details for the AFM experiments are described.
Abstract
AFM imaging is a powerful technique for the study of protein-DNA interactions. This single molecule method allows the simultaneous resolution of different molecules and molecular assemblies in a heterogeneous sample. In the particular context of DNA interacting protein systems, different protein complex forms and their corresponding binding positions on target sites containing DNA fragments can thus be distinguished. Here, an application of AFM to the study of DNA lesion recognition in the prokaryotic and eukaryotic nucleotide excision DNA repair (NER) systems is presented. The procedures of DNA and protein sample preparations are described and experimental as well as analytical details of the experiments are provided. The data allow important conclusions on the strategies by which target site verification may be achieved by the NER proteins. Interestingly, they indicate different approaches of lesion recognition and identification for the eukaryotic NER system, depending on the type of lesion. Furthermore, distinct structural properties of the two different helicases involved in prokaryotic and eukaryotic NER result in and explain the different strategies observed for these two systems. Importantly, these experimental and analytical approaches can be applied not only to the study of DNA repair but also very similarly to other DNA interacting protein systems such as those involved in replication or transcription processes.
Introduction
المجهر القوة الذرية (عفم) هو تقنية قوية لتحليل التفاعلات البروتين الحمض النووي 1 ، 2 ، 3 ، 4 ، 5 ، 6 ، 7 ، 8 ، 9 . وهو يتطلب كميات قليلة فقط من المواد عينة لتصور مباشرة عينات غير متجانسة مع قرار على مستوى جزيء واحد. يمكن أن ينتج عدم التجانس من مختلف حالات التوافقية أو أوليغوميريك من البروتين. على وجه الخصوص، في سياق عينات من الحمض النووي للبروتين، يمكن أن المجمعات البروتين عرض مختلف الكيمياء المتجانسة و / أو التوافق الناجم عن الحمض النووي ملزمة بشكل عام أو ملزمة لموقع مستهدف معين داخل الحمض النووي. قد تحتوي عينات غير متجانسة أيضا اثنين (أو أكثر) أنواع مختلفة من البروتينات، ومختلفة البروتين معقدةأشكال (على سبيل المثال ، تتكون من نوع واحد فقط من البروتين مقابل المجمعات هيتيروميريك) قد تتفاعل بشكل مختلف مع الحمض النووي. الدراسات التي نوقشت هنا استغلال التصوير عفم في الهواء على عينات ثابتة، المجففة من البروتينات إصلاح الحمض النووي إلى طويلة (~ 900 أزواج قاعدة، بي بي) شظايا الحمض النووي التي تحتوي على الآفة، وهو ما يمثل هدفا لهذه البروتينات. القرار الجزيئي العالي ل عفم يسمح بالتمييز بين أنواع مختلفة من المجمعات البروتينية وتحديد المواقف الملزمة للبروتينات على شظايا الحمض النووي. الأهم من ذلك، يتم إدخال الآفات في ركائز الحمض النووي في مواقف محددة جيدا. لأنه من المعروف موقع موقع الآفة في الحمض النووي، وتوزيعات البروتينات ملزمة على الحمض النووي توفر نظرة ثاقبة (مختلفة) خصائص التعرف على الآفات من (مختلف) المجمعات البروتين، على سبيل المثال ، مدى أنها تعترف نوع معين من الآفة (مقارنة إلى الحمض النووي غير التالفة) 2 ، 3 ، 4 ، 5 ، 6 . مواقفهم على الحمض النووي تسمح أيضا التمييز بين المجمعات البروتين ملزمة على وجه التحديد في الآفات والمجمعات ملزمة غير محددة في أماكن أخرى على الحمض النووي. توصيف منفصل من هذه الأنواع المعقدة المختلفة (المجمعات المرتبطة تحديدا في الآفة مقابل المجمعات غير محددة) يمكن أن تكشف عن التغييرات التوافقية المحتملة في المجمعات التي يسببها تحديد الموقع المستهدف.
البروتينات إصلاح الحمض النووي التي تركز على هنا هي هيليكاسيس التي هي المسؤولة عن التعرف على الآفات في إصلاح ختان النوكليوتيدات (نر) المسار. في البكتيريا، ويتحقق نر من البروتينات أوفرا، أوفرب، و أوفرك. أوفرا هو المسؤول عن استشعار الآفة الأولية في أوفا 2 / أوفرب 2 الحمض النووي المسح الضوئي المعقدة. بعد التحقق من الآفة من قبل أوفرب تحول هذا المجمع إلى أوفرب أحادية الحد ملزمة في موقع الآفة وهذا المجمع المحدد يمكن بعد ذلك تجنيد pداء النواة النواة نونوكلياز نونوكلياز. أوفرك يكيس قصيرة (12-13 نت) تمتد من الحمض النووي الذين تقطعت بهم السبل واحد (سدنا) التي تحتوي على الآفة. ثم يتم إعادة ملء المفقود من قبل البلمرة الحمض النووي. وأخيرا، دنا يغاز الأختام تمتد حديثا تمدد مع الحمض النووي الأصلي 9 ، 10 . في حقيقيات النوى، معظم البروتينات من سلسلة نر هي جزء من مجمع النسخ مولتيمريك كبير H H (تفيه) معقدة. بعد الآفة الأولية الاستشعار عن طريق مجمع CEN2-شيك-HR23B تريمي، يتم تعيين تفييه إلى موقع الهدف الحمض النووي. عندما شد داخل مجمع يتحقق من وجود آفة الهدف نر، يتم تعيين إندونوكليس نر حقيقية النواة النواة زغ و زف لاستئصال قصيرة (24-32 نت) تمتد من سدنا تحتوي على الآفة 9 ، 10 . هنا، على وجه التحديد، و هيليكاسيس أوفرب و شد من بدائية النواة و نر حقيقية النواة، على التوالي، ودرس. هذه هيليكاسيس تتطلب منطقة غير مزدحمة فيالحمض النووي (فقاعة الحمض النووي) لخيوط على واحد من اثنين من الحمض النووي فروع واحدة وترجمة بعد ذلك على طول هذا حبلا تغذيها أتب التحلل. بالإضافة إلى آفات الحمض النووي، وبالتالي تم إدخال فقاعة الحمض النووي في ركائز التي تعمل كموقع تحميل للبروتينات.
وقد وصف الإجراء لإعداد ركائز الحمض النووي الآفة محددة سابقا 11 . فإنه يتطلب بناء دنا دائري (البلازميد) مع اثنين من مواقع تقييد متباعدة عن كثب لنيكاز. في سياق هذه الدراسة، تم استخدام البلازميد pUC19N (2729 بي بي) (التي تم إنشاؤها بواسطة مختبر S. ويلسون، نيهس). هذا البلازميد يحتوي على ثلاثة مواقع تقييد متباعدة عن كثب ل Nt.BstNBI النيك التي تأطير 48 النيوكليوتيدات (نت) تمتد. بعد الحضانة مع نيكيس، يمكن إزالة امتداد سدنا بين هذه المواقع واستبدالها من قبل قليل النوكليوتيدات التي تحتوي على أي ميزة الهدف. بعد كل خطوة، يتم اختبار الهضم الأنزيمي الكامل عن طريق هلام الاغاروزالكهربائي. يمكن تمييز الحمض النووي دائري نيكيد بسبب انخفاض التنقل الكهربي مقارنة البلازميد سوبيركويلد الأصلي. ويمكن تقييم الفجوات من الحمض النووي واستبدال تمتد إزالتها من قبل قليل النوكليوتيد الركيزة محددة عن طريق الهضم مع انزيم تقييد الذي يحرض الركيزة حصرا داخل المنطقة بين النكات. ومن ثم سيتم قمع الخطي من البلازميد دائري من قبل الانزيم للحمض النووي المنبثق واستعادة بعد إدراج أوليغونكلأوتيد محددة. وأخيرا، فإن اثنين من مواقع تقييد نوكلياز داخلية (من الناحية المثالية القواطع واحدة) تسمح لتوليد الركيزة الحمض النووي الخطي، مع طول كما هو مطلوب ومع موقع الهدف المحدد في موقف محدد وكذلك فقاعة الحمض النووي على مسافة من الآفة إما في 5 'أو 3' الاتجاه.
التعرف على الآفات التي كتبها نيل هيليكاسيس يمكن التحقيق عن طريق التصوير عفم. توقفت الحمض النووي نقل من هيليكاسيس في رموقع الآفة مرئية كذروة في توزيع موقف البروتين على الحمض النووي ويشير إلى التعرف على الآفات. لأن نقل الحمض النووي لهذه هيليكاسيس هو علاوة على ذلك الاتجاه، مع 5 إلى 3 'قطبية، والاعتماد على الاعتراف الآفة على موقع موقع التحميل (فقاعة دنا المنبع أو المصب من الآفة) يشير أيضا إلى ما إذا كانت الآفة معترف بها بشكل تفضيلي على ترانزلاتوكاتد أو على العكس، غير ترانزلاتوكات سدنا حبلا 5 ، 9 . في الأقسام التالية، سيتم عرض الأساليب المستخدمة وسيتم مناقشة النتائج الرئيسية من هذه التجارب بإيجاز. الأهم من ذلك، على غرار العمل المثالي على إصلاح الحمض النووي هو مبين هنا، والتصوير عفم يمكن تطبيقها على دراسة مختلف النظم المتفاعلة الحمض النووي، مثل تكرار الحمض النووي أو النسخ 8 و 12 و 13 و 14 .
Protocol
1. إعداد العينة
- إعداد ركائز الحمض النووي 11
- توليد سدنا الفجوة في البلازميد
- هضم تماما عينة من البلازميد (هنا: تعديل pUC19، pUC19N) في أنبوب التفاعل مع النكاز المناسب (هنا: Nt.BstNBI) تليها انزيم الحرارة تعطيل، وذلك باستخدام الظروف وفقا لبروتوكول الشركة المصنعة (انظر الشكل 1 للتخطيطي عرض). تبدأ مع ~ 50 ميكرولتر و ~ 500 نانومتر البلازميد للحصول على عائد كاف.
- التحقق من البلازميد النك بواسطة الاغاروز هلام الكهربائي على عينات المخفف (~ 20 نانومتر) 15 . ارتداء قفازات للحماية من الحمض النووي ملزمة صبغ تستخدم لتصور الحمض النووي.
ملاحظة: التحركات الكهربي مختلفة تسمح التمييز بين النيكل (استرخاء) و سوبيركويلد دائري البلازميد دائري ( الشكل 1 ). - إزالة تمتد سدنا المحطمة (بين مواقع نيك) من البلازميد من قبلالحضانة مع ~ 10 أضعاف الزائدة من أوليغونكلأوتيد التكميلية (أوليغونوكلأوتيد 1 في الجدول 1 )، والهز في 300 دورة في الدقيقة لمدة 30 دقيقة بالقرب من درجة حرارة انصهار أوليغونكلأوتيد (هنا: 68 درجة مئوية ل pUC19N) في كتلة الحرارة ( الشكل 1 ).
- فصل البلازميد الانفجارات من شظايا الحمض النووي أصغر باستخدام 50 كيلو دالتون الوزن الجزيئي قطع (موكو) تصفية بواسطة الطرد المركزي لمدة 10 دقيقة في 10000 x ج في الطرد المركزي الجدول ( الشكل 1 ). لاستخراج الحمض النووي المركزة من مرشح، عكس مرشح وإدراجها في أنبوب رد فعل 1.5 مل جديد. أجهزة الطرد المركزي لمدة 3 دقائق في 1000 × ز.
- إعادة ملء العينة الحمض النووي المركزة الناتجة إلى 500 ميكرولتر مع منزوع الأيونات، المياه التي تمت تصفيتها، إضافة إضافية ~ 5 أضعاف الزائدة من قليل النوكليوتيد التكميلي وكرر الخطوات 1.1.1.3 و 1.1.1.4 3 مرات على الأقل.
- اختبار للفجوات كاملة من الحمض النووي عن طريق الحضانة مع انزيم تقييد مناسب (هنا: شوي أو بغلي) باستخدام كونديتيأونس وفقا لوصف الشركة المصنعة. استخدام المخفف (نيك مقابل مقابل) عينات الحمض النووي (~ 20 نانومتر). تشغيل الاغاروز هلام الكهربائي للتمييز بين البلازميد الخطي (التي لا تحتوي على فجوة سدنا) والحمض النووي غير منقوشة (الحمض النووي غاب) باستخدام الحمض النووي نك كما السيطرة الإيجابية (تشمل سلم الحمض النووي كمرجع، الشكل 1 ). ارتداء القفازات.
- إعادة ملء الفجوة مع أوليغونوكلأوتيدس سدنا تعديل
- أنال البلازميد عن طريق الحاضنة مع زيادة ~ 25 أضعاف من 5 'أوليغونوكليوتيد فوسفهوريلاتيد الذي يحتوي على موقع الهدف المحدد (ق) من الاختيار، واحتضان في ~ 45 درجة مئوية لمدة 4 ساعات ( الشكل 1 ). هنا، استخدم 48 نت سدنا تحتوي على آفة، إما فلوريسئين ثيمين أدوكتد أو سيكلوبوتان بيريميدين ديمر (كبد)، وبالإضافة إلى ذلك قصيرة (8 نت) تسلسل غير تكميلي لإنتاج فقاعات الحمض النووي (انظر الجدول 1 ).
- تساهميا ربط إدراج صلب إلى البلازميد التي كتبها إنكوبامع T4 الحمض النووي ليغاز بين عشية وضحاها في درجة حرارة الغرفة وفقا لبروتوكول الشركة المصنعة ( الشكل 1 ). لهذا التفاعل، إضافة أتب تحتوي على محلول المخزون العازلة المركزة لإنتاج الظروف العازلة مناسبة لليغاز (على سبيل المثال ، استخدام العازلة رد أوفرب: 50 ملي تريس هكل درجة الحموضة 7.5، 10 ملي مغكل 2 ، 50 ملي بوكل، 5 ملي دت، 1 ملم ATP).
- اختبار لإدراج أوليغونكلأوتيد محددة في البلازميد عن طريق الهضم من العينات المخففة مع انزيم تقييد مناسبة (كما هو الحال في 1.1.1.6) وفقا لبروتوكول الشركة المصنعة تليها الكهربائي هلام الاغاروز (كما هو الحال في 1.1.1.6، الشكل 1 ).
- إعداد الركيزة الحمض النووي الخطي
- هضم تعديل الحمض النووي البلازميد مع الانزيمات تقييد (من الناحية المثالية مع موقع تقييد واحد في البلازميد) باستخدام الظروف الموصى بها من قبل الشركة المصنعة ( الشكل 1 ). تنتج هذه الخطوة شظايا خطية تحتويإدخال التعديل المدرج في موضع محدد. هنا، استخدم سسبي وقطع بسبكي في مواقف 613 و 255 نقطة أساس المنبع والمصب من إدراج، على التوالي، مما أدى إلى 916 شظية الحمض النووي جزء يحتوي على موقع الآفة محددة في ~ 30٪ من طول الحمض النووي.
ملاحظة: لتجارب التصوير عفم كما هو موضح هنا، شظايا الحمض النووي مع أطوال بين ~ 200 بب و ~ 2000 بب هي ركائز مناسبة. - عزل شظية الهدف عبر الاغاروز هلام الكهربائي واستخراج هلام باستخدام مجموعة التجارية. ارتداء قفازات للحماية.
- اختياريا، وقطع هلام الاغاروز مع مشرط لفصل سلم الحمض النووي والممرات عينة المخفف (الممرات الأولى) من الممرات التي تحتوي على الحمض النووي المركزة لتنقيته. فقط تعريض جزء هلام مع الممرات الأولين للأشعة فوق البنفسجية عن طريق وضع هذا الجزء فقط من هلام على طاولة الأشعة فوق البنفسجية.
- قطع الفرقة المقابلة لجزء من الاختيار مع مشرط. إعادة توحيد اثنين من أجزاء هلام (قبالة علامة التبويب الأشعة فوق البنفسجيةلو). استخدام موقف الفرقة استئصال من العينة المخففة والتوجه لموقف الفرقة من الركيزة الحمض النووي المطلوب. حساب لتركيز أعلى من خلال قطع شرائح أوسع قليلا من السيطرة المخففة.
ملاحظة: لأنه هنا، تم التحقيق من التعرف على الأشعة فوق البنفسجية الآفات، وإدخال الآفات الأشعة فوق البنفسجية إضافية عن طريق أشعة فوق البنفسجية تم تجنبها بعناية في الركيزة الحمض النووي من خلال هذا النهج.
- قطع الفرقة المقابلة لجزء من الاختيار مع مشرط. إعادة توحيد اثنين من أجزاء هلام (قبالة علامة التبويب الأشعة فوق البنفسجيةلو). استخدام موقف الفرقة استئصال من العينة المخففة والتوجه لموقف الفرقة من الركيزة الحمض النووي المطلوب. حساب لتركيز أعلى من خلال قطع شرائح أوسع قليلا من السيطرة المخففة.
- حساب تركيز الحمض النووي ج من امتصاص في 260 نانومتر يقاس من قبل طيف الأشعة فوق البنفسجية فيس باستخدام قانون لامبرت البيرة مع الحمض النووي المزدوج تقطعت بهم السبل (دسدنا) متوسط معامل الانقراض المولي من ε 260nm ~ 6،700 M - 1 سم - 1 لكل بب:
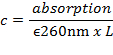
حيث L هو طول المسير (طول غرفة القياس، عادة 1 سم).
- هضم تعديل الحمض النووي البلازميد مع الانزيمات تقييد (من الناحية المثالية مع موقع تقييد واحد في البلازميد) باستخدام الظروف الموصى بها من قبل الشركة المصنعة ( الشكل 1 ). تنتج هذه الخطوة شظايا خطية تحتويإدخال التعديل المدرج في موضع محدد. هنا، استخدم سسبي وقطع بسبكي في مواقف 613 و 255 نقطة أساس المنبع والمصب من إدراج، على التوالي، مما أدى إلى 916 شظية الحمض النووي جزء يحتوي على موقع الآفة محددة في ~ 30٪ من طول الحمض النووي.
- توليد سدنا الفجوة في البلازميد
- التعبير وتنقية البروتينات
- ريكومبينانتلي التعبير عن أوفرب في E. القولونية وتنقية البروتين عن طريق معيار الكيتين تقارب حبة 15 وحجم الاستبعاد اللوني 15 كما هو موضح سابقا 16 .
ملاحظة: تم استنساخ الجين أوفرب من عصيات كالدوتيناكس في ناقلات pTYB1. - التعبير عن شد في E. القولونية وتنقية البروتين عن طريق معيار النيكل إيدا تقارب 15 واللون استبعاد اللوني 15 تليها أنيون التبادل اللوني 15 ، كما هو موضح سابقا 17 .
ملاحظة: تم استنساخ الجين ل شيتوميوم ثيروفوفيلوم زيروديرما بيغمينتوسوم المجموعة D البروتين (شد) في ناقلات pBADM11. C. كان ثيرموفيلوم p44 هدية نوع من مختبر كارولين كيسكر، وأعرب ونقيكما ورد وصفه 17 .
- ريكومبينانتلي التعبير عن أوفرب في E. القولونية وتنقية البروتين عن طريق معيار الكيتين تقارب حبة 15 وحجم الاستبعاد اللوني 15 كما هو موضح سابقا 16 .
2. تجربة عفم
- إعداد عينة
- اختياريا، قبل احتضان الركيزة الحمض النووي عند 65 درجة مئوية لمدة 10 دقيقة في كتلة الحرارة لإزالة بلورات الملح الصغيرة المحتملة التي قد تكونت خلال التخزين في الثلاجة.
- إعداد العازلة رد فعل (ق) في تركيز 10 أضعاف (10X مخازن).
ملاحظة: رد فعل رد شد في تركيز 1X الواردة 20 ملي تريس-حمض الهيدروكلوريك درجة الحموضة 7.5، 10 ملي بوكل، 5 ملي مغكل 2 ، 1 مم تسيب، 2 ملم أتب. 1x أوفرب رد فعل العازلة الواردة 50 ملي تريس-حمض الهيدروكلوريك درجة الحموضة 7.5، 50 ملي بوكل، 10 ملي مغكل 2 ، 5 ملي دت، و 1 ملم أتب. - قبل احتضان البروتينات في تركيز أعلى في ظروف حضانة مناسبة لتعزيز تشكيل معقدة. قبل تمييع حجم صغير (على سبيل المثال ، 1 ميكرولتر) من البروتينات الفردية في 1X العازلة رد فعل البروتين إلى التركيز المطلوب وتخلط كميات صغيرة (على سبيل المثال ، 1 ميكرولتر) من روقال انه حلول البروتين الفردية في أنبوب رد فعل 0.5 مل.
- وضع أنبوب في كتلة الحرارة لدرجات حرارة الحضانة أعلى من درجة حرارة الغرفة. اختيار نسبة تركيز اعتمادا على الكيمياء المتكافئة المعقدة المتوقعة، إما متساوي الأقطاب أو تركيزات المقابلة. هنا، احتضان 1 ميكرولتر شد (20 ميكرومتر في 1 × رد فعل رد شد) و 1 P44 ميكرولتر (20 ميكرومتر في 1X رد فعل رد شد) في 10 ميكرومتر كل لمدة 10 دقيقة عند 37 درجة مئوية.
- احتضان عينات في البروتينات المناسبة وتركيزات الحمض النووي في المخزن المؤقت رد فعل البروتين في أنبوب رد فعل 0.5 مل. هنا، استخدم 500 نانومتر أوفرب أو 1 ميكرومتر شد + 1 ميكرومتر P44 و 100 نانومتر الحمض النووي. ماصة أحجام صغيرة لحفظ المواد، على سبيل المثال ، 0.25-0.5 بروتين ميكرولتر (المخفف مسبقا إلى 10 أضعاف تركيز الحضانة) والحمض النووي في حجم إجمالي 2.5-5 ميكرولتر من 1X العازلة رد فعل البروتين. هنا، احتضان لمدة 30 دقيقة عند 37 درجة مئوية في كتلة الحرارة. تدور باستمرار في أنبوب رد فعل لفترة وجيزة (~ 1 ثانية) في الطرد المركزي الجدوله لضمان خلط أحجام صغيرة.
- عينة ترسب
- إعداد الركيزة الميكا: قطع ما يقرب من 1 × 1 سم 2 قطعة من الميكا من شرائط أكبر باستخدام مشرط. تخرج من الطبقات العليا للطبقة المعدنية الميكا متعددة الطبقات باستخدام شريط لاصق للكشف عن نظيفة، مسطحة، وعلى نحو سلس ركيزة سطح الركيزة.
ملاحظة: قطعة الميكا يمكن إعادة استخدامها لتجارب متعددة عن طريق تجريد طبقة أخرى (ق). - إعداد العازلة ترسب عفم مع منزوع الأيونات، المياه التي تمت تصفيتها، على سبيل المثال ، 25 ملي هيبيس الرقم الهيدروجيني 7.5، 25 ملي نا خلات، 10 ملم مغ أسيتات. تصفية من خلال مرشح حقنة 0.02 ميكرون.
ملاحظة: الكاتيونات ثنائي التكافؤ في المخزن المؤقت ترسب تعمل على خلب جزيئات الحمض النووي سالبة الشحنة إلى سطح الميكا، وهو أيضا مشحونة سلبا في الرقم الهيدروجيني محايد. إذا كانت عالية نسبيا مغ 2 + تركيز أيون في المخزن المؤقت ترسب يطرح مشكلة لنظام معين من البروتين الحمض النووي، بدلا من ذلك،يمكن أن يكون سطح الميكا محملة مسبقا مع مجموعات الأمينية باستخدام الكيمياء القائمة على سيلاتران لتوفير رسوم سطح إيجابية لرسو 18 . ويمكن بعد ذلك ترسب عينة يتم تنفيذها في المخزن المؤقت الذي يحتوي على أو كميات قليلة فقط من الكاتيونات ثنائي التكافؤ. - تمييع العينة (انظر 2.1) لترسب فوري على الميكا في العازلة ترسب. إيداع حجم صغير (هنا: 20 ميكرولتر).
ملاحظة: عوامل التخفيف تعتمد على تركيز العينة. هنا، تمييع عينات 50-100x. كقاعدة من الإبهام، ~ 1 نانومتر النتائج الحمض النووي في تغطية سطح جيدة ل ~ 1000 نقطة أساس. - على الفور شطف عينة ثلاث إلى أربع مرات مع بضعة ملليلتر من المياه المصفاة منزوع الأيونات، وصمة عار قبالة السائل الزائد، وضربة الجافة في تيار لطيف من النيتروجين. العملية برمتها من عينة ترسب إلى عينات المجففة يمكن تنفيذها ضمن أقل من 30 ثانية.
- إصلاح قطعة من الميكا على شريحة المجهر باستخدام شريط لاصق في حوافها.
ملاحظة: أنظمة عفم مختلفة لديها دمتطلبات إفيرنت لتحديد العينة إلى المرحلة. وتمتلك أدمس أخرى مراحل مغناطيسية، ويتم تثبيت قطع الميكا على الأقراص المغناطيسية، على سبيل المثال ، باستخدام الغراء الحراري. تفاصيل نظام عفم المستخدمة هنا يمكن العثور عليها في جدول المواد.
- إعداد الركيزة الميكا: قطع ما يقرب من 1 × 1 سم 2 قطعة من الميكا من شرائط أكبر باستخدام مشرط. تخرج من الطبقات العليا للطبقة المعدنية الميكا متعددة الطبقات باستخدام شريط لاصق للكشف عن نظيفة، مسطحة، وعلى نحو سلس ركيزة سطح الركيزة.
- عفم التصوير
- وضع العينة (انظر 2.2.5) مركزيا على مرحلة عفم وإصلاح شريحة المجهر على خشبة المسرح مع منصات المغناطيسي.
- إدراج طرف عفم في حامل طرف. استخدام الكابولي مع حاد (<10 نانومتر) عفم التحقيق مناسبة للتأرجح، متقطع وضع الاتصال الاتصال في الهواء. استخدام تحقيقات عفم ، على سبيل المثال، كما هو موضح في جدول المواد . هنا (يعتمد على عفم)، إصلاح طرف في حامل تحت المشبك عن طريق تشديد المسمار المشبك (إصبع ضيق). أدخل حامل طرف في رأس قياس عفم. بقية الرأس على ظهره لهذه الخطوة.
- وضع رأس قياس عفم على رأس العينة. التفاصيل تعتمد على نموذج عفم. هنا، متأكد من أن الرأس يقف مستقر مع ساقيه داخل المسافات البادئة. تأكد من أن الميكا تقع على خشبة المسرح حيث سوف تلميح عفم تحوم مباشرة فوقه. مسامير ميكرومتر على يمين المرحلة تسمح لتحديد المواقع بدقة من العينة.
- محاذاة الليزر عفم على الجزء الخلفي من ناتئ لقوة إشارة الأمثل من موقف فوتوديتكتور حساسة على ينعكس الليزر. التفاصيل تعتمد على نموذج عفم.
- هنا، وتحويل العجلات في الجانب الأيمن والجزء الخلفي من رئيس قياس عفم لضبط X- و Y- مواقف الليزر عفم لتوجيهه مركزيا على نهاية ناتئ. مشاهدة إشارة الانعكاس في إطار الفيديو عفم (إذا كان متوفرا، هنا: اضغط على أيقونة الكاميرا واختر الإدخال: سفيديو).
- مرة واحدة في وضع فظيع، وتحسين إشارة مجموع الكشف (مجموع في مجموع ونافذة متر نافذة في برنامج عفم) عن طريق ضبط دقيق موقف الليزر مع عجلتين (البقاء في نهاية ناتئ،لتر يعتمد على عفم ونوع ناتئ، هنا: الهدف للمبلغ> 5).
- صفر إشارة الفرق من الثنائيات العلوية والسفلية من مجموعة الكاشف (هنا: إشارة الانحراف في مجموع ونافذة متر نافذة) عن طريق توجيه انعكاس الليزر عفم على مركز الكشف (هنا: تحويل عجلة في الجانب الأيسر من قياس عفم رئيس).
ملاحظة: الانحرافات من الصفر ثم تشير إلى انحراف ناتئ بسبب التفاعلات السطحية، والتي يتم ترجمتها إلى معلومات الارتفاع من قبل عفم. - تحديد تردد الرنين ناتئ بواسطة لحن تردد تنفيذها في برنامج عفم (هنا: قيادة السيارات لحن في لوحة ماستر / نافذة اللحن). اختيار السعة المقابلة إلى 1 V المدخلات لبيزو الذي يدفع التذبذب ناتئ. تعيين تردد التذبذب إلى قليلا (5٪) أقل من تردد الرنين وصفر مرحلة من التذبذب.
- نهج طرف إلى سطح العينة باستخدام الخام الانخراط وزارة الدفاعe (هير: كوماند إنغاج في نافذة اللوحة الرئيسية) حتى يتم الوصول إلى إعداد الحماية (نقطة مجموعة). استخدام ~ 2٪ قطع من السعة الحرة التذبذب مستوى كنقطة مجموعة (هنا، أدخل تعيين نقطة 980 مف في اللوحة الرئيسية).
- غرامة إشراك طرف عفم مع سطح العينة عن طريق خفض نقطة مجموعة باستخدام برنامج عفم. تهدف للتصوير وضع التنافر مع مرحلة التذبذب ناتئ (المرحلة في مجموع وانحراف متر نافذة) فقط تحت مرحلة مستوى الحرة (قبل الانخراط، وهنا عادة ~ 70). هنا، استخدام نقاط مجموعة النهائية النموذجية على الترتيب من 70-80٪ من السعة مستوى الحرة (1 V).
- قبل المسح الضوئي، اختر الإشارات للتسجيل في لوحة القناة الرئيسية. اختر الارتفاع (هت) والسعة (آم).
- بدء المسح الضوئي عينة (هنا: الأمر مسح في إطار اللوحة الرئيسية). استخدام سرعة المسح الضوئي على سبيل المثال، 2.5 ميكرون / ثانية (الأمر سرعة المسح الضوئي في اللوحة الرئيسية) إلى مناطق سطح الصورة من 4 ميكرون × 4 ميكرون أو 8 ميكرون × 8 ميكرون (أدخل حجم المسح الضوئي في الجزء الرئيسيل) مع دقة بكسل من 2،048 أو 4،096 (نقاط المسح وخطوط المسح الضوئي في اللوحة الرئيسية)، على التوالي.
- حفظ ملف الصورة (الأمر حفظ الصورة في اللوحة الرئيسية) مع عدم وجود تعديلات المناسب (اختيار لا شيء في لوحة القناة الرئيسية / حفظ الطائرة صالح).
- لمزيد من التحليل، معالجة الصورة المحفوظة. تحميل الصورة (الأمر استعراض البيانات المحفوظة في القائمة تحليل عفم). افتح لوحة التعديل (اضغط على M في القسم العلوي من الصورة). تطبيق بلانيفيت في x و y أبعاد إلى صورة الارتفاع (ملحق هتر) (الأمر زي في نافذة بلانيفيت في لوحة تعديل، اختيار بلانفيت النظام 3). ثم تسطيح الصورة (الأمر تتسطح في نافذة فلاتن في تعديل لوحة) اختيار تسطيح النظام 3.
- تصدير الصورة كملف تيف (اضغط على أوامر في الجزء العلوي من الصورة، واختيار تيف تصدير 2X المقابلة ل 2048 بكسل القرار) لمزيد من التحليل.
3. تحليل عفم
- البروتين حجم مجمع
- ما قبلحدد المجمعات البروتين الحمض النووي ذات الصلة عن طريق الفحص البصري المباشر من الصور في برنامج عفم، وذلك باستخدام نظام الألوان المناسبة لتحقيق أقصى قدر من المظاهر المختلفة من المجمعات حجم مختلفة (انظر ألوان وأحجام مختلفة من المجمعات المختلفة في الشكل 2 ؛ هنا: نظام الألوان سيلانداندفير في و أفمسوفتوار). بالنسبة لعينات شد، شملت المضاعفات المحتملة شد / p44-دنا وكذلك شد -دنا و p44-دنا، مع أحجام مختلفة بشكل واضح بسبب الكتلة الجزيئية الكبيرة نسبيا والمختلفة من شد (~ 95 كيلو دالتون) و p44 (~ 40 كيلو دالتون) .
- قياس كميات من قمم البروتين الفردية على الحمض النووي للتحقق من نوع معقد. ويمكن تحقيق ذلك مع برنامج صورة مختلفة (هنا: أداة القسم من برنامج عفم).
- قياس ارتفاع (ح) وقطر (د) من أقسام ذروة البروتين مع مؤشر نافذة القسم. قياس القطر على مقربة من قاعدة قسم الجسيمات.
- تحديد حجم (V) من البازلاءكس باستخدام نماذج رياضية بسيطة، على سبيل المثال ، باستخدام الصيغة التالية، التي تقوم على نموذج غطاء كروي:
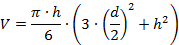
- ترجمة الأحجام تقاس في كتلة البروتين الجزيئي التقريبي.
- في البداية، ومعايرة نظام عفم للتحجم إلى الوزن الجزيئي (مو) باستخدام مجموعة من البروتينات ذات الوزن الجزيئي المعروف (هنا: ما مجموعه 12 التجارب مع ما بين 25 و 851 نقطة البيانات كل أجريت على 5 بروتينات مختلفة في أحادية، ديميريك ، تريميريك، أو تيترامريك الدول) 4 ، 19 ، 20 . بروتينات الإيداع والصورة (كما هو موضح أعلاه، في 2.2 و 2.3) وقياس أحجامها كما هو موضح أعلاه (3.1.2). رسم الأحجام على الأوزان الجزيئية المعروفة. ويظهر الرسم البياني الناتج علاقة خطية بين V و مو، يمكن الحصول على معادلة من خلالهاخط يصلح للبيانات. بالنسبة لنظام عفم المستخدم هنا، تم الحصول على العلاقة التالية 19 :
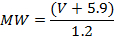
ملاحظة: هذه الخطوة لا يجب أن تتكرر قبل كل قياس، ولكن يتم فقط مرة واحدة. ويمكن تطبيق حجم مو المعايرة على الصور التي تم الحصول عليها في ظل ظروف التصوير مماثلة باستخدام تحقيقات عفم مع أقطار متفاوتة قليلا. - استنادا إلى حجمها المقاسة (3.1.2) ومعايرة V- إلى- مو (3.1.3.1)، وتحديد مو التقريبية من المجمعات البروتين 20 ، الذي يوفر معلومات عن المحتوى الجزيئي لها. على سبيل المثال ، بالنسبة لعينات شد / p44، ~ 50 كيلو دالتون، ~ 100 كيلو دالتون، و ~ 140 كيلو دالتون تم الحصول عليها، بما يتفق مع المجمعات دنا p44 (أو قمم الناجمة عن مجرد البنية الفوقية الحمض النووي)، شد قمم فقط، و شد / P44 قمم، على التوالي .
- في البداية، ومعايرة نظام عفم للتحجم إلى الوزن الجزيئي (مو) باستخدام مجموعة من البروتينات ذات الوزن الجزيئي المعروف (هنا: ما مجموعه 12 التجارب مع ما بين 25 و 851 نقطة البيانات كل أجريت على 5 بروتينات مختلفة في أحادية، ديميريك ، تريميريك، أو تيترامريك الدول) 4 ، 19 ، 20 . بروتينات الإيداع والصورة (كما هو موضح أعلاه، في 2.2 و 2.3) وقياس أحجامها كما هو موضح أعلاه (3.1.2). رسم الأحجام على الأوزان الجزيئية المعروفة. ويظهر الرسم البياني الناتج علاقة خطية بين V و مو، يمكن الحصول على معادلة من خلالهاخط يصلح للبيانات. بالنسبة لنظام عفم المستخدم هنا، تم الحصول على العلاقة التالية 19 :
- المواقف المعقدة البروتين على الحمض النووي
- تحديد الحمض النوويأطوال الشظايا.
- تتبع شظايا الحمض النووي في الصور عفم، على سبيل المثال ، مع وظيفة خط يدوي من برنامج تحليل صورة مناسبة (انظر على سبيل المثال جدول المواد) وقياس طول الخط.
ملاحظة: استبعاد المجاميع الحمض النووي وكذلك شظايا قطعت من قبل هوامش الصورة. - بن ورسم أطوال الحمض النووي من التجربة برمتها في الرسم البياني، وذلك باستخدام تحليل البيانات المناسبة والرسوم البيانية البرمجيات (على سبيل المثال ، انظر جدول المواد ).
- تناسب توزيع طول مع منحنى غاوس في تحليل البيانات والرسوم البيانية البرمجيات لتحديد طول شظايا الحمض النووي. من أجل معرفة موقع الموقع المستهدف للدنا (انظر 1.1)، لا تشمل سوى شظايا الحمض النووي ذات الطول الصحيح ضمن انحرافين معياريين عن مركز منحنى غاوس (y = y 0 + z إكس (-2 (شكس c ) 2 / w 2 )، حيث z هو عامل القاعدة و x c و w هي المركز وكامل أقصى نصف العرض من غاوس) في مزيد من التحليلات.
ملاحظة: طول الحمض النووي في وسط منحنى غاوس يجب أن يكون قريبا من الطول النظري لشظية الحمض النووي (محسوبة باستخدام 0.34 نانومتر / بي بي). أطوال تصل إلى 10٪ أقصر من القيمة النظرية هي نموذجية ومن المرجح أن تسببها حدود القرار عفم.
- تتبع شظايا الحمض النووي في الصور عفم، على سبيل المثال ، مع وظيفة خط يدوي من برنامج تحليل صورة مناسبة (انظر على سبيل المثال جدول المواد) وقياس طول الخط.
- تحديد مواقف قمم البروتين على ركائز الحمض النووي.
- قياس المسافة من قمم البروتين من نهاية جزء الحمض النووي أقرب، كما هو موضح في 3.2.1.1 وتقسيم طول الحمض النووي الكلي للحصول على مسافات في وحدات من جزء من طول الحمض النووي.
- بن ورسم المسافات المقاسة في الرسم البياني باستخدام تحليل البيانات المناسبة والرسوم البيانية البرمجيات (انظر على سبيل المثال ، الشكل 3 ). وكقاعدة عامة، اختر حجم بن الذي يعطي حاويات √n تقريبا لنقاط البيانات n.
ملاحظة: منذ ينتهي الحمض النووي لا يمكن تمييز هنا، مؤامرة فقط لجزء من طول الحمض النووي 0.5 (مركز دنا فراجمنة). لأن نهاية الحمض النووي ملزمة ليست محور الدراسة، استبعاد ينتهي الحمض النووي من التحليلات عن طريق البدء في بن البيانات الموقف قليلا بعد موقف 0 (على سبيل المثال ، بينينغ من جزء طول الحمض النووي من 0.02).
- تحديد خصوصية الموقع المستهدف من البروتين توزيعات الموقف المعقدة.
- تناسب الحد الأقصى (أحداث الربط المعززة) في توزيع المواضع بمنحنى غاوس كما هو موضح في الفقرة 3.2.1.3 ولكن تم تثبيته على ارتفاع ارتباط الخلفية (انظر الشكل 3 ).
- حساب خصوصية S لموقع معين مقابل الخلفية الحمض النووي غير محددة (جزيئات البروتين ملزمة لمواقع الحمض النووي غير محددة) مع الصيغة التالية 21
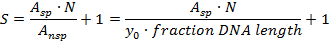
A سب: عدد من المجمعات محددة (منطقة تحت منحنى غاوسيان)
A نسب : عدد من المجمعات غير محددة (منطقة الخلفية، أي (طول الحمض النووي الكسر) x (متوسط ارتفاع الخلفية y 0 )؛ وجزء من طول الحمض النووي المغطاة هنا هو 0.48 للرسم البياني بدءا من 0.02 طول الحمض النووي)
N: عدد مواقع ربط الحمض النووي المحتملة (هنا: N = 914 باستثناء نهايات الحمض النووي)
- تحديد الحمض النوويأطوال الشظايا.
- زوايا منحنى الحمض النووي
- باستخدام أداة زاوية في برنامج تحليل صورة مناسبة، وقياس زاوية β بين سطرين وضعت مركزيا على طول العمود الفقري الحمض النووي وتركز في ذروة البروتين (انظر على سبيل المثال ، أقحم في الشكل 4 ). قياس الزوايا لعدد ذات الصلة إحصائيا من المجمعات الحمض النووي للبروتين (> 50، من الناحية المثالية> 100) 2 ، 3 ، 5 .
ملاحظة: يتم تعريف زاوية منحنى الحمض النووي كما 180 ° -β 2 ، 3 ، 5 . - استخدام تحليل البيانات والرسوم البيانية البرمجيات(انظر جدول المواد) تنتج منحنى توزيع زاوية الانحناء من قبل بينينغ زوايا منحنى الحمض النووي.
- تناسب توزيع زاوية الانحناء مع منحنى غاوس. إذا كان أكثر من الحد الأقصى هو واضح في التوزيع، واختيار متعددة الذروة غاوس صالح. مركز (مراكز) منحنى غاوس (ق) تعطي (ق) متوسط حالة زاوية الانحناء من الأنواع المحددة.
- إذا كان التحول في زاوية الانحناء (ق) (ماكسيما من تناسب غاوس) s هو واضح للتوزيعات، على سبيل المثال، من مختلف المتغيرات البروتين أو أنواع مختلفة من البروتين معقدة، وتطبيق الطالب t -test لتقييم مستوى أهمية التغييرات.
- باستخدام أداة زاوية في برنامج تحليل صورة مناسبة، وقياس زاوية β بين سطرين وضعت مركزيا على طول العمود الفقري الحمض النووي وتركز في ذروة البروتين (انظر على سبيل المثال ، أقحم في الشكل 4 ). قياس الزوايا لعدد ذات الصلة إحصائيا من المجمعات الحمض النووي للبروتين (> 50، من الناحية المثالية> 100) 2 ، 3 ، 5 .
النتائج
التمييز بين أنواع معقدة مختلفة على أساس وحدات التخزين المعقدة البروتين
يتم تنشيط النشاط هيليكاس من بدائية النواة نر هيليكاس أوفرب من الحمض النووي ملزمة 22 ، 23 . أو...
Discussion
تحليلات إحصائية عفم من المواقف ملزمة من البروتينات على شظايا الحمض النووي طويلة التي تحتوي على مواقع مستهدفة محددة يمكن أن تكشف عن تفاصيل مثيرة للاهتمام حول الاستراتيجيات المحددة المستخدمة من قبل البروتين للتعرف على هذه المواقع 2 ، 3 ...
Disclosures
الكتاب ليس لديهم ما يكشف.
Acknowledgements
PUC19N، أوليغونوكلأوتيدس التي تحتوي على كبد، و p44 تم توفيرها من قبل صمويل ويلسون، كوربينيان هيل وتوماس كاريل، و غودرون ميشلز وكارولين كيسكر، على التوالي. وأيد هذا العمل من المنح المقدمة من دويتشه فورسشونغزجيمينشافت (دفغ) FZ82 و تي-671/4 لتكنولوجيا المعلومات.
Materials
| Name | Company | Catalog Number | Comments |
| Molecular Force Probe (MFP) 3D | Asylum Research | N/A | atomic force microscope (AFM) |
| Precision 390 | DELL | N/A | computer |
| ThermoMixer and 1.5 mL block | Eppendorf | 5382000015 | heat block for DNA preparation |
| Rotilabo Block-Heater H 250 & blocks for 0.5 mL tubes | Carl Roth GmbH | Y264.1 & Y267.1 | heat block for protein-DNA incubations |
| Mini-Sub Cell GT | Bio-Rad Laboratories GmbH | 1704467 | electrophoresis chamber with gel caster and power supply |
| Power Pac Basic | Bio-Rad Laboratories GmbH | 1645050 | electrophoresis power supply |
| Centifuge 5415 D with rotor | Eppendorf | 2262120-3 | table centrifuge |
| Ultra-Lum electronic UV transillumonator MEB-15 | Ultralum | 900-1322-02 | UV irradiation table |
| NanoDrop ND-1000 | VWR International / PEQLAB Biotechnologie GmbH | N/A | UV spectrophotometer |
| TKAX-CAD with 0.2 μm capsule filter | Unity Lab Services | N/A | water deionization and filter unit |
| Name | Company | Catalog number | Comments |
| Software | |||
| MFP software on Igor Pro | Asylum Research | N/A | AFM software |
| ImageJ (open source Java image processing) | NIH Image | N/A | Image analysis software |
| Excel (Microsoft Office) | Microsoft Corporation | N/A | data analysis software |
| Origin9 / Origin2016 | OriginLab Corporation | N/A | statistical data analysis and graphing software |
| Name | Company | Catalog number | Comments |
| Material | |||
| OMCL-AC240TS | Olympus | OMCL-AC240TS | AFM cantilevers |
| grade V-5 muscovite | SPI Supplies | 1805 | mica sheets |
| Amicon Ultra 0.5 mL 50k Ultracell | Millipore Ireland Ltd. | UFC505096 | centrifuge filters |
| NucleoSpin Extract II | Macherey-Nagel GmbH | 740 609.250 | Agarose gel extraction kit |
| Rotilabo cellulose paper type 111A | Carl Roth GmbH | AP59.1 | AFM deposition blotting paper |
| Anatop 25 (0.02 μm) | Whatman GmbH | 6809-2102 | syringe filter |
| SSpI, BspQI | New England Biolabs (NEB) | R0132, R0712 | restriction enzymes for DNA substrate preparation |
| XhoI, BglII | R0146, R0144 | restriction enzymes for DNA preparation controls | |
| nicking restriction enzyme Nt.BstNBI | New England Biolabs (NEB) | R0607 | nickase |
| T4 DNA ligase | New England Biolabs (NEB) | M0202S | Ligase |
| Tris, HEPES | Carl Roth GmbH | 4855, 9105 | buffer chemicals |
| NaCl, MgCl2, KCl, MgAcetate | Carl Roth GmbH | 3957, HN03, HN02, P026 | salt chemicals |
| NaAc | Sigma-Aldrich Chemie GmbH | 32318 | salt chemicals |
| DTT, TCEP, EDTA | 6908, HN95, 8040 | chemicals/reagents | |
| agarose, acetic acid, HCl | Carl Roth GmbH | 2267, 3738, K025 | reagents |
| ATP | Carl Roth GmbH | K054 | nucleotides |
| oligonucleotide #1 in Table 1 | Biomers | custom | complementary DNA oligonucleotide |
| oligonucleotides #2, #3, and #6 in Table 1 | Integrated DNA Technologies (IDT) | custom | fluorescein containing oligonucleotides |
| oligonucleotides #4 and #5 in Table 1 | private (available from e.g. TriLink or GlenResearch) | CPD containing oligonucleotides | |
| SafeSeal reaction tube 0.5 mL and 1.5 mL | Sarstedt | 72.704 and 72.706 | incubation tubes |
| GeneRuler 1 kb | Thermo Scientific | SM0311 | DNA ladder |
| 6x concentrate gel loading dye purple | New England Biolabs (NEB) | 51406 | DNA loading dye |
| Midori Green | Nippon Genetics Europe GmbH | 999MG28055 | DNA stain |
References
- Janicijevic, A., Ristic, D., Wyman, C. The molecular machines of DNA repair: scanning force microscopy analysis of their architecture. J. Microsc. 212 (3), 264-272 (2003).
- Wang, H., et al. DNA bending and unbending by MutS govern mismatch recognition and specificity. Proc. Natl. Acad. Sci. USA. 100 (25), 14822-14827 (2003).
- Tessmer, I., et al. Mechanism of MutS searching for DNA mismatches and signaling repair. J. Biol. Chem. 283 (52), 36646-36654 (2008).
- Wagner, K., Moolenaar, G., van Noort, J., Goosen, N. Single-molecule analysis reveals two separate DNA-binding domains in the Escherichia coli UvrA dimer. Nucleic Acids Res. 37 (6), 1962-1972 (2009).
- Buechner, C. N., et al. Strand-specific recognition of DNA damages by XPD provides insights into nucleotide excision repair substrate versatility. J Biol. Chem. 289 (6), 3613-3624 (2014).
- Van der Linden, E., Sanchez, H., Kinoshita, E., Kanaar, R., Wyman, C. RAD50 and NBS1 form a stable complex functional in DNA binding and tethering. Nucleic Acids Res. 37 (5), 1580-1588 (2009).
- Fuentes-Perez, M. E., Dillingham, M., Moreno-Herrero, F. AFM volumetric methods for the characterization of proteins and nucleic acids. Methods. 60, 113-121 (2013).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Miyagi, A., Lyubchenko, Y. L. Specificity of binding of single-stranded DNA-binding protein to its target. Biochemistry. 51, 1500-1509 (2012).
- Wirth, N., et al. Conservation and Divergence in Nucleotide Excision Repair Lesion Recognition. J. Biol. Chem. 291 (36), 18932-18946 (2016).
- Kuper, J., Kisker, C. Damage recognition in nucleotide excision DNA repair. Curr. Opin. Struct. Biol. 22, 88-93 (2012).
- Buechner, C. N., Tessmer, I. DNA substrate preparation for atomic force microscopy studies of protein-DNA interactions. J. Mol. Recognit. 26 (12), 605-617 (2013).
- Sun, Z., Tan, H. Y., Bianco, P. R., Lyubchenko, Y. L. Remodeling of RecG Helicase at the DNA Replication Fork by SSB Protein. Sci. Rep. 5, 9625 (2015).
- Billingsley, D. J., Bonass, W. A., Crampton, N., Kirkham, J., Thomson, N. H. Single-molecule studies of DNA transcription using atomic force microscopy. Phys. Biol. 9 (2), 021001 (2012).
- Maurer, S., Fritz, J., Muskhelishvili, G., Travers, A. RNA polymerase and an activator form discrete subcomplexes in a transcription initiation complex. EMBO J. 25 (16), 3784-3790 (2006).
- Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Theis, K., Chen, P. J., Skorvaga, M., Van Houten, B., Kisker, C. Crystal structure of UvrB, a DNA helicase adapted for nucleotide excision repair. EMBO J. 18 (24), 6899-6907 (1999).
- Kuper, J., et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 12 (9), e1001954 (2014).
- Shlyakhtenko, L. S., et al. Silatrane-based surface chemistry for immobilization of DNA, protein-DNA complexes and other biological materials. Ultramicroscopy. 97, 279-287 (2003).
- Roth, H. M., et al. XPB helicase regulates DNA incision by the Thermoplasma acidophilum endonuclease Bax1. DNA Repair. 11 (3), 286-293 (2012).
- Ratcliff, G. C., Erie, D. A. A Novel Single-Molecule Study to Determine Protein-Protein Association Constants. J. Am. Chem. Soc. 123 (24), 5632-5635 (2001).
- Yang, Y., Sass, L. E., Du, C., Hsieh, P., Erie, D. A. Determination of protein-DNA binding constants and specificities from statistical analyses of single molecules: MutS-DNA interactions. Nucleic Acids Res. 33 (13), 4322-4334 (2005).
- Caron, P. R., Grossman, L. Involvement of a cryptic ATPase activity of UvrB and its proteolysis product, UvrB* in DNA repair. Nucleic Acids Res. 16 (22), 10891-10902 (1988).
- Wang, H., et al. UvrB domain 4, an autoinhibitory gate for regulation of DNA binding and ATPase activity. J. Biol. Chem. 281 (22), 15227-15237 (2006).
- Chammas, O., Billingsley, D. J., Bonass, W. A., Thomson, N. H. Single-stranded DNA loops as fiducial markers for exploring DNA-protein interactions in single molecule imaging. Methods. 60 (2), 122-130 (2013).
- Truglio, J. J., et al. Structural basis for DNA recognition and processing by UvrB. Nat. Struct. Mol. Biol. 13 (4), 360-364 (2006).
- Kuper, J., Wolski, S. C., Michels, G., Kisker, C. Functional and structural studies of the nucleotide excision repair helicase XPD suggest a polarity for DNA translocation. EMBO J. 31 (2), 494-502 (2012).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Goosen, N. The presence of two UvrB subunits in the UvrAB complex ensures damage detection in both DNA strands. EMBO J. 21 (15), 4196-4205 (2002).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Hoeijmakers, J. H., Goosen, N. Architecture of nucleotide excision repair complexes: DNA is wrapped by UvrB before and after damage recognition. EMBO J. 20 (3), 601-611 (2001).
- Moolenaar, G. F., et al. The Role of ATP Binding and Hydrolysis by UvrB during Nucleotide Excision Repair. J. Biol. Chem. 275, 8044-8050 (2000).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved