È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Microscopia a forza atomica Ricerche del riconoscimento delle lesioni del DNA nella riparazione di excisione dei nucleotidi
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Here, the study of different DNA lesion recognition approaches via single molecule AFM imaging is demonstrated with the nucleotide excision repair system as an example. The procedures of DNA and protein sample preparations and experimental as well as analytical details for the AFM experiments are described.
Abstract
AFM imaging is a powerful technique for the study of protein-DNA interactions. This single molecule method allows the simultaneous resolution of different molecules and molecular assemblies in a heterogeneous sample. In the particular context of DNA interacting protein systems, different protein complex forms and their corresponding binding positions on target sites containing DNA fragments can thus be distinguished. Here, an application of AFM to the study of DNA lesion recognition in the prokaryotic and eukaryotic nucleotide excision DNA repair (NER) systems is presented. The procedures of DNA and protein sample preparations are described and experimental as well as analytical details of the experiments are provided. The data allow important conclusions on the strategies by which target site verification may be achieved by the NER proteins. Interestingly, they indicate different approaches of lesion recognition and identification for the eukaryotic NER system, depending on the type of lesion. Furthermore, distinct structural properties of the two different helicases involved in prokaryotic and eukaryotic NER result in and explain the different strategies observed for these two systems. Importantly, these experimental and analytical approaches can be applied not only to the study of DNA repair but also very similarly to other DNA interacting protein systems such as those involved in replication or transcription processes.
Introduzione
La microscopia a forza atomica (AFM) è una tecnica potente per l'analisi delle interazioni proteine-DNA 1 , 2 , 3 , 4 , 5 , 6 , 7 , 8 , 9 . Richiede solo basse quantità di materiale di campionamento per visualizzare direttamente campioni eterogenei con una risoluzione a livello di singola molecola. L'eterogeneità può derivare da diversi stati conformazionali o oligomerici di una proteina. In particolare, nel contesto di campioni di proteine-DNA, i complessi proteici possono mostrare diverse stoichiometrie e / o conformazioni indotte dal legame del DNA in generale o che si legano ad un sito target specifico all'interno del DNA. I campioni eterogenei possono anche contenere due (o più) diversi tipi di proteine e diversi complessi proteiciForme ( ad esempio , costituite da un solo tipo di proteine contro complessi eteromerici) possono interagire in modo diverso con il DNA. Gli studi qui discussi sfruttano l'imaging AFM in aria su campioni statici e secchi di proteine di riparazione del DNA legate a lunghi frammenti di DNA (~ 900 base paia, bp) che contengono una lesione che rappresenta un obiettivo di queste proteine. L'elevata risoluzione molecolare di AFM consente di distinguere tra diversi tipi di complessi proteici e di determinare le posizioni di legame delle proteine sui frammenti del DNA. Importante, le lesioni vengono introdotte nei substrati del DNA a posizioni ben definite. Poiché la posizione del sito di lesione nel DNA è nota, le distribuzioni di proteine legate al DNA forniscono una visione delle proprietà di riconoscimento delle diverse lesioni (differenti) dei complessi proteici (diversi), ad esempio , quanto bene riconoscono un particolare tipo di lesione A DNA non danneggiato) 2 , 3 , 4 , 5 , 6 . Le loro posizioni sul DNA permettono anche la distinzione tra i complessi proteici legati specificamente alle lesioni e ai complessi legati in modo non specifico altrove sul DNA. La caratterizzazione separata di questi diversi tipi complessi (complessi legati specificamente al complesso lesionario o non specifico) possono rivelare potenziali variazioni conformazionali nei complessi indotti dall'identificazione del sito di destinazione.
Le proteine di riparazione del DNA concentrate qui sono le elicasi che sono responsabili del riconoscimento delle lesioni nel percorso di riparazione delle escisioni del nucleotide (NER). Nei batteri, il NER è ottenuto dalle proteine UvrA, UvrB e UvrC. UvrA è responsabile della rilevazione iniziale delle lesioni in un complesso di scansione DNA di UvaA 2 / UvrB 2 . Alla verifica della lesione da UvrB questo complesso si converte in UvrB monomerico legato al sito della lesione e questo complesso specifico può quindi reclutare la pRocaryotic NER endonucleasi UvrC. UvrC eccisce un breve tratto (12-13 nt) di DNA a singolo filamento (ssDNA) contenente la lesione. Il tratto mancante viene poi riempito dalla DNA polimerasi. Infine, la ligasi DNA sigilla il tratto appena sintetizzato con il DNA originale 9 , 10 . Negli eucarioti, la maggior parte delle proteine della cascata NER fa parte del complesso multimerico di trascrizione II H (TFIIH). Dopo la rilevazione iniziale della lesione attraverso il complesso trimerico CEN2-XPC-HR23B, TFIIH viene reclutato nel sito di destinazione del DNA. Quando XPD all'interno del complesso verifica la presenza di una lesione target NER, le endonucleasi eerichee NER XPG e XPF vengono reclutate per accusare un breve tratto (24-32 nt) di ssDNA contenente la lesione 9 , 10 . Qui, in particolare, sono state studiate le elicasi UvrB e XPD da NER prokaryotico e eucariotico. Queste elicasi richiedono una regione non distinta nellaDNA (una bolla del DNA) per filettare su uno dei due singoli filamenti del DNA e successivamente traslocare lungo questa fili alimentata da idrolisi ATP. Oltre alle lesioni del DNA, una bolla del DNA è stata quindi introdotta nei substrati che funge da luogo di caricamento per le proteine.
La procedura per la preparazione di substrati specifici del DNA della lesione è stata descritta in precedenza 11 . Richiede un costrutto circolare del DNA (plasmide) con due siti di restrizione strettamente distanziati per un nickase. Nel contesto di questo studio è stato utilizzato il plasmide pUC19N (2729 bp) (creato dal laboratorio di S. Wilson, NIEHS). Questo plasmide contiene tre siti di restrizione strettamente distanziati per la nickel Nt.BstNBI che costituiscono un tratto di 48 nucleotidi (nt). Dopo l'incubazione con il nickase, il tratto di ssDNA tra questi siti può essere rimosso e sostituito da un oligonucleotide contenente qualsiasi caratteristica di destinazione. Dopo ogni passaggio, la digestione enzimatica completa viene testata mediante gel agarosioelettroforesi. Il DNA circolare nichelato può essere distinto a causa della sua minore mobilità elettroforetica rispetto al plasmide originale supercoiled. Lo scorrimento del DNA e la sostituzione del tratto rimosso con l'oligonucleotide specifico del substrato possono essere valutati tramite la digestione con un enzima di restrizione che incide il substrato esclusivamente all'interno della regione tra i nicchie. La linearizzazione del plasmide circolare da parte dell'enzima verrà quindi soppressa per il DNA scompattato e ripristinato dopo l'inserimento del oligonucleotide specifico. Infine, due siti di restrizione endonucleasi (idealmente taglienti singoli) consentono la generazione di un substrato lineare del DNA, con lunghezza come desiderato e con il sito target specifico in una posizione definita, nonché una bolla del DNA a distanza dalla lesione in 5 'O 3' direzione.
Il riconoscimento delle lesioni da parte delle elicasi NER può essere studiato tramite l' imaging AFM. La traslocazione del DNA degli alligati a tIl sito della lesione è visibile come un picco nella distribuzione della posizione proteica sul DNA e indica il riconoscimento della lesione. Poiché la traslocazione del DNA di queste elicasi è inoltre direzionale, con polarità 5'-3 ', la dipendenza del riconoscimento della lesione sulla posizione del sito di carico (bolla del DNA a monte oa valle della lesione) indica anche se la lesione è preferenzialmente riconosciuta Sullo strato ssDNA traslocato o al contrario, non traslocato , 5 , 9 . Nelle sezioni seguenti vengono introdotti i metodi utilizzati e saranno discussi brevemente i principali risultati di questi esperimenti. Importante, analogo al lavoro esemplare sulla riparazione del DNA mostrato qui, l'imaging AFM può essere applicato allo studio di diversi sistemi interattivi del DNA, come la replicazione del DNA o la trascrizione 8 , 12 , 13 , 14 .
Protocollo
1. Preparazione del campione
- Preparazione dei substrati del DNA 11
- Generare un gap di ssDNA nel plasmide
- Completamente digerire un campione del plasmide (qui: modificato pUC19, pUC19N) in un tubo di reazione con una appropriata nickasi (qui: Nt.BstNBI) seguita da inattivazione del calore enzimatico, utilizzando condizioni secondo il protocollo del produttore (si veda la figura 1 per uno schema presentazione). Iniziare con ~ 50 μL e ~ 500 nM plasmide per una resa sufficiente.
- Verificare il plasmide di nicking mediante elettroforesi di agarosio su campioni diluiti (~ 20 nM) 15 . Indossare guanti per la protezione contro il colorante vincolante utilizzato per la visualizzazione del DNA.
NOTA: Le differenti mobilitazioni elettroforetiche permettono di distinguere tra il DNA plasmidico circolare (rivelato) e il DNA circolare supercoiled ( Figura 1 ). - Rimuovere il tratto ssDNA inciso (tra i siti nick) dal plasmide daIncubazione con un eccesso di 10 volte l'oligonucleotide complementare (oligonucleotide 1 nella tabella 1 ) agitando a 300 giri / min per 30 minuti vicino alla temperatura di fusione dell'oligonucleotide (qui: 68 ° C per pUC19N) in un blocco termico ( Figura 1 ).
- Separare il plasmide gapped dai frammenti di DNA più piccoli usando un filtro da taglio di peso molecolare di 50 kDa (MWCO) mediante centrifugazione per 10 min a 10.000 xg in una centrifuga da tavola ( Figura 1 ). Per estrarre il DNA concentrato dal filtro, invertire il filtro e inserire in un nuovo tubo di reazione da 1,5 ml. Centrifugare per 3 min a 1.000 x g.
- Riempire il campione di DNA concentrato risultante a 500 μL con acqua deionizzata e filtrata, aggiungere un ulteriore eccesso di 5 volte l'oligonucleotide complementare e ripetere le fasi 1.1.1.3 e 1.1.1.4 almeno 3 volte.
- Test per la completa eliminazione del DNA mediante incubazione con un idoneo enzima di restrizione (qui: XhoI o BglII) utilizzando conditiIn base alla descrizione del costruttore. Utilizzare campioni di DNA diluiti (nicked versus gapped) (~ 20 nM). Eseguire un'elettroforesi con gel agarosio per distinguere tra il plasmide linearizzato (che non contiene ssDNA gap) e il DNA non inciso (DNA gapped) utilizzando il DNA nicked come controllo positivo (include una scala del DNA per riferimento, figura 1 ). Indossare i guanti.
- Riempire il divario con gli oligonucleotidi ssDNA modificati
- Incollare il plasmide gapped mediante incubazione con un eccesso di 25 volte di un oligonucleotide fosforilato 5 'che contiene i siti specifici di scelta, incubandosi a ~ 45 ° C per 4 ore ( figura 1 ). Qui usiamo 48 ssDNA nt contenenti una lesione, o una dimina addizionata di fluoresceina o un dimero pirimidina ciclobutano (CPD), e in aggiunta una breve sequenza non complimentale (8 nt) per produrre le bolle del DNA (cfr. Tabella 1 ).
- Collegare in modo covalente l'inserto ricotto con il plasmide per incubatoCon la ligasi del DNA di T4 per notte a temperatura ambiente secondo il protocollo del produttore ( Figura 1 ). Per questa reazione, aggiungere ATP contenente una soluzione concentrata di buffer tampone per produrre opportune condizioni di tampone per la ligasi ( ad esempio , utilizzare buffer di reazione UvrB: 50 mM Tris-HCl pH 7,5, 10 mM MgCl2, 50 mM KCl, 5 mM DTT, 1 mM ATP).
- Prova per l'inserimento dell'ogigonucleotide specifico nel plasmide gapped mediante digestione di campioni diluiti con un idoneo enzima di restrizione (come al punto 1.1.1.6) secondo il protocollo del produttore seguita da un'elettroforesi del gel agarosio (come al punto 1.1.1.6, figura 1 ).
- Preparazione del substrato lineare del DNA
- Digestare il DNA plasmidico modificato con gli enzimi di restrizione (idealmente con un unico sito di restrizione nel plasmide) utilizzando le condizioni come raccomandato dal produttore ( Figura 1 ). Questa fase produce frammenti lineari che contengonoLa modifica inserita in una posizione definita. Qui usa il taglio SspI e BspQI alle posizioni 613 e 255 bp rispettivamente a monte ea valle dell'inserto, ottenendo un frammento di DNA di 916 bp contenente il sito specifico di lesione a ~ 30% della lunghezza del DNA.
NOTA: Per gli esperimenti di imaging AFM come descritto qui, i frammenti di DNA con lunghezze tra ~ 200 bp e ~ 2000 bp sono substrati adatti. - Isolare il frammento di bersaglio mediante elettroforesi del gel agarosico e l'estrazione del gel utilizzando un kit commerciale. Indossare guanti protettivi.
- Facoltativamente, tagliare il gel agarosio con uno scalpello per separare la scala del DNA e corsie del campione diluito (prime due corsie) dalle corsie contenenti il DNA concentrato da purificare. Soltanto esporre la parte del gel con le prime due corsie all'irraggiamento UV, posizionando solo questa parte del gel su un tavolo UV.
- Tagliare la banda corrispondente al frammento di scelta con un bisturi. Riagganciare le due parti del gel (fuori dalla linguetta UVle). Utilizzare la posizione della fascia eccitata dal campione diluito come orientamento per la posizione della banda del substrato DNA desiderato. Considera la concentrazione più elevata tagliando fette leggermente più ampie rispetto al controllo diluito.
NOTA: Poiché qui è stato studiato il riconoscimento delle lesioni UV, l'introduzione di ulteriori lesioni UV mediante irraggiamento UV è stata accuratamente evitata nel substrato del DNA da questo approccio.
- Tagliare la banda corrispondente al frammento di scelta con un bisturi. Riagganciare le due parti del gel (fuori dalla linguetta UVle). Utilizzare la posizione della fascia eccitata dal campione diluito come orientamento per la posizione della banda del substrato DNA desiderato. Considera la concentrazione più elevata tagliando fette leggermente più ampie rispetto al controllo diluito.
- Calcolare la concentrazione del DNA c dall'assorbimento a 260 nm misurata con uno spettrofotometro UV-Vis utilizzando la legge di Lambert-Beer con un coefficiente di estinzione molare media di ε 260nm ~ 6.700 M - 1 cm - 1 per bp:
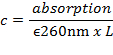
Dove L è la lunghezza del percorso (lunghezza della camera di misura, tipicamente 1 cm).
- Digestare il DNA plasmidico modificato con gli enzimi di restrizione (idealmente con un unico sito di restrizione nel plasmide) utilizzando le condizioni come raccomandato dal produttore ( Figura 1 ). Questa fase produce frammenti lineari che contengonoLa modifica inserita in una posizione definita. Qui usa il taglio SspI e BspQI alle posizioni 613 e 255 bp rispettivamente a monte ea valle dell'inserto, ottenendo un frammento di DNA di 916 bp contenente il sito specifico di lesione a ~ 30% della lunghezza del DNA.
- Generare un gap di ssDNA nel plasmide
- Espressione e purificazione delle proteine
- Esprimono in modo recombinante UvrB in E. coli e purificano la proteina mediante l' affinità standard di chitin perline 15 e la cromatografia di esclusione di dimensioni 15 come descritto in precedenza 16 .
NOTA: Il gene uvrB da Bacillus caldotenax era stato clonato nel vettore pTYB1. - Esprimere XPD in E. coli e purificare la proteina mediante l' affinità standard 15 del nickel IDA e la cromatografia di esclusione di dimensioni 15 seguita da una cromatografia di scambio anionico 15 , come descritto in precedenza 17 .
NOTA: Il gene per Chaetomium thermophilum Xeroderma pigmentosum proteina del gruppo D (XPD) era stato clonato nel vettore pBADM11. C. thermophilum p44 era un regalo gentile del laboratorio di Caroline Kisker, espresso e puricoCome descritto 17 .
- Esprimono in modo recombinante UvrB in E. coli e purificano la proteina mediante l' affinità standard di chitin perline 15 e la cromatografia di esclusione di dimensioni 15 come descritto in precedenza 16 .
2. Esperimento AFM
- preparazione del campione
- Facoltativamente, pre-incubare il substrato del DNA a 65 ° C per 10 minuti in un blocco termico per rimuovere i potenziali cristalli di sale microscopico che potrebbero essere formate durante lo stoccaggio nel frigorifero.
- Preparare tampone di reazione a una concentrazione di 10 volte (tamponi 10x).
NOTA: il tampone di reazione XPD a concentrazione 1x conteneva 20 mM Tris-HCl pH 7,5, 10 mM KCl, 5 mM MgCl2, 1 mM TCEP, 2 mM ATP; 1x buffer di reazione UvrB conteneva 50 mM Tris-HCl pH 7,5, 50 mM KCl, 10 mM MgCl2, 5 mM DTT e 1 mM ATP. - Pre-incubare le proteine ad una concentrazione più elevata in condizioni di incubazione appropriate per aumentare la formazione del complesso. Pre-diluire un piccolo volume ( ad esempio , 1 μL) delle singole proteine in tampone di reazione di proteina 1x alla concentrazione desiderata e mescolare piccoli volumi ( ad esempio , 1 μL) di tHa soluzioni proteiche individuali in un tubo di reazione di 0,5 mL.
- Mettere il tubo in un blocco termico per le temperature di incubazione superiori alla temperatura ambiente. Scegliere un rapporto di concentrazione in funzione della stechiometria complessa prevista, sia di equimolarità che di corrispondenti concentrazioni. Qui incubare 1 μl di XPD (20 μM in 1 x tampone di reazione XPD) e 1 μl p44 (20 μM in 1x tampone di reazione XPD) a 10 μM ciascuno per 10 minuti a 37 ° C.
- Incubare i campioni ad adeguate concentrazioni di proteine e DNA in tampone di reazione proteica in un tubo di reazione da 0,5 mL. Qui usi 500 nM UvrB o 1 μM XPD + 1 μM p44 e 100 nM DNA. Pipettare piccoli volumi per salvare materiale, ad esempio 0,25-0,5 μL di proteine (pre-diluito a una concentrazione di 10 volte di incubazione) e DNA in un volume totale di 2,5-5 μL di tampone di reazione di proteine 1x. Qui, incubare per 30 minuti a 37 ° C in un blocco termico. Spin giù in un tubo di reazione brevemente (~ 1 s) in un centrifugo da tavoloE per garantire la miscelazione di piccoli volumi.
- Deposizione di campione
- Preparare un substrato mica: tagliare un pezzo di mica di circa 1 x 1 cm 2 da strisce più grandi usando un bisturi. Strizza gli strati superiori del pezzo minerale mica multistrato usando nastro adesivo per rivelare una superficie di substrato liscia, piatta e atomica.
NOTA: il pezzo di mica può essere riutilizzato per più esperimenti rimuovendo ulteriormente gli strati. - Preparare il tampone di deposizione AFM con acqua deionizzata e filtrata, ad esempio 25 mM HEPES pH 7,5, 25 mM Na-acetato, 10 mM Mg-acetato. Filtro attraverso un filtro da siringa da 0,02 μm.
NOTA: I cationi divalenti nel tampone di deposizione servono a chelare le molecole del DNA caricate negativamente alla superficie di mica, che è anche caricata negativamente al pH neutro. Se la concentrazione di ioni di Mg 2+ relativamente elevata nel buffer di deposizione costituisce un problema per un determinato sistema proteico-DNA, in alternativa,La superficie della mica può essere pre-caricata con amino gruppi usando la chimica a base di silatrane per fornire cariche posteriori positive per l'ancoraggio 18 . La deposizione di campione può quindi essere effettuata in un tampone che non contiene solo o basse quantità di cationi bivalenti. - Diluire il campione (vedere 2.1) per la deposizione immediata sulla mica nel tampone di deposizione. Depositare un piccolo volume (qui: 20 μL).
NOTA: i fattori di diluizione dipendono dalla concentrazione del campione. Qui, diluire i campioni 50-100x. In linea di principio, il DNA di 1 nM ha una buona copertura superficiale per ~ 1.000 bp. - Risciacquare immediatamente il campione da tre a quattro volte con qualche millilitro di acqua filtrata, deionizzata, spazzare via l'eccesso di liquido e soffiare in un leggero flusso di azoto. L'intero processo dalla deposizione di campioni ai campioni essiccati può essere effettuato entro meno di 30 s.
- Fissare il pezzo di mica su uno scivolo a microscopio con nastro adesivo ai suoi bordi.
NOTA: diversi sistemi AFM hanno dRequisiti diversi per fissare il campione sul palco. Altri AFM dispongono di stadi magnetici e le parti di mica sono fissate su dischi magnetici, ad esempio con colla termica. I dettagli del sistema AFM utilizzato qui si trovano nella tabella dei materiali.
- Preparare un substrato mica: tagliare un pezzo di mica di circa 1 x 1 cm 2 da strisce più grandi usando un bisturi. Strizza gli strati superiori del pezzo minerale mica multistrato usando nastro adesivo per rivelare una superficie di substrato liscia, piatta e atomica.
- AFM imaging
- Posizionare il campione (vedi 2.2.5) centralmente sullo stadio AFM e fissare lo scivolo a microscopio sul palco con i pattini magnetici.
- Inserire la punta AFM nel supporto della punta. Usare i cantilever con una sonda AFM tagliente (<10 nm) adatta per l'immagine oscillante e intermittente della modalità di contatto in aria. Utilizzare le sonde AFM, ad esempio, come indicato nella tabella dei materiali . Qui (dipende da AFM), fissare la punta nel supporto sotto un morsetto stringendo la vite di serraggio (dito stretto). Inserire il supporto della punta nella testina di misura AFM. Riposiziona la testa sulla schiena per questo passo.
- Posizionare la testata di misurazione AFM sulla parte superiore del campione. I dettagli dipendono dal modello AFM. Qui, mAssicuratevi che la testa sia stabile con le gambe all'interno delle fessure del palco. Assicurarsi che la mica si trovi sul palco in cui la punta AFM si sposterà direttamente sopra di essa. Le viti a micrometro a destra dello stadio consentono un posizionamento fine del campione.
- Allineare il laser AFM sul retro del cantilever per una resistenza ottimale del segnale dal fotorivelatore sensibile alla posizione su cui si riflette il laser. I dettagli dipendono dal modello AFM.
- Qui ruotare le ruote sul lato destro e posteriore della testina di misura AFM per regolare le posizioni x e y del laser AFM per dirigere centralmente sull'estremità della sommità. Guarda il segnale di riflessione nella finestra video AFM (se disponibile, qui: premere l'icona della fotocamera e scegliere l'ingresso: Svideo).
- Una volta posizionata in modo crudo, ottimizzare il segnale di somma del rilevatore (Sum in Sum e Deflection Meter window nel software AFM) regolando bene la posizione del laser con le due ruote (rimanere alla fine del cantilever, il segnale sumL dipende dal tipo AFM e dal tipo di cantilever, qui: obiettivo per somma> 5).
- Segnare il segnale di differenza dai diodi superiori e inferiori dell'array del rilevatore (qui: segnale di deflessione nella finestra Sum e Deflection Meter), orientando la riflessione laser AFM sul centro rivelatore (qui: ruotare la ruota a sinistra della misura AFM capo).
NOTA: Le deviazioni da zero indicano quindi la deflessione del cantilever a causa di interazioni di superficie, che sono tradotte in informazioni di altezza da parte dell'AFM. - Determinare la frequenza della risonanza a cantilena con una frequenza di sintonia implementata nel software AFM (qui: comando automatico in Master Panel / Tune). Scegliere un'ampiezza corrispondente ad un ingresso da 1 V per il piezo che guida l'oscillazione a cantilever. Impostare la frequenza di oscillazione leggermente (5%) inferiore alla frequenza di risonanza e zero la fase dell'oscillazione.
- Avvicinare la punta alla superficie del campione usando il grezzo impegno modE (qui: comando Engage nella finestra del pannello principale) fino a raggiungere l'impostazione di protezione (set-point). Utilizzare il taglio del ~ 2% dell'ampiezza di oscillazione a livello libero come set-point (qui, immettere Set Point 980 mV nel pannello Master).
- Fine impegnare la punta AFM con la superficie del campione abbassando il setpoint utilizzando il software AFM. Obiettivo per l'imaging in modalità repulsiva con la fase di oscillazione a cantilever (fase in somma e finestra del deflettore) appena sotto la fase a livello libero (prima di impegnarsi, qui tipicamente ~ 70). Qui, utilizzare punti tipici tipici finali sull'ordine del 70-80% dell'ampiezza del livello libero (1 V).
- Prima della scansione, selezionare i segnali da registrare nel pannello canale master. Scegli l'altezza (Ht) e l'ampiezza (Am).
- Inizia la scansione del campione (qui: comando Do Scan nella finestra del pannello principale). Utilizzare una velocità di scansione di 2,5 μm / s (velocità di scansione del comando in Master Panel) in aree di immagine di 4 μm x 4 μm o 8 μm x 8 μm (inserire la dimensione di scansione nel Master PaneL) con risoluzioni di pixel di 2.048 o 4.096 (punti di scansione e linee di scansione nel pannello master) rispettivamente.
- Salvare il file di immagine (comando Salva immagine nel pannello master) senza modifiche apportate (scegliere Nessuno nel pannello canale master / Salva piani adatta).
- Per ulteriori analisi, elaborare l'immagine salvata. Caricare l'immagine (comando Sfoglia dati salvati nel menu Analisi AFM). Aprire il pannello Modifica (premere M nella parte superiore dell'immagine). Applicare un piano di dimensioni x e y all'immagine in altezza (estensione HtR) (comando XY nella finestra di Planefit nel pannello Modifica, scegliere Ordine 3). Quindi appiattire l'immagine (comando Flatten in finestra Flatten in Pannello Modifica) scegliendo Ordine Ordine 3.
- Esportare l'immagine come file TIFF (premere Comandi nella sezione superiore dell'immagine, scegliere Esportazione TIFF 2x corrispondente a 2.048 pixel di risoluzione) per ulteriori analisi.
3. Analisi AFM
- Volume del complesso proteico
- Pre-Selezionare complessi proteina-DNA rilevanti attraverso l'ispezione visiva diretta delle immagini nel software AFM, utilizzando un adeguato schema di colori per ottimizzare le diverse apparenze di complessi di dimensioni diverse (vedere colori e dimensioni differenti di diversi complessi in figura 2 ; qui: schema di colori SeaLandAndFire in L'AFMsoftware). Per i campioni XPD, i possibili complessi comprendevano XPD / p44-DNA e XPD-DNA e p44-DNA, con dimensioni decisamente diverse a causa delle masse molecolari relativamente grandi e diverse di XPD (~ 95 kDa) e p44 (~ 40 kDa) .
- Misurare i volumi di picchi di proteine individuali sul DNA per verificare il tipo complesso. Ciò può essere ottenuto con diversi software di immagine (qui: lo strumento di sezione del software AFM).
- Misurare l'altezza (h) e il diametro (d) delle sezioni di picco di proteine con i cursori della finestra di sezione. Misurare il diametro vicino alla base della sezione delle particelle.
- Determinare il volume (V) del piselloKs utilizzando semplici modelli matematici, ad esempio utilizzando la seguente formula, basata su un modello a sfera sferica:
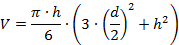
- Trasformare i volumi misurati in una massa molecolare approssimativa della proteina.
- Inizialmente calibrare il sistema AFM per la conversione di volume a peso molecolare (MW) utilizzando una gamma di proteine con peso molecolare noto (qui: sono stati condotti complessivamente 12 esperimenti con 25 e 851 punti dati su 5 diverse proteine in monomerica, dimerica , Stati trimerici o tetramerici) 4 , 19 , 20 . Le proteine di deposito e di immagine (come descritto sopra, in 2.2 e 2.3) e misurare i loro volumi come sopra descritto (3.1.2). Tracciare i volumi sopra i loro pesi molecolari noti. Il grafico risultante mostrerà una relazione lineare tra V e MW, l'equazione per cui può essere ottenuta daUna linea adatta ai dati. Per il sistema AFM utilizzato qui, è stata ottenuta la seguente relazione 19 :
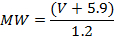
NOTA: questo passaggio non deve essere ripetuto prima di ogni misurazione, ma viene eseguito solo una volta. La calibrazione da volume a MW può essere applicata alle immagini ottenute in condizioni di imaging simile utilizzando sonde AFM con diametri leggermente variabili. - Sulla base dei loro volumi misurati (3.1.2) e della calibrazione V-to-MW (3.1.3.1), determinare il MW approssimativo dei complessi proteici 20 , che fornisce informazioni sul suo contenuto molecolare. Ad esempio , per i campioni XPD / p44 sono stati ottenuti ~ 50 kDa, ~ 100 kDa e ~ 140 kDa, coerenti con i complessi p44-DNA (o picchi causati da semplice sovrastruttura del DNA), picchi di XPD e picchi XPD / p44 .
- Inizialmente calibrare il sistema AFM per la conversione di volume a peso molecolare (MW) utilizzando una gamma di proteine con peso molecolare noto (qui: sono stati condotti complessivamente 12 esperimenti con 25 e 851 punti dati su 5 diverse proteine in monomerica, dimerica , Stati trimerici o tetramerici) 4 , 19 , 20 . Le proteine di deposito e di immagine (come descritto sopra, in 2.2 e 2.3) e misurare i loro volumi come sopra descritto (3.1.2). Tracciare i volumi sopra i loro pesi molecolari noti. Il grafico risultante mostrerà una relazione lineare tra V e MW, l'equazione per cui può essere ottenuta daUna linea adatta ai dati. Per il sistema AFM utilizzato qui, è stata ottenuta la seguente relazione 19 :
- Posizioni complesse di proteine sul DNA
- Determinare il DNALunghezze di frammento.
- Traccia i frammenti di DNA nelle immagini AFM, ad esempio con la funzione di linea libera di un idoneo software di analisi delle immagini (vedere ad esempio Tabella dei materiali) e misurare la lunghezza della linea.
NOTA: escludere gli aggregati del DNA così come i frammenti tagliati dai margini dell'immagine. - Staccare e tracciare le lunghezze del DNA dall'intero esperimento in un istogramma, utilizzando un'adeguata analisi dei dati e software di grafica ( ad esempio , vedere tabella dei materiali ).
- Adattare le distribuzioni di lunghezza con una curva Gaussian nell'analisi dei dati e software di grafica per determinare la lunghezza dei frammenti di DNA. Per conoscere la posizione del sito di DNA inserito (vedere 1.1), includere solo i frammenti di DNA con la giusta lunghezza entro due deviazioni standard dal centro della curva Gauss (y = y 0 + z exp (-2 (xx c ) 2 / w 2 ), dove z è un fattore normale e x c e w sono il centro eMassima piena larghezza massima della Gaussiana) in ulteriori analisi.
NOTA: La lunghezza del DNA al centro della curva di Gauss deve essere vicina alla lunghezza teorica del frammento di DNA (calcolato con 0,34 nm / bp). Le lunghezze fino al 10% più corte del valore teorico sono tipiche e sono probabilmente causate da limiti di risoluzione AFM.
- Traccia i frammenti di DNA nelle immagini AFM, ad esempio con la funzione di linea libera di un idoneo software di analisi delle immagini (vedere ad esempio Tabella dei materiali) e misurare la lunghezza della linea.
- Determinare le posizioni di picchi di proteine sui substrati del DNA.
- Misurare la distanza dei picchi proteici dal finale più stretto del DNA come descritto al punto 3.2.1.1 e dividere per la lunghezza totale del DNA per ottenere distanze in unità di frazione della lunghezza del DNA.
- Bin e tracciare le distanze misurate in un istogramma utilizzando un'adeguata analisi dei dati e software di grafica (si veda ad esempio , figura 3 ). Come regola generale, scegliere una dimensione del contenitore che fornisce circa √n i contenitori per n punti dati.
NOTA: poiché non si distinguono qui i brani del DNA, la trama solo alla frazione della lunghezza del DNA 0,5 (centro del DNA fragmentment). Poiché il legame del DNA non è il focus dello studio, escludere le estremità del DNA dalle analisi iniziando a rimuovere i dati di posizione leggermente dopo la posizione 0 ( ad esempio , qui: il frammento di una frazione di lunghezza del DNA di 0,02).
- Determinare la specificità del sito di destinazione dalle distribuzioni di posizione complesse di proteine.
- Adattare il massimo (incrementato occorrenza di legame) nella distribuzione di posizione con una curva di Gauss come al punto 3.2.1.3, ma in piedi sull'altezza del legame di sfondo (v. Figura 3 ).
- Calcolare la specificità S per il sito specifico rispetto allo sfondo del DNA non specifico (molecole proteiche legate a siti di DNA non specifici) con la seguente formula 21
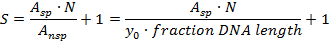
A sp : Numero di complessi specifici (area sotto la curva gaussiana)
A nsp : numero di complessi non specifici (area dello sfondo, Cioè (frazione DNA lunghezza) x (altezza media di fondo y 0 ); La frazione della lunghezza del DNA qui riportata è di 0,48 per un istogramma a partire da 0,02 di lunghezza del DNA)
N: numero di possibili siti di legame del DNA (qui: N = 914 escluse le estremità del DNA)
- Determinare il DNALunghezze di frammento.
- Angoli di curvatura del DNA
- Utilizzando l'utensile ad angolo in un idoneo software di analisi delle immagini, misurare l'angolo β tra due linee posizionate centralmente lungo la spina dorsale del DNA e centrando al picco della proteina (vedere ad esempio , inserto in figura 4 ). Misurare gli angoli per un numero statisticamente rilevante di complessi protein-DNA (> 50, idealmente> 100) 2 , 3 , 5 .
NOTA: L'angolo di curvatura del DNA è definito come 180 ° -β 2 , 3 , 5 . - Utilizzando l'analisi dei dati e il software di grafica(Vedi Tabella dei Materiali) producono un istogramma di distribuzione angolare della curvatura, facendo in modo che gli angoli di curvatura del DNA venissero creati.
- Adattare la distribuzione dell'angolo di curvatura con una curva di Gauss. Se nella distribuzione è visibile più di un massimo, scegliere più punti di picco di Gauss. Il centro (s) della curva Gaussian (s) dà (s) lo stato di angolo di curvatura media della specie particolare.
- Se uno spostamento nell'angolo (i) di curvatura (maxima di adattamento Gaussiano) è evidente per le distribuzioni, ad esempio, di diverse varianti di proteine o di diverse specie di proteine complesse, applicare un test t studente per valutare il livello di significatività del i cambiamenti.
- Utilizzando l'utensile ad angolo in un idoneo software di analisi delle immagini, misurare l'angolo β tra due linee posizionate centralmente lungo la spina dorsale del DNA e centrando al picco della proteina (vedere ad esempio , inserto in figura 4 ). Misurare gli angoli per un numero statisticamente rilevante di complessi protein-DNA (> 50, idealmente> 100) 2 , 3 , 5 .
Risultati
Distinguere tra diversi tipi complessi basati su volumi complessi di proteine
L'attività elicolare dell'elicasica NER prokaryotica UvrB è stimolata dal legame del DNA 22 , 23 . UvrB richiede una regione non distinta nel DNA (una bolla del DNA) per caricare correttamente su uno dei due singoli fili di ssDNA. In vivo , questa struttura del DNA è fornita ...
Discussione
Analisi statistiche AFM di posizioni di legame di proteine su lunghi frammenti di DNA che contengono siti target specifici possono rivelare dettagli interessanti sulle strategie particolari impiegate dalla proteina per riconoscere questi siti 2 , 3 , 4 , 5 , 6 . Per interpretare le distribuzioni di posizione risultanti, le posizioni degli obiettivi nel ...
Divulgazioni
Gli autori non hanno niente da rivelare.
Riconoscimenti
PUC19N, oligonucleotidi contenenti CPD e p44 sono stati gentilmente forniti da Samuel Wilson, Korbinian Heil e Thomas Carell, e rispettivamente da Gudrun Michels e Caroline Kisker. Questo lavoro è stato sostenuto da sovvenzioni della Deutsche Forschungsgemeinschaft (DFG) FZ82 e TE-671/4 a IT.
Materiali
| Name | Company | Catalog Number | Comments |
| Molecular Force Probe (MFP) 3D | Asylum Research | N/A | atomic force microscope (AFM) |
| Precision 390 | DELL | N/A | computer |
| ThermoMixer and 1.5 mL block | Eppendorf | 5382000015 | heat block for DNA preparation |
| Rotilabo Block-Heater H 250 & blocks for 0.5 mL tubes | Carl Roth GmbH | Y264.1 & Y267.1 | heat block for protein-DNA incubations |
| Mini-Sub Cell GT | Bio-Rad Laboratories GmbH | 1704467 | electrophoresis chamber with gel caster and power supply |
| Power Pac Basic | Bio-Rad Laboratories GmbH | 1645050 | electrophoresis power supply |
| Centifuge 5415 D with rotor | Eppendorf | 2262120-3 | table centrifuge |
| Ultra-Lum electronic UV transillumonator MEB-15 | Ultralum | 900-1322-02 | UV irradiation table |
| NanoDrop ND-1000 | VWR International / PEQLAB Biotechnologie GmbH | N/A | UV spectrophotometer |
| TKAX-CAD with 0.2 μm capsule filter | Unity Lab Services | N/A | water deionization and filter unit |
| Name | Company | Catalog number | Comments |
| Software | |||
| MFP software on Igor Pro | Asylum Research | N/A | AFM software |
| ImageJ (open source Java image processing) | NIH Image | N/A | Image analysis software |
| Excel (Microsoft Office) | Microsoft Corporation | N/A | data analysis software |
| Origin9 / Origin2016 | OriginLab Corporation | N/A | statistical data analysis and graphing software |
| Name | Company | Catalog number | Comments |
| Material | |||
| OMCL-AC240TS | Olympus | OMCL-AC240TS | AFM cantilevers |
| grade V-5 muscovite | SPI Supplies | 1805 | mica sheets |
| Amicon Ultra 0.5 mL 50k Ultracell | Millipore Ireland Ltd. | UFC505096 | centrifuge filters |
| NucleoSpin Extract II | Macherey-Nagel GmbH | 740 609.250 | Agarose gel extraction kit |
| Rotilabo cellulose paper type 111A | Carl Roth GmbH | AP59.1 | AFM deposition blotting paper |
| Anatop 25 (0.02 μm) | Whatman GmbH | 6809-2102 | syringe filter |
| SSpI, BspQI | New England Biolabs (NEB) | R0132, R0712 | restriction enzymes for DNA substrate preparation |
| XhoI, BglII | R0146, R0144 | restriction enzymes for DNA preparation controls | |
| nicking restriction enzyme Nt.BstNBI | New England Biolabs (NEB) | R0607 | nickase |
| T4 DNA ligase | New England Biolabs (NEB) | M0202S | Ligase |
| Tris, HEPES | Carl Roth GmbH | 4855, 9105 | buffer chemicals |
| NaCl, MgCl2, KCl, MgAcetate | Carl Roth GmbH | 3957, HN03, HN02, P026 | salt chemicals |
| NaAc | Sigma-Aldrich Chemie GmbH | 32318 | salt chemicals |
| DTT, TCEP, EDTA | 6908, HN95, 8040 | chemicals/reagents | |
| agarose, acetic acid, HCl | Carl Roth GmbH | 2267, 3738, K025 | reagents |
| ATP | Carl Roth GmbH | K054 | nucleotides |
| oligonucleotide #1 in Table 1 | Biomers | custom | complementary DNA oligonucleotide |
| oligonucleotides #2, #3, and #6 in Table 1 | Integrated DNA Technologies (IDT) | custom | fluorescein containing oligonucleotides |
| oligonucleotides #4 and #5 in Table 1 | private (available from e.g. TriLink or GlenResearch) | CPD containing oligonucleotides | |
| SafeSeal reaction tube 0.5 mL and 1.5 mL | Sarstedt | 72.704 and 72.706 | incubation tubes |
| GeneRuler 1 kb | Thermo Scientific | SM0311 | DNA ladder |
| 6x concentrate gel loading dye purple | New England Biolabs (NEB) | 51406 | DNA loading dye |
| Midori Green | Nippon Genetics Europe GmbH | 999MG28055 | DNA stain |
Riferimenti
- Janicijevic, A., Ristic, D., Wyman, C. The molecular machines of DNA repair: scanning force microscopy analysis of their architecture. J. Microsc. 212 (3), 264-272 (2003).
- Wang, H., et al. DNA bending and unbending by MutS govern mismatch recognition and specificity. Proc. Natl. Acad. Sci. USA. 100 (25), 14822-14827 (2003).
- Tessmer, I., et al. Mechanism of MutS searching for DNA mismatches and signaling repair. J. Biol. Chem. 283 (52), 36646-36654 (2008).
- Wagner, K., Moolenaar, G., van Noort, J., Goosen, N. Single-molecule analysis reveals two separate DNA-binding domains in the Escherichia coli UvrA dimer. Nucleic Acids Res. 37 (6), 1962-1972 (2009).
- Buechner, C. N., et al. Strand-specific recognition of DNA damages by XPD provides insights into nucleotide excision repair substrate versatility. J Biol. Chem. 289 (6), 3613-3624 (2014).
- Van der Linden, E., Sanchez, H., Kinoshita, E., Kanaar, R., Wyman, C. RAD50 and NBS1 form a stable complex functional in DNA binding and tethering. Nucleic Acids Res. 37 (5), 1580-1588 (2009).
- Fuentes-Perez, M. E., Dillingham, M., Moreno-Herrero, F. AFM volumetric methods for the characterization of proteins and nucleic acids. Methods. 60, 113-121 (2013).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Miyagi, A., Lyubchenko, Y. L. Specificity of binding of single-stranded DNA-binding protein to its target. Biochemistry. 51, 1500-1509 (2012).
- Wirth, N., et al. Conservation and Divergence in Nucleotide Excision Repair Lesion Recognition. J. Biol. Chem. 291 (36), 18932-18946 (2016).
- Kuper, J., Kisker, C. Damage recognition in nucleotide excision DNA repair. Curr. Opin. Struct. Biol. 22, 88-93 (2012).
- Buechner, C. N., Tessmer, I. DNA substrate preparation for atomic force microscopy studies of protein-DNA interactions. J. Mol. Recognit. 26 (12), 605-617 (2013).
- Sun, Z., Tan, H. Y., Bianco, P. R., Lyubchenko, Y. L. Remodeling of RecG Helicase at the DNA Replication Fork by SSB Protein. Sci. Rep. 5, 9625 (2015).
- Billingsley, D. J., Bonass, W. A., Crampton, N., Kirkham, J., Thomson, N. H. Single-molecule studies of DNA transcription using atomic force microscopy. Phys. Biol. 9 (2), 021001 (2012).
- Maurer, S., Fritz, J., Muskhelishvili, G., Travers, A. RNA polymerase and an activator form discrete subcomplexes in a transcription initiation complex. EMBO J. 25 (16), 3784-3790 (2006).
- Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Theis, K., Chen, P. J., Skorvaga, M., Van Houten, B., Kisker, C. Crystal structure of UvrB, a DNA helicase adapted for nucleotide excision repair. EMBO J. 18 (24), 6899-6907 (1999).
- Kuper, J., et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 12 (9), e1001954 (2014).
- Shlyakhtenko, L. S., et al. Silatrane-based surface chemistry for immobilization of DNA, protein-DNA complexes and other biological materials. Ultramicroscopy. 97, 279-287 (2003).
- Roth, H. M., et al. XPB helicase regulates DNA incision by the Thermoplasma acidophilum endonuclease Bax1. DNA Repair. 11 (3), 286-293 (2012).
- Ratcliff, G. C., Erie, D. A. A Novel Single-Molecule Study to Determine Protein-Protein Association Constants. J. Am. Chem. Soc. 123 (24), 5632-5635 (2001).
- Yang, Y., Sass, L. E., Du, C., Hsieh, P., Erie, D. A. Determination of protein-DNA binding constants and specificities from statistical analyses of single molecules: MutS-DNA interactions. Nucleic Acids Res. 33 (13), 4322-4334 (2005).
- Caron, P. R., Grossman, L. Involvement of a cryptic ATPase activity of UvrB and its proteolysis product, UvrB* in DNA repair. Nucleic Acids Res. 16 (22), 10891-10902 (1988).
- Wang, H., et al. UvrB domain 4, an autoinhibitory gate for regulation of DNA binding and ATPase activity. J. Biol. Chem. 281 (22), 15227-15237 (2006).
- Chammas, O., Billingsley, D. J., Bonass, W. A., Thomson, N. H. Single-stranded DNA loops as fiducial markers for exploring DNA-protein interactions in single molecule imaging. Methods. 60 (2), 122-130 (2013).
- Truglio, J. J., et al. Structural basis for DNA recognition and processing by UvrB. Nat. Struct. Mol. Biol. 13 (4), 360-364 (2006).
- Kuper, J., Wolski, S. C., Michels, G., Kisker, C. Functional and structural studies of the nucleotide excision repair helicase XPD suggest a polarity for DNA translocation. EMBO J. 31 (2), 494-502 (2012).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Goosen, N. The presence of two UvrB subunits in the UvrAB complex ensures damage detection in both DNA strands. EMBO J. 21 (15), 4196-4205 (2002).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Hoeijmakers, J. H., Goosen, N. Architecture of nucleotide excision repair complexes: DNA is wrapped by UvrB before and after damage recognition. EMBO J. 20 (3), 601-611 (2001).
- Moolenaar, G. F., et al. The Role of ATP Binding and Hydrolysis by UvrB during Nucleotide Excision Repair. J. Biol. Chem. 275, 8044-8050 (2000).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon