É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Investigações por microscopia de força atômica para o reconhecimento de lesões de DNA no reparo por excisão de nucleotídeos
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Here, the study of different DNA lesion recognition approaches via single molecule AFM imaging is demonstrated with the nucleotide excision repair system as an example. The procedures of DNA and protein sample preparations and experimental as well as analytical details for the AFM experiments are described.
Resumo
AFM imaging is a powerful technique for the study of protein-DNA interactions. This single molecule method allows the simultaneous resolution of different molecules and molecular assemblies in a heterogeneous sample. In the particular context of DNA interacting protein systems, different protein complex forms and their corresponding binding positions on target sites containing DNA fragments can thus be distinguished. Here, an application of AFM to the study of DNA lesion recognition in the prokaryotic and eukaryotic nucleotide excision DNA repair (NER) systems is presented. The procedures of DNA and protein sample preparations are described and experimental as well as analytical details of the experiments are provided. The data allow important conclusions on the strategies by which target site verification may be achieved by the NER proteins. Interestingly, they indicate different approaches of lesion recognition and identification for the eukaryotic NER system, depending on the type of lesion. Furthermore, distinct structural properties of the two different helicases involved in prokaryotic and eukaryotic NER result in and explain the different strategies observed for these two systems. Importantly, these experimental and analytical approaches can be applied not only to the study of DNA repair but also very similarly to other DNA interacting protein systems such as those involved in replication or transcription processes.
Introdução
A microscopia de força atômica (AFM) é uma poderosa técnica para a análise das interações proteína-DNA 1 , 2 , 3 , 4 , 5 , 6 , 7 , 8 , 9 . Requer apenas baixas quantidades de material de amostra para visualizar diretamente amostras heterogêneas com uma resolução no nível de molécula única. A heterogeneidade pode resultar de diferentes estados conformacionais ou oligoméricos de uma proteína. Em particular, no contexto de amostras de proteína-ADN, os complexos de proteínas podem exibir estequiometrias e / ou conformações diferentes induzidas por ligação ao ADN em geral ou ligação a um local alvo específico dentro do ADN. As amostras heterogéneas também podem conter dois (ou mais) tipos diferentes de proteínas e diferentes complexos de proteínas( Por exemplo , consistindo de apenas um tipo de proteína versus complexos heteroméricos) podem interagir de forma diferente com o DNA. Os estudos aqui discutidos exploram imagens AFM no ar em amostras estáticas e secas de proteínas de reparo de DNA ligadas a fragmentos de DNA longos (~ 900 pares de bases, bp) que contêm uma lesão, o que representa um alvo dessas proteínas. A alta resolução molecular do AFM permite a distinção entre diferentes tipos de complexos protéicos e determinar as posições de ligação das proteínas nos fragmentos de DNA. É importante notar que as lesões são introduzidas nos substratos de ADN em posições bem definidas. Uma vez que a posição do local da lesão no DNA é conhecida, as distribuições de proteínas ligadas ao DNA proporcionam uma visão das propriedades (diferentes) de reconhecimento de lesões dos complexos de proteínas (diferentes), por exemplo , como reconhecem um tipo particular de lesão A ADN não danificado) 2 , 3 , 4 , 5 , 6 . Suas posições no DNA também permitem a distinção entre complexos de proteínas ligados especificamente às lesões e complexos ligados inespecificamente em qualquer outra parte do DNA. A caracterização separada destes diferentes tipos complexos (complexos ligados especificamente à lesão versus complexos não específicos) pode revelar alterações conformacionais potenciais nos complexos induzidas na identificação do local alvo.
As proteínas de reparo do DNA focadas aqui são helicases que são responsáveis pelo reconhecimento de lesão na via de reparo de excisão de nucleotídeos (NER). Em bactérias, o NER é conseguido pelas proteínas UvrA, UvrB e UvrC. UvrA é responsável pela detecção inicial de lesões num complexo de varrimento de ADN UvaA 2 / UvrB 2 . Após a verificação da lesão por UvrB, este complexo converte-se em UvrB monomérico ligado no local da lesão e este complexo específico pode então recrutar o pEndonuclease NER de rokaryotic UvrC. A UvrC excisa um segmento curto (12-13 nt) de ADN de cadeia simples (ssDNA) contendo a lesão. O estiramento em falta é então reenchido pela ADN polimerase. Finalmente, a ADN ligase sela o estiramento recentemente sintetizado com o DNA original 9 , 10 . Em eucariotas, a maioria das proteínas da cascata NER são parte do grande complexo de transcrição do complexo II H (TFIIH). Após detecção de lesão inicial através do complexo trimérico CEN2-XPC-HR23B, TFIIH é recrutado para o local alvo de ADN. Quando XPD dentro do complexo verifica a presença de uma lesão alvo NER, as endonucleases EER eucarióticas XPG e XPF são recrutadas para excisar um trecho curto (24-32 nt) de ssDNA contendo a lesão 9,10 . Aqui, especificamente, foram estudadas as helicases UvrB e XPD de NER procariótico e eucariótico, respectivamente. Estas helicases requerem uma região nãoDNA (uma bolha de ADN) para enfiar numa das duas cadeias simples de ADN e subsequentemente translocar ao longo desta cadeia alimentada por hidrólise de ATP. Além das lesões de DNA, foi introduzida uma bolha de ADN nos substratos que funciona como local de carga para as proteínas.
O procedimento para a preparação de substratos específicos de DNA de lesão já foi descrito anteriormente 11 . Requer uma construção de ADN circular (plasmídeo) com dois locais de restrição estreitamente espaçados para uma nickase. No contexto deste estudo, utilizou-se o plasmídeo pUC19N (2729 pb) (criado pelo laboratório de S. Wilson, NIEHS). Este plasm�eo cont� tr� locais de restri�o espa�dos para a nickase Nt.BstNBI que enquadram um alongamento de 48 nucle�idos (nt). Após incubação com a nquase, o trecho de ssDNA entre estes locais pode ser removido e substituído por um oligonucleótido contendo qualquer característica alvo. Após cada passo, a digestão enzimática completa é testada através de gel de agaroseEletroforese. O ADN circundado circular pode ser distinguido devido à sua mobilidade electroforética inferior em comparação com o plasmídeo super-enrolado original. A separação do ADN e a substituição do estiramento removido pelo oligonucleótido de substrato específico podem ser avaliadas através de digestão com uma enzima de restrição que incisa o substrato exclusivamente dentro da região entre os entalhes. A linearização do plasmídeo circular pela enzima será assim suprimida para o ADN gapped e restaurada após a inserção do oligonucleótido específico. Finalmente, dois locais de restrição de endonuclease (idealmente cortadores únicos) permitem a geração de um substrato de ADN linear, com o comprimento desejado e com o local alvo específico numa posição definida assim como uma bolha de ADN a uma distância da lesão, quer em 5 'Ou 3'.
O reconhecimento das lesões pelas helicases NER pode ser investigado através de imagens AFM. A translocação de ADN bloqueado das helicases em tO local da lesão é visível como um pico na distribuição da posição da proteína no DNA e indica o reconhecimento da lesão. Uma vez que a translocação do ADN destas helicases é ainda direccional, com uma polaridade de 5 'a 3', a dependência do reconhecimento da lesão na posição do local de carga (bolha de ADN a montante ou a jusante da lesão) também indica se a lesão é preferencialmente reconhecida Na cadeia de ssDNA não translocada ou no lado oposto, não translocada 5 , 9 . Nas seções a seguir, os métodos utilizados serão introduzidos e os principais achados dessas experiências serão brevemente discutidos. Importante, analogamente ao trabalho exemplar sobre o reparo de DNA mostrado aqui, a imagiologia AFM pode ser aplicada ao estudo de diferentes sistemas de interação de DNA, tais como replicação ou transcrição de DNA 8 , 12 , 13 , 14 .
Protocolo
1. Preparação da amostra
- Preparação de substratos de ADN 11
- Gerando um gap ssDNA no plasmídeo
- Digerir completamente uma amostra do plasmídeo (aqui: pUC19 modificado, pUC19N) num tubo de reacção com uma nickase apropriada (aqui: Nt.BstNBI) seguida por inactivação por calor enzimático, utilizando condições de acordo com o protocolo do fabricante (ver a Figura 1 para um esquema apresentação). Comece com ~ 50 μL e ~ 500 nM plasmídeo para um rendimento suficiente.
- Verificar plasmídeo entalhamento por electroforese em gel de agarose em amostras diluídas (~ 20 nM) 15 . Usar luvas para proteção contra o corante de ligação ao DNA usado para a visualização do DNA.
NOTA: Diferentes mobilidades electroforéticas permitem a distinção entre ADN de plasmídeo circular cortado (relaxado) e super-enrolado ( Figura 1 ). - Remova o estiramento de ssDNA incisado (entre os locais de entalhe) do plasmídeo porIncubação com um excesso de 10 vezes do oligonucleótido complementar (oligonucleótido 1 na Tabela 1 ), agitação a 300 rpm durante 30 min perto da temperatura de fusão do oligonucleótido (aqui: 68 ° C para pUC19N) num bloco de aquecimento ( Figura 1 ).
- Separar o plasmídeo gapped dos fragmentos de ADN menores utilizando um filtro de corte de peso molecular de 50 kDa (MWCO) por centrifugação durante 10 min a 10 000 xg numa centrífuga de mesa ( Figura 1 ). Para extrair o ADN concentrado do filtro, inverter o filtro e inserir num novo tubo de reacção de 1,5 mL. Centrifugar durante 3 min a 1000 x g.
- Reabastecer a amostra de ADN concentrada resultante para 500 μL com água desionizada e filtrada, adicionar um excesso adicional de 5 vezes de oligonucleótido complementar e repetir os passos 1.1.1.3 e 1.1.1.4 pelo menos 3 vezes.
- Ensaio para detecção completa do ADN por incubação com uma enzima de restrição adequada (aqui: XhoI ou BglII) utilizando conditiOns de acordo com a descrição do fabricante. Utilizar amostras de ADN diluídas (cortadas contra folheadas) (~ 20 nM). Executar uma electroforese em gel de agarose para distinguir entre o plasmídeo linearizado (que não contém intervalo ssDNA) e o ADN não recortado (ADN bloqueado) utilizando o ADN cortado como controlo positivo (incluir uma escada de ADN para referência, Figura 1 ). Use luvas.
- Reabastecimento da lacuna com oligonucleótidos modificados de ssDNA
- Recozimento do plasmídeo gapped através de incubação com um excesso ~ 25 vezes de um oligonucleótido fosforilado 5 'que contém o local alvo específico escolhido, incubando a ~ 45 ° C durante 4 h ( Figura 1 ). Neste caso, utilizar ssDNA de 48 nt contendo uma lesão, quer uma timina aduzida por fluoresceína quer um dímero de pirimidina de ciclobutano (CPD), e adicionalmente uma sequência não complementar curta (8 nt) para produzir bolhas de ADN (ver Tabela 1 ).
- Ligar covalentemente o inserto recozido ao plasmídeo por incubaCom T4 DNA ligase durante a noite à temperatura ambiente de acordo com o protocolo do fabricante ( Figura 1 ). Para esta reacção, adicione-se ATP contendo solução tampão concentrada para produzir condições de tampão adequadas para a ligase ( por exemplo , use tampão de reacção UvrB: Tris-HCl 50 mM pH 7,5, MgCl2 10 mM, KCl 50 mM, DTT 5 mM, ATP).
- Ensaio para a inserção do oligonucleótido específico no plasmídeo bloqueado por digestão de amostras diluídas com uma enzima de restrição adequada (o mesmo que em 1.1.1.6) de acordo com o protocolo do fabricante seguido de uma electroforese em gel de agarose (como em 1.1.1.6, Figura 1 ).
- Preparação de substrato de ADN linear
- Digerir o ADN plasmídico modificado com enzimas de restrição (idealmente com um único local de restrição no plasmídeo) utilizando as condições recomendadas pelo fabricante ( Figura 1 ). Esta etapa produz fragmentos lineares que contêmModificação inserida numa posição definida. Aqui, utilizar o corte SspI e BspQI nas posições 613 e 255 pb a montante e a jusante do inserto, respectivamente, resultando num fragmento de ADN de 916 pb contendo o local da lesão específica a ~ 30% do comprimento do ADN.
NOTA: Para experiências de imagiologia com AFM como aqui descrito, fragmentos de ADN com comprimentos entre ~ 200 pb e ~ 2 000 pb são substratos adequados. - Isolar o fragmento alvo através de electroforese em gel de agarose e extracção de gel utilizando um kit comercial. Usar luvas para proteção.
- Opcionalmente, corte o gel de agarose com um bisturi para separar a escada de ADN e as pistas de amostra diluídas (duas primeiras pistas) das pistas que contêm o ADN concentrado a ser purificado. Apenas exponha a parte do gel com as duas primeiras pistas à irradiação UV colocando apenas esta parte do gel numa mesa UV.
- Cortar a banda correspondente ao fragmento de escolha com um bisturi. Re-unir as duas partes do gel (fora da aba UVLe). Utilizar a posição da banda excisada da amostra diluída como orientação para a posição da banda do substrato de ADN desejado. Considere a maior concentração cortando fatias ligeiramente mais largas do que para o controlo diluído.
NOTA: Uma vez que aqui foi investigado o reconhecimento de lesões UV, a introdução de lesões UV adicionais através da irradiação UV foi cuidadosamente evitada no substrato de ADN por esta abordagem.
- Cortar a banda correspondente ao fragmento de escolha com um bisturi. Re-unir as duas partes do gel (fora da aba UVLe). Utilizar a posição da banda excisada da amostra diluída como orientação para a posição da banda do substrato de ADN desejado. Considere a maior concentração cortando fatias ligeiramente mais largas do que para o controlo diluído.
- Calcular a concentração de ADN c da absorção a 260 nm medida por um espectrofotómetro UV-Vis utilizando a lei de Lambert-Beer com um coeficiente de extinção molar médio de ADN de cadeia dupla (dsDNA) de ε 260 nm ~ 6.700 M -1 cm -1 por pb:
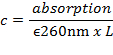
Onde L é o comprimento do percurso (comprimento da câmara de medição, tipicamente 1 cm).
- Digerir o ADN plasmídico modificado com enzimas de restrição (idealmente com um único local de restrição no plasmídeo) utilizando as condições recomendadas pelo fabricante ( Figura 1 ). Esta etapa produz fragmentos lineares que contêmModificação inserida numa posição definida. Aqui, utilizar o corte SspI e BspQI nas posições 613 e 255 pb a montante e a jusante do inserto, respectivamente, resultando num fragmento de ADN de 916 pb contendo o local da lesão específica a ~ 30% do comprimento do ADN.
- Gerando um gap ssDNA no plasmídeo
- Expressão e purificação de proteínas
- Recombinantemente expressar UvrB em E. coli e purificar a proteína via padrão afinidade de talão de quitina 15 e cromatografia de exclusão de tamanho 15 como descrito anteriormente 16 .
NOTA: O gene uvrB de Bacillus caldotenax tinha sido clonado no vector pTYB1. - Expressar XPD em E. coli e purificar a prote�a atrav� de afinidade n�uel IDA de n�uel 15 e cromatografia de exclus� de tamanho 15 seguida por cromatografia de permuta ani�ica 15 , como anteriormente descrito 17 .
NOTA: O gene para a proteína do grupo D de Chaetomium thermophilum Xeroderma pigmentosum (XPD) tinha sido clonado no vector pBADM11. C. thermophilum p44 foi uma espécie de presente do laboratório de Caroline Kisker, expressa e puriConforme descrito 17 .
- Recombinantemente expressar UvrB em E. coli e purificar a proteína via padrão afinidade de talão de quitina 15 e cromatografia de exclusão de tamanho 15 como descrito anteriormente 16 .
2. Experiência AFM
- Preparação de amostra
- Opcionalmente, pré-incubar o substrato de ADN a 65 ° C durante 10 minutos num bloco de calor para remover potenciais cristais de micro-sal que podem ter-se formado durante o armazenamento no frigorífico.
- Preparar o (s) tampão (s) de reacção a uma concentração de 10 vezes (10 x tampões).
NOTA: O tampão de reacção XPD à concentração 1x continha Tris-HCl 20 mM pH 7,5, KCl 10 mM, MgCl2 5 mM, TCEP 1 mM, ATP 2 mM; 1x tampão de reacção UvrB continha 50 mM de Tris-HCl a pH 7,5, 50 mM de KCl, 10 mM de MgCl2, 5 mM de DTT e 1 mM de ATP. - Pré-incuba as proteínas a uma concentração mais elevada em condições de incubação adequadas para aumentar a formação do complexo. Pré-diluir um pequeno volume ( por exemplo , 1 μL) das proteínas individuais em 1x tampão de reacção de proteína para a concentração desejada e misturar pequenos volumes ( eg , 1 μL) de tIndividual de proteínas num tubo de reacção de 0,5 mL.
- Coloque o tubo num bloco de calor para temperaturas de incubação superiores à temperatura ambiente. Escolha uma razão de concentração dependendo da estequiometria complexa esperada, quer equimolar, quer das concentrações correspondentes. Aqui, incubar 1 μL de XPD (20 μM em 1 x tampão de reacção XPD) e 1 μL de p44 (20 μM em tampão de reacção 1x XPD) a 10 μM cada 10 min a 37 ° C.
- Incubar amostras em concentrações adequadas de proteína e DNA em tampão de reação protéica em um tubo de reação de 0,5 mL. Aqui, use UvrB 500 nM ou 1 μM XPD + 1 μM p44 e ADN 100 nM. Pipetar pequenos volumes para economizar material, por exemplo , 0,25-0,5 μL de proteína (pré-diluída até uma concentração de incubação de 10 vezes) e ADN num volume total de 2,5-5 μL de tampão de reacção de proteína 1x. Aqui, incubar durante 30 min a 37 ° C num bloco de calor. Girar rapidamente para baixo num tubo de reacção (~ 1 s) num centrifugador de mesaE para garantir a mistura de pequenos volumes.
- Depósito da amostra
- Preparar um substrato de mica: cortar um pedaço aproximadamente 1 x 1 cm 2 de mica de tiras maiores usando um bisturi. Retirar as camadas superiores da peça de mica mineral multicamadas usando fita adesiva para revelar uma superfície de substrato limpa, plana e atômica.
NOTA: A peça de mica pode ser reutilizada para experiências múltiplas por remoção de camada (s) adicional (is). - Preparar o tampão de deposição de AFM com água filtrada desionizada, eg , HEPES 25 mM pH 7,5, Na-acetato 25 mM, acetato Mg 10 mM. Filtrar através de um filtro de seringa de 0,02 μm.
NOTA: Os catiões divalentes no tampão de deposição servem para quelar as moléculas de ADN negativamente carregadas para a superfície de mica, que também é carregada negativamente a pH neutro. Se a concentração relativamente elevada de ião Mg2 + no tampão de deposição coloca um problema para um sistema de proteína-ADN particular, alternativamente,A superfície de mica pode ser pré-carregada com grupos amino usando química baseada em silatrane para fornecer cargas de superfície positivas para ancoragem 18 . A deposição da amostra pode então ser realizada num tampão que não contém ou apenas pequenas quantidades de catiões divalentes. - Diluir a amostra (ver 2.1) para deposição imediata sobre mica em tampão de deposição. Deposite um pequeno volume (aqui: 20 μL).
NOTA: Os fatores de diluição dependem da concentração da amostra. Aqui, diluir as amostras 50-100x. Como regra geral, ~ 1 nM DNA resulta numa boa cobertura superficial para ~ 1.000 pb. - Imediatamente enxaguar a amostra de três a quatro vezes com alguns mililitros de água desionizada filtrada, remover o excesso de líquido e secar com um leve fluxo de azoto. Todo o processo, desde a deposição da amostra até às amostras secas, pode ser realizado em menos de 30 s.
- Fixar o pedaço de mica em um slide microscópio com fita adesiva em suas bordas.
NOTA: Diferentes sistemas AFM têm dPara a fixação da amostra no palco. Outros AFMs possuem fases magnéticas, e as peças de mica são fixadas em discos magnéticos, por exemplo , usando cola térmica. Detalhes do sistema AFM usado aqui podem ser encontrados na Tabela de Materiais.
- Preparar um substrato de mica: cortar um pedaço aproximadamente 1 x 1 cm 2 de mica de tiras maiores usando um bisturi. Retirar as camadas superiores da peça de mica mineral multicamadas usando fita adesiva para revelar uma superfície de substrato limpa, plana e atômica.
- Imagem AFM
- Colocar a amostra (ver 2.2.5) centralmente no estágio AFM e fixar a lâmina do microscópio no palco com pastilhas magnéticas.
- Insira a ponta AFM no suporte da ponta. Use cantilevers com uma ponta AFM afiada (<10 nm) adequada para oscilar, imagem de modo de contato intermitente no ar. Use sondas AFM, por exemplo, conforme listado na Tabela de Materiais . Aqui (dependendo do AFM), fixe a ponta no suporte sob um grampo apertando o parafuso de aperto (apertado com os dedos). Insira o suporte da ponta na cabeça de medição AFM. Descanse a cabeça em suas costas para esta etapa.
- Coloque a cabeça de medição AFM no topo da amostra. Os detalhes dependem do modelo AFM. Aqui mA certeza de que a cabeça fica estável com as pernas dentro do estágio indentations. Certifique-se de que a mica está localizada no palco onde a ponta AFM paira diretamente acima dela. Os parafusos de micrômetro à direita da platina permitem o posicionamento fino da amostra.
- Alinhe o laser AFM na parte traseira do cantilever para a força de sinal ideal do fotodetector sensível à posição em que o laser é refletido. Os detalhes dependem do modelo AFM.
- Aqui, gire as rodas no lado direito e traseiro da cabeça de medição AFM para ajustar as posições x e y do laser AFM para direcioná-lo centralmente para a extremidade do cantilever. Observe o sinal de reflexão na janela de vídeo AFM (se disponível, aqui: pressione o ícone da câmera e escolha a entrada: Svideo).
- Uma vez posicionados grosseiramente, otimize o sinal de soma do detector (soma em soma e janela de medidor de deflexão no software AFM) ajustando a posição do laser com as duas rodas (fique no final do cantilever, a soma signaL depende do tipo AFM e cantilever, aqui: apontar para soma> 5).
- Zero o sinal de diferença dos diodos superior e inferior da matriz do detector (aqui: sinal de deflexão na janela Sum and Deflection Meter) direcionando a reflexão do laser AFM para o centro do detector (aqui: gire a roda no lado esquerdo da medição AFM cabeça).
NOTA: Os desvios de zero indicam então a deflexão do cantilever devido às interações superficiais, que são traduzidas em informações de altura pelo AFM. - Determine a freqüência de ressonância em cantilever por uma melodia de freqüência implementada no software AFM (aqui: auto-ajuste de comando na janela Master Panel / Tune). Escolha uma amplitude correspondente à entrada de 1V para o piezo que aciona a oscilação do cantilever. Ajuste a frequência de oscilação para um valor ligeiramente inferior (5%) à frequência de ressonância e zero à fase de oscilação.
- Aproxime a ponta para a superfície da amostra usando o engE (aqui: command Engage na janela do Master Panel) até que a configuração de proteção (set-point) seja alcançada. Use ~ 2% de corte da amplitude de oscilação de nível livre como o set-point (aqui, entre em Set Point 980 mV no Master Panel).
- Fixe a ponta AFM com a superfície da amostra, baixando o ponto de ajuste usando o software AFM. Aponte para a imagem de modo repulsivo com a fase de oscilação do cantilever (fase na janela Sum and Deflection Meter) logo abaixo da fase de nível livre (antes de engatar, aqui normalmente ~ 70). Aqui, use pontos de ajuste finais típicos na ordem de 70-80% da amplitude de nível livre (1 V).
- Antes de digitalizar, escolha os sinais para gravação no Painel Mestre de Canal. Escolha altura (Ht) e amplitude (Am).
- Inicie a varredura de amostra (aqui: comando Fazer varredura na janela do Painel Principal). Use uma velocidade de varredura de, por exemplo, 2,5 μm / s (comando Velocidade de varredura no painel mestre) para áreas de superfície de imagem de 4 μm x 4 μm ou 8 μm x 8 μm (insira o tamanho de digitalização no painel mestreL) com resoluções de pixel de 2.048 ou 4.096 (Pontos de Varredura e Linhas de Varredura no Painel Principal), respectivamente.
- Salve o arquivo de imagem (comando Salvar Imagem no Painel Principal) sem modificações de ajuste (escolha Nenhum no Painel de Mestre / Salvar Ajuste de Plano).
- Para análise adicional, processe a imagem salva. Carregue a imagem (comando Procurar dados salvos no menu Análise do AFM). Abra o Painel de Modificação (pressione M na parte superior da imagem). Aplique um planefit em dimensões xey para a altura da imagem (extensão HtR) (comando XY na janela Planefit no painel Modify, escolha Planefit Order 3). Em seguida, aplainar a imagem (comando Aplastar na janela achatada no painel Modificar) escolhendo Ordem achatada 3.
- Exporte a imagem como um arquivo TIFF (pressione Comandos na parte superior da imagem, escolha Exportação TIFF 2x correspondente a resolução de 2.048 pixels) para análise posterior.
3. Análise do AFM
- Volume do complexo proteico
- Pré-Selecionar complexos proteína-DNA relevantes por inspeção visual direta das imagens no software AFM, usando um esquema de cores adequado para maximizar as diferentes aparências de diferentes complexos de tamanho (ver diferentes cores e tamanhos de diferentes complexos na Figura 2) , aqui: esquema de cores SeaLandAndFire in O software AFM). Para as amostras de XPD, os complexos possíveis incluíam o ADN de XPD / p44 , bem como o ADN de XPD e p44-DNA, com tamanhos distintamente diferentes devido às massas moleculares relativamente grandes e diferentes de XPD (~ 95 kDa) e p44 (~ 40 kDa) .
- Medir volumes de picos de proteínas individuais no DNA para verificar o tipo complexo. Isso pode ser conseguido com software de imagem diferente (aqui: a ferramenta de seção do software AFM).
- Medir a altura (h) e diâmetro (d) das seções de pico de proteína com os cursores de janela de seção. Meça o diâmetro próximo da base da seção de partículas.
- Determine o volume (V) da ervilhaKs usando modelos matemáticos simples, por exemplo , usando a seguinte fórmula, que é baseado em um modelo de tampão esférico:
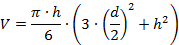
- Traduzir volumes medidos em uma massa molecular de proteína aproximada.
- Inicialmente, calibrar o sistema AFM para a conversão de volume em peso molecular (MW) utilizando uma gama de proteínas com peso molecular conhecido (aqui: um total de 12 experiências com entre 25 e 851 pontos de dados foram conduzidos em 5 proteínas diferentes em monómeros, diméricos , Trim�ico ou tetram�ico) 4 , 19 , 20 . Depósito e proteínas de imagem (como descrito acima, em 2.2 e 2.3) e medir seus volumes como descrito acima (3.1.2). Traçar os volumes sobre os pesos moleculares conhecidos. O gráfico resultante mostrará uma relação linear entre V e MW, cuja equação pode ser obtida porUma linha adequada aos dados. Para o sistema AFM aqui utilizado, foi obtida a seguinte relação 19 :
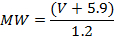
NOTA: Este passo não tem de ser repetido antes de cada medição, mas só é feito uma vez. A calibração de volume para MW pode ser aplicada a imagens obtidas em condições de imagem semelhantes utilizando sondas AFM com diâmetros ligeiramente variáveis. - Com base nos volumes medidos (3.1.2) e na calibração V-MW (3.1.3.1), determine o MW aproximado dos complexos protéicos 20 , que fornece informações sobre seu conteúdo molecular. Para amostras de XPD / p44, obtiveram-se ~ 50 kDa, ~ 100 kDa e ~ 140 kDa, consistentes com complexos de p44-DNA (ou picos causados por mera superestrutura de ADN), picos de XPD e picos de XPD / p44 respectivamente .
- Inicialmente, calibrar o sistema AFM para a conversão de volume em peso molecular (MW) utilizando uma gama de proteínas com peso molecular conhecido (aqui: um total de 12 experiências com entre 25 e 851 pontos de dados foram conduzidos em 5 proteínas diferentes em monómeros, diméricos , Trim�ico ou tetram�ico) 4 , 19 , 20 . Depósito e proteínas de imagem (como descrito acima, em 2.2 e 2.3) e medir seus volumes como descrito acima (3.1.2). Traçar os volumes sobre os pesos moleculares conhecidos. O gráfico resultante mostrará uma relação linear entre V e MW, cuja equação pode ser obtida porUma linha adequada aos dados. Para o sistema AFM aqui utilizado, foi obtida a seguinte relação 19 :
- Posições complexas de proteínas no DNA
- Determinar o DNAComprimento dos fragmentos.
- Trace os fragmentos de DNA nas imagens AFM, por exemplo , com a função de linha livre de um software de análise de imagem adequado (veja por exemplo Tabela de Materiais) e meça o comprimento da linha.
NOTA: Excluir agregados de DNA bem como fragmentos cortados pelas margens da imagem. - Bin e traçar os comprimentos de DNA de toda a experiência em um histograma, usando análise de dados adequada e software gráfico ( por exemplo , ver Tabela de Materiais ).
- Ajustar as distribuições de comprimento com uma curva gaussiana na análise de dados e software gráfico para determinar o comprimento dos fragmentos de DNA. Para se conhecer a posição do sítio alvo de ADN inserido (ver 1.1), inclua apenas fragmentos de ADN com o comprimento correcto dentro de dois desvios padrão do centro da curva de Gauss (y = y0 + z exp (-2xxc ) 2 / w 2 ), onde z é um fator norma e x c w são o centro eLargura total máxima do Gaussiano) em análises adicionais.
NOTA: O comprimento do ADN no centro da curva de Gauss deve estar próximo do comprimento teórico do fragmento de ADN (calculado utilizando 0,34 nm / pb). Comprimentos de até 10% mais curtos do que o valor teórico são típicos e são provavelmente causados por limites de resolução AFM.
- Trace os fragmentos de DNA nas imagens AFM, por exemplo , com a função de linha livre de um software de análise de imagem adequado (veja por exemplo Tabela de Materiais) e meça o comprimento da linha.
- Determinar posições de picos de proteína em substratos de DNA.
- Medir a distância dos picos de proteína do final do fragmento de DNA mais próximo, como descrito em 3.2.1.1 e dividir pelo comprimento total do DNA para obter distâncias em unidades de fração de comprimento de DNA.
- Bin e traçar as distâncias medidas em um histograma usando um software adequado de análise de dados e gráficos (ver, por exemplo , Figura 3 ). Como regra geral, escolha um tamanho de escaninho que forneça aproximadamente √n escaninhos para n pontos de dados.
NOTA: Uma vez que as extremidades do ADN não podem ser distinguidas aqui, trace apenas uma fracção do comprimento de ADN 0,5 (centro do fragmento de ADNMento). Como a ligação à extremidade do DNA não é o foco do estudo, exclua as extremidades do DNA das análises, começando a separar os dados da posição ligeiramente após a posição 0 ( por exemplo , aqui: binning de uma fração de comprimento de DNA de 0,02).
- Determinar a especificidade do local alvo a partir de distribuições de posição complexas de proteínas.
- Ajustar o máximo (ocorrências de ligação melhoradas) na distribuição de posição com uma curva de Gauss como em 3.2.1.3 mas com base na altura da ligação de fundo (ver Figura 3 ).
- Calcular a especificidade S para o local específico versus o fundo de ADN n� espec�ico (mol�ulas de prote�a ligadas a s�ios de ADN n� espec�icos) com a seguinte f�mula 21
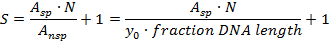
A sp : Número de complexos específicos (área sob a curva gaussiana)
A nsp : Número de complexos não específicos (área do fundo, Ie (comprimento de ADN da fracção) x (altura de fundo média y0); A fracção do comprimento de ADN aqui coberto é 0,48 para um histograma começando com 0,02 de comprimento de ADN)
N: número de locais de ligação ao ADN possíveis (aqui: N = 914 excluindo extremidades de ADN)
- Determinar o DNAComprimento dos fragmentos.
- Ângulos de dobra do DNA
- Usando a ferramenta de ângulo em um software de análise de imagem adequado, meça o ângulo β entre duas linhas colocadas centralmente ao longo do backbone de DNA e centrando no pico de proteína (veja, por exemplo , a inserção na Figura 4 ). Medir os ângulos para um número estatisticamente relevante de complexos proteína-DNA (> 50, idealmente> 100) 2 , 3 , 5 .
NOTA: O ângulo de dobra do ADN é definido como 180 ° -p2 , 3 , 5 . - Usando análise de dados e software gráfico(Ver Tabela de Materiais) produzem um histograma de distribuição do ângulo de dobra dividindo os ângulos de dobra do DNA.
- Ajuste a distribuição do ângulo de dobra com uma curva de Gauss. Se mais de um máximo for aparente na distribuição, escolha o ajuste de pico de Gauss múltiplo. O (s) centro (s) da (s) curva (s) gaussiana (s) dá (m) o estado médio do ângulo de dobra da espécie particular.
- Se um deslocamento no ângulo de dobra (s) (máximo do ajuste gaussiano) for aparente para distribuições, por exemplo, de diferentes variantes de proteína ou espécies de complexos de proteínas diferentes, aplique um teste t de Student para avaliar o nível de significância do alterar.
- Usando a ferramenta de ângulo em um software de análise de imagem adequado, meça o ângulo β entre duas linhas colocadas centralmente ao longo do backbone de DNA e centrando no pico de proteína (veja, por exemplo , a inserção na Figura 4 ). Medir os ângulos para um número estatisticamente relevante de complexos proteína-DNA (> 50, idealmente> 100) 2 , 3 , 5 .
Resultados
Distinguir entre diferentes tipos complexos baseados em volumes complexos de proteínas
A actividade de helicase da NER helicase UvrB procariótica é estimulada pela ligação ao ADN 22 , 23 . UvrB requer uma região não emparelhada no ADN (uma bolha de ADN) de modo a carregar correctamente numa das duas cadeias simples de ADNcs. In vivo , esta estrutura de ADN...
Discussão
As análises estatísticas de AFM de posições de ligação de proteínas em fragmentos de ADN longos que contêm locais alvo específicos podem revelar detalhes interessantes sobre as estratégias particulares utilizadas pela proteína para reconhecer estes sítios 2 , 3 , 4 , 5 , 6 . Para interpretar as distribuições de posição resultantes, as posiçõ...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
PUC19N, oligonucleótidos contendo CPD e p44 foram gentilmente fornecidos por Samuel Wilson, Korbinian Heil e Thomas Carell, e Gudrun Michels e Caroline Kisker, respectivamente. Este trabalho foi apoiado por doações da Deutsche Forschungsgemeinschaft (DFG) FZ82 e TE-671/4 para TI.
Materiais
| Name | Company | Catalog Number | Comments |
| Molecular Force Probe (MFP) 3D | Asylum Research | N/A | atomic force microscope (AFM) |
| Precision 390 | DELL | N/A | computer |
| ThermoMixer and 1.5 mL block | Eppendorf | 5382000015 | heat block for DNA preparation |
| Rotilabo Block-Heater H 250 & blocks for 0.5 mL tubes | Carl Roth GmbH | Y264.1 & Y267.1 | heat block for protein-DNA incubations |
| Mini-Sub Cell GT | Bio-Rad Laboratories GmbH | 1704467 | electrophoresis chamber with gel caster and power supply |
| Power Pac Basic | Bio-Rad Laboratories GmbH | 1645050 | electrophoresis power supply |
| Centifuge 5415 D with rotor | Eppendorf | 2262120-3 | table centrifuge |
| Ultra-Lum electronic UV transillumonator MEB-15 | Ultralum | 900-1322-02 | UV irradiation table |
| NanoDrop ND-1000 | VWR International / PEQLAB Biotechnologie GmbH | N/A | UV spectrophotometer |
| TKAX-CAD with 0.2 μm capsule filter | Unity Lab Services | N/A | water deionization and filter unit |
| Name | Company | Catalog number | Comments |
| Software | |||
| MFP software on Igor Pro | Asylum Research | N/A | AFM software |
| ImageJ (open source Java image processing) | NIH Image | N/A | Image analysis software |
| Excel (Microsoft Office) | Microsoft Corporation | N/A | data analysis software |
| Origin9 / Origin2016 | OriginLab Corporation | N/A | statistical data analysis and graphing software |
| Name | Company | Catalog number | Comments |
| Material | |||
| OMCL-AC240TS | Olympus | OMCL-AC240TS | AFM cantilevers |
| grade V-5 muscovite | SPI Supplies | 1805 | mica sheets |
| Amicon Ultra 0.5 mL 50k Ultracell | Millipore Ireland Ltd. | UFC505096 | centrifuge filters |
| NucleoSpin Extract II | Macherey-Nagel GmbH | 740 609.250 | Agarose gel extraction kit |
| Rotilabo cellulose paper type 111A | Carl Roth GmbH | AP59.1 | AFM deposition blotting paper |
| Anatop 25 (0.02 μm) | Whatman GmbH | 6809-2102 | syringe filter |
| SSpI, BspQI | New England Biolabs (NEB) | R0132, R0712 | restriction enzymes for DNA substrate preparation |
| XhoI, BglII | R0146, R0144 | restriction enzymes for DNA preparation controls | |
| nicking restriction enzyme Nt.BstNBI | New England Biolabs (NEB) | R0607 | nickase |
| T4 DNA ligase | New England Biolabs (NEB) | M0202S | Ligase |
| Tris, HEPES | Carl Roth GmbH | 4855, 9105 | buffer chemicals |
| NaCl, MgCl2, KCl, MgAcetate | Carl Roth GmbH | 3957, HN03, HN02, P026 | salt chemicals |
| NaAc | Sigma-Aldrich Chemie GmbH | 32318 | salt chemicals |
| DTT, TCEP, EDTA | 6908, HN95, 8040 | chemicals/reagents | |
| agarose, acetic acid, HCl | Carl Roth GmbH | 2267, 3738, K025 | reagents |
| ATP | Carl Roth GmbH | K054 | nucleotides |
| oligonucleotide #1 in Table 1 | Biomers | custom | complementary DNA oligonucleotide |
| oligonucleotides #2, #3, and #6 in Table 1 | Integrated DNA Technologies (IDT) | custom | fluorescein containing oligonucleotides |
| oligonucleotides #4 and #5 in Table 1 | private (available from e.g. TriLink or GlenResearch) | CPD containing oligonucleotides | |
| SafeSeal reaction tube 0.5 mL and 1.5 mL | Sarstedt | 72.704 and 72.706 | incubation tubes |
| GeneRuler 1 kb | Thermo Scientific | SM0311 | DNA ladder |
| 6x concentrate gel loading dye purple | New England Biolabs (NEB) | 51406 | DNA loading dye |
| Midori Green | Nippon Genetics Europe GmbH | 999MG28055 | DNA stain |
Referências
- Janicijevic, A., Ristic, D., Wyman, C. The molecular machines of DNA repair: scanning force microscopy analysis of their architecture. J. Microsc. 212 (3), 264-272 (2003).
- Wang, H., et al. DNA bending and unbending by MutS govern mismatch recognition and specificity. Proc. Natl. Acad. Sci. USA. 100 (25), 14822-14827 (2003).
- Tessmer, I., et al. Mechanism of MutS searching for DNA mismatches and signaling repair. J. Biol. Chem. 283 (52), 36646-36654 (2008).
- Wagner, K., Moolenaar, G., van Noort, J., Goosen, N. Single-molecule analysis reveals two separate DNA-binding domains in the Escherichia coli UvrA dimer. Nucleic Acids Res. 37 (6), 1962-1972 (2009).
- Buechner, C. N., et al. Strand-specific recognition of DNA damages by XPD provides insights into nucleotide excision repair substrate versatility. J Biol. Chem. 289 (6), 3613-3624 (2014).
- Van der Linden, E., Sanchez, H., Kinoshita, E., Kanaar, R., Wyman, C. RAD50 and NBS1 form a stable complex functional in DNA binding and tethering. Nucleic Acids Res. 37 (5), 1580-1588 (2009).
- Fuentes-Perez, M. E., Dillingham, M., Moreno-Herrero, F. AFM volumetric methods for the characterization of proteins and nucleic acids. Methods. 60, 113-121 (2013).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Miyagi, A., Lyubchenko, Y. L. Specificity of binding of single-stranded DNA-binding protein to its target. Biochemistry. 51, 1500-1509 (2012).
- Wirth, N., et al. Conservation and Divergence in Nucleotide Excision Repair Lesion Recognition. J. Biol. Chem. 291 (36), 18932-18946 (2016).
- Kuper, J., Kisker, C. Damage recognition in nucleotide excision DNA repair. Curr. Opin. Struct. Biol. 22, 88-93 (2012).
- Buechner, C. N., Tessmer, I. DNA substrate preparation for atomic force microscopy studies of protein-DNA interactions. J. Mol. Recognit. 26 (12), 605-617 (2013).
- Sun, Z., Tan, H. Y., Bianco, P. R., Lyubchenko, Y. L. Remodeling of RecG Helicase at the DNA Replication Fork by SSB Protein. Sci. Rep. 5, 9625 (2015).
- Billingsley, D. J., Bonass, W. A., Crampton, N., Kirkham, J., Thomson, N. H. Single-molecule studies of DNA transcription using atomic force microscopy. Phys. Biol. 9 (2), 021001 (2012).
- Maurer, S., Fritz, J., Muskhelishvili, G., Travers, A. RNA polymerase and an activator form discrete subcomplexes in a transcription initiation complex. EMBO J. 25 (16), 3784-3790 (2006).
- Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Theis, K., Chen, P. J., Skorvaga, M., Van Houten, B., Kisker, C. Crystal structure of UvrB, a DNA helicase adapted for nucleotide excision repair. EMBO J. 18 (24), 6899-6907 (1999).
- Kuper, J., et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 12 (9), e1001954 (2014).
- Shlyakhtenko, L. S., et al. Silatrane-based surface chemistry for immobilization of DNA, protein-DNA complexes and other biological materials. Ultramicroscopy. 97, 279-287 (2003).
- Roth, H. M., et al. XPB helicase regulates DNA incision by the Thermoplasma acidophilum endonuclease Bax1. DNA Repair. 11 (3), 286-293 (2012).
- Ratcliff, G. C., Erie, D. A. A Novel Single-Molecule Study to Determine Protein-Protein Association Constants. J. Am. Chem. Soc. 123 (24), 5632-5635 (2001).
- Yang, Y., Sass, L. E., Du, C., Hsieh, P., Erie, D. A. Determination of protein-DNA binding constants and specificities from statistical analyses of single molecules: MutS-DNA interactions. Nucleic Acids Res. 33 (13), 4322-4334 (2005).
- Caron, P. R., Grossman, L. Involvement of a cryptic ATPase activity of UvrB and its proteolysis product, UvrB* in DNA repair. Nucleic Acids Res. 16 (22), 10891-10902 (1988).
- Wang, H., et al. UvrB domain 4, an autoinhibitory gate for regulation of DNA binding and ATPase activity. J. Biol. Chem. 281 (22), 15227-15237 (2006).
- Chammas, O., Billingsley, D. J., Bonass, W. A., Thomson, N. H. Single-stranded DNA loops as fiducial markers for exploring DNA-protein interactions in single molecule imaging. Methods. 60 (2), 122-130 (2013).
- Truglio, J. J., et al. Structural basis for DNA recognition and processing by UvrB. Nat. Struct. Mol. Biol. 13 (4), 360-364 (2006).
- Kuper, J., Wolski, S. C., Michels, G., Kisker, C. Functional and structural studies of the nucleotide excision repair helicase XPD suggest a polarity for DNA translocation. EMBO J. 31 (2), 494-502 (2012).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Goosen, N. The presence of two UvrB subunits in the UvrAB complex ensures damage detection in both DNA strands. EMBO J. 21 (15), 4196-4205 (2002).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Hoeijmakers, J. H., Goosen, N. Architecture of nucleotide excision repair complexes: DNA is wrapped by UvrB before and after damage recognition. EMBO J. 20 (3), 601-611 (2001).
- Moolenaar, G. F., et al. The Role of ATP Binding and Hydrolysis by UvrB during Nucleotide Excision Repair. J. Biol. Chem. 275, 8044-8050 (2000).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados