Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Atommikroskopie Untersuchungen zur DNA-Läsionserkennung bei der Nucleotide Excision Repair
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Here, the study of different DNA lesion recognition approaches via single molecule AFM imaging is demonstrated with the nucleotide excision repair system as an example. The procedures of DNA and protein sample preparations and experimental as well as analytical details for the AFM experiments are described.
Zusammenfassung
AFM imaging is a powerful technique for the study of protein-DNA interactions. This single molecule method allows the simultaneous resolution of different molecules and molecular assemblies in a heterogeneous sample. In the particular context of DNA interacting protein systems, different protein complex forms and their corresponding binding positions on target sites containing DNA fragments can thus be distinguished. Here, an application of AFM to the study of DNA lesion recognition in the prokaryotic and eukaryotic nucleotide excision DNA repair (NER) systems is presented. The procedures of DNA and protein sample preparations are described and experimental as well as analytical details of the experiments are provided. The data allow important conclusions on the strategies by which target site verification may be achieved by the NER proteins. Interestingly, they indicate different approaches of lesion recognition and identification for the eukaryotic NER system, depending on the type of lesion. Furthermore, distinct structural properties of the two different helicases involved in prokaryotic and eukaryotic NER result in and explain the different strategies observed for these two systems. Importantly, these experimental and analytical approaches can be applied not only to the study of DNA repair but also very similarly to other DNA interacting protein systems such as those involved in replication or transcription processes.
Einleitung
Atomkraftmikroskopie (AFM) ist eine leistungsfähige Technik für die Analyse der Protein-DNA-Wechselwirkungen 1 , 2 , 3 , 4 , 5 , 6 , 7 , 8 , 9 . Es erfordert nur geringe Mengen an Probenmaterial, um heterogene Proben direkt mit einer Auflösung auf Einzelmolekülebene zu visualisieren. Heterogenität kann aus verschiedenen konformationellen oder oligomeren Zuständen eines Proteins resultieren. Insbesondere können im Rahmen von Protein-DNA-Proben Proteinkomplexe unterschiedliche Stöchiometrien und / oder Konformationen aufweisen, die durch DNA-Bindung im Allgemeinen induziert werden oder an eine spezifische Zielstelle innerhalb der DNA binden. Heterogene Proben können auch zwei (oder mehrere) verschiedene Arten von Proteinen und verschiedene Proteinkomplexe enthaltenFormen ( zB bestehend aus nur einer Art von Protein gegenüber heteromeren Komplexen) können unterschiedlich mit DNA interagieren. Die hier diskutierten Studien nutzten die AFM-Bildgebung in der Luft auf statischen, getrockneten Proben von DNA-Reparaturproteinen, die an lange (~ 900 Basenpaare, bp) DNA-Fragmente gebunden sind, die eine Läsion enthalten, die ein Ziel dieser Proteine darstellt. Die hohe molekulare Auflösung von AFM ermöglicht die Unterscheidung zwischen verschiedenen Arten von Proteinkomplexen und die Bestimmung der Bindungspositionen der Proteine an den DNA-Fragmenten. Wichtig ist, dass die Läsionen in gut definierten Positionen in die DNA-Substrate eingebracht werden. Da die Position der Läsionsstelle in der DNA bekannt ist, liefern die Verteilungen von Proteinen, die an DNA gebunden sind, einen Einblick in (verschiedene) Läsionserkennungseigenschaften der (verschiedenen) Proteinkomplexe, zB wie gut sie eine bestimmte Art von Läsion erkennen (verglichen Zu nicht beschädigter DNA) 2 , 3 , 4 , 5 , 6 . Ihre Positionen auf der DNA erlauben auch die Unterscheidung zwischen Proteinkomplexen, die spezifisch an den Läsionen und Komplexen gebunden sind, die unspezifisch anderswo an der DNA gebunden sind. Eine getrennte Charakterisierung dieser verschiedenen komplexen Typen (Komplexe, die spezifisch an der Läsion gegenüber unspezifischen Komplexen gebunden sind) können potentielle Konformationsänderungen in den Komplexen zeigen, die bei der Zielortidentifizierung induziert werden.
Die DNA-Reparaturproteine, die hier konzentriert sind, sind Helicasen, die für die Läsionserkennung in der Nucleotide Exzision Reparatur (NER) Weg verantwortlich sind. In Bakterien wird NER durch die Proteine UvrA, UvrB und UvrC erreicht. UvrA ist verantwortlich für die initiale Läsionserfassung in einem UvaA 2 / UvrB 2 DNA-Scan-Komplex. Nach der Läsionsüberprüfung durch UvrB wandelt sich dieser Komplex in monomeres UvrB um, das an der Läsionsstelle gebunden ist, und dieser spezifische Komplex kann dann das p rekrutierenRokaryotische NER Endonuklease UvrC. UvrC verbreitet eine kurze (12-13 nt) Strecke einzelsträngiger DNA (ssDNA), die die Läsion enthält. Die fehlende Dehnung wird dann durch DNA-Polymerase nachgefüllt. Schließlich versiegelt die DNA-Ligase die neu synthetisierte Dehnung mit der ursprünglichen DNA 9 , 10 . Bei Eukaryonten sind die meisten Proteine der NER-Kaskade Teil des großen, multimeren Transkriptionsfaktors II H (TFIIH) -Komplexes. Nach der ersten Läsionserfassung über den trimeren CEN2-XPC-HR23B-Komplex wird TFIIH an die DNA-Zielstelle rekrutiert. Wenn XPD innerhalb des Komplexes die Anwesenheit einer NER-Zielläsion verifiziert, werden die eukaryotischen NER-Endonukleasen XPG und XPF rekrutiert, um eine kurze (24-32 nt) Strecke von ssDNA, die die Läsion 9 , 10 enthält , zu exzimieren. Hier wurden insbesondere die Helicasen UvrB und XPD aus prokaryotischem und eukaryotischem NER untersucht. Diese Helicases erfordern eine ungepaarte Region in derDNA (eine DNA-Blase), um auf einen der beiden DNA-Einzelstränge zu fädeln und anschließend entlang diesem durch ATP-Hydrolyse angefeuerten Strang zu translozieren. Zusätzlich zu den DNA-Läsionen wurde daher eine DNA-Blase in die Substrate eingeführt, die als Ladestelle für die Proteine fungieren.
Das Verfahren zur Herstellung von spezifischen Läsions-DNA-Substraten wurde zuvor beschrieben 11 . Es erfordert ein kreisförmiges DNA-Konstrukt (Plasmid) mit zwei eng beabstandeten Restriktionsstellen für eine Nickase. Im Rahmen dieser Studie wurde das Plasmid pUC19N (2729 bp) verwendet (erstellt von S. Wilsons Laboratorium, NIEHS). Dieses Plasmid enthält drei eng beabstandete Restriktionsstellen für die Nickel Nt.BstNBI, die eine 48 Nukleotid (nt) Streckung bilden. Nach der Inkubation mit der Nickel kann die Strecke der ssDNA zwischen diesen Stellen entfernt und durch ein Oligonukleotid ersetzt werden, das ein beliebiges Zielmerkmal enthält. Nach jedem Schritt wird die vollständige enzymatische Verdauung über Agarosegel getestetElektrophorese Die vernetzte zirkuläre DNA kann aufgrund ihrer geringeren elektrophoretischen Beweglichkeit gegenüber dem ursprünglichen supercoiled Plasmid unterschieden werden. Das Anhängen der DNA und das Ersetzen der entfernten Dehnung durch das spezifische Substrat-Oligonukleotid können durch Verdauung mit einem Restriktionsenzym ausgewertet werden, das das Substrat ausschließlich innerhalb des Bereichs zwischen den Nicks aufnimmt. Die Linearisierung des zirkulären Plasmids durch das Enzym wird daher für die gapped DNA unterdrückt und nach der Insertion des spezifischen Oligonukleotids wiederhergestellt. Schließlich erlauben zwei Endonuklease-Restriktionsstellen (idealerweise Einzelschneider) die Erzeugung eines linearen DNA-Substrats mit der gewünschten Länge und mit der spezifischen Zielstelle an einer definierten Position sowie einer DNA-Blase in einem Abstand von der Läsion entweder in 5 'Oder 3' Richtung.
Die Erkennung der Läsionen durch die NER-Helicasen kann mittels AFM-Bildgebung untersucht werden . Stabile DNA-Translokation der Helicasen bei tEr Läsionsstelle ist als Peak in der Proteinpositionsverteilung auf der DNA sichtbar und zeigt eine Läsionserkennung an. Da die DNA-Translokation dieser Helicinen weiterhin mit einer 5'-bis-3'-Polarität gerichtet ist, zeigt die Abhängigkeit der Läsionserkennung an der Position der Ladestelle (DNA-Blase stromaufwärts oder stromabwärts der Läsion) auch an, ob die Läsion bevorzugt erkannt wird Auf dem translozierten oder auf dem entgegengesetzten, nicht translozierten ssDNA-Strang 5 , 9 . In den folgenden Abschnitten werden die angewandten Methoden eingeführt und wichtige Erkenntnisse aus diesen Experimenten werden kurz diskutiert. Wichtig ist, dass analog zur beispielhaften Arbeit zur hier beschriebenen DNA-Reparatur die AFM-Bildgebung auf die Untersuchung verschiedener DNA-interagierender Systeme wie der DNA-Replikation oder der Transkription 8 , 12 , 13 , 14 angewendet werden kann .
Protokoll
1. Probenvorbereitung
- Herstellung von DNA-Substraten 11
- Erzeugung einer ssDNA-Lücke im Plasmid
- Vollständig eine Probe des Plasmids (hier: modifiziertes pUC19, pUC19N) in einem Reaktionsgefäß mit einer geeigneten Nickel (hier: Nt.BstNBI), gefolgt von Enzymwärmeinaktivierung unter Verwendung von Bedingungen gemäß dem Protokoll des Herstellers (siehe Abbildung 1 für ein Schema) Präsentation). Beginnen Sie mit ~ 50 μl und ~ 500 nM Plasmid für eine ausreichende Ausbeute.
- Überprüfen Sie das Plasmid-Nicking durch Agarose-Gelelektrophorese auf verdünnte Proben (~ 20 nM) 15 . Tragen Sie Handschuhe zum Schutz gegen den DNA-Bindungsfarbstoff, der für die DNA-Visualisierung verwendet wird.
HINWEIS: Verschiedene elektrophoretische Mobilitäten erlauben die Unterscheidung zwischen nickter (entspannter) und supercoiled zirkulärer Plasmid-DNA (Abbildung 1 ). - Entfernen Sie die eingeschnittene ssDNA-Strecke (zwischen den Nickstellen) aus dem Plasmid durchInkubation mit einem ~ 10-fachen Überschuss des komplementären Oligonukleotids (Oligonukleotid 1 in Tabelle 1 ), Schütteln bei 300 U / min für 30 min nahe der Schmelztemperatur des Oligonukleotids (hier: 68 ° C für pUC19N) in einem Wärmeblock (Abbildung 1 ).
- Trennen Sie das gapped Plasmid aus den kleineren DNA-Fragmenten unter Verwendung eines 50 kDa-Molekulargewichts-Cutoff (MWCO) -Filters durch Zentrifugation für 10 min bei 10.000 xg in einer Tabellenzentrifuge (Abbildung 1 ). Um die konzentrierte DNA aus dem Filter zu extrahieren, den Filter umdrehen und in ein neues 1,5 ml Reaktionsrohr geben. 5 min bei 1000 x g zentrifugieren
- Nachfüllen der resultierenden konzentrierten DNA-Probe auf 500 μl mit deionisiertem, gefiltertem Wasser, fügt ein zusätzliches ~ 5-faches überschüssiges komplementäres Oligonukleotid hinzu und wiederholt die Schritte 1.1.1.3 und 1.1.1.4 mindestens 3 mal.
- Testen Sie auf vollständiges Spalten der DNA durch Inkubation mit einem geeigneten Restriktionsenzym (hier: XhoI oder BglII) unter Verwendung von conditiOns nach der Beschreibung des Herstellers. Verwenden Sie verdünnte (vernickelte versus gapped) DNA Proben (~ 20 nM). Führen Sie eine Agarose-Gelelektrophorese aus, um zwischen linearisiertem Plasmid (ohne ssDNA-Lücke) und nicht-eingeschnittener DNA (gapped DNA) unter Verwendung der nickten DNA als Positivkontrolle zu unterscheiden (einschließlich einer DNA-Leiter als Referenz, Abbildung 1 ). Trag Handschuhe.
- Nachfüllen der Lücke mit modifizierten ssDNA-Oligonukleotiden
- Glätten Sie das gapped Plasmid durch Inkubation mit einem ~ 25-fachen Überschuss eines 5'-phosphorylierten Oligonukleotids, das die spezifische Zielstelle (n) der Wahl enthält, wobei sie bei 45 ° C für 4 h inkubiert (Abbildung 1 ). Hierbei verwenden Sie 48 nt ssDNA, die eine Läsion enthält, entweder ein Fluorescein-adduziertes Thymin oder ein Cyclobutan-Pyrimidindimer (CPD) und zusätzlich eine kurze (8 nt) nicht komplementäre Sequenz zur Herstellung von DNA-Blasen (siehe Tabelle 1 ).
- Kovalent verknüpfen Sie den getemperten Einsatz mit dem Plasmid durch incubaMit T4-DNA-Ligase über Nacht bei Raumtemperatur nach dem Protokoll des Herstellers (Abbildung 1 ). Für diese Reaktion wird ATP mit konzentrierter Puffer-Stammlösung zur Herstellung geeigneter Pufferbedingungen für die Ligase zugegeben ( z. B. UvrB-Reaktionspuffer: 50 mM Tris-HCl pH 7,5, 10 mM MgCl 2 , 50 mM KCl, 5 mM DTT, 1 mM ATP).
- Test für die Insertion des spezifischen Oligonukleotids in das gapped Plasmid durch Verdauung von verdünnten Proben mit einem geeigneten Restriktionsenzym (wie in 1.1.1.6) gemäß dem Protokoll des Herstellers gefolgt von einer Agarose-Gelelektrophorese (wie in 1.1.1.6, Abbildung 1 ).
- Herstellung von linearem DNA-Substrat
- Digitieren Sie die modifizierte Plasmid-DNA mit Restriktionsenzymen (idealerweise mit einer einzigen Restriktionsstelle im Plasmid) unter Verwendung von vom Hersteller empfohlenen Bedingungen (Abbildung 1 ). Dieser Schritt erzeugt lineare Fragmente enthaltenDie eingefügte Änderung an einer definierten Position. Hierbei verwenden Sie SspI- und BspQI-Schnitt an den Positionen 613 und 255 bp stromaufwärts und stromabwärts des Inserts, was zu einem 916 bp-DNA-Fragment führt, das die spezifische Läsionsstelle bei ~ 30% der DNA-Länge enthält.
HINWEIS: Für AFM-Bildgebungsexperimente, wie hier beschrieben, sind DNA-Fragmente mit Längen zwischen ~ 200 bp und ~ 2000 bp geeignete Substrate. - Isolieren Sie das Zielfragment über Agarose-Gelelektrophorese und Gel-Extraktion mit einem kommerziellen Kit. Schutzhandschuhe tragen.
- Gegebenenfalls geschnittenes Agarosegel mit einem Skalpell, um die DNA-Leiter und verdünnte Probenspuren (erste zwei Bahnen) von den Bahnen zu trennen, die die zu reinigende konzentrierte DNA enthalten. Setzen Sie den Gelteil nur mit den ersten beiden Bahnen auf UV-Bestrahlung aus, indem Sie nur diesen Teil des Gels auf einen UV-Tisch legen.
- Schneide die Band entsprechend dem Fragment der Wahl mit einem Skalpell aus. Die beiden Gelteile (aus der UV-Lasche) wieder zusammenbringenLe). Verwenden Sie die Position des ausgeschnittenen Bandes aus der verdünnten Probe als Orientierung für die Position des Bandes des gewünschten DNA-Substrats. Berücksichtigung der höheren Konzentration durch Ausschneiden von etwas breiteren Scheiben als für die verdünnte Kontrolle.
HINWEIS: Weil hier die Erkennung von UV-Läsionen untersucht wurde, wurde durch das erfindungsgemäße Einbringen zusätzlicher UV-Läsionen durch UV-Bestrahlung im DNA-Substrat sorgfältig vermieden.
- Schneide die Band entsprechend dem Fragment der Wahl mit einem Skalpell aus. Die beiden Gelteile (aus der UV-Lasche) wieder zusammenbringenLe). Verwenden Sie die Position des ausgeschnittenen Bandes aus der verdünnten Probe als Orientierung für die Position des Bandes des gewünschten DNA-Substrats. Berücksichtigung der höheren Konzentration durch Ausschneiden von etwas breiteren Scheiben als für die verdünnte Kontrolle.
- Berechnen Sie die DNA-Konzentration c aus der Absorption bei 260 nm, gemessen mit einem UV-Vis-Spektrophotometer unter Verwendung des Lambert-Beer-Gesetzes mit einem doppelsträngigen DNA (dsDNA) durchschnittlichen molaren Extinktionskoeffizienten von ε 260nm ~ 6,700 M - 1 cm - 1 pro bp:
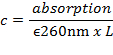
Wobei L die Weglänge ist (Messkammerlänge, typischerweise 1 cm).
- Digitieren Sie die modifizierte Plasmid-DNA mit Restriktionsenzymen (idealerweise mit einer einzigen Restriktionsstelle im Plasmid) unter Verwendung von vom Hersteller empfohlenen Bedingungen (Abbildung 1 ). Dieser Schritt erzeugt lineare Fragmente enthaltenDie eingefügte Änderung an einer definierten Position. Hierbei verwenden Sie SspI- und BspQI-Schnitt an den Positionen 613 und 255 bp stromaufwärts und stromabwärts des Inserts, was zu einem 916 bp-DNA-Fragment führt, das die spezifische Läsionsstelle bei ~ 30% der DNA-Länge enthält.
- Erzeugung einer ssDNA-Lücke im Plasmid
- Expression und Reinigung von Proteinen
- Rekombinant exprimieren UvrB in E. coli und reinigen das Protein über Standard-Chitin-Perlen-Affinität 15 und Größenausschlusschromatographie 15 wie zuvor beschrieben 16 .
HINWEIS: Das uvrB- Gen aus Bacillus caldotenax wurde in den pTYB1-Vektor kloniert. - Express-XPD in E. coli und reinigen das Protein über Standard-Nickel-IDA-Affinität 15 und Größenausschlusschromatographie 15, gefolgt von Anionenaustauschchromatographie 15 , wie zuvor beschrieben 17 .
HINWEIS: Das Gen für Chaetomium thermophilum Xeroderma pigmentosum Gruppe D Protein (XPD) wurde in den pBADM11 Vektor kloniert. C. thermophilum p44 war ein freundliches Geschenk von Caroline Kiskers Labor, ausgedrückt und puriWie beschrieben 17 .
- Rekombinant exprimieren UvrB in E. coli und reinigen das Protein über Standard-Chitin-Perlen-Affinität 15 und Größenausschlusschromatographie 15 wie zuvor beschrieben 16 .
2. AFM-Experiment
- Probenvorbereitung
- Gegebenenfalls das DNA-Substrat bei 65 ° C für 10 min in einem Hitzeblock vorinkubieren, um potentielle Mikrosalzkristalle zu entfernen, die sich während der Lagerung im Kühlschrank gebildet haben können.
- Bereiten Sie den Reaktionspuffer bei einer 10-fachen Konzentration (10x Puffer) vor.
HINWEIS: XPD-Reaktionspuffer bei 1x-Konzentration enthielt 20 mM Tris-HCl pH 7,5, 10 mM KCl, 5 mM MgCl 2 , 1 mM TCEP, 2 mM ATP; 1x UvrB-Reaktionspuffer enthielt 50 mM Tris-HCl pH 7,5, 50 mM KCl, 10 mM MgCl 2 , 5 mM DTT und 1 mM ATP. - Vor-Inkubieren von Proteinen in einer höheren Konzentration in geeigneten Inkubationsbedingungen, um die Komplexbildung zu verbessern Ein kleines Volumen ( zB 1 μl) der einzelnen Proteine in 1x Protein-Reaktionspuffer auf die gewünschte Konzentration verdünnen und kleine Volumina ( zB 1 μl) von t mischenEr einzelne Proteinlösungen in einem 0,5 mL Reaktionsrohr.
- Legen Sie das Röhrchen in einen Hitzeblock für Inkubationstemperaturen höher als Raumtemperatur. Wählen Sie ein Konzentrationsverhältnis in Abhängigkeit von der erwarteten komplexen Stöchiometrie, entweder äquimolare oder entsprechende Konzentrationen. Hier inkubieren 1 & mgr; l XPD (20 & mgr; M in 1 × XPD-Reaktionspuffer) und 1 & mgr; l p44 (20 & mgr; M in 1x XPD-Reaktionspuffer) bei 10 & mgr; M jeweils für 10 min bei 37 ° C.
- Inkubieren von Proben bei geeigneten Protein- und DNA-Konzentrationen in Protein-Reaktionspuffer in einem 0,5-ml-Reaktionsrohr. Hier verwenden Sie 500 nM UvrB oder 1 μM XPD + 1 μM p44 und 100 nM DNA. Pipettieren Sie kleine Volumina, um Material zu sparen, z. B. 0,25-0,5 μl Protein (vorverdünnt auf eine 10-fache Inkubationskonzentration) und DNA in einem Gesamtvolumen von 2,5-5 μl 1x Proteinreaktionspuffer. Hier inkubieren für 30 min bei 37 ° C in einem Hitzeblock. In einem Reaktionsschlauch kurz (~ 1 s) in eine Tafelzentrifuge gebenUm das Mischen von kleinen Volumina zu gewährleisten.
- Probenablagerung
- Bereiten Sie ein Glimmer-Substrat vor: schneiden Sie etwa 1 x 1 cm 2 Stück Glimmer aus größeren Streifen mit einem Skalpell. Streichen Sie die obersten Schichten des mehrschichtigen Glimmermineralstücks mit Klebeband ab, um eine saubere, flache und atomar glatte Substratoberfläche zu zeigen.
ANMERKUNG: Das Glimmerstück kann für mehrfache Experimente wiederverwendet werden, indem man weitere Schicht (e) abstreift. - Den AFM-Abscheidungspuffer mit deionisiertem, gefiltertem Wasser, zB 25 mM HEPES pH 7,5, 25 mM Na-Acetat, 10 mM Mg-Acetat, herstellen. Filter durch einen 0,02 μm Spritzenfilter.
HINWEIS: Zweiwertige Kationen im Abscheidungspuffer dienen dazu, die negativ geladenen DNA-Moleküle auf die Glimmeroberfläche zu chelatisieren, was ebenfalls bei neutralem pH negativ geladen ist. Wenn die relativ hohe Mg 2+ -Ionenkonzentration in dem Abscheidungspuffer ein Problem für ein bestimmtes Protein-DNA-System darstellt,Kann die Glimmeroberfläche mit Aminogruppen unter Verwendung von Silatrane-basierter Chemie vorbelastet werden, um positive Oberflächenladungen für die Verankerung zu liefern. Die Probenabscheidung kann dann in einem Puffer durchgeführt werden, der keine oder nur geringe Mengen an zweiwertigen Kationen enthält. - Die Probe (siehe 2.1) zur sofortigen Abscheidung auf Glimmer in Abscheidungspuffer verdünnen. Ein kleines Volumen einlegen (hier: 20 μL).
HINWEIS: Verdünnungsfaktoren hängen von der Probenkonzentration ab. Hier verdünnen Sie Proben 50-100x. Als Faustregel gilt ~ 1 nM DNA zu einer guten Flächendeckung für ~ 1000 bp. - Sofort die Probe drei bis viermal mit einigen Millilitern gefiltertem, deionisiertem Wasser abspülen, überschüssige Flüssigkeit abtupfen und in einem sanften Stickstoffstrom trocknen lassen. Der gesamte Prozess von der Probenablagerung zu getrockneten Proben kann innerhalb von weniger als 30 s durchgeführt werden.
- Fixieren Sie das Glimmerstück auf einem Mikroskop-Objektträger mit Klebeband an den Kanten.
HINWEIS: Verschiedene AFM-Systeme haben dIfferent Anforderungen für die Festsetzung der Probe auf der Bühne. Andere AFMs besitzen magnetische Stufen, und die Glimmerstücke sind auf Magnetplatten befestigt, z. B. mit thermischem Leim. Details zum hier verwendeten AFM-System finden Sie in der Werkstoffliste.
- Bereiten Sie ein Glimmer-Substrat vor: schneiden Sie etwa 1 x 1 cm 2 Stück Glimmer aus größeren Streifen mit einem Skalpell. Streichen Sie die obersten Schichten des mehrschichtigen Glimmermineralstücks mit Klebeband ab, um eine saubere, flache und atomar glatte Substratoberfläche zu zeigen.
- AFM-Bildgebung
- Legen Sie die Probe (siehe 2.2.5) mittig auf die AFM-Stufe und fixieren Sie den Mikroskop-Objektträger mit Magnetkissen auf die Bühne.
- Setzen Sie die AFM-Spitze in den Spitzenhalter ein. Verwenden Sie Ausleger mit einer scharfen (<10 nm) AFM-Sonde, die für oszillierende, intermittierende Kontaktmodus-Bildgebung in Luft geeignet ist. Verwenden Sie zum Beispiel AFM-Sonden, wie in der Werkstoffliste aufgeführt . Hier (abhängig von AFM), fixieren Sie die Spitze in der Halterung unter einer Klemme durch Anziehen der Klemmschraube (fingerdicht). Setzen Sie den Spitzenhalter in den AFM Messkopf ein. Legen Sie den Kopf auf den Rücken für diesen Schritt.
- Legen Sie den AFM-Messkopf auf die Probe. Details hängen vom AFM-Modell ab. Hier, mAke sicher, der Kopf steht stabil mit den Beinen in den Bühneneinbauten. Vergewissern Sie sich, dass sich der Glimmer auf der Bühne befindet, wo die AFM-Spitze direkt darüber liegt. Mikrometer-Schrauben auf der rechten Seite der Bühne ermöglichen eine feine Positionierung der Probe.
- Richten Sie den AFM-Laser auf der Rückseite des Cantilevers für eine optimale Signalstärke aus dem positionsempfindlichen Photodetektor aus, auf den der Laser reflektiert wird. Details hängen vom AFM-Modell ab.
- Hier drehen Sie die Räder an der rechten und hinteren Seite des AFM-Messkopfes, um die x- und y-Positionen des AFM-Lasers einzustellen, um ihn zentral auf das Ende des Cantilevers zu richten. Beobachten Sie das Reflexionssignal im AFM-Videofenster (falls vorhanden, hier: drücken Sie das Kamerasymbol und wählen Sie den Eingang: Svideo).
- Einmal grob positioniert, optimiere das Detektorsummensignal (Summe in Summen- und Defduktionszähler-Fenster in AFM-Software) durch Feinabstimmung der Laserposition mit den beiden Rädern (am Ende des Cantilevers bleiben, die Summe signaL hängt von der AFM und Cantilever Typ, hier: Ziel für Summe> 5).
- Nullen Sie das Differenzsignal von den oberen und unteren Dioden des Detektorarrays (hier: Ablenksignal im Summen- und Ablenkzähler-Fenster), indem Sie die AFM-Laserreflexion auf das Detektorzentrum richten (hier: drehen Sie das Rad auf der linken Seite der AFM-Messung Kopf).
HINWEIS: Abweichungen von Null geben dann die Auslenkung des Cantilevers aufgrund von Oberflächenwechselwirkungen an, die vom AFM in Höheninformationen übersetzt werden. - Bestimmen Sie die Cantilever-Resonanzfrequenz durch eine Frequenzstimmung, die in die AFM-Software implementiert ist (hier: Befehlsautomatisierung im Master Panel / Tune-Fenster). Wählen Sie eine Amplitude, die 1 V Eingang für das Piezo entspricht, das die Cantilever-Oszillation antreibt. Setzen Sie die Oszillationsfrequenz auf etwas (5%) niedriger als die Resonanzfrequenz und null die Phase der Oszillation.
- Nähern Sie sich die Spitze an die Probenoberfläche mit dem rohen Eingriff modE (hier: Befehl In Master Panel Fenster einschalten) bis die Schutzeinstellung (Sollwert) erreicht ist. Verwenden Sie ~ 2% Schneiden der freien Pegel-Oszillationsamplitude als Sollwert (hier geben Sie Set Point 980 mV im Master Panel ein).
- Fassen Sie die AFM-Spitze mit der Probenoberfläche ein, indem Sie den Sollwert mit der AFM-Software absenken. Ziel für die Abstoßungs-Bildgebung mit der Cantilever-Oszillationsphase (Phase in Summe und Deflection Meter-Fenster) knapp unterhalb der freien Pegelphase (vor dem Eingriff, hier typischerweise ~ 70). Verwenden Sie hier typische Endpunkte in der Größenordnung von 70-80% der freien Pegelamplitude (1 V).
- Vor dem Scannen wählen Sie die Signale für die Aufnahme im Master Channel Panel. Wähle Höhe (Ht) und Amplitude (Am).
- Beginne das Sample Scanning (hier: Befehl Do Scan im Master Panel Fenster). Verwenden Sie eine Scan-Geschwindigkeit von zB 2,5 μm / s (Befehl Scan Speed in Master Panel) auf Bildflächen von 4 μm x 4 μm oder 8 μm x 8 μm (geben Sie Scan Size in Master Pane einL) mit Pixelauflösungen von 2.048 oder 4.096 (Scan Points und Scan Lines im Master Panel).
- Speichern Sie die Bilddatei (Befehl Bild speichern im Master Panel) ohne passende Änderungen (wählen Sie im Master Channel Panel / Save Plane Fit keine).
- Zur weiteren Analyse verarbeiten Sie das gespeicherte Bild. Laden Sie das Bild (Befehl Durchsuchen gespeicherte Daten im Menü AFM-Analyse). Öffnen Sie das Modify Panel (drücken Sie M im oberen Teil des Bildes). Tragen Sie eine Ebene in x und y Dimensionen auf die Höhe Bild (Erweiterung HtR) (Befehl XY in Planefit Fenster in Modify Panel, wählen Sie Planefit Auftrag 3). Dann flach das Bild (Befehl Flatten in Flatten Fenster in Modify Panel) wählen Flatten Order 3.
- Exportieren Sie das Bild als TIFF-Datei (drücken Sie Befehle im oberen Bildbereich, wählen Sie TIFF Export 2x entsprechend 2,048 Pixel Auflösung) für weitere Analysen.
3. AFM-Analyse
- Protein komplexes Volumen
- Vor-Selektieren relevante Protein-DNA-Komplexe durch direkte visuelle Inspektion der Bilder in der AFM-Software unter Verwendung eines geeigneten Farbschemas, um unterschiedliche Erscheinungen unterschiedlicher Größenkomplexe zu maximieren (siehe verschiedene Farben und Größen verschiedener Komplexe in Abbildung 2 , hier: Farbschema SeaLandAndFire in Die AFMsoftware). Für XPD-Proben gehörten sowohl die XPD / p44-DNA als auch XPD-DNA und p44-DNA mit deutlich unterschiedlichen Größen aufgrund der relativ großen, unterschiedlichen Molekülmassen von XPD (~ 95 kDa) und p44 (~ 40 kDa) .
- Messen Sie die Volumina der einzelnen Proteinspitzen auf der DNA, um den komplexen Typ zu überprüfen. Dies kann mit einer anderen Bildsoftware erreicht werden (hier: das Schnittwerkzeug der AFM-Software).
- Messen Sie die Höhe (h) und den Durchmesser (d) der Protein-Peak-Abschnitte mit den Abschnitt Fenster-Cursor. Messen Sie den Durchmesser in der Nähe der Basis des Partikelabschnitts.
- Bestimmen Sie das Volumen (V) der ErbseKs mit einfachen mathematischen Modellen, zB mit der folgenden Formel, die auf einem sphärischen Cap-Modell basiert:
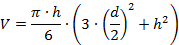
- Übersetzen der gemessenen Volumen in eine ungefähre Proteinmolekülmasse.
- Zuerst kalibriere das AFM-System für die Volumen-zu-Molekulargewichts- (MW) -Umwandlung unter Verwendung einer Reihe von Proteinen mit bekanntem Molekulargewicht (hier wurden insgesamt 12 Experimente mit jeweils 25 bis 851 Datenpunkten an 5 verschiedenen Proteinen in monomeren, dimeren, , Trimere oder tetramere Zustände) 4 , 19 , 20 . Ablagerungs- und Bildproteine (wie oben in 2.2 und 2.3 beschrieben) und messen ihre Volumina wie oben beschrieben (3.1.2). Zeichnen Sie die Bände über ihre bekannten Molekulargewichte. Der daraus resultierende Graph zeigt eine lineare Beziehung zwischen V und MW, die Gleichung, für die man erhalten kannEine Linie passt zu den Daten. Für das hier verwendete AFM-System wurde die folgende Beziehung erhalten: 19 :
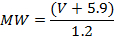
HINWEIS: Dieser Schritt muss nicht vor jeder Messung wiederholt werden, sondern nur einmal durchgeführt. Die Lautstärke zur MW-Kalibrierung kann auf Bilder angewendet werden, die unter ähnlichen Bildgebungsbedingungen unter Verwendung von AFM-Sonden mit leicht variierenden Durchmessern erhalten wurden. - Basierend auf ihren gemessenen Volumina (3.1.2) und der V-zu-MW-Kalibrierung (3.1.3.1), bestimmen die ungefähren MW der Proteinkomplexe 20 , die Informationen über ihren molekularen Gehalt liefern. ZB wurden für XPD / p44-Proben ~ 50 kDa, ~ 100 kDa und ~ 140 kDa erhalten, im Einklang mit p44-DNA-Komplexen (oder Peaks, die durch bloße DNA-Überstrukturen verursacht wurden), XPD-Peaks und XPD / p44-Peaks .
- Zuerst kalibriere das AFM-System für die Volumen-zu-Molekulargewichts- (MW) -Umwandlung unter Verwendung einer Reihe von Proteinen mit bekanntem Molekulargewicht (hier wurden insgesamt 12 Experimente mit jeweils 25 bis 851 Datenpunkten an 5 verschiedenen Proteinen in monomeren, dimeren, , Trimere oder tetramere Zustände) 4 , 19 , 20 . Ablagerungs- und Bildproteine (wie oben in 2.2 und 2.3 beschrieben) und messen ihre Volumina wie oben beschrieben (3.1.2). Zeichnen Sie die Bände über ihre bekannten Molekulargewichte. Der daraus resultierende Graph zeigt eine lineare Beziehung zwischen V und MW, die Gleichung, für die man erhalten kannEine Linie passt zu den Daten. Für das hier verwendete AFM-System wurde die folgende Beziehung erhalten: 19 :
- Protein komplexe Positionen auf DNA
- Bestimmen Sie die DNAFragmentlängen
- Trace die DNA-Fragmente in den AFM-Bildern, zB mit der Freihand-Line-Funktion einer geeigneten Bildanalyse-Software (siehe zB Materialtabelle) und messen die Länge der Linie.
HINWEIS: Ausschluss von DNA-Aggregaten sowie von den Bildrändern abgeschnittene Fragmente. - Bin und zeichne die DNA-Längen aus dem gesamten Experiment in einem Histogramm, mit geeigneten Datenanalyse und Grafik-Software (siehe zB Tabelle der Materialien ).
- Passen Sie die Längenverteilungen mit einer Gaußschen Kurve in die Datenanalyse und Grafik-Software, um die Länge der DNA-Fragmente zu bestimmen. Um die Position der eingefügten DNA-Zielstelle zu kennen (siehe 1.1), enthalten nur DNA-Fragmente mit der korrekten Länge innerhalb von zwei Standardabweichungen vom Zentrum der Gauss-Kurve (y = y 0 + z exp (-2 (xx c ) 2 / w 2 ), wobei z ein Normfaktor ist und x c und w die Mitte undVolle maximale halbe Breite des Gaußschen) in weiteren Analysen.
HINWEIS: Die DNA-Länge im Zentrum der Gauss-Kurve muss nahe der theoretischen Länge des DNA-Fragments liegen (berechnet unter Verwendung von 0,34 nm / bp). Längen von bis zu 10% kürzer als der theoretische Wert sind typisch und werden wahrscheinlich durch AFM-Auflösung begrenzt.
- Trace die DNA-Fragmente in den AFM-Bildern, zB mit der Freihand-Line-Funktion einer geeigneten Bildanalyse-Software (siehe zB Materialtabelle) und messen die Länge der Linie.
- Bestimmen Sie Positionen von Proteinpeaks auf DNA-Substraten.
- Messen Sie den Abstand der Proteinspitzen vom näheren DNA-Fragmentende, wie in 3.2.1.1 beschrieben, und teilen Sie die Gesamt-DNA-Länge, um Abstände in Einheiten der Fraktion der DNA-Länge zu erhalten.
- Bin und zeichnen Sie die gemessenen Abstände in einem Histogramm mit einer geeigneten Datenanalyse und Grafik-Software (siehe zB Abbildung 3 ). Als Faustregel gilt eine Bin-Größe, die ca. √n Bins für n Datenpunkte gibt.
HINWEIS: Da DNA-Enden hier nicht unterschieden werden können, nur auf Fraktion der DNA-Länge 0,5 (Zentrum der DNA-Fraktion)). Da die DNA-Endbindung nicht der Fokus der Studie ist, schließen die DNA-Enden aus den Analysen aus, indem sie die Positionsdaten leicht nach der Position 0 beginnen ( z. B. hier: Binning aus einem DNA-Längenanteil von 0,02).
- Bestimmen Sie die Zielort-Spezifität aus Protein-Komplex-Positionsverteilungen.
- Setzen Sie die maximale (verbesserte Bindungsereignisse) in der Positionsverteilung mit einer Gauss-Kurve wie in 3.2.1.3, aber footed auf der Höhe der Hintergrundbindung (siehe Abbildung 3 ).
- Berechnen Sie die Spezifität S für die spezifische Stelle gegenüber dem unspezifischen DNA-Hintergrund (Proteinmoleküle, die an unspezifische DNA-Stellen gebunden sind) mit der folgenden Formel 21
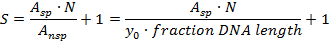
A sp : Anzahl der spezifischen Komplexe (Bereich unter der Gaußschen Kurve)
A nsp : Anzahl der unspezifischen Komplexe (Bereich des Hintergrundes, Dh (Fraktions-DNA-Länge) x (durchschnittliche Hintergrundhöhe y 0 ); Der Bruchteil der hier abgedeckten DNA-Länge beträgt 0,48 für ein Histogramm beginnend bei 0,02 DNA-Länge)
N: Anzahl möglicher DNA-Bindungsstellen (hier: N = 914 ohne DNA-Enden)
- Bestimmen Sie die DNAFragmentlängen
- DNA-Biegewinkel
- Mit dem Winkelwerkzeug in einer geeigneten Bildanalyse-Software messen Sie den Winkel β zwischen zwei Linien, die zentral entlang des DNA-Rückgrats liegen und am Proteinpeak zentrieren (siehe zB Einfügung in Abbildung 4 ). Messen Sie die Winkel für eine statistisch relevante Anzahl von Protein-DNA-Komplexen (> 50, idealerweise> 100) 2 , 3 , 5 .
HINWEIS: Der DNA-Biegewinkel ist definiert als 180 ° -β 2 , 3 , 5 . - Mit Datenanalyse und Grafik-Software(Siehe Tabelle der Materialien) ein Biegewinkelverteilungshistogramm durch Binning der DNA-Biegewinkel erzeugen.
- Die Biegewinkelverteilung mit einer Gauss-Kurve anpassen. Wenn mehr als ein Maximum in der Verteilung sichtbar ist, wählen Sie mehrere Peak Gauss fit. Die Mitte (n) der Gaußschen Kurve (n) gibt (n) den durchschnittlichen Biegewinkelzustand der jeweiligen Spezies.
- Wenn eine Verschiebung des Biegewinkels (s) (Maxima der Gaußschen Passung (en)) für Verteilungen, z. B. von verschiedenen Proteinvarianten oder verschiedenen Proteinkomplexarten, offensichtlich ist, wenden Sie einen Schülertest an, um das Signifikanzniveau des Änderungen.
- Mit dem Winkelwerkzeug in einer geeigneten Bildanalyse-Software messen Sie den Winkel β zwischen zwei Linien, die zentral entlang des DNA-Rückgrats liegen und am Proteinpeak zentrieren (siehe zB Einfügung in Abbildung 4 ). Messen Sie die Winkel für eine statistisch relevante Anzahl von Protein-DNA-Komplexen (> 50, idealerweise> 100) 2 , 3 , 5 .
Ergebnisse
Unterscheidung zwischen verschiedenen komplexen Typen auf der Basis von Protein komplexen Volumina
Die Helicaseaktivität der prokaryotischen NER-Helicase UvrB wird durch DNA-Bindung 22 , 23 stimuliert. UvrB benötigt eine ungepaarte Region in der DNA (eine DNA-Blase), um korrekt auf einen der beiden einzelnen ssDNA-Stränge zu laden. In vivo wird diese DNA-Struk...
Diskussion
AFM statistische Analysen von Bindungspositionen von Proteinen auf langen DNA-Fragmenten, die spezifische Zielstellen enthalten, können interessante Details über die speziellen Strategien des Proteins zeigen, um diese Stellen 2 , 3 , 4 , 5 , 6 zu erkennen. Um die daraus resultierenden Positionsverteilungen zu interpretieren, müssen die Positionen der Targe...
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
PUC19N, CPD-haltige Oligonukleotide und p44 wurden freundlicherweise von Samuel Wilson, Korbinian Heil und Thomas Carell und Gudrun Michels bzw. Caroline Kisker bereitgestellt. Diese Arbeit wurde durch Stipendien der Deutschen Forschungsgemeinschaft (DFG) FZ82 und TE-671/4 an IT unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Molecular Force Probe (MFP) 3D | Asylum Research | N/A | atomic force microscope (AFM) |
| Precision 390 | DELL | N/A | computer |
| ThermoMixer and 1.5 mL block | Eppendorf | 5382000015 | heat block for DNA preparation |
| Rotilabo Block-Heater H 250 & blocks for 0.5 mL tubes | Carl Roth GmbH | Y264.1 & Y267.1 | heat block for protein-DNA incubations |
| Mini-Sub Cell GT | Bio-Rad Laboratories GmbH | 1704467 | electrophoresis chamber with gel caster and power supply |
| Power Pac Basic | Bio-Rad Laboratories GmbH | 1645050 | electrophoresis power supply |
| Centifuge 5415 D with rotor | Eppendorf | 2262120-3 | table centrifuge |
| Ultra-Lum electronic UV transillumonator MEB-15 | Ultralum | 900-1322-02 | UV irradiation table |
| NanoDrop ND-1000 | VWR International / PEQLAB Biotechnologie GmbH | N/A | UV spectrophotometer |
| TKAX-CAD with 0.2 μm capsule filter | Unity Lab Services | N/A | water deionization and filter unit |
| Name | Company | Catalog number | Comments |
| Software | |||
| MFP software on Igor Pro | Asylum Research | N/A | AFM software |
| ImageJ (open source Java image processing) | NIH Image | N/A | Image analysis software |
| Excel (Microsoft Office) | Microsoft Corporation | N/A | data analysis software |
| Origin9 / Origin2016 | OriginLab Corporation | N/A | statistical data analysis and graphing software |
| Name | Company | Catalog number | Comments |
| Material | |||
| OMCL-AC240TS | Olympus | OMCL-AC240TS | AFM cantilevers |
| grade V-5 muscovite | SPI Supplies | 1805 | mica sheets |
| Amicon Ultra 0.5 mL 50k Ultracell | Millipore Ireland Ltd. | UFC505096 | centrifuge filters |
| NucleoSpin Extract II | Macherey-Nagel GmbH | 740 609.250 | Agarose gel extraction kit |
| Rotilabo cellulose paper type 111A | Carl Roth GmbH | AP59.1 | AFM deposition blotting paper |
| Anatop 25 (0.02 μm) | Whatman GmbH | 6809-2102 | syringe filter |
| SSpI, BspQI | New England Biolabs (NEB) | R0132, R0712 | restriction enzymes for DNA substrate preparation |
| XhoI, BglII | R0146, R0144 | restriction enzymes for DNA preparation controls | |
| nicking restriction enzyme Nt.BstNBI | New England Biolabs (NEB) | R0607 | nickase |
| T4 DNA ligase | New England Biolabs (NEB) | M0202S | Ligase |
| Tris, HEPES | Carl Roth GmbH | 4855, 9105 | buffer chemicals |
| NaCl, MgCl2, KCl, MgAcetate | Carl Roth GmbH | 3957, HN03, HN02, P026 | salt chemicals |
| NaAc | Sigma-Aldrich Chemie GmbH | 32318 | salt chemicals |
| DTT, TCEP, EDTA | 6908, HN95, 8040 | chemicals/reagents | |
| agarose, acetic acid, HCl | Carl Roth GmbH | 2267, 3738, K025 | reagents |
| ATP | Carl Roth GmbH | K054 | nucleotides |
| oligonucleotide #1 in Table 1 | Biomers | custom | complementary DNA oligonucleotide |
| oligonucleotides #2, #3, and #6 in Table 1 | Integrated DNA Technologies (IDT) | custom | fluorescein containing oligonucleotides |
| oligonucleotides #4 and #5 in Table 1 | private (available from e.g. TriLink or GlenResearch) | CPD containing oligonucleotides | |
| SafeSeal reaction tube 0.5 mL and 1.5 mL | Sarstedt | 72.704 and 72.706 | incubation tubes |
| GeneRuler 1 kb | Thermo Scientific | SM0311 | DNA ladder |
| 6x concentrate gel loading dye purple | New England Biolabs (NEB) | 51406 | DNA loading dye |
| Midori Green | Nippon Genetics Europe GmbH | 999MG28055 | DNA stain |
Referenzen
- Janicijevic, A., Ristic, D., Wyman, C. The molecular machines of DNA repair: scanning force microscopy analysis of their architecture. J. Microsc. 212 (3), 264-272 (2003).
- Wang, H., et al. DNA bending and unbending by MutS govern mismatch recognition and specificity. Proc. Natl. Acad. Sci. USA. 100 (25), 14822-14827 (2003).
- Tessmer, I., et al. Mechanism of MutS searching for DNA mismatches and signaling repair. J. Biol. Chem. 283 (52), 36646-36654 (2008).
- Wagner, K., Moolenaar, G., van Noort, J., Goosen, N. Single-molecule analysis reveals two separate DNA-binding domains in the Escherichia coli UvrA dimer. Nucleic Acids Res. 37 (6), 1962-1972 (2009).
- Buechner, C. N., et al. Strand-specific recognition of DNA damages by XPD provides insights into nucleotide excision repair substrate versatility. J Biol. Chem. 289 (6), 3613-3624 (2014).
- Van der Linden, E., Sanchez, H., Kinoshita, E., Kanaar, R., Wyman, C. RAD50 and NBS1 form a stable complex functional in DNA binding and tethering. Nucleic Acids Res. 37 (5), 1580-1588 (2009).
- Fuentes-Perez, M. E., Dillingham, M., Moreno-Herrero, F. AFM volumetric methods for the characterization of proteins and nucleic acids. Methods. 60, 113-121 (2013).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Miyagi, A., Lyubchenko, Y. L. Specificity of binding of single-stranded DNA-binding protein to its target. Biochemistry. 51, 1500-1509 (2012).
- Wirth, N., et al. Conservation and Divergence in Nucleotide Excision Repair Lesion Recognition. J. Biol. Chem. 291 (36), 18932-18946 (2016).
- Kuper, J., Kisker, C. Damage recognition in nucleotide excision DNA repair. Curr. Opin. Struct. Biol. 22, 88-93 (2012).
- Buechner, C. N., Tessmer, I. DNA substrate preparation for atomic force microscopy studies of protein-DNA interactions. J. Mol. Recognit. 26 (12), 605-617 (2013).
- Sun, Z., Tan, H. Y., Bianco, P. R., Lyubchenko, Y. L. Remodeling of RecG Helicase at the DNA Replication Fork by SSB Protein. Sci. Rep. 5, 9625 (2015).
- Billingsley, D. J., Bonass, W. A., Crampton, N., Kirkham, J., Thomson, N. H. Single-molecule studies of DNA transcription using atomic force microscopy. Phys. Biol. 9 (2), 021001 (2012).
- Maurer, S., Fritz, J., Muskhelishvili, G., Travers, A. RNA polymerase and an activator form discrete subcomplexes in a transcription initiation complex. EMBO J. 25 (16), 3784-3790 (2006).
- Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Theis, K., Chen, P. J., Skorvaga, M., Van Houten, B., Kisker, C. Crystal structure of UvrB, a DNA helicase adapted for nucleotide excision repair. EMBO J. 18 (24), 6899-6907 (1999).
- Kuper, J., et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 12 (9), e1001954 (2014).
- Shlyakhtenko, L. S., et al. Silatrane-based surface chemistry for immobilization of DNA, protein-DNA complexes and other biological materials. Ultramicroscopy. 97, 279-287 (2003).
- Roth, H. M., et al. XPB helicase regulates DNA incision by the Thermoplasma acidophilum endonuclease Bax1. DNA Repair. 11 (3), 286-293 (2012).
- Ratcliff, G. C., Erie, D. A. A Novel Single-Molecule Study to Determine Protein-Protein Association Constants. J. Am. Chem. Soc. 123 (24), 5632-5635 (2001).
- Yang, Y., Sass, L. E., Du, C., Hsieh, P., Erie, D. A. Determination of protein-DNA binding constants and specificities from statistical analyses of single molecules: MutS-DNA interactions. Nucleic Acids Res. 33 (13), 4322-4334 (2005).
- Caron, P. R., Grossman, L. Involvement of a cryptic ATPase activity of UvrB and its proteolysis product, UvrB* in DNA repair. Nucleic Acids Res. 16 (22), 10891-10902 (1988).
- Wang, H., et al. UvrB domain 4, an autoinhibitory gate for regulation of DNA binding and ATPase activity. J. Biol. Chem. 281 (22), 15227-15237 (2006).
- Chammas, O., Billingsley, D. J., Bonass, W. A., Thomson, N. H. Single-stranded DNA loops as fiducial markers for exploring DNA-protein interactions in single molecule imaging. Methods. 60 (2), 122-130 (2013).
- Truglio, J. J., et al. Structural basis for DNA recognition and processing by UvrB. Nat. Struct. Mol. Biol. 13 (4), 360-364 (2006).
- Kuper, J., Wolski, S. C., Michels, G., Kisker, C. Functional and structural studies of the nucleotide excision repair helicase XPD suggest a polarity for DNA translocation. EMBO J. 31 (2), 494-502 (2012).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Goosen, N. The presence of two UvrB subunits in the UvrAB complex ensures damage detection in both DNA strands. EMBO J. 21 (15), 4196-4205 (2002).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Hoeijmakers, J. H., Goosen, N. Architecture of nucleotide excision repair complexes: DNA is wrapped by UvrB before and after damage recognition. EMBO J. 20 (3), 601-611 (2001).
- Moolenaar, G. F., et al. The Role of ATP Binding and Hydrolysis by UvrB during Nucleotide Excision Repair. J. Biol. Chem. 275, 8044-8050 (2000).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten