需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
原子力显微镜检查DNA损伤识别核苷酸切除修复
摘要
Here, the study of different DNA lesion recognition approaches via single molecule AFM imaging is demonstrated with the nucleotide excision repair system as an example. The procedures of DNA and protein sample preparations and experimental as well as analytical details for the AFM experiments are described.
摘要
AFM imaging is a powerful technique for the study of protein-DNA interactions. This single molecule method allows the simultaneous resolution of different molecules and molecular assemblies in a heterogeneous sample. In the particular context of DNA interacting protein systems, different protein complex forms and their corresponding binding positions on target sites containing DNA fragments can thus be distinguished. Here, an application of AFM to the study of DNA lesion recognition in the prokaryotic and eukaryotic nucleotide excision DNA repair (NER) systems is presented. The procedures of DNA and protein sample preparations are described and experimental as well as analytical details of the experiments are provided. The data allow important conclusions on the strategies by which target site verification may be achieved by the NER proteins. Interestingly, they indicate different approaches of lesion recognition and identification for the eukaryotic NER system, depending on the type of lesion. Furthermore, distinct structural properties of the two different helicases involved in prokaryotic and eukaryotic NER result in and explain the different strategies observed for these two systems. Importantly, these experimental and analytical approaches can be applied not only to the study of DNA repair but also very similarly to other DNA interacting protein systems such as those involved in replication or transcription processes.
引言
原子力显微镜(AFM)是分析蛋白质 - DNA相互作用1,2,3,4,5,6,7,8,9的有力技术。它只需要少量的样品材料,以单分子水平的分辨率直接观察异质样品。蛋白质的不同构象或寡聚状态可能产生异质性。特别地,在蛋白质-DNA样品的上下文中,蛋白质复合物可以显示通常的DNA结合诱导的不同化学计量学和/或构象,或者结合DNA中的特定靶位点。不均匀的样品也可能含有两种(或多种)不同种类的蛋白质和不同的蛋白质复合物形式( 例如 ,仅由一种类型的蛋白质与异源复合物组成)可能与DNA不同的相互作用。这里讨论的研究利用空气中的AFM成像技术,将DNA修复蛋白的静态,干燥样品结合到长(〜900个碱基对,bp)的含有病变的DNA片段,其代表这些蛋白质的靶标。 AFM的高分子分辨率允许区分不同类型的蛋白质复合物并确定蛋白质在DNA片段上的结合位置。重要的是,将损伤引入到确定位置的DNA底物中。因为DNA中的病变部位的位置是已知的,所以与DNA结合的蛋白质的分布提供了(不同的)蛋白质复合物的(不同的)病变识别特性的洞察, 例如 ,它们如何识别特定类型的病变(比较对未损伤的DNA) 2,3 , 4,5,6 。他们在DNA上的位置也可以区分特异性结合病变的蛋白质复合物和DNA上非特异性结合的复合物。这些不同复杂类型(复合物特异性结合病变与非特异性复合物)的分离表征可以揭示在靶位点鉴定中诱导的复合物中潜在的构象变化。
在这里聚集的DNA修复蛋白是解旋酶,其负责核苷酸切除修复(NER)途径中的病变识别。在细菌中,NER通过蛋白质UvrA,UvrB和UvrC实现。 UvrA负责UvaA 2 / UvrB 2 DNA扫描复合物中的初始病变感测。通过UvrB的病变验证,该复合物转化为在病变部位结合的单体UvrB,然后该特异性复合物可以招募p核型NER核酸内切酶UvrC。 UvrC切除含有病变的短(12-13nt)单链DNA(ssDNA)。然后通过DNA聚合酶重新填充缺失的片段。最后,DNA连接酶用原始DNA 9,10密封新合成的片段。在真核生物中,NER级联的大多数蛋白质是大的多聚体转录因子II H(TFIIH)复合物的一部分。 通过三聚体CEN2-XPC-HR23B复合物进行初始病变感染后,TFIIH被引入DNA靶位点。当复合物中的XPD验证NER靶病变的存在时,招募真核NER核酸内切酶XPG和XPF以消除含有病变9,10的短(24-32nt)片段的ssDNA。具体来说,研究了分离自原核和真核NER的解旋酶UvrB和XPD。这些解剖器需要不配对的区域DNA(DNA气泡)连接到两条DNA单链之一上,随后沿着ATP水解促进的该链转移。除DNA损伤之外,DNA泡也被引入作为蛋白质负载位点的底物中。
以前已经描述了用于制备特定病变DNA底物的方法11 。它需要一个循环DNA构建体(质粒),其具有用于切口酶的两个紧密间隔的限制性位点。在本研究的上下文中,使用质粒pUC19N(2729bp)(由S.Wilson的实验室,NIEHS创建)。该质粒含有三个紧密间隔的限制性位点,用于构建48个核苷酸(nt)延伸的切口酶Nt.BstNBI。与切口酶孵育后,可以除去这些位点之间的ssDNA片段,并用含有任何靶标特征的寡核苷酸代替。在每个步骤之后,通过琼脂糖凝胶测试完全酶消化电泳。由于与原来的超螺旋质粒相比,镍环状DNA的电泳迁移率较低,因此可以区别。通过用特异性底物寡核苷酸替代除去的拉伸的替代物可以通过限制酶进行消化来进行评估,所述限制酶将底物专门在切口之间的区域内切割。因此,通过酶对圆形质粒的线性化将被抑制,并且在插入特异性寡核苷酸后恢复。最后,两个核酸内切酶限制性位点(理想的是单个切割器)允许产生线性DNA底物,其长度根据需要和特定的靶位点在确定的位置以及与病变相距一定距离的DNA气泡'或3'方向。
可以通过 AFM成像来检查NER解旋酶对病变的识别。在t处的解旋酶失调的DNA易位他的病变部位作为DNA上的蛋白质位置分布的峰值可见,表明病变识别。因为这些解旋酶的DNA易位进一步是定向的,具有5'至3'极性,病变识别对加载位点的位置(病毒上游或下游的DNA气泡)的依赖性还指示是否优先识别病变在易位或相对的非易位的ssDNA链5,9上 。在以下部分中,将介绍所使用的方法,并简要讨论这些实验的主要发现。重要的是,类似于本文所示的DNA修复的示范性工作,AFM成像可以应用于不同DNA相互作用系统的研究,例如DNA复制或转录8,12,13,14 。
研究方案
样品制备
- DNA底物的制备11
- 在质粒中产生ssDNA间隙
- 在具有合适的切口酶(这里为Nt.BstNBI)的反应管中完全消化质粒样品(这里:修饰的pUC19,pUC19N),然后使用根据制造商的方案的条件进行酶热灭活(参见图1的示意图介绍)。以〜50μL和〜500nM质粒开始,得到足够的产率。
- 通过琼脂糖凝胶电泳验证稀释样品(〜20nM) 15质粒切口。戴上手套以防止用于DNA可视化的DNA结合染料。
注意:不同的电泳迁移率允许区分切口(松弛)和超螺旋环状质粒DNA( 图1 )。 - 从质粒中除去切割的ssDNA片段(切口位点之间)与互补寡核苷酸( 表1中的寡核苷酸1 )的〜10倍过量孵育,在加热块中的寡核苷酸的熔解温度(对应于pUC19N为68℃)以300rpm振荡30分钟( 图1 )。
- 使用50kDa分子量截留(MWCO)过滤器,在距离离心机中以10000xg离心10分钟( 图1 ),将有间隙的质粒与较小的DNA片段分开。为了从过滤器中提取浓缩的DNA,将过滤器反转并插入新的1.5 mL反应管中。以1,000 x g离心3分钟。
- 将所得的浓缩DNA样品用去离子水,过滤水补充至500μL,再加入约5倍过量的互补寡核苷酸,并重复步骤1.1.1.3和1.1.1.4至少3次。
- 通过使用条件通过与合适的限制酶(这里为XhoI或BglII)温育来测试DNA完全间隔根据制造商的描述。使用稀释(切口与间隙)DNA样品(〜20 nM)。使用琼脂糖凝胶电泳来区分线性化质粒(不含ssDNA间隙)和使用缺口DNA作为阳性对照的非切割DNA(带隙DNA)(包括用于参考的DNA梯形图, 图1 )。戴手套。
- 用修饰的ssDNA寡核苷酸填充间隙
- 通过与约25倍过量的含有特定目标位点的5'磷酸化寡核苷酸一起孵育,获得缺口的质粒,在〜45℃孵育4小时( 图1 )。在这里,使用含有病变的48nt ssDNA,荧光素加合胸腺嘧啶或环丁烷嘧啶二聚体(CPD),另外还使用短(8nt)非互补序列产生DNA气泡( 参见表1 )。
- 通过孵育将退火的插入物与质粒共价连接根据制造商的方案在室温下用T4DNA连接酶过夜( 图1 )。对于该反应,加入含有浓缩缓冲液储备溶液的ATP以产生用于连接酶的合适的缓冲液条件( 例如 ,使用UvrB反应缓冲液:50mM Tris-HCl pH7.5,10mM MgCl 2,50mM KCl,5mM DTT,1mM ATP)。
- 通过根据制造商的方案,用合适的限制酶(与1.1.1.6相同)消化稀释的样品,然后进行琼脂糖凝胶电泳(如1.1.1.6, 图1中所示 ),测试将特异性寡核苷酸插入缺口质粒中)。
- 线性DNA底物的制备
- 使用制造商推荐的条件( 图1 ),用限制酶(理想情况下,使用质粒中的单个限制性位点)纯化修饰的质粒DNA。此步骤产生包含的线性片段在所定义的位置插入修改。在这里,分别在插入片段上游和下游的位置613和255bp处使用SspI和BspQI切割,导致在DNA长度的〜30%处含有特异性病变位点的916bp DNA片段。
注意:对于如本文所述的AFM成像实验,长度在约200bp至约2,000bp之间的DNA片段是合适的底物。 - 通过琼脂糖凝胶电泳和使用商业试剂盒凝胶提取分离靶片段。戴手套保护。
- 任选地,用手术刀切割琼脂糖凝胶以从含有待纯化的浓缩DNA的泳道分离DNA梯子和稀释的样品泳道(前两个泳道)。仅将这部分凝胶放置在紫外线台上,才能将前两条通道的凝胶部分暴露于紫外线照射。
- 用解剖刀切出与选择片段对应的乐队。将两个凝胶部分重新组合(离开UV标签乐)。使用来自稀释样品的切除带的位置作为所需DNA底物带的位置的取向。通过切割稍微更宽的切片比稀释对照更高的浓度。
注意:因为这里,通过这种方法在DNA底物中仔细地避免了紫外线损伤的识别, 通过 UV照射引入额外的紫外线损伤。
- 用解剖刀切出与选择片段对应的乐队。将两个凝胶部分重新组合(离开UV标签乐)。使用来自稀释样品的切除带的位置作为所需DNA底物带的位置的取向。通过切割稍微更宽的切片比稀释对照更高的浓度。
- 用紫外 - 可见分光光度计测定的260nm处吸收的DNA浓度c ,使用Lambert-Beer定律,双链DNA(dsDNA)的平均摩尔消光系数为ε260nm〜6,700M - 1cm -1 ,每bp:
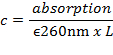
其中L是路径长度(测量室长度,通常为1cm)。
- 使用制造商推荐的条件( 图1 ),用限制酶(理想情况下,使用质粒中的单个限制性位点)纯化修饰的质粒DNA。此步骤产生包含的线性片段在所定义的位置插入修改。在这里,分别在插入片段上游和下游的位置613和255bp处使用SspI和BspQI切割,导致在DNA长度的〜30%处含有特异性病变位点的916bp DNA片段。
- 在质粒中产生ssDNA间隙
- 蛋白质的表达和纯化
- 在大肠杆菌中重组表达UvrB,并通过标准几丁质珠亲和力15和尺寸排阻层析15纯化蛋白质,如前所述。
注意:来自芽孢杆菌的uvrB基因已经克隆到pTYB1载体中。 - 在大肠杆菌中表达 XPD,并通过标准镍IDA亲和力15和尺寸排阻色谱法15纯化蛋白质,然后如前所述17进行阴离子交换层析15 。
注意: 嗜热毛霉菌色素性色素蛋白D蛋白(XPD)的基因已经克隆到pBADM11载体中。 C.嗜热菌 p44是Caroline Kisker实验室的一种礼物,表达和纯化如17所述。
- 在大肠杆菌中重组表达UvrB,并通过标准几丁质珠亲和力15和尺寸排阻层析15纯化蛋白质,如前所述。
AFM实验
- 样品制备
- 任选地,在热块中将DNA底物在65℃预孵育10分钟以除去在冰箱中储存期间可能形成的潜在的微盐晶体。
- 以10倍浓度(10倍缓冲液)制备反应缓冲液。
注意:1x浓度的XPD反应缓冲液含有20mM Tris-HCl pH7.5,10mM KCl,5mM MgCl 2,1mM TCEP,2mM ATP; 1x UvrB反应缓冲液含有50mM Tris-HCl pH7.5,50mM KCl,10mM MgCl 2,5mM DTT和1mM ATP。 - 在合适的孵育条件下,以更高的浓度预孵育蛋白质以增强复合物的形成。将1x蛋白反应缓冲液中的少量体积( 例如 1μL)的各个蛋白质预稀释至所需浓度,并将小体积( 例如 1μL)的t他单独的蛋白质溶液在0.5 mL反应管中。
- 将管放置在高于室温的孵育温度的热块中。根据预期的复合化学计量选择浓度比,即等摩尔或相应的浓度。在这里,在37℃下以10μM的速度孵育1μLXPD(1×XPD反应缓冲液中20μM)和1μLp44(1x XPD反应缓冲液中20μM)10分钟。
- 在0.5 mL反应管中,将蛋白质反应缓冲液中合适的蛋白质和DNA浓度的样品孵育。在这里,使用500nM UvrB或1μMXPD +1μMP44和100nM DNA。移取小体积以节省材料, 例如 0.25-0.5μL蛋白质(预稀释至10倍孵育浓度)和DNA,总体积为2.5-5μL的1x蛋白质反应缓冲液。在这里,在37℃下在热块中孵育30分钟。在表离心机中短暂(〜1秒)在反应管中旋转e确保小批量混合。
- 样品沉积
- 准备云母基材:使用手术刀从较大的条切割大约1×1厘米2的云母。使用胶带剥离多层云母矿物片的顶层,以露出清洁,平坦和原子光滑的基底表面。
注意:通过剥离更多的层,云母片可以重复使用多个实验。 - 用去离子水,过滤水( 例如 25mM HEPES pH7.5,25mM乙酸钠,10mM醋酸镁)制备AFM沉积缓冲液。通过0.02微米注射器过滤器过滤。
注意:沉积缓冲液中的二价阳离子用于将带负电的DNA分子螯合到云母表面,云母表面也在中性pH下带负电荷。如果沉积缓冲液中相对较高的Mg 2+离子浓度对于特定的蛋白质-DNA系统构成问题,或者,云母表面可以使用基于硅烷的化学物质预加载氨基,以提供用于锚定的正面荷电18 。然后可以在不含或仅含有少量二价阳离子的缓冲液中进行样品沉积。 - 稀释样品(见2.1),以便立即沉积在沉积缓冲液中的云母上。沉积少量(这里:20μL)。
注意:稀释因子取决于样品浓度。在这里,稀释样品50-100x。作为经验法则,〜1nM DNA导致约1,000bp的良好的表面覆盖。 - 立即用几毫升过滤的去离子水冲洗样品三到四次,清除过量的液体,并在温和的氮气流中吹干。从样品沉积到干燥样品的整个过程可以在不到30秒的时间内进行。
- 使用胶带在其显微镜载玻片边缘固定云母片。
注意:不同的AFM系统有d将样品固定到舞台上的不同要求。其他AFM具有磁性阶段,云母片固定在磁盘上, 例如使用热胶。这里使用的AFM系统的细节可以在材料表中找到。
- 准备云母基材:使用手术刀从较大的条切割大约1×1厘米2的云母。使用胶带剥离多层云母矿物片的顶层,以露出清洁,平坦和原子光滑的基底表面。
- AFM成像
- 将样品(见2.2.5)放置在AFM舞台上,并将显微镜载玻片固定在带有磁垫的舞台上。
- 将AFM尖端插入刀头座。使用尖锐(<10 nm)AFM探头的悬臂,适用于空气中的振荡,间歇接触模式成像。使用AFM探针,例如材料表中列出的 。这里(取决于AFM),通过拧紧夹紧螺丝(手指紧固)将尖端固定在夹具下的夹具中。将刀片座插入AFM测量头。将头靠在这一步上。
- 将AFM测量头放在样品的顶部。细节取决于AFM模型。在这里,m确保头部稳定地站立在台阶缩进之内。确保云母位于舞台上,AFM尖端将悬停在其上方。台面右侧的千分尺螺丝可以精确定位样品。
- 将AFM激光对准悬臂的背面,以便从位于激光被反射的位置敏感光电检测器获得最佳的信号强度。细节取决于AFM模型。
- 在这里,转动AFM测量头右侧和后面的车轮,以调整AFM激光器的x和y位置,将其中心地引导到悬臂的末端。在AFM视频窗口中观察反射信号(如果可用,请在此处按下相机图标并选择输入:Svideo)。
- 一旦粗定位,通过微调两个轮的激光位置(停留在悬臂的末端,总和信号)来优化检测器和信号(AFM软件中的Sum和Deflection Meter窗口中的Sum)l取决于AFM和悬臂类型,这里:目标为sum> 5)。
- 通过将AFM激光反射引导到检测器中心(这里:转动AFM测量左侧的车轮),将来自检测器阵列的顶部和底部二极管的差分信号(这里:Sum和Deflection Meter窗口中的偏转信号)置零头)。
注意:与零的偏差表示由于表面相互作用引起的悬臂的偏转,这些相互作用被AFM转换成高度信息。 - 通过在AFM软件中实现的频率曲线来确定悬臂共振频率(这里:在主面板/调谐窗口中自动调整命令)。为驱动悬臂振荡的压电体选择对应于1 V输入的振幅。将振荡频率设置为比谐振频率略低(5%),并将振荡的相位置零。
- 使用粗制模具接近样品表面的尖端e(这里:命令参与主面板窗口),直到达到保护设置(设定点)。使用〜2%的自由电平振荡幅度作为设定点(此处,在主面板中输入设定点980 mV)。
- 通过使用AFM软件降低设定点,将AFM尖端与样品表面接合良好。目标是在悬臂振荡阶段(Sum和偏转仪窗口中的相位)正好在自由水平相位之下(在啮合之前,这里通常为〜70)的排斥模式成像。在这里,使用大约70-80%自由电平振幅(1 V)的典型最终设定点。
- 扫描之前,请选择主频道面板中要录制的信号。选择高度(Ht)和振幅(Am)。
- 开始样品扫描(这里:命令在主面板窗口中进行扫描)。使用例如2.5μm/ s的扫描速度(在主面板中指令扫描速度)可以对4μmx 4μm或8μmx 8μm的表面区域进行成像(在主窗格中输入扫描尺寸l)像素分辨率分别为2,048或4,096(主面板中的扫描点和扫描线)。
- 保存图像文件(在主面板中命令保存图像),无需进行适配修改(在主通道面板/保存平面拟合中选择无)。
- 为进一步分析,处理保存的图像。加载图像(命令在AFM分析菜单中浏览保存的数据)。打开修改面板(按图像顶部的M)。将x和y维度的平面应用应用于高度图像(扩展名HtR)("修改面板"中Planefit窗口中的命令XY,选择Planefit Order 3)。然后平铺图像(在"修改面板"中的"平整窗口中的"展平"命令)中选择"平铺顺序3"。
- 将图像导出为TIFF文件(按图像顶部的命令,选择TIFF导出2x对应于2048像素分辨率)进一步分析。
AFM分析
- 蛋白质复合体积
- 预通过直接目视检查AFM软件中的图像来选择相关的蛋白质-DNA复合物,使用合适的配色方案来最大化不同大小复合物的不同外观(参见图2中不同颜色和不同复合物的大小);这里:颜色方案SeaLandAndFire in AFMsoftware)。 对于XPD样品,可能的复合物包括XPD / p44-DNA以及XPD-DNA和p44-DNA,由于XPD (〜95kDa)和p44(〜40kDa)的分子量相对较大,具有明显不同的大小, 。
- 测量DNA上单个蛋白质峰的体积以验证复合物类型。这可以通过不同的图像软件(这里是AFM软件的部分工具)来实现。
- 用截面窗口光标测量蛋白质峰部分的高度(h)和直径(d)。测量靠近颗粒部分底部的直径。
- 确定豌豆的体积(V)ks使用简单的数学模型, 例如 ,使用以下公式,其基于球形帽模型:
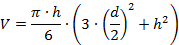
- 将测量体积翻译成近似的蛋白质分子量。
- 最初,使用一系列具有已知分子量的蛋白质校准AFM系统的体积与分子量(MW)转换(这里:总共12个实验,每个在25和851个数据点之间进行5个不同的蛋白质单体,二聚体,三聚体或四聚体状态)4,19,20。沉积和图像蛋白(如上所述,在2.2和2.3中),并如上所述测量其体积(3.1.2)。绘制体积超过其已知的分子量。得到的曲线图将显示V与MW之间的线性关系,其方程式可以由此得到一条适合数据的线。对于这里使用的AFM系统,获得以下关系19 :
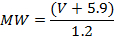
注意:在每次测量之前,不必重复此步骤,但只能进行一次。可以使用具有稍微变化的直径的AFM探针将体积与MW校准应用于在相似成像条件下获得的图像。 - 基于它们的测量体积(3.1.2)和V-to-MW校准(3.1.3.1),确定蛋白质复合物20的近似MW,其提供了其分子内容的信息。 例如 ,对于XPD / p44样品,获得约50kDa,〜100kDa和〜140kDa,与p44-DNA复合物(或仅由DNA上部结构引起的峰),XPD仅峰值和XPD / p44峰分别一致。
- 最初,使用一系列具有已知分子量的蛋白质校准AFM系统的体积与分子量(MW)转换(这里:总共12个实验,每个在25和851个数据点之间进行5个不同的蛋白质单体,二聚体,三聚体或四聚体状态)4,19,20。沉积和图像蛋白(如上所述,在2.2和2.3中),并如上所述测量其体积(3.1.2)。绘制体积超过其已知的分子量。得到的曲线图将显示V与MW之间的线性关系,其方程式可以由此得到一条适合数据的线。对于这里使用的AFM系统,获得以下关系19 :
- DNA上的蛋白质复合物位置
- 确定DNA片段长度。
- 跟踪AFM图像中的DNA片段, 例如 ,使用合适的图像分析软件的手绘线功能(参见例如材料表)并测量线的长度。
注意:排除DNA聚集以及由图像边缘切割的碎片。 - Bin,并使用合适的数据分析和图形软件( 例如参见表格 ),绘制直方图中整个实验的DNA长度。
- 在数据分析和图形软件中使用高斯曲线拟合长度分布,以确定DNA片段的长度。为了了解插入的DNA靶位点的位置(见1.1),只包括与Gauss曲线中心的两个标准偏差范围内的正确长度的DNA片段(y = y 0 + z exp(-2(xx c ) 2 / w 2 ),其中z是范数因子,x c和w是中心全高最大半高宽)进一步分析。
注意:高斯曲线中心的DNA长度必须接近DNA片段的理论长度(用0.34 nm / bp计算)。长度比理论值短10%是典型的,可能是由AFM分辨率限制引起的。
- 跟踪AFM图像中的DNA片段, 例如 ,使用合适的图像分析软件的手绘线功能(参见例如材料表)并测量线的长度。
- 确定DNA底物上蛋白质峰的位置。
- 测量距离较近的DNA片段末端的蛋白质峰的距离,如3.2.1.1所述,并除以总DNA长度以获得DNA长度分数单位的距离。
- Bin并使用适当的数据分析和图形软件绘制直方图中的测量距离(参见例如 图3 )。根据经验,选择一个bin大小,为n个数据点提供大约√n个bin。
注意:由于DNA末端不能在此区分,仅绘制DNA长度0.5(DNA片段中心)的分数MENT)。由于DNA末端结合不是研究的焦点,因此通过开始在位置0后略微排列位置数据( 例如 ,从DNA长度分数为0.02)进行分组,将DNA末端排除在分析之外。
- 从蛋白质复合物位置分布确定靶位点特异性。
- 使用高斯曲线拟合位置分布中的最大值(增强绑定事件),如3.2.1.3所示,但是基于背景绑定的高度( 见图3 )。
- 计算特异性位点与非特异性DNA背景(结合非特异性DNA位点的蛋白质分子)的特异性S ,具有以下公式21
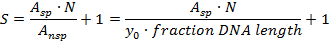
A sp :特定配合物的数量(高斯曲线下的面积)
A nsp :非特异性复合物的数量(背景的面积, (分数DNA长度)×(平均背景高度y 0 );对于从0.02 DNA长度开始的直方图,这里覆盖的DNA长度的分数为0.48)
N:可能的DNA结合位点数(这里:N = 914,不包括DNA末端)
- 确定DNA片段长度。
- DNA弯角
- 在合适的图像分析软件中使用角度测量工具,测量沿DNA骨架中心放置的两条线之间的角度β,并以蛋白质峰值为中心(参见图4中的插图)。测量统计学相关数量的蛋白质-DNA复合物的角度(> 50,理想的> 100)2,3,5。
注意:DNA弯曲角度定义为180°-β2,3,5。 - 使用数据分析和绘图软件(参见表材料)通过分组DNA弯曲角度产生弯角分布直方图。
- 用高斯曲线拟合弯角分布。如果分布中有多个最大值,则选择多峰高斯拟合。高斯曲线的中心给出了特定物种的平均弯角状态。
- 如果弯曲角度的偏移(高斯拟合的最大值)对于例如不同蛋白质变体或不同蛋白质复合物种的分布是明显的,则应用学生t检验来评估变化。
- 在合适的图像分析软件中使用角度测量工具,测量沿DNA骨架中心放置的两条线之间的角度β,并以蛋白质峰值为中心(参见图4中的插图)。测量统计学相关数量的蛋白质-DNA复合物的角度(> 50,理想的> 100)2,3,5。
结果
基于蛋白质复合体积区分不同复杂类型
原核NER解旋酶UvrB的解旋酶活性受DNA结合22,23刺激。 UvrB需要DNA中的未配对区域(DNA气泡)才能正确地加载到两条单链DNA链之一上。 在体内 ,该DNA结构通过NER级联的另一种蛋白质的DNA相互作用提供;在体外实验中,气泡可以人造地引入到与目标病变附近的dsDNA片?...
讨论
蛋白质在含有特定靶位点的长DNA片段上的结合位置的AFM统计分析可以揭示蛋白质识别这些位点2,3,4,5,6的特定策略的有趣细节。为了解释所得到的位置分布,DNA中靶标的位置需要精确地知道。这是通过将定义明确的序列位置的特定位点引入循环质粒DNA并使用限制酶技术切割包含该特异位点的DNA片段来实现的。在蛋白质结合位置的AFM图像分析中,只有具有正确长度的DNA片段(与切出的片段的全长一致?...
披露声明
作者没有什么可以披露的。
致谢
PUC19N,含CPD的寡核苷酸和p44分别由Samuel Wilson,Korbinian Heil和Thomas Carell以及Gudrun Michels和Caroline Kisker提供。这项工作得到德意志民主共和国(DFG)FZ82和TE-671/4向IT部门的资助。
材料
| Name | Company | Catalog Number | Comments |
| Molecular Force Probe (MFP) 3D | Asylum Research | N/A | atomic force microscope (AFM) |
| Precision 390 | DELL | N/A | computer |
| ThermoMixer and 1.5 mL block | Eppendorf | 5382000015 | heat block for DNA preparation |
| Rotilabo Block-Heater H 250 & blocks for 0.5 mL tubes | Carl Roth GmbH | Y264.1 & Y267.1 | heat block for protein-DNA incubations |
| Mini-Sub Cell GT | Bio-Rad Laboratories GmbH | 1704467 | electrophoresis chamber with gel caster and power supply |
| Power Pac Basic | Bio-Rad Laboratories GmbH | 1645050 | electrophoresis power supply |
| Centifuge 5415 D with rotor | Eppendorf | 2262120-3 | table centrifuge |
| Ultra-Lum electronic UV transillumonator MEB-15 | Ultralum | 900-1322-02 | UV irradiation table |
| NanoDrop ND-1000 | VWR International / PEQLAB Biotechnologie GmbH | N/A | UV spectrophotometer |
| TKAX-CAD with 0.2 μm capsule filter | Unity Lab Services | N/A | water deionization and filter unit |
| Name | Company | Catalog number | Comments |
| Software | |||
| MFP software on Igor Pro | Asylum Research | N/A | AFM software |
| ImageJ (open source Java image processing) | NIH Image | N/A | Image analysis software |
| Excel (Microsoft Office) | Microsoft Corporation | N/A | data analysis software |
| Origin9 / Origin2016 | OriginLab Corporation | N/A | statistical data analysis and graphing software |
| Name | Company | Catalog number | Comments |
| Material | |||
| OMCL-AC240TS | Olympus | OMCL-AC240TS | AFM cantilevers |
| grade V-5 muscovite | SPI Supplies | 1805 | mica sheets |
| Amicon Ultra 0.5 mL 50k Ultracell | Millipore Ireland Ltd. | UFC505096 | centrifuge filters |
| NucleoSpin Extract II | Macherey-Nagel GmbH | 740 609.250 | Agarose gel extraction kit |
| Rotilabo cellulose paper type 111A | Carl Roth GmbH | AP59.1 | AFM deposition blotting paper |
| Anatop 25 (0.02 μm) | Whatman GmbH | 6809-2102 | syringe filter |
| SSpI, BspQI | New England Biolabs (NEB) | R0132, R0712 | restriction enzymes for DNA substrate preparation |
| XhoI, BglII | R0146, R0144 | restriction enzymes for DNA preparation controls | |
| nicking restriction enzyme Nt.BstNBI | New England Biolabs (NEB) | R0607 | nickase |
| T4 DNA ligase | New England Biolabs (NEB) | M0202S | Ligase |
| Tris, HEPES | Carl Roth GmbH | 4855, 9105 | buffer chemicals |
| NaCl, MgCl2, KCl, MgAcetate | Carl Roth GmbH | 3957, HN03, HN02, P026 | salt chemicals |
| NaAc | Sigma-Aldrich Chemie GmbH | 32318 | salt chemicals |
| DTT, TCEP, EDTA | 6908, HN95, 8040 | chemicals/reagents | |
| agarose, acetic acid, HCl | Carl Roth GmbH | 2267, 3738, K025 | reagents |
| ATP | Carl Roth GmbH | K054 | nucleotides |
| oligonucleotide #1 in Table 1 | Biomers | custom | complementary DNA oligonucleotide |
| oligonucleotides #2, #3, and #6 in Table 1 | Integrated DNA Technologies (IDT) | custom | fluorescein containing oligonucleotides |
| oligonucleotides #4 and #5 in Table 1 | private (available from e.g. TriLink or GlenResearch) | CPD containing oligonucleotides | |
| SafeSeal reaction tube 0.5 mL and 1.5 mL | Sarstedt | 72.704 and 72.706 | incubation tubes |
| GeneRuler 1 kb | Thermo Scientific | SM0311 | DNA ladder |
| 6x concentrate gel loading dye purple | New England Biolabs (NEB) | 51406 | DNA loading dye |
| Midori Green | Nippon Genetics Europe GmbH | 999MG28055 | DNA stain |
参考文献
- Janicijevic, A., Ristic, D., Wyman, C. The molecular machines of DNA repair: scanning force microscopy analysis of their architecture. J. Microsc. 212 (3), 264-272 (2003).
- Wang, H., et al. DNA bending and unbending by MutS govern mismatch recognition and specificity. Proc. Natl. Acad. Sci. USA. 100 (25), 14822-14827 (2003).
- Tessmer, I., et al. Mechanism of MutS searching for DNA mismatches and signaling repair. J. Biol. Chem. 283 (52), 36646-36654 (2008).
- Wagner, K., Moolenaar, G., van Noort, J., Goosen, N. Single-molecule analysis reveals two separate DNA-binding domains in the Escherichia coli UvrA dimer. Nucleic Acids Res. 37 (6), 1962-1972 (2009).
- Buechner, C. N., et al. Strand-specific recognition of DNA damages by XPD provides insights into nucleotide excision repair substrate versatility. J Biol. Chem. 289 (6), 3613-3624 (2014).
- Van der Linden, E., Sanchez, H., Kinoshita, E., Kanaar, R., Wyman, C. RAD50 and NBS1 form a stable complex functional in DNA binding and tethering. Nucleic Acids Res. 37 (5), 1580-1588 (2009).
- Fuentes-Perez, M. E., Dillingham, M., Moreno-Herrero, F. AFM volumetric methods for the characterization of proteins and nucleic acids. Methods. 60, 113-121 (2013).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Miyagi, A., Lyubchenko, Y. L. Specificity of binding of single-stranded DNA-binding protein to its target. Biochemistry. 51, 1500-1509 (2012).
- Wirth, N., et al. Conservation and Divergence in Nucleotide Excision Repair Lesion Recognition. J. Biol. Chem. 291 (36), 18932-18946 (2016).
- Kuper, J., Kisker, C. Damage recognition in nucleotide excision DNA repair. Curr. Opin. Struct. Biol. 22, 88-93 (2012).
- Buechner, C. N., Tessmer, I. DNA substrate preparation for atomic force microscopy studies of protein-DNA interactions. J. Mol. Recognit. 26 (12), 605-617 (2013).
- Sun, Z., Tan, H. Y., Bianco, P. R., Lyubchenko, Y. L. Remodeling of RecG Helicase at the DNA Replication Fork by SSB Protein. Sci. Rep. 5, 9625 (2015).
- Billingsley, D. J., Bonass, W. A., Crampton, N., Kirkham, J., Thomson, N. H. Single-molecule studies of DNA transcription using atomic force microscopy. Phys. Biol. 9 (2), 021001 (2012).
- Maurer, S., Fritz, J., Muskhelishvili, G., Travers, A. RNA polymerase and an activator form discrete subcomplexes in a transcription initiation complex. EMBO J. 25 (16), 3784-3790 (2006).
- Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Theis, K., Chen, P. J., Skorvaga, M., Van Houten, B., Kisker, C. Crystal structure of UvrB, a DNA helicase adapted for nucleotide excision repair. EMBO J. 18 (24), 6899-6907 (1999).
- Kuper, J., et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 12 (9), e1001954 (2014).
- Shlyakhtenko, L. S., et al. Silatrane-based surface chemistry for immobilization of DNA, protein-DNA complexes and other biological materials. Ultramicroscopy. 97, 279-287 (2003).
- Roth, H. M., et al. XPB helicase regulates DNA incision by the Thermoplasma acidophilum endonuclease Bax1. DNA Repair. 11 (3), 286-293 (2012).
- Ratcliff, G. C., Erie, D. A. A Novel Single-Molecule Study to Determine Protein-Protein Association Constants. J. Am. Chem. Soc. 123 (24), 5632-5635 (2001).
- Yang, Y., Sass, L. E., Du, C., Hsieh, P., Erie, D. A. Determination of protein-DNA binding constants and specificities from statistical analyses of single molecules: MutS-DNA interactions. Nucleic Acids Res. 33 (13), 4322-4334 (2005).
- Caron, P. R., Grossman, L. Involvement of a cryptic ATPase activity of UvrB and its proteolysis product, UvrB* in DNA repair. Nucleic Acids Res. 16 (22), 10891-10902 (1988).
- Wang, H., et al. UvrB domain 4, an autoinhibitory gate for regulation of DNA binding and ATPase activity. J. Biol. Chem. 281 (22), 15227-15237 (2006).
- Chammas, O., Billingsley, D. J., Bonass, W. A., Thomson, N. H. Single-stranded DNA loops as fiducial markers for exploring DNA-protein interactions in single molecule imaging. Methods. 60 (2), 122-130 (2013).
- Truglio, J. J., et al. Structural basis for DNA recognition and processing by UvrB. Nat. Struct. Mol. Biol. 13 (4), 360-364 (2006).
- Kuper, J., Wolski, S. C., Michels, G., Kisker, C. Functional and structural studies of the nucleotide excision repair helicase XPD suggest a polarity for DNA translocation. EMBO J. 31 (2), 494-502 (2012).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Goosen, N. The presence of two UvrB subunits in the UvrAB complex ensures damage detection in both DNA strands. EMBO J. 21 (15), 4196-4205 (2002).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Hoeijmakers, J. H., Goosen, N. Architecture of nucleotide excision repair complexes: DNA is wrapped by UvrB before and after damage recognition. EMBO J. 20 (3), 601-611 (2001).
- Moolenaar, G. F., et al. The Role of ATP Binding and Hydrolysis by UvrB during Nucleotide Excision Repair. J. Biol. Chem. 275, 8044-8050 (2000).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。