Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Nükleotid Eksizyon Onarımında DNA Lezyon Tanınması için Atomik Kuvvet Mikroskopi Araştırmaları
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Here, the study of different DNA lesion recognition approaches via single molecule AFM imaging is demonstrated with the nucleotide excision repair system as an example. The procedures of DNA and protein sample preparations and experimental as well as analytical details for the AFM experiments are described.
Özet
AFM imaging is a powerful technique for the study of protein-DNA interactions. This single molecule method allows the simultaneous resolution of different molecules and molecular assemblies in a heterogeneous sample. In the particular context of DNA interacting protein systems, different protein complex forms and their corresponding binding positions on target sites containing DNA fragments can thus be distinguished. Here, an application of AFM to the study of DNA lesion recognition in the prokaryotic and eukaryotic nucleotide excision DNA repair (NER) systems is presented. The procedures of DNA and protein sample preparations are described and experimental as well as analytical details of the experiments are provided. The data allow important conclusions on the strategies by which target site verification may be achieved by the NER proteins. Interestingly, they indicate different approaches of lesion recognition and identification for the eukaryotic NER system, depending on the type of lesion. Furthermore, distinct structural properties of the two different helicases involved in prokaryotic and eukaryotic NER result in and explain the different strategies observed for these two systems. Importantly, these experimental and analytical approaches can be applied not only to the study of DNA repair but also very similarly to other DNA interacting protein systems such as those involved in replication or transcription processes.
Giriş
Atomik kuvvet mikroskobu (AFM), protein-DNA etkileşimlerinin analizi için güçlü bir tekniktir 1 , 2 , 3 , 4 , 5 , 6 , 7 , 8 , 9 . Tek molekül düzeyinde bir çözünürlükle heterojen örnekleri doğrudan görselleştirmek için yalnızca düşük miktarda numune malzemesi gerekir. Heterojenlik, bir proteinin farklı konformasyonel veya oligomerik durumlarından kaynaklanabilir. Özellikle, protein-DNA örnekleri bağlamında, protein kompleksleri genel olarak DNA bağlanması veya DNA içindeki spesifik bir hedef bölgeye bağlanma ile uyarılan farklı stoikiometri ve / veya konformasyonlar gösterebilir. Heterojen numuneler ayrıca iki (veya daha fazla) farklı protein türü ve farklı protein kompleksi içerebilirFormlar ( örn . Heteromerik komplekslere karşı yalnızca bir protein türünü kapsar) DNA ile farklı etkileşime girebilirler. Burada tartışılan çalışmalar, bu proteinlerin bir hedefini temsil eden bir lezyon içeren uzun (~ 900 baz çiftli, bp) DNA fragmanlarına bağlı DNA onarım proteinlerinin statik, kurutulmuş örnekleri üzerindeki havadaki AFM görüntülemesini kullanmaktadır. AFM'nin yüksek, moleküler çözünürlüğü, farklı protein kompleksleri türleri arasında ayrım yapmayı ve proteinlerin DNA fragmanları üzerindeki bağlanma yerlerini belirlemeyi sağlar. Önemli olan, lezyonlar, DNA substratlarına iyi tanımlanmış pozisyonlarda eklenir. DNA'daki lezyon yerinin konumu bilindiğinden, DNA'ya bağlı olan proteinlerin dağılımı, (farklı) protein komplekslerinin (farklı) lezyon tanıma özelliklerine, örneğin belirli bir lezyon tipini ne kadar iyi tanıdıklarını (karşılaştırıldığında) Hasar görmemiş DNA'ya) 2 , 3 , 4 , 5 , 6 . DNA'daki pozisyonları ayrıca lezyonlara spesifik olarak bağlanan protein kompleksleri ile DNA'nın başka yerlerinde spesifik olmayan şekilde bağlanmış olan kompleksler arasındaki ayrımı da mümkün kılar. Bu farklı karmaşık türlerin (lezyona spesifik olmayan komplekslere spesifik olarak bağlanan kompleksler) ayrı karakterizasyonu, hedef bölge tanımlamasında indüklenen komplekslerde potansiyel konformasyonel değişiklikleri ortaya çıkarabilir.
Burada odaklanan DNA tamir proteinleri, nükleotid eksizyon tamiri (NER) yolunda lezyonun tanınmasından sorumlu helikazlardır. Bakterilerde NER, UvrA, UvrB ve UvrC proteinleriyle elde edilir. UvrA, bir UvaA 2 / UvrB 2 DNA tarama kompleksinde başlangıç lezyon algılamasından sorumludur. UvrB lezyon doğrulamasının ardından bu kompleks, lezyon yerinde bağlı monomerik UvrB'ye dönüşür ve bu spesifik kompleks pRokaryotik NER endonükleaz UvrC. UvrC, lezyonu içeren kısa (12-13 nt) bir tek sarmallı DNA (ssDNA) uzatır. Eksik streç daha sonra DNA polimeraz ile doldurulur. Son olarak, DNA ligazı yeni sentezlenmiş gerdirmeyi orijinal DNA 9 , 10 ile sızdırmaz hale getirir. Ökaryotlarda, NER basamaklarının çoğunun proteinleri büyük, multimerik transkripsiyon faktörü II H (TFIIH) kompleksinin bir parçasıdır. Trimerik CEN2-XPC-HR23B kompleksi yoluyla ilk lezyon algılamasından sonra, TFIIH DNA hedef bölgeye yönlendirilir. Kompleks içindeki XPD, bir NER hedef lezyon varlığını doğruladığında, ökaryot NER endonükleazlar XPG ve XPF, lezyon 9 , 10 içeren kısa (24-32 nt) bir ssDNA serisini tüketmek üzere görevlendirilir. Burada, özellikle sırasıyla prokaryotik ve ökaryotik NER'den gelen UvrB ve XPD helikazları incelenmiştir. Bu helikazlar, eşleştirilmemiş bir bölge gerektirir.DNA (bir DNA kabarcığı) iki DNA tek sarmalından birine bağlanıp daha sonra ATP hidroliziyle beslenen bu sarmal boyunca yer değiştirir. DNA lezyonlarına ilaveten, proteinler için yükleme yeri olarak işlev gören substratlara bir DNA kabarcığı ilave edildi.
Spesifik lezyon DNA substratlarının hazırlanması prosedürü daha önce 11 açıklanmıştır. Bir lümen için iki birbirine yakın aralıklı kısıtlama bölgeleri olan dairesel bir DNA yapısı (plazmid) gerektirir. Bu çalışma bağlamında plazmid pUC19N (2729 bp) kullanıldı (S. Wilson laboratuarı, NIEHS tarafından hazırlandı). Bu plazmit, 48 nükleotit (nt) bir gerilimi çerçeve içine alan Nt.BstNBI nickazı için birbirine yakın mesafeli üç sınırlama bölgesi içerir. Lipaz ile inkübe edildikten sonra, bu alanlar arasındaki ssDNA'nın uzantısı uzaklaştırılabilir ve herhangi bir hedef özelliği içeren bir oligonükleotid ile değiştirilebilir. Her adımdan sonra, enzimatik sindirim, agaroz jeliElektroforez. Yuvarlak dairesel DNA, orijinal süper-sargılı plazmid ile karşılaştırıldığında daha düşük elektroforetik hareketliliğinden dolayı ayırdedilebilir. DNA'nın boşluk bırakılması ve çıkarılan gerilmenin spesifik alt tabaka oligonükleotidiyle değiştirilmesi, alt tabakayı münhasıran çentikler arasındaki bölgede kesen bir kısıtlama enzimi ile sindirim yoluyla değerlendirilebilir. Dolayısıyla, dairesel plazmitin enzim tarafından doğrusallaştırılması, çakıştırılmış DNA için bastırılacak ve spesifik oligonükleotidin eklenmesinden sonra restore edilecektir. Son olarak, iki endonükleaz sınırlama alanı (ideal olarak tek kesiciler), arzu edilen uzunlukta ve belirlenmiş bir konumda spesifik hedef alanı ve aynı zamanda lezyondan belirli bir mesafede bulunan bir DNA kabarcık ile, doğrusal bir DNA substratının oluşturulmasına izin verir. 'Veya 3' yönünde.
Lezyonların NER helikazları tarafından tanınması AFM görüntüleme yoluyla araştırılabilir. Tepedeki helikazların duraklatılmış DNA translokasyonuLezyon bölgesi, DNA üzerindeki protein pozisyon dağılımında zirve olarak görünür ve lezyon tanımayı gösterir. Bu helikazların DNA translokasyonu ayrıca yönlü, 5'-3 'polarite ile lezyon tanımanın yükleme yerine (DNA kabarcık yukarı akış veya lezyonun aşağısında) bağımlılığı da lezyonun öncelikli olarak tanınıp onaylanmadığını gösterir Translokasyona uğratılmış veya tersine, translokasyona uğramamış ssDNA iplikçikleri 5 , 9 . Sonraki bölümlerde, kullanılan yöntemler tanıtılacak ve bu deneylerden elde edilen önemli bulgular kısaca tartışılacaktır. Önemli olarak, burada gösterilen DNA tamiri ile ilgili örnek çalışmalara benzeyen AFM görüntüleme, DNA replikasyonu veya transkripsiyon 8 , 12 , 13 , 14 gibi farklı DNA etkileşen sistemlerin çalışmasına uygulanabilir Sup.
Protokol
1. Numune Hazırlama
- DNA substratlarının hazırlanması 11
- Plazmidde bir ssDNA boşluğu üretme
- Üreticinin protokolüne göre koşulları kullanarak, enzim ısısı inaktivasyonunu takiben uygun bir lümenli bir reaksiyon tüpünde (burada: Nt.BstNBI) plazmidden bir numuneyi (burada modifiye edilmiş pUC19, pUC19N) tamamen sindirin (şematik için Şekil 1'e bakın) sunum). Yeterli bir verim için ~ 50 uL ve ~ 500 nM plazmid ile başlayın.
- Seyreltilmiş numuneler (~ 20 nM) üzerinde agaroz jel elektroforezi ile plazmit nicking doğrulayın. DNA görselleştirme için kullanılan DNA bağlayıcı boyaya karşı koruma için eldivenler takın.
NOT: Farklı elektroforetik hareketlilik, çentikli (rahat) ve süper-sargılı dairesel plazmid DNA'yı birbirinden ayırır ( Şekil 1 ). - Kesilen ssDNA gerilimini (nick alanları arasında) plazmiddenBir ısı bloğundaki oligonükleotidin erime sıcaklığının (burada pUC19N için 68 ° C) 300 rpm'de çalkalanarak tamamlayıcı oligonükleotitin ( Tablo 1'deki oligonükleotid l'in) ~ 10 kat fazlasıyla inkübe edildi ( Şekil 1 ).
- Bir masa santrifüjünde 10,000 xg'de 10 dakika süreyle santrifüje edilerek 50 kDa molekül ağırlığı kesilmiş (MWCO) filtre kullanarak daha küçük DNA fragmanlarından ayrılmış plazmid ayrılır ( Şekil 1 ). Filtreden konsantre DNA'yı çıkarmak için filtreyi tersine çevirin ve yeni bir 1.5 mL reaksiyon tüpüne yerleştirin. 1,000 x g'de 3 dakika santrifüjleyin.
- Elde edilen konsantre DNA örneğini, iyonu alınmış, filtrelenmiş su ile 500 uL'ye doldurun, ek bir ~ 5 misli fazla tamamlayıcı oligonükleotid ilave edin ve adımlar 1.1.1.3 ve 1.1.1.4'ü en az 3 kez tekrarlayın.
- Conditi kullanarak uygun bir restriksiyon enzimi (burada: XhoI veya BglII) ile kuluçka yapılarak DNA'nın tamamen boşluğunu test edinÜreticinin açıklamasına göre. Seyreltilmiş (çentikli ve boşluklu) DNA örnekleri (~ 20 nM) kullanın. Pozitif kontrol olarak çizgili DNA kullanarak doğrusallaştırılmış plasmid (hiçbir ssDNA boşluğu içermeyen) ve kesilmiş DNA (boşluk bırakılmış DNA) arasında ayrım yapmak için bir agaroz jel elektroforezi çalıştırın (referans için bir DNA merdiveni ekleyin, Şekil 1 ). Eldiven giy.
- Aralığı değiştirilmiş ssDNA oligonükleotidleri ile doldurma
- Çakıştırılmış plazmid, ~ 45 ° C'de 4 saat süreyle inkübe edilerek ( Şekil 1 ) seçilen spesifik hedef bölgeyi içeren 5 'fosforillenmiş bir oligonükleotitin 25 misli fazlalığı ile inkübasyon yoluyla tavlanır. Burada, DNA kabarcıkları üretmek için flöresin adisyonlu timin veya siklobutan pirimidin dimer (CPD) ve ayrıca kısa (8 nt) bir lezyon içeren 48 nts ssDNA kullanın (bkz. Tablo 1 ).
- Tavlanmış ekin plazmaya kuvertür bağlanması için incubaT4 DNA ligazı ile oda sıcaklığında üreticinin protokolüne göre gece boyunca eklendi ( Şekil 1 ). Bu reaksiyon için, ligaz için uygun tampon koşulları üretmek için konsantre tampon stok solüsyonu içeren ATP ilave edin ( örn. , UvrB reaksiyon tamponu: 50 mM Tris-HC1 pH 7.5, 10 mM MgCl2, 50 mM KCI, 5 mM DTT, 1 mM ATP).
- Seyreltilmiş örneklerin uygun bir kısıtlama enzimi (1.1.1.6'da olduğu gibi) ile imalatçının protokolüne göre sindirerek spesifik oligonükleotidin çakıştırılmış plasmid içine sokulması için test edin ve bunu takiben bir agaroz jel elektroforezi (1.1.1.6, Şekil 1 ).
- Doğrusal DNA substratının hazırlanması
- Üreticinin önerdiği koşulları kullanarak modifiye plazmid DNA'yı (ideal olarak plazmitteki tek bir kısıtlama bölgesi olan) restriksiyon enzimleri ile sindirin ( Şekil 1 ). Bu adım,Yerleştirilen modifikasyonun tanımlanmış bir konuma getirilmesi. Burada, DNA uzunluğunun ~% 30'unda spesifik lezyon bölgesi içeren 916 bp'lik bir DNA fragmanına neden olan, sırasıyla 613 ve 255 bp'lik sıranın yukarı ve aşağı kısımlarında kesme SspI ve BspQI kullanınız.
NOT: Burada açıklanan AFM görüntüleme deneyleri için, ~ 200 bp ile ~ 2000 bp arasında uzunluklardaki DNA parçaları uygun substratlardır. - Hedef parçayı, ticari bir kit kullanarak agaroz jel elektroforezi ve jel ekstraksiyonu yoluyla izole edin. Koruyucu eldiven giyin.
- İsteğe bağlı olarak, saflaştırılacak konsantre DNA içeren şeritlerden DNA merdiveni ve seyreltilmiş numune şeritlerini (ilk iki şerit) ayırmak için bir neşterle agaroz jelini kesin. Sadece jelin bir kısmını bir UV masasına yerleştirerek ilk iki şeritteki jel bölümünü UV radyasyona maruz bırakın.
- Bir bisturi ile seçilen parçaya tekabül eden bandı kesin. İki jel parçasını yeniden birleştirin (UV sekmesinden çıkarın)le). İstenilen DNA substratının bandı konumu için seyreltilmiş numunedeki eksize bandın pozisyonunu kullanın. Seyreltik kontrol için olduğundan biraz daha geniş dilim keserek daha yüksek konsantrasyonu hesaplayın.
NOT: Burada, UV-lezyonlarının tanınması araştırıldığından UV ışınlaması yoluyla ilave UV lezyonlarının sokulması DNA substratında bu yaklaşımla dikkatle önlenmiştir.
- Bir bisturi ile seçilen parçaya tekabül eden bandı kesin. İki jel parçasını yeniden birleştirin (UV sekmesinden çıkarın)le). İstenilen DNA substratının bandı konumu için seyreltilmiş numunedeki eksize bandın pozisyonunu kullanın. Seyreltik kontrol için olduğundan biraz daha geniş dilim keserek daha yüksek konsantrasyonu hesaplayın.
- 260 nm ~ 6,700 M - 1 cm - 1 bp'de bir çift sicimli DNA (dsDNA) ortalama molar sönme katsayısı ile Lambert-Beer yasasını kullanarak bir UV-Vis spektrofotometre ile ölçülen 260 nm'lik absorbsiyona ait DNA konsantrasyonunu hesaplayın:
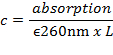
Burada L yol uzunluğudur (ölçüm odası uzunluğu, tipik olarak 1 cm'dir).
- Üreticinin önerdiği koşulları kullanarak modifiye plazmid DNA'yı (ideal olarak plazmitteki tek bir kısıtlama bölgesi olan) restriksiyon enzimleri ile sindirin ( Şekil 1 ). Bu adım,Yerleştirilen modifikasyonun tanımlanmış bir konuma getirilmesi. Burada, DNA uzunluğunun ~% 30'unda spesifik lezyon bölgesi içeren 916 bp'lik bir DNA fragmanına neden olan, sırasıyla 613 ve 255 bp'lik sıranın yukarı ve aşağı kısımlarında kesme SspI ve BspQI kullanınız.
- Plazmidde bir ssDNA boşluğu üretme
- Proteinlerin ekspresyonu ve saflaştırılması
- E. coli içinde rekombinant şekilde UvrB'yi eksprese edin ve proteini , daha önce tarif edildiği gibi 16 standart kütin boncuk eğilimi 15 ve boyut uzaklaştırma kromatografisi yoluyla saflaştırın 16 .
NOT: Bacillus caldotenax'tan uvrB geni pTYB1 vektörüne klonlanmıştı. - E. coli'de XPD'yi eksprese edin ve proteini standart nikel IDA afinitesi 15 ve boyut uzaklaştırma kromatografisi 15, ardından anyon değişim kromatografisi 15 , daha önce tarif edildiği gibi 17 ile saflaştırın.
NOT: Chaetomium thermophilum Xeroderma pigmentosum D grubu protein (XPD) geni, pBADM11 vektörüne klonlanmıştı . C. thermophilum p44, Caroline Kisker'in laboratuvarından bir tür armağan, ifade edildi ve puriAçıklandığı gibi atılmış 17 .
- E. coli içinde rekombinant şekilde UvrB'yi eksprese edin ve proteini , daha önce tarif edildiği gibi 16 standart kütin boncuk eğilimi 15 ve boyut uzaklaştırma kromatografisi yoluyla saflaştırın 16 .
2. AFM Deneyi
- örnek hazırlama
- İsteğe bağlı olarak, buzdolabında saklanırken oluşabilecek olası mikro-tuzlu kristalleri çıkarmak için, DNA alt-tabakasını bir ısı bloğunda 65 ° C'de önceden inkübe edin.
- Reaksiyon tamponu (leri) 10 kat konsantrasyonda (10x tampon) hazırlayın.
NOT: 1x konsantrasyonda XPD reaksiyon tamponu 20 mM Tris-HC1 pH 7.5, 10 mM KC1, 5 mM MgCl2, 1 mM TCEP, 2 mM ATP; 1x UvrB reaksiyon tamponu, 50 mM Tris-HCI pH 7.5, 50 mM KCI, 10 mM MgCl2, 5 mM DTT ve 1 mM ATP ihtiva etti. - Kompleks oluşumunu arttırmak için proteinleri uygun inkübasyon koşullarında daha yüksek bir konsantrasyonda ön inkübe edin. 1x protein reaksiyonu tamponu içindeki tek tek proteinlerin küçük bir hacmini ( örn. , 1 μL) önceden istenen konsantrasyona kadar seyreltin ve küçük hacimlerde ( örn. , 1 μL) t0.5 mL reaksiyon tüpünde bireysel protein solüsyonları.
- Tüp, oda sıcaklığından daha yüksek inkübasyon sıcaklıkları için bir ısı bloğuna yerleştirilir. Beklenen kompleks stokiyometriye, ya eşmolar ya da buna tekabül eden konsantrasyonlara bağlı olarak bir konsantrasyon oranı seçin. Burada, 1 uL XPD'yi (1 x XPD reaksiyon tamponu içinde 20 uM) ve 10 uM'de her biri için 10 dakika 37 ° C'de 1 uL p44 (1x XPD reaksiyonu tamponu içinde 20 uM) inkübe edin.
- 0.5 mL reaksiyon tüpünde protein reaksiyonu tamponu içinde uygun protein ve DNA konsantrasyonlarında örnekleri inkübe edin. Burada, 500 nM UvrB veya 1 uM XPD + 1 μM p44 ve 100 nM DNA kullanın. Malzemeyi, örneğin 0.25-0.5 μL protein (önceden 10 kat inkübasyon konsantrasyonuna seyreltilmiş olarak) ve DNA'yı 2.5-5 μL 1x protein reaksiyon tamponunun toplam hacminde kaydetmek için küçük hacimlerde pipetleyin. Burada, bir ısı bloğunda 37 ° C'de 30 dakika inkübe edin. Bir tepkime tüpünde bir masa santrifüjünde kısaca (~ 1 s) döndürünE küçük hacimlerin karıştırılmasını sağlamak.
- Örnek depozisyon
- Bir mika alt tabakası hazırlayın: bir bisturi yardımıyla daha büyük şeritlerden yaklaşık 1 x 1 cm 2 lik bir mika kesin. Çok katmanlı mika mineral parçasının üst katmanlarını yapışkan bantla soyarak temiz, düz ve atomik olarak pürüzsüz bir yüzey elde edin.
NOT: Mika parçası daha fazla tabaka (ları) soyarak çoklu denemeler için yeniden kullanılabilir. - AFM çökelme tamponunu deiyonize filtrelenmiş su, örneğin 25 mM HEPES pH 7.5, 25 mM Na-asetat, 10 mM Mg-asetat ile hazırlayın. 0.02 μm şırınga filtresi ile filtre edin.
NOT: Biriktirme tamponundaki çift değerli katyonlar, negatif yüklü DNA moleküllerini, nötr pH'da negatif yüklü mika yüzeyine kenetlemek için kullanılır. Eğer çökelme tamponundaki nispeten yüksek Mg2 + iyon konsantrasyonu, belirli bir protein-DNA sistemi için bir sorun teşkil ediyorsa, alternatif olarak,Mika yüzeyi ankraj 18 için pozitif yüzey yükleri sağlamak için silatran bazlı kimya kullanarak amino grupları ile önceden yüklenebilir. Numune birikimi, daha sonra, az ya da çok miktarda iki değerli katyon içeren bir tamponda gerçekleştirilebilir. - Depolama tamponu içindeki mika üzerine derhal çökelmek için numuneyi seyreltin (2.1'e bakın). Küçük bir hacim bırakın (burada: 20 μL).
NOT: Seyreltme faktörleri numune konsantrasyonuna bağlıdır. Burada, numuneleri 50-100x seyreltin. Bir kural olarak, ~ 1 nM DNA ~ 1,000 bp için iyi bir yüzey kaplaması sağlar. - Numuneyi, birkaç mililitre süzülmüş, iyonsuzlaştırılmış su ile üç ila dört kez tekrar durulayın, fazla sıvıyı lekeleyip nazik bir nitrojen akışı ile kurutun. Numune biriktirme işleminden kurutulmuş numunelere kadar olan süreç 30 saniyeden daha kısa sürede gerçekleştirilebilir.
- Mika parçasını, kenarlarında yapışkan bant kullanarak mikroskop slayt üzerinde düzeltin.
NOT: Farklı AFM sistemlerinde dNumunenin sahneye sabitlenmesi için gereken şartlar. Diğer AFM'ler manyetik aşamalara sahiptir ve mika parçaları manyetik disklere, örneğin termal tutkal kullanılarak sabitlenir. Burada kullanılan AFM sisteminin ayrıntıları Malzeme Tablosu'nda bulunabilir.
- Bir mika alt tabakası hazırlayın: bir bisturi yardımıyla daha büyük şeritlerden yaklaşık 1 x 1 cm 2 lik bir mika kesin. Çok katmanlı mika mineral parçasının üst katmanlarını yapışkan bantla soyarak temiz, düz ve atomik olarak pürüzsüz bir yüzey elde edin.
- AFM görüntüleme
- Numuneyi (bkz. 2.2.5) AFM aşamasında merkezi olarak yerleştirin ve manyetik pedlerle sahnedeki mikroskop slaydı sabitleyin.
- AFM ucunu uç tutucuya yerleştirin. Havada titreşen, aralıklı temas modu görüntülemesi için keskin (<10 nm) bir AFM probu bulunan konsolları kullanın. Örneğin, Malzeme Tablosunda listelenen gibi AFM probları kullanın. Burada (AFM'ye bağlıdır) kelepçe vidasını sıkıştırarak (parmakla sıkıştırın) ucu tutucuya bir kelepçe altına sabitleyin. Uç tutucuyu AFM ölçüm kafasına takın. Bu adım için başını sırt üstünde dinlendirin.
- AFM ölçüm kafasını numunenin üzerine yerleştirin. Ayrıntılar AFM modeline bağlıdır. Burada, mKafanın sahne girintileri içindeki bacaklarıyla stabil durduğundan emin olun. Mika'nın, AFM ucunun doğrudan üzerinde bulunduğu sahnede bulunduğundan emin olun. Sahnenin sağındaki mikrometre vidaları, numunenin hassas konumlandırılmasına olanak tanır.
- Lazerin üzerine yansıdığı konuma duyarlı fotodetektöre optimum sinyal gücü için konsolun arkasındaki AFM lazerini hizalayın. Ayrıntılar AFM modeline bağlıdır.
- Burada, AFM lazerinin x ve y konumlarını konsolun ucuna merkezi olarak yönlendirmek için AFM ölçüm kafasının sağ tarafındaki ve arkasındaki tekerlekleri çevirin. AFM video penceresindeki yansıma sinyalini izleyin (varsa, burada: kamera simgesine basın ve girişi seçin: Svideo).
- Kaba bir şekilde yerleştirildikten sonra, iki tekerleğin lazer konumunu ince ayarlayarak (konsolun sonundaki toplam sinyali) dedektör toplam sinyalini (Toplamda Sum ve Sapma Ölçer penceresinde AFM yazılımı) optimize edinL AFM ve konsol türüne, burada: toplamı hedefliyor> 5).
- AFM lazer yansımasını dedektör merkezine (burada: tekerleği AFM ölçümünün sol tarafında çevirin) yönlendirerek detektör dizisinin üst ve alt diyotlarından gelen fark sinyalini sıfırlayın (burada: Toplam ve Sinyal Saptır penceresinde Saptırma sinyali penceresi) kafa).
NOT: Daha sonra sıfırdan sapmalar, AFM tarafından yükseklik bilgilerine çevrilen yüzey etkileşimlerinden dolayı kirişin sapmasına işaret eder. - Kanton rezonans frekansını, AFM yazılımında uygulanan bir frekans tonu ile belirleyin (burada: Master Panel / Tune penceresinde komut otomatik ayarlama). Konsol salınımını sağlayan piezo için 1 V girişine karşılık gelen bir genlik seçin. Osilasyon frekansını rezonans frekansından biraz düşük (% 5) ve osilasyon fazını sıfırlayın.
- Ham kavrama modunu kullanarak ucu örnek yüzeyine yaklaştırınKoruyucu ayar (ayar noktası) ulaşılana kadar (burada: Ana Panelde Engage komut penceresi). Serbest düzey salınım amplitüdünün ayar noktası olarak ~% 2 kesimini kullanın (burada, Ana Panelde Ayar Noktası 980 mV girin).
- AFM yazılımını kullanarak ayar noktasını düşürerek AFM ucu numune yüzeyi ile iyice tutun. Serbest düzey aşamanın hemen altındaki konsol salınım fazı (Toplam Faz ve Sapma Ölçer penceresinde) (çekmeden önce, burada tipik olarak ~ 70) ile itici mod görüntüleme için amaç edin. Burada, serbest seviye amplitüdünün (1 V)% 70-80'i düzeyinde tipik nihai ayar noktalarını kullanın.
- Taramadan önce, Ana Kanal Panelinde kayıt için sinyalleri seçin. Yüksekliği (Ht) ve genliği (Am) seçin.
- Örnek taramaya başlayın (burada: Ana Panelde Tarama Yap penceresi). 4 μm x 4 μm veya 8 μm x 8 μm görüntü yüzey alanlarına 2,5 μm / s (Master Panel'de komut Tarama Hızı) gibi bir tarama hızı kullanın (Master Pane'de Tarama Boyutu girinL) 2,048 piksel çözünürlüğü veya 4,096 (Master Panel'de Tarama Noktaları ve Tarama Satırı) ile sırasıyla.
- Resim dosyasını herhangi bir değişiklik yapmadan kaydedin (Ana Kanal Panelinde Yok seçeneğini seçin / Düzlemi Kaydet).
- Daha fazla analiz için kayıtlı görüntüyü işleyin. Görüntüyü yükleyin (AFM Analiz menüsünde Kayıtlı Veri Göz At komutunu kullanın). Değiştirme Panelini açın (resmin üst bölümünde M tuşuna basın). Yükseklik görüntüsüne x ve y boyutlarında bir planefit uygulayın (uzantı HtR) (Modify Panel'deki Planefit penceresindeki XY komutunu, Planefit Order 3'ü seçin). Daha sonra Düzleştir Siparişini seçerek görüntüyü düzleştirin (Düzleştirme Penceresinde Düzleştir komutu Modify Panelinde Düzleştir komutunu seçin).
- Görüntüyü bir TIFF dosyası olarak dışa aktarın (daha fazla analiz için resmin üst kısmındaki Komutlara basın, 2,048 piksel çözünürlüğüne karşılık gelen TIFF Dışa Aktarma 2x seçeneğini seçin).
3. AFM Analizi
- Protein kompleks hacmi
- öncesiFarklı büyüklükteki komplekslerin farklı görünümlerini en üst düzeye çıkarmak için uygun bir renk şeması kullanarak, AFM yazılımındaki görüntülerin doğrudan görsel denetimi ile ilgili protein-DNA komplekslerini seçin (bkz. Şekil 2'deki farklı komplekslerin farklı renkleri ve boyutları; burada renk şeması SeaLandAndFire AFM yazılımı). XPD örnekleri için muhtemel kompleksler XPD (~ 95 kDa) ve p44 (~ 40 kDa) nispeten büyük, farklı moleküler kitleleri nedeniyle XPD / p44-DNA'nın yanı sıra XPD-DNA ve p44-DNA'yı da içeriyordu. .
- Kompleks türünü doğrulamak için DNA'daki tek tek protein zirvelerinin hacimlerini ölçün. Bu, farklı görüntü yazılımlarıyla (burada: AFM yazılımının kesit aracı) başarılabilir.
- Kesit penceresi imleçleri ile protein tepe kesitlerinin yüksekliğini (h) ve çapını (d) ölçün. Partikül bölümünün tabanına yakın çapı ölçün.
- Bezelye hacmini (V) belirleyinKs, basit bir matematiksel model kullanarak, örneğin , aşağıdaki formülü kullanarak, küresel bir başlık modelini temel alan:
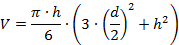
- Ölçülen hacimleri, yaklaşık protein molekül kütlesi haline getirin.
- Başlangıçta, bilinen molekül ağırlığına sahip bir dizi protein kullanarak moleküler ağırlığa (MW) dönüştürme hacmi için AFM sistemini kalibre edin (burada: 25 ve 851 veri noktası arasındaki toplam 12 deney, monomerik, dimerik olarak 5 farklı protein üzerinde gerçekleştirildi. , Trimerik ya da tetramerik haller) 4 , 19 , 20 . Biriktirme ve görüntü proteinleri (yukarıda tarif edildiği gibi 2.2 ve 2.3'te) ve yukarıda tarif edildiği gibi hacimlerini ölçün (3.1.2). Bilinen molekül ağırlığının üzerine hacim çizin. Ortaya çıkan grafik V ve MW arasında doğrusal bir ilişki gösterecektir, denklemi şu şekilde elde edilebilir:Verilere uyan bir çizgi. Burada kullanılan AFM sistemi için aşağıdaki ilişki elde edilmiştir 19 :
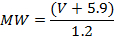
NOT: Bu adım her ölçümden önce tekrarlanmasına gerek yoktur, ancak yalnızca bir kez yapılır. MW kalibrasyon hacmi, hafifçe değişen çaplara sahip AFM probları kullanılarak benzer görüntüleme koşulları altında elde edilen görüntülere uygulanabilir. - Ölçülen hacimlerine (3.1.2) ve V'den MW'a kalibrasyona (3.1.3.1) dayanarak, moleküler içeriği hakkında bilgi veren protein komplekslerinin yaklaşık MW'sını belirleyin. Örneğin , XPD / p44 örnekleri için, p44-DNA kompleksleri (veya yalnızca DNA üst yapısından kaynaklanan tepe noktaları) ile tutarlı olarak, ~ 50 kDa, ~ 100 kDa ve ~ 140 kDa, XPD tek zirveleri ve XPD / p44 zirveleri elde edildi .
- Başlangıçta, bilinen molekül ağırlığına sahip bir dizi protein kullanarak moleküler ağırlığa (MW) dönüştürme hacmi için AFM sistemini kalibre edin (burada: 25 ve 851 veri noktası arasındaki toplam 12 deney, monomerik, dimerik olarak 5 farklı protein üzerinde gerçekleştirildi. , Trimerik ya da tetramerik haller) 4 , 19 , 20 . Biriktirme ve görüntü proteinleri (yukarıda tarif edildiği gibi 2.2 ve 2.3'te) ve yukarıda tarif edildiği gibi hacimlerini ölçün (3.1.2). Bilinen molekül ağırlığının üzerine hacim çizin. Ortaya çıkan grafik V ve MW arasında doğrusal bir ilişki gösterecektir, denklemi şu şekilde elde edilebilir:Verilere uyan bir çizgi. Burada kullanılan AFM sistemi için aşağıdaki ilişki elde edilmiştir 19 :
- DNA üzerinde protein kompleks pozisyonları
- DNA'yı belirleParça uzunlukları.
- DNA parçalarını AFM görüntülerinde takip edin, örneğin uygun bir görüntü analiz yazılımının serbest el çizgisi fonksiyonuyla (bkz. Malzeme Tablosu) ve çizginin uzunluğunu ölçün.
NOT: DNA yığınlarının yanı sıra görüntü kenar boşluklarına göre kesilmiş parçaları çıkarın. - Uygun veri analizi ve grafik yazılımı kullanarak ( örn . Malzeme Tablosu'na bakınız ) tüm deneyin DNA uzunluklarını histogramda çizin ve çizin .
- DNA parçalarının uzunluğunu belirlemek için veri analizi ve grafik yazılımında uzunluk dağılımlarını bir Gauss eğrisi ile uydurun. Eklenen DNA hedef alanının konumunu bilmek için (bkz. 1.1), Gauss eğrisinin merkezinden iki standart sapma içinde doğru uzunluktaki DNA fragmanlarını ekleyin (y = y 0 + z exp (-2 (xx c ) 2 / w 2 ), burada z bir norm faktörüdür ve x c ve w merkezi,Gaussian'ın tam maksimum yarı genişliği) daha ileri analizlerde.
NOT: Gauss eğrisinin merkezindeki DNA uzunluğu, DNA fragmanının teorik uzunluğuna yakın olmalıdır (0.34 nm / bp kullanılarak hesaplanmıştır). Teorik değerden% 10 daha kısa olan uzunluklar tipiktir ve muhtemelen AFM çözünürlük limitlerinden kaynaklanmaktadır.
- DNA parçalarını AFM görüntülerinde takip edin, örneğin uygun bir görüntü analiz yazılımının serbest el çizgisi fonksiyonuyla (bkz. Malzeme Tablosu) ve çizginin uzunluğunu ölçün.
- DNA substratlarındaki protein doruklarının konumlarını belirleyin.
- 3.2.1.1'de açıklandığı gibi yakın DNA fragmanı ucundaki protein zirvelerinin mesafesini ölçün ve DNA uzunluğunun fraksiyonu birimlerinde mesafeler elde etmek için toplam DNA uzunluğuna bölün.
- Ölçülen mesafeleri uygun bir veri analizi ve grafik yazılımı kullanarak bir histogramda çizin (bkz . Şekil 3 ). Kural olarak, n veri noktası için yaklaşık √ n kutu veren bir kutu boyutu seçin.
NOT: Burada DNA uçları ayırt edilemediğinden sadece DNA uzunluğunun fraksiyonu için 0.5 (DNA fragmanı merkeziMENT). DNA bitiş bağlama çalışmanın odak noktası olmadığından, 0 konumundan biraz sonra konum verilerini boşaltmaya başlayarak DNA bitiş analizlerini hariç tutun ( ör . Buradaki: 0,02'lik bir DNA uzunluk fraksiyonundan binleme).
- Protein kompleksi konum dağılımlarından hedef alan özgüllüğünü belirleyin.
- 3.2.1.3'deki gibi bir Gauss eğrisi ile konum dağılımındaki azami (güçlendirilmiş bağlanma olaylarını) uyuz, ancak arka plan bağlamanın yüksekliğine dayandırın (bkz. Şekil 3 ).
- Spesifikliği belirlemek için spesifik alana karşı S spesifik olmayan DNA zeminine (spesifik olmayan DNA sahalarına bağlı protein molekülleri) karşı hesaplayın 21
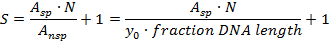
A sp : Belirli komplekslerin sayısı (Gauss eğrisinin altındaki alan)
A nsp: Belirgin olmayan komplekslerin sayısı (arka planın alanı, Yani (fraksiyon DNA uzunluğu) x (ortalama arka plan yüksekliği y 0 ); Burada kapsanan DNA uzunluğunun fraksiyonu 0.02 DNA uzunluğunda başlayan bir histogram için 0.48'dir)
N: olası DNA bağlama alanları sayısı (burada: DNA uçları hariç N = 914)
- DNA'yı belirleParça uzunlukları.
- DNA eğilme açıları
- Açı aletini uygun bir görüntü analiz yazılımında kullanarak, DNA omurgası boyunca merkezi olarak yerleştirilen ve protein zirvesine ortalanmış iki çizgi arasındaki açı β'yı ölçün (bkz . Ör. , Şekil 4'te yer alan ). İstatistiksel olarak ilgili sayıda protein-DNA kompleksi (> 50, ideal olarak> 100) için açılar ölçün 2 , 3 , 5 .
NOT: DNA bükme açısı 180 ° -β 2 , 3 , 5 olarak tanımlanır. - Veri analizi ve grafik yazılımı kullanma(Malzeme Tablosuna bakınız), DNA bükülme açısını binleyerek bükülme açısı dağılım histogramı üretmektedir.
- Eğilme açısı dağılımını bir Gauss eğrisi ile uydurun. Dağılımda birden fazla maksimum belirgindir, çoklu pik Gauss uyumu seçin. Gauss eğrisinin merkez (leri), belirli türlerin ortalama eğilme açısı durumunu verir.
- Örneğin farklı protein varyantlarının veya farklı protein kompleks türlerinin dağılımları için viraj açısında (Gauss fit (ler) in maxima) bir kayma belirgindeyse, bir protein testinin önem derecesini değerlendirmek için bir öğrenci t testi uygulayın. değişir.
- Açı aletini uygun bir görüntü analiz yazılımında kullanarak, DNA omurgası boyunca merkezi olarak yerleştirilen ve protein zirvesine ortalanmış iki çizgi arasındaki açı β'yı ölçün (bkz . Ör. , Şekil 4'te yer alan ). İstatistiksel olarak ilgili sayıda protein-DNA kompleksi (> 50, ideal olarak> 100) için açılar ölçün 2 , 3 , 5 .
Sonuçlar
Protein kompleks hacimlerine dayanan farklı karmaşık tipleri ayırt etme
Prokaryot NER helikaz UvrB'nin helikaz aktivitesi DNA bağlanması 22 , 23 tarafından uyarıldı. UvrB, iki adet tek ssDNA diziliminden birinin üzerine doğru yüklemek için DNA'da (DNA kabarcığı) eşleştirilmemiş bir bölgenin olmasını gerektirir. In vivo , bu DNA yapı...
Tartışmalar
Belirli hedef alanları içeren uzun DNA fragmanları üzerindeki proteinlerin bağlanma pozisyonlarının AFM istatistiksel analizleri, proteinin bu siteleri tanıması için kullanılan stratejilere ilginç ayrıntılar 2 , 3 , 4 , 5 , 6 gösterebilir. Ortaya çıkan konum dağılımlarını yorumlamak için DNA'daki hedeflerin pozisyonları tam olarak ...
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
PUC19N, CPD içeren oligonükleotidler ve p44 sırasıyla Samuel Wilson, Korbinian Heil ve Thomas Carell, ve Gudrun Michels ve Caroline Kisker tarafından nazikçe sağlandı. Bu çalışma Deutsche Forschungsgemeinschaft (DFG) FZ82 ve TE-671 / 4'ten BT'ye hibelerle desteklendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Molecular Force Probe (MFP) 3D | Asylum Research | N/A | atomic force microscope (AFM) |
| Precision 390 | DELL | N/A | computer |
| ThermoMixer and 1.5 mL block | Eppendorf | 5382000015 | heat block for DNA preparation |
| Rotilabo Block-Heater H 250 & blocks for 0.5 mL tubes | Carl Roth GmbH | Y264.1 & Y267.1 | heat block for protein-DNA incubations |
| Mini-Sub Cell GT | Bio-Rad Laboratories GmbH | 1704467 | electrophoresis chamber with gel caster and power supply |
| Power Pac Basic | Bio-Rad Laboratories GmbH | 1645050 | electrophoresis power supply |
| Centifuge 5415 D with rotor | Eppendorf | 2262120-3 | table centrifuge |
| Ultra-Lum electronic UV transillumonator MEB-15 | Ultralum | 900-1322-02 | UV irradiation table |
| NanoDrop ND-1000 | VWR International / PEQLAB Biotechnologie GmbH | N/A | UV spectrophotometer |
| TKAX-CAD with 0.2 μm capsule filter | Unity Lab Services | N/A | water deionization and filter unit |
| Name | Company | Catalog number | Comments |
| Software | |||
| MFP software on Igor Pro | Asylum Research | N/A | AFM software |
| ImageJ (open source Java image processing) | NIH Image | N/A | Image analysis software |
| Excel (Microsoft Office) | Microsoft Corporation | N/A | data analysis software |
| Origin9 / Origin2016 | OriginLab Corporation | N/A | statistical data analysis and graphing software |
| Name | Company | Catalog number | Comments |
| Material | |||
| OMCL-AC240TS | Olympus | OMCL-AC240TS | AFM cantilevers |
| grade V-5 muscovite | SPI Supplies | 1805 | mica sheets |
| Amicon Ultra 0.5 mL 50k Ultracell | Millipore Ireland Ltd. | UFC505096 | centrifuge filters |
| NucleoSpin Extract II | Macherey-Nagel GmbH | 740 609.250 | Agarose gel extraction kit |
| Rotilabo cellulose paper type 111A | Carl Roth GmbH | AP59.1 | AFM deposition blotting paper |
| Anatop 25 (0.02 μm) | Whatman GmbH | 6809-2102 | syringe filter |
| SSpI, BspQI | New England Biolabs (NEB) | R0132, R0712 | restriction enzymes for DNA substrate preparation |
| XhoI, BglII | R0146, R0144 | restriction enzymes for DNA preparation controls | |
| nicking restriction enzyme Nt.BstNBI | New England Biolabs (NEB) | R0607 | nickase |
| T4 DNA ligase | New England Biolabs (NEB) | M0202S | Ligase |
| Tris, HEPES | Carl Roth GmbH | 4855, 9105 | buffer chemicals |
| NaCl, MgCl2, KCl, MgAcetate | Carl Roth GmbH | 3957, HN03, HN02, P026 | salt chemicals |
| NaAc | Sigma-Aldrich Chemie GmbH | 32318 | salt chemicals |
| DTT, TCEP, EDTA | 6908, HN95, 8040 | chemicals/reagents | |
| agarose, acetic acid, HCl | Carl Roth GmbH | 2267, 3738, K025 | reagents |
| ATP | Carl Roth GmbH | K054 | nucleotides |
| oligonucleotide #1 in Table 1 | Biomers | custom | complementary DNA oligonucleotide |
| oligonucleotides #2, #3, and #6 in Table 1 | Integrated DNA Technologies (IDT) | custom | fluorescein containing oligonucleotides |
| oligonucleotides #4 and #5 in Table 1 | private (available from e.g. TriLink or GlenResearch) | CPD containing oligonucleotides | |
| SafeSeal reaction tube 0.5 mL and 1.5 mL | Sarstedt | 72.704 and 72.706 | incubation tubes |
| GeneRuler 1 kb | Thermo Scientific | SM0311 | DNA ladder |
| 6x concentrate gel loading dye purple | New England Biolabs (NEB) | 51406 | DNA loading dye |
| Midori Green | Nippon Genetics Europe GmbH | 999MG28055 | DNA stain |
Referanslar
- Janicijevic, A., Ristic, D., Wyman, C. The molecular machines of DNA repair: scanning force microscopy analysis of their architecture. J. Microsc. 212 (3), 264-272 (2003).
- Wang, H., et al. DNA bending and unbending by MutS govern mismatch recognition and specificity. Proc. Natl. Acad. Sci. USA. 100 (25), 14822-14827 (2003).
- Tessmer, I., et al. Mechanism of MutS searching for DNA mismatches and signaling repair. J. Biol. Chem. 283 (52), 36646-36654 (2008).
- Wagner, K., Moolenaar, G., van Noort, J., Goosen, N. Single-molecule analysis reveals two separate DNA-binding domains in the Escherichia coli UvrA dimer. Nucleic Acids Res. 37 (6), 1962-1972 (2009).
- Buechner, C. N., et al. Strand-specific recognition of DNA damages by XPD provides insights into nucleotide excision repair substrate versatility. J Biol. Chem. 289 (6), 3613-3624 (2014).
- Van der Linden, E., Sanchez, H., Kinoshita, E., Kanaar, R., Wyman, C. RAD50 and NBS1 form a stable complex functional in DNA binding and tethering. Nucleic Acids Res. 37 (5), 1580-1588 (2009).
- Fuentes-Perez, M. E., Dillingham, M., Moreno-Herrero, F. AFM volumetric methods for the characterization of proteins and nucleic acids. Methods. 60, 113-121 (2013).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Miyagi, A., Lyubchenko, Y. L. Specificity of binding of single-stranded DNA-binding protein to its target. Biochemistry. 51, 1500-1509 (2012).
- Wirth, N., et al. Conservation and Divergence in Nucleotide Excision Repair Lesion Recognition. J. Biol. Chem. 291 (36), 18932-18946 (2016).
- Kuper, J., Kisker, C. Damage recognition in nucleotide excision DNA repair. Curr. Opin. Struct. Biol. 22, 88-93 (2012).
- Buechner, C. N., Tessmer, I. DNA substrate preparation for atomic force microscopy studies of protein-DNA interactions. J. Mol. Recognit. 26 (12), 605-617 (2013).
- Sun, Z., Tan, H. Y., Bianco, P. R., Lyubchenko, Y. L. Remodeling of RecG Helicase at the DNA Replication Fork by SSB Protein. Sci. Rep. 5, 9625 (2015).
- Billingsley, D. J., Bonass, W. A., Crampton, N., Kirkham, J., Thomson, N. H. Single-molecule studies of DNA transcription using atomic force microscopy. Phys. Biol. 9 (2), 021001 (2012).
- Maurer, S., Fritz, J., Muskhelishvili, G., Travers, A. RNA polymerase and an activator form discrete subcomplexes in a transcription initiation complex. EMBO J. 25 (16), 3784-3790 (2006).
- Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Theis, K., Chen, P. J., Skorvaga, M., Van Houten, B., Kisker, C. Crystal structure of UvrB, a DNA helicase adapted for nucleotide excision repair. EMBO J. 18 (24), 6899-6907 (1999).
- Kuper, J., et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 12 (9), e1001954 (2014).
- Shlyakhtenko, L. S., et al. Silatrane-based surface chemistry for immobilization of DNA, protein-DNA complexes and other biological materials. Ultramicroscopy. 97, 279-287 (2003).
- Roth, H. M., et al. XPB helicase regulates DNA incision by the Thermoplasma acidophilum endonuclease Bax1. DNA Repair. 11 (3), 286-293 (2012).
- Ratcliff, G. C., Erie, D. A. A Novel Single-Molecule Study to Determine Protein-Protein Association Constants. J. Am. Chem. Soc. 123 (24), 5632-5635 (2001).
- Yang, Y., Sass, L. E., Du, C., Hsieh, P., Erie, D. A. Determination of protein-DNA binding constants and specificities from statistical analyses of single molecules: MutS-DNA interactions. Nucleic Acids Res. 33 (13), 4322-4334 (2005).
- Caron, P. R., Grossman, L. Involvement of a cryptic ATPase activity of UvrB and its proteolysis product, UvrB* in DNA repair. Nucleic Acids Res. 16 (22), 10891-10902 (1988).
- Wang, H., et al. UvrB domain 4, an autoinhibitory gate for regulation of DNA binding and ATPase activity. J. Biol. Chem. 281 (22), 15227-15237 (2006).
- Chammas, O., Billingsley, D. J., Bonass, W. A., Thomson, N. H. Single-stranded DNA loops as fiducial markers for exploring DNA-protein interactions in single molecule imaging. Methods. 60 (2), 122-130 (2013).
- Truglio, J. J., et al. Structural basis for DNA recognition and processing by UvrB. Nat. Struct. Mol. Biol. 13 (4), 360-364 (2006).
- Kuper, J., Wolski, S. C., Michels, G., Kisker, C. Functional and structural studies of the nucleotide excision repair helicase XPD suggest a polarity for DNA translocation. EMBO J. 31 (2), 494-502 (2012).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Goosen, N. The presence of two UvrB subunits in the UvrAB complex ensures damage detection in both DNA strands. EMBO J. 21 (15), 4196-4205 (2002).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Hoeijmakers, J. H., Goosen, N. Architecture of nucleotide excision repair complexes: DNA is wrapped by UvrB before and after damage recognition. EMBO J. 20 (3), 601-611 (2001).
- Moolenaar, G. F., et al. The Role of ATP Binding and Hydrolysis by UvrB during Nucleotide Excision Repair. J. Biol. Chem. 275, 8044-8050 (2000).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır