Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Атомно-силовая микроскопия Исследования распознавания повреждения ДНК при восстановлении эксцизии нуклеотидов
* Эти авторы внесли равный вклад
В этой статье
Резюме
Here, the study of different DNA lesion recognition approaches via single molecule AFM imaging is demonstrated with the nucleotide excision repair system as an example. The procedures of DNA and protein sample preparations and experimental as well as analytical details for the AFM experiments are described.
Аннотация
AFM imaging is a powerful technique for the study of protein-DNA interactions. This single molecule method allows the simultaneous resolution of different molecules and molecular assemblies in a heterogeneous sample. In the particular context of DNA interacting protein systems, different protein complex forms and their corresponding binding positions on target sites containing DNA fragments can thus be distinguished. Here, an application of AFM to the study of DNA lesion recognition in the prokaryotic and eukaryotic nucleotide excision DNA repair (NER) systems is presented. The procedures of DNA and protein sample preparations are described and experimental as well as analytical details of the experiments are provided. The data allow important conclusions on the strategies by which target site verification may be achieved by the NER proteins. Interestingly, they indicate different approaches of lesion recognition and identification for the eukaryotic NER system, depending on the type of lesion. Furthermore, distinct structural properties of the two different helicases involved in prokaryotic and eukaryotic NER result in and explain the different strategies observed for these two systems. Importantly, these experimental and analytical approaches can be applied not only to the study of DNA repair but also very similarly to other DNA interacting protein systems such as those involved in replication or transcription processes.
Введение
Атомно-силовая микроскопия (АСМ) является мощным методом анализа взаимодействий белок-ДНК 1 , 2 , 3 , 4 , 5 , 6 , 7 , 8 , 9 . Это требует только небольшого количества материала образца, чтобы непосредственно визуализировать гетерогенные образцы с разрешением на уровне одной молекулы. Неоднородность может быть результатом различных конформационных или олигомерных состояний белка. В частности, в контексте образцов белковой ДНК белковые комплексы могут отображать различные стехиометрии и / или конформации, индуцированные связыванием ДНК в целом или связываясь с конкретным сайтом-мишенью в ДНК. Гетерогенные образцы также могут содержать два (или более) различных вида белков и различный белковый комплексФормы ( например , состоящие только из одного типа белковых и гетеромерных комплексов) могут по-разному взаимодействовать с ДНК. Обсуждаемые здесь исследования используют AFM-визуализацию в воздухе на статических, высушенных образцах репарационных ДНК-белков, связанных с длинными фрагментами ДНК (~ 900 пар оснований, bp), которые содержат поражение, которое представляет собой мишень этих белков. Высокое молекулярное разрешение AFM позволяет различать различные типы белковых комплексов и определять позиции связывания белков на фрагментах ДНК. Важно отметить, что повреждения вносятся в ДНК-субстраты в четко определенных положениях. Поскольку положение участка поражения в ДНК известно, распределения белков, связанных с ДНК, обеспечивают понимание различительных (распознающих) свойств распознавания поражения (различных) белковых комплексов, например , насколько хорошо они распознают определенный тип поражения (по сравнению К неразрушенной ДНК) 2 , 3 , 4 , 5 , 6 . Их положения в ДНК также позволяют различать белковые комплексы, специфически связанные с поражениями и комплексами, связанными неспецифически в другом месте ДНК. Отдельная характеристика этих различных сложных типов (комплексы, специфически связанные с повреждением против неспецифических комплексов) может выявить потенциальные конформационные изменения в комплексах, индуцированных при идентификации целевого сайта.
Рекомбинантные белки репарации ДНК - это геликазы, которые ответственны за распознавание поражения в пути восстановления иссекающих нуклеотидов (NER). В бактериях NER достигается белками UvrA, UvrB и UvrC. UvrA отвечает за первичное зондирование поражения в ДНК-сканирующем комплексе UvaA 2 / UvrB 2 . После проверки на повреждение UvrB этот комплекс превращается в мономерный UvrB, связанный на участке повреждения, и этот специфический комплекс может затем набирать pРокариотическая эндонуклеаза NER UvrC. UvrC вырезает короткий (12-13 NT) участок одноцепочечной ДНК (оцДНК), содержащей поражение. Отсутствующее растяжение затем снова заполняется ДНК-полимеразой. Наконец, лигаза ДНК уплотняет вновь синтезированное растение с исходной ДНК 9 , 10 . У эукариот большинство белков каскада NER являются частью большого, мультимерного комплекса транскрипционного фактора II H (TFIIH). После первичного обнаружения поражения через тримерный комплекс CEN2-XPC-HR23B, TFIIH рекрутируется в целевой участок ДНК. Когда XPD внутри комплекса проверяет наличие поражения NER-мишени, эукариотические эндонуклеазы NER XPG и XPF рекрутируются для акциза короткого (24-32 nt) участка оцДНК, содержащего поражение 9,10. Здесь, в частности, изучались геликазы UvrB и XPD из прокариот и эукариотических NER, соответственно. Эти геликазы нуждаются в непарномДНК (пузырь ДНК) наматывается на одну из двух одиночных нитей ДНК и затем транслоцируется вдоль этой цепи, подпитываемой гидролизом АТФ. В дополнение к повреждениям ДНК, пузырь ДНК был, следовательно, введен в субстраты, которые функционируют как место загрузки белков.
Процедура получения специфических субстратов ДНК поражения описана ранее 11 . Для этого требуется кольцевая ДНК-конструкция (плазмида) с двумя близко расположенными рестрикционными сайтами для нимазы. В контексте этого исследования была использована плазмида pUC19N (2729 bp) (создана лабораторией С. Вильсона, NIEHS). Эта плазмида содержит три близко расположенные рестрикционные сайты для нимазы Nt.BstNBI, которые образуют 48-нуклеотидное (nt) растяжение. После инкубации с нимазой участок ссДНК между этими сайтами можно удалить и заменить олигонуклеотидом, содержащим любую целевую особенность. После каждой стадии полное ферментативное расщепление испытывают через агарозный гельэлектрофорез. Никеированная кольцевая ДНК может быть различена из-за ее более низкой электрофоретической подвижности по сравнению с исходной суперспирализованной плазмидой. Промежуток ДНК и замещение удаленной вытяжки специфическим олигонуклеотидом субстрата можно оценить путем переваривания ферментом рестрикции, который вырезает субстрат исключительно в области между никами. Таким образом, линеаризация круговой плазмиды ферментом будет подавлена для разрыхленной ДНК и восстановлена после вставки специфического олигонуклеотида. Наконец, два сайта редукции эндонуклеазами (в идеале одиночные резцы) позволяют получить линейный ДНК-субстрат с желаемой длиной и с определенным сайтом-мишенью в определенном положении, а также пузырь ДНК на расстоянии от очага поражения либо в 5 'Или 3' направлении.
Распознавание поражений геликазными НЕР можно исследовать с помощью визуализации АСМ. Замедленная транслокация ДНК геликаз при tОн участок поражения виден как пик в расположении положения белка на ДНК и указывает на распознавание поражения. Поскольку транслокация ДНК этих геликаз является, кроме того, направленной, с полярностью 5'-к-3 ', зависимость распознавания поражения от положения места загрузки (пузырь ДНК вверх или вниз по течению от повреждения) также указывает, является ли поражение преимущественно распознаваемым На транслоцированной или на противоположной, неперемещенной нити оцДНК 5 , 9 . В следующих разделах будут использованы используемые методы, и основные результаты этих экспериментов будут кратко обсуждаться. Важно отметить, что аналогично примерной работе по восстановлению ДНК, показанной здесь, АФМ-визуализация может быть применена к изучению различных взаимодействующих с ДНК систем, таких как репликация ДНК или транскрипция 8 , 12 , 13 , 14 .
протокол
1. Подготовка образцов
- Приготовление ДНК субстратов 11
- Создание щели оцДНК в плазмиде
- Полностью переварить образец плазмиды (здесь: модифицированный pUC19, pUC19N) в реакционной трубке с подходящей никазой (здесь: Nt.BstNBI) с последующей инактивацией тепла инактивацией фермента, используя условия в соответствии с протоколом производителя (см. презентация). Начните с ~ 50 мкл и ~ 500 нМ плазмиды с достаточным выходом.
- Проверьте наложение плазмиды электрофорезом в агарозном геле на разбавленных образцах (~ 20 нМ) 15 . Наденьте перчатки для защиты от ДНК-связывающего красителя, используемого для визуализации ДНК.
ПРИМЕЧАНИЕ. Различные электрофоретические подвижности позволяют различать никелевую (релаксированную) и сверхскрученную кольцевую плазмидную ДНК ( рис. 1 ). - Удалите вырезанное оцДНК-стрейч (между сайтами ников) из плазмиды с помощьюИнкубацию с 10-кратным избытком комплементарного олигонуклеотида (олигонуклеотид 1 в таблице 1 ), встряхиванием при 300 об / мин в течение 30 мин вблизи температуры плавления олигонуклеотида (здесь: 68 ° С для pUC19N) в тепловом блоке ( фиг.1 ).
- Отделяют плазмиду с зазором от меньших фрагментов ДНК с использованием фильтра с молекулярной массой 50 кДа (MWCO) центрифугированием в течение 10 мин при 10000 мкг в таблетной центрифуге ( фиг.1 ). Чтобы извлечь концентрированную ДНК из фильтра, инвертируйте фильтр и вставьте в новую 1,5 мл реакционную трубку. Центрифуга в течение 3 мин при 1000 х g.
- Заполните полученный концентрированный образец ДНК до 500 мкл деионизированной, отфильтрованной водой, добавьте дополнительный ~ 5-кратный избыток комплементарного олигонуклеотида и повторите шаги 1.1.1.3 и 1.1.1.4 не менее 3 раз.
- Протестируйте для полного разрыва ДНК путем инкубации с подходящим рестрикционным ферментом (здесь: XhoI или BglII), используя conditiВ соответствии с описанием производителя. Используйте образцы ДНК с разведенным (никированным или пропахшим) (~ 20 нМ). Проведите электрофорез в агарозном геле, чтобы отличить линеаризованную плазмиду (не содержащую щели оцДНК) и неврезанную ДНК (прорезанную ДНК), используя ДНК с никами в качестве положительного контроля (включая опорную лестницу для ДНК, см. Рисунок 1 ). Носить перчатки.
- Повторное заполнение щели модифицированными олигонуклеотидами оцДНК
- Отцеливают прорезанную плазмиду путем инкубации с 25-кратным избытком 5'-фосфорилированного олигонуклеотида, который содержит специфический целевой сайт (ы) выбора, инкубируя при ~ 45 ° C в течение 4 ч ( фиг.1 ). Здесь используют 48 нт оцДНК, содержащую поражение, либо аддуктированный с флюоресцеином тимин, либо димер циклобутанового пиримидина (CPD), и, кроме того, короткую (8 NT) некомплементарную последовательность для продуцирования ДНК-пузырьков (см. Таблицу 1 ).
- Ковалентно связывают отожженную вставку с плазмидой с помощью инкубаС ДНК-лигазой Т4 в течение ночи при комнатной температуре в соответствии с протоколом производителя ( рис. 1 ). Для этой реакции добавляют ATP, содержащий концентрированный буферный основной раствор, для получения подходящих буферных условий для лигазы ( например , используют буфер для реакции UvrB: 50 мМ Tris-HCl pH 7,5, 10 мМ MgCl 2 , 50 мМ KCl, 5 мМ DTT, 1 мМ АТФ).
- Тест на введение специфического олигонуклеотида в плазму, заполненную пробелами, путем переваривания разбавленных образцов подходящим рестрикционным ферментом (тот же, что в 1.1.1.6) в соответствии с протоколом производителя, с последующим электрофорезом в агарозном геле (как в 1.1.1.6, Figure 1 ).
- Приготовление линейного ДНК-субстрата
- Переваривают модифицированную плазмидную ДНК рестрикционными ферментами (в идеале с единственным сайтом рестрикции в плазмиде), используя условия, рекомендованные производителем ( рисунок 1 ). Этот шаг дает линейные фрагменты, содержащиеВставляя вставленную модификацию в определенное положение. Здесь используют срезы SspI и BspQI в положениях 613 и 255 bp выше и ниже по течению от вставки, соответственно, в результате чего фрагмент ДНК размером 916 пар оснований, содержащий специфический участок поражения, составляет ~ 30% от длины ДНК.
ПРИМЕЧАНИЕ. Для экспериментов по визуализации АСМ, как описано здесь, подходящими субстратами являются фрагменты ДНК длиной ~ 200 п.н. и ~ 2000 п.о. - Изолируйте целевой фрагмент с помощью электрофореза в агарозном геле и экстракции геля с использованием коммерческого набора. Надевайте защитные перчатки.
- Необязательно, разрезайте агарозный гель скальпелем, чтобы отделить лестницу ДНК и разбавленные пробы (первые две дорожки) от дорожек, содержащих концентрированную ДНК, которую необходимо очистить. Только подвергайте гелевую часть первыми двумя дорожками воздействию ультрафиолетового излучения, помещая только эту часть геля на УФ-стол.
- Вырежьте полоску, соответствующую фрагменту выбора, с помощью скальпеля. Повторно объединить две части геля (с УФ-ярлыкале). Используйте положение вырезанной полосы из разбавленного образца в качестве ориентации для положения полосы желаемого ДНК-субстрата. Учитывайте более высокую концентрацию, вырезая немного более широкие срезы, чем для разбавленного контроля.
ПРИМЕЧАНИЕ. Поскольку здесь было обнаружено распознавание УФ-повреждений, при использовании этого подхода в субстрате ДНК было тщательно предотвращено введение дополнительных УФ-лучей посредством УФ-облучения.
- Вырежьте полоску, соответствующую фрагменту выбора, с помощью скальпеля. Повторно объединить две части геля (с УФ-ярлыкале). Используйте положение вырезанной полосы из разбавленного образца в качестве ориентации для положения полосы желаемого ДНК-субстрата. Учитывайте более высокую концентрацию, вырезая немного более широкие срезы, чем для разбавленного контроля.
- Вычислить концентрацию ДНК c в поглощении на 260 нм, измеренную с помощью спектрофотометра UV-Vis с использованием закона Ламберта-Биера с коэффициентом средней молярной экстинкции двойной двунитевой ДНК (dsDNA) ε 260 нм -6 700 М - 1 см - 1 на 1 п.о .:
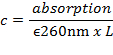
Где L - длина пути (длина измерительной камеры, как правило, 1 см).
- Переваривают модифицированную плазмидную ДНК рестрикционными ферментами (в идеале с единственным сайтом рестрикции в плазмиде), используя условия, рекомендованные производителем ( рисунок 1 ). Этот шаг дает линейные фрагменты, содержащиеВставляя вставленную модификацию в определенное положение. Здесь используют срезы SspI и BspQI в положениях 613 и 255 bp выше и ниже по течению от вставки, соответственно, в результате чего фрагмент ДНК размером 916 пар оснований, содержащий специфический участок поражения, составляет ~ 30% от длины ДНК.
- Создание щели оцДНК в плазмиде
- Экспрессия и очистка белков
- Рекомбинантно экспрессировать UvrB в E. coli и очищать белок посредством стандартного сродства к хитиновым шарикам 15 и эксклюзионной хроматографии 15, как описано ранее 16 .
ПРИМЕЧАНИЕ: ген uvrB от Bacillus caldotenax был клонирован в вектор pTYB1. - Express XPD в Е. coli и очищают белок через стандартное сродство к никелю IDA 15 и эксклюзионную хроматографию 15 с последующей анионообменной хроматографией 15 , как описано ранее 17 .
ПРИМЕЧАНИЕ : ген для белка группы D Chaetomium thermophilum Xeroderma piggyosum (XPD) был клонирован в вектор pBADM11. C. thermophilum p44 был добрым подарком из лаборатории Кэролайн Кискер, выраженной и пуриFied, как описано 17 .
- Рекомбинантно экспрессировать UvrB в E. coli и очищать белок посредством стандартного сродства к хитиновым шарикам 15 и эксклюзионной хроматографии 15, как описано ранее 16 .
2. Эксперимент АСМ
- Базовые приготовления
- Необязательно предварительно инкубировать ДНК-субстрат при 65 ° С в течение 10 мин в тепловом блоке для удаления потенциальных кристаллов микросолей, которые могли образоваться во время хранения в холодильнике.
- Готовят реакционный буфер (ы) при 10-кратной концентрации (10х буферы).
ПРИМЕЧАНИЕ. Буфер для XPD-реакции при концентрации 1х содержал 20 мМ Трис-HCl pH 7,5, 10 мМ KCl, 5 мМ MgCl 2 , 1 мМ TCEP, 2 мМ АТФ; 1x буферный раствор UvrB содержал 50 мМ Tris-HCl pH 7,5, 50 мМ KCl, 10 мМ MgCl 2 , 5 мМ DTT и 1 мМ АТФ. - Предварительно инкубировать белки в более высокой концентрации в подходящих условиях инкубации для усиления комплексообразования. Предварительно развести небольшой объем (1 мкл) отдельных белков в 1x белковом реакционном буфере до желаемой концентрации и смешать небольшие объемы ( например , 1 мкл) tОн индивидуальных белковых растворов в 0,5 мл реакционной трубки.
- Поместите трубку в тепловой блок для температур инкубации выше комнатной температуры. Выберите коэффициент концентрации в зависимости от ожидаемой комплексной стехиометрии, эквимолярной или соответствующей концентрации. Здесь инкубируют 1 мкл XPD (20 мкМ в 1 х XPD реакционном буфере) и 1 мкл p44 (20 мкМ в 1х XPD реакционном буфере) при 10 мкМ каждый в течение 10 мин при 37 ° С.
- Инкубируйте образцы в подходящих концентрациях белка и ДНК в белковом реакционном буфере в 0,5 мл реакционной пробирке. Здесь используют 500 нМ UvrB или 1 мкМ XPD + 1 мкМ р44 и 100 нМ ДНК. Пипетируйте небольшие объемы для сохранения материала, например , 0,25-0,5 мкл белка (предварительно разведенного до 10-кратной концентрации инкубации) и ДНК в общем объеме 2,5-5 мкл 1x белкового реакционного буфера. Здесь, инкубировать в течение 30 мин при 37 ° С в тепловом блоке. Спирально вниз в реакционной трубе кратко (~ 1 с) в столовом центрифугеE для обеспечения смешивания небольших объемов.
- Отбор образцов
- Подготовьте слюдяную подложку: отрежьте кусочек слюды приблизительно 1 x 1 см 2 от более крупных полосок с помощью скальпеля. Слейте верхние слои многослойной слюдяной части минерала с помощью клейкой ленты, чтобы выявить чистую, плоскую и атомарно гладкую поверхность подложки.
ПРИМЕЧАНИЕ: кусок слюды можно повторно использовать для нескольких экспериментов путем удаления дополнительного слоя (слоев). - Подготовьте буфер для осаждения АСМ деионизированной, отфильтрованной водой, например , 25 мМ HEPES pH 7,5, 25 мМ Na-ацетата, 10 мМ Mg-ацетата. Фильтр через шприцевой фильтр 0,02 мкм.
ПРИМЕЧАНИЕ. Двухвалентные катионы в буфере осаждения служат для хелатного взаимодействия отрицательно заряженных молекул ДНК с поверхностью слюды, которая также отрицательно заряжается при нейтральном рН. Если относительно высокая концентрация ионов Mg 2+ в буфере осаждения создает проблему для конкретной системы белок-ДНК, альтернативно,Поверхность слюды может быть предварительно загружена аминогруппами с использованием химии на основе силатрана для обеспечения положительных поверхностных зарядов для закрепления 18 . Затем осаждение образца может быть проведено в буфере, который не содержит или содержит только небольшие количества двухвалентных катионов. - Разбавьте пробу (см. 2.1) для немедленного осаждения на слюду в буфере осаждения. Вложите небольшой объем (здесь: 20 мкл).
ПРИМЕЧАНИЕ: Коэффициенты разведения зависят от концентрации образца. Здесь разбавленные образцы 50-100х. Как правило, ~ 1 нМ ДНК приводит к хорошему покрытию поверхности для ~ 1000 пар оснований. - Тщательно прополощите образец три-четыре раза с помощью нескольких миллилитров фильтрованной деионизированной воды, залейте лишнюю жидкость и высушите в слабом потоке азота. Весь процесс от осаждения образца до высушенных образцов может быть выполнен в течение менее 30 с.
- Закрепите кусок слюды на предметном стекле микроскопа, используя клейкую ленту по краям.
ПРИМЕЧАНИЕ. Различные системы АСМ имеют dIfferent требования для крепления образца к этапу. Другие АСМ обладают магнитными ступенями, а части слюды закреплены на магнитных дисках, например , с использованием термического клея. Подробности используемой здесь системы АСМ можно найти в таблице материалов.
- Подготовьте слюдяную подложку: отрежьте кусочек слюды приблизительно 1 x 1 см 2 от более крупных полосок с помощью скальпеля. Слейте верхние слои многослойной слюдяной части минерала с помощью клейкой ленты, чтобы выявить чистую, плоскую и атомарно гладкую поверхность подложки.
- Отображение AFM
- Поместите образец (см. 2.2.5) централизованно на этапе AFM и закрепите подставку для микроскопа на сцене магнитными подушечками.
- Вставьте наконечник АСМ в держатель наконечника. Используйте кантилеверы с острым (<10 нм) AFM-зонд, подходящим для колебательных, прерывистых контактных изображений в воздухе. Используйте , например, AFM-зонды, как указано в таблице материалов . Здесь (в зависимости от АСМ) закрепите наконечник в держателе под зажимом, затянув зажимной винт (затяните палец). Вставьте держатель наконечника в измерительную головку AFM. Положите голову на спину для этого шага.
- Поместите измерительную головку AFM сверху образца. Детали зависят от модели AFM. Здесь mУбедитесь, что голова устойчиво стоит со своими ногами внутри углублений в кадре. Убедитесь, что слюда находится на той стадии, где кончик АСМ будет парить прямо над ней. Винты с микрометрами в правой части сцены обеспечивают точное позиционирование образца.
- Совместите лазер AFM на задней части кантилевера для оптимальной мощности сигнала с позиционно-чувствительного фотоприемника, на который отражается лазер. Детали зависят от модели AFM.
- Здесь поверните колеса с правой и с задней стороны измерительной головки AFM, чтобы отрегулировать положение x и y для AFM-лазера, чтобы направлять его центрально на конец кантилевера. Посмотрите сигнал отражения в окне видео AFM (если доступно, нажмите здесь значок камеры и выберите вход: Svideo).
- После грубого позиционирования оптимизируйте суммарный сигнал детектора (Sum in Sum и Deflection Meter window в программном обеспечении AFM) путем тонкой настройки положения лазера двумя колесами (остановитесь в конце кантилевера, суммой signaL зависит от АСМ и типа кантилевера, здесь: цель для суммы> 5).
- Нулевой сигнал разности от верхнего и нижнего диодов матрицы детектора (здесь: сигнал отклонения в окне суммирования и отклонения) путем направления лазерного отражения АСМ на центр детектора (здесь: поверните колесо в левой части измерения АСМ глава).
ПРИМЕЧАНИЕ. Отклонения от нуля указывают на отклонение кантилевера из-за поверхностных взаимодействий, которые AFM преобразует в информацию о высоте. - Определите резонансную частоту кантилевера с помощью частотной настройки, реализованной в программном обеспечении AFM (здесь: команда автоматической настройки в окне Master Panel / Tune). Выберите амплитуду, соответствующую входу 1 В для пьезодатчика, который управляет колебаниями кантилевера. Установите частоту колебаний немного (5%) ниже резонансной частоты и обнулите фазу колебаний.
- Приблизьте наконечник к поверхности образца, используя сырой модE (здесь: команда Включить в окно главной панели) до достижения защитного параметра (уставки). Используйте ~ 2% отрезки амплитуды колебаний свободного уровня в качестве уставки (здесь введите контрольную точку 980 мВ в мастер-панели).
- Мелко зацепите наконечник АСМ с поверхностью образца, опустив заданное значение с помощью программного обеспечения AFM. Стремитесь к отображению отталкивающего режима с фазой осцилляций кантилевера (окно «Фаза в сумме» и «Отклонение») чуть ниже фазы свободного уровня (перед включением, здесь обычно ~ 70). Здесь используйте типичные конечные уставки порядка 70-80% от амплитуды свободного уровня (1 В).
- Перед сканированием выберите сигналы для записи на панели главного канала. Выберите высоту (Ht) и амплитуду (Am).
- Начните сканирование образцов (здесь: команда Do Scan in Master Panel). Используйте скорость сканирования, например, 2,5 мкм / с (команда Скорость сканирования на главной панели), чтобы получить области поверхности изображения 4 мкм × 4 мкм или 8 мкм × 8 мкм (введите размер сканирования в главной панелиЛ) с разрешением пикселов 2048 или 4096 (точки сканирования и линии сканирования на главной панели) соответственно.
- Сохраните файл изображения (команда «Сохранить изображение на главной панели») без изменений фитинга (выберите «Нет» на панели «Мастер-канал» / «Сохранить плоскость»).
- Для дальнейшего анализа обработайте сохраненное изображение. Загрузите изображение (команда Просмотр сохраненных данных в меню Анализ АСМ). Откройте панель Modify Panel (нажмите M в верхней части изображения). Примените плоскость в размерах x и y к изображению высоты (расширение HtR) (команда XY в окне Planefit в панели Modify, выберите Planefit Order 3). Затем сгладьте изображение (команда Flatten в окне Flatten в панели Modify), выбрав Flatten Order 3.
- Экспортируйте изображение в виде файла TIFF (нажмите «Команды» в верхней части изображения, выберите «Экспорт TIFF 2х», соответствующий разрешению 2048 пикселей) для дальнейшего анализа.
3. АФМ-анализ
- Объем белкового комплекса
- пред-Выбирать соответствующие комплексы белок-ДНК путем прямого визуального контроля изображений в программном обеспечении AFM с использованием подходящей цветовой схемы для максимизации различных проявлений комплексов разного размера (см. Различные цвета и размеры различных комплексов на рисунке 2 , здесь: цветовая схема SeaLandAndFire в Программное обеспечение AFM). Для образцов XPD возможные комплексы включали ДНК XPD / p44 , а также XPD-ДНК и p44-ДНК с отчетливо различными размерами из-за относительно больших молекулярных масс XPD (~ 95 кДа) и p44 (~ 40 кДа) ,
- Измерьте объемы отдельных пиков белка на ДНК для проверки сложного типа. Этого можно достичь с помощью другого графического программного обеспечения (здесь: инструмент раздела программного обеспечения АСМ).
- Измерьте высоту (h) и диаметр (d) участков пиков белка с помощью курсоров оконного окна. Измерьте диаметр близко к основанию частица.
- Определить объем (V) горохаKs, используя простые математические модели, например , используя следующую формулу, которая основана на модели шарового колпачка:
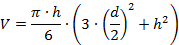
- Переведите измеренные объемы в приблизительную молекулярную массу белка.
- Первоначально откалибруйте систему AFM для преобразования объема в молекулярную массу (MW) с использованием ряда белков с известной молекулярной массой (здесь: всего было проведено 12 экспериментов, каждый из которых проводился между 25 и 851 точками данных на 5 различных белках в мономерных, димерных , Тримерные или тетрамерные состояния) 4 , 19 , 20 . Депозитные и имиджевые белки (как описано выше, в 2.2 и 2.3) и измеряют их объемы, как описано выше (3.1.2). Постройте объемы по их известным молекулярным весам. Результирующий график покажет линейную зависимость между V и MW, уравнение которой можно получить изЛиния, соответствующая данным. Для используемой здесь АСМ-системы было получено следующее соотношение: 19 :
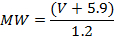
ПРИМЕЧАНИЕ. Этот шаг не должен повторяться перед каждым измерением, но выполняется только один раз. Калибровка объема до МВ может быть применена к изображениям, полученным в аналогичных условиях визуализации с использованием AFM-зондов с небольшим изменением диаметров. - Основываясь на измеренных объемах (3.1.2) и калибровке V-MW (3.1.3.1), определите приблизительную молекулярную массу белковых комплексов 20 , которая предоставляет информацию о ее молекулярном содержании. Например , для образцов XPD / p44 были получены ~ 50 кДа, ~ 100 кДа и ~ 140 кДа, в соответствии с комплексами ДНК p44 (или пиками, вызванными простой надстройкой ДНК), только пики XPD и пики XPD / p44 соответственно ,
- Первоначально откалибруйте систему AFM для преобразования объема в молекулярную массу (MW) с использованием ряда белков с известной молекулярной массой (здесь: всего было проведено 12 экспериментов, каждый из которых проводился между 25 и 851 точками данных на 5 различных белках в мономерных, димерных , Тримерные или тетрамерные состояния) 4 , 19 , 20 . Депозитные и имиджевые белки (как описано выше, в 2.2 и 2.3) и измеряют их объемы, как описано выше (3.1.2). Постройте объемы по их известным молекулярным весам. Результирующий график покажет линейную зависимость между V и MW, уравнение которой можно получить изЛиния, соответствующая данным. Для используемой здесь АСМ-системы было получено следующее соотношение: 19 :
- Положения белкового комплекса на ДНК
- Определить ДНКДлины фрагментов.
- Проследите фрагменты ДНК в AFM-изображениях, например , с помощью функции свободной руки от подходящего программного обеспечения анализа изображений (см., Например, Table of Materials) и измерьте длину линии.
ПРИМЕЧАНИЕ. Исключайте агрегаты ДНК, а также фрагменты, обрезанные по краям изображения. - Bin и начертите длины ДНК из всего эксперимента на гистограмме, используя подходящий анализ данных и графическое программное обеспечение (см. Таблицу материалов ).
- Подгоните распределение длин по гауссовой кривой в программном анализе и графическом анализе для определения длины фрагментов ДНК. Чтобы узнать положение вставленного участка ДНК-ДНК (см. 1.1), включайте только фрагменты ДНК с правильной длиной в пределах двух стандартных отклонений от центра кривой Гаусса (y = y 0 + z exp (-2 (xx c ) 2 / w 2 ), где z - нормальный фактор, а x c и w - центр иПолная максимальная полуширина гауссова) в дальнейших анализах.
ПРИМЕЧАНИЕ. Длина ДНК в центре кривой Гаусса должна быть близка к теоретической длине фрагмента ДНК (рассчитанная с использованием 0,34 нм / п.о.). Длины до 10% короче теоретического значения являются типичными и, вероятно, вызваны ограничениями разрешения АСМ.
- Проследите фрагменты ДНК в AFM-изображениях, например , с помощью функции свободной руки от подходящего программного обеспечения анализа изображений (см., Например, Table of Materials) и измерьте длину линии.
- Определить положения пиков белка на ДНК-субстратах.
- Измерьте расстояние пиков белка от более близкого конца фрагмента ДНК, как описано в 3.2.1.1, и разделите на полную длину ДНК, чтобы получить расстояния в единицах доли длины ДНК.
- Bin и постройте график измеренных расстояний в гистограмме, используя подходящий анализ данных и графическое программное обеспечение (см., Например , рис. 3 ). Как правило, выберите размер бункера, который дает приблизительно √n бункеров для n точек данных.
ПРИМЕЧАНИЕ: Так как концы ДНК не могут быть выделены здесь, график только фракции длины ДНК 0.5 (центр фрагмента ДНКМент). Поскольку связывание конца ДНК не является фокусом исследования, исключить концы ДНК из анализов, начав бин данные позиции немного после позиции 0 ( например , здесь: бинирование из доли ДНК длиной 0,02).
- Определить целевую специфичность сайта по распределению белковых комплексных позиций.
- Установите максимальное (усиленные вхождения связывания) в распределение по положению с кривой Гаусса, как в 3.2.1.3, но ногами на высоте привязки фона (см . Рис. 3 ).
- Вычислите специфичность S для конкретного участка по сравнению с неспецифическим фоновым ДНК (молекулы белка, связанные с неспецифическими сайтами ДНК), со следующей формулой 21
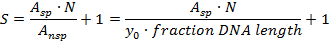
A sp : количество конкретных комплексов (площадь под гауссовой кривой)
A nsp : количество неспецифических комплексов (площадь фона, Т.е. (длина ДНК фракции) x (средняя высота фона y 0 ); Доля покрытой здесь длины ДНК составляет 0,48 для гистограммы, начинающейся с 0,02 длины ДНК)
N: количество возможных участков связывания ДНК (здесь: N = 914, исключая концы ДНК)
- Определить ДНКДлины фрагментов.
- Углы изгиба ДНК
- Используя угловой инструмент в подходящем программном обеспечении для анализа изображений, измерьте угол β между двумя линиями, расположенными в центре вдоль основной цепи ДНК и центрирующими на пике белка (см., Например , вставка на рисунке 4 ). Измерьте углы для статистически значимого числа комплексов белок-ДНК (> 50, в идеале> 100) 2 , 3 , 5 .
ПРИМЕЧАНИЕ. Угол изгиба ДНК определяется как 180 ° -β 2 , 3 , 5 . - Использование программного обеспечения для анализа и графического анализа данных(См. Таблицу материалов) создают гистограмму распределения угла изгиба, разбивая углы изгиба ДНК.
- Подгоните распределение угла изгиба кривой Гаусса. Если в распределении присутствует более одного максимума, выберите несколько пиков Гаусса. Центр (ы) гауссовой кривой (ы) дают (ют) среднее угловое состояние изгиба конкретного вида.
- Если сдвиг угла изгиба (s) (максимумы гауссовой посадки) проявляется для распределений, например, различных вариантов белка или разных видов сложных белков, применяют t-критерий ученика для оценки уровня значимости изменения.
- Используя угловой инструмент в подходящем программном обеспечении для анализа изображений, измерьте угол β между двумя линиями, расположенными в центре вдоль основной цепи ДНК и центрирующими на пике белка (см., Например , вставка на рисунке 4 ). Измерьте углы для статистически значимого числа комплексов белок-ДНК (> 50, в идеале> 100) 2 , 3 , 5 .
Результаты
Выделение различных комплексных типов на основе комплексных объемов белка
Геликазная активность прокариотной геликазной НЕРВ UvrB стимулируется связыванием ДНК 22 , 23 . UvrB требует неспаренной области в ДН?...
Обсуждение
AFM статистический анализ положения связывания белков на длинных фрагментах ДНК, которые содержат специфические сайты-мишени, может показать интересные подробности о конкретных стратегиях, используемых белком для распознавания этих сайтов 2 , 3 ,
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
PUC19N, CPD-содержащие олигонуклеотиды и p44 были любезно предоставлены Сэмюэлем Уилсоном, Корбинианом Хейлом и Томасом Карелл, Гудрун Михельс и Кэролайн Кискер соответственно. Эта работа была поддержана грантами Deutsche Forschungsgemeinschaft (DFG) FZ82 и TE-671/4 для ИТ.
Материалы
| Name | Company | Catalog Number | Comments |
| Molecular Force Probe (MFP) 3D | Asylum Research | N/A | atomic force microscope (AFM) |
| Precision 390 | DELL | N/A | computer |
| ThermoMixer and 1.5 mL block | Eppendorf | 5382000015 | heat block for DNA preparation |
| Rotilabo Block-Heater H 250 & blocks for 0.5 mL tubes | Carl Roth GmbH | Y264.1 & Y267.1 | heat block for protein-DNA incubations |
| Mini-Sub Cell GT | Bio-Rad Laboratories GmbH | 1704467 | electrophoresis chamber with gel caster and power supply |
| Power Pac Basic | Bio-Rad Laboratories GmbH | 1645050 | electrophoresis power supply |
| Centifuge 5415 D with rotor | Eppendorf | 2262120-3 | table centrifuge |
| Ultra-Lum electronic UV transillumonator MEB-15 | Ultralum | 900-1322-02 | UV irradiation table |
| NanoDrop ND-1000 | VWR International / PEQLAB Biotechnologie GmbH | N/A | UV spectrophotometer |
| TKAX-CAD with 0.2 μm capsule filter | Unity Lab Services | N/A | water deionization and filter unit |
| Name | Company | Catalog number | Comments |
| Software | |||
| MFP software on Igor Pro | Asylum Research | N/A | AFM software |
| ImageJ (open source Java image processing) | NIH Image | N/A | Image analysis software |
| Excel (Microsoft Office) | Microsoft Corporation | N/A | data analysis software |
| Origin9 / Origin2016 | OriginLab Corporation | N/A | statistical data analysis and graphing software |
| Name | Company | Catalog number | Comments |
| Material | |||
| OMCL-AC240TS | Olympus | OMCL-AC240TS | AFM cantilevers |
| grade V-5 muscovite | SPI Supplies | 1805 | mica sheets |
| Amicon Ultra 0.5 mL 50k Ultracell | Millipore Ireland Ltd. | UFC505096 | centrifuge filters |
| NucleoSpin Extract II | Macherey-Nagel GmbH | 740 609.250 | Agarose gel extraction kit |
| Rotilabo cellulose paper type 111A | Carl Roth GmbH | AP59.1 | AFM deposition blotting paper |
| Anatop 25 (0.02 μm) | Whatman GmbH | 6809-2102 | syringe filter |
| SSpI, BspQI | New England Biolabs (NEB) | R0132, R0712 | restriction enzymes for DNA substrate preparation |
| XhoI, BglII | R0146, R0144 | restriction enzymes for DNA preparation controls | |
| nicking restriction enzyme Nt.BstNBI | New England Biolabs (NEB) | R0607 | nickase |
| T4 DNA ligase | New England Biolabs (NEB) | M0202S | Ligase |
| Tris, HEPES | Carl Roth GmbH | 4855, 9105 | buffer chemicals |
| NaCl, MgCl2, KCl, MgAcetate | Carl Roth GmbH | 3957, HN03, HN02, P026 | salt chemicals |
| NaAc | Sigma-Aldrich Chemie GmbH | 32318 | salt chemicals |
| DTT, TCEP, EDTA | 6908, HN95, 8040 | chemicals/reagents | |
| agarose, acetic acid, HCl | Carl Roth GmbH | 2267, 3738, K025 | reagents |
| ATP | Carl Roth GmbH | K054 | nucleotides |
| oligonucleotide #1 in Table 1 | Biomers | custom | complementary DNA oligonucleotide |
| oligonucleotides #2, #3, and #6 in Table 1 | Integrated DNA Technologies (IDT) | custom | fluorescein containing oligonucleotides |
| oligonucleotides #4 and #5 in Table 1 | private (available from e.g. TriLink or GlenResearch) | CPD containing oligonucleotides | |
| SafeSeal reaction tube 0.5 mL and 1.5 mL | Sarstedt | 72.704 and 72.706 | incubation tubes |
| GeneRuler 1 kb | Thermo Scientific | SM0311 | DNA ladder |
| 6x concentrate gel loading dye purple | New England Biolabs (NEB) | 51406 | DNA loading dye |
| Midori Green | Nippon Genetics Europe GmbH | 999MG28055 | DNA stain |
Ссылки
- Janicijevic, A., Ristic, D., Wyman, C. The molecular machines of DNA repair: scanning force microscopy analysis of their architecture. J. Microsc. 212 (3), 264-272 (2003).
- Wang, H., et al. DNA bending and unbending by MutS govern mismatch recognition and specificity. Proc. Natl. Acad. Sci. USA. 100 (25), 14822-14827 (2003).
- Tessmer, I., et al. Mechanism of MutS searching for DNA mismatches and signaling repair. J. Biol. Chem. 283 (52), 36646-36654 (2008).
- Wagner, K., Moolenaar, G., van Noort, J., Goosen, N. Single-molecule analysis reveals two separate DNA-binding domains in the Escherichia coli UvrA dimer. Nucleic Acids Res. 37 (6), 1962-1972 (2009).
- Buechner, C. N., et al. Strand-specific recognition of DNA damages by XPD provides insights into nucleotide excision repair substrate versatility. J Biol. Chem. 289 (6), 3613-3624 (2014).
- Van der Linden, E., Sanchez, H., Kinoshita, E., Kanaar, R., Wyman, C. RAD50 and NBS1 form a stable complex functional in DNA binding and tethering. Nucleic Acids Res. 37 (5), 1580-1588 (2009).
- Fuentes-Perez, M. E., Dillingham, M., Moreno-Herrero, F. AFM volumetric methods for the characterization of proteins and nucleic acids. Methods. 60, 113-121 (2013).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Miyagi, A., Lyubchenko, Y. L. Specificity of binding of single-stranded DNA-binding protein to its target. Biochemistry. 51, 1500-1509 (2012).
- Wirth, N., et al. Conservation and Divergence in Nucleotide Excision Repair Lesion Recognition. J. Biol. Chem. 291 (36), 18932-18946 (2016).
- Kuper, J., Kisker, C. Damage recognition in nucleotide excision DNA repair. Curr. Opin. Struct. Biol. 22, 88-93 (2012).
- Buechner, C. N., Tessmer, I. DNA substrate preparation for atomic force microscopy studies of protein-DNA interactions. J. Mol. Recognit. 26 (12), 605-617 (2013).
- Sun, Z., Tan, H. Y., Bianco, P. R., Lyubchenko, Y. L. Remodeling of RecG Helicase at the DNA Replication Fork by SSB Protein. Sci. Rep. 5, 9625 (2015).
- Billingsley, D. J., Bonass, W. A., Crampton, N., Kirkham, J., Thomson, N. H. Single-molecule studies of DNA transcription using atomic force microscopy. Phys. Biol. 9 (2), 021001 (2012).
- Maurer, S., Fritz, J., Muskhelishvili, G., Travers, A. RNA polymerase and an activator form discrete subcomplexes in a transcription initiation complex. EMBO J. 25 (16), 3784-3790 (2006).
- Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Theis, K., Chen, P. J., Skorvaga, M., Van Houten, B., Kisker, C. Crystal structure of UvrB, a DNA helicase adapted for nucleotide excision repair. EMBO J. 18 (24), 6899-6907 (1999).
- Kuper, J., et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 12 (9), e1001954 (2014).
- Shlyakhtenko, L. S., et al. Silatrane-based surface chemistry for immobilization of DNA, protein-DNA complexes and other biological materials. Ultramicroscopy. 97, 279-287 (2003).
- Roth, H. M., et al. XPB helicase regulates DNA incision by the Thermoplasma acidophilum endonuclease Bax1. DNA Repair. 11 (3), 286-293 (2012).
- Ratcliff, G. C., Erie, D. A. A Novel Single-Molecule Study to Determine Protein-Protein Association Constants. J. Am. Chem. Soc. 123 (24), 5632-5635 (2001).
- Yang, Y., Sass, L. E., Du, C., Hsieh, P., Erie, D. A. Determination of protein-DNA binding constants and specificities from statistical analyses of single molecules: MutS-DNA interactions. Nucleic Acids Res. 33 (13), 4322-4334 (2005).
- Caron, P. R., Grossman, L. Involvement of a cryptic ATPase activity of UvrB and its proteolysis product, UvrB* in DNA repair. Nucleic Acids Res. 16 (22), 10891-10902 (1988).
- Wang, H., et al. UvrB domain 4, an autoinhibitory gate for regulation of DNA binding and ATPase activity. J. Biol. Chem. 281 (22), 15227-15237 (2006).
- Chammas, O., Billingsley, D. J., Bonass, W. A., Thomson, N. H. Single-stranded DNA loops as fiducial markers for exploring DNA-protein interactions in single molecule imaging. Methods. 60 (2), 122-130 (2013).
- Truglio, J. J., et al. Structural basis for DNA recognition and processing by UvrB. Nat. Struct. Mol. Biol. 13 (4), 360-364 (2006).
- Kuper, J., Wolski, S. C., Michels, G., Kisker, C. Functional and structural studies of the nucleotide excision repair helicase XPD suggest a polarity for DNA translocation. EMBO J. 31 (2), 494-502 (2012).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Goosen, N. The presence of two UvrB subunits in the UvrAB complex ensures damage detection in both DNA strands. EMBO J. 21 (15), 4196-4205 (2002).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Hoeijmakers, J. H., Goosen, N. Architecture of nucleotide excision repair complexes: DNA is wrapped by UvrB before and after damage recognition. EMBO J. 20 (3), 601-611 (2001).
- Moolenaar, G. F., et al. The Role of ATP Binding and Hydrolysis by UvrB during Nucleotide Excision Repair. J. Biol. Chem. 275, 8044-8050 (2000).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены