JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
핵 포스 현미경 검사에 의한 DNA 절편 검사에서의 DNA 병변 인식 연구
요약
Here, the study of different DNA lesion recognition approaches via single molecule AFM imaging is demonstrated with the nucleotide excision repair system as an example. The procedures of DNA and protein sample preparations and experimental as well as analytical details for the AFM experiments are described.
초록
AFM imaging is a powerful technique for the study of protein-DNA interactions. This single molecule method allows the simultaneous resolution of different molecules and molecular assemblies in a heterogeneous sample. In the particular context of DNA interacting protein systems, different protein complex forms and their corresponding binding positions on target sites containing DNA fragments can thus be distinguished. Here, an application of AFM to the study of DNA lesion recognition in the prokaryotic and eukaryotic nucleotide excision DNA repair (NER) systems is presented. The procedures of DNA and protein sample preparations are described and experimental as well as analytical details of the experiments are provided. The data allow important conclusions on the strategies by which target site verification may be achieved by the NER proteins. Interestingly, they indicate different approaches of lesion recognition and identification for the eukaryotic NER system, depending on the type of lesion. Furthermore, distinct structural properties of the two different helicases involved in prokaryotic and eukaryotic NER result in and explain the different strategies observed for these two systems. Importantly, these experimental and analytical approaches can be applied not only to the study of DNA repair but also very similarly to other DNA interacting protein systems such as those involved in replication or transcription processes.
서문
원자 힘 현미경 (AFM)은 단백질 -DNA 상호 작용 1 , 2 , 3 , 4 , 5 , 6 , 7 , 8 , 9 의 분석을위한 강력한 기술입니다. 단일 분자 수준의 분해능으로 이종 샘플을 직접 시각화하기 위해서는 적은 양의 시료가 필요합니다. 이질성은 단백질의 상이한 형태 또는 올리고머 상태에 기인 할 수있다. 특히, 단백질 -DNA 샘플과 관련하여, 단백질 복합체는 일반적으로 DNA 결합 또는 DNA 내의 특정 표적 부위에 결합함으로써 유도 된 상이한 화학 양론 및 / 또는 형태를 나타낼 수있다. 이종 샘플은 또한 2 종 (또는 그 이상)의 상이한 종류의 단백질 및 상이한 단백질 복합체형태 ( 예를 들어 , 하나의 유형의 단백질 대 헤테로 메릭 복합체로 이루어진)는 DNA와 다르게 상호 작용할 수있다. 여기서 논의 된 연구는 공기 중의 AFM 이미징을 이용하여이 단백질의 표적을 나타내는 병변을 포함하는 긴 (~ 900 염기쌍, bp) DNA 단편에 결합 된 DNA 복구 단백질의 정적 건조 샘플을 이용한다. AFM의 분자 분해능이 높기 때문에 여러 종류의 단백질 복합체를 구별 할 수 있으며 DNA 조각에 결합하는 단백질의 결합 위치를 결정할 수 있습니다. 중요하게도, 병변은 잘 정의 된 위치에서 DNA 기질에 도입됩니다. DNA에 병변 부위의 위치가 알려지기 때문에 DNA에 결합 된 단백질의 분포는 (상이한) 단백질 복합체의 (상이한) 병변 인식 특성, 예를 들어 그들이 특정 유형의 병변을 얼마나 잘인지하는지에 대한 통찰력을 제공한다 손상되지 않은 DNA에) 2 , 3 , 4 , 5 , 6 . DNA에 대한 그들의 위치는 또한 병변에서 특이 적으로 결합 된 단백질 복합체와 DNA의 다른 곳에서 비특이적으로 결합 된 복합체 사이의 구별을 허용한다. 이들 상이한 복합 형 (병변 및 비 특이성 복합체에서 특이 적으로 결합 된 복합체)의 분리 된 특성화는 표적 부위 확인시 유도 된 복합체에서의 잠재적 인 구조 변화를 나타낼 수있다.
여기에 초점을 맞춘 DNA 복구 단백질은 핵산 절삭 수선 (NER) 경로에서 병변 인식을 담당하는 헬리 카아 제입니다. 박테리아에서 NER은 단백질 UvrA, UvrB 및 UvrC에 의해 달성됩니다. UvrA는 UvaA 2 / UvrB 2 DNA 스캐닝 복합체에서 초기 병변 감지를 담당합니다. UvrB에 의한 병변 검증시,이 복합체는 병변 부위에서 결합 된 단량체 UvrB로 전환되고,이 특정 복합체는 p원핵 세포 NER 엔도 뉴 클레아 제 UvrC. UvrC는 병변이있는 짧은 (12-13nt) 스트레치의 단일 가닥 DNA (ssDNA)를 절단합니다. 누락 된 스트레치는 DNA 폴리머 라제에 의해 재충전됩니다. 마지막으로, DNA 리가 제가 원래 합성 된 DNA 9,10 으로 새로 합성 된 신축성을 봉인합니다. 진핵 생물에서 NER 캐스케이드의 대부분의 단백질은 대형의 다중 전사 인자 II H (TFIIH) 복합체의 일부입니다. 삼량 체 CEN2-XPC-HR23B 복합체 를 통한 초기 병변 감지 후, TFIIH는 DNA 표적 부위로 모집된다. 복합체 내의 XPD가 NER 표적 병변의 존재를 확인하면, 진핵 생물 NER 엔도 뉴 클레아 제인 XPG 및 XPF는 병변 9 , 10을 함유 한 짧은 (24-32 nt) ssDNA 스트레치를 모집하도록 모집된다. 여기서는 원핵 생물과 진핵 생물 NER의 헬리 케이즈 UvrB와 XPD를 연구했습니다. 이 헬리콥터에는DNA (DNA bubble)는 두 개의 DNA 단일 가닥 중 하나에 연결되고,이어서 ATP 가수 분해에 의해 연료 가닥을 따라 이동합니다. 따라서 DNA 병변 이외에 DNA 버블이 단백질의 로딩 위치로 기능하는 기질에 도입되었습니다.
특정 병변 DNA 기판의 준비 절차는 이전에 11에서 설명 했습니다. 그것은 니카아제를위한 2 개의 밀접하게 이격 된 제한 부위를 갖는 원형 DNA 구조체 (플라스미드)를 필요로한다. 이 연구의 맥락에서, 플라스미드 pUC19N (2729 BP)이 사용되었다 (S. Wilson의 실험실, NIEHS에 의해 만들어졌다). 이 플라스미드는 48 뉴클레오타이드 (nt) 스트레치를 구성하는 니카타아제 Nt.BstNBI를위한 3 개의 밀접하게 이격 된 제한 사이트를 포함한다. 니카아제로 배양 한 후, 이들 부위 사이의 ssDNA의 스트레치를 제거하고 임의의 표적 특징을 함유하는 올리고 뉴클레오타이드로 대체 할 수있다. 각 단계 후, 완전한 효소 소화를 아가 로즈 겔로 시험한다전기 영동. Nicked 원형 DNA는 원래 supercoiled 플라스미드에 비해 낮은 전기 영동 이동성으로 인해 구별하실 수 있습니다. DNA의 갭핑 및 제거 된 스트레치의 특정 기질 올리고 뉴클레오타이드에 의한 대체는 닉 사이의 영역 내에서만 기질을 절개하는 제한 효소로 분해함으로써 평가 될 수있다. 효소에 의한 원형 플라스미드의 선형화는 따라서 갭 DNA에 대해 억제되고 특정 올리고 뉴클레오타이드의 삽입 후에 회복 될 것이다. 마지막으로, 두 endonuclease 제한 사이트 (이상적으로 단일 커터) 원하는대로 길이와 정의 된 위치뿐만 아니라 병변에서 거리에있는 DNA 버블에서 특정 대상 사이트와 선형 DNA 기판의 생성을 허용 5 '또는 3'방향.
NER 헬리 케이즈에 의한 병변의인지 는 AFM 영상 을 통해 조사 할 수 있습니다. t에서 헬리 카스의 DNA 전좌가 스톨 된그 병변 부위는 DNA상의 단백질 위치 분포의 첨단으로 볼 수 있으며 병변 인식을 나타낸다. 이 헬리 카제의 DNA 전좌가 5 '에서 3'극성으로 나아가 방향성을 갖기 때문에 로딩 위치 (병변의 상류 또는 하류의 DNA 버블)의 위치에 대한 병변 인식의 의존성도 병변이 우선적으로인지되는지 여부를 나타냅니다 (translocated) 또는 반대쪽으로, 비 - 전위 된 ssDNA 가닥 5 , 9 . 다음 섹션에서는 사용 된 방법을 소개하고이 실험의 주요 결과를 간단히 설명합니다. 중요한 것은 여기에 제시된 DNA 수리에 대한 모범 사례와 유사하게 AFM 이미징은 DNA 복제 또는 전사 8 , 12 , 13 , 14 와 같은 서로 다른 DNA 상호 작용 시스템의 연구에 적용될 수 있습니다 .
프로토콜
1. 샘플 준비
- DNA 기판 준비 11
- 플라스미드에서 ssDNA 갭 생성
- 적절한 니카아제 (여기 Nt.BstNBI)와 효소 열 불 활성화와 반응 튜브에서 플라스미드 (여기에 : 수정 된 pUC19, pUC19N)의 샘플을 완전히 소화기 제조 업체의 프로토콜에 따라 조건을 사용하여 소화 (그림 1 참조 설계도 표시). 충분한 수율을 얻기 위해 ~ 50 μL와 ~ 500 nM 플라스미드로 시작하십시오.
- 묽게 한 견본 (~ 20 nM) 15 에 아가로 오스 젤 전기 이동 법에 의하여 플라스마 새싹을 확인하십시오. DNA 시각화에 사용되는 DNA 결합 염료에 대한 보호용 장갑을 착용하십시오.
참고 : 다른 전기 영동 이동성은 nicked (relaxed)와 supercoiled 원형 플라스미드 DNA ( 그림 1 )를 구분할 수 있습니다. - 플라스미드로부터 절개 된 ssDNA 스트레치 (닉 사이트 사이)를열 블록 ( 도 1 )에서 올리고 뉴클레오타이드의 용융 온도 (여기서는 68 ℃, pUC19N의 경우) 근처에서 30 분 동안 300rpm으로 흔들어 ~ 10 배 초과하는 상보 적 올리고 뉴클레오타이드 ( 표 1 의 올리고 뉴클레오타이드 1) ).
- 테이블 원심 분리기 ( 그림 1 )에서 10,000 XG에서 10 분 동안 원심 분리하여 50 kDa 분자량 차단 (MWCO) 필터를 사용하여 작은 DNA 조각에서 gapped 플라스미드를 분리합니다. 필터에서 농축 된 DNA를 추출하려면 필터를 뒤집고 새로운 1.5 mL 반응 튜브에 삽입하십시오. 1,000 x g에서 3 분간 원심 분리하십시오.
- 추출한 여과수로 500μL로 농축 된 DNA 샘플을 보충하고, 상보 적 올리고 뉴클레오타이드를 ~ 5 배 초과하여 추가로 3 회 이상 단계 1.1.1.3과 1.1.1.4를 반복한다.
- conditi를 사용하여 적절한 제한 효소 (여기 : XhoI 또는 BglII)와 함께 항온 배양하여 DNA의 완전한 갭을 테스트합니다.제조업체의 설명에 따라 작동합니다. 희석 된 (흠집 대 갭이있는) DNA 샘플을 사용하십시오 (~ 20nM). nicked DNA를 양성 대조군으로 사용하여 선형화 된 플라스미드 (ssDNA 간격 포함)와 비 절개 DNA (gapped DNA)를 구별하기 위해 아가로 오스 겔 전기 영동을 실행하십시오 (참조 용 DNA 사다리 포함, 그림 1 ). 장갑을 착용하십시오.
- 변형 된 ssDNA 올리고 뉴클레오타이드와의 갭 재충전
- 선택한 특정 표적 부위가 들어있는 5 '인산화 올리고 뉴클레오티드의 ~ 25 배 초과 배양액을 사용하여 갭 플라스 미를 제거한 후 ~ 45 ° C에서 4 시간 동안 배양합니다 ( 그림 1 ). 여기에서, 병변 (형광 증식 된 티민 또는 시클로 부탄 피리 미딘 다이머 (CPD)) 및 DNA 버블 생성을위한 짧은 (8nt) 비 상보 서열을 함유하는 48 nt ssDNA를 사용한다 ( 표 1 참조).
- 인큐베이터로 어닐링 한 인서트를 플라스미드에 공유 결합T4 DNA ligase로 상온에서 하룻밤 동안 배양 하였다 ( 그림 1 ). 이 반응을 위해, 농축 완충액 원액을 포함하는 ATP를 첨가하여 리가 아제 ( 예 : UvrB 반응 완충액 : 50 mM Tris-HCl pH 7.5, 10 mM MgCl 2 , 50 mM KCl, 5 mM DTT, 1 mM ATP).
- 희석 된 시료를 적절한 제한 효소 (1.1.1.6에서와 동일)로 소화시켜 제조사의 프로토콜에 따라 특정 올리고 뉴클레오타이드를 삽입 한 플라스미드에 삽입 한 후 아가로 오스 겔 전기 영동 (1.1.1.6, 그림 1 ).
- 선형 DNA 기질의 제조
- 제한 효소 (이상적으로 플라스미드의 단일 제한 사이트와 함께) 수정 된 플라스미드 DNA를 제조사에서 권장 한 조건을 사용하여 분해하십시오 ( 그림 1 ). 이 단계는 다음을 포함하는 선형 조각을 생성합니다.삽입 된 수정을 정의 된 위치에 놓습니다. 여기에서 SspI와 BspQI를 삽입물의 상류와 하류의 위치 613과 255bp에서 절단하여 DNA 길이의 ~ 30 %에 특정 병변 부위를 포함하는 916 bp DNA 단편을 만듭니다.
참고 : 여기에 설명 된 AFM 이미징 실험의 경우 ~ 200bp와 ~ 2,000bp 사이의 길이를 갖는 DNA 조각이 적합한 기질입니다. - 상업용 키트를 사용하여 아가 로스 겔 전기 영동 및 겔 추출을 통해 표적 단편 을 단리한다. 보호 장갑을 착용하십시오.
- 선택적으로 메스로 아가로 오스 겔을 절단하여 DNA 사다리와 희석 된 샘플 레인 (처음 2 레인)을 정제 할 농축 DNA가 들어있는 레인에서 분리합니다. 첫 번째 두 차선이있는 젤 부분 만 UV 젤에이 젤 부분을 두어 UV 조사에 노출 시키십시오.
- 선택한 조각에 해당하는 밴드를 메스로 잘라냅니다. 두 개의 젤 부품을 다시 결합하십시오 (UV 탭에서 분리하십시오).르). 원하는 DNA 기질의 밴드 위치에 대한 방향으로 희석 샘플에서 excised 밴드의 위치를 사용하십시오. 묽게 한 통제보다는 경미하게 더 넓은 조각을 잘라서 더 높은 농도를 고려하십시오.
참고 : 여기에서 UV 병소의 인식이 조사되었으므로 UV 조사 를 통한 추가 UV 병소의 도입은이 방법으로 DNA 기질에서 조심스럽게 회피되었습니다.
- 선택한 조각에 해당하는 밴드를 메스로 잘라냅니다. 두 개의 젤 부품을 다시 결합하십시오 (UV 탭에서 분리하십시오).르). 원하는 DNA 기질의 밴드 위치에 대한 방향으로 희석 샘플에서 excised 밴드의 위치를 사용하십시오. 묽게 한 통제보다는 경미하게 더 넓은 조각을 잘라서 더 높은 농도를 고려하십시오.
- UV-Vis 분광 광도계에 의해 측정 된 260 nm에서의 흡수로부터 DNA 농도 c 를 계산한다. 이중 가닥 DNA (dsDNA) 평균 몰 흡광 계수 ε 260nm ~ 6,700 M - 1 cm -1 bp 당 Lambert-Beer의 법칙을 사용한다 :
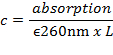
여기서 L 은 경로 길이 (측정 챔버 길이, 일반적으로 1cm)입니다.
- 제한 효소 (이상적으로 플라스미드의 단일 제한 사이트와 함께) 수정 된 플라스미드 DNA를 제조사에서 권장 한 조건을 사용하여 분해하십시오 ( 그림 1 ). 이 단계는 다음을 포함하는 선형 조각을 생성합니다.삽입 된 수정을 정의 된 위치에 놓습니다. 여기에서 SspI와 BspQI를 삽입물의 상류와 하류의 위치 613과 255bp에서 절단하여 DNA 길이의 ~ 30 %에 특정 병변 부위를 포함하는 916 bp DNA 단편을 만듭니다.
- 플라스미드에서 ssDNA 갭 생성
- 단백질 발현 및 정제
- E. coli 에서 UvrB를 재조합 발현하고 표준 키틴 비드 친 화성 15 및 크기 배제 크로마토 그래피 15 를 통해 단백질 을 정제한다.
참고 : Bacillus caldotenax 의 uvrB 유전자는 pTYB1 벡터에 클로닝되었습니다. - 대장균 에서 XPD를 익스프레션하고 표준 니켈 IDA 친화도 15 및 크기 배제 크로마토 그래피 15, 이어서 음이온 교환 크로마토 그래피 15 를 통해 단백질 을 정제한다.
참고 : Chaetomium thermophilum Xeroderma pigmentosum 그룹 D 단백질 (XPD) 유전자는 pBADM11 벡터에 클로닝되었습니다. C. thermophilum p44는 캐롤라인 키스 커 (Caroline Kisker)의 실험실에서 표현한 선물이었고 puri17 .
- E. coli 에서 UvrB를 재조합 발현하고 표준 키틴 비드 친 화성 15 및 크기 배제 크로마토 그래피 15 를 통해 단백질 을 정제한다.
2. AFM 실험
- 샘플 준비
- 선택적으로, 가열 블록에서 65 ℃에서 10 분간 DNA 기질을 사전 배양하여 냉장고에 보관하는 동안 형성 될 수있는 잠재적 인 미세 염 결정을 제거합니다.
- 10 배 농도 (10x 버퍼)에서 반응 버퍼를 준비합니다.
참고 : 1X 농도의 XPD 반응 완충액은 20 mM Tris-HCl pH 7.5, 10 mM KCl, 5 mM MgCl 2 , 1 mM TCEP, 2 mM ATP; 1x UvrB 반응 완충액은 50 mM Tris-HCl pH 7.5, 50 mM KCl, 10 mM MgCl2, 5 mM DTT 및 1 mM ATP를 함유 하였다. - 복잡한 형성을 향상시키기 위해 적절한 배양 조건에서보다 높은 농도로 단백질을 미리 배양하십시오. 원하는 농도로 1x 단백질 반응 완충액에 소량의 단백질 ( 예 : 1 μL)을 희석하고 작은 양 ( 예 : 1 μL)의 t그는 0.5 mL 반응 튜브에서 개별 단백질 용액을 얻었다.
- 튜브를 실온보다 높은 배양 온도를위한 열 블록에 넣으십시오. 등몰 또는 해당 농도의 복잡한 화학 양론에 따라 농도 비율을 선택하십시오. 여기에 1 μL XPD (1 x XPD 반응 완충액에 20 μM)와 1 μL p44 (1 x XPD 반응 완충액에 20 μM)를 10 μM 씩 37 ° C에서 10 분간 품어 둔다.
- 0.5 ML 반응 튜브에 단백질 반응 버퍼에 적절한 단백질과 DNA 농도 샘플을 품다. 여기에 500 nM UvrB 또는 1 μM XPD + 1 μM p44와 100 nM DNA를 사용하십시오. 피펫 작은 볼륨, 예를 들어 , 0.25-0.5 μL 단백질 (10 배 배양 농도에 미리 희석) 및 DNA 1x 단백질 반응 버퍼 2.5-5 μL의 총 볼륨에 저장합니다. 여기, 열 블록에서 37 ° C에서 30 분 품어. 원심 분리기에서 반응 튜브에서 잠깐 (~ 1 초) 스핀 다운작은 부피의 혼합을 보장한다.
- 샘플 증착
- 운모 기질 준비 : 메스를 사용하여 큰 스트립에서 약 1 x 1cm 2 운모를 자릅니다. 점착 테이프를 사용하여 다층 운모 광물 조각의 상단 레이어를 벗겨 깨끗하고 평평하며 원자 적으로 평탄한 소재 표면을 나타냅니다.
참고 : 운모 조각은 여러 층을 벗겨내어 여러 번 실험을 할 때 다시 사용할 수 있습니다. - 25 MM HEPES pH 7.5, 25 MM Na 아세테이트, 10 MM Mg 아세테이트와 같은 탈 이온수로 AFM 증착 버퍼를 준비하십시오. 0.02 μm 주사기 필터를 통해 여과하십시오.
참고 : 증착 버퍼의 2가 양이온은 마이너스로 대전 된 DNA 분자를 운모 표면에 킬레이트시키는 역할을하며, 운모 표면은 또한 중성 pH에서 음으로 대전됩니다. 침전 완충액 중 비교적 높은 Mg 2+ 이온 농도가 특정 단백질 -DNA 시스템에 문제를 제기한다면,운모 표면은 앵커 링을위한 양전하를 제공하기 위해 실라 트레인 기반 화학을 사용하여 아미노 그룹으로 사전로드 될 수 있습니다. 샘플 증착은 2가 양이온을 함유하지 않거나 단지 적은 양의 양이온을 함유하는 완충액에서 수행 될 수있다. - 침전 완충액에있는 운모에 즉시 침착시키기 위해 시료를 희석한다 (2.1 참조). 작은 양의 침전물 (여기에는 20 μL).
참고 : 희석 인자는 시료 농도에 따라 다릅니다. 여기, 희석 샘플 50-100x. 경험적으로 ~ 1nM의 DNA는 ~ 1,000bp의 우수한 표면 커버리지를 제공합니다. - 몇 밀리리터의 여과 된 탈 이온수로 시료를 즉시 3 ~ 4 회 헹구고 과량의 액체를 털어 내고 부드러운 질소 흐름으로 불어 건조시킵니다. 샘플 증착에서부터 건조 샘플까지의 전체 프로세스는 30 초 이내에 수행 될 수 있습니다.
- 가장자리에 접착 테이프를 사용하여 현미경 슬라이드에 운모 조각을 고정시킵니다.
참고 : 다른 AFM 시스템에는 d무대에 샘플을 고정하기위한 다른 요구 사항. 다른 AFM은 자기 단계를 가지고 있으며, 운모 조각은 열 접착제를 사용하여 자기 디스크에 고정됩니다. 여기에 사용 된 AFM 시스템의 세부 사항은 재료 표에서 찾을 수 있습니다.
- 운모 기질 준비 : 메스를 사용하여 큰 스트립에서 약 1 x 1cm 2 운모를 자릅니다. 점착 테이프를 사용하여 다층 운모 광물 조각의 상단 레이어를 벗겨 깨끗하고 평평하며 원자 적으로 평탄한 소재 표면을 나타냅니다.
- AFM 이미징
- 샘플을 (2.2.5 참조) AFM 스테이지 중앙에 놓고 자석 패드가 달린 스테이지에 현미경 슬라이드를 고정시킵니다.
- AFM 팁을 팁 홀더에 삽입하십시오. 공기 중 진동, 간헐적 접촉 모드 이미징에 적합한 날카로운 (<10 nm) AFM 프로브가있는 캔틸레버를 사용하십시오. 예를 들어, 재료 표에 나열된 AFM 프로브를 사용하십시오. 여기 (AFM에 따라 다름) 클램프 나사를 조여 (손으로 조여서) 클램프 아래의 홀더에 팁을 고정시킵니다. 팁 홀더를 AFM 측정 헤드에 삽입하십시오. 이 단계를 위해 머리를 뒤로 눕혀 라.
- AFM 측정 헤드를 샘플 위에 놓습니다. 세부 사항은 AFM 모델에 따라 다릅니다. 여기서, m무대 안의 안쪽에 머리가 안정적으로 놓여 있는지 확인하십시오. 운모가 AFM 팁이 위에 놓일 무대에 있는지 확인하십시오. 스테이지 오른쪽에있는 마이크로 미터 나사를 사용하여 샘플을 정밀하게 배치 할 수 있습니다.
- 레이저가 반사되는 위치 감지 광 검출기의 최적 신호 강도를 위해 AFM 레이저를 캔틸레버 뒷면에 맞 춥니 다. 세부 사항은 AFM 모델에 따라 다릅니다.
- 여기에서 AFM 측정 헤드의 오른쪽과 뒤쪽의 휠을 돌려서 AFM 레이저의 x 및 y 위치를 조정하여 캔틸레버의 끝 부분에 초점을 맞 춥니 다. AFM 비디오 창에서 반사 신호를 봅니다 (사용 가능한 경우 여기 : 카메라 아이콘을 누르고 입력 : Svideo를 선택).
- 조잡한 위치에 놓은 후 두 바퀴로 레이저 위치를 미세 조정하여 검출기 합 신호 (AFM 소프트웨어의 누적 합계 및 편향 미터 창에서 합계)를 최적화합니다 (캔틸레버 끝 부분에 머물러 있으십시오.AFM과 캔틸레버 유형에 따라 달라집니다. 여기서는 sum> 5)를 목표로합니다.
- AFM 레이저 반사를 감지기 중심으로 향하게하여 감지기 배열의 상단 및 하단 다이오드 (여기서는 편차 및 편향 미터 창에서의 편향 신호)와의 차이 신호를 제로화합니다 (여기에서는 AFM 측정의 왼쪽에서 휠을 돌립니다). 머리).
참고 : 0에서의 편차는 AFM에 의해 높이 정보로 변환되는 표면 상호 작용으로 인한 캔틸레버의 처짐을 나타냅니다. - AFM 소프트웨어에 구현 된 주파수 튠에 의해 캔틸레버 공진 주파수를 결정합니다 (여기에서는 마스터 패널 / 튜닝 윈도우의 명령 자동 튜닝). 캔틸레버 진동을 일으키는 피에조에 대한 1V 입력에 해당하는 진폭을 선택하십시오. 발진 주파수를 공진 주파수보다 약간 (5 %) 낮추고 발진의 위상을 0으로 설정하십시오.
- Crucial engaged 모드를 사용하여 팁을 샘플 표면에 접근e (여기 : 마스터 패널 창에서 명령) 보호 설정 (설정치)에 도달 할 때까지 기다립니다. 프리 레벨 진동 진폭의 ~ 2 %를 설정 점으로 사용하십시오 (여기서는 마스터 패널에서 설정 점 980mV로 입력하십시오).
- AFM 소프트웨어를 사용하여 설정 포인트를 낮춤으로써 AFM 팁을 샘플 표면에 미세하게 맞 춥니 다. 프리 레벨 위상 바로 아래의 캔틸레버 진동 위상 (Phase in Sum and Deflection Meter window)을 사용하여 반발 모드 이미징을 목표로하십시오. 여기에서 프리 레벨 진폭 (1V)의 70-80 % 정도의 순서로 전형적인 최종 설정 점을 사용하십시오.
- 스캔하기 전에 마스터 채널 패널에 녹음 할 신호를 선택하십시오. 높이 (Ht)와 진폭 (Am)을 선택하십시오.
- 샘플 스캐닝을 시작하십시오 (여기에서는 마스터 패널 창에서 명령 수행). 2.5 μm / s 스캔 속도 (마스터 패널의 스캔 속도 명령)를 사용하여 4 μm x 4 μm 또는 8 μm x 8 μm의 표면 영역을 이미지화합니다 (마스터 패널의 스캔 크기 입력).l)의 픽셀 해상도가 2,048 또는 4,096 (마스터 패널의 스캔 포인트 및 스캔 라인)입니다.
- 피팅 수정없이 이미지 파일 (마스터 패널에 이미지 저장 명령)을 저장하십시오 (마스터 채널 패널에서 없음 / 평면 저장을 선택하십시오).
- 추가 분석을 위해 저장된 이미지를 처리하십시오. 이미지를 불러옵니다 (AFM 분석 메뉴에서 저장된 데이터 찾아보기 명령). 수정 패널을여십시오 (이미지의 맨 위 섹션에서 M을 누르십시오). 높이 이미지 (확장 HtR)에 x 및 y 치수로 planefit을 적용합니다 (Modify Panel의 Planefit 창에서 XY를 명령하고 Planefit Order 3을 선택합니다). 그런 다음 이미지 병합 (Modify Panel의 Flatten 창에서 Flatten 명령)을 선택하여 Flatten Order 3을 선택하십시오.
- 추가 분석을 위해 이미지를 TIFF 파일로 내 보냅니다 (이미지의 상단 섹션에있는 명령을 누르고 2,048 픽셀 해상도에 해당하는 TIFF 내보내기 2x를 선택하십시오).
3. AFM 분석
- 단백질 복합체 부피
- 사전 -AFM 소프트웨어의 이미지를 직접 육안 검사하여 관련 단백질 -DNA 복합체를 선택하고, 서로 다른 크기의 복합체의 다양한 모습을 최대화하는 데 적합한 색상 구성표를 사용합니다 ( 그림 2 의 다양한 색상 및 크기의 복합체 참조, 그림 : SeaLandAndFire AFM 소프트웨어). XPD 샘플의 경우 XPD (~ 95 kDa)와 p44 (~ 40 kDa)의 상대적으로 큰 분자량으로 인해 크기가 뚜렷한 XPD / DNA 와 p44-DNA 는 물론 XPD / p44- .
- 복합 형을 확인하기 위해 DNA상의 개별 단백질 피크의 부피를 측정합니다. 이것은 다른 이미지 소프트웨어 (여기에서는 AFM 소프트웨어의 섹션 도구)를 사용하여 수행 할 수 있습니다.
- 섹션 윈도우 커서를 사용하여 단백질 피크 섹션의 높이 (h)와 직경 (d)을 측정합니다. 입자 섹션의 바닥에 가까운 직경을 측정하십시오.
- 완두콩의 부피 (V)를 결정하십시오구형 캡 모델을 기반으로하는 다음 수식을 사용하여 간단한 수학 모델을 사용하여 ks를 계산합니다.
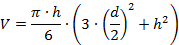
- 측정 된 부피를 대략 단백질 분자량으로 변환합니다.
- 분자량이 알려진 단백질의 범위를 사용하여 분자량 (MW)의 부피를 측정하기 위해 AFM 시스템을 보정하십시오. 여기에서 각각 25와 851 데이터 포인트를 갖는 총 12 개의 실험이 단량체, 이량 체 (dimeric) , 삼량 체 또는 사량 체 상태) 4 , 19 , 20 . 위에서 설명한 바와 같이 (2.2 및 2.3에서) 단백질을 축적하고 이미지화하고 (3.1.2) 위에서 설명한대로 볼륨을 측정합니다. 알려진 분자량에 걸쳐 볼륨을 플롯합니다. 결과 그래프는 V와 MW 사이의 선형 관계를 보여 주며,이 방정식은데이터에 맞는 선. 여기에 사용 된 AFM 시스템의 경우, 다음과 같은 관계가 얻어졌다. 19 :
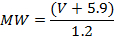
참고 :이 단계는 각 측정 전에 반복하지 않아도되지만 한 번만 수행됩니다. MW 보정의 부피는 직경이 약간 다른 AFM 프로브를 사용하여 유사한 이미징 조건에서 얻어진 이미지에 적용 할 수 있습니다. - 측정 된 부피 (3.1.2)와 V-to-MW 보정 (3.1.3.1)을 기반으로 분자 복합체의 분자량에 대한 정보를 제공하는 단백질 복합체 20 의 근사 분자량을 결정합니다. 예 를 들어 XPD / p44 샘플의 경우 p44-DNA 복합체 (또는 단순한 DNA 상부 구조에 의한 피크), XPD만의 피크 및 XPD / p44 피크와 각각 일치하여 ~ 50kDa, ~ 100kDa 및 ~ 140kDa가 얻어졌습니다 .
- 분자량이 알려진 단백질의 범위를 사용하여 분자량 (MW)의 부피를 측정하기 위해 AFM 시스템을 보정하십시오. 여기에서 각각 25와 851 데이터 포인트를 갖는 총 12 개의 실험이 단량체, 이량 체 (dimeric) , 삼량 체 또는 사량 체 상태) 4 , 19 , 20 . 위에서 설명한 바와 같이 (2.2 및 2.3에서) 단백질을 축적하고 이미지화하고 (3.1.2) 위에서 설명한대로 볼륨을 측정합니다. 알려진 분자량에 걸쳐 볼륨을 플롯합니다. 결과 그래프는 V와 MW 사이의 선형 관계를 보여 주며,이 방정식은데이터에 맞는 선. 여기에 사용 된 AFM 시스템의 경우, 다음과 같은 관계가 얻어졌다. 19 :
- DNA의 단백질 복합체 위치
- DNA를 결정하십시오.조각 길이.
- AFM 이미지의 DNA 조각을 추적하십시오 ( 예 : 적절한 이미지 분석 소프트웨어의 프리 핸드 라인 기능 (예 : 표표 참조)). 그리고 선의 길이를 측정하십시오.
참고 : DNA 집합체와 이미지 여백으로 잘라낸 조각은 제외하십시오. - 적절한 데이터 분석 및 그래프 작성 소프트웨어를 사용하여 히스토그램에서 전체 실험의 DNA 길이를 표시하고 플롯합니다 ( 예 : 표 재료 참조).
- 데이터 분석 및 그래프 작성 소프트웨어에서 가우스 곡선을 사용하여 길이 분포를 맞추어 DNA 단편의 길이를 결정합니다. 삽입 된 DNA 표적 위치 (1.1 참조)의 위치를 알기 위해서는, 가우스 곡선의 중심으로부터 2 표준 편차 이내의 정확한 길이를 갖는 DNA 단편만을 포함시킨다 (y = y 0 + z exp (-2 (xx c ) 2 / w 2 )이며, 여기서 z는 표준 인자이고 x c 와 w는 중심이며Gaussian의 최대 최대 반값 폭).
참고 : 가우스 곡선의 중심에있는 DNA 길이는 DNA 단편의 이론적 길이 (0.34 nm / bp를 사용하여 계산)에 근접해야합니다. 이론 값보다 최대 10 % 더 짧은 길이가 일반적이며 AFM 분해능 한계로 인해 발생할 수 있습니다.
- AFM 이미지의 DNA 조각을 추적하십시오 ( 예 : 적절한 이미지 분석 소프트웨어의 프리 핸드 라인 기능 (예 : 표표 참조)). 그리고 선의 길이를 측정하십시오.
- DNA 기질의 단백질 피크 위치를 결정하십시오.
- 3.2.1.1에서 설명한대로 가까운 DNA 단편 말단에서 단백질 피크의 거리를 측정하고 총 DNA 길이로 나누어 DNA 길이 분율의 단위로 거리를 구하십시오.
- 적절한 데이터 분석 및 그래프 작성 소프트웨어 ( 그림 3 참조)를 사용하여 히스토그램에서 측정 된 거리를 저장하고 플롯합니다. 일반적으로 n 개의 데이터 포인트에 대해 대략 √ bin을 제공하는 bin 크기를 선택하십시오.
참고 : DNA 끝은 여기에서 구별 할 수 없으므로 DNA 길이의 0.5 분의 1까지만 작도하십시오 (DNA frag의 중심ment). DNA 최종 바인딩은 연구의 초점이 아니기 때문에, 위치 0 이후 약간 위치 데이터를 bin에 시작하여 분석에서 DNA 끝 부분을 제외하십시오 ( 예 : 여기에서 : DNA 길이 부분 0.02에서부터 binning).
- 단백질 복합체 위치 분포로부터 목표 부위 특이성을 결정하십시오.
- 3.2.1.3에서와 같이 Gauss 커브를 사용하여 위치 분포에서 최대 값 (향상된 바인딩 발생)을 맞추지 만 배경 바인딩의 높이에 맞추십시오 ( 그림 3 참조).
- 특정 사이트에 대한 특이성 S 대 비특이적 DNA 배경 (비특이적 인 DNA 사이트에 결합 된 단백질 분자)을 다음의 수식 21로 계산하십시오
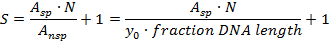
A sp : 특정 복합물의 수 (가우시안 곡선 아래 영역)
A nsp : 비특이적 복합체의 수 (배경 영역, ie (DNA 길이 분율) x (평균 배경 높이 y 0 ); 여기에 포함 된 DNA 길이의 부분은 0.02 DNA 길이에서 시작하는 히스토그램의 경우 0.48입니다)
N : 가능한 DNA 결합 부위의 수 (여기서는 DNA 말단을 제외한 N = 914)
- DNA를 결정하십시오.조각 길이.
- DNA 벤드 각도
- 적절한 이미지 분석 소프트웨어에서 각도 도구를 사용하여 DNA 백본을 따라 중앙에 위치하고 단백질 피크를 중심으로 두 선 사이의 각도 β를 측정합니다 ( 그림 4의 삽화 참조). 통계적으로 적절한 수의 단백질 -DNA 복합체 (> 50 이상, 이상적으로는> 100) 2 , 3 , 5에 대한 각도를 측정합니다.
참고 : DNA 굽힘 각도는 180 ° -β 2 , 3 , 5 로 정의됩니다. - 데이터 분석 및 그래프 작성 소프트웨어 사용(재료 표 참조)는 DNA 굽힘 각도를 비닝하여 굽힘 각도 분포 히스토그램을 생성합니다.
- 굽힘 각도 분포를 가우스 곡선에 맞 춥니 다. 분포에서 최대 값이 두 개 이상이면 여러 가우스 피크 적합성을 선택하십시오. 가우시안 곡선의 중심은 특정 종의 평균 굴곡 각 상태를 나타냅니다.
- 굴곡 각 (Gaussian fit (s))의 변화가 다른 단백질 변이체 또는 다른 단백질 복합체와 같은 분포에 대해 명백한 경우 학생의 t- 테스트를 적용하여 변경.
- 적절한 이미지 분석 소프트웨어에서 각도 도구를 사용하여 DNA 백본을 따라 중앙에 위치하고 단백질 피크를 중심으로 두 선 사이의 각도 β를 측정합니다 ( 그림 4의 삽화 참조). 통계적으로 적절한 수의 단백질 -DNA 복합체 (> 50 이상, 이상적으로는> 100) 2 , 3 , 5에 대한 각도를 측정합니다.
결과
단백질 복합체 부피를 기준으로 다른 복합 형을 구별
원핵 생물 NER 헬리 케이즈 UvrB의 헬리 케이즈 활성은 DNA 결합에 의해 자극된다 22 , 23 . UvrB는 두 개의 단일 ssDNA 가닥 중 하나에 올바르게로드하기 위해 DNA에 짝을 이루지 않은 영역 (DNA 버블)이 필요합니다. 생체 내 에서,이 DN...
토론
특정 표적 부위를 포함하는 긴 DNA 단편에 대한 단백질의 결합 위치에 대한 AFM 통계 분석은 단백질이 이들 부위 2 , 3 , 4 , 5 , 6 을 인식하기 위해 사용하는 특정 전략에 대한 흥미로운 세부 사항을 나타낼 수있다. 결과적인 위치 분포를 해석하기 위해, DNA 내 표적의 위치를 ?...
공개
저자는 공개 할 것이 없습니다.
감사의 말
PUC19N, CPD- 함유 올리고 뉴클레오타이드 및 p44는 Samuel Wilson, Korbinian Heil 및 Thomas Carell, Gudrun Michels 및 Caroline Kisker에 의해 각각 제공되었다. 이 연구는 도이체 Forschungsgemeinschaft (DFG) FZ82 및 TE-671 / 4에서 IT 보조금으로 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| Molecular Force Probe (MFP) 3D | Asylum Research | N/A | atomic force microscope (AFM) |
| Precision 390 | DELL | N/A | computer |
| ThermoMixer and 1.5 mL block | Eppendorf | 5382000015 | heat block for DNA preparation |
| Rotilabo Block-Heater H 250 & blocks for 0.5 mL tubes | Carl Roth GmbH | Y264.1 & Y267.1 | heat block for protein-DNA incubations |
| Mini-Sub Cell GT | Bio-Rad Laboratories GmbH | 1704467 | electrophoresis chamber with gel caster and power supply |
| Power Pac Basic | Bio-Rad Laboratories GmbH | 1645050 | electrophoresis power supply |
| Centifuge 5415 D with rotor | Eppendorf | 2262120-3 | table centrifuge |
| Ultra-Lum electronic UV transillumonator MEB-15 | Ultralum | 900-1322-02 | UV irradiation table |
| NanoDrop ND-1000 | VWR International / PEQLAB Biotechnologie GmbH | N/A | UV spectrophotometer |
| TKAX-CAD with 0.2 μm capsule filter | Unity Lab Services | N/A | water deionization and filter unit |
| Name | Company | Catalog number | Comments |
| Software | |||
| MFP software on Igor Pro | Asylum Research | N/A | AFM software |
| ImageJ (open source Java image processing) | NIH Image | N/A | Image analysis software |
| Excel (Microsoft Office) | Microsoft Corporation | N/A | data analysis software |
| Origin9 / Origin2016 | OriginLab Corporation | N/A | statistical data analysis and graphing software |
| Name | Company | Catalog number | Comments |
| Material | |||
| OMCL-AC240TS | Olympus | OMCL-AC240TS | AFM cantilevers |
| grade V-5 muscovite | SPI Supplies | 1805 | mica sheets |
| Amicon Ultra 0.5 mL 50k Ultracell | Millipore Ireland Ltd. | UFC505096 | centrifuge filters |
| NucleoSpin Extract II | Macherey-Nagel GmbH | 740 609.250 | Agarose gel extraction kit |
| Rotilabo cellulose paper type 111A | Carl Roth GmbH | AP59.1 | AFM deposition blotting paper |
| Anatop 25 (0.02 μm) | Whatman GmbH | 6809-2102 | syringe filter |
| SSpI, BspQI | New England Biolabs (NEB) | R0132, R0712 | restriction enzymes for DNA substrate preparation |
| XhoI, BglII | R0146, R0144 | restriction enzymes for DNA preparation controls | |
| nicking restriction enzyme Nt.BstNBI | New England Biolabs (NEB) | R0607 | nickase |
| T4 DNA ligase | New England Biolabs (NEB) | M0202S | Ligase |
| Tris, HEPES | Carl Roth GmbH | 4855, 9105 | buffer chemicals |
| NaCl, MgCl2, KCl, MgAcetate | Carl Roth GmbH | 3957, HN03, HN02, P026 | salt chemicals |
| NaAc | Sigma-Aldrich Chemie GmbH | 32318 | salt chemicals |
| DTT, TCEP, EDTA | 6908, HN95, 8040 | chemicals/reagents | |
| agarose, acetic acid, HCl | Carl Roth GmbH | 2267, 3738, K025 | reagents |
| ATP | Carl Roth GmbH | K054 | nucleotides |
| oligonucleotide #1 in Table 1 | Biomers | custom | complementary DNA oligonucleotide |
| oligonucleotides #2, #3, and #6 in Table 1 | Integrated DNA Technologies (IDT) | custom | fluorescein containing oligonucleotides |
| oligonucleotides #4 and #5 in Table 1 | private (available from e.g. TriLink or GlenResearch) | CPD containing oligonucleotides | |
| SafeSeal reaction tube 0.5 mL and 1.5 mL | Sarstedt | 72.704 and 72.706 | incubation tubes |
| GeneRuler 1 kb | Thermo Scientific | SM0311 | DNA ladder |
| 6x concentrate gel loading dye purple | New England Biolabs (NEB) | 51406 | DNA loading dye |
| Midori Green | Nippon Genetics Europe GmbH | 999MG28055 | DNA stain |
참고문헌
- Janicijevic, A., Ristic, D., Wyman, C. The molecular machines of DNA repair: scanning force microscopy analysis of their architecture. J. Microsc. 212 (3), 264-272 (2003).
- Wang, H., et al. DNA bending and unbending by MutS govern mismatch recognition and specificity. Proc. Natl. Acad. Sci. USA. 100 (25), 14822-14827 (2003).
- Tessmer, I., et al. Mechanism of MutS searching for DNA mismatches and signaling repair. J. Biol. Chem. 283 (52), 36646-36654 (2008).
- Wagner, K., Moolenaar, G., van Noort, J., Goosen, N. Single-molecule analysis reveals two separate DNA-binding domains in the Escherichia coli UvrA dimer. Nucleic Acids Res. 37 (6), 1962-1972 (2009).
- Buechner, C. N., et al. Strand-specific recognition of DNA damages by XPD provides insights into nucleotide excision repair substrate versatility. J Biol. Chem. 289 (6), 3613-3624 (2014).
- Van der Linden, E., Sanchez, H., Kinoshita, E., Kanaar, R., Wyman, C. RAD50 and NBS1 form a stable complex functional in DNA binding and tethering. Nucleic Acids Res. 37 (5), 1580-1588 (2009).
- Fuentes-Perez, M. E., Dillingham, M., Moreno-Herrero, F. AFM volumetric methods for the characterization of proteins and nucleic acids. Methods. 60, 113-121 (2013).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Miyagi, A., Lyubchenko, Y. L. Specificity of binding of single-stranded DNA-binding protein to its target. Biochemistry. 51, 1500-1509 (2012).
- Wirth, N., et al. Conservation and Divergence in Nucleotide Excision Repair Lesion Recognition. J. Biol. Chem. 291 (36), 18932-18946 (2016).
- Kuper, J., Kisker, C. Damage recognition in nucleotide excision DNA repair. Curr. Opin. Struct. Biol. 22, 88-93 (2012).
- Buechner, C. N., Tessmer, I. DNA substrate preparation for atomic force microscopy studies of protein-DNA interactions. J. Mol. Recognit. 26 (12), 605-617 (2013).
- Sun, Z., Tan, H. Y., Bianco, P. R., Lyubchenko, Y. L. Remodeling of RecG Helicase at the DNA Replication Fork by SSB Protein. Sci. Rep. 5, 9625 (2015).
- Billingsley, D. J., Bonass, W. A., Crampton, N., Kirkham, J., Thomson, N. H. Single-molecule studies of DNA transcription using atomic force microscopy. Phys. Biol. 9 (2), 021001 (2012).
- Maurer, S., Fritz, J., Muskhelishvili, G., Travers, A. RNA polymerase and an activator form discrete subcomplexes in a transcription initiation complex. EMBO J. 25 (16), 3784-3790 (2006).
- Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Theis, K., Chen, P. J., Skorvaga, M., Van Houten, B., Kisker, C. Crystal structure of UvrB, a DNA helicase adapted for nucleotide excision repair. EMBO J. 18 (24), 6899-6907 (1999).
- Kuper, J., et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 12 (9), e1001954 (2014).
- Shlyakhtenko, L. S., et al. Silatrane-based surface chemistry for immobilization of DNA, protein-DNA complexes and other biological materials. Ultramicroscopy. 97, 279-287 (2003).
- Roth, H. M., et al. XPB helicase regulates DNA incision by the Thermoplasma acidophilum endonuclease Bax1. DNA Repair. 11 (3), 286-293 (2012).
- Ratcliff, G. C., Erie, D. A. A Novel Single-Molecule Study to Determine Protein-Protein Association Constants. J. Am. Chem. Soc. 123 (24), 5632-5635 (2001).
- Yang, Y., Sass, L. E., Du, C., Hsieh, P., Erie, D. A. Determination of protein-DNA binding constants and specificities from statistical analyses of single molecules: MutS-DNA interactions. Nucleic Acids Res. 33 (13), 4322-4334 (2005).
- Caron, P. R., Grossman, L. Involvement of a cryptic ATPase activity of UvrB and its proteolysis product, UvrB* in DNA repair. Nucleic Acids Res. 16 (22), 10891-10902 (1988).
- Wang, H., et al. UvrB domain 4, an autoinhibitory gate for regulation of DNA binding and ATPase activity. J. Biol. Chem. 281 (22), 15227-15237 (2006).
- Chammas, O., Billingsley, D. J., Bonass, W. A., Thomson, N. H. Single-stranded DNA loops as fiducial markers for exploring DNA-protein interactions in single molecule imaging. Methods. 60 (2), 122-130 (2013).
- Truglio, J. J., et al. Structural basis for DNA recognition and processing by UvrB. Nat. Struct. Mol. Biol. 13 (4), 360-364 (2006).
- Kuper, J., Wolski, S. C., Michels, G., Kisker, C. Functional and structural studies of the nucleotide excision repair helicase XPD suggest a polarity for DNA translocation. EMBO J. 31 (2), 494-502 (2012).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Goosen, N. The presence of two UvrB subunits in the UvrAB complex ensures damage detection in both DNA strands. EMBO J. 21 (15), 4196-4205 (2002).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Hoeijmakers, J. H., Goosen, N. Architecture of nucleotide excision repair complexes: DNA is wrapped by UvrB before and after damage recognition. EMBO J. 20 (3), 601-611 (2001).
- Moolenaar, G. F., et al. The Role of ATP Binding and Hydrolysis by UvrB during Nucleotide Excision Repair. J. Biol. Chem. 275, 8044-8050 (2000).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유