このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ヌクレオチド切除修復におけるDNA損傷認識の原子間力顕微鏡検査
要約
Here, the study of different DNA lesion recognition approaches via single molecule AFM imaging is demonstrated with the nucleotide excision repair system as an example. The procedures of DNA and protein sample preparations and experimental as well as analytical details for the AFM experiments are described.
要約
AFM imaging is a powerful technique for the study of protein-DNA interactions. This single molecule method allows the simultaneous resolution of different molecules and molecular assemblies in a heterogeneous sample. In the particular context of DNA interacting protein systems, different protein complex forms and their corresponding binding positions on target sites containing DNA fragments can thus be distinguished. Here, an application of AFM to the study of DNA lesion recognition in the prokaryotic and eukaryotic nucleotide excision DNA repair (NER) systems is presented. The procedures of DNA and protein sample preparations are described and experimental as well as analytical details of the experiments are provided. The data allow important conclusions on the strategies by which target site verification may be achieved by the NER proteins. Interestingly, they indicate different approaches of lesion recognition and identification for the eukaryotic NER system, depending on the type of lesion. Furthermore, distinct structural properties of the two different helicases involved in prokaryotic and eukaryotic NER result in and explain the different strategies observed for these two systems. Importantly, these experimental and analytical approaches can be applied not only to the study of DNA repair but also very similarly to other DNA interacting protein systems such as those involved in replication or transcription processes.
概要
原子間力顕微鏡(AFM)は、タンパク質-DNA相互作用1,2,3,4,5,6,7,8,9の解析に強力な技術です。単一分子レベルの分解能で異種試料を直接視覚化するには、少量の試料材料しか必要としない。異種性は、タンパク質の異なる立体配座またはオリゴマー状態から生じ得る。特に、タンパク質-DNA試料に関しては、タンパク質複合体は、一般にDNA結合によって誘導されるか、またはDNA内の特定の標的部位に結合する異なる化学量論および/またはコンホメーションを示すことができる。異種試料はまた、2種類(またはそれ以上)の異なる種類のタンパク質および異なるタンパク質複合体形態( 例えば 、1つのタイプのタンパク質対ヘテロメリック複合体のみからなる)はDNAと異なって相互作用することができる。ここで論じた研究は、これらのタンパク質の標的を表す病変を含む長い(約900塩基対、bp)DNA断片に結合したDNA修復タンパク質の静的乾燥サンプルの空気中でのAFMイメージングを利用する。 AFMの高い分子分解能は、異なるタイプのタンパク質複合体の間の区別を可能にし、DNA断片上のタンパク質の結合位置を決定する。重要なことに、病変は、明確な位置でDNA基質に導入される。 DNA中の病変部位の位置は既知であるので、DNAに結合したタンパク質の分布は、(異なる)タンパク質複合体の(異なる)病変認識特性、 例えばそれらが特定のタイプの病変をどれくらいよく認識するか( 例えば 、非損傷DNAに対する) 4,5,6。 DNA上のそれらの位置はまた、病変部に特異的に結合したタンパク質複合体とDNA上の他の場所に非特異的に結合した複合体との区別を可能にする。これらの異なる複合型(病変部と非特異的複合体に特異的に結合した複合体)の別個の特徴付けは、標的部位の同定に誘導された複合体における立体配座変化を明らかにする可能性がある。
ここで焦点を当てたDNA修復タンパク質は、ヌクレオチド切除修復(NER)経路における病変認識の原因となるヘリカーゼである。細菌では、NERはタンパク質UvrA、UvrBおよびUvrCによって達成される。 UvrAは、UvaA 2 / UvrB 2 DNAスキャン複合体における初期病変感知の原因である。 UvrBによる病変確認の際、この複合体は病変部位で結合した単量体UvrBに変換され、この特定の複合体はp原核生物NERエンドヌクレアーゼUvrC。 UvrCは、病変を含む一本鎖DNA(ssDNA)の短い(12-13nt)ストレッチを切除する。失われたストレッチは、DNAポリメラーゼによって再充填される。最後に、DNAリガーゼは新しく合成されたストレッチを元のDNA 9,10 で密封する。真核生物では、NERカスケードの大部分のタンパク質は、大きな多量体転写因子II H(TFIIH)複合体の一部である。三量体CEN2-XPC-HR23B複合体による初期病変感知の後、TFIIHをDNA標的部位に補充する。複合体内のXPDがNER標的病変の存在を確認すると、真核生物のNERエンドヌクレアーゼXPGおよびXPFが病変を含むssDNAの短い(24〜32nt)ストレッチを切除するために補充される9,10 。ここでは、具体的には、原核生物および真核生物NER由来のヘリカーゼUvrBおよびXPDをそれぞれ研究した。これらのヘリカーゼは、2つのDNA一本鎖のうちの1つに糸を通し、続いてATP加水分解によってこの鎖に沿って転位するDNA(DNAバブル)。したがって、DNA損傷に加えて、DNAバブルが、タンパク質の負荷部位として機能する基質に導入された。
特異的病変DNA基質の調製手順は以前に記載されている11 。それは、ニカナーゼのための2つの密接に隔てられた制限部位を有する環状DNA構築物(プラスミド)を必要とする。この研究の文脈では、プラスミドpUC19N(2729bp)を使用した(S. Wilsonの実験室、NIEHSによって作製された)。このプラスミドは、48ヌクレオチド(nt)のストレッチを構成するニカナーゼNt.BstNBIの3つの密接に隔てられた制限部位を含む。ニカナーゼとのインキュベーション後、これらの部位間のssDNAのストレッチを除去し、任意の標的特性を含むオリゴヌクレオチドで置き換えることができる。各工程の後、完全な酵素消化をアガロースゲル電気泳動。ニッキングされた環状DNAは、元のスーパーコイルプラスミドと比較してその電気泳動移動度が低いために区別することができる。 DNAのギャップ付けおよび除去されたストレッチの特定の基質オリゴヌクレオチドによる置換は、基質のみをニックの間の領域内で切断する制限酵素による消化によって評価することができる。したがって、酵素による環状プラスミドの直線化は、ギャップを有するDNAについて抑制され、特異的オリゴヌクレオチドの挿入後に回復する。最後に、2つのエンドヌクレアーゼ制限部位(理想的には単一のカッター)は、線状DNA基質の生成を可能にし、長さは所望の通りであり、特定の標的部位と定義された位置にDNA泡があり、 'または3'方向。
NERヘリカーゼによる病変の認識は、AFMイメージングによって調べることができる。 tにおけるヘリカーゼのDNA転位の停止病変部位は、DNA上のタンパク質位置分布のピークとして見え、病変認識を示す。これらのヘリカーゼのDNA転位はさらに5 'から3'の極性で方向性があるため、装填部位(病変の上流または下流のDNAバブル)の位置に対する病変認識の依存性も病変が優先的に認識されるかどうかを示す転位されたまたは反対の、非転座のssDNA鎖5,9。以下のセクションでは、使用される方法が紹介され、これらの実験からの主要な所見が簡単に議論される。重要なことに、ここに示すDNA修復の例示的な研究に類似して、AFMイメージングは、DNA複製または転写などの異なるDNA相互作用系の研究に適用することができる8,12,13,14
プロトコル
サンプル調製
- DNA基質の調製11
- プラスミド中のssDNAギャップの生成
- 製造者のプロトコールに従った条件を用いて、適切なニカナーゼ(ここではNt.BstNBI)と反応チューブ中のプラスミド(ここでは改変pUC19、pUC19N)のサンプルを完全に消化した後、酵素熱不活性化する(図式プレゼンテーション)。 〜50μLおよび〜500nMのプラスミドから十分な収率で開始する。
- 希釈サンプル(約20 nM)でアガロースゲル電気泳動でプラスミドのニッキングを確認する15 。 DNAの可視化に使用されるDNA結合色素に対する保護用の手袋を着用してください。
注:異なる電気泳動移動度により、ニック(緩和)およびスーパーコイル状のプラスミドDNA( 図1 )を区別することができます。 - プラスミドから切開したssDNAストレッチ(ニック部位の間)をヒートブロック中のオリゴヌクレオチドの融解温度付近(ここでは68℃、pUC19Nについては68℃)で300rpmで30分間振とうして〜10倍過剰の相補オリゴヌクレオチド( 表1のオリゴヌクレオチド1)とインキュベートする( 図1 )。
- ギャップ遠心分離したプラスミドを、テーブル遠心分離機( 図1 )で10,000 xgで10分間遠心分離することにより、50 kDaの分子量カットオフ(MWCO)フィルターを使用して、より小さなDNA断片から分離する。フィルターから濃縮DNAを抽出するには、フィルターを逆さにして新しい1.5 mL反応チューブに挿入します。 1,000 x gで3分間遠心分離する。
- 得られた濃縮DNAサンプルを脱イオン水で500μLに補充し、〜5倍過剰の相補的オリゴヌクレオチドを添加し、ステップ1.1.1.3および1.1.1.4を少なくとも3回繰り返す。
- コンディションを用いた適切な制限酵素(ここではXhoIまたはBglII)とのインキュベーションによるDNAの完全なギャップの試験製造元の説明書に従ってください。希釈した(ニッティングされたものとギャップがあるもの)DNAサンプルを使用する(約20nM)。アガロースゲル電気泳動を行って、陽性対照としてニックを入れたDNA(参考のためにDNAラダーを含む、 図1 )を用いて線状プラスミド(ssDNAギャップを含まない)と非切開DNA(ギャップDNA)を区別する。手袋を着用してください。
- 修飾されたssDNAオリゴヌクレオチドによるギャップの再充填
- 約45℃で4時間インキュベートする( 図1 )、選択された特定の標的部位を含む約25倍過剰の5 'リン酸化オリゴヌクレオチドと共にインキュベートすることにより、ギャップ付きプラスミドをアニーリングする。ここでは、病変、フルオレセイン付加チミンまたはシクロブタンピリミジン二量体(CPD)、さらに短い(8nt)非相補的配列を含む48ntsDNAを使用して、DNAバブルを生成する( 表1参照)。
- インキュベーションによってアニールされたインサートをプラスミドに共有結合させるT4 DNAリガーゼを用いて、室温で一晩、製造者のプロトコール( 図1 )に従いインキュベートする。この反応のために、濃縮緩衝液ストック溶液を含むATPを添加して、リガーゼ( 例えば 、UvrB反応緩衝液:50mM Tris-HCl pH 7.5,10mM MgCl 2、50mM KCl、5mM DTT、1mM ATP)。
- 希釈したサンプルを適切な制限酵素(1.1.1.6と同じ)で製造業者のプロトコールに従って消化し、続いてアガロースゲル電気泳動(1.1.1.6、 図1のように)により、特異的オリゴヌクレオチドのギャップ付きプラスミドへの挿入について試験する。 )。
- 線状DNA基質の調製
- 制限酵素(理想的にはプラスミド中の単一の制限部位を有する)で修飾されたプラスミドDNAを、製造業者の推奨する条件を用いて消化する( 図1 )。このステップは、挿入された変更を定義された位置に挿入します。ここでは、SspIおよびBspQIを挿入物の上流および下流の613および255bpの位置でそれぞれ切断して、DNA長の〜30%の特定の病変部位を含む916bpのDNA断片を得る。
注記:ここに記載したようなAFMイメージング実験では、〜200bp〜2000bpの長さを有するDNA断片が適切な基質である。 - 市販のキットを用いてアガロースゲル電気泳動およびゲル抽出によって標的断片を単離する。手袋を着用して保護する。
- 必要に応じて、メスでアガロースゲルを切断して、精製すべき濃縮DNAを含むレーンからDNAラダーおよび希釈サンプルレーン(最初の2レーン)を分離する。ゲルのこの部分のみをUVテーブルに置き、最初の2つのレーンでゲル部分をUV照射にさらしてください。
- 選択した断片に対応するバンドをメスで切り取る。 2つのゲル部分を再結合する(UVタブから外すル)。所望のDNA基質のバンドの位置の方向として、希薄試料から切除されたバンドの位置を使用する。希薄コントロールよりもわずかに広いスライスを切り取って、より高い濃度を考慮する。
注:ここでは、UV病変の認識が調査されたので、UV照射による追加のUV病変の導入は、このアプローチによってDNA基質において注意深く回避された。
- 選択した断片に対応するバンドをメスで切り取る。 2つのゲル部分を再結合する(UVタブから外すル)。所望のDNA基質のバンドの位置の方向として、希薄試料から切除されたバンドの位置を使用する。希薄コントロールよりもわずかに広いスライスを切り取って、より高い濃度を考慮する。
- 2本鎖DNA(dsDNA)の平均モル吸光係数ε260nm〜6,700 M -1 cm -1 / bpを有するLambert-Beerの法則を用いてUV-Vis分光光度計で測定した260 nmでの吸収からDNA濃度cを計算する:
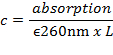
ここで、 Lは経路の長さ(測定室の長さ、典型的には1cm)である。
- 制限酵素(理想的にはプラスミド中の単一の制限部位を有する)で修飾されたプラスミドDNAを、製造業者の推奨する条件を用いて消化する( 図1 )。このステップは、挿入された変更を定義された位置に挿入します。ここでは、SspIおよびBspQIを挿入物の上流および下流の613および255bpの位置でそれぞれ切断して、DNA長の〜30%の特定の病変部位を含む916bpのDNA断片を得る。
- プラスミド中のssDNAギャップの生成
- タンパク質の発現と精製
- 大腸菌で UvrBを組換え発現し、標準的なキチンビーズ親和性15およびサイズ排除クロマトグラフィー15 を介してタンパク質を精製する。
注: Bacillus caldotenax由来の uvrB遺伝子は、pTYB1ベクターにクローン化されていた。 - 大腸菌で XPDを発現させ、標準的なニッケルIDA親和性15およびサイズ排除クロマトグラフィー15、続いて陰イオン交換クロマトグラフィー15 を介してタンパク質を精製する。
注: Chaetomium thermophilum Xeroderma色素群Dタンパク質(XPD)の遺伝子は、pBADM11ベクターにクローニングされています。 C. thermophilum p44は、Caroline Kiskerの研究室から寄贈されたものであり、記載されているようにした17 。
- 大腸菌で UvrBを組換え発現し、標準的なキチンビーズ親和性15およびサイズ排除クロマトグラフィー15 を介してタンパク質を精製する。
2. AFM実験
- サンプル調製
- 必要に応じて、ヒートブロックで65℃で10分間プレインキュベートして、冷蔵庫で保存中に形成された潜在的な微塩結晶を除去します。
- 10倍濃度(10×バッファー)で反応バッファーを調製する。
注:1X濃度のXPD反応緩衝液は、20mMトリス-HCl pH7.5,10mM KCl、5mM MgCl 2、1mM TCEP、2mM ATP; 1×UvrB反応緩衝液は、50mM Tris-HCl pH7.5,50mM KCl、10mM MgCl 2、5mM DTT、および1mM ATPを含有した。 - 適切なインキュベーション条件でより高い濃度でタンパク質をプレインキュベートして、複合体の形成を促進する。 1xタンパク質反応バッファー中の少量( 例えば 、1μL)の個々のタンパク質を所望の濃度に予め希釈し、少量( 例えば 、1μL)のt彼は個々のタンパク質溶液を0.5mL反応管に入れた。
- チューブを室温より高いインキュベーション温度のヒートブロックに入れてください。等モルまたは対応する濃度のいずれかで、予想される複雑な化学量論に応じて濃度比を選択します。ここでは、1μLのXPD(1×XPD反応バッファーで20μM)と1μLのp44(1×XPD反応バッファーで20μM)をそれぞれ10μMで37℃で10分間インキュベートする。
- 0.5mLの反応チューブ中のタンパク質反応バッファー中の適切なタンパク質およびDNA濃度で試料をインキュベートする。ここでは、500nMのUvrBまたは1μMのXPD +1μMのp44と100nMのDNAを使用します。 例えば 、0.25〜0.5μLのタンパク質(10倍のインキュベーション濃度まで予め希釈)および1×タンパク質反応緩衝液の2.5〜5μLの全容量中のDNAを保存するために、小容量のピペットをピペットに入れる。ここでは、37℃で30分間インキュベートします。テーブルの遠心分離機で短時間(〜1秒)反応管にスピンダウン少量の混合を確実にする。
- サンプル堆積
- マイカ基板を準備する:メスを使用して、大きなストリップから約1×1cm 2の雲母片を切断する。粘着テープを使用して多層のマイカミネラルピースの最上層を剥ぎ取って、清潔で平らで原子的に滑らかな基材表面を明らかにする。
注:雲母片は、別の層を取り除いて複数回の実験に再使用することができます。 - AFM沈着緩衝液を、脱イオン水、 例えば 25mM HEPES pH7.5,25mM Na-アセテート、10mM Mg-アセテートで調製する。 0.02μmシリンジフィルターで濾過する。
注:沈着緩衝液中の二価陽イオンは、負に荷電したDNA分子を、中性pHで負に帯電しているマイカ表面にキレートするのに役立つ。沈着緩衝液中の比較的高いMg 2+イオン濃度が特定のタンパク質-DNA系に問題を提起する場合、雲母表面には、固定化のための正の表面電荷を提供するために、シトラートベースの化学反応を用いてアミノ基を予め装填することができる( 18) 。次いで、サンプル沈着を、2価の陽イオンを含まないか、またはわずかしか含まないバッファー中で実施することができる。 - サンプルを希釈して(2.1を参照)、瞬時に析出用バッファー中のマイカに析出させる。少量(ここでは20μL)を預けます。
注:希釈係数はサンプル濃度に依存します。ここでは、サンプルを50〜100倍に希釈します。経験則として、約1nMのDNAは〜1,000bpの良好な表面カバレージをもたらす。 - 直ちにサンプルを数ミリリットルのろ過脱イオン水で3〜4回リンスし、余分な液体をブロットし、穏やかな窒素気流でブロー乾燥する。試料の析出から乾燥した試料までの全プロセスは、30秒以内に行うことができます。
- 接着剤テープを使用して顕微鏡スライド上の雲母片をその端に固定する。
注:異なるAFMシステムにはdサンプルをステージに固定するための異なる要件。他のAFMは磁気ステージを有しており、マイカ片は例えばサーマルグルーを用いて磁気ディスク上に固定されている。ここで使用されるAFMシステムの詳細は、材料表に記載されています。
- マイカ基板を準備する:メスを使用して、大きなストリップから約1×1cm 2の雲母片を切断する。粘着テープを使用して多層のマイカミネラルピースの最上層を剥ぎ取って、清潔で平らで原子的に滑らかな基材表面を明らかにする。
- AFMイメージング
- 試料をAFMステージの中央に置き(2.2.5参照)、顕微鏡スライドを磁気パッド付きのステージ上に固定する。
- AFMチップをチップホルダーに挿入します。カンチレバーには、空気中で振動、間欠接触モードイメージングに適した鋭い(<10 nm)AFMプローブを使用してください。たとえば、材料表に記載されているAFMプローブを使用します。ここでは(AFMに依存)、クランプねじを締めて(クランプの下で)、クランプの下のホルダにチップを固定します。チップホルダーをAFM測定ヘッドに挿入します。このステップのために頭を背中に置きます。
- AFM測定ヘッドをサンプルの上に置きます。詳細はAFMモデルに依存します。ここで、mステージの窪みの中に脚を立てて頭が安定していることを確認してください。雲母がAFMの先端がその上にホバーするステージ上にあることを確認してください。ステージの右側にあるマイクロメータのネジは、サンプルの精密な位置決めを可能にします。
- カンチレバーの背面にAFMレーザーを位置合わせして、レーザーが反射する位置検出光検出器からの最適な信号強度を得ます。詳細はAFMモデルに依存します。
- ここで、AFM測定ヘッドの右側と後ろの車輪を回してAFMレーザーのxとyの位置を調整してカンチレバーの端に中央に向けます。反射信号をAFMビデオウィンドウで見る(利用可能な場合は、カメラアイコンを押し、入力:Svideoを選択)。
- 粗い位置に配置したら、2つのホイールでレーザ位置を微調整することにより、検出器の和信号(AFMソフトウェアのSum and Deflection Meterウィンドウの合計)を最適化します(カンチレバーの端に留まり、lはAFMとカンチレバータイプに依存し、ここではsum> 5)。
- AFMレーザー反射を検出器中心に向けることによって、検出器アレイの上部および下部ダイオードからの差信号(ここでは、合計および偏向メーターウィンドウの偏向信号)をゼロにする(ここでは、AFM測定の左側で車輪を回転させる頭)。
注:ゼロからの偏差は、表面相互作用によるカンチレバーのたわみを示し、AFMによって高さ情報に変換されます。 - カンチレバーの共振周波数は、AFMソフトウェアに実装されている周波数調整(ここでは、マスターパネル/チューンウィンドウのコマンド自動チューン)によって決定します。カンチレバー振動を駆動するピエゾの1V入力に対応する振幅を選択します。発振周波数を共振周波数よりわずかに(5%)低く設定し、発振の位相をゼロにします。
- 粗結合モードを使用してチップをサンプル表面に近づけますe(ここでは、マスターパネルのウィンドウでコマンドを実行)をクリックして保護設定(設定値)に達するまで待ちます。自由振動振幅の約2%のカットを設定点として使用します(ここでは、マスターパネルで設定点980mVを入力します)。
- AFMソフトウェアを使用して設定点を下げることによって、AFMチップをサンプル表面にきめ細かく固定します。カンチレバー振動フェーズ(フェーズ・イン・アンド・デフレーション・メータ・ウィンドウ)をフリー・レベル・フェーズの直前(係合前、ここでは通常〜70)の反発モードイメージングを目指します。ここでは、自由レベル振幅(1V)の70〜80%のオーダの典型的な最終設定点を使用します。
- スキャンする前に、マスターチャンネルパネルに録音する信号を選択します。高さ(Ht)と振幅(Am)を選択します。
- サンプルスキャンを開始します(ここでは、マスターパネルウィンドウでDo Scanを実行します)。 2.5μm/ sのスキャン速度(Master PanelのコマンドScan Speed)を使用して、4μmx 4μmまたは8μmx 8μmの表面領域をイメージします(Master PaneのScan Sizeと入力します)。1)のピクセル解像度が2,048または4,096(マスターパネルのスキャンポイントおよびスキャンライン)である。
- フィッティングを変更せずに画像ファイルを保存します(マスターパネルに画像を保存するコマンド)。マスターチャンネルパネルではNoneを選択し、面で保存するにはSaveを選択します。
- さらに分析するには、保存した画像を処理します。イメージをロードします([AFM Analysis]メニューで[Browse Saved Data]コマンドを実行します)。 Modifyパネルを開きます(画像の上部にあるMを押してください)。高さイメージ(拡張HtR)にxとy次元のplanefitを適用します(ModifyパネルのPlanefitウィンドウのXYコマンド、Planefit Order 3を選択します)。次に、「Flatten Order 3」を選択して、イメージを平坦化します(Modify PanelのFlattenウィンドウでFlattenコマンドを実行します)。
- イメージをTIFFファイルとしてエクスポートします(イメージの上部セクションにあるコマンドを押し、2,048ピクセル解像度に対応するTIFF Export 2xを選択してください)。
3. AFM分析
- タンパク質複合体の体積
- 事前に、異なるサイズの複合体の異なる外観を最大にするために適切な配色を使用して、AFMソフトウェアの画像を直接視覚検査することにより、関連するタンパク質-DNA複合体を選択する( 図2の異なる複合体の異なる色およびサイズを参照;ここでは、SeaLandAndFire AFMソフトウェア)。 XPDサンプルについては、 XPD (〜95kDa)およびp44(〜40kDa)の比較的大きな異なる分子量のために、サイズがはっきりと異なるXPD / p44-DNAならびにXPD-DNAおよびp44-DNAを含む可能性のある複合体が、 。
- 複合型を確認するために、DNA上の個々のタンパク質ピークの量を測定する。これは、異なる画像ソフトウェア(ここでは、AFMソフトウェアのセクションツール)で実現できます。
- セクションウィンドウのカーソルを使用してタンパク質ピークセクションの高さ(h)および直径(d)を測定する。パーティクルセクションのベースに近い直径を測定します。
- エンドウ豆の量(V)を決定する球形のキャップモデルに基づく以下の式を使用して、単純な数学的モデルを使用して、
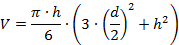
- 測定された体積をおおよそのタンパク質分子量に変換する。
- 分子量が既知のタンパク質の範囲(ここでは、25〜851のデータ点を有する計12回の実験をそれぞれ単量体、二量体の5つの異なるタンパク質で行った)を使用して、体積から分子量(MW)への変換をAFMシステムで較正する。 、三量体、または四量体状態)4,19,20。 (上記の2.2および2.3に記載のように)沈着およびイメージタンパク質を測定し、上記(3.1.2)のようにそれらの量を測定する。既知の分子量を超えて体積をプロットする。得られたグラフは、VとMWとの間の線形関係を示し、その方程式はデータにフィットする線。ここで用いたAFMシステムでは、以下の関係が得られた19 。
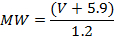
注記:このステップは、各測定の前に繰り返す必要はありませんが、一度だけ行います。体積からMWへの較正は、わずかに変化する直径を有するAFMプローブを用いて同様の画像形成条件下で得られた画像に適用することができる。 - 測定された体積(3.1.2)およびV-to-MW較正(3.1.3.1)に基づいて、分子複合体の情報を提供するタンパク質複合体20の近似MWを決定する。 例えば 、p44-DNA複合体(または単なるDNA上部構造に起因するピーク)、XPDのみのピーク、およびXPD / p44ピークとそれぞれ一致して、XPD / p44試料について〜50kDa、〜100kDaおよび〜140kDaが得られた。
- 分子量が既知のタンパク質の範囲(ここでは、25〜851のデータ点を有する計12回の実験をそれぞれ単量体、二量体の5つの異なるタンパク質で行った)を使用して、体積から分子量(MW)への変換をAFMシステムで較正する。 、三量体、または四量体状態)4,19,20。 (上記の2.2および2.3に記載のように)沈着およびイメージタンパク質を測定し、上記(3.1.2)のようにそれらの量を測定する。既知の分子量を超えて体積をプロットする。得られたグラフは、VとMWとの間の線形関係を示し、その方程式はデータにフィットする線。ここで用いたAFMシステムでは、以下の関係が得られた19 。
- DNA上のタンパク質複合体の位置
- DNAを決定する断片の長さ。
- 適切な画像解析ソフトウェア(例えば、材料表を参照)のフリーハンドライン機能を用いて、AFM画像のDNA断片をトレースし、線の長さを測定する。
注:DNAの集合体と画像の余白で切り取られた断片を除外します。 - 適切なデータ解析およびグラフ作成ソフトウェア( 例えば 、 表の表を参照 )を使用して、ヒストグラム内の実験全体からのDNAの長さをビンしてプロットする。
- データ解析およびグラフ作成ソフトウェアで長さ分布をガウス曲線で近似し、DNA断片の長さを決定します。挿入されたDNA標的部位(1.1参照)の位置を知るために、ガウス曲線の中心から2標準偏差以内の正しい長さのDNA断片のみを含める(y = y 0 + z exp(-2(xx c ) 2 / w 2 )ここで、zはノルム因子であり、x cとwは中心であり、ガウシアンの全半値全幅)を用いて計算される。
注:ガウス曲線の中央のDNA長は、DNA断片の理論上の長さに近くなければなりません(0.34 nm / bpを使用して計算)。理論値よりも最大10%短い長さが一般的であり、おそらくAFM分解能の限界によって引き起こされる。
- 適切な画像解析ソフトウェア(例えば、材料表を参照)のフリーハンドライン機能を用いて、AFM画像のDNA断片をトレースし、線の長さを測定する。
- DNA基質上のタンパク質ピークの位置を決定する。
- 3.2.1.1に記載されているように、より近いDNA断片末端からのタンパク質ピークの距離を測定し、全DNA長で割り、DNA長の断片単位で距離を得る。
- 適切なデータ分析とグラフ作成ソフトウェア( 図3参照)を使用して、測定された距離をヒストグラムにビンしてプロットします。経験則として、n個のデータポイントに対して約√nビンを与えるビンサイズを選択します。
注:ここでDNA末端を区別することはできないので、DNA長0.5の画分にのみプロットする(DNA断片の中心)。 DNA末端結合は研究の焦点ではないので、位置0の少し後に位置データをビンに入れ始めることにより、分析からDNA末端を除外する( 例えば 、ここでは、DNA長の割合0.02からのビニング)。
- タンパク質複合体位置分布から標的部位特異性を決定する。
- 3.2.1.3のようにGaussカーブを使って位置分布に最大値(拘束力の強化)を合わせますが、バックグラウンドバインディングの高さに足を当てます( 図3参照)。
- 非特異的DNAバックグラウンド(非特異的DNA部位に結合したタンパク質分子)に対する特異的部位の特異性Sを以下の式21で計算する
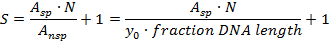
A sp :特定の複合体の数(ガウス曲線の下の面積)
A nsp :非特異的複合体の数(バックグラウンド領域、 すなわち(DNA長の断片)x(平均背景の高さy 0 );ここでカバーされるDNA長の割合は、0.02DNA長で開始するヒストグラムについて0.48である)
N:可能なDNA結合部位の数(ここではDNA末端を除くN = 914)
- DNAを決定する断片の長さ。
- DNAベンド角
- 適切な画像解析ソフトウェアでアングルツールを使用して、DNAバックボーンに沿って中央に配置され、タンパク質ピークを中心とする2本の線の間の角度βを測定する( 図4の挿入図参照 )。統計学的に関連する数のタンパク質-DNA複合体の角度(> 50、理想的には> 100)2,3,5を測定する。
注:DNAベンド角は180°-β2、3,5と定義されています。 - データ分析とグラフ作成ソフトウェアの使用(材料の表を参照)は、DNAの曲げ角度をビニングすることによって曲げ角度分布ヒストグラムを生成する。
- ガウス曲線で曲げ角度分布を合わせます。分布内に複数の最大値が現れる場合は、複数のピークガウスフィットを選択します。ガウス曲線の中心は、特定の種の平均曲げ角度状態を与える。
- 例えば、異なるタンパク質変異体または異なるタンパク質複合体種の分布について、曲がり角度(ガウスフィットの最大値)のシフトが明らかである場合は、スチューデントのt検定を適用して、変更。
- 適切な画像解析ソフトウェアでアングルツールを使用して、DNAバックボーンに沿って中央に配置され、タンパク質ピークを中心とする2本の線の間の角度βを測定する( 図4の挿入図参照 )。統計学的に関連する数のタンパク質-DNA複合体の角度(> 50、理想的には> 100)2,3,5を測定する。
結果
タンパク質複合体の体積に基づいて異なる複合型を区別する
原核生物のNERヘリカーゼUvrBのヘリカーゼ活性は、DNA結合によって刺激される22,23 。 UvrBは、2つの単一のssDNA鎖のうちの1つに正確にロードするために、DNA(DNAバブル)中に不対領域を必要とする。 インビボでは、このDNA構造は、NERカスケー?...
ディスカッション
特定の標的部位を含む長いDNA断片上のタンパク質の結合位置のAFM統計分析は、これらの部位2,3,4,5,6を認識するためにタンパク質が用いる特定の戦略に関する興味深い詳細を明らかにすることができる。得られた位置分布を解釈するために、DNA中の標的の位置を正確に知る必要がある。これは、明確な配列位置に特異的部位を環状プラスミドDNAに導入し、この特異的部位を含むDNAの断片を制限?...
開示事項
著者は何も開示することはない。
謝辞
PUC19N、CPD含有オリゴヌクレオチド、およびp44は、Samuel Wilson、Korbinian HeilおよびThomas Carell、およびGudrun MichelsおよびCaroline Kiskerによりそれぞれ提供された。この研究は、ドイツForschungsgemeinschaft(DFG)FZ82およびTE-671/4からITへの助成金によって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| Molecular Force Probe (MFP) 3D | Asylum Research | N/A | atomic force microscope (AFM) |
| Precision 390 | DELL | N/A | computer |
| ThermoMixer and 1.5 mL block | Eppendorf | 5382000015 | heat block for DNA preparation |
| Rotilabo Block-Heater H 250 & blocks for 0.5 mL tubes | Carl Roth GmbH | Y264.1 & Y267.1 | heat block for protein-DNA incubations |
| Mini-Sub Cell GT | Bio-Rad Laboratories GmbH | 1704467 | electrophoresis chamber with gel caster and power supply |
| Power Pac Basic | Bio-Rad Laboratories GmbH | 1645050 | electrophoresis power supply |
| Centifuge 5415 D with rotor | Eppendorf | 2262120-3 | table centrifuge |
| Ultra-Lum electronic UV transillumonator MEB-15 | Ultralum | 900-1322-02 | UV irradiation table |
| NanoDrop ND-1000 | VWR International / PEQLAB Biotechnologie GmbH | N/A | UV spectrophotometer |
| TKAX-CAD with 0.2 μm capsule filter | Unity Lab Services | N/A | water deionization and filter unit |
| Name | Company | Catalog number | Comments |
| Software | |||
| MFP software on Igor Pro | Asylum Research | N/A | AFM software |
| ImageJ (open source Java image processing) | NIH Image | N/A | Image analysis software |
| Excel (Microsoft Office) | Microsoft Corporation | N/A | data analysis software |
| Origin9 / Origin2016 | OriginLab Corporation | N/A | statistical data analysis and graphing software |
| Name | Company | Catalog number | Comments |
| Material | |||
| OMCL-AC240TS | Olympus | OMCL-AC240TS | AFM cantilevers |
| grade V-5 muscovite | SPI Supplies | 1805 | mica sheets |
| Amicon Ultra 0.5 mL 50k Ultracell | Millipore Ireland Ltd. | UFC505096 | centrifuge filters |
| NucleoSpin Extract II | Macherey-Nagel GmbH | 740 609.250 | Agarose gel extraction kit |
| Rotilabo cellulose paper type 111A | Carl Roth GmbH | AP59.1 | AFM deposition blotting paper |
| Anatop 25 (0.02 μm) | Whatman GmbH | 6809-2102 | syringe filter |
| SSpI, BspQI | New England Biolabs (NEB) | R0132, R0712 | restriction enzymes for DNA substrate preparation |
| XhoI, BglII | R0146, R0144 | restriction enzymes for DNA preparation controls | |
| nicking restriction enzyme Nt.BstNBI | New England Biolabs (NEB) | R0607 | nickase |
| T4 DNA ligase | New England Biolabs (NEB) | M0202S | Ligase |
| Tris, HEPES | Carl Roth GmbH | 4855, 9105 | buffer chemicals |
| NaCl, MgCl2, KCl, MgAcetate | Carl Roth GmbH | 3957, HN03, HN02, P026 | salt chemicals |
| NaAc | Sigma-Aldrich Chemie GmbH | 32318 | salt chemicals |
| DTT, TCEP, EDTA | 6908, HN95, 8040 | chemicals/reagents | |
| agarose, acetic acid, HCl | Carl Roth GmbH | 2267, 3738, K025 | reagents |
| ATP | Carl Roth GmbH | K054 | nucleotides |
| oligonucleotide #1 in Table 1 | Biomers | custom | complementary DNA oligonucleotide |
| oligonucleotides #2, #3, and #6 in Table 1 | Integrated DNA Technologies (IDT) | custom | fluorescein containing oligonucleotides |
| oligonucleotides #4 and #5 in Table 1 | private (available from e.g. TriLink or GlenResearch) | CPD containing oligonucleotides | |
| SafeSeal reaction tube 0.5 mL and 1.5 mL | Sarstedt | 72.704 and 72.706 | incubation tubes |
| GeneRuler 1 kb | Thermo Scientific | SM0311 | DNA ladder |
| 6x concentrate gel loading dye purple | New England Biolabs (NEB) | 51406 | DNA loading dye |
| Midori Green | Nippon Genetics Europe GmbH | 999MG28055 | DNA stain |
参考文献
- Janicijevic, A., Ristic, D., Wyman, C. The molecular machines of DNA repair: scanning force microscopy analysis of their architecture. J. Microsc. 212 (3), 264-272 (2003).
- Wang, H., et al. DNA bending and unbending by MutS govern mismatch recognition and specificity. Proc. Natl. Acad. Sci. USA. 100 (25), 14822-14827 (2003).
- Tessmer, I., et al. Mechanism of MutS searching for DNA mismatches and signaling repair. J. Biol. Chem. 283 (52), 36646-36654 (2008).
- Wagner, K., Moolenaar, G., van Noort, J., Goosen, N. Single-molecule analysis reveals two separate DNA-binding domains in the Escherichia coli UvrA dimer. Nucleic Acids Res. 37 (6), 1962-1972 (2009).
- Buechner, C. N., et al. Strand-specific recognition of DNA damages by XPD provides insights into nucleotide excision repair substrate versatility. J Biol. Chem. 289 (6), 3613-3624 (2014).
- Van der Linden, E., Sanchez, H., Kinoshita, E., Kanaar, R., Wyman, C. RAD50 and NBS1 form a stable complex functional in DNA binding and tethering. Nucleic Acids Res. 37 (5), 1580-1588 (2009).
- Fuentes-Perez, M. E., Dillingham, M., Moreno-Herrero, F. AFM volumetric methods for the characterization of proteins and nucleic acids. Methods. 60, 113-121 (2013).
- Shlyakhtenko, L. S., Lushnikov, A. Y., Miyagi, A., Lyubchenko, Y. L. Specificity of binding of single-stranded DNA-binding protein to its target. Biochemistry. 51, 1500-1509 (2012).
- Wirth, N., et al. Conservation and Divergence in Nucleotide Excision Repair Lesion Recognition. J. Biol. Chem. 291 (36), 18932-18946 (2016).
- Kuper, J., Kisker, C. Damage recognition in nucleotide excision DNA repair. Curr. Opin. Struct. Biol. 22, 88-93 (2012).
- Buechner, C. N., Tessmer, I. DNA substrate preparation for atomic force microscopy studies of protein-DNA interactions. J. Mol. Recognit. 26 (12), 605-617 (2013).
- Sun, Z., Tan, H. Y., Bianco, P. R., Lyubchenko, Y. L. Remodeling of RecG Helicase at the DNA Replication Fork by SSB Protein. Sci. Rep. 5, 9625 (2015).
- Billingsley, D. J., Bonass, W. A., Crampton, N., Kirkham, J., Thomson, N. H. Single-molecule studies of DNA transcription using atomic force microscopy. Phys. Biol. 9 (2), 021001 (2012).
- Maurer, S., Fritz, J., Muskhelishvili, G., Travers, A. RNA polymerase and an activator form discrete subcomplexes in a transcription initiation complex. EMBO J. 25 (16), 3784-3790 (2006).
- Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Theis, K., Chen, P. J., Skorvaga, M., Van Houten, B., Kisker, C. Crystal structure of UvrB, a DNA helicase adapted for nucleotide excision repair. EMBO J. 18 (24), 6899-6907 (1999).
- Kuper, J., et al. In TFIIH, XPD helicase is exclusively devoted to DNA repair. PLoS Biol. 12 (9), e1001954 (2014).
- Shlyakhtenko, L. S., et al. Silatrane-based surface chemistry for immobilization of DNA, protein-DNA complexes and other biological materials. Ultramicroscopy. 97, 279-287 (2003).
- Roth, H. M., et al. XPB helicase regulates DNA incision by the Thermoplasma acidophilum endonuclease Bax1. DNA Repair. 11 (3), 286-293 (2012).
- Ratcliff, G. C., Erie, D. A. A Novel Single-Molecule Study to Determine Protein-Protein Association Constants. J. Am. Chem. Soc. 123 (24), 5632-5635 (2001).
- Yang, Y., Sass, L. E., Du, C., Hsieh, P., Erie, D. A. Determination of protein-DNA binding constants and specificities from statistical analyses of single molecules: MutS-DNA interactions. Nucleic Acids Res. 33 (13), 4322-4334 (2005).
- Caron, P. R., Grossman, L. Involvement of a cryptic ATPase activity of UvrB and its proteolysis product, UvrB* in DNA repair. Nucleic Acids Res. 16 (22), 10891-10902 (1988).
- Wang, H., et al. UvrB domain 4, an autoinhibitory gate for regulation of DNA binding and ATPase activity. J. Biol. Chem. 281 (22), 15227-15237 (2006).
- Chammas, O., Billingsley, D. J., Bonass, W. A., Thomson, N. H. Single-stranded DNA loops as fiducial markers for exploring DNA-protein interactions in single molecule imaging. Methods. 60 (2), 122-130 (2013).
- Truglio, J. J., et al. Structural basis for DNA recognition and processing by UvrB. Nat. Struct. Mol. Biol. 13 (4), 360-364 (2006).
- Kuper, J., Wolski, S. C., Michels, G., Kisker, C. Functional and structural studies of the nucleotide excision repair helicase XPD suggest a polarity for DNA translocation. EMBO J. 31 (2), 494-502 (2012).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Goosen, N. The presence of two UvrB subunits in the UvrAB complex ensures damage detection in both DNA strands. EMBO J. 21 (15), 4196-4205 (2002).
- Verhoeven, E. E., Wyman, C., Moolenaar, G. F., Hoeijmakers, J. H., Goosen, N. Architecture of nucleotide excision repair complexes: DNA is wrapped by UvrB before and after damage recognition. EMBO J. 20 (3), 601-611 (2001).
- Moolenaar, G. F., et al. The Role of ATP Binding and Hydrolysis by UvrB during Nucleotide Excision Repair. J. Biol. Chem. 275, 8044-8050 (2000).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved